È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Una schermata di High Throughput per Biomining attività Cellulase da librerie metagenomiche
In questo articolo
Riepilogo
Questo protocollo descrive uno schermo ad alta velocità per l'attività cellulosolitici da una libreria metagenomica espressa in Escherichia coli. Lo schermo è soluzione basata e altamente automatizzata, ed utilizza una pentola chimica nel 384 micropiastre bene con la lettura finale come una misura di assorbanza.
Abstract
Cellulosa, la fonte più ricca di carbonio organico del pianeta, ha applicazioni industriali di ampio respiro con enfasi crescente sulla produzione di biocarburanti 1. Metodi chimici per modificare o degradare la cellulosa in genere richiedono acidi forti e temperature elevate. Come tale, i metodi enzimatici sono diventati di primo piano nel processo di bioconversione. Mentre l'individuazione delle cellulasi attivo da ceppi di batteri e funghi è stata in qualche modo efficace, la stragrande maggioranza dei microbi in natura resistere coltura di laboratorio. Genomica ambientale, noto anche come approcci metagenomica, lo screening ha una grande promessa per colmare il divario coltivazione nella ricerca di enzimi bioconversione romanzo. Approcci di screening metagenomica hanno recuperato con successo cellulasi romanzo da ambienti diversi, come i terreni 2, bufali rumine 3 e la termite posteriori-gut 4 con carbossimetilcellulosa (CMC) piastre di agar colorate con colorante rosso congo (basato sul metodo del Teatro e Wood 5). Tuttavia, il metodo CMC è limitata in velocità, non è quantitativo e manifesta un basso rapporto segnale rumore 6. Altri metodi sono stati segnalati 7,8 ma entrambi utilizzare un piatto a base di test di agar, che è indesiderabile per high-throughput screening di grandi biblioteche genomiche inserire. Qui vi presentiamo una soluzione a base di schermo per l'attività cellulasi utilizzando un dinitrofenolo cromogenico (DNP)-cellobioside substrato 9. La nostra biblioteca è stato clonato nel controllo pCC1 copia fosmid per aumentare la sensibilità del test attraverso l'induzione copia numero 10. Il metodo utilizza una pentola chimica in 384 pozzetti con la lettura finale fornito come misura dell'assorbanza. Questa lettura è quantitativa, sensibile e automatizzati con un throughput fino a 100X piastre da 384 pozzetti al giorno, utilizzando un gestore di liquidi e lettore di piastre con annesso sistema di impilaggio.
Protocollo
Prima di iniziare questo protocollo, è necessario libreria metagenomica memorizzati in un formato 384 pozzetti. Nel nostro studio, abbiamo usato il pCC1 controllo vettoriale fosmid copia in combinazione con il fago T1-resistente TransforMax EPI300 T1-R E. coli come host biblioteca e conservati i nostri piatti a -80 ° C 11.
1. La replica dei piatti Biblioteca metagenomica
- Scongelare i piatti contenenti la libreria a 37 ° C per circa 20 minuti, o fino a quando tutti i pozzetti siano scongelati.
- Utilizzare la funzione di luce UV di sterilizzazione della qPix2 di sterilizzare il robot per 15 minuti.
- Preparare il brodo LB con cloramfenicolo ad una concentrazione finale di 12.5ug/mL e arabinosio a 100ug/mL in una bottiglia di reagente 500mL. Ogni piatto utilizzerà circa 20 mL, oltre a fare un ulteriore 50mL per consentire volume morto.
- Impostare il qFill3 come istruzioni del produttore, con la bottiglia media associati al collettore attraverso il tubo sterile. Programma che per la quantità appropriata di media, e impostare per riempire un volume 45uL in ciascun pozzetto.
- Spurgare l'aria dal tubo e collettore utilizzando la funzione di eliminazione del robot è visibile fino a quando i media provenienti da ogni pin del collettore.
- Riempire il numero desiderato di piastre con i media LB utilizzando il qFill3. Ogni piatto richiede circa 20 secondi per riempire.
- Caricare le piastre biblioteca e le piastre fresca in apposite aree del robot qPix2. Riempire il bagno di pulizia con i reagenti appropriati, 2% Micro90 nella vasca da bagno posteriore, autoclave acqua distillata nella vasca centrale, l'80% di etanolo nel bagno di fronte.
- Utilizzare il programma "replica" del qPix2. Nel software, selezionare la testa, il numero ed i tipi di piatti di origine e di piastre di destinazione. Inoltre istituito la testa per pulire tra le repliche, di solito 6 cicli di ogni bagno è sufficiente. Capacità della macchina è di 10 piatti alla volta, e ci vogliono circa 15 minuti per replicare tutti con una testa di spillo 384 (o circa 50 minuti con una testa 96 pin).
Nota: Vi è la possibilità di "Stir Fonte" o "Stir di destinazione". Si raccomanda di non utilizzare tali opzioni problemi sono già verificati con il nostro robot li utilizzano. - Una volta che i piatti vengono replicati, crescono le piastre a 37 ° C per 24 ore in una scatola di umidità. Perché siamo inducendo un elevato numero di copie fosmid con aggiunta di arabinosio, i cloni tendono a crescere più lentamente rispetto ai non-indotta cloni. Restituire le piastre biblioteca a -80 ° C.
Nota: L'incubazione in una scatola di umidità garantisce l'evaporazione dei mezzi di comunicazione non si verifica nei pozzi sul bordo delle piastre, mantenendo un ambiente uniforme per tutti i pozzetti. - Pulire il qFill3 robot prima purificazione con l'acqua (~ 50 mL) e poi a 80% di etanolo (~ 50 mL). Smontare i componenti, e pulita e autoclave la bottiglia, coperchio e tubi avvolti in carta stagnola.
2. Misurare la crescita di E. coli cloni
- Rimuovere le piastre da 37 ° C incubatore. Togliere e mettere da parte le palpebre, e le piastre posto sulla piattaforma di carico rivista fornito con il RapidStak, fino ad un massimo di 25 piastre.
Nota: La piastra sul fondo della pila sarà il primo ad essere letto. Assicurati di tenere traccia dell'ordine delle piastre, in quanto il software non registra questo. - Rimuovere una rivista dal Stak rapida, assicurarsi che sia vuoto, e spingerlo in cima alla pila di piatti da leggere. Afferrarlo per la maniglia, e le piastre dovrebbero essere caricati nel magazzino. Caricare la rivista nel RapidStak.
Nota: La rivista ha una piccola vite in un angolo che denota che i pozzi A1 angolo delle piastre dovrebbe andare. La rivista sarà solo montare in RapidStak in un unico orientamento. - Aprire il programma RE SkanIt sul computer collegato alle macchine. Selezionare "New Session" e un nome appropriato. Scegliere "Corning a fondo piatto da 384 pozzetti" come tipo di piatto.
- In "Layout Plate", selezionare il pulsante "Wizard", e scegliere per aggiungere 384 incognite al tuo piatto.
- Nell'area "Protocollo", selezionare l'opzione "Bene Loop" opzione e immettere per 384 pozzetti. Un "Beh Loop" icona apparirà nella flow-albero sul lato sinistro dello schermo. Selezionare l'icona e fare clic per aggiungere "di misura fotometrica". Impostato per leggere a 600nm.
- Salvare il protocollo con un nome univoco, e chiudere il programma SkanIt RE.
- Sul computer, aprire il programma PolaraRS.
- Nella pagina principale ci sarà tabella che elenca gli strumenti collegati al dispositivo RapidStak. Il VarioSkan dovrebbe essere sul lato sinistro di questa tabella. Sotto il titolo VarioSkan, ci saranno due link: "RunSession" e "incubare". Fare clic sull'opzione "RunSession".
- Una nuova finestra, la finestra del test, apparirà con un diagramma di flusso nel mezzo. Selezionare la voce "RunSession" (dovrebbe essere l'unica voce presente). Sul lato destro dello schermo un menu a discesaapparirà. Trova il nome del RE SkanIt salvato eseguito in questo menu.
Nota: Non sembra esserci nessun ordinamento razionale a questo menu. Non è in ordine alfabetico, o per data di aggiunta, o in relazione al punto in cui il computer il protocollo è salvato. Questo significa che si deve guardare attraverso tutti i protocolli prima di trovare il proprio, che è un dolore. - Dopo aver selezionato il protocollo, in alto a destra cliccare su "Esegui questo test".
- Viene visualizzata una finestra si aprirà, che chiede di indicare quale rivista da utilizzare come fonte, e che la rivista è vuoto. La casella in basso sullo schermo rispetto alla rivista anteriore. [Opzionale: Si chiede di inserire il numero di lastre caricate a determinare una stima per la quantità di tempo ci vorrà. Questa stima è normalmente lontano.]
- Garantire sia la RapidStak e VarioSkan sono accesi, e premere il tasto "OK".
- Il RapidStak caricherà automaticamente il vostro piatti nel lettore di piastre, consentendo misurazioni in continuo di lastre. Per leggere 25 piastre è di circa 50 minuti.
Nota: Si raccomanda di osservare il primo caricamento piastra nel VarioSkan per assicurare un corretto allineamento delle macchine. Se il RapidStak non carica il lettore di piastre correttamente, utilizzare la funzione "Pause questo saggio" pulsante nell'angolo in alto a destra della finestra PolaraRS. - Una volta che il VarioSkan è finito di leggere tutti i piatti, rimuovere il caricatore pieno e posizionarlo sul pianale di carico rivista. Sollevare il rettangolo esterno della piattaforma, e la rivista dovrebbe scivolare via, lasciando i piatti in piedi in un mucchio in mezzo. Sostituire i coperchi alle piastre appropriato.
3. Aggiunta del saggio Mix per ogni piatto
- Preparare mix test utilizzando premade magazzino 10x mix di lisi (10% Triton X-100, 100mM RIS, 10mM EDTA) in 50 mM tampone acetato di potassio a pH 5,5. Preparare uno stock di fino a 75mg/mL DNP-cellobioside substrato in DMSO, assicurandosi che il substrato è completamente sciolto. Aggiungi DNP-cellobioside soluzione madre di mescolare test ad una concentrazione finale di 0.1mg/mL. Ogni piatto utilizzerà circa 20 mL di soluzione, oltre a fare un ulteriore 50mL per consentire volume morto.
Nota: DNP-Cellobioside non è facilmente solubile in acqua, in modo da sciogliere in DMSO aumenta la solubilità. La presenza di DMSO nella soluzione di saggio finale non ha effetti osservabili. - Impostare il qFill3 come istruzioni del produttore, con la bottiglia media associati al collettore attraverso il tubo sterile. Programma che per la quantità appropriata di media, e impostare per riempire un volume 45uL in ciascun pozzetto.
- Spurgare l'aria dal tubo e collettore utilizzando la funzione di eliminazione del robot è visibile fino a quando i media provenienti da ogni pin del collettore.
- Aggiungi mix di test a ogni piatto con il qFill3. Ogni piatto richiede circa 20 secondi per riempire.
- Una volta mix saggio è aggiunto, incubare le piastre a 37 ° C in una scatola di umidità per 12-16 ore.
Nota: Over-incubazione delle piastre quando l'impasto saggio è aggiunto provoca l'evaporazione nei pozzetti esterni della piastra, ottenendo valori di assorbanza superiore a quello visto per i pozzi all'interno. Questo può essere compensato da incubando le piastre in una camera umida.
4. Assorbanza lettura dei cloni dosato
- Rimuovere le piastre da 37 ° C incubatore. Togliere e mettere da parte le palpebre, e le piastre posto sulla piattaforma di carico rivista fornito con il RapidStak, fino ad un massimo di 25 piastre.
Nota: La piastra sul fondo della pila sarà il primo ad essere letto. Assicurati di tenere traccia dell'ordine delle piastre, in quanto il software non registra questo. - Rimuovere una rivista dal Stak rapida, assicurarsi che sia vuoto, e spingerlo in cima alla pila di piatti da leggere. Afferrarlo per la maniglia, e le piastre dovrebbero essere caricati nel magazzino. Caricare la rivista nel RapidStak.
Nota: La rivista ha una piccola vite in un angolo che denota che i pozzi A1 angolo delle piastre dovrebbe andare. La rivista sarà solo montare in RapidStak in un unico orientamento. - Aprire il programma RE SkanIt sul computer collegato alle macchine. Selezionare "New Session" e un nome appropriato. Scegliere "Corning a fondo piatto da 384 pozzetti" come tipo di piatto.
- In "Layout Plate", selezionare il pulsante "Wizard", e scegliere per aggiungere 384 incognite al tuo piatto.
- Nell'area "Protocollo", selezionare l'opzione "Bene Loop" opzione e immettere per 384 pozzetti. Un "Beh Loop" icona apparirà nella flow-albero sul lato sinistro dello schermo. Selezionare l'icona e fare clic per aggiungere "di misura fotometrica". Impostarlo per leggere a 400nm.
- Salvare il protocollo con un nome univoco, e chiudere il programma SkanIt RE.
- Sul computer, aprire il programma PolaraRS.
- Nella pagina principale ci sarà tabella che elenca gli strumenti collegati al dispositivo RapidStak. Il VarioSkan dovrebbe essere sul lato sinistro di questa tabella. Sotto il titolo VarioSkan, ci saranno due link: "RunSession" e "Incubate ". Fare clic su" "opzione RunSession.
- Una nuova finestra, la finestra del test, apparirà con un diagramma di flusso nel mezzo. Selezionare la voce "RunSession" (dovrebbe essere l'unica voce presente). Sul lato destro dello schermo un menu a discesa appare. Trova il nome del RE SkanIt salvato eseguito in questo menu.
- Dopo aver selezionato il protocollo, in alto a destra cliccare su "Esegui questo test".
- Viene visualizzata una finestra si aprirà, che chiede di indicare quale rivista da utilizzare come fonte, e che la rivista è vuoto. La casella in basso sullo schermo rispetto alla rivista anteriore. [Opzionale: Si chiede di inserire il numero di lastre caricate a determinare una stima per la quantità di tempo ci vorrà. Questa stima è normalmente lontano.]
- Garantire sia la RapidStak e VarioSkan sono accesi, e premere "OK"
- Il RapidStak caricherà automaticamente il vostro piatti nel lettore di piastre, consentendo misurazioni in continuo di lastre.
Nota: Si raccomanda di osservare il primo caricamento piastra nel VarioSkan per assicurare un corretto allineamento delle macchine. Se il RapidStak non carica il lettore di piastre correttamente, utilizzare la funzione "Pause questo saggio" pulsante nell'angolo in alto a destra della finestra PolaraRS. - Una volta che il VarioSkan è finito di leggere tutti i piatti, rimuovere il caricatore pieno e posizionarlo sul pianale di carico rivista. Sollevare il rettangolo esterno della piattaforma, e la rivista dovrebbe scivolare via, lasciando i piatti in piedi in un mucchio in mezzo. Le lastre possono essere smaltiti in base alle proprie procedure di laboratorio.
- Per esportare le letture di assorbanza, di software open RE SkanIt, e scegliere "Apri un file esistente".
- Selezionare la directory dove avete salvato la sessione, ha colpito il simbolo "+" per espandere l'elenco e sotto la voce titolo verrà opzioni etichettato come "contenitore 1" a "Container N" a seconda del numero di piatti. Selezionare la prima piastra da analizzare.
- Nella barra dei menu nella parte superiore della pagina, selezionare "Elaborazione Dati> Rapporto / Export"
- Scegliere le opzioni desiderate per l'esportazione.
Nota: Io di solito selezionare "Layout piastra" e "fotometrici" dati per l'esportazione. - Fare clic su "Visualizza rapporto" e poi "File> Salva". Scegliere la directory desiderata. Il formato di output è un foglio di calcolo Microsoft Excel
5. Rappresentante Risultati
Un esempio di valori di assorbanza da una singola 384 pozzetti contenenti un clone positivo è mostrato nella Figura 2. Cloni positivi mostrano un marcato aumento di assorbanza più di quelli che non esprimono l'attività cellulasi. Differenze nel tempo di analisi, l'ubicazione e sulla piastra, o concentrazione DNP (può essere introdotto filtrando un-dissolto DNP) potrebbero influenzare le letture assolute. Letture dell'assorbanza relativa, come la differenza di assorbanza al di sopra della media piastra o media colonna, sono un metodo più affidabile per identificare cloni cellulasi positivo.
A seguito di identificazione di cloni positivi dalle piastre biblioteca, si consiglia di replicare tutti i cloni positivi in una nuova piastra per lo screening secondario. Questo elimina gli effetti derivanti dalla posizione bene o variazione targa e consente una maggiore confronto diretto tra i cloni positivi.

Figura 1. Diagramma di flusso del test high-throughput per una libreria metagenomica clonato in E. coli e conservati a -80 ° C.
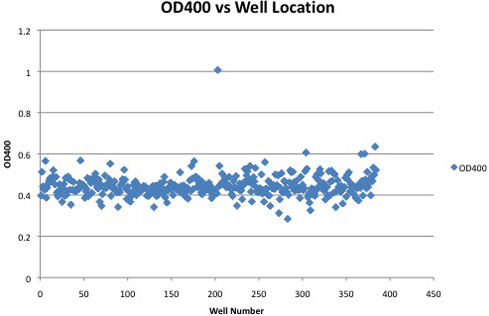
Figura 2. Letture di assorbanza da una piastra da 384 pozzetti contenenti un clone positivo. Cellulasi cloni positivi possono essere identificati mediante assorbimento significativamente aumentato nel corso cloni negativo.
Discussione
Uno schermo ad alto rendimento per la rapida individuazione di attività cellulosolitici da una grande libreria genomica metagenomica di DNA espressa in E. coli è descritto in questo protocollo. Questo metodo è un miglioramento rispetto al CMC / Congo saggio Rosso comunemente utilizzati in letteratura. Si tratta di una soluzione basata, e consente di one-pot di screening chimico a 384 pozzetti, con il risultato finale come le letture di assorbanza da un lettore di piastre consentendo analisi quantitativa. L&#...
Divulgazioni
Riconoscimenti
Gli autori desiderano ringraziare il Dott. Steve Withers e Hong-Ming Chen per la fornitura di DNP-Cellobioside substrato.
Materiali
| Name | Company | Catalog Number | Comments |
| qPix2 | Genetix | With 384-pin gridding head | |
| qFill3 | Genetix | With 384-well manifold | |
| Varioskan | Thermo Fisher Scientific, Inc. | ||
| RapidStak | Thermo Fisher Scientific, Inc. | Connected to Varioskan | |
| Micro90 Detergent | Cole-Parmer | 18100-00 | Diluted to 2% in water |
| Ethanol | Major Lab Supplier | Diluted to 80% in water | |
| Chloramphenicol | Sigma-Aldrich | C0378 | 12.5mg/mL in ethanol |
| LB broth, Miller | Fisher Scientific | BP1426-2 | 25g/L, autoclaved |
| 384-well flat bottom plates | Corning | 3680 | |
| L-(+)-Arabinose | Sigma-Aldrich | A3256 | 100mg/mL in water |
| Potassium Acetate | Fisher Scientific | P171 | 50mM in water, autoclaved, adjusted to pH 5.5 with HCl |
| Triton X-100 | Fisher Scientific | BP151 | |
| Trizma hydrochloride | Sigma-Aldrich | T3253 | In TE buffer solution, 100mM |
| EDTA disodium salt | Sigma-Aldrich | E5134 | In TE buffer solution, 10mM |
| 2,4-dinitrophenyl cellobioside | Provided by Dr. Steve Withers, UBC | ||
| Dimethyl Sulfoxide | Sigma-Aldrich | D8418 |
Riferimenti
- Rubin, E. M. Genomics of cellulosic biofuels. Nature. 454, 841-845 (2008).
- Voget, S., Steele, H. L., Streit, W. R. Characterization of a metagenome derived halotolerant cellulase. J. Biotechnol. 126, 26-36 (2006).
- Duan, C. J. Isolation and partial characterization of novel genes encoding acidic cellulases from metagenomes of buffalo rumens. J. Appl. Microbiol. 107, 245-256 (2009).
- Zhang, Y. H. P. Outlook for cellulase improvement: Screening and selection strategies. Biotech. Advances. 24, 452-481 (2006).
- Teather, R. M., Wood, P. J. Use of congo red-polysaccharide interactions in enumeration and characterization of cellulolytic bacteria from the bovine rumen. Appl. And Environ. Microbiol. 43, 777-780 (1982).
- Sharrock, K. R. Cellulase assay methods: a review. J. Biochem and Biophys Methods. 17, 81-106 (1988).
- Kasana, R. C., Salwan, R., Dhar, H., Dutt, S., Gulati, A. A rapid and easy method for detection of microbial cellulases on agar plates using Gram's Iodine. Curr. Microbiology. 57, 503-507 (2008).
- Huang, J. S., Tang, J. Sensitive assay for cellulase and dextranase. Anal. Biochem. 73, 369-377 (1976).
- Tull, D., Withers, S. G. Mechanisms of cellulases and xylanases: A detailed kinetic study of the exo-beta-1,4-glycanase from Cellulomonas fimi. Biochemistry. 33, 6363-6370 (1994).
- Martinez, A., Bradley, A. S., Waldbauer, J. R., Summons, R. E., DeLong, E. F. Proteorhodopsin photosystem gene expression enables photophosphorylation in a heterologous host. PNAS. 104, 5590-5595 (2007).
- Taupp, M., Lee, S., Hawley, A., Yang, J., Hallam, S. J. Large Insert Environmental Genomic Library Production. J Vis Exp. , (2009).
- Martinez, A., Tyson, G. W., DeLong, E. F. Widespread known and novel phosphonate utilization pathways in marine bacteria revealed by functional screening and metagenomic analyses. Environ Microbiol. 12, 222-238 (2010).
- Wild, J., Hradecna, Z., Szybalski, W. Conditionally amplifiable BACs: Switching from single-copy to high-copy vectors and genomic clones. Genome Research. 12, 1434-1444 (2002).
- Johnson, E. A. Sacchirification of complex cellulosic substrates by the cellulase system from Clostridium thermocellum. Appl Environ Microbiology. 43, 1125-1132 (1982).
- Stutzenberger, F. J. Cellulase Production by Thermomonospora curvata isolated from municipal solid waste compost. Appl Environ Microbiol. 22, 147-152 (1971).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon