Method Article
Cromosoma Trappola associate per identificare interazioni a lungo raggio del DNA
In questo articolo
Riepilogo
La trappola dei cromosomi associati (ACT) saggio è un nuovo metodo imparziale per identificare interazioni a lungo raggio del DNA. La caratterizzazione delle interazioni a lungo raggio del DNA ci permette di determinare la relazione di architettura nucleare per l'espressione genica sia in fisiologia normale e in stati malati.
Abstract
Informazione genetica codificata dal DNA è organizzato in una struttura complessa e altamente regolamentato cromatina. Ogni cromosoma occupa un territorio specifico, che può cambiare a seconda dello stadio di sviluppo o del ciclo cellulare. L'espressione genica può avvenire in impianti specializzati dove trascrizionale segmenti cromatina può ciclo fuori dai territori cromosoma varie, portando a co-localizzazione di segmenti di DNA che possono esistere su cromosomi diversi o distanti sullo stesso cromosoma. The Associated Trappola cromosoma (ACT) test fornisce una metodologia efficace per identificare questi a lungo raggio associazioni del DNA in modo distorti da estendere e modificare la conformazione tecnica del cromosoma cattura. Il test ACT rende possibile per noi di indagare i meccanismi di regolazione trascrizionale in trans, e può aiutare a spiegare il rapporto tra architettura nucleare per l'espressione genica nella fisiologia normale e durante stati di malattia.
Protocollo
1. Fissazione di formaldeide di interazioni a lungo raggio cromatina
- Linea cellulare cultura umana HL-60 in RPMI1640 media con FBS 15% e 1 x penicillina / streptomicina al 80-90% confluenza in un incubatore in dotazione con il 5% CO 2 a 37 ° C.
- Raccogliere le cellule in una provetta da 50 ml Nunc, centrifugare a 1200 rpm per 15 minuti, quindi rimuovere il supporto per aspirazione
- Aggiungere 5 ml di terreno di coltura per risospendere il pellet cellulare, e contare le cellule utilizzando un emocitometro. Richiede circa 1 x 10 7 cellule ad un volume di 40 ml con RPMI1640 / 10% FBS, quindi aggiungere 1,7 ml di formaldeide al 37% per fissare la cromatina diluito.
- Incubare a temperatura ambiente per 10 minuti agitando delicatamente, e poi spegnere con 2,4 ml di 2M glicina. Centrifugare per 15 minuti a 1200 rpm a 4 ° C, e rimuovere il surnatante. Lavare il pellet una volta con 40 ml di PBS freddo ghiaccio, poi rallenta il precipitato ed eliminare la PBS.
2. Lisi cellulare per isolare nuclei
- Risospendere le cellule in 40 ml di tampone di lisi freddo ghiaccio (10 mM Tris-HCl, pH8.0, 10 mM NaCl, 0,2% NP-40) con inibitori della proteasi appena aggiunto (diluizione 1:500) e 0,1 mM PMSF. Incubare in una stanza fredda con rotazione per 90 minuti, centrifugare a 2.500 giri per 15 minuti, e rimuovere il surnatante.
3. Digestione con enzimi di restrizione Bgl II
- Risospendere i nuclei in 0,5 ml di 1 x ONA tampone 3, e aggiungere 15 μlof 10% SDS. Incubare a 37 ° C per 1 ora con agitazione, poi aggiungere 45 ml di 20% Triton X-100 di segregare l'SDS. Incubare a 37 ° C per 1 ora con agitazione.
- Utilizzare un aliquota di 1 x 10 6 nuclei (circa 15 mg, un decimo delle cellule originali) per enzimi di restrizione. Estrarre 55 ml di soluzione di nuclei a partire dal punto 3.1 e portare a 500 microlitri con 433 ml di buffer di 1 x NEB 3 e 12 ml di Bgl II (50U/ul). Incubare a 37 ° C durante la notte.
4. Legatura di segmenti di DNA che interagiscono
- Inattivare l'enzima di restrizione con l'aggiunta di 95 ml di 10% SDS, e denaturare per riscaldamento a 65 ° C per 20 minuti in un bagno d'acqua.
- Aggiungere 7 ml di buffer di 1 legatura x (30 mM Tris-HCl, pH 8,0, 10 mM MgCl 2, 10 mM DTT, 1 mM ATP) e 360 microlitri del 20% Triton X-100 e incubare a 37 ° C per 1 ora .
- Abbassare la temperatura a 16 ° C e aggiungere 50 ml di 400 U / mL DNA ligasi T4. Incubare il campione a 16 ° C per 4 ore e poi a temperatura ambiente per 30 minuti.
5. Purificazione del DNA
- Aggiungere 300 mg di proteinasi K, e incubare a 65 ° C durante la notte.
- Aggiungere 5 mg di RNasi A e incubare a 37 ° C per 30 minuti
- Purificare il DNA per estrazione fenolo / cloroformio, e il DNA precipitato in isopropanolo. Sciogliere il DNA in 150 ml di acqua distillata sterile.
6. Digestione con Msp I e legatura con linker oligonucleotide
- Incubare 2μg di DNA purificato con 5 unità di Msp io a 37 ° C per 4-6 ore. Inattivare Msp io a 65 ° C per 10 minuti, poi precipitare il DNA in etanolo con 1 ml di 5 mg / ml di glicogeno. Sciogliere il pellet di DNA in 50 ml di acqua distillata sterile.
- Mescolare 50 ml di Msp I trattati di DNA con 2 ml di uno L 20 oligonucleotide mM linker (5'-gctgaccctgaattcgcacgtgcctgtcgttagcggacacagggcgattcac-3 '), 1 l di un oligonucleotide 20 micron S (5'-cggtgaatc-3'), 1 ml di acqua distillata sterile e 6 ml di buffer di 10 T4 DNA ligasi x. Coprire il composto con cera liquida.
- Oligonucleotidi denaturare a 50 ° C per 1 minuto e lasciar raffreddare gradualmente a 10 ° C a 0,5 A ° C / minuto gradiente in un termociclatore.
- Aggiungere 1 ml di 400 U / mL DNA ligasi T4 e incubare a 15 ° C durante la notte. Purificare il linker-legatura del DNA utilizzando un kit di purificazione QIAquick PCR, ed eluire in 50 ml di acqua distillata sterile.
7. PCR amplificazione e analisi di sequenza
- Scegli un fondo per la regione specifica di DNA che si vuole esaminare per interazioni a lungo raggio (cioè, il 'esca'). In questo esempio, utilizziamo ABL-1.
- Prendere 1 ml di DNA purificato per eseguire il primo round di PCR utilizzando 1 ml di 20 mM ABL -1 primer specifico # 4656 (5'-gttcaagcgattctcctgcctcga-3 '), 1 ml di 20 mM linker primer specifico # 2963 (5'- gctgaccctgaattcgcacgtgcct-3 '), 3μl di 3 x Klen Taq polimerasi del DNA I cocktail e 3 ml di acqua distillata sterile. Due ml di 32 P-dCTP è aggiunta a 100 ml di 3 x Klen DNA polimerasi Taq I cocktail prima PCR. Il ciclo termico per un inizio PCR caldo è 72 ° C per 2 minuti, 95 ° Cfor 2 minuti, 18 cicli di 95 ° C per 20 secondi, 65 ° C per 40 secondi e 72 ° C per 1 minuto, per l'ultima proroga viene eseguita a 72 ° Cper 5 minuti.
- Purificare il primo turno prodotti PCR utilizzando un kit di purificazione QIAquick PCR e DNA eluire in 30 ml di acqua distillata sterile.
- Prendere 1 ml di 100 x diluito primo prodotto della PCR per effettuare un secondo ciclo di PCR con primer nested con l'aggiunta di 1 ml di 20 mM ABL-1 primer specifico # 4626 (5'-ggagaatttttatctgcctctgtga-3 ') (Figura 1a), 1 ml di 20 mM di linker primer specifico # 2961 (5'-gtcgttagcggacacagggcgattc-3 '), 3 ml di 3 x Klen Taq polimerasi del DNA I cocktail e 3 ml di acqua distillata sterile. Il programma cicli termici è di 25 cicli di 95 ° C per 20 secondi, 67 ° C per 40 secondi e 72 ° C per 1 minuto, seguito da un'estensione a 72 ° C per 5 minuti.
- Visualizza i prodotti PCR eseguendo un 5% di urea-PAGE gel e la scansione dello schermo esposti in un PhosphoImager (Figura 1b). Ogni banda PCR possono essere riciclati dal gel, sciogliendo le strisce di gel in una provetta Eppendorf contenente 60 ml di acqua distillata sterile, e incubazione a 95 ° C per 5 minuti. Centrifugare brevemente a 10.000 giri per 10 secondi per raccogliere tutti i campioni. Togliere 1 ml da utilizzare come modello di DNA per eseguire PCR con la coppia di primer 2961/4626 utilizzando le stesse condizioni sopra descritte. Analisi della sequenza può essere eseguita dopo la depurazione utilizzando un kit di purificazione QIAquick PCR.
- Sequenze di DNA vengono analizzati utilizzando un tool online per determinare la loro posizione cromosomica sul sito web: http://genome.ucsc.edu (Figura 1c). Fare clic su 'BLAT' per inserire una nuova finestra che permette di incollare una sequenza di DNA, una nuova finestra compare dopo aver cliccato su 'submit', e mostra la 'Risultati della ricerca BLAT'. Fare clic su 'browser' del colpo con il 100% di identità per entrare in una finestra accanto per mostrare la posizione della sequenza di DNA.
8. Rappresentante Risultati
1. ACT test con ABL-1 regione come esca per determinare le sue interazioni a lungo raggio del DNA
Come illustrato nella figura 1a, due Bgl siti II e una BamH io sito sono stati scelti per il test ACT. Nel secondo turno della PCR, set di primer 4626/2961 è stato utilizzato per amplificare ABL-M1, 4630/2961 è stato utilizzato per ABL-M2, e 4636/2961 è stato utilizzato per ABL-M3. Un modello tipico mostra un gel a diverse band (Figura 1b). Ogni frammento di un test ACT si compone di due segmenti di DNA combinati: un segmento deriva dalla regione del DNA esca, mentre il secondo segmento deriva dal partner associato, che è stato unito al segmento regione esca dalla prima sequenza di riconoscimento degli enzimi di restrizione. La seconda sequenza di riconoscimento degli enzimi apparirà alla fine della sequenza partner associati (Figura 1c). Il clonato ABL-M1 frammento contiene il DNA dalla regione di ABL1 situato sul cromosoma 9q32.4 da +133,592,306-133,592,399, e il partner associati si trova al cromosoma 3p13 da +71,869,882-71,870,107. Le identità dei partner associati sono scoperti da sabbiatura loro sequenze utilizzando il browser UCSC genoma (GRCh7/hg19 rilasciato nel febbraio 2009). PROK2 è stato identificato come il partner associati a livello del locus chr3p13.
Analogamente, il ABL-M2 partner associato, è stato localizzato a chr5q21.1, mentre il clone ABL-M3 è stato identificato come un intra-cromosomica associazione nei pressi della ABL-1 locus.
2. Determinazione meno diffuso interazioni a lungo raggio tramite test ACT
Altri test sulla base di 3C hanno riportato molti partner di interagire più che abbiamo mostrato in figura 1 e al IGF2 / H19 locus 12. La metodologia che abbiamo delineato selezionerà le interazioni più frequenti lungo raggio. Tuttavia, aumentando il numero di cicli di PCR, è possibile individuare ulteriori, associazioni meno frequenti e (Figura 2).
3. Le differenze nelle interazioni a lungo raggio nelle cellule tumorali rispetto ai tessuti normali.
Il test ACT può essere utilizzato anche per identificare le differenze di architettura nucleare e di interazioni a lungo raggio tra cellule normali e cellule tumorali (Figura 3). Queste differenze possono riflettere i cambiamenti architettura nucleare che si verificano durante la trasformazione delle cellule e, quindi, in ultima analisi, questo test può essere applicabili per scopi diagnostici. I modelli gel simili che si verificano in tessuti normali e tumorali indicano che il test ACT è affidabile e ripetibile. Mentre il test ACT in grado di identificare i partner a lungo raggio del DNA, ulteriori analisi, come il pesce e saggi ChIP, sono tenuti a verificare la presenza delle associazioni individuate fra loci distanti. Studi genetici, fisiologici e biochimici può essere eseguita per chiarire le implicazioni biologiche di queste associazioni di DNA lungo raggio.
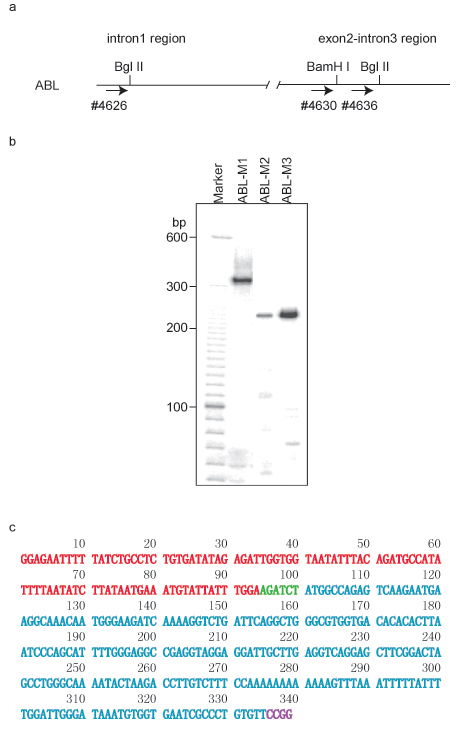
Figura 1 test ACT con ABL-1 regione del DNA esca in cellule HL-60. a. Struttura del DNAURE nella ABL-1 regione. L'enzima di restrizione la prima volta nel ACT test è stato Bgl II. I primer per il secondo turno di PCR sono anche etichettati da frecce e numeri fondo corrispondenti. b. Modello Gel del test ACT 5% di urea-PAGE. Primer coppia 4626/2961 è stato utilizzato per amplificare clone ABL-M1 nel nested PCR, 4630/2961 è stato utilizzato per clonare ABL-M2, e 4636/2961 è stato utilizzato per clonare ABL-M3. c. Sequenza del DNA del clone ABL-M1. Frammento di DNA da ABL-1 DNA esca è marcato in rosso, e il partner del DNA associate è etichettato in ciano. Bgl II sito (AGACTC) è stato etichettato in verde, e io Msp sito (CCGG) è etichettato in viola.

Figura 2 cicli di PCR influenzare i risultati del test ACT. Utilizzando la regione di controllo dell'imprinting (ICR) a livello del locus Igf2/H19 come esca DNA in cellule di fibroblasti di topo, diversi programmi ciclistici sono stati applicati nei turni primo e secondo della PCR nel test ACT. 18-20 cicli al primo turno della PCR non amplificare il segnale abbastanza per essere visualizzati. Venticinque cicli nei risultati secondo PCR in bande chiare, mentre aumenta il numero di cicli per secondo turno della PCR indotto un modello di striscio, oltre a più bande.

Figura 3 Rilevamento delle differenze di architettura nucleare tra il tessuto normale del colon e il cancro del colon in tessuto test ACT. A. Struttura del DNA della regione ICR a IGF2/H19 locus genico e IGF2. DPN siti II per test ACT sono etichettati. Primer utilizzati nel secondo turno di PCR sono etichettati da frecce e numeri in diversi colori che corrispondono a ciascuna corsia in b pannello della figura. b. Modello Gel del test ACT 5% di urea-PAGE. Tessuto del colon normale MAD03-1423 e il cancro dei tessuti del colon MAD04-149 è stato ottenuto da Cooperativa Network tessuto umano (CHTN) Western Division. Dopo ogni tessuto del colon è stato omogeneizzato, test ACT è stata effettuata seguendo le procedure descritte nel presente documento. Corsia 1 rappresenta i risultati PCR utilizzando coppia di primer 2961and # # 4161 (5'-tctgcgccatcagggcagtgagac-3 ') etichettati in rosa nel pannello a; corsia 2 rappresenta i risultati PCR utilizzando coppia di primer # 2961 e # 4163 (5'-gccgcgcggccacttccgattcc-3 ') etichettati in arancione in un pannello, corsia 3 rappresenta il risultato PCR utilizzando coppia di primer # 2961 e # 5145 (5'-gccatgcaggtaggatttgagctg-3') etichettati in blu nel pannello a; corsia 4 rappresenta i risultati PCR utilizzando coppia di primer # 2961 e # 5151 (5'-gtctcaaataggggccagctagcttgg-3 ') etichettati in verde nel pannello a. Gruppi unici che appaiono solo nel tessuto del colon normale sono etichettati da frecce gialle, e quelle band che è apparso solo nel tessuto del cancro del colon sono etichettati in frecce rosse. ICR, regione di controllo imprinting, DMR, diversa regione metilato.
Discussione
Dekker et al. Sviluppato la conformazione cromosoma cattura (3C) approccio per rilevare la frequenza di interazione tra due loci genomici 1, e 3C è stato ampiamente utilizzato per studiare le associazioni intra-e inter-cromosomiche cromosomiche tra le due regioni del DNA in cellule di mammifero conosciuto 2 -9. Anche se di recente sviluppo Hi-C metodologia può essere applicata per lo studio del genoma del DNA associazione, test ACT è ancora una tecnica efficace per lo studio del locus-specifiche interazioni DNA 10-11. Abbiamo modificato questo approccio per identificare le regioni sconosciute del DNA che sono associati a una regione del DNA conosciuto nel topo e cellule umane in coltura (Figura 1). Abbiamo chiamato questo metodo la trappola dei cromosomi associati (ACT) saggio, come ci ha fornito un metodo affidabile e riproducibile per identificare nuovi partner DNA sconosciuto che associano con una regione del DNA bersaglio nota 12. Un test di successo 3C con controlli sia eseguito correttamente prima di eseguire il romanzo aspetti del test ACT 13. Per tofind come molte regioni del DNA associate possibile, è necessario utilizzare diverse combinazioni di enzimi di restrizione prima e seconda. E 'particolarmente importante utilizzare gli enzimi di restrizione che sono insensibili alla metilazione CpG di effettuare il primo passo legatura 3C. Legame con le proteine e la metilazione del DNA può anche influenzare l'efficienza degli enzimi di restrizione digestione e possono portare al fallimento della legatura delle regioni del DNA associate per digestioni alcuni enzimi di restrizione. Il verificarsi di legatura intra-o inter-cromosomiche dipende dalla proteina-DNA cross-linking e appropriato mappe fisiche di entrambe le regioni di DNA associate. Così, alcuni esperimenti preliminari sono essenziali per stabilire una concentrazione di formaldeide efficace e praticabile tempo di trattamento nel test ACT. Una gamma di concentrazioni finali di formaldeide (from1.5% al 2%) sono stati utilizzati in molti laboratori durante la fase 3C del dosaggio 7,9. In alternativa, gli oligonucleotidi usati come linker può essere progettato per abbinare la fine coesa tagliato dall'enzima di restrizione secondo. Anche se abbiamo scoperto che i cicli di 18-20 nel primo turno della PCR e 20-25 cicli al secondo turno della PCR potrebbe fornire bande chiare, è necessario stabilire le migliori condizioni di PCR per ogni esperimento (Figura 2). Intra-molecola ricottura tra 5'-end e 3'-end adattatore sequenza complementare di un filamento di DNA si può verificare in PCR, inibisce l'adattatore specifico fondo di ricottura con la molecola di DNA, e aumentare l'efficienza dell'amplificazione molto più bassa nei primi cicli diversi. Dopo che il DNA bersaglio è stato amplificato per i cicli, il suo importo può essere molto più grande di queste reazioni non specifiche, e può facilitare la concorrenza di primer ricottura di molecole di DNA bersaglio. È anche per questo possiamo vedere l'amplificazione di fondo, e perché il primo prodotto della PCR turno deve essere diluito per diminuire amplification.It sfondo è importante rimuovere i primer in eccesso dalla reazione di PCR al fine di diminuire il fondo nel secondo turno della PCR. Come in tutti gli esperimenti di PCR-based, è di vitale importanza per la progettazione primer che non si trovano in regioni sequenza di ripetizione, che costituiscono la maggior parte del DNA umano e del mouse. Anche se di recente sviluppo Hi-C metodologia può essere applicata per lo studio del genoma del DNA associazione, test ACT è ancora una tecnica efficace per lo studio del locus-specifici di interazione DNA.
Divulgazioni
Riconoscimenti
Ringraziamo Adelle Murell e Wolf Reik molto per condividere il loro protocollo 3C. Questo lavoro è stato sostenuto dal Dipartimento della Difesa e il Servizio di Ricerca del Department of Veterans Affairs.
Materiali
| Name | Company | Catalog Number | Comments |
| RPMI1640 medium | Invitrogen | 22400-105 | |
| acrylamide | Invitrogen | 15512-023 | |
| ATP solution, 10mM | Invitrogen | AM8110G | |
| fetal bovine serum | Invitrogen | 16000-044 | |
| penicillin-streptomycin | Invitrogen | 15140-122 | |
| 1M Tris pH8.0 | Invitrogen | AM9856 | |
| RNase A | Invitrogen | 12091-039 | |
| SDS | Invitrogen | 15525-017 | |
| urea | Invitrogen | 15505-035 | |
| BamH I | New England Biolabs | R0136T | |
| Bgl II | New England Biolabs | R0144M | |
| Dpn II | New England Biolabs | R0543T | |
| Msp I | New England Biolabs | R0106S | |
| dNTPs | New England Biolabs | N0447L | |
| proteinase K | New England Biolabs | P8102S | |
| T4 DNA ligase | New England Biolabs | M0202T | |
| 37% formaldehyde | Sigma-Aldrich | F8775 | |
| Bis-acrylamide | Sigma-Aldrich | 146072 | |
| dithiothreitol | Sigma-Aldrich | 43815 | |
| glycine | Sigma-Aldrich | 50046 | |
| PMSF | Sigma-Aldrich | 93482 | |
| proteinase inhibitor | Sigma-Aldrich | S8830 | |
| Nonidet P-40 | Roche Group | 11754599001 | |
| KlenTaq1 | Ab peptides | 1001 | |
| dCTP alpha P32 | PerkinElmer, Inc. | BLU513H250UC | |
| PTC-100 Thermal Cycler | MJ Research | mjptc100 | |
| Power Supply | Bio-Rad | 164-5056 | |
| OmniPAGE Maxi | Aurogene Life Science | VS20D | |
| Typhoon 9400 | GE Healthcare | 63-0055-78 |
Riferimenti
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing chromosome conformation. Science. 295, 1306-1311 (2002).
- Apostolou, E., Thanos, D. Virus Infection Induces NF-kappaB-dependent interchromosomal associations mediating monoallelic IFN-beta gene expression. Cell. 134, 85-96 (2008).
- Duan, Z. A three-dimensional model of the yeast genome. Nature. 465, 363-367 (2010).
- Lomvardas, S. Interchromosomal interactions and olfactory receptor choice. Cell. 126, 403-413 (2006).
- Murrell, A., Heeson, S., Reik, W. Interaction between differentially methylated regions partitions the imprinted genes Igf2 and H19 into parent-specific chromatin loops. Nat Genet. 36, 889-893 (2004).
- Schoenfelder, S. Preferential associations between co-regulated genes reveal a transcriptional interactome in erythroid cells. Nat Genet. 42, 53-61 (2010).
- Spilianakis, C. G., Lalioti, M. D., Town, T., Lee, G. R., Flavell, R. A. Interchromosomal associations between alternatively expressed loci. Nature. 435, 637-645 (2005).
- Vakoc, C. R. Proximity among distant regulatory elements at the beta-globin locus requires GATA-1 and FOG-1. Mol Cell. 17, 453-462 (2005).
- Xu, N., Tsai, C. L., Lee, J. T. Transient homologous chromosome pairing marks the onset of X inactivation. Science. 311, 1149-1152 (2006).
- Lieberman-Aiden, E. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326, 289-293 (2009).
- Berkum, N. L. v. a. n. Hi-C: a method to study the three-dimensional architecture of genomes. J Vis Exp. , (2010).
- Ling, J. Q. CTCF mediates interchromosomal colocalization between Igf2/H19 and Wsb1/Nf1. Science. 312, 269-272 (2006).
- Dekker, J. The three 'C's of chromosome conformation capture: controls, controls, controls. Nat Methods. 3, 17-21 (2006).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon