È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
ampliPHOX rilevazione colorimetrica su un DNA microarray per l'influenza
In questo articolo
Riepilogo
ampliPHOX tecnologia di rilevamento colorimetrico viene presentato come un'alternativa economica a fluorescenza per microarray. Sulla base di fotopolimerizzazione, ampliPHOX produce spot polimero solido visibile ad occhio nudo in pochi minuti. I risultati sono poi ripreso e interpretato automaticamente con un pacchetto software semplice ma potente.
Abstract
DNA microarray sono emerse come un potente strumento per la rilevazione di agenti patogeni. 1-5 Per esempio, molti esempi della capacità di tipo e sottotipo di virus influenzale sono stati dimostrati. 6-11 L'identificazione e la tipizzazione di influenza sul DNA microarray ha applicazioni sia in pubblico salute e la clinica per la diagnosi precoce, l'intervento rapido, e riducendo al minimo l'impatto di una pandemia influenzale. Fluorescenza tradizionale è attualmente il più utilizzato metodo di rilevamento microarray. Tuttavia, come la tecnologia dei microarray progredisce verso l'uso clinico, 1 sostituzione di strumentazione costosa tecnologia di rilevamento a basso costo che presentano caratteristiche di prestazioni simili a fluorescenza renderà saggi microarray più attraente e conveniente.
La rilevazione ampliPHOX colorimetrico di tecnologia destinata ad applicazioni di ricerca, ed ha un limite di rilevazione all'interno di un ordine di grandezza dei tradizionali fluorescenza 11, con un vantaggio principale è un approssimativo costo dieci volte inferiore rispetto a strumento del microarray scanner confocale necessari per fluorescenza microarray rilevamento. Un altro vantaggio è la compattezza dello strumento che consente la portabilità e flessibilità, a differenza dei tradizionali strumenti di fluorescenza. Perché la tecnologia di polimerizzazione non è così intrinsecamente lineare come rivelazione a fluorescenza, tuttavia, è più adatto per le applicazioni di densità più bassa microarray, in cui è desiderato un sì / no risposta per la presenza di una certa sequenza, come per gli array di rilevazione degli agenti patogeni. Attualmente la densità del punto massimo compatibile con la rilevazione ampliPHOX è ~ 1800 punti / array. A causa delle limitazioni densità posto, microarrays ad alta densità non sono adatti per il rilevamento ampliPHOX.
Qui, vi presentiamo la tecnologia di rilevamento colorimetrico ampliPHOX come metodo di amplificazione del segnale su un microarray a bassa densità sviluppato per la rilevazione e la caratterizzazione dei virus influenzali (FluChip). Anche se questo protocollo utilizza il FluChip (un DNA microarray) come una specifica applicazione di ampliPHOX rilevazione, ogni microarray incorporando obiettivo biotinilato possono essere etichettati e rilevati in modo analogo. Il design microarray e biotinilazione del bersaglio da catturare sono sotto la responsabilità dell'utente. Una volta che il bersaglio biotinilato è stato catturato sulla matrice, il rilevamento ampliPHOX può essere eseguito da tagging primo array con un coniugato streptavidina-label (ampliTAG). Su esposizione alla luce di usare lo strumento ampliPHOX Reader, polimerizzazione di una soluzione di monomero (ampliPHY) si verifica solo nelle regioni contenenti ampliTAG marcato obiettivi. Il polimero formato può essere successivamente colorato con un non-tossico soluzione per migliorare il contrasto visivo, seguita da immagini e analisi utilizzando un semplice pacchetto di software (ampliVIEW). Il test FluChip intero da un-estratto campione risultato può essere eseguita in circa 6 ore, e le fasi di rilevamento ampliPHOX descritto sopra può essere completata in circa 30 min.
Protocollo
1. Esempio di amplificazione con RT-PCR
- Estrarre l'RNA virale da materiale clinico o da un isolato virale del virus usando il kit Qiagen MinElute Spin in collaborazione con il QIAcube piattaforma automatizzata estrazione degli acidi nucleici. Estrazioni vengono effettuate su 200 campioni microlitri con un volume di eluizione finale di 60 microlitri. Estratti di conservare a -70 ° C o inferiore per un uso successivo.
- In un modello free-zona, preparare la RT-PCR master mix su ghiaccio secondo il protocollo del produttore. Per incorporare biotina Durante la RT-PCR, utilizzare una miscela biotinilati dNTP al posto del produttore fornito mix di dNTP. Il costo di utilizzo di una miscela biotinilato dNTP è meno di $ 1 USD / test. Metodi alternativi di incorporazione biotina come l'uso di un primer biotinilato può anche essere usato. Aggiungi fondo mescola a concentrazioni finale di 1,0 mM per l'influenza A, 1,0 mM per B Flu, e 0,14 mM per il controllo interno. L'influenza Un insieme Primer amplifica il segmento gene matrice (1032 prodotto nt) e l'influenza di fondo B amplifica un non-strutturali del segmento genico (811 prodotti nt). Ogni set fondo FluChip contiene un fondo con un 5 'gruppo fosforil per facilitare la generazione di DNA a singolo filamento tramite digestione enzimatica con esonucleasi lambda dopo PCR. Il prodotto risultante singolo filamento richiede tempi molto più brevi di ibridazione o superiore al controvalore a doppio filamento e riduce significativamente il tempo complessivo di analisi. Un pre-preparato miscela di questi primer e il modello di controllo interno RNA sono disponibili da InDevR ai ricercatori interessati su una base limitata.
- Mescolare brevemente nel vortex e distribuire 18 l di mix master in tubi con pareti sottili PCR.
- Trasferimento tubi sul ghiaccio ad un'area di lavoro più adatto per modello e modello di aggiungere 2 microlitri di ciascun tubo di reazione.
- Trasferimento provette PCR ad un termociclatore ed eseguire il seguente profilo termico: la trascrizione inversa a 50 ° C per 30 minuti, l'inattivazione dell'enzima / attivazione a 95 ° C per 15 min, 40 cicli di PCR di 95 ° C per 30 s, 55 ° C per 30 secondi e 72 ° C per 1 min, ed una estensione finale a 72 ° C per 10 min.
2. Ibridazione di RT-PCR prodotti microarray a bassa densità
- Al fine di generare DNA a singolo filamento per l'ibridazione FluChip, preparare la miscela digestione enzimatica, combinando 1,0 microlitri enzima esonucleasi lambda, 2,2 l di buffer di reazione di accompagnamento, e 0,8 l di acqua priva di nucleasi. Tali importi si riferiscono a un singolo campione, ma può essere semplicemente scalati per il numero totale di campioni per essere digerito. Rimuovere campioni dal termociclatore e aggiungere 4 ml di miscela pronta a ogni reazione a digerire il filone fosforilata del prodotto della PCR. Ritorna campioni al termociclatore e programmare il termociclatore a 37 ° C per 15 minuti seguita da 95 gradi Celsius per 10 minuti per completare la digestione enzimatica e passi la frammentazione di calore.
- I microarray influenza personalizzati utilizzati sono stampati su lastre di vetro aldeide funzionalizzati da Applied Microarrays Inc. (Tempe, AZ). 5'-amino sequenze di acquisizione terminato sono combinati con un tampone ottimizzato spotting e stampati ad una concentrazione finale di 20 mM (tranne che per la sequenza di controllo positivo, che ha inoltre un 3'-biotina modifica e viene avvistato a una concentrazione finale di 500 nM) . Un non-contatto spotting metodo viene utilizzato, con un diametro dello spot ottimale di 300 micron e il centro a passo centro di 700 micron.
- Rimuovere microarray dalla casella di stoccaggio e applicare monouso pozzi ibridazione in tutto il microarray rimuovendo il foglio protettivo e premendo con decisione intorno al perimetro bene.
- Collocare i vetrini in un bidone lavare contenente 110 ml di acqua purificata per un min 5 pre-ibridazione lavare con un agitatore orbitale a 60-90 giri al minuto. Asciugare la matrice delicatamente toccando un tergicristallo tessuto al bordo del pozzo e permettendo all'acqua di essere malvagio distanza.
- Unire 22 ml di tampone di ibridazione 2x con ciascuno dei prodotti frammentato ssDNA, e pipetta 40 ml di soluzioni di ibridazione nei pozzetti microarray.
- Permettere che i vetrini a ibridare in camera umida chiuso per 60 minuti.
- Rimuovere i vetrini dalla camera di umidità, brevemente risciacquare array con tampone di lavaggio D in un risciacquo bottiglia prima di inserire nel rack di diapositive. Di solito il volume di risciacquo di tampone di lavaggio D è di 2 ml per array. Luogo cremagliera diapositiva contenente le diapositive di bin lavare contenente 110 ml di Wash Buffer bin A. Mettere su agitatore orbitale a 60-90 rpm per 1 minuto.
- Rimuovere il rack di scivolare dal tampone di lavaggio A, brevemente risciacquare con tampone di lavaggio D, far scorrere cremagliera trasferimento in un bidone contenente tampone di lavaggio B, e si agita a 60-90 rpm per 5 minuti.
- Asciugare delicatamente l'array in ogni diapositiva, e il luogo diapositive essiccato in camera umida in preparazione ampliPHOX fasi di rilevamento colorimetrico.
3. ampliPHOX: etichettatura dei prodotti ibridato e patatine calibrazione con ampliTAG
- Combinare 10 ml di ampliTAG, 20 ml di tampone ampliTAG 2x, e 10 ml di acqua purificata per ogni array da elaborare. I volumi dei reagenti possono essere semplicemente scalati per il numero totale di campioni. Assicurarsi di preparare impasto abbastanza etichettatura per tenere conto di tutti gli array campione e gli array di taratura necessarie. Il numero di chip di taratura necessarie dipende dal fatto che si sta eseguendo una calibrazione completa (per uno strumento nuovo o nuovo lotto di reagenti), o semplicemente un controllo dello strumento. Per una calibrazione completa, 3 chip di taratura devono essere etichettati, e per un controllo reattivo, solo un singolo chip di taratura devono essere etichettati. Si prega di notare che prima della calibrazione array vengono etichettati, devono passare attraverso la pre-ibridazione fase di lavaggio che è stato precedentemente descritto per gli array influenza.
- Trasferire 40 ml di miscela etichetta per ciascuna matrice e consentono l'uso di reazione di procedere in una camera umida chiuso per 5 minuti.
- Sciacquare immediatamente gli array con tampone di lavaggio D in un risciacquo bottiglia prima di mettere le diapositive in un rack di diapositive. Trasferimento cremagliera in bin contenente 110 ml di tampone di lavaggio C, e agitare a 60-90 rpm per 5 minuti.
- Utilizzando un secondo bidone lavare con acqua purificata, eseguire tre cadute consecutive acqua breve per rimuovere i residui di sale. Secco gli array delicatamente toccando un tergicristallo tessuto al bordo dei pozzetti.
- I microarray sono ora correttamente etichettati con ampliTAG, e il resto della procedura di rilevamento ampliPHOX può essere eseguita. Fotoattivazione e l'imaging di array etichetta dovrebbe essere completato entro 24 ore, e gli array aggiuntivi possono essere memorizzati in una scatola buia scivolare fino al momento dell'uso.
4. ampliPHOX: calibrazione, amplificazione del segnale, e di imaging
- Girare ampliPHOX lettore e verificare ampliVIEW software è pronto per fotoattivazione.
- Determinare il momento ottimale fotoattivazione utilizzando chip di calibrazione. In aggiunta alla breve introduzione che segue, questa procedura è descritta dettagliatamente nel manuale d'uso ampliPHOX. I chip di calibrazione contiene una serie di diluizioni di una sequenza di controllo biotinilato che vengono utilizzati per ottimizzare la sensibilità del test, con l'obiettivo di massimizzare il numero di righe del chip di calibrazione che producono un segnale positivo, come determinato dal software.
- Rimuovere ampliPHY da 4 ° C, permettono di riscaldare a temperatura ambiente, e vortice brevemente per mescolare. Pipettare 3 ml di ampliPHY enhancer nel flaconcino ampliPHY, e vortice a fondo per 10 secondi.
- Uniformemente trasferimento 40 ml di soluzione ampliPHY nel microarray contenente il bene ampliTAG marcato array, assicurando che non siano presenti bolle. Chiudere la fiala ampliPHY tra ogni applicazione. Inserire il vetrino microarray nella baia fotoattivazione del lettore ampliPHOX.
- Per l'array prima calibrazione, utilizzare il tempo fotoattivazione predefinito nella casella 'Time' sul pannello sinistro del software, e fare clic sul pulsante verde 'Start' per avviare il fotoattivazione. Una volta completato, rimuovere l'array e sciacquare con acqua purificata per rimuovere ampliPHY in eccesso. Chiara la formazione di polimeri in alcuni dei luoghi dovrebbe essere visibile.
- Lasciare i punti polimero ad asciugare per 2 minuti, quindi distribuire due gocce di ampliRED sul array e colorazione permettono di procedere per 2 minuti.
- Poi, rapidamente microarray risciacquare con acqua purificata e asciugare con un tessuto pulire.
- Inserire microarray nella baia di imaging del lettore ampliPHOX, e clicca sul pulsante 'Image Capture Nuovo' nella scheda Imaging. Una volta ripreso, regolare il raccolto e salvare l'immagine.
- Nella scheda di analisi, selezionare la maschera Chip di calibrazione e il pulsante 'Placement Auto' per avviare l'analisi. Il software produrrà automaticamente un 'resoconto sommario' che mostra i risultati quantificati. Sulla base di questi risultati, il tempo fotoattivazione è regolata da 10 secondi per la matrice di calibrazione secondo, e il procedimento viene ripetuto.
- Una volta che il tempo fotoattivazione ottimale è stato determinato, gli array campione può essere elaborati utilizzando l'amplificazione del segnale e lo stesso protocollo di imaging utilizzata per la calibrazione array.
- Una volta che l'immagine viene acquisita per gli array campione, una maschera per il layout matrice particolare, come la struttura del sistema influenza qui descritto può essere created.The ampliVIEW software è in grado di eseguire l'analisi automatizzata delle immagini e generare risultati dell'influenza sottotipizzazione per ogni campione.
5. Rappresentante dei risultati:
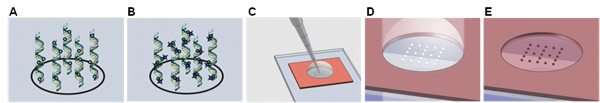
Figura 1. Illustrazione schematica del metodo di rilevazione ampliPHOX colorimetrico. (A) DNA bersaglio biotinilato è ibridato da ogni punto della matrice, e (B) marcato con ampliTAG. (C) soluzione ampliPHY è poi aggiunto, e (D) esposti alla luce per formare macchie polimero visibile. (E) Gli spot polimero formato vengono successivamente tinto con un colorante non tossico per migliorare il contrasto.
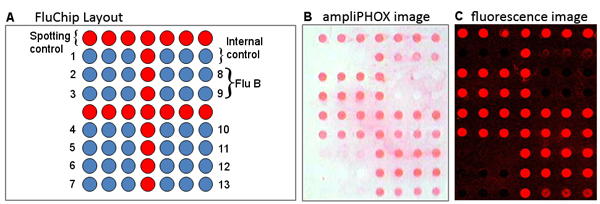
Figura 2. (A) influenza il layout microarray a bassa densità. Sequenze di 1-7 e 10-13 influenza bersaglio A, e le sequenze di 8, 9 bersaglio il virus dell'influenza B. (B) ampliPHOX e (C) immagini di fluorescenza di un romanzo H1N1 2009 ('influenza suina') del campione che mostra lo stesso modello di rilevazione sia metodi.
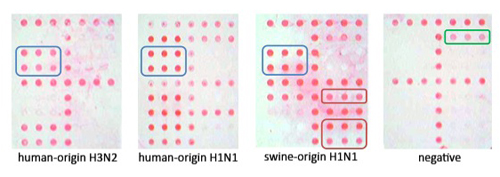
Figura 3. Da sinistra a destra, immagini rappresentative ampliPHOX per l'influenza A H3N2, di origine umana H1N1, 2009 H1N1 (di origine suina), e un campione negativo. Tutti e 3 i sottotipi di mostrare visivamente i modelli distinti sulla matrice. Si noti che in senso negativo solo il controllo interno MS2 è visto, indicando la RT-PCR l'amplificazione non è stato inibito.
Discussione
La rilevazione ampliPHOX colorimetrico tecnologia qui presentata è una rapida, alternativa economica alla rilevazione unico colore a fluorescenza per applicazioni a bassa densità microarray. Mostrato schematicamente nella Figura 1, il principio di rilevazione si basa sull'utilizzo di una etichetta fotoiniziatore (1B). In presenza di un monomero contenente soluzione (1C), l'esposizione alla luce fa sì che il fotoiniziatore (ampliTAG) per innescare una reazione di polimerizzazione solo nelle re...
Divulgazioni
Riconoscimenti
InDevR riconosce NIH / NIAID U01AI070276 e R43AI077112 per il finanziamento di tale opera.
Materiali
| Name | Company | Catalog Number | Comments |
| Reagente / attrezzature | Fabbricante | Catalogo | Commenti |
|---|---|---|---|
| Qiagen MinElute Virus Kit Spin | Qiagen | 57704 | singola 60 microlitri di eluizione |
| QIAcube | Qiagen | 9001292 | opzionale |
| ABI 9800 Veloce termociclatore | Applied Biosystems | 4441166 | |
| Qiagen OneStep RT-PCR kit | Qiagen | 210210 | Kit dNTP non utilizzato |
| 2x Buffer Spotting | InDevR Inc. | MI-5007 | |
| Biotinilato dNTP Mix | InDevR Inc. | MI-5009 | |
| Lambda esonucleasi | Epicentre Biotecnologie | LE032K | 2500 U, 10U/μl |
| FluChip fondo mix | InDevR | N / A | non sono ancora disponibili per la vendita |
| Shaker orbitale | Madell Tecnologia | ZD-9556-A | |
| Lavare Bins | InDevR Inc. | MI-4002 | |
| Lavare Rack | InDevR Inc. | MI-4003 | |
| Ibridazione buffer 2x | InDevR Inc. | MI-5004 | |
| Calibrazione Chips | InDevR Inc. | AP-5006 | |
| Lavare Buffer dC | InDevR Inc. | MI-5005 | |
| ampliRED | InDevR Inc. | AP-5004 | |
| ampliTAG | InDevR Inc. | AP-5001 | |
| 2x ampliTAG Buffer | InDevR Inc. | AP-5002 | |
| ampliPHY, ampliPHY enhancer | InDevR Inc. | AP-5003 |
Riferimenti
- Kumar, R. M. The Widely Used Diagnostics "DNA-Microarray"-A Review. Amer J Inf Dis. 5, 207-218 (2009).
- Miller, M. B., Tang, Y. W. Basic Concepts of Microarrays and Potential Applications in Clinical Microbiology. Clin Microbiol Rev. 22, 611-633 (2009).
- Mikhailovich, V., Gryadunov, D., Kolchinsky, A., Makarov, A. A., Zasedatelev, A. DNA microarrays in the clinic: infectious diseases. BioEssays. 30, 673-682 (2008).
- Call, D. R. Challenges and opportunities for pathogen detection using DNA microarrays. Crit Rev Microbiol. 31, 91-99 (2005).
- Raoult, D., Fournier, P. E., Drancourt, M. What does the future hold for clinical microbiology. Nat Rev Microbiol. 2, 151-159 (2004).
- Dawson, E. D., Rowlen, K. L., Wang, Q., Tao, Y. J. MChip: A Single Gene Diagnostic for Influenza A. Influenza: Molecular Virology. , (2010).
- Gall, A., Hoffman, B., Harder, T., Grund, C., Ehricht, R., Beer, M. Rapid hemagglutinin subtyping and pathotyping of avian influenza viruses by a DNA microarray. J Virol Meth. 160, 200-205 (2009).
- Townsend, M. B., Dawson, E. D., Mehlmann, M., Smagala, J. A., Dankbar, D. M., Moore, C. L., Smith, C. B., Cox, N. J. FluChip: Experimental evaluation of a diagnostic influenza microarray. J Clin Microbiol. 44, 2863-2871 (2006).
- Wang, Z., Daum, L. T., Vora, G. J., Metzgar, D., Walter, E. A., Canas, L. C., Malanosky, A. P., Lin, B., Stenger, D. A. Identifying influenza viruses with resequencing arrays. Emerg Inf Dis. 12, 638-646 (2006).
- Kessler, N., Ferraris, O., Palmer, K., Marsh, W., Steel, A. Use of the DNA Flow-Thru Chip, a three-dimensional biochip, for typing and subtyping of influenza viruses. J Clin Microbiol. 42, 2173-2185 (2004).
- Kuck, L. R., Taylor, A. W. Photopolymerization as an innovative detection technique for low-density microarrays. Biotechniques. 45, 179-186 (2008).
- Avens, H. J., Bowman, C. N. Development of fluorescent polymerization-based signal amplification for sensitive and non-enzymatic biodetection in antibody arrays. Acta Biomat. 6, 83-89 (2010).
- Sikes, H. D., Jenison, R., Bowman, C. N. Antigen detection using polymerization-based amplification. Lab on a Chip. 9, 653-656 (2008).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon