Method Article
Schermi competitivo genomica delle Biblioteche di lievito con codice a barre
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Abbiamo sviluppato completa, imparziale genoma schermate per capire gene-farmaco e interazioni gene-ambiente. Metodi per lo screening queste collezioni mutanti sono presentati.
Abstract
In virtù dei progressi nelle tecnologie di sequenziamento di nuova generazione, abbiamo accesso alle sequenze del genoma nuovo quasi ogni giorno. Il tempo di questi progressi sta accelerando, promettendo una maggiore profondità e ampiezza. Alla luce di questi progressi straordinari, la necessità di una rapida, metodi paralleli per definire la funzione del gene diventa sempre più importante. Collezioni di genome-wide mutanti di delezione in lieviti ed E. coli sono serviti come cavalli da tiro per la caratterizzazione funzionale della funzione del gene, ma questo approccio non è scalabile, attuale gene-cancellazione approcci richiedono ciascuno delle migliaia di geni che costituiscono il genoma da eliminare e verificato. Solo dopo questo lavoro è completo possiamo perseguire high-throughput fenotipizzazione. Negli ultimi dieci anni, il nostro laboratorio ha perfezionato un portafoglio di competitivo, miniaturizzati, ad alta produttività genome-wide test che possono essere eseguite in parallelo. Questo parallelizzazione è possibile perché l'inclusione dei 'tag' del DNA, o 'codici a barre,' in ogni mutante, con il codice a barre che funge da proxy per la mutazione e si può misurare l'abbondanza di codici a barre per valutare idoneità mutante. In questo studio, cerchiamo di colmare il divario tra sequenza del DNA e codice a barre collezioni di mutanti. Per fare questo introduciamo un combinato trasposone interruzione-codici a barre approccio che apre saggi di codici a barre parallele di nuova sequenza, ma poco caratterizzato microbi. Per illustrare questo approccio vi presentiamo una nuova collezione di Candida albicans interruzione codice a barre e di descrivere come sia microarray-based e sequenziamento di nuova generazione basati su piattaforme possono essere utilizzate per raccogliere 10.000 - 1.000.000 gene-gene e le interazioni farmaco-gene in un singolo esperimento.
Protocollo
1. Informazioni di base
Ci sono diversi modi per generare mutanti che portano i tag codice a barre. Il gold standard attuale è il KnockOut lievito (YKO) collezione creata da un consorzio di laboratori e completato nel 2002 1. Poiché il YKO originale è stato introdotto, collezioni lievito altri sono stati generati, in contesti diversi ceppo, utilizzando sovra-espressione costruisce, e in altri microbi quali E. coli 2. In parallelo, lo sforzo di creare librerie di codice a barre shRNA sta procedendo rapidamente, e in effetti, molti dei principi di progettazione di queste collezioni di mammiferi sono state adottate dal lievito. Per dimostrare come trasposoni codice a barre può essere una rapida strategia ampiamente applicabile per la creazione di collezioni sistematiche mutante, ci concentriamo su una raccolta di recente abbiamo creato nel fungo patogeno per l'uomo, la Candida albicans. Il nostro lavoro su Candida si basa sul successo degli schermi codice a barre a S. cerevisiae, e viene qui utilizzato come organismo esempio. Il protocollo campione, con piccole modifiche possono essere usati per lo screening qualsiasi organismo che può essere coltivata in coltura sospensione. Perché gli organismi pochi hanno il requisito alti tassi di trasformazione e ricombinazione mitotica efficienti necessari per creare mutanti di delezione perfetto, di conseguenza abbiamo sviluppato un protocollo che utilizza trasposone mutagenesi in vitro mutagenize una libreria genomica del DNA, e quindi trasformato questi frammenti genomici codice a barre in Candida albicans 3 , 4. Ispirato dal successo della collezione originale YKO e il suo ruolo fondamentale nelle scoperte sulla natura del gene reti di 5-8, genome-wide aploinsufficienza 9, bersaglio di droga e meccanismo di azione 10,11, e l'essenzialità di tutti i geni nel genoma 12 prevediamo ampliando questo approccio per altri microbi sarà estremamente fruttuosa.
Il protocollo qui di seguito presuppone che la raccolta desiderato mutante è stata creata (ad esempio YKO o Candida albicans interruzione di raccolta) ed è disponibile come ceppi singolarmente archiviati. Per una descrizione dettagliata della costruzione ceppo vedere 1,13,14.
2. Combina mutanti singoli in un unico pool
- Consentire una settimana per generare aliquote di cellule in pool (possono essere conservati a tempo indeterminato a -80 ° C).
- Scongelare gli stock di glicerolo congelati per i ceppi di interesse del tutto, ma non lasciare che le cellule rimangono scongelato per> 2 ore.
- Sterilizzare uno strumento pin a 96 pozzetti, immergere lo strumento perno in acqua per rimuovere tutte le cellule rimanenti, seguito da 2 tuffi in vasche etanolo al 70% (ad esempio pipetta coperchi scatola punta), la fiamma lo strumento perno e raffreddare per 1 minuto. Fate attenzione alla fiamma lo strumento pin minuti dai bagni di etanolo. Il livello dei bagni di etanolo dovrebbe superare il livello del bagno d'acqua per garantire che tutti riporto cellule sono fiammato e rimossi. Sostituire l'acqua ogni 4-6 pinnings.
- Inserire lo strumento sterile 96-pin bene in un scongelati piastra a 96 pozzetti, agitare delicatamente e trasferire le cellule ad un vassoio contenente YPD Nunc Omni-agar compreso l'antibiotico appropriato. Crescere colonie fino a raggiungere dimensioni massime a 30 ° C (2-3d). Per conservare piatti, troviamo più utili per consolidare quattro piastre a 96 pozzetti su un singolo Omni-vassoio con ~ 384 ceppi.
- Dopo le colonie sono cresciute, si noti qualsiasi ceppi mancanti o crescita lenta e Repin questi a ~ 2X massa cellulare, come il resto dei ceppi.
- Lavorare in un ambiente di microbiologia (con la fiamma e da laboratorio sterile, piatto inondazione con i media 5-10 ml, immergere per 5 minuti e colonie risospendere con una spatola cellula. Versare il liquido più cellule in una provetta da 50 ml per centrifuga coniche e aggiungere al glicerolo 15% o DMSO al 7% (vol / vol).
- Misurare il diametro esterno 600 della piscina e regolare (per diluizione o centrifugazione) ad una concentrazione finale di 50 OD 600 / ml con supporti contenenti glicerolo 15% o 7% di DMSO.
- Aliquota in 40 volumi microlitri in tubi striscia di PCR e congelare a -80 ° C.
3. Piscina crescita sperimentale
Questa procedura è illustrata in figura 1.
- Scongelare aliquote piscina (nei tubi PCR) su ghiaccio. Se non si utilizza la robotica, passare al punto 5.
- Immediatamente diluire (dolcemente!) la piscina in media con droga o condizione di scelta, inoculando in un OD 600 del 0,0625 in un volume totale di 700 microlitri in 48 pozzetti. Includere almeno un controllo appropriato solvente sul piatto. Per gli esperimenti che si estendono oltre 5 generazioni di crescita (cioè più di 1 bene), riempire i pozzi adiacenti con i media o la condizione di scelta, ma NO cellule.
- Sigillare con un sigillo piatto di plastica, se la condizione richiede la crescita aerobica (per esempio, non fermentescibili fonti di carbonio), utilizzare un 21 gauge per perforare i fori nella guarnizione della membrana verso il lato di ogni bene.
- Crescere in uno spettrofotometro, agitazione a 30 ° C con una determinazione sperimentalened agitazione regime (per esempio 14 min a scuotere impostazione più alta (o temperatura controllata di vibrazione), leggere pozzi, riprendere scuotimento). Una parte della sospensione di cellule possono essere raccolte dal robot e salvate su un piatto freddo sul ponte robotica in punti generazione di tempo definiti dall'utente, tipicamente 5, 10, 15 e 20 generazioni di crescita. (Http://med.stanford.edu/sgtc/technology/access.html, per i dettagli di contatto C. Nislow o G. Giaever).
- Per la crescita cellulare manuale, inoculare un 50 ml per la cultura di partenza OD 600 di 0,002 in una beuta da 250 ml cultura. Agitare a 30 ° C a 250 giri al minuto fino a quando le cellule raggiungono una finale OD 600 di 2.0 (per cerevisiae o Candida) per circa 10 generazioni di crescita. Ulteriori generazioni di crescita può essere ottenuta con cellule diluendo in un OD600 di 2,0 tornare a 0,02 in un pallone di fresco.
- Raccolta di almeno 2 OD ~ 600 unità di cellule per ogni campione / time-point in Safe-Lock tubi microcentrifuga.
Nota: Sempre raccogliere un campione di cellule di partenza (cioè un "T0 punto temporale") per valutare la rappresentanza iniziale sforzo in qualsiasi piscina appena creato con l'aggiunta di 1-2 OD 600 di piscina direttamente dal freezer aliquote in una provetta da 1,5 ml e la lavorazione come descritto di seguito.
4. Estrazione del DNA genomico, PCR e ibridazione microarray o sequenziamento
- Purificare DNA genomico da ~ 2 OD 600 di celle con la ricerca Zymo YeaStar kit in base alle istruzioni del produttore (Protocollo I se si utilizza il DNA di lievito), o con altro metodo adeguato specifico per l'organismo di interesse (standard di estrazione fenolo / cloroformio seguita da precipitazione alcool opere bene per i microbi vari). Se si utilizza il kit YeaStar, eluire il DNA con 300 ml di 0,1 X. TE invece del 60 microlitri di TE 1X specificati nel protocollo. Il DNA genomico può essere conservato indefinitamente a -80 ° C.
- Impostare due reazioni PCR per ogni campione, uno per il uptags e uno per il downtags, con le condizioni di reazione come segue: 33 microlitri DDH 2 O, 6 microlitri di buffer 10X PCR senza MgCl 2, 3 ml 50 mM MgCl 2, 1,2 microlitri 10 mM dNTPs, 1,2 microlitri 50 micron su o giù miscela di primer, 0,6 microlitri 5 U / mL Taq polimerasi, ~ 0.1 mg DNA genomico in 15 microlitri. Volume totale è di 60 microlitri. Thermocycle alle seguenti condizioni: 94 ° C 3 min, 30 cicli di 94 ° C, 30s, 30s 55 ° C, 72 ° C, 30 anni, poi 72 ° C 3 minuti, e tenere a 4 ° C. Controllare i prodotti risultanti PCR su un gel di 60 bp prodotto per entrambe le PCR è previsto per ampliconi utilizzato per l'ibridazione e 130bp per amplificati per il sequenziamento del codice a barre). I prodotti di PCR possono quindi essere conservati a -80 ° C a tempo indeterminato.
- Preriscaldare la temperatura del forno di ibridazione a 42 ° C e creare un bagno di acqua bollente e secchiello per il ghiaccio contenente un impasto di acqua e ghiaccio.
- Pre-umido gli array lentamente riempiendo con 120 microlitri di buffer di ibridazione 1X.
- Incubare nel buffer di ibridazione a 42 ° C e 20 giri per 10 minuti.
- Preparare 90 ml di mix di ibridazione per esempio, oltre a più di un buffer di uno, come segue: 75 microlitri di buffer di ibridazione 2X, 0,5 microlitri B213 oligonucleotidi di controllo (0,2 fm / mL), 12 microlitri oligonucleotidi misto (12:05 / mL), 3 microlitri soluzione 50X Denhardt) nel blocco superiore provette da 0,5 ml.
- Aggiungere 30 microlitri uptag PCR e 30 microlitri downtag PCR a 120 microlitri mix di ibridazione per un volume totale di 150 microlitri. Fate bollire per 2 minuti e messo in acqua ghiacciata per almeno 2 minuti. In breve giro i tubi prima dell'uso.
- Rimuovere la pre-ibridazione buffer dalla array e ibridazione aggiungere 90 microlitri / mix PCR. Per evitare l'evaporazione, coprire le guarnizioni array con un duro-Spot. Ibridare per 16 ore a 42 ° C, 20 min.
- Appena preparare 600 microlitri gamma di marchi biotina per campione, più uno in più, come segue: 180 microlitri 20X SSPE, 12 microlitri 50X Denhardt, 6 microlitri 1% Tween 20 (vol / vol), 1 ml 1 mg / ml streptavidina-ficoeritrina, 401 microlitri DDH 2 O. Conservare tutti i streptavidina-PE campioni al buio. Aliquota 600 microlitri in 2 ml provette. Rimuovere Tough-Spots da trucioli.
- Rimuovere lentamente mix di ibridazione dagli array di pipetta e riempire microarray con 120 microlitri Lavare A. Primo stazione Affymetrix fluidica.
- Lavare gli array utilizzando un Affymetrix fluidica stazione secondo le istruzioni del produttore, utilizzando il protocollo "Gene-Flex_Sv3_450" con le seguenti modifiche: 1 punto in più con lavaggio A (1 ciclo, 2 mix) prima della colorazione, temperatura di lavaggio B 42 ° C invece di 40 ° C, macchia a 42 ° C invece di 25 ° C. E 'anche possibile effettuare il lavaggio dopo ibridazione, la colorazione biotina, e il lavaggio colorazione posta manualmente (vedi p. 396 in riferimento 15). A seguito di operazioni di fluidica, eseguire la stazione fluidica protocollo "SHUTDOWN_450".
- Dopo il lavaggio, assicurarsi che non siano presenti bolle d'aria. Se necessario aggiungere 90 microlitri Lavare A e pipetta lentamente finché le bolle non scompaiono. Se ci sono segni o smudges sulla superficie dell'array, pulire il vetro con isopropanolo e un panno privo di tessuto. Applicare fresco Tough-Spots oltre le guarnizioni / setti e luogo gli array nello scanner.
- Scansione in uno scanner Affymetrix GeneArray ad una lunghezza d'onda di emissione di 560 nm.
5. Array di analisi (vedi Figura 2 per un esempio ottenuto usando l'interruzione Candida albicans raccolta)
- Mascheramento outlier: dato l'array Affymetrix TAG4 contiene 5 repliche di ciascun complemento di codici a barre dispersi in modo casuale qualsiasi sonda codice a barre che devia dalla replica può essere mascherato e scartati. Per fare questo, per ogni caratteristica array che sembra essere un outlier in base al suo segnale rispetto alle altre 4 repliche di questa funzione, il nostro software esamina anzitutto le 5 caratteristiche che circondano il sospetto outlier, producendo una matrice di 25 funzioni con il sospetto caratteristica al centro. Se> 13/25 sonde in questa regione diversa da ciascuna delle loro singole loro media replicare tagliati (la media delle tre repliche al centro, escludendo il più alto e più basso si replica) di oltre il 10%, questa sonda viene poi scartato da ulteriori analisi. Poiché tali valori anomali sono spesso il risultato di post-ibridazione incongruenze lavaggio, si espandono o "pad" la regione contenente sonde sospetto. Sonde pad tale includendo tutte le sonde all'interno di un raggio di 5-sonda, secondo la definizione di ((x 1-x 2) 2 + (y 1-y 2) 2) ½ <6 dove x 1, x 2, y 1, e y 2 sono le coordinate x e y per le due funzioni. Infine, scartare funzioni per le quali la deviazione standard (incluso nel. Cel file per gli array Affymetrix) di pixel caratteristica / media pixel funzione. Dopo aver rimosso anomali, valori medi l'intensità per tutti i rimanenti repliche.
- Rimozione di tag inutilizzabile: Tag con bassa intensità valori darà scarsa qualità dei risultati e devono essere rimossi. Un taglio di esclusione per queste sonde a bassa intensità può essere calcolato come segue:
- Per qualsiasi prestazione per il controllo coppia di array, calcolare log 2 ((i c-b g) / (i t-b g)) per ogni tag, dove i c è l'intensità di controllo, i t è l'intensità del trattamento, e b g è l'intensità media delle sonde etichetta assegnata.
- La coppia uptag e rapporti downtag da sforzo e per ogni coppia di tag, prendere l'intensità minima per i due tag nei due campioni. Ordina le coppie rapporto con questa intensità minima.
- Utilizzare una dimensione della finestra scorrevole di 50 sulle coppie di rapporto classificato, calcolare la correlazione di uptag e downtag coppie di rapporto all'interno della finestra. Anche calcolare la media delle intensità minima calcolata nel passaggio precedente.
- Far scorrere la finestra di 25 coppie, e ripetere il passaggio precedente fino a quando tutte le coppie sono state incrociate.
- Plot l'intensità media minima rispetto al uptag-downtag di correlazione per tutte le finestre.
- Infine, scegliere una soglia di intensità, in genere si usa il valore di intensità in cui la correlazione raggiunge prima l'80% del suo livello massimo. Bandiera e togliere da ulteriori analisi nessun tag di sotto di questa soglia.
- Correzione di saturazione: Perché ogni funzione sul microarray codice a barre può diventare satura, il segnale sulla matrice TAG4 non è correlata linearmente alla concentrazione tag. Per correggere questa saturazione seguire il protocollo descritto nel riferimento 16.
- Normalizzazione matrice: per ogni array, normalizzare il uptags e downtags separatamente. Per normalizzare quantile, il rango dei valori ottenuti da ciascuna matrice per uptags e downtags in ordine di intensità crescente. Per dire normalizzare per ogni set di uptags e downtags, dividere per la media. La normalizzazione media di tutti gli array è un secondo passo seguente trasformare i dati grezzi per la media di ciascun array.
- Calcolare i punteggi di sensibilità per il controllo del trattamento confronti: Per utilizzare log 2 rapporti come metrica di sensibilità: per ogni ceppo, calcolare log 2 ((μ g c-b) / (μ g t-b)), dove c è il μ intensità media per i campioni di controllo, μ t è l'intensità media per i campioni di trattamento, e b g è l'intensità media delle sonde non assegnati. Ceppi con un positivo rapporto di log 2 sono sensibili al trattamento, e quelli che sono resistenti sono negativi log 2 rapporti.
6. Valutare l'idoneità dei ceppi di lievito con codice a barre mediante sequenziamento
- Isolare il DNA dalle piscine cancellazione come descritto per microarray.
- Amplificare ogni 20-mer codice a barre uptag con primer composito costituito da sequenze di primer comune codice a barre e le sequenze necessarie per l'ibridazione di cella a flusso Illumina (Vedi Tabella dei primer Illumina e la Figura 3 per un diagramma del amplicone). Questeprimer può essere utilizzato dissalate senza ulteriore purificazione. PCR viene eseguita in 100μL, utilizzando Invitrogen Platinum PCR Supermix (Cat. No. 11306-016) con le seguenti condizioni: 95 ° C / 3 min, 25 cicli di 94 ° C/30 sec, 55 ° C/30 sec, 68 ° C/30 secondi, seguiti da 68 ° C/10 min.
- Purificare il prodotto di PCR (~ 130bp) con Qiagen MinElute 96 UF PCR Purification Kit (Cat. N. 28051).
- A seguito di purificazione PCR, quantificare il DNA con la Quant-iT Invitrogen dsDNA BR Assay Kit (Cat. No. Q32853). Non contare su 260/280 letture!
- Normalizzare la concentrazione del DNA per volumi 10μg/ml e piscina pari del DNA normalizzata.
- Separare il DNA in pool su un gel di poliacrilamide al 12% TBE per 3-4 ore a seconda del voltaggio utilizzato.
- Macchia gel con bromuro di etidio (SYBR Green dovrebbe funzionare così) per 30 minuti.
- Individuare la banda di interesse su un lightbox UV a onde lunghe (indossando un'adeguata protezione viso), tagliato fuori ed estratto il DNA utilizzando la calca e godersi il metodo seguito dalla precipitazione 17 etanolo.
- Conferma che il DNA dimensione appropriata (130bp) è stato isolato e che inneschi sono stati rimossi con il Agilent Bioanalyzer High Sensitivity DNA kit (Cat. No. 5067-4626).
- Esempio di sequenza:
- Illumina GAIIx piattaforma:
- Generare cluster su un singolo Leggi cella di flusso utilizzando il CBOT e Single-Read Kit Generation Cluster (Cat. No. GD-300-1001). Per Leggi 1, SU e GIU-tag primer modificato sequenziamento sono raggruppate ad una concentrazione magazzino 100um e aggiunto una striscia-tube (0.6μL di ciascun primer di sequenziamento 100um in 120 microlitri HT1). SR_Amp_Block_StripTubeHyb_v7.0 ricetta è usato per generare cluster R1.
- Sequenziamento del genoma Analyzer IIx. A seguito di 18 cicli di sequenziamento, il appaiati fine modulo è utilizzato per togliere il filamento sintetizzato primo e rehybridize la cella di flusso, con la R1 Illumina (sotto). I cluster sono rigenerati e in sequenza per 5 cicli di catturare il tag di indice.
- Illumina GAIIx piattaforma:
- La sequenza di tag indice è utilizzato per le sequenze bin in contenitori sperimentali.
- All'interno di ogni bin sperimentale, le sequenze di lievito di codici a barre sono conteggiati per un numero totale di conteggi per ogni codice a barre.
- Conta sono normalizzati quantile in modo che ogni esperimento ha la stessa distribuzione di conteggio. Per analogia con gli esperimenti microarray codice a barre fitness, rapporti di idoneità difetto per ogni ceppo sono calcolati ed espressi come rapporto log 2 (controllo / trattamento). Punteggi positivi difetto di fitness significare una diminuzione della tensione abbondanza durante il trattamento di droga e suggeriscono che la wild-type versione del gene soppresso in questa tensione è necessario per la resistenza a quel farmaco o inibitore.
Nota: Considerando Bar-ss come alternativa alla ibridazione di array. Con i costi di high-throughput sequencing in calo, con high-throughput sequencing come una lettura di abbondanza tag è sempre fattibile e in molti casi, è più conveniente 18. In questo modo, amplificato prodotto di PCR viene misurato direttamente come 'conta' piuttosto che come intensità del segnale come ibridato ad un array. Questo elimina i falsi negativi e positivi che derivano da tag contaminazione incrociata, saturazione o problemi derivanti da intensità di segnale molto alta o molto bassa. Inoltre, gli esperimenti multipli possono essere combinati prima del sequenziamento con l'aggiunta di una base del DNA 4-8 indice 19. Perché i codici a barre lievito sono 20 bp, una singola, 2-step letto di 26-28 basi cattura sia indice di multiplex e codice a barre unico, consentendo estrema 100 + multiplexing. Al momento in cui scriviamo, Bar-seq offre un vantaggio di costo su codici a barre microarrays, e, inoltre, Bar-seq è intrinsecamente flessibili, che come il numero di aumenti di legge / run, il livello di multiplexing può aumentare a diminuire ulteriormente costs . Diversi "mid-capacità" sequenziatori da tutti i produttori più importante piattaforma ulteriormente democratizzare Bar-seq, con sequenziamento rischia di diventare la lettura di scelta.
Questo protocollo è stato anche convalidato il Illumina HiSeq2000.
Un'eccellente dimostrazione dell'uso di Bar-seq per affrontare una questione fondamentale nel controllo della crescita biologica Saccharomyces cerevisiae è presentato in un recente studio di Gresham et al. 20 che hanno delineare alcuni importanti sperimentale di design e le linee guida di interpretazione.
7. Validazione dei dati di screening in pool
I risultati da qualsiasi schermata di genomica funzionale deve essere verificata utilizzando i ceppi isolati nella cultura individuale. Poiché ogni esperimento sarà diversa in termini di numero di ceppi sensibili, selezionando il numero di ceppi candidato per confermare è alquanto arbitraria. Come guida, classifica i ceppi più sensibili per il rapporto di log 2 o z-score e testare la parte superiore del 25-50% dei candidati (che si traduce di solito a 2-3 standard deviations dalla media di tutti i ceppi in piscina) è un buon equilibrio tra costi e benefici. Conferme individuali può essere eseguita in qualsiasi fiasco ma eseguire questi test da 5 generazioni di crescita in 96 pozzetti utilizzando un inoculo di partenza di 0,06 OD 600 in 100 l di media in uno spettrofotometro agitazione, le misurazioni ogni 15 minuti (vedi figura 4) .
8. Rappresentante Risultati
Una volta che un genome-wide a schermo è completo, e gli array sono stati normalizzati e il comportamento di ogni ceppo rispetto ad un trattamento di controllo (ad esempio confrontando intensità microarray o sequenziamento conteggi / ceppo) i dati sono più facilmente manipolata in un file excel con genes classificato dal log2 rapporti di controllo / esperimento. In questo modo, maggiore è il rapporto log2 negativo, il più sensibile che il ceppo particolare è la condizione di test. Questi file Excel possono essere tracciati in una varietà di pacchetti software grafici. Lo troviamo più semplice per tracciare i rapporti log2 sull'asse Y e il gene oi nomi ORF sull'asse X. Nell'esempio mostrato nella Figura 2a, come una trama di trattamento clotrimazolo (un noto agente antimicotico) viene visualizzato. Tutte le tensioni che sono significativamente sensibili al trattamento con un rapporto di 2 log2 sono evidenziati in rosso, e noi in genere verificare molti di tali ceppi in analisi la crescita individuale di ogni mutante, in presenza della stessa concentrazione di farmaco. In questo esempio, 4 ceppi sono evidenziati, NCP1, ERG2 e 2 alleli indipendente ERG11, l'obiettivo proteina nota di clotrimazolo. Ognuno di questi 4 geni è direttamente coinvolto nella biosintesi dell'ergosterolo, il lievito equivalente di colesterolo. Per esempio, NCP1 codifica una NADP-reduttasi del citocromo P450 che è coinvolto nella biosintesi dell'ergosterolo e che è associata e coordinatamente regolato con Erg11. Questo esempio evidenzia il fatto che l'obiettivo farmaco noto (Erg11) viene identificato in questa schermata imparziali, così come molti altri componenti chiave del percorso di destinazione. Infine, molti dei geni evidenziato in rosso rappresentano i geni che possono essere coinvolti nella biosintesi dell'ergosterolo o in distinti processi biologici. Come accennato in precedenza, ogni ceppo individuato come sensibili in una schermata in pool deve essere verificata per la sua sensibilità nel dosaggio crescita individuale. Nell'esempio mostrato nella Figura 2b, quattro ceppi sono confermati ad essere sensibili al clotrimazolo in base alla loro crescita è diminuito rispetto al ceppo selvatico genitore, BWP17. Queste curve di crescita individuale in evidenza una caratteristica importante di tale pool genico-droga schermi, cioè il grado assoluto di un particolare ceppo non riflette necessariamente il suo esatto livello di sensibilità. Inoltre, la Figura 2b mostra anche il valore di avere più alleli per ogni gene, in questo caso, i due mutanti erg11 disagi sono leggermente diverse sensibilità. Correlare la natura di queste interruzioni con il grado di sensibilità può fornire un'ulteriore visione delle droghe il meccanismo d'azione.
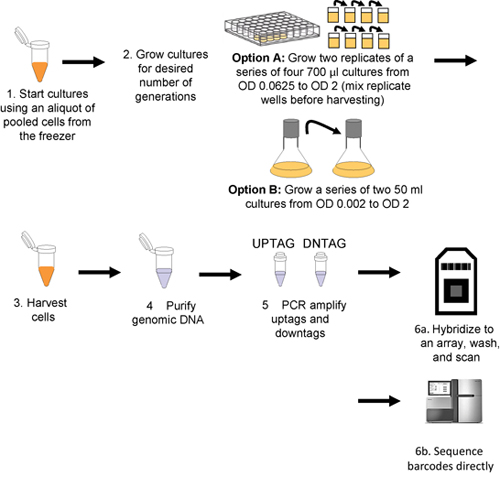
Figura 1. Flusso di lavoro per il saggio di crescita in pool e la rilevazione di codici a barre. Colture vengono inoculate con aliquote scongelati pool di cellule (fase 1), e poi coltivate per il numero desiderato di generazioni (punto 2) o robot (opzione A) o manualmente (opzione B). Le cellule sono raccolte mediante centrifugazione (fase 3) e del DNA genomico è poi isolato dalle cellule raccolte (fase 4), uptags e downtags sono amplificati in modo indipendente (punto 5), e ibridato ad un array (passo 6a o in sequenza direttamente passo 6b).
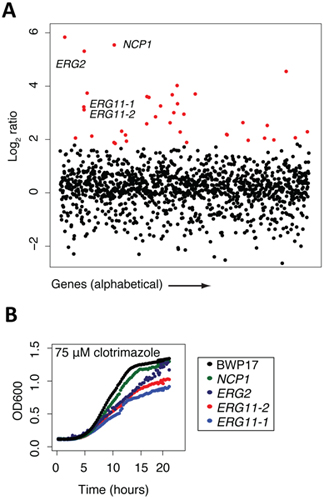
Figura 2. I dati di esempio raccolti in alcuni punti del protocollo. (A) i dati del campione da risultati dello screening (adattato da 13). Il pool di mutanti tag è stato coltivato per 20 generazioni in presenza di clotrimazolo e DMSO (controllo). Log 2 rapporto (controllo dell'intensità / trattamento intensità) è stato calcolato e tracciato in funzione del gene. Ceppi ad alta sensibilità (rosso) incluso l'obiettivo nota di clotrimazolo, ERG11p. Si noti che questa analisi rivela spesso altri mutanti sensibili in aggiunta al bersaglio vero del composto. In genere, questi mutanti sono quelli che agiscono sinteticamente con il target, quelli che fanno parte di un generale sforzo / risposta al trattamento, o sono falsi positivi che non riescono a confermare. (B) Esempio di dati conferma (adattato da 13). I risultati del test di crescita in pool può essere convalidato da una crescente la tensione nella cultura individuale e confrontati con wild-type di crescita (nero).
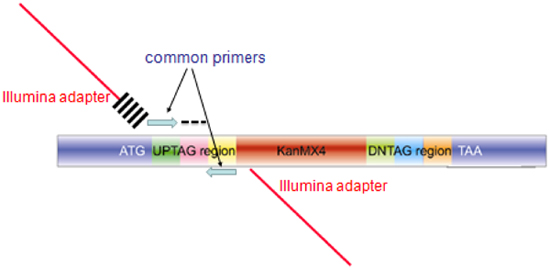
Figura 3. Struttura del amplicone ottenuto da pool di saggi di codici a barre per l'ibridazione di microarray o sequenziamento codici a barre. L'amplicone prodotte per ogni mutante in la raccolta contiene omologia con il genoma per l'integrazione (regioni blu etichettato ATG e TAA), codici a barre unico (con l'etichetta AG e indicati da un trattino nero). Per l'ibridazione di microarray, i primer blu comune sono usati per amplificare una sonda per l'ibridazione microarray 60bp. Per il sequenziamento di codici a barre, primer esteso vengono utilizzati nella reazione di PCR, composto da sequenze di codifica della scheda Illumina (barra rossa) e indice di 6bases cross-portelli) e il fondo azzurro comune per il primer a monte, e il primer stesso composito (meno i 6 indice di base) per il fondo secondo.
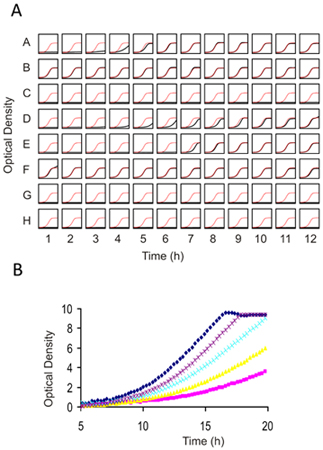
Figura 4. Saggi di crescita individuale per 1) Prescreening composti contro la wild-type lievito per determinare una dose adeguata di genome-wide screening e 2) Conferma dei risultati da schermi di genome-wide. (A) A 96 piatto fondo e piatto è riempito con 100 ml di sospensione cellulare con un diametro esterno di 0,062. Ogni bene può contenere lo stesso ceppo (per la dose-determinazione) o diverse combinazioni di tensione e di droghe (per i test di conferma). 2 ml di composto (tipicamente sciolto in DMSO) viene aggiunto e le cellule sono coltivate con costante agitazione per 16-20 ore a 30 ° C. La concentrazione finale di DMSO non deve superare il 2%. In questo esempio, in ciascun pozzetto della piastra la curva di crescita è tracciato in nero contro un complotto della curva di controllo della crescita in rosso. (B) immagine ad alta risoluzione di prescreens diversi ottenuti con un farmaco esempio sovrapposti uno sopra l'altro. In questa serie di titolazione, un IC 10-15 si ottiene con la dose viola e sarebbe opportuno per il profiling la cancellazione (e HIP HOP). A causa della non linearità alle alte densità ottiche, Tecan (o qualsiasi altro lettore di piastre simili) OD deve essere calibrato con quelli ottenuti con un "tradizionale" 1 millimetro di lunghezza percorso cuvetta.
Discussione
Qui, abbiamo delineato un protocollo che, con modeste modifiche, può essere facilmente adattato ad una vasta gamma di collezioni esistenti di codici a barre collezioni mutante di microrganismi diversi per creare tag collezioni mutante. Sottolineiamo che, mentre noi abbiamo riportato un protocollo di mutagenesi trasposone tag per i patogeni lievito C. albicans, un protocollo molto simile potrebbe essere adattato ad una grande varietà di funghi unicellulari. Modificato, questo protocollo funziona bene nei batteri 13, e attualmente collezioni per una serie di ulteriori genomi fungini e batterici sono in costruzione. Al momento, questo test fornisce l'unica completa, genome-wide a schermo imparziali per gene-piccole interazioni molecola. Una caratteristica particolarmente interessante del saggio è che nessuna conoscenza preliminare del gene o piccola molecola è richiesto. Nonostante la portata e la potenza di questi test, la loro trasferibilità in altri laboratori è stato ostacolato in qualche modo con l'investimento di capitale iniziale e gli strumenti informatici per l'analisi dei risultati. Con l'adozione di una lettura prossima sequenza di generazione combinata con strumenti affidabili per l'analisi, ci aspettiamo che la loro adozione ad aumentare.
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Ringraziamo Ron Davis, Adam Deutschbauer, e l'intero laboratorio HIP HOP presso l'Università di Toronto per le discussioni e consigli. NC è sostenuto da finanziamenti della National Human Genome Research Institute (Grant Numero HG000205), RO1 HG003317, CIHR MOP-84305, e Canadian Cancer Society (# 020380). JO è stato sostenuto dal Programma di Stanford Formazione Genoma (Grant Numero T32 HG00044 dal National Human Genome Research Institute) e il National Institutes of Health (Grant Numero P01 GH000205). GG è supportato dal RO1 NHGRI HG003317 e la Canadian Cancer Society, Grant # 020380, TD e il Donnelly Sequencing Center è sostenuto in parte da finanziamenti della Fondazione canadese per l'innovazione di Drs. Brenda e Jack Andrews Greenblatt. AMS è supportato da una borsa di studio dell'Università di Toronto Open.
Materiali
| Name | Company | Catalog Number | Comments |
| Antibiotici | Vendor e catalogo numeri | ||
| Carbenicillina | Sigma, parte # C1613 | ||
| Kanamicina | Sigma, parte # K1876 | ||
| spectinomicina | Sigma, parte # S0692 | ||
| Cloramfenicolo | Sigma, parte # C0378 | ||
| DNA di pulizia e kit di concentrazione | |||
| QIAprep Spin Miniprep Kit | Qiagen, parte # 27106 | ||
| HiSpeed Kit Maxi plasmidi | Qiagen, parte # 12663 | ||
| QIAquick PCR Purification Kit | Qiagen, parte # 28106 | ||
| QIAquick Gel Extraction Kit | Qiagen, parte # 28704 | ||
| PCR e reagenti elettroforesi | |||
| Taq DNA polimerasi (Mg-free) Buffer | New England Biolabs, parte # M0320L | ||
| Soluzione Mix desossinucleotide | New England Biolabs, parte # N0447L | ||
| 25 mM MgCl 2 | Sigma, parte # 63036 | ||
| Agarosio, carico colorante e acido nucleico macchia adatto per elettroforesi su gel | Vari | ||
| 10X tampone TAE | Sigma, parte # T8280 | ||
| 1 Kb Inoltre DNA Ladder | Invitrogen, parte # 10787026 | ||
| YPD brodo | |||
| 10 g di estratto di lievito | Sigma, parte # Y1625 | ||
| 20 g Bacto peptone | BD Biosciences, parte # 211677 | ||
| 20 g di destrosio | Sigma, parte # D9434 | ||
| Da laboratorio | |||
| Pipette multicanale (1000, 200, e 20 mL) | Vari | ||
| Monouso serbatoi pipettaggio | Vari | ||
| 15 e 50 provette ml | Vari | ||
| 96 - e pozzetti 384-pozzo profondo | Axygen Scientifico, parte # P-2ML-SQ-CS & P-384240SQCS | ||
| 96-bene e 384 pozzetti PCR e film di tenuta | Vari | ||
| Rullo di lamiera per la sigillatura a più pozzetti | Sigma, parte # R1275 | ||
| 30 ° C e 37 ° C agitando incubatori per la crescita di batteri e lieviti su piastre e tubi | Vari | ||
| In vitro trasposone mutagenesi | |||
| EZ-Tn5 trasposasi | Biotecnologie Epicentre, parte # TNP92110 | ||
| High-throughput trasformazione | |||
| Seqprep 96 HT Prep Kit plasmidi | Bordo Biosystems, parte # 84359 | ||
| polietilene glicole, peso molecolare 3350 | Vari | ||
| acetato di litio | Sigma, parte # 517992 | ||
| 6-pozzetti, sterile | Corning, parte # 3335 | ||
| 50 mg / ml uridina | Sigma, parte # U3750 | ||
| 100X Tris-EDTA soluzione tampone | Sigma, parte # T9285 | ||
| 1X TE/0.1M LiOAc | Vari | ||
| salmone testicolo DNA | Sigma, parte # 1626 | ||
| La crescita delle collezioni barcode | |||
| 48-pozzetti, qualora la crescita culture in lastre | Greiner, parte # M9437 | ||
| Sigillante adesivo | ABgene, parte # AB-0580 | ||
| 200 mL cultura fiaschi | Vari | ||
| Spettrofotometro in grado di assorbanza | Vari | ||
| A temperatura controllata shaker per 250 ml o palloni spettrofotometro agitazione | Vari | ||
| Safe-Lock provetta, 2 ml | Eppendorf, parte # 0030 120.094 | ||
| Ibridazione attrezzature | |||
| Ibridazione Forno 640 | Affymetrix, parte # 800138 | ||
| GeneChip fluidico Stazione 450 | Affymetrix, parte # 00-0079 | ||
| GeneArray Scanner 3000 | Affymetrix, parte # 00-0212 | ||
| Bagno di acqua bollente con cremagliera galleggiante | Vari | ||
| Ibridazione di consumo | |||
| Genflex Tag 16K Array v2 | Affymetrix, parte # 511331 | ||
| Soluzione Denhardt, il concentrato di 50X | Sigma, parte # D2532 | ||
| Streptavidina, R-ficoeritrina coniugato (SAPE) | Invitrogen, parte # S866 | ||
| Safe-Lock provetta, 0,5 ml | Eppendorf, parte # 0030 123.301 | ||
| Teeny Tough-Spots | Diversificato Biotech, parte # LTTM-1000 | ||
| 0,5 M EDTA | BioRad, parte # 161-0729 | ||
| 10% Tween | Sigma, parte # T2700 | ||
| MES monoidrato acido libero | Sigma, parte # M5287 | ||
| MES sale di sodio | Sigma, parte # M5057 | ||
| 5 M NaCl | Sigma, parte # 71386 | ||
| 20X SSPE | Sigma, parte # S2015 | ||
| Biologia molecolare acqua grado | Sigma, parte # W4502 | ||
| Ibridazione Primer | Vari fornitori (dissalazione di serie) | ||
| Uptag | 5 'GATGTCCACGAGGTCTCT 3' | ||
| Buptagkanmx4 | 5 'biotina-GTCGACCTGCAGCGTACG 3' | ||
| Dntag | 5 'CGGTGTCGGTCTCGTAG 3' | ||
| Bdntagkanmx4 | 5 'biotina-GAAAACGAGCTCGAATTCATCG 3' | ||
| B213 | 5 'biotina-CTGAACGGTAGCATCTTGAC 3' | ||
| Uptagkanmx | 5 'GTCGACCTGCAGCGTACG 3' | ||
| Dntagkanmx | 5'GAAAACGAGCTCGAATTCATCG 3 ' | ||
| Uptagcomp | 5'AGAGACCTCGTGGACATC 3 ' | ||
| Dntagcomp | 5'CTACGAGACCGACACCG 3 ' | ||
| Upkancomp | 5'CGTACGCTGCAGGTCGAC 3 ' | ||
| Dnkancomp | 5'CGATGAATTCGAGCTCGTTTTC 3 ' | ||
| Sequencing Primer: Piattaforma Illumina | Vari fornitori | ||
| UpTag Avanti (100um) | 5 'CAA GCA GAA GAC GGC ATA CGA GCT CTT CCG ATC T GAT GTC CAC GAG GTC TCT 3' | ||
| UpTag Reverse (100um) | 5 'AAT GAT ACG GCG ACC ACC GAC ACT CTT CCT CTA CAC GAC GCT CTT CCG ATC T NNNNN GTC GAC CTG CAG CGT ACG 3' | ||
| DownTag Avanti (100um) | 5 'CAA GCA GAA GAC GGC ATA CGA GCT CTT CCG ATC T GAA AAC GAG CTC GAA TTC ATC G 3' | ||
| DownTag Reverse (100um) | 5 'AAT GAT ACG GCG ACC ACC GAC ACT CTT CCT CTA CAC GAC GCT CTT CCG ATC T NNNNN CGG TGT CGG TCT CGT AG 3 ` | ||
| Leggi 1 UP-tag seguenti primer (100um) | 5 'GTC GAC CTG CAG CGT ACG 3' | ||
| Leggi 1 GIU-tag Primer seq (100um) | 5 'CGG TGT CGG TCT CGT AG 3' | ||
| Leggi 2 Primer Sequencing Index (standard Illumina fondo R1) (100um) | 5 'AC ATTO CTT CCT CTA CAC GAC GCT CTT CCG ATC T 3' | ||
| Reagenti Sequencing Ulteriori / attrezzature | |||
| Qiagen MinElute 96 UF Kit di purificazione PCR | Qiagen, parte # 29051 | ||
| Pompa del vuoto | Qualsiasi fornitore | ||
| Macherey-Nagel Vacuum Manifold | Macherey-Nagel, parte # 740 681 | ||
| Invitrogen Quant-iT dsDNA BR Assay Kit | Invitrogen, parte # Q32853 | ||
| Invitrogen Qubit saggio tubi | Invitrogen, parte # Q32856 | ||
| Acrilamide 40% più 1% di N, N'-metilene-bis-acrilamide, 37.5:1 | Bio Rad, part # 161-0148 | ||
| Tris Base | Sigma, parte # T1503-1KG | ||
| Acido borico | Sigma, parte # B6768-500G | ||
| 0.5M EDTA, pH 8,0 | Teknova, parte # E0306 | ||
| Ammonio persolfato | Sigma, parte # A3678-25G | ||
| TEMED | Bioshop, parte # TEM001.25 | ||
| Bromuro di etidio Soluzione | Bioshop, parte # ETB444.10 | ||
| Ammonio acetato 0.5M | Teknova, parte # A2000 | ||
| 10mM acetato di magnesio tetraidrato | Sigma, parte # M0631-100G | ||
| 1mM EDTA, pH 8,0 | Vedi 0.5M EDTA, pH 8,0 | ||
| Etanolo | Vari | ||
| Di sodio acetato, pH 5,2 | Teknova, parte # S0297 | ||
| Velocità a vuoto | Vari | ||
| Leggi singolo cluster Kit Generation | Illumina, parte # GD-300-1001 | ||
| 36 quater Sequencing Kit v4 | Illumina, parte # FC-104-4002 | ||
TBE ricetta 10X
| Importi | Reagenti |
| 108 grammi | Tris Base |
| 55 grammi | Acido borico |
| 40ml | 0.5M EDTA (pH 8,0) |
| Aggiungi dH 2 O a 1L marchio | |
12% di poliacrilammide ricetta gel
| Volumi | Reagenti |
| 5,8 ml | Acrilamide 40% più 1% di N, N'-metilene-bis-acrilamide, 37.5:1 |
| 12 ml | dH 2 O |
| 2 ml | TBE 10X |
| 140 microlitri | Persolfato di ammonio 10% |
| Volume totale: 20ml |
Uptag mix di fondo:
Risospendere Uptag e Buptagkanmx4 ciascuno in DDH 2 O a 100 micron, quindi mescolare in un rapporto di 1:1 per una concentrazione finale di 50 micron ciascuna. Conservare a -20 ° C.
Downtag mix di fondo:
Risospendere Dntag e Bdntagkanmx4 ciascuno in DDH 2 O a 100 micron, quindi mescolare in un rapporto di 1:1 per una concentrazione finale di 50 micron ciascuna. Conservare a -20 ° C.
Oligonucleotidi mista:
Risospendere ciascuna delle seguenti otto oligos (standard dissalate) in DDH 2 O a 100 mM:
Uptag, Dntag, Uptagkanmx, Dntagkanmx, Uptagcomp, Dntagcomp, Upkancomp, Dnkancomp.
Mescolare un volume equivalente di otto oligonucleotidi per una concentrazione finale di 12,5 mM ciascuno.
12X MES magazzino:
Per 10 ml, sciogliere 0,7 g monoidrato acido libero MES e 1,93 g di sale di sodio MES in 8 acqua biologia molecolare grado ml. Dopo aver mescolato bene, controllare il pH e regolare, se necessario, a pH 6,5-6,7. Aggiungere acqua per un volume totale di 10 ml. Filtro sterilizzare e conservare a 4 ° C al riparo dalla luce (ad esempio, avvolgere il tubo in alluminio). Sostituire se visibilmente soluzione diventa gialla o dopo 6 mesi.
Ibridazione tampone 2X:
Per 50 ml, miscelare 8,3 ml di 12X MES magazzino (dal 2.9.14), 17,7 ml di 5 M NaCl, 4,0 ml di 0.5 M EDTA, 0,1 ml di 10% di Tween 20 (vol / vol), e 19,9 ml di filtrato DDH 2 O. Filtro sterilizzare.
Lavare A: Mescolare 300 ml 20X SSPE, 1 ml di Tween 10% (vol / vol), 699 ml di DDH 2 O. Filtro sterilizzare.
Lavare B: Mescolare 150 ml 20X SSPE, 1 ml di Tween 10% (vol / vol), 849 ml di DDH 2 O. Filtro sterilizzare.
Codice a barre sequenza primer
In UP-tag sequenze primer 5 'coda (in grassetto) sono Illumina sequenze specifico adattatore incorporato nel fondo F e R. La sequenza variabile (corsivo) rappresenta il 5-mer tag di indicizzazione utilizzato in multiplexing / index read-out. Coda di 3 '(sottolineato) rappresenta l'innesco comune che fiancheggiano il codice a barre uptag ed è necessario per amplificare i codici a barre lievito.
In GIU-tag Primer sequenze 5 'coda è identico a 5' coda di UP-tag primer (Illumina sequenza specifica), tuttavia, il 3 'coda (sottolineato), è sostituito con il primer comuni che vengono utilizzati per amplificare il DOWN- tag codici a barre.
Riferimenti
- Giaever, G. Functional profiling of the Saccharomyces cerevisiae genome. Nature. 418, 387-391 (2002).
- Baba, T. Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection. Molecular systems biology. 2, 2006.0008-2006.0008 (2006).
- Claus, H., Frosch, M., Vogel, U. Identification of a hotspot for transformation of Neisseria meningitidis by shuttle mutagenesis using signature-tagged transposons. Mol Gen Genet. 259, 363-371 (1998).
- Hava, D. L., Camilli, A. Large-scale identification of serotype 4 Streptococcus pneumoniae virulence factors. Molecular microbiology. 45, 1389-1406 (2002).
- Costanzo, M. The Genetic Landscape of a Cell. Science. 327, 425-431 (2010).
- Tong, A. H. Systematic genetic analysis with ordered arrays of yeast deletion mutants. Science. 294, 2364-2368 (2001).
- Pan, X. A robust toolkit for functional profiling of the yeast genome. Molecular cell. 16, 487-496 (2004).
- Schuldiner, M. Exploration of the Function and Organization of the Yeast Early Secretory Pathway through an Epistatic Miniarray Profile. Cell. 123, 507-519 (2005).
- Deutschbauer, A. M. Mechanisms of haploinsufficiency revealed by genome-wide profiling in yeast. Genetics. 169, 1915-1925 (2005).
- Giaever, G. Chemogenomic profiling: identifying the functional interactions of small molecules in yeast. Proceedings of the National Academy of Sciences of the United States of America. 101, 793-798 (2004).
- Lum, P. Y. Discovering modes of action for therapeutic compounds using a genome-wide screen of yeast heterozygotes. Cell. 116, 121-137 (2004).
- Hillenmeyer, M. E. The chemical genomic portrait of yeast: uncovering a phenotype for all genes. Science. 320, 362-365 (2008).
- Oh, J. A universal TagModule collection for parallel genetic analysis of microorganisms. Nucleic acids research. 38, e146-e146 (2010).
- Oh, J. Gene annotation and drug target discovery in Candida albicans with a tagged transposon mutant collection. PLoS pathogens. 6, (2010).
- Nislow, C., Giaever, G., Stark, I., Stansfields, M. J. R. Chapter 387. Yeast Gene Analysis. , 387-414 (2007).
- Pierce, S. E., Davis, R. W., Nislow, C., Giaever, G. Genome-wide analysis of barcoded Saccharomyces cerevisiae gene-deletion mutants in pooled cultures. Nature protocols. 2, 2958-2974 (2007).
- Sambrook, J., Russell, D. W. . Molecular cloning : a laboratory manual. , (2001).
- Smith, A. M. Quantitative phenotyping via deep barcode sequencing. Genome Res. , (2009).
- Hamady, M., Walker, J. J., Harris, J. K., Gold, N. J., Knight, R. Error-correcting barcoded primers for pyrosequencing hundreds of samples in multiplex. Nature. 5, 235-237 (2008).
- Gresham, D. System-Level Analysis of Genes and Functions Affecting Survival During Nutrient Starvation in Saccharomyces cerevisiae. Genetics. 187, 299-317 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon