Method Article
A PCR-based metodo di genotipizzazione di distinguere tra le varietà selvatiche di tipo ornamentale e di Imperata cylindrica
In questo articolo
Riepilogo
Forniamo un protocollo conveniente e rapido genotipizzazione molecolare che impiega varietà-specifici primer PCR che bersaglio differenze di sequenza del DNA all'interno della regione cloroplasto trnL-F distanziatore per differenziare varietà di Imperata cylindrica (Cogongrass) che non può essere distinto da sola morfologia. Queste varietà sono l'erba federale elencati nocive, cogongrass ed affine, diffusa varietà ornamentale, I. cylindrica Var. Koenigii (Erba sangue giapponese).
Abstract
Wild-type I. cylindrica (cogongrass) è uno dei dieci peggiori piante invasive al mondo, un impatto negativo risorse agricole e naturali in 73 paesi diversi in tutta l'Africa, Asia, Europa, Nuova Zelanda, Oceania e Americhe 1-2. Cogongrass forma rapida diffusione, stand monodominant che dislocano una grande varietà di specie vegetali autoctone e, a sua volta minacciano gli animali autoctoni che dipendono dalle specie vegetali native per sfollati foraggio e riparo. Per aggiungere al problema, una varietà ornamentale [I. cylindrica var. koenigii (Retzius)] è ampiamente commercializzato con i nomi di 'Rubra' Imperata cylindrica, Red Baron, e l'erba del sangue giapponese (JBG). Questa varietà è putativamente sterile e non invasiva ed è considerato uno ornamentale auspicabile per le sue foglie di colore rosso. Tuttavia, in condizioni ottimali, JBG in grado di produrre semi vitali (Carol Holko, 2009 comunicazione personale) e può tornare a un verde iforma nvasive che è spesso indistinguibile da cogongrass in quanto assume le caratteristiche distintive della wild-type varietà invasivo 4 (Figura 1). Questo rende l'identificazione con morfologia un compito difficile anche per i ben addestrati tassonomi vegetali. Tendenza della JBG ad un fenotipo aggressivo verde non è un evento raro. Usando confronti di sequenze di codifica e regioni variabili del DNA, sia nucleare e dei cloroplasti, abbiamo confermato che la JBG è tornato alla invasiva verde all'interno degli Stati del Maryland, South Carolina e Missouri. JBG è stata venduta e piantati in stato di quasi tutti negli Stati Uniti continentali in cui non vi è una infestazione attiva cogongrass. L'entità del problema tornare in non ben compreso perché le piante sono tornati privi di documenti e spesso distrutti.
L'applicazione di questo protocollo molecolare fornisce un metodo per identificare JBG ritorna e può aiutare a mantenere queste varietà di co-occorrenti unoND possibilmente ibridare. Cogongrass è un outcrosser obbligato e, se incrociate con un genotipo diverso, in grado di produrre vento disperse vitale semi che si diffondono cogongrass su grandi distanze 5-7. JBG ha un genotipo leggermente diversa cogongrass e può essere in grado di formare ibridi vitali con cogongrass. Per aggiungere al problema, JBG è più freddo e tollera l 'ombra di cogongrass 8-10, e il flusso genico tra queste due varietà è in grado di generare ibridi che sono più aggressive, ombra tollerante e resistente freddo di wild-type cogongrass. Mentre wild-type cogongrass infesta attualmente oltre 490 milioni di ettari nel mondo, negli Stati Uniti sud-est che infesta più di 500.000 ettari ed è in grado di occupare la maggior parte degli Stati Uniti come si diffonde rapidamente verso nord a causa della sua ampia nicchia geografica e potenzialità 3,7,11. Il potenziale di un incrocio genetico è una seria preoccupazione per l'USDA-APHIS programma settimanale federale nocivi. Attualmente, l'USDA-APHIS vieta JBG negli Stati whprima che ci sono maggiori infestazioni cogongrass (ad esempio, Florida, Alabama, Mississippi). Tuttavia, impedendo ai due varietà di combinare può rivelarsi più difficile in quanto cogongrass e JBG espandere le loro distribuzioni. Inoltre, la distribuzione della JBG ripristinare è attualmente sconosciuto e senza la capacità di identificare questi varietà attraverso morfologia, alcuni infestazioni cogongrass può essere il risultato di JBG ritorna. Sfortunatamente, gli attuali metodi molecolari di identificazione tipicamente si basano su AFLP (Polimorfismi dei frammenti amplificati) e sequenziamento del DNA, che sono entrambi tempo e costoso. Qui vi presentiamo il primo costo-efficace e affidabile PCR-based metodo molecolare genotipizzazione di distinguere con precisione tra cogongrass e JBG ripristinare.
Protocollo
1. Raccolta e Conservazione
Questo metodo è stato sviluppato e testato utilizzando freschi, congelati e tessuti fogliari di recente secchi.
- Identificare cogongrass e / o JBG tessuti con l'aiuto di un tassonomista specializzata nell'identificazione delle specie erba. Con le sue foglie di colore rosso vivo, JBG ornamentale è facile distinguere visivamente da wild-type cogongrass e JBG tornare, tuttavia, cogongrass e JBG tornare sono quasi indistinguibili gli uni dagli altri. Cogongrass e il fenotipo JBG hanno ripristinato verdi, foglie, rizomi molto più grandi e più a lungo, e l'area fogliare più di JBG 12 (Figura 1).
- Tessuto fogliare Fresh offre il DNA qualità più abbondante e più alto e può essere raccolto dal campo o serra cresciuta I. cylindrica piante. Se il tessuto fresco deve essere utilizzato, estrarre il DNA entro 3 ore dalla raccolta per aiutare a prevenire il degrado. Altrimenti, preparare il tessuto per lo stoccaggio, keeping il tessuto fresco e dalla luce solare diretta.
- Per memorizzare i tessuti di estrazione del DNA in una data successiva, il metodo più ottimale è di congelare il tessuto e conservare a -80 ° C immediatamente. Non permettere che il tessuto congelato a scongelare prima dell'estrazione del DNA. Trasferire il tessuto congelato in azoto liquido prima delle fasi di macinazione di estrazione del DNA per evitare lo scongelamento.
- Se un congelatore a -80 ° C non è disponibile, asciutto il tessuto immediatamente. Per lo stoccaggio secco, posizionare il tessuto in una busta di carta e memorizzare la busta in gel di silice disidratati o altri materiali essiccanti attivi a temperatura ambiente. Una piccola quantità di silice indicatore misto a non indica silice garantirà che la silice è completamente disidratato e adatto essiccazione tessuti vegetali.
- Utilizzare almeno 10 volte superiore a gel di silice tessuto della foglia fresca in peso. Tessuto vegetale deve asciugare entro 24 ore. La qualità e la quantità di DNA viene ridotto mediante stoccaggio a secco nel tempo (come nel caso del campione erbarios).
2. Estrazione del DNA
Per estrarre il DNA da tessuto vegetale, seguire la DNeasy Plant Mini Kit (Qiagen, Valencia, CA; Cat # 69104 o 69106) le istruzioni del produttore con una modifica minore. Invece di utilizzare il suggerito meno di 100 mg di tessuto fresco o inferiore a 20 mg di tessuto secco, per ciascuna colonna, macinare superiore a 100 mg, e poi trasferire 100 mg di tessuto fresco o congelato (o> 20 mg di tessuto asciutto) idonee provette per l'estrazione. DNA nucleare e plastidio viene estratto simultaneamente.
- Prima di iniziare queste procedure, verificare che l'etanolo è stato aggiunto al buffer AP3 / E e AW.
- Macinare> 100 mg di tessuto fogliare fresche o congelate (o> 20 mg di tessuto fogliare secca) di una polvere fine con tre cicli di macinazione con azoto liquido in un mortaio e pestello refrigerati. Interruzione insufficiente del materiale di partenza o lisi insufficiente possono anche determinare una riduzione dei rendimenti di DNA. Con attenzione macinare il TISSue e non sovraccaricare le colonne con i tessuti troppo.
- Trasferire 100 mg di polvere congelata da tessuto fresco o congelato (o 20 mg di polvere secca da tessuto) in una provetta da 1,5 ml contenente 400 pl di tampone AP1 e 4 pl di RNasi A. Ogni tubo può essere collocato in un rack su un piccolo equilibrio per monitorare il peso corretto del tessuto per provetta.
- Campione Vortex o shake (s) per miscelare e incubare per 10 min a 65 ° C, invertendo il tubo (s) 2-3 volte durante l'incubazione.
- Aggiungere 130 ul di tampone AP2 a ciascun campione. Miscelare capovolgendo la provetta (s) più volte, e incubare per 5 min in ghiaccio.
- Pipettare ogni lisato in una colonna separata rotazione Mini QIAshredder, e posizionare ogni colonna in un tubo di raccolta 2 ml (fornito con il kit). Centrifuga colonna (s) per 2 min a 20000 g (circa 14000 rpm), e trasferire ogni flusso-through frazione in un nuovo tubo (non fornito con il kit) senza interrompere alcuna forma pellet.
- Aggiungere 1,5 volumi di Buffer AP3 /E, e mescolare pipettaggio.
- Trasferire 650 ul di miscela in una colonna DNeasy giro Mini in una provetta 2 ml di raccolta. Centrifuga colonna (s) per 1 min a 6.000 g (~ 8000 rpm), ed eliminare il flusso-through. Ripetere questo passaggio con l'impasto rimanente per ogni campione.
- Porre la colonna spin (s) in un nuovo tubo di raccolta 2 ml (s), e aggiungere 500 microlitri di Buffer AW alla parte superiore di ogni colonna. Centrifuga colonna (s) per 1 min a 6.000 xg (~ 8000 rpm), e scartare flusso passante.
- Aggiungere 500 pl di un tampone AW alla parte superiore di ciascuna colonna. Centrifugare per 2 min a 20000 g (circa 14000 rpm). Questo passaggio asciugare la colonna, eliminando così qualsiasi etanolo residua contenuta nel buffer che possono inibire PCR.
- Trasferire ogni colonna di selezione per una nuova provetta da microcentrifuga 1,5 ml. Aggiungere 100 pl AE Buffer all'inizio di ogni colonna di eluizione, e incubare colonna (s) per 5 min a temperatura ambiente. Centrifuga colonna (s) per 1 min a 6.000 xg (circa 8000 rpm) per raccogliere il DNA.
- Ripetere questi passaggi eluizione una volta, eluendo il DNA nella stessa provetta 1,5 ml per microcentrifuga per dare 200 pl di campione. I campioni di DNA Conservare a -20 ° C fino all'uso. Concentrazioni di DNA dipendono dal tipo di tessuto e le condizioni di conservazione. Rese ottimali si ottengono quando eluendo DNA con un totale di 200 pl di tampone AE, tuttavia, le concentrazioni può essere aumentato se volumi di eluizione vengono ridotte a un minimo di 50 pl.
3. Verifica della qualità e quantità del DNA
- Testare la qualità e la quantità di DNA estratto prima configurazione PCR utilizzando uno spettrofotometro o fluorometro e elettroforesi su gel. Ciò contribuirà a garantire il successo delle fasi successive.
- Utilizzando uno, spettrofotometro qualità e quantità del DNA di prova. Buone rese DNA dovrebbe essere tra 50 e 150 ng / ml con 260/280 e 230/280 rapporto vicino a 2,0. Come esempio, la Figura 2 mostra risultati di buona qualità utilizzando lo spettrofotometro NanoDrop (ThermoScientific, Wilmington, DE).
- Effettuare elettroforesi utilizzando uno standard di gel di agarosio all'1%. Verificare la presenza di bande relativamente grandi (> 10 Kb) senza striature di poco da contaminazione RNA (Figura 3).
- Se la concentrazione di DNA è alta, campioni di DNA diluiti a 70 ng / pl per le fasi successive.
4. Primer PCR
I primer PCR usati in questo protocollo si basano sulle differenze di sequenza tra il plastidio trnL-F regione spacer di genotipi cogongrass e JBG. Queste differenze si presentano sotto forma di SNP (polimorfismi a singolo nucleotide) e indels (Inserimenti ed eliminazioni) che hanno permesso lo sviluppo di varietà di primer specifici, individuando i primer nei siti di sequenze uniche (Figura 4).
- La qualità dei primer può avere un impatto significativo sui risultati PCR. Primer ordine da una società rispettabile. Ordiniamo i nostri primer da Oblique Bio, Inc. (:/ / Www.obliquebio.com/web/ "target =" _blank "> http://www.obliquebio.com/web/, Huntsville, AL), richiedendo solo procedure standard dissalazione.
- trnL-F primer di controllo positivi: Questa serie di primer amplifica il plastidio trnL-F regione distanziatore di taxa più erba 11-13 e serve come un buon controllo positivo (risultante in una banda di 890 bp).
Nome Sequenza TRNF (GAA)-F 5'-ATTTGAACTGGTGACACGAG-3 ' trnL (5 'esone)-C 5'-CGAAATCGGTAGACGCTACG-3 ' - Wild-type primers cogongrass: Questa serie di primer è specifico per cogongrass e non amplificare il trnL-F regione JBG genotipi. I risultati esposti in una band che è 595 bp.
Nome Sequence trnLF-C-F1 5'-TCCACTTTTTTGAAAAAACAAGTGCAA-3 ' trnLF-C-R1 5'-GCCGATACTCTAATAAATAAAAAAAAAAAAGAAAT-3 ' - JBG e JBG Revert primers: Questo set di primer specifico per JBG genotipo e non amplifica il trnL-F regione cogongrass. I risultati esposti in una band che è di 594 bp.
Nome Sequenza trnLF-R-F2 5'-CCAAATCCACTTTTTTGAAAAAACAAGTGGTT-3 ' trnLF-R-R2 5'-CGAGATTCCTTGCCGATACTCTAATAAAA-3 ' - Risospendere ciascun primer in un volume sufficiente di nucleasi libera ddh 2 O per ottenere una soluzione stock di 100 mm che possono essere conservati a -20 ° C, a lungo termine.
- Diluire ogni stock innesco a 12 mm prima delle operazioni di messa in PCR.
5. PCR Setup
Estrazioni DNA sono amplificate utilizzando ciascuna delle serie di primer sopra nelle reazioni PCR. Includere un controllo positivo per assicurare che tutti i reagenti PCR stanno lavorando bene e in grado di generare una band. Includere un controllo negativo per garantire che nessuno dei reagenti sono contaminati con il DNA indesiderato. Il controllo negativo contiene no-modello e dovrebbe portare a nessuna produzione band.
- Preparare tutte le reazioni a tubi con pareti sottili PCR per consentire il trasferimento del calore tra il blocco termociclatore e il campione. Si consiglia di utilizzare commercialmente disponibili aerosol privi di punte di pipetta per evitare la contaminazione.
- Per ciascun campione di DNA isolato, costituito da 50 pl reazioni PCR utilizzando ciascuno dei set di primer di cui sopra in 0,2 ml parete sottile provette PCR aggiungendo i seguenti reagenti on Ice nell'ordine elencato di seguito. Se più campioni vengono preparati, un cocktail contenente tutti i reagenti trannedello stampo di DNA a stabilire condizioni uniformi tra tutte le reazioni.
PCR Reagente Volume Usato Concentramento finale Priva di nucleasi ddh 2 O 40,5 pl Vantaggio 2 10X PCR Buffer (Clonetech, CA) 5,0 pl 10% (v / v) Advantage ultrapura PCR dNTP Mix (10 mm ciascuno, Clonetech, CA) 1,0 pl 0,2 mM Primer 1 (12μM in ddh 2 O) 1,0 pl 0.24 pM Primer 2 (12μM in ddh 2 O) 1,0 pl 0.24 pM Vantaggio 2 Mix polimerasi (Clonetech, CA) 0,5 pl 1% (v / v) Estrazione del DNA(70 ng / reazione, regolare il volume ddh 2 O, se necessario) 1,0 pl 1,4 ng / ul Totale: 50,0 pl - Per garantire che tutti i reagenti PCR stanno funzionando bene, impostare il controllo positivo utilizzando i primer di controllo positivo. Questo set di fondo funziona altrettanto bene per cogongrass, JBG, JBG ripristinare e altre erbe e si tradurrà in una band che è 890 bp.
- Impostare il controllo negativo utilizzando il primer di controllo stessa impostato come controllo positivo, utilizzando ddh 2 O invece l'estrazione del DNA. Se tutti i reagenti sono privi di DNA contamina, questa reazione comporta alcuna banda.
- Se la concentrazione di DNA è bassa, più DNA può essere aggiunto a ciascuna reazione, regolando la quantità di priva di nucleasi ddh 2 O utilizzato per portare il volume totale di reazione di 50 pl. Non utilizzare più di 5 pl di DNA (10% dei il volume totale) per reazione, come possibili impurità contenute nei campioni di DNA può inibire reazioni PCR.
6. PCR Ciclismo
- Effettuare amplificazioni di PCR in un termociclatore dotato di un coperchio riscaldato utilizzando i seguenti parametri di ciclismo PCR. Usiamo il Mastercycle pro S termociclatore (Eppendorf, Hauppauge, NY) impostato per operare con condizioni standard Ramping temperatura. Qualsiasi termociclatore qualità dovrebbe funzionare bene.
Ciclo Denaturazione di ricottura Polimerizzazione 1 2 minuti a 95 ° C 2 30 sec a 95 ° C 30 secondi a 61 ° C 90 sec a 68 ° C 35 cicli 3 5 min a 68 ° C Mantenere a 4 ° C fino a quando il campione viene rimosso - Ottimizzare le condizioni di PCR (inclusa la temperatura di ricottura innesco, i tempi di estensione, e il numero di cicli) come necessario in base alla qualità del DNA, primer, polimerasi Taq o tipo di termociclatore utilizzato. Si consiglia di utilizzare un termociclatore gradiente per determinare il grado ottimale ricottura temperature.
- Se il termociclatore utilizzata non ha un coperchio riscaldato, aggiungere 1 goccia di olio minerale all'inizio di ciascun campione per impedire l'evaporazione durante i cicli PCR.
7. Elettroforesi su gel dei prodotti PCR
Per visualizzare i risultati dell'analisi, separate prodotti di PCR su un gel di agarosio all'1% mediante elettroforesi standard.
- Combinare 2 microlitri di tampone standard carico DNA (tipicamente una soluzione 5x o 6x) con 5 pl di ciascun prodotto amplificato PCR.
- Caricare i campioni su un gel di agarosio 1% contenente EtBr (bromuro di etidio per la colorazione del DNA) e realizzato conither TAE o SB (borato di sodio) sistemi tampone 16. Usiamo 1 pl di una mg / ml 10 soluzione madre EtBr per 100 ml di 1% agarosio (0,1 mcg / ml).
- Eseguire campioni a ~ 120V finché il fronte del colorante raggiunge ¾ della lunghezza totale del gel.
- Sotto la luce UV (per esempio un breve box onda della luce UV), controllare le bande risultanti per vedere se un frammento del caso è stato amplificato.
- Documentare il gel e le fasce risultanti disponibili utilizzando un sistema di documentazione fotografica o una telecamera.
8. Risultati rappresentativi
Dopo visualizzazione dei prodotti PCR, cogongrass ha un modello unico bande rispetto a quella di JBG o ripristinato JBG (Figura 5). Per ciascun campione di DNA, il trnL-F positive set di primer di controllo dovrebbe sfociare in una singola ad alta intensità di banda a ~ 890 bp. Questo verifica che tutti i reagenti PCR stanno funzionando bene. Allo stesso modo, il controllo negativo (senza maschera) reazione dovrebbe contenere bande per ogni s innescoet utilizzato. Ciò verifica che nessuno dei reagenti sono stati contaminati.
Se il campione di DNA è derivato dal tipo selvatico cogongrass, una reazione di PCR utilizzando il cogongrass-specifica serie di primer si tradurrà in una singola banda ~ 595 bp, mentre i primer specifici JBG comporta alcuna banda. Analogamente, se il campione di DNA è derivata da JBG o ripristinato JBG, una reazione di PCR utilizzando il JBG-specifica serie di primer si tradurrà in una singola banda ~ 594 bp, mentre i primer specifici cogongrass comporta alcuna banda. Perché JBG, tornando JBG hanno identica sequenza di acido nucleico, avranno quindi hanno identici modelli di bande. Se molti campioni devono essere confrontati su un gel al tempo stesso, si consiglia di eseguire tutti i campioni derivati da ciascun primer uno accanto all'altro, rendendo così più facile per la scansione dei campioni di risultati positivi.
Differenze morfologiche tra JBG e JBG ritorna sono abbastanza evidenti (ad esempio il colore rosso delle foglie e piccoli di statura JB G contro la colorazione verde, più grande statura e la crescita aggressiva della JBG tornare), così mentre i risultati PCR sarà lo stesso, le varietà JBG sono facili da distinguere utilizzando morfologia delle piante.

Figura 1. Confronto tra effetto serra cresciuta Imperata cylindrica var. Koenigii (erba sangue giapponese), Annullate I. cylindrica var. koenigii (JBG Ripristina) e I. cylindrica (Wild-type cogongrass).
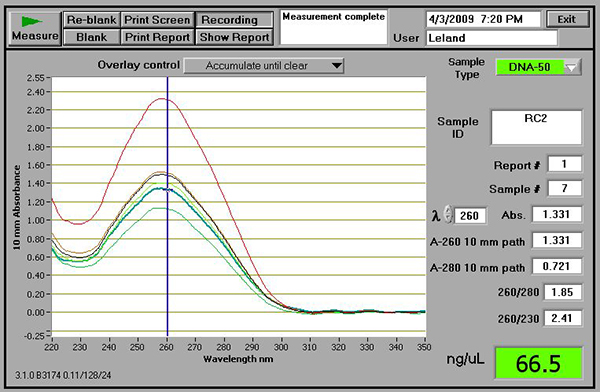
Figura 2. Un esempio di campioni di DNA verificate utilizzando uno spettrofotometro NanoDrop. Si noti che, indipendentemente dalla spettrofotometro utilizzata, il rapporto di 260/280 deve essere vicino a 1,8 e il 260/230 dovrebbe rapporto vicino a 2,0.
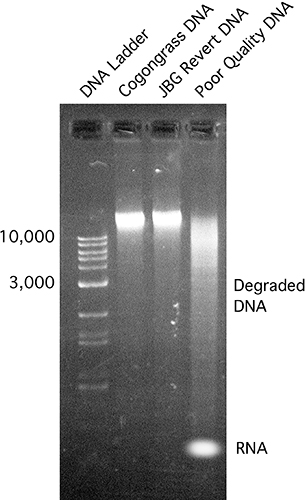
Campioni di DNA Figura 3. Verificata usando standard di gel elettroforesi su un gel di agarosio all'1%. Un marcatore del DNA commerciale è stato utilizzato per l'analisi dimensioni. Lane # 4 è un esempio di campione di DNA scarsa qualità, mostrando sbavature e qualche contaminazione RNA.
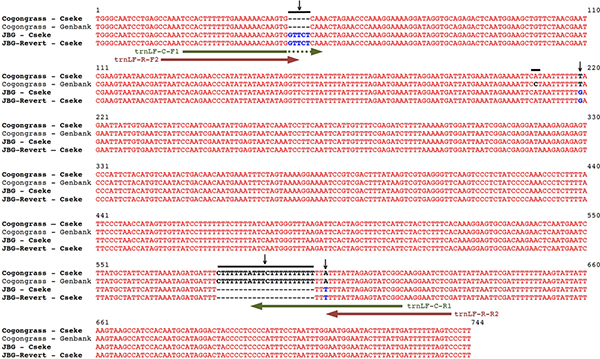
Allineamenti di sequenza Figura 4. Delle trnL-F regioni Imperata cylindrica var. Koenigii (erba sangue giapponese), Annullate I. cylindrica var. koenigii (JBG Ripristina) e I. cylindrica (Wild-type cogongrass). Verticali frecce nere indicano differenze nelle sequenze risultanti dal SNP e indels. Orizzontali frecce verdi indicano le posizioni delle wild-type primers cogongrass utilizzati per cogongrass-PCR specifica. Orizzontali frecce rosse indicano il Poquisizioni della JBG e JBG Ripristina primers utilizzati per JBG-PCR specifica.
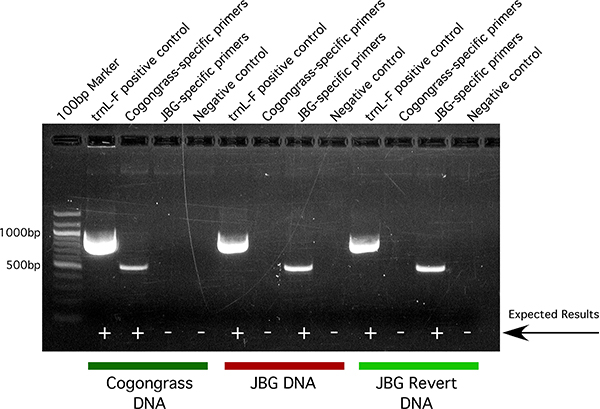
Figura 5. Risultato rappresentativa di elettroforesi su gel di prodotti di PCR derivati da cogongrass, JBG e JBG ripristinare campioni di DNA combinati con cogongrass-e JBG primer specifici nonché la trnL-F controllo positivo e un modello nessun controllo negativo.
Discussione
Il vivaio degli Stati Uniti e le industrie del paesaggio prosperano sulla coltivazione e la vendita di specie di piante esotiche e romanzo. Questo, unito alla crescente globalizzazione del commercio, aumenta le probabilità che una specie di piante invasive entreranno, stabilire e diffondere negli Stati Uniti la capacità di regolare federale tali impianti si basa su informazioni che spesso non è disponibile, compreso il potenziale per diventare invasive , tassonomia corretta e distinzione genetica taxa nativi e naturalizzati. Perché la nostra conoscenza delle piante invasive è spesso limitata, con piante importate nascosti caratteristiche invasive sono stati volontariamente introdotti solo per scoprire più tardi che invadono le nostre risorse agricole e naturali. Questo protocollo ha lo scopo di affrontare i problemi associati alla I. varietà cylindrica fornendo il primo metodo semplificato molecolare che può accuratamente distinguere tra wild-type e la forma cogongrass ritornò della sua controparte JBG ornamentali.
jove_content "> Per lo sviluppo di questo protocollo, wild-type cogongrass sono stati raccolti da popolazioni naturalizzate presso l'Unità di Pond Creek forestale di Santa Rosa nei pressi di Jay County, Florida, nel giugno del 2008. JBG è stato acquistato presso un vivaio commerciale (Bluebird Nursery, Inc ..) nel giugno 2008, nonché dalla collezione di una casa in Columbia, MO JBG torna sono stati ottenuti dal cortile del Museo Campbell Geological alla Clemson University, SC nel giugno 2008, presso l'Università Park a Riverdale, MA nel mese di giugno 2009; dal cortile di una casa in Columbia, MO nel 2009 (identificato da Leland Cseke). Tutte le piante sono state mantenute in una serra situata presso l'Università di Alabama a Huntsville (Huntsville, AL).Sequenziamento genetico di DNA raccolti da queste piante incluso il confronto in profondità di 9 regioni di DNA indipendenti comunemente usati per le piante di codici a barre 2. In tutti i casi, le sequenze di JBG erano una corrispondenza del 100% a quelle del JBG ripristinare, contribuendo così averificare che JBG effettivamente tornare ad un verde, forma invasiva. Solo il suo programma nucleare e le trnL-F regioni cloroplasti sono differenze che possono essere utilizzati per distinguere geneticamente tra cogongrass e JBG. La regione ITS ha un totale di 3 SNP (polimorfismi a singolo nucleotide) tra cogongrass e JBG, mentre il trnL-F regione ha due SNPs e 2 indels (inserimenti ed eliminazioni). Queste differenze genetiche ha permesso variety-PCR primer specifici da sviluppare in grado di distinguere tra wild-type cogongrass e JBG ritorna. I risultati più attendibili sono venuti da primers derivati dalla plastidio trnL-F regione. Così, questo protocollo si basa sulle differenze di sequenza tra le trnL-F regioni del genoma di cloroplasto cogongrass, JBG e tornati JBG (Figura 4).
Per contribuire a generare primers che sono più specifici per le varietà in questione e per evitare falsi positivi da specie strettamente correlate, All noti trnL-F sequenze di I. varietà cylindrica sono stati confrontati con trnL-F sequenze da specie erbacee legate (43 sequenze indipendenti provenienti da 29 specie, ad esempio, Cymbopogon citratus, i Sorghastrum incompletum e Coix lacryma-jobi, i Miscanthus sinensis e Saccharum officinarum e halepense Sorghum). Sebbene, abbiamo esaminato varietà specifiche sequenze di primer in 29 specie di erbe, la specificità dei vari primer specifici non è stato completamente esaminato per la sua capacità di amplificare il DNA dalla maggior parte delle specie erbacee altre. Di conseguenza, il tessuto usato per l'estrazione del DNA deve essere attentamente identificato come I. cylindrica prima di iniziare questo protocollo. Se l'erba non può essere identificato sia come cogongrass o JBG, allora vi consigliamo il sequenziamento del prodotto di PCR per assicurarsi che le sequenze sono una corrispondenza esatta per cogongrass o JBG. Attualmente, il metodo più accurato per verificare l'identità di un campione erba è dato effettuare PCR sia sul tRNL-F e le sue regioni, seguita dalla verifica della sequenza dei prodotti di PCR e confronto delle sequenze a sequenze note da taxa identificato con precisione. Il DNA può essere amplificato utilizzando primer di controllo descritte in questo protocollo (per il trnL-F regione) o inneschi altre disponibili in altre pubblicazioni 13-15. Sequencing è molto più lavoro e costosa rispetto all'utilizzo di nostra procedura semplificata.
La qualità dei primer utilizzati per la PCR è fondamentale per il successo della procedura. Abbiamo fatto i primer per questa procedura disponibile presso Oblique Bio, Inc. ( http://www.obliquebio.com/web/ , Huntsville, AL). Il vantaggio di ordinare i primer da Bio Oblique è quella di immagazzinare un gran numero di aliquote di ciascun primer dalla serie identiche di produzione del campione come primer che abbiamo usato per l'ottimizzazione nel nostro laboratorio. Di conseguenza, non solo inneschi hanno la stessa sequenza, ma they provengono dalla partita esattamente stessa produzione che è stato utilizzato in questo protocollo. Usando primer dallo stesso lotto, può evitare variabili estranee nel procedimento che può derivare da differenze nella qualità di primer PCR. Analogamente, mentre altre polimerasi Taq dovrebbe funzionare bene per PCR, la qualità della polimerasi Taq utilizzato avranno un impatto sulla qualità dei risultati di PCR. Per consentire una migliore coerenza nella reagenti PCR, abbiamo ottimizzato il protocollo utilizzando reagenti da Clontech. Il 2 polimerasi Advantage (Clontech, Mountain View, CA, Cat # 639201 o 639202) è una miscela di un robusto, hot-start Taq polimerasi e una prova di lettura enzima che aiuta a fornire una elevata specificità e amplificazioni più accurate.
Poiché questo protocollo si basa sul DNA dei cloroplasti, che è ereditato dalla madre in erba, eventi di ibridazione tra cogongrass e genotipi JBG non possono essere catturate con la nostra procedura di identificazione molecolare. Nei casi in cui è hypridization suspected, si consiglia di utilizzare le regioni nucleari che vengono ereditati da entrambi i genitori. Il più comunemente usato non plastidio regione variabile da considerare nella genotipizzazione impianto è la centrale nucleare ribosomale ITS regione 13-15,17. Attualmente, stiamo facendo progressi verso il multiplexing l'amplificazione della regione cloroplasto trnL-F con quello della centrale nucleare di regione ITS nella stessa provetta PCR. Multiplexing plastidio con le regioni del DNA nucleare potrebbe potenzialmente aggirare le limitazioni di utilizzo sia da solo, tuttavia, tali metodi richiedono l'ottimizzazione e la valutazione aggiuntivo per determinare la fattibilità caso per caso. L'uso di quantitative real-time PCR (qPCR) e più recenti tecnologie, quali gli apparecchi di sonde molecolari (primer fluorescenti), sono anche in corso di valutazione come metodi di piante fail-safe e accurate di genotipizzazione.
Il protocollo qui presentata fornisce un approccio veloce e affidabile per distinguere la JBG tornare da quella di wild-type cogongrass. Incoraggiamo utilizzarers di questo protocollo a contattarci per riportare i risultati derivanti dall'utilizzo di questo protocollo. Tali informazioni condivise contribuirà a fornire informazioni sulla distribuzione della JBG ritorna. Questo aiuterà anche regolatori a USDA prendere decisioni informate sulle azioni che possono essere necessari per aggirare la diffusione e l'ibridazione potenzialità delle varietà altamente invasive cogongrass.
Divulgazioni
Non abbiamo nulla da rivelare.
Riconoscimenti
Ringraziamo Alan Tasker (USDA-APHIS, Riverdale, MD), Stephen Compton (Clemson), Sherry Aultman (Clemson), Craig Ramsey (USDA-APHIS, Fort Collins, CO), e Betty Marose (UMD) per l'assistenza per l'ottenimento campioni . Ringraziamo gli studenti Andrew Adrian (UA-Huntsville) e Derek Thacker (UA-Huntsville) per la loro assistenza nel testare questo protocollo, e Giuseppe Herdy per il suo lavoro nelle riprese del video. Questo lavoro è stato finanziato dal National Fish e Wildlife Foundation.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reattivo | Azienda | Numero di catalogo | Comments |
| DNeasy Plant Mini Kit | Qiagen, Valencia, CA | 69104 o 69106 | Qualsiasi reputazione impianto kit DNA genomico che produce DNA di buona qualità dovrebbe funzionare bene per queste procedure. |
| TRNF (GAA)-F | Oblique Bio, Inc. | 3-0578 | Controllo primer forward positivo |
| trnL (5 'esone)-C | Oblique Bio, Inc. | 3-0579 | Primer reverse di controllo positivo |
| trnLF-C-F1 | Oblique Bio, Inc. | 3-0864 | Forward wild-type cogongrass fondo |
| trnLF-C-R1 | Oblique Bio, Inc. | 3-0865 | Invertirewild-type cogongrass fondo |
| trnLF-R-F2 | Oblique Bio, Inc. | 3-0866 | Forward JBG e JBG tornare fondo |
| trnLF-R-R2 | Oblique Bio, Inc. | 3-0867 | Reverse JBG e JBG tornare fondo |
| Advantage ultrapura PCR dNTP Mix | Clontech, Mountain View, CA | 639125 | |
| Vantaggio 2 polimerasi | Clontech, Mountain View, CA | 639201 o 639202 | Una buona correzione di bozze, hot-start Taq polimerasi |
Riferimenti
- Holm, L. G., Pancho, J. V., Herberger, J. P., Plucknett, D. L. . A Geographical Atlas of World Weeds. , (1979).
- CABI, Crop Protection Compendium. Commonwealth Agricultural Bureau International (CABI). , (2007).
- MacDonald, G. E. Cogongrass (Imperata cylindrica) - Biology, ecology, and management. Critical Reviews in Plant Sciences. 23, 367-380 (2004).
- Talley, S. M., Cseke, L. J., Zink, R. Molecular diagnostic technologies for invasive plants. CPHST Fort Collins Laboratory 2009 Annual Report. , 27-28 (2009).
- Dozier, H., Gaffney, J. F., McDonald, S. -. K., Johnson, E. R. R. L., Shilling, D. G. Cogongrass in the United States: History, ecology, impacts, and management. Weed Technology. 12, 737-743 (1998).
- Chikoye, D., Ekeleme, F. Weed flora and soil seedbanks in fields dominated by Imperata cylindrica in the moist savannah of West Africa. Weed Research. 41, 475-490 (2001).
- Weber, E. . Invasive Plant Species of the World: A Reference Guide to Environmental Weeds. , (2003).
- Patterson, D. T. Shading effects on growth and partitioning of plant biomass in Cogongrass (Imperata cylindrica) from shaded and exposed habitats. Weed Science. 28, 735-740 (1980).
- Cole, J. T., Cole, J. C. Ornamental grass growth response to three shade intensities. Journal of Environmental Horticulture. 18, 18-22 (2000).
- Bryson, C. T., Koger, C. H., Byrd, J. D. Effects of temperature and exposure period to heat on cogongrass (Imperata cylindrica) viability. Weed Technology. 21, 141-144 (2007).
- Capo-chichi, L. J. A., Faircloth, W. H., Williamson, A. G., Patterson, M. G., Miller, J. H., van Santen, E. Invasion Dynamics and Genotypic Diversity of Cogongrass (Imperata cylindrica) at the Point of Introduction in the Southeastern United States. Invasive Plant Science and Management. 1 (2), 133-141 (2008).
- Talley, S. M., Ramsey, C. L., Zink, R. Experimentally assessing the invasive potential of plants. CPHST Laboratory 2009 Annual Report. , 29-30 (2009).
- Hodkinson, T. R., Chase, M. W., Lledó, M. D., Salamin, N., Renvoize, S. A. Phylogenetics of Miscanthus, Saccharum and related genera (Saccharinae, Andropogoneae, Poaceae) based on DNA sequences from ITS nuclear ribosomal DNA and plastid trnLintron and trnL-F intergenic spacers. J. Plant Res. 115, 381-392 (2002).
- Kress, W. J., Wurdack, K. J., Zimmer, E. A., Weigt, L. A., Janzen, D. H. Use of DNA barcodes to identify flowering plants. Proc. Natl. Acad. Sci. U.S.A. 102 (23), 8369-8374 (2005).
- Roodt-Wilding, R., Spies, J. J. Phylogenetic relationships in southern African chloridoid grasses (Poaceae) based on nuclear and chloroplast sequence data. Systematics and Biodiversity. 4, 401-415 (2006).
- Brody, J. R., Kern, S. E. Sodium boric acid: a Tris-free, cooler conductive medium for DNA electrophoresis. BioTechniques. 36, 214-216 (2004).
- Chou, C. -. H., Tsai, C. C. Genetic variation in the intergenic spacer of ribosomal DNA of Imperata cylindrica (L.) Beauv. var. major (Cogongrass) populations in Taiwan. Botanical Bulletin of Academia Sinica (Taipei). 40, 319-332 (1999).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon