È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Rilevazione multiplex di batteri in campioni clinici complessi ed Ambientali con oligonucleotidi accoppiati microsfere fluorescenti
In questo articolo
Riepilogo
Descriviamo un metodo multiplex per la rilevazione di microrganismi in un campione con oligonucleotide ad accoppiamento perline fluorescenti. Amplificato da tutti gli organismi in un campione è ibridato ad un pannello di sonda-perline accoppiate. Uno strumento Luminex o Bio-Plex è usato per interrogare ogni perla per il tipo di perline e segnale di ibridazione.
Abstract
Vaginosi batterica (BV) è una sindrome polimicrobica ricorrente che è caratterizzata da un cambiamento nel microbiota "normale" da Lactobacillus dominato ad un microbiota dominato da un numero di specie batteriche, tra cui Gardnerella vaginalis, Atopobium vaginae, e altri 1-3. Questa condizione è associata a una serie di esiti negativi per la salute, compreso l'HIV acquisizione 4, e può essere difficile da gestire clinicamente 5. Inoltre, la diagnosi di BV ha invocato l'uso della colorazione di Gram di strisci tampone vaginale che sono incise su vari criteri numerici 6,7. Anche se questa diagnosi è semplice, economico e adatto a risorse limitate, può soffrire di problemi legati ad interpretazioni soggettive e non dà un profilo dettagliato della composizione del microbiota vaginale 8. I recenti sforzi di sequenziamento profondo hanno rivelato una ricca e diversificata flora batterica vaginale, conchiare differenze tra campioni prelevati da individui che sono diagnosticati con BV rispetto a quegli individui che sono considerati normali 9,10, che ha portato alla identificazione di un numero di potenziali bersagli per la diagnosi molecolare di BV 11,12. Questi studi hanno fornito una grande quantità di informazioni utili, ma sequenziamento profondo non è ancora pratico come metodo diagnostico in ambito clinico. Abbiamo recentemente descritto un metodo rapido per il profiling del microbiota vaginale in un formato multiplex con oligonucleotidi accoppiati perline fluorescenti con rilevamento su una piattaforma Luminex 13. Questo metodo, come la corrente colorazione di Gram metodi basati, è rapido e semplice, ma aggiunge l'ulteriore vantaggio di sfruttare le conoscenze molecolari derivanti da studi di sequenziamento nella progettazione della sonda. Questo metodo fornisce quindi un modo per profilo i microrganismi più importanti che sono presenti in un tampone vaginale che può essere utilizzato per diagnosticare BV con elevata specificità e sensibilità compared a macchia Gram fornendo ulteriori informazioni sulla presenza e abbondanza delle specie in modo semi-quantitativo e rapida. Questo metodo multiplex è espandibile ben oltre la portata della corrente analisi PCR quantitativa per gli organismi particolari, che attualmente è limitato a 5 o 6 test differenti in un singolo campione 14. È importante sottolineare che il metodo non si limita alla rilevazione di batteri nei tamponi vaginali e può essere facilmente adattato al profilo rapidamente quasi tutte le comunità microbica di interesse. Per esempio, abbiamo recentemente iniziato ad applicare questa metodologia per lo sviluppo di strumenti diagnostici per l'impiego in impianti di trattamento delle acque reflue.
Protocollo
Questo metodo è stato utilizzato nella ricerca riportata in Dumonceaux et al. J. Clin. . Microbiol 47, 4067-4077, doi: 10.1128/jcm.00112-09 (2009).
Un diagramma schematico raffigurante la procedura generale è presentato nella figura 1.
1. Perlina di accoppiamento
Questo descrive i metodi da utilizzare per l'accoppiamento sonde oligonucleotidiche al polistirolo perle di Luminex (vedi tabella 2). I volumi sono leggermente adattati per la valutazione delle sonde di cattura nuovo in prova, questi volumi sono indicati tra parentesi.
- Rimuovere 1-etil-3-(3-dimethylamiopropyl) carbodiimmide HCl (EDC) in polvere da -20 ° C e caldo essiccatore a temperatura ambiente.
- Risospendere le microsfere da sonciation in un sonicatore bagnomaria per 20 secondi, quindi vortex circa 20 secondi.
- Trasferire 400 microlitri (100 microlitri) di microsfere di una provetta Eppendorf (ciò corrisponde a 5x10 6 o 1.25x10 6 microsfere). Luminex consiglia l'utilizzo di tubi in microcentrifuga a basso legame con le proteine (per esempio tubi Eppendorf LoBind proteine, numero di catalogo 0030 108,094) per evitare che le perle non accoppiato di attaccarsi ai tubi e interferendo con recupero tallone. Non abbiamo trovato che questo è un problema utilizzando le normali tubi da microcentrifuga in polipropilene, che si utilizzano in genere (ad esempio, numero di catalogo VWR 87003-298).
- Pellet a 14000 xg per 1 minuto. Rimuovere e scartare il surnatante.
- Risospendere le microsfere in 50 microlitri (12,5 mL) di temperatura ambiente 0,1 M 2 - (N-Morpholino) etano acido (MES) pH 4.5.
- Preparare una soluzione fresca di EDC a 10 mg / ml in acqua.
- Preparare meno di 1 ml di questa soluzione pesando 5-10 mg di EDC su una bilancia analitica, poi l'aggiunta di acqua a 10 mg / ml.
- Aggiungere 1 nmol di 5'-amino oligonucleotide catturare C12-modificato (Tabella 2) a microsfere e mescolare nel vortex. 1 nmol è di 5 ml di 200 mM oligonucleotide.
- Aggiungere 2,5 ml di soluzione fresca EDC per le microsfere e mescolare nel vortex per 5 secondi.
- Incubare a temperatura ambiente per 30 minuti al buio.
- Scartare la soluzione EDC (passo 1,6) e preparare un nuovo campione dei 10 mg / ml EDC in acqua come sopra (passo 1,6).
- Aggiungere altri 2,5 ml di soluzione fresca EDC per le microsfere e vortex per 5 secondi.
- Incubare a temperatura ambiente per 30 minuti al buio.
- Lavare le perle con l'aggiunta di 1 ml di 0,02% di Tween 20. Vortex (opzionale ultrasuoni per 20 secondi) in modo da risospendere le sfere.
- Centrifugare 14000 xg 1 minuto. Rimuovere e scartare il surnatante.
- Lavare le perle di nuovo con l'aggiunta di 1 ml di solfato di sodio 0,1% dodecil (SDS). Vortex (opzionale ultrasuoni per 20 secondi) in modo da risospendere le sfere.
- Centrifugare 14000 xg 1 minuto. Rimuovere e scartare il surnatante.
- Risospendere le sfere in 100 l (25 mL) di Tris-EDTA (TE) buffer [10 mM Tris-Cl pH 8.0, 1 mM EDTA, pH 8.0].
- Enumerare le perle in un emocitometro o contatore Coulter per determinare la concentrazione stock.
- Preparare un Mix microsfere Maestro diluendo ogni perla ad una concentrazione finale di 100 perline / mL in TE buffer. Piscina accoppiato perline corrispondente al complesso desiderata del test (ad esempio per un 10-plex test, mix 10 perle diverse unita ad una concentrazione finale di 100/μl di ogni perla).
- Conservare il Mix microsfere Maestro a 4 ° C al buio. L'impasto può essere conservato per mesi se mantenuto in queste condizioni.
2. Chaperonine 60 universale destinazione (cpn60 UT) amplicone produzione e generazione di singoli filamenti.
- Generare la reazione a catena della polimerasi (PCR) del prodotto per ogni campione. Includere le fosforotioato-e impostare fondo biotina modificato 5 '(Tabella 1). Vedere la Tabella 3 per i volumi per miscelare e concentrazioni.
- Subito dopo la PCR è completa, aggiungete 2 microlitri (20 unità) di T7 exonuclease a ciascuna provetta PCR (PCR buffer sarà sufficiente per la reazione T7). Incubare la reazione a temperatura ambiente (~ 22-25 ° C) per 40 minuti.
- Al termine di questa incubazione, aggiungere 12,5 ml di 0,5 M etilene diammina (EDTA) pH 8,0 e mescolare. Questo dà un totale di ~ 64.5μl del singolo filamento prodotto della PCR.
3. Ibridazione di singolo filamento prodotto PCR di oligonucleotidi ad accoppiamento perle di polistirene.
- Preriscaldare lo strumento a 60 ° C per mantenere la temperatura di ibridazione durante l'analisi. Accendere il riscaldatore piattaforma utilizzando il software dello strumento e assicurarsi di utilizzare il blocco di riscaldamento in ottone che si inserisce la PCR-stile piatto che contiene la miscela ibridato tallone.
- Risospendere microsfere Master Mix (step 1,20) con una pipetta, una quantità appropriata dispensare in una provetta Eppendorf, tappo del tubo, e sonicare in un sonicatore bagnomaria per 2 minuti. In alternativa, per garantire la coerenza assoluta nel tallone resuspeESPANSIONE, la master mix microsfere possono essere risospese vortex e pipettaggio, poi sonicazione come sopra, poi dispensare la quantità desiderata in un tubo di polipropilene Eppendorf.
- Dispensare 17 ml di prodotto a singolo filamento PCR (punto 2.2) nei rispettivi pozzetti, di un basso profilo da 96 pozzetti Pozzetto piastra PCR (Tabella 2). Aggiungere 33 ml di risospeso, miscela sonciated perlina in ogni pozzetto. Coprire con coperchio in silicone (Tabella 2) e toccare delicatamente.
- Mettere la piastra nel termociclatore con un programma di: 95 ° C per 5 min, 60 ° C per 10 min, 60 ° C attesa, 60 ° C per 5 minuti, fine. Avviare il programma.
- Fai fresca streptavidina-ficoeritrina (SA-PE) soluzione; avrete bisogno di 25 microlitri per bene (fare diversi pozzi extra). Fai la SA-PE soluzione diluendo magazzino SA-PE (1 mg / ml) 1:50 a 20 mcg / ml con 1x cloruro tetrametilammonio (TMAC) buffer (3 M TMAC; Sarkosyl 0,1%; 50 mM Tris-HCl, pH 8,0 , 4 mM EDTA, pH 8,0).
- Quando il termociclatore arriva al 60 ° tenere passaggio C,aprire il coperchio, togliere il coperchio di silicone e aggiungere SA-PE soluzione direttamente a ciascun bene (non prendere la piastra dal termociclatore). Riposizionare il coperchio di silicone, chiudere il coperchio termociclatore e riprendere il programma.
- Quando il programma è completo, prendere il piatto fuori e velocemente trasferire alla macchina Bioplex da leggere. Piastra deve essere letta entro 10 minuti. Leggi a 60 ° C, in modo che il Bioplex preriscaldata a questa temperatura. Essere certi che l'altezza della sonda è stato adattato per ospitare la piastra utilizzato, come descritto nel manuale d'uso per lo strumento utilizzato.
- Per un accurato tallone e la rilevazione del segnale, le impostazioni cancello sulla Bioplex deve essere impostato in base al tipo di microsfere in uso. Perle di polistirene BioRad richiedono un'impostazione cancello di 4,335-10,000 mentre microsfere magnetiche richiedono un ambiente di 5.000-25.000. C'è anche la possibilità di utilizzare la piastra utilizzando l'impostazione Alta PMT che aumenta il valore di guadagno reporter. Ciò che possoncrease l'intensità dei segnali inferiore tuttavia è importante includere un controllo appropriato negativo come segnale di fondo sarà anche aumentata.
4. Rappresentante dei risultati:
Uno dei nostri obiettivi nello sviluppo e attuazione del presente saggio è stato quello di renderlo il più semplice e snella possibile. Abbiamo quindi ottimizzato la critica post-amplificazione fasi, compreso il tempo di trattamento esonucleasi T7 e il tempo di ibridazione al fine di sviluppare un metodo che può essere completato in un arco di tempo ragionevole. Come mostrato nella Figura 2A, il trattamento T7 dell'amplicone è essenziale per la generazione del segnale, come la maggior parte delle sonde avevano poco o nessun segnale con breve o nessun trattamento T7. Il segnale aumenta in modo lineare fino a circa 40 minuti, momento in cui l'aumento del segnale di rallentamento. Nessun degrado del segnale è stata osservata anche a 2 ore di tempo di trattamento T7, indicando che la modifica fosforotioati dei primer è altamente efficace nel prevenire Targedegrado filone t come descritto 15. Abbiamo scelto 40 minuti per tempo di trattamento T7 ridurre al minimo il tempo complessivo di protocollo, ma è chiaro dalla Figura 2A che il trattamento T7 può andare avanti per molto più tempo. Abbiamo anche determinato l'effetto del tempo sulla ibridazione generazione del segnale (Figura 2B) ed ha trovato che 10 minuti sono stati sufficienti per il massimo del segnale, in quanto non ulteriore aumento del segnale è stata osservata anche dopo 4 ore di ibridazione. Quindi, un passo ibridazione 10 minuti è stato scelto, ancora una volta per minimizzare il tempo complessivo di analisi. Con questi risultati in mente, e con tecniche rapide di estrazione del DNA come InstaGene (Bio-Rad), l'analisi globale, compresa l'estrazione del DNA, PCR, e analisi Luminex può essere completato in 4-5 ore.
Su una nota di cautela, il livello di aggregazione delle perle di polistirene durante una corsa o Luminex Bioplex può avere un impatto importante sulla efficacia del test. Il software Bioplex mostrerà il livello di aggregazione tallone, che si verifica quandopiù di una perla viene rilevato nel percorso laser e comporta l'esclusione del segnale di ibridazione dal aggregato. Apparente aggregazione tallone può anche essere causato da qualsiasi materiale particolato che non è la dimensione corretta per un unico tallone, e può anche essere causato da bolle d'aria. In uno di questi eventi, il segnale proveniente dal aggregato perlina, bolla d'aria, o particella viene scartato. Nella maggior parte dei casi, troviamo che l'aggregazione tallone è minimo (figura 3A) e il livello mirato di 100 eventi contare per ogni perlina è facilmente realizzabile. Occasionalmente, tuttavia, le perle mostrano un moderato (Figura 3B) o grave (Figura 3C) livello di aggregazione. In questi casi, poiché la maggior parte dei dati viene scartato, lo strumento potrebbe avere problemi a raggiungere 100 eventi per ogni tipo di perla e il risultato può essere discutibile. Il passo sonicazione (passo 3.2) ha lo scopo di minimizzare l'aggregazione tallone. Inoltre, l'archiviazione le perle come un mix diluire maestro microsfere (punto 1.20) può aiutare. Abbiamo notato che TMAC escludendo dalla hybritampone dization - aggiungerla alla SA-PE diluente invece (passo 3.5) - aiuta a minimizzare l'aggregazione tallone. Inoltre, mentre non abbiamo testato questo, le sfere magnetiche più recenti disponibili da Luminex o BioRad sono pensati per visualizzare meno di una tendenza ad aggregarsi.
I risultati dell'applicazione di un 5-complesso serie Luminex targeting G. vaginalis, A. vaginae, L. iners, L. crispatus, e L. gasseri con corrispondente Gram strisci colorati tampone vaginale sono mostrati in figura 4. Questi campioni sono stati prelevati da un singolo individuo in momenti diversi. Al tempo 0, l'individuo è stato diagnosticato un BV sulla base di colorazione di Gram (Figura 4A) ed i risultati Luminex da questo stesso campione (Figura 4B) mostrano che degli organismi rappresentati nella matrice, G. vaginalis è più diffusa, mentre A. vaginae e L. iners sono stati positivi. Nove giorni dopo, l'individuo era ancora BV positivo e il signal per G. vaginalis sono notevolmente aumentate, mentre A. vaginae e L. iners erano ancora positivi. È importante sottolineare che, dopo questo tempo l'individuo ha cominciato a transizione verso un microbiota normale come bacilli Gram positivi è diventato rilevabile nella strisci (Figura 4A) mentre il segnale di G. vaginalis svaniva ed il segnale per L. iners aumentato (Figura 4B). Con la nostra definizione originale di BV con questo metodo (campioni positivi per G. vaginalis e / o A. vaginae stati considerati positivi BV) 13, questo individuo era BV positivo a tutti i tempi, anche se la colorazione di Gram non è riuscito a rilevare G. vaginalis in questi ultimi due punti del tempo. Con il metodo descritto qui Luminex, le tendenze possono essere facilmente rispetto al sequenziamento metodi basati, ed i risultati del test confermano Luminex tipicamente colorazione di Gram 13, fornendo ulteriori informazioni sull'identità organismo e abbondanza.

Figura 1. Schema del protocollo Luminex per la determinazione del profilo microbiota di un campione clinico complesso o ambientali. Il protocollo inizia con il DNA modello che è stato estratto dal campione di interesse. (1) Generare prodotto di PCR da DNA stampo utilizzando filo-specifico biotinilato, fosforotioato modificati cpn60 UT primer PCR accoppiata con non modificato cpn60 UT primer PCR (Tabella 1), (2) Questo genera un pool di PCR prodotto che rappresenta il microbiota con la biotina- fosforotioato modifica su un filo, (3) Digest double-stranded prodotto PCR con esonucleasi T7, che non può degradare la fosforotioato modificato filo e genera quindi a singolo filamento di DNA che si modifica al 5 'con biotina, (4) Coppia perle di polistirene a specie-specifico cpn60 sonde UT - ogni perla ha un indirizzo univoco spettrale (indicata dal colore perla) che idistinguibili dal Luminex o Bio-Plex strumento s; (5) ibridare il singolo filamento prodotto di PCR generato dal campione di interesse per il suite di specie-specifico oligonucleotide accoppiato perle; (6) Aggiungere streptavidina-ficoeritrina coniugato che si lega alla il biotinilato a singolo filamento prodotto di PCR e agisce come un indicatore di ibridazione; (7) Determinare l'indirizzo spettrale (tallone d'identità) e l'intensità del segnale di ibridazione con un Luminex o Bio-Plex strumento. Almeno 100 perline vengono conteggiati per ciascuna identità tallone e l'intensità media di fluorescenza (MFI) del segnale ficoeritrina viene segnalato come l'uscita. Fino a 100 diversi tipi cordone possono essere valutate contemporaneamente, ma un saggio che mostra la discriminazione di tre tipi tallone è illustrata. (8) Quando il MFI di PCR replica generata dallo stesso campione è significativamente maggiore rispetto al controllo negativo per un dato tallone (una coda studente t-test, p <0,05), il campione è considerato positivo per tale organismo.
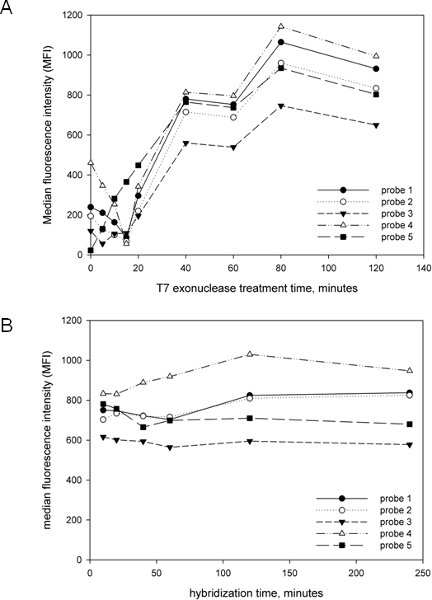
Figura 2. Ottimizzazione dei parametri di test Luminex. Cinque diverse sonde mirati per anaerobius Peptostreptococcus sono stati utilizzati con ampliconi generati da un modello misto composto plasmidi contenenti il clonato cpn60 UT di 20 batteri noti per essere associati con la vagina, tra cui P. anaerobius. (A). Effetto del tempo di trattamento esonucleasi T7. Il protocollo sopra descritto è stato seguito, ma il tempo di trattamento con esonucleasi T7 è stata variata prima di ibridazione e l'intensità media di fluorescenza (MFI) è stata determinata per tutte le sonde. (B). Effetto del tempo di ibridazione. Il protocollo sopra descritto è stato seguito con una varietà di tempi ibridazione. Gli stessi set di sonde sono state usate con un amplicon generato utilizzando lo stesso modello come in A e l'intensità media di fluorescenza (MFI) è stata determinata per tutte le sonde.
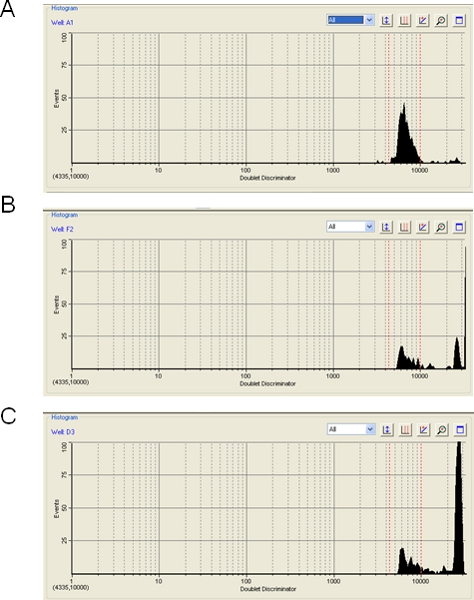
Figura 3. Determinazione di aggregazione tallone utilizzando il software Bioplex. Tre esempi sono dati di piste Bioplex in cui il livello del tallone aggregazione è (A), accettabile (5%), (B), borderline (50%) e (C) inaccettabili (80%).
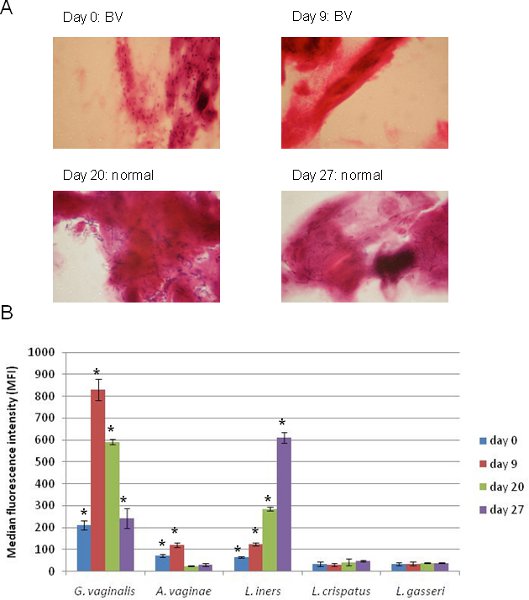
Figura 4. Applicazione di un 5-complesso serie Luminex alla diagnosi BV sequenziale in campioni prelevati da uno stesso individuo. (A). Gram-vetrini colorati che mostrano il tradizionale diagnostici utilizzati per valutare ogni campione di BV. (B). Applicazione di un 5-complesso serie Luminex preparata ed eseguita come descritto in questo protocollo per gli stessi quattro esemplari mostrati in (A). Obiettivi batterica IFM cui segnale è stato significativamente positivo per la nostra definizione (una coda studente t-test, p <0,05) sono contrassegnati da un asterisco (*).
Discussione
La specificità di generazione del segnale è di importanza critica, si deve essere sicuri che il segnale osservato rispecchia in pieno la rilevazione degli ampliconi generati da tale organismo. Software come PrimerPlex (Premier Biosoft) può aiutare a progettare delle sonde che ibridano in modo efficiente, ma può o non può cross-ibridare a specie non bersaglio. Quando universale primer PCR sono utilizzati come descritto in questo protocollo, è importante tenere a mente che l'ha generato amplicone rappresenta tut...
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Ringraziamo Alberto Severini e Vanessa Goleski per facilitare lo sviluppo di analisi e commenti critici su questo manoscritto. Questo lavoro è stato finanziato dall'Agenzia di sanità pubblica del Canada e degli Industrial Research Assistance Program (National Research Council of Canada). Ulteriore supporto è stato ottenuto presso l'Università di Saskatchewan Fondo di pubblicazione.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome oligonucleotide | Azienda | Sequenza 1 | |
| H279BP | Invitrogen, IDT, o altri | Biotina-OEFO GAIIIIGCIGGIGAYGGIACIACIAC | |
| H280 | YKIYKITCICCRAAICCIGGIGCYTT | ||
| H1612BP | Biotina-OEFO GAIIIIGCIGGYGACGGYACSACSAC | ||
| H1613 | CGRCGRTCRCCGAAGCCSGGIGCCTT | ||
| 1O, fosforotioato-C, E, fosforotioato-G, F, fosforotioato-A, io, inosina, Y, C o T, R, A o G, K, T, G, S, C o G. | |||
Sequenze Tabella 1. Dei oligonucleotidi modificati per universale cpn60 PCR.
| Nome del reagente | Azienda | Numero di catalogo | Commenti |
| 1-etil-3-(3-dimethylamiopropyl) carbodiimmide HCl (EDC) | Perforare | 22980 | |
| Perline fluorescenti di polistirene: Microplex Microsfere (Luminex), o Bio-Plex COOH Bead (Bio-Rad) | Luminex o Bio-Rad | Bio-Rad: 171-506xxx dove xxx corrisponde al tallone identificatore | Biglie magnetiche sono sempre disponibili per l'accoppiamento oligonucleotidi e può offrire alcuni vantaggi. Non sono stati provati da questi autori. |
| Oligonucleotidi di cattura (5 'amino C12 modificata) | Invitrogen, IDT, o altri | varie | Livello di purezza dissalate è accettabile. Sequenze delle sonde di cattura utilizzati per caratterizzare i tamponi vaginali sono forniti nel manoscritto su cui si basa questo protocollo 13. |
| T7esonucleasi | New England Biolabs | M0263S | |
| Streptavidina-R-ficoeritrina (SA-PE) | Invitrogen | S-866 | Fare attenzione ad ottenere elevata purezza SA-PE, questo numero di catalogo è consigliato |
| Pozzetto 96 pozzetti PCR | Pescatore | CS006509 | rientrano in entrambe le termociclatore a 96 pozzetti e la macchina Bioplex |
| Pozzetto di tenuta tappeto | Pescatore | CS006555 | Possono essere riutilizzati; lavare con acqua e sapone, lavare bene e asciugare |
| TMAC 5M | Sigma | T3411 | |
| Bio-Plex o strumento Luminex | Bio-Rad o Luminex | Bio-Rad: 171-000201 | |
| Software per la progettazione PrimerPlex sonda | Premier Biosoft | www.premierbiosoft.com | Consigliato per Luminex sonda desegno, anche se piattaforme software possono essere utilizzati altri |
Tabella 2. Reagenti e attrezzature specifiche.
| componente | microlitri / saggio | saggi μl/100 | concentrazione finale |
| 10x PCR buffer (Invitrogen) | 5 | 500 | 1x |
| 50 mM MgCl 2 (Invitrogen) | 2,5 | 250 | 2,5 mM |
| 10 mM dNTP | 1 | 100 | 0,2 mm ciascuna |
| H279BP, 25 mM | 0,25 | 25 | 375 nm |
| H1612BP, 25 mM | 0,75 | 75 | 125 Nm |
| H280, 25 mM | 0,25 | 25 | 375 nm |
| H1613,25 mM | 0,75 | 75 | 125 Nm |
| Acqua | 34 | 3400 | --- |
| Totali | 44,5 | 4450 |
Tabella 3. Consigliati miscele per PCR con primer modificato cpn60 UT (Tabella 1). Il test è costituito da 5 ml di DNA stampo e 0,5 microlitri (2,5 U) di Taq DNA polimerasi (Invitrogen). Normalmente grandi volumi sono preparati (ad esempio, sufficiente per 100 test) e conservati a -20 ° C.
Riferimenti
- Hale, L. P., Swidsinski, A., Mendling, W. Bacteria associated with bacterial vaginosis. N. Engl. J. Med. 354, 202-203 (2006).
- Hay, P. Life in the littoral zone: lactobacilli losing the plot. Sex. Transm. Infect. 81, 100-102 (2005).
- Morris, M., Nicoll, A., Simms, I., Wilson, J., Catchpole, M. Bacterial vaginosis: a public health review. Brit. J. Obstet. Gynecol. 108, 439-450 (2001).
- Myer, L., Kuhn, L., Stein, Z. A., Wright, T. C., Denny, L. Intravaginal practices, bacterial vaginosis, and women's susceptibility to HIV infection: epidemiological evidence and biological mechanisms. Lancet. Infect. Dis. 5, 786-794 (2005).
- Wilson, J. Managing recurrent bacterial vaginosis. Sex. Transm. Infect. 80, 8-11 (2004).
- Nugent, R. P., Krohn, M. A., Hillier, S. L. Reliability of diagnosing bacterial vaginosis is improved by a standardized method of gram stain interpretation. J. Clin. Microbiol. 29, 297-301 (1991).
- Ison, C. A., Hay, P. E. Validation of a simplified grading of Gram stained vaginal smears for use in genitourinary medicine clinics. Sex. Transm. Infect. 78, 413-415 (2002).
- Money, D. The laboratory diagnosis of bacterial vaginosis. Can. J. Infect. Dis. Med. Microbiol. 16, 77-79 (2005).
- Schellenberg, J. Pyrosequencing of the chaperonin-60 universal target as a tool for determining microbial community composition. Appl. Environ. Microbiol. 75, 2889-2898 (2009).
- Spear, G. T. Comparison of the diversity of the vaginal microbiota in HIV-infected and HIV-uninfected women with or without bacterial vaginosis. J. Infect. Dis. 198, 1131-1140 (2008).
- Brotman, R. M., Ravel, J. Ready or not: the molecular diagnosis of bacterial vaginosis. Clin. Infect. Dis. 47, 44-46 (2008).
- Fredricks, D. N., Fiedler, T. L., Marrazzo, J. M. Molecular identification of bacteria associated with bacterial vaginosis. N. Engl. J. Med. 353, 1899-1911 (2005).
- Dumonceaux, T. J. Multiplex detection of bacteria associated with normal microbiota and with bacterial vaginosis in vaginal swabs by use of oligonucleotide-coupled fluorescent microspheres. J. Clin. Microbiol. 47, 4067-4077 (2009).
- Molenkamp, R., van der Ham, A., Schinkel, J., Beld, M. Simultaneous detection of five different DNA targets by real-time Taqman PCR using the Roche LightCycler480: Application in viral molecular diagnostics. J. Virol. Meth. 141, 205-211 (2007).
- Nikiforov, T. T., Rendle, R. B., Kotewicz, M. L., Rogers, Y. H. The use of phosphorothioate primers and exonuclease hydrolysis for the preparation of single-stranded PCR products and their detection by solid-phase hybridization. PCR. Methods. Appl. 3, 285-291 (1994).
- Hill, J. E., Penny, S. L., Crowell, K. G., Goh, S. H., Hemmingsen, S. M. cpnDB: A Chaperonin Sequence Database. Genome. Res. 14, 1669-1675 (2004).
- Bradshaw, C. S. The association of Atopobium vaginae and Gardnerella vaginalis with bacterial vaginosis and recurrence after oral metronidazole therapy. J. Infect. Dis. 194, 828-836 (2006).
- Dumonceaux, T. J. Enumeration of specific bacterial populations in complex intestinal communities using quantitative PCR based on the chaperonin-60 target. J. Microbiol. Meth. 64, 46-62 (2006).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon