È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Immunoprecipitazione della cromatina (ChIP) con Drosophila Tessuto
In questo articolo
Riepilogo
Recentemente high-throughput tecnologia di sequenziamento ha notevolmente aumentato la sensibilità di immunoprecipitazione della cromatina (ChIP) esperimento e ha spinto la sua applicazione con cellule purificate o tessuti sezionati. Qui delineare un metodo per usare la tecnica ChIP con Drosophila Tessuto, che può definire la condizione cromatina endogeno in un sistema ben caratterizzato biologico.
Abstract
L'epigenetica rimane un settore in rapida evoluzione che gli studi come lo stato della cromatina contribuisce alla espressione genica differenziale in diversi tipi di cellule a diversi stadi di sviluppo. Regolazione epigenetica contribuisce ad un ampio spettro di processi biologici, tra cui la differenziazione cellulare durante lo sviluppo embrionale e l'omeostasi in età adulta. Una strategia critico negli studi epigenetici è quello di esaminare come le varie modificazioni degli istoni e fattori di cromatina regolare l'espressione genica. Per risolvere questo problema, immunoprecipitazione della cromatina (ChIP) è ampiamente utilizzato per ottenere una fotografia della associazione di fattori particolari con DNA nelle cellule di interesse.
ChIP tecnica utilizza comunemente cellule coltivate come materiale di partenza, che può essere ottenuto in abbondanza e omogeneità per generare dati riproducibili. Tuttavia, vi sono diversi avvertimenti: Innanzitutto, l'ambiente far crescere le cellule in piastre di Petri è diversa da quella in vivo, così puònon rispecchiano lo stato endogena cromatina delle cellule in un organismo vivente. In secondo luogo, non tutti i tipi di cellule possono essere coltivate ex vivo. Ci sono solo un numero limitato di linee cellulari, da cui la gente può ottenere abbastanza materiale per l'analisi ChIP.
Qui si descrive un metodo per fare esperimenti ChIP utilizzando tessuti di Drosophila. Il materiale di partenza è sezionato da un tessuto animale vivente, quindi in grado di rispecchiare con precisione lo stato endogena cromatina. L'adattabilità di questo metodo con diversi tipi di tessuto permetterà ai ricercatori di affrontare molto più biologicamente questioni rilevanti in materia di regolazione epigenetica in vivo 1, 2. Combinando questo metodo con high-throughput sequencing (ChIP-seq) ulteriormente permetterà ai ricercatori di ottenere un paesaggio epigenomic.
Protocollo
(La procedura intero chip richiede circa due giorni. Preparazione di librerie di chip per alta sequenziamento richiede altri 2-3 giorni.)
1. Sezionare e prepara il tessuto per Experiment ChIP (circa 1 milione di cellule)
- Dissect tessuto di interesse (ad esempio, 200 paia di Drosophila testicoli) in PBS freddo inibitore della proteasi + 1x (sciogliere 1 pellet di cocktail inibitore della proteasi in 1,5 ml di PBS 1x a 7x ottenendo così la soluzione stock) + PMSF (concentrazione finale 100 pg / mL). Risciacquare tessuto doppio tessuto e risospendere in 200 pl della stessa soluzione di PBS con inibitori.
- Per risolvere il campione aggiungere 5,5 microlitri di formaldeide al 37% (formaldeide calda in bagno d'acqua a 37 ° C per 1 minuto prima di usare). Incubare a 37 ° C per 15 minuti, vortex ogni 5 minuti in mezzo.
- Mettere il campione sul ghiaccio per il tessuto riposare per 2 minuti. Sciacquare 2x con 450 microlitri di PBS (con inibitori della proteasi e PMSF). Esempio può essere ora negoziod a -20 ° C. Ripetere più volte dissezione per ottenere materiale sufficiente per ChIP.
2. Preparare supernatante con la proteina-DNA Coniugazione per l'analisi ChIP
- Combina campione abbastanza dal punto 1.3 in una provetta eppendorf 1,75 mL.
- Rimuovere dalla PBS campione di tessuto. Aggiungere 200 pl di tampone di lisi (50 mM Tris-HCl, pH 7,6, 1 mM CaCl2, 0,2% Triton X-100 o NP-40, 5 mM butirrato, e 1x cocktail inibitore di proteinasi). Aggiungi fresca soluzione madre PMSF al tampone di lisi (PMSF concentrazione finale di 0,5 mM). Omogeneizzare con il blu omogeneizzatore (Cat Fisher # 749521-1590) fino a quando i tessuti si dissociano completamente senza alcuna aggregazione, incubare a RT (temperatura ambiente) per 10 minuti.
- Sonicazione: microtip sonicatore uso (Misonix HS-XL200) alla potenza 20. Con ultrasuoni per 10 secondi e di riposo in ghiaccio per 50 sec. Ripetere 4-5 volte (tempo di sonicazione ottimale dovrebbe essere determinata empiricamente, come la maggior sonicazione produrrà frammenti più piccoli. Per ChIP con anticorpi contro Histonmodifiche elettroniche, che normalmente ultrasuoni cromatina di circa 200-300 bp; per il fattore di trascrizione o di regolatore di cromatina (ad esempio proteine Polycomb), che normalmente sonicare cromatina a 200-1000 bp. Tuttavia, la dimensione del frammento ottimale deve essere testato empiricamente. Nota: non rimuovere la provetta in ghiaccio ANCHE DURANTE sonicazione PER EVITARE ESEMPIO DA RISCALDAMENTO!
- Diluire il campione con l'aggiunta di 1,8 mL di tampone RIPA (10 mM Tris-HCl, pH 7,6, 1 mM EDTA, 0,1% SDS, 0,1% Na-desossicolato, 1% Triton X-100, con inibitori della proteasi PMSF e alla stessa concentrazione descritta in precedenza). Salvare 40 microlitri come ingresso di controllo sommando 2 microlitri 5M NaCl e incubare a 65 ° C per una notte (O / N) per invertire reticolazione.
- Per anticorpo coniugato a perline, aggiungere 40 ul Proteina A perline in una provetta eppendorf da 1,5 ml, quindi aggiungere 600 microlitri di PBS e roccia a 4 ° C per 2 minuti, perline applicano al magnete e rimuovere supernatante. Aggiungere 100 pl di PBS e anticorpo di interesse (quantità di anticorpo dovrebbe essere determinata empirismocamente). Incubare a RT per 1 ora o 4 ° C per 4 ore.
- Rimuovere il surnatante da perline di magnete e aggiungere 1 ml di estratto di cromatina dal passo 2,4 ai talloni. Ruota a 4 ° CO / N.
- Applicare il campione ChIP al magnete e rimuovere il surnatante. Lavare le perline con il seguente tampone a 4 ° C per 10 minuti ciascuno:
2x con 1 ml di tampone RIPA [1,89 mL 'tampone RIPA' + 315 pl inibitori della proteasi 7x + 20 pl PMSF];
2x con 1 ml di tampone RIPA + 0,3 M NaCl [1,89 mL di tampone RIPA + 220 microlitri 3 M NaCl];
2x con 1 mL di tampone LiCl (0,25 M LiCl, 0,5% NP40, 0,5% NaDOC);
1x con 1 ml di TE 1x + 0,2% Triton X-100;
1x con 1 ml di TE 1x. - Per invertire reticolazione, risospendere le sfere in 100 microlitri tampone TE + 3 microlitri 10% SDS + 5 pl di proteinasi K (20 mg / mL). Incubare a 65 ° CO / N.
- Applicare perline al magnete e trasferire supernatante in un nuovo tubo. Il supernatante CONTIENE IL CAMPIONE DNA. Lavare perline con 100 microlitri TE + 0,5 MNaCl e combinare le due surnatante.
- Fenolo-cloroformio estrarre il campione di DNA. Aggiungere 200 ul Fenolo: Chlorofrom: IAA (25:24:1), mescolare e vortex. Spin a 14k per 5 minuti a RT. In alternativa, il DNA può essere estratto usando il kit di purificazione PCR Qiagen.
- Trasferire il surnatante / acquoso in una nuova provetta, e aggiungere acrilammide lineare ad una concentrazione finale di 20 ug / mL (In alternativa, 1 pl di glicogeno a 20 mg / ml per ogni 1 ml di surnatante possono anche essere utilizzati. Glicogeno viene utilizzato per qPCR successivo o analisi PCR, mentre acrilammide lineare viene utilizzato per il sequenziamento saggi.) Aggiungere 20 microlitri di 3M NaOAc e 500 microlitri EtOH 100%. Mescolare bene ed incubare a 80 ° C per 10 minuti. Centrifuga alla massima velocità per 20 minuti a 4 ° C.
- Rimuovere il surnatante e lavare con 300 microlitri EtOH 70%. Aria campione secco e risospendere in 50 microlitri tampone TE. Esempio possono essere conservati a -20 ° C. Esempio può essere usato per la PCR quantitativa (qPCR) test (Figura 1).
3. Analizzare ed ChIP-DNA
3 bis. Analizzare ChIP-DNA ed utilizzando qPCR
- Diluire DNA genomico al seguente concentrazione di una curva standard: non diluito, 1/10, 1/100, 1/1000, 1/5000.
- Per ogni coppia di primer, istituito qPCR in duplice esemplare in una piastra a 96 pozzetti con il DNA genomico da 3a.1 passo, il controllo degli ingressi e ChIP-ed DNA:
10 L 2x PCR SYBR mix (Fermentas, K0222);
1 ul 10 uM innesco in avanti;
1 pl 10 pM primer reverse;
x ul-ChIP ed DNA (controllo di input, o DNA genomico);
y pl priva di nucleasi H 2 O per regolare il volume di reazione di 20 pl. - Far girare la piastra a 96 pozzetti con Mini piastra Spinner (Labnet).
- Eseguire qPCR con ABI 7300 PCR in tempo reale di sistema (o altri sistemi in tempo reale PCR) utilizzando la seguente condizione:
Fase 1: 50 ° C per 2 min, 1 ciclo;
Fase 2: 95 ° C per 10 min, 1 ciclo;
Fase 3: 95 ° C per 15 s, 60 ° C per 1 minuto, 40 cycles;
Fase 4 (fase di dissociazione): 95 ° C per 15 s, 60 ° C per 1 minuto, 95 ° C per 15 s. - Controllare la curva di dissociazione: Un picco al tracciato termico dissociazione suggerisce un singolo amplicone dalla reazione PCR. Più di un picco indica non specifico prodotto con la coppia di primer, i dati qPCR non devono essere utilizzati.
- Determinare la fase lineare di amplificazione esponenziale di reazione PCR per ciascuna serie di primer, calcolando la formula che risolve quantità di DNA secondo l'(soglia ciclo) Ct valore, che si basa sulla curva standard utilizzando la serie di diluito DNA genomico in 3a. 1.
- Se il valore di Ct ed ChIP-DNA e controllo di input sono all'interno della gamma lineare del valore Ct, calcolare l'arricchimento di ChIP-DNA rispetto ed input, a seconda del fattore di diluizione della ChIP-DE DNA e controllo di input in 2.4.
3b. Amplifica ChIP-per il sequenziamento del DNA ed elevato throughput.
- End-riparazione di ChIP-ed mix DNA (UsaEpicentre DNA END-Repair kit), costituita il ghiaccio:
1-34 DNA genomico microlitri da passo 2,12 (da 0,1 a 5 pg)
5 microlitri tampone 10x End riparazione
5 microlitri mix 2,5 mM dNTP
5 microlitri 10 mM ATP
x pl H 2 O per regolare il volume di reazione di 49 microlitri
1 pl End-Repair mix enzima (DNA polimerasi T4 + polinucleotide chinasi T4)
Incubare per 45 minuti a RT. Purificare reazione con Qiagen reazione MinElute
Cleanup kit. Eluire in 30 microlitri tampone di eluizione (EB). - Aggiungere A-sovrasta all'estremità 3 ':
30 pl di DNA prodotto eluito dalla 3b.1 passo
Buffer 5 microlitri NEB 10x # 2
10 pl 1 mM dATP
2 microlitri dH 2 O
3 microlitri 5 U / pl frammento di Klenow (3 '→ 5' Exo-)
Incubare per 30 minuti a 37 ° C. Purificare reazione con MinElute kit ReactionCleanup. Eluire in 10 pl EB. - Solexa legatura linker:
10 pl di DNA eluito dalla 3b.2 passo
10 pl ddh 2 O
2.5 μ l 10x tampone di T4 DNA ligasi
1 ml mix oligo Adattatore da Illumina (1/10-diluted dal magazzino)
2,5 microlitri DNA ligasi T4 (400 unità / pl)
Incubare per 1 ora a RT. Purificare reazione con MinElute kit ReactionCleanup. Eluire in 20 pl EB. Esempio possono essere conservati a -20 ° C. - Eseguire il campione eluito da DNA 3B.3 passo utilizzando l'indirizzo e-gel apparecchio. Isolare la banda di 300-500 bp nel gel e purificare mediante il Gel Extraction kit Qiagen (questo passaggio elimina i ~ 125 bp auto-legato linker dalla ligazione dell'adattatore oligo). Eluire in 12 pl EB.
- Amplifica libreria tramite accoppiato-end (PE) primer da Illumina:
10,5 pl di DNA eluito dalla 3B.4 passo
12,5 microlitri di master mix (2X Phusion HF, Finnzymes)
1 ml di primer PCR PE1 1
1 ml di primer PCR PE2 2
PCR condizione:
98 ° C per 30 sec
(98 ° C 10 sec, 65 ° C 30 sec, 72 ° C 30 sec), 20 cicli si ripetono
72 ° C per 5 minuti. - Campioni di 3b.5 passo può essere utilizzato per ChIP-seq dopo aver selezionato il DNA di dimensioni adeguate (300-500 bp) utilizzando lo standard 2% gel di agarosio. Esempio possono essere conservati a -20 ° C. E 'meglio isolare ogni campione ChIP su un unico gel per prevenire la contaminazione. I campioni possono essere presentate per high-throughput sequencing.
3c. Solexa pipeline di analisi
- Il 25-bp sequenziamento legge sono stati ottenuti dal genoma Illumina Analyzer (GA) pipeline.
- Tutte le letture sono state allineate al genoma di Drosophila (dm3) utilizzando l'antilope (Efficient Allineamento locale dei dati Nucleotide) software, che consente fino a due discrepanze con la sequenza di riferimento.
- Letture in modo univoco mappato sono state selezionate per l'analisi a valle. Per letture multiple identici, fino a tre copie sono state conservate per ridurre la possibilità di distorsioni di amplificazione PCR.
- L'uscita del Pipeline GA è stato convertito in dati estendibili del browser (BED) file.
- Per generarei file di parrucca per caricare al browser per la visualizzazione UCSC, abbiamo usato una bisaccia python che è stato descritto in una precedente pubblicazione 3 con 4 bp, come la dimensione della finestra e 160 bp, come la dimensione del frammento di DNA.
- Per classificare i geni nella Drosophila geni silenziosi e espressa, abbiamo utilizzato un numero digitale chiamato RPKM (letture / per kilobasi fusa regione exonic / per milione mappata letture). I geni con RPKM = 0 sono stati classificati come gruppo silenzioso e geni con RPKM ≥ 1 sono stati classificati nei gruppi espresse, che possono essere classificate in tre sottogruppi come i geni con livelli bassi di espressione, moderato e alto, e ogni gruppo ha circa lo stesso numero di geni.
- Per confrontare il livello di modificazione degli istoni e l'espressione genica, abbiamo utilizzato uno script python che è stato descritto in una precedente pubblicazione 3.
4. Risultati rappresentativi
Esempi di ChIP-qPCR risultati utilizzando Bam (sacchetto di biglie) mutante testicoli sono mostrati in Figura 1 4. In Bam testicoli, si verifica un errore nel passaggio dal spermatogoni proliferativa a differenziare spermatociti 5, 6. Usiamo bam testicoli come fonte di cellule germinali indifferenziate, che sono arricchiti in questo tipo di tessuto. Differenziazione dei geni necessari per la differenziazione degli spermatozoi, come maschio-specifico fattore di trascrizione 87 (mst87F), Don Juan (dj), e la cipolla fuzzy (fzo) non sono espressi in Bam testicoli. Questi geni sono altamente arricchito con la repressione H3K27me3 modifica istoni 7 (Figura 1A), ma sono privi del attivo H3K4me3 modifica istoni 8 (Figura 1B), una firma cromatina abbiamo chiamato 7 'monovalente'. Arricchimento di una o H3K27me3 H3K4me3 è determinata dalla normalizzazione ad una ciclina costitutivamente espressa A (CYCA) del gene 4.
contenuto "> ChIP-seq analisi utilizzando lo stesso set di anticorpi (cioè repressiva H3K27me3 e attiva H3K4me3) con bam testicoli mutanti (Figura 2) qPCR convalidato i risultati illustrati nella Figura 1. Per i tre geni testati differenziazione terminale mst87F, dj e fzo , le regioni genomiche sono altamente arricchito con H3K27me3 ma non H3K4me3 (Figura 2A-2C). In contrasto, il gene espresso costitutivamente CYCA ha H3K4me3 significativo ma poco H3K27me3 vincolante in prossimità del suo sito di inizio trascrizione (TSS) (Figura 2D).Inoltre, i profili di ChIP H3K27me3 e H3K4me3 vicino ai TSSS di quattro classi di geni differenzialmente espressi sono coerenti con la funzione di ogni modificazione degli istoni. Come mostrato in figura 3A, l'arricchimento di H3K27me3 valle del TSSS è inversamente correlato al livello di espressione genica. I geni silenziosi hanno il più alto H3K27me3 mentre ilgeni altamente espressi non hanno H3K27me3 vincolanti. Questi dati sono coerenti con il ruolo repressivo di H3K27me3 sull'espressione del gene. Al contrario, l'arricchimento delle H3K4me3 intorno alle TSSS hanno mostrato la correlazione opposta con il livello di espressione genica (Figura 3B), coerente con il ruolo attivo di H3K4me3 sull'espressione genica.
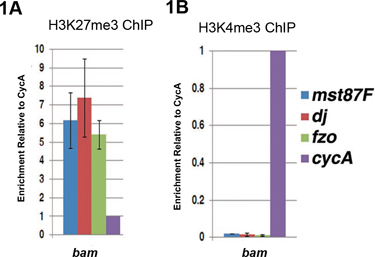
Figura 1. Analisi qPCR di ChIP-ed utilizzando anticorpi contro il DNA sia il repressiva H3K27me3 modificazione degli istoni (A) o attivo H3K4me3 modificazione degli istoni (B) nei testicoli cellule indifferenziate mutanti arricchito bam. (A) a Bam testicoli, i geni di differenziazione (Mst87F, dj, fzo) sono arricchiti con la modifica H3K27me3 repressiva degli istoni. (B) geni di differenziazione si esauriscono con H3K4me3 marchio attivo. Il livello di DNA ChIP (DNA ChIP / ingresso) al gene bersaglio (Mst87F, dj o fzo) per la prima volta normalizzata la Lev el DNA di ChIP al gene di controllo CYCA. Le barre di errore indicano la deviazione standard da tre repliche indipendenti biologica.
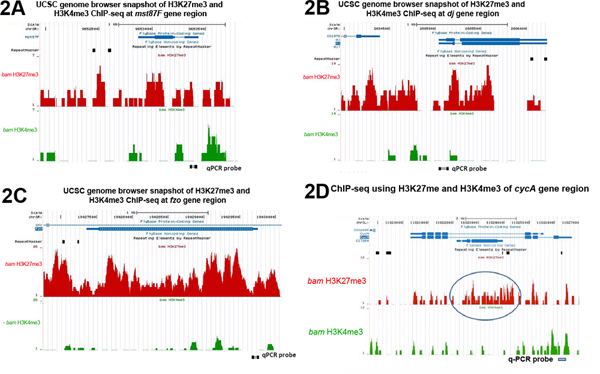
Figura 2. UCSC Genome Browser istantanee di H3K27me3 e H3K4me3 arricchimento attraverso le intere regioni genomiche di (A) Mst87F (B), dj e (C) fzo, (D) geni CYCA. La A cerchiata H3K27me3 arricchita di regione (D) riflette lo stato della cromatina una sovrapposizione gene CG7264, che si esprime umile bam testicoli (RPKM = 1), ma altamente espresso nel wild-type testicoli (RPKM = 130) 8 (ChIP-seq dati provenienti da 7). Sonde utilizzate in analisi PCR quantitativa dei risultati chip in figura 1 sono classificati nella parte inferiore di ogni parcella. Clicca qui per ingrandire la figura .
tp_upload/3745/3745fig3.jpg "/>
Figura 3. ChIP-seq profili utilizzando H3K27me3 e H3K4me3 in bam testicoli 7. I quattro gruppi di geni (7.509 geni) sono stati classificati in base ai livelli di espressione genica loro basati su RNA-seq 8 risultati. Le classi rappresentative dei geni sono tracciate per l'arricchimento di una particolare modificazione degli istoni, utilizzando sequenze da 3kb 3kb a monte a valle dei siti di trascrizione loro inizio (TSSS). Questo genera un profilo di arricchimento (A) H3K27me3 (K27) e (B) H3K4me3 (K4) ChIP-seq analisi di ciascun gruppo. Clicca qui per ingrandire la figura .
Access restricted. Please log in or start a trial to view this content.
Discussione
La versatilità di analisi ChIP discussi in questo protocollo può essere utilizzato su diversi tessuti, che fornisce l'opportunità di studiare lo stato cromatina in un sistema biologicamente rilevante. Esperimenti chip utilizzando cellule coltivate sono sistemi conveniente eseguire a causa grande quantità di cellule può essere ottenuto facilmente. Tuttavia, cellule coltivate non riflette necessariamente cellule in un multi-cellulare ambiente. Con lo sviluppo di questa tecnica usando tessuti sezionati da animali ...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Non abbiamo nulla da rivelare.
Riconoscimenti
Gli autori desiderano ringraziare il Dott. Keji Zhao lab (NIH / NHLBI) per il loro aiuto nel fornire i risultati di sequenziamento. Vorremmo anche ringraziare il progetto genoma UCSC per l'utilizzo di Browser per visualizzare sequenziamento del genoma mappato legge.
Questo lavoro è stato sostenuto dal Pathway R00HD055052 NIH al Premio Indipendenza e R01HD065816 dal NICHD, il Lucile Parkard Foundation, e la Johns Hopkins University di start-up fondi per XC
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reattivo | Azienda | Numero di catalogo | Comments |
| Complete Mini cocktail inibitore della proteasi | Roche | 11836153001 | |
| La formaldeide (37%) | Supelco | 47.083-U | |
| PMSF | Sigma | 78830 | |
| Kontes pellet pestello | Fischer Scientific | K749521-1590 | |
| PCR Purification Kit | Qiagen | 28104 | |
| Poliacrilammide lineare | Sigma | 56575-1 ml | |
| Glicogeno | Qiagen | 158930 | |
| SYBR green / ROX qPCR Master Mix | Fermentas | K0223 | |
| Mini Piastra filatrice | Labnet | Z723533 | |
| Real time PCR System | Applied Biosystem | 4351101 | |
| Volume piccolo processore ad ultrasuoni | Misonix | HS-XL2000 | Modello interrotto |
| Dynabeads, Proteina A | Invitrogen | 100-01D | |
| Dynamag magnete | Invitrogen | 123-21D | |
| Fenolo: Chlorofrom: IAA | Invitrogen | 15593-049 | |
| Epicentre DNA END-Repair Kit | Epicentre Biotecnologie | ER0720 | |
| MinElute Reaction Cleanup Kit | Qiagen | 28204 | |
| Klenow Fragment (3 '→ 5' exo-) | New England Biolabs | M0212S | |
| DNA ligasi T4 | Promega Corporation | M1794 | |
| Adattatore oligonucleotidi | Illumina | PE-400-1001 | |
| Abbinato-End Primer 1.0 e 2.0 | Illumina | 1001783 1001 784 | |
| E-Gel Electorphoresis sistema | Invitrogen | G6512ST | |
| 2X Phusion HF Mastermix | Finnzymes | F-531 |
Riferimenti
- Kharchenko, P. V., Alekseyenko, A. A., Schwartz, Y. B., Minoda, A., Riddle, N. C., Ernst, J., Sabo, P. J., Larschan, E., Gorchakov, A. A., Gu, T. Comprehensive analysis of the chromatin landscape in Drosophila melanogaster. Nature. 471, 480-485 (2011).
- Filion, G. J., van Bemmel, J. G., Braunschweig, U., Talhout, W., Kind, J., Ward, L. D., Brugman, W., de Castro, I. J., Kerkhoven, R. M., Bussemaker, H. J., van Steensel, B. Systematic protein location mapping reveals five principal chromatin types in Drosophila cells. Cell. 143, 212-224 (2010).
- Barski, A., Cuddapah, S., Cui, K., Roh, T. Y., Schones, D. E., Wang, Z., Wei, G., Chepelev, I., Zhao, K. High-resolution profiling of histone methylations in the human genome. Cell. 129, 823-837 (2007).
- Chen, X., Lu, C., Prado, J. R., Eun, S. H., Fuller, M. T. Sequential changes at differentiation gene promoters as they become active in a stem cell lineage. Development. 138, 2441-2450 (2011).
- Gonczy, P., Matunis, E., DiNardo, S. bag-of-marbles and benign gonial cell neoplasm act in the germline to restrict proliferation during Drosophila spermatogenesis. Development. 124, 4361-4371 (1997).
- McKearin, D. M., Spradling, A. C. bag-of-marbles: a Drosophila gene required to initiate both male and female gametogenesis. Genes Dev. 4, 2242-2251 (1990).
- Gan, Q., Schones, D. E., Eun, S. H., Wei, G., Cui, K., Zhao, K., Chen, X. Monovalent and unpoised status of most genes in undifferentiated cell-enriched Drosophila testis. Genome Biol. 11, 42-42 (2010).
- Gan, Q., Chepelev, I., Wei, G., Tarayrah, L., Cui, K., Zhao, K., Chen, X. Dynamic regulation of alternative splicing and chromatin structure in Drosophila gonads revealed by RNA-seq. Cell Res. 7, 763-783 (2010).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon