Method Article
Profiling tiolo Proteome Redox Utilizzando tag spettrometria di massa isotopica
In questo articolo
Riepilogo
Livello di specie reattive di ossigeno è elevata quando le cellule incontrano condizioni di stress. Qui si mostra l'esempio di 3'-3 'colorazione diaminobenzidina così come l'etichettatura cysTMT e spettrometria di massa per analizzare il proteoma redox in Pseudomonas syringae Trattati foglie di pomodoro.
Abstract
Pseudomonas syringae pv. Pomodoro DC3000 ceppo non solo provoca la malattia batterica speck in Solanum lycopersicum, ma anche su specie di Brassica, nonché su Arabidopsis thaliana, un organismo geneticamente trattabili 1,2 pianta ospite. L'accumulo di specie reattive dell'ossigeno (ROS) in cotiledoni inoculati con DC3000 indica un ruolo dei ROS nella modulazione morte delle cellule necrotiche durante la malattia batterica granello di pomodoro 3. Il perossido di idrogeno, un componente di ROS, viene prodotto dopo l'inoculazione di piante di pomodoro con Pseudomonas 3. Il perossido di idrogeno può essere rilevato utilizzando un istochimica macchia 3'-3 'diaminobenzidina (DAB) 4. Colorazione DAB reagisce con perossido di idrogeno per produrre una macchia scura il 4 tessuto fogliare. ROS ha un ruolo normativo l'ambiente cellulare redox, che può modificare lo stato redox di alcune proteine 5. La cisteina è un importante aminoacido sensibilemodifiche redox. Sotto blanda ossidazione, ossidazione reversibile di cisteina gruppi solfidrilici funge redox sensori e trasduttori di segnale che regolano una varietà di processi fisiologici 6,7. Di massa tandem tag (TMT) reagenti consentire l'identificazione simultanea e multiplex quantificazione delle proteine in diversi campioni con spettrometria di massa tandem 8,9. Le cisteina-reattivi TMT (cysTMT) reagenti consentono etichettatura selettiva e quantificazione relativa di cisteina contenenti peptidi da un massimo di sei campioni biologici. Ogni tag cysTMT isobarica ha la stessa massa madre nominale ed è composto da un sulfidrile reattivo, un gruppo MS-neutro braccio spaziatore e un MS / MS giornalista 10. Dopo etichettatura, i campioni sono stati sottoposti a digestione proteasica. I peptidi cisteina-marcati sono stati arricchiti con una resina contenente anticorpi anti-TMT. Durante MS / MS, una serie di ioni del reporter (cioè, 126-131 Da) emergono nella regione di bassa massa, fornisce informazioni sulla quantificazione relativa.Il flusso di lavoro è efficace per ridurre la complessità dei campioni, migliorando gamma dinamica e studiare modifiche cisteina. Qui vi presentiamo redox analisi proteomica del pomodoro Pst DC3000 trattati (Rio Grande) lascia utilizzando la tecnologia cysTMT. Questo elevato throughput metodo ha il potenziale per essere applicata a studiare altri processi fisiologici redox-regolati.
Protocollo
1. Piantine in crescita e batteri Preparazione
- Piantine sono germinati in metromix 500 miscela di sporco (Aziende BWI) in una camera di crescita (160 fotoni micromoli m -2 s -1 con un fotoperiodo di 8 ore di luce solare a 22 ° C e 16 ore di buio a 20 ° C per 1 settimana. Relativa umidità è stato impostato al 70%. piantine sono state annaffiate con acqua di rubinetto come necessario per mantenere il terreno diventi secca.
- Due pianticelle di dimensioni simili vengono trapiantate a 4 "vasi contenenti il diametro metromix 500 terreno e coltivate per ulteriori 3 settimane nelle stesse condizioni 1,1.
- Pseudomonas syringae (Pst) è inizialmente T-striato sul Kings B Medio (KBM) piastre per litro: 20 g PROTEOSE Peptone, 0,1975 g MgSO 4, 1% glicerolo, 1,5 g di K 2 HPO 4, Agar 18 g) e coltivate per due giorni a 28 ° C. Una singola colonia viene raccolto, mescolato in 300μl Kings liquido B Medium (20 g PROTEOSE peptone, 0,1975 g MgSO 4 , glicerolo 10 mL, 10 mL di 100x K 2 HPO 4 archivio (1,5 g di K 2 HPO 4 in 10 mL di H 2 O), e diffuso su una piastra Kings B medio. Questo viene coltivato durante la notte a 28 ° C.
- L'inoculo viene preparato batteri raschiando i batteri coltivati dalla piastra KBM in 20 mL H 2 O. Un OD 600 di 0,03 (~ 10 6 cfu / ml) viene preparato in 1 L di tampone di inoculazione (10 mM MgCl 2 + 400 microlitri Silwett-70). L'inoculazione del buffer meno batteri è stata preparata per la soluzione di controllo.
2. Inoculazione di pomodoro con Pst e H 2 O 2 istochimica Stain
- Quattro settimane di età piante sono state inoculate mediante immersione nella soluzione inoculazione sopra per 30 secondi. Sacchetti di plastica sono state messe sopra le piante immediatamente dopo l'inoculazione.
- 3'-3 'diaminobenzidina (DAB) macchia 11 è stato preparato (1 mg / ml DAB in H 2 O, pH 3,8 (HCl)) tre days prima dell'uso per avere solubilità completa. La soluzione è stata scossa per miscelare a temperatura ambiente.
- Ventiquattro ore dopo il trattamento, la foglia completamente espansa è stato rimosso, posto in DAB lato macchiato epidermica, e infiltrato vuoto per 15 min. Le foglie sono poi stati messi al buio a temperatura ambiente per tutta la notte per la colorazione.
- Le foglie sono stati bolliti in 95% etanolo per 10 minuti, e quindi tenuti in etanolo al 75%.
3. Estrazione delle proteine
- Dopo la raccolta, foglia di pomodoro sono state polverizzato con azoto liquido usando mortaio e pestello.
- Per 0,5 g di tessuto, aggiungere 1,25 ml di Tris pH 8,8 tamponata fenolo e 1,25 mL / 0,5 g di tampone di estrazione (0,1 M Tris-HCl pH 8,8, 10 mM EDTA, 0,9 M saccarosio), poi proseguire macinazione per qualche minuto in un più cappa.
- Trasferire l'estratto di centrifuga tubo Oakridge (Thermo Fisher Scientific Company Nalgene) ed agitare per 2 ore a temperatura ambiente.
- Centrifugare a 5000 xge 15 ° C per 10 min. Trasferire la fase superiore fenolo in una nuova provetta pulita.
- Back-estrae la fase acquosa estratta con un volume uguale tampone fenolo; agitate su un agitatore per 30 min. Centrifugare e trasferire l'estratto in una nuova provetta Falcon.
- Ripetere il passaggio precedente ancora una volta e trasferire l'estratto di nuovo tubo Oakridge.
- Fenolo precipitare le proteine estratte con l'aggiunta di 5 volumi di acetato di ammonio 0,1 M in metanolo 100% (conservato a -20 ° C). Vortex e incubare a -20 ° C per il pernottamento.
- Raccogliere la proteina pellet mediante centrifugazione a 20.000 xg, a 4 ° C per 20 min.
- Lavare i pellet 2 volte con il freddo acetato di ammonio 0,1 M in metanolo e 2 volte con l'80% acetone freddo. Aggiungere 1,5 ml di etanolo freddo al 70% di pellet e sospendere. Trasferire in una provetta da microcentrifuga da 2 mL (USA Scientific) e centrifugare a 14.000 rpm, 4 ° C per 20 min. Eliminare l'etanolo.
- Asciugare brevemente il pellet in un concentratore SpeedVac e si dissolvono in un extr proteineun'azione tampone, ad esempio ReadyPrep Protein Reagent Kit di estrazione 3 (8 M urea, 4% CHAPS, 40 mM Tris-base, tiourea 2 M) (Bio-Rad). Centrifugare a 14.000 rpm e 20 ° C per 30 minuti per formare un pellet. Raccogliere il surnatante.
- Misurare la concentrazione di proteine con un CB-X dosaggio di proteine in base al manuale del costruttore (Geno Technology).
4. Preparazione del campione e Labeling Peptide con cysTMTs
- Preparare il campione di proteine ad una concentrazione di 2-5 mg / pl. Qui usiamo 100 microgrammi di proteine per ogni tag di massa, 20 campioni ul ul-50. Per questo esperimento, tutti i tag 6 sono stati utilizzati.
- Aggiungere volume uguale di tampone alchilazione (100 mM Tris-HCl pH7.5, 200 mM di iodoacetammide) per il campione di proteina per bloccare il gruppo tiolo libero. Buffer deve essere preparato fresco. Alchilazione viene effettuata a 37 ° C per 1 ora.
- Precipitare la proteina aggiungendo 1 ml di acetone freddo 80% a -20 ° C per una notte. La proteina pellet mediante centrifugazione a 14.000rpm, 4 ° C per 20 min. Lavare 3 volte a pellet con l'80% Acetone vortex ogni volta. Rimuovere il surnatante e permettere all'aria quando asciutto su ghiaccio.
- Risospendere il precipitato in 50 microlitri di tampone di lisi (6 M urea, 50 mM Tris base, 1 mM EDTA). 0,5 pl di 100 mM Tris (2-carbossietil) fosfina (TCEP) viene poi aggiunto per ridurre i legami disolfuro mediante incubazione per un'ora a temperatura ambiente.
- Preparare i tag con l'aggiunta di 20 microlitri di acetonitrile ai tubi di reagenti per solubilizzare i tag. Vortex e centrifugare fino in fondo.
- Lavare TCEP usando un dispositivo Microcon centrifuga 3KD filtro (Millipore). Aggiungere 50 microlitri di campione colonna Microcon 3KD, e 50 pl di tampone di lisi alla colonna. Centrifugare per 15 min a 10.000 xg e 4 ° C. Ripetere questo passaggio per un totale di tre volte. Rimuovere colonna e invertire in una provetta pulita. Centrifugare per 5 min a 1000 xga 4 ° C.
- Controllare il pH. Se necessario, regolare il pH a 7,0-8,0 con 1 M HCl.
- Aggiungere 5 pl della cysTMReagente T per ogni campione (Il kit reagenti cysTMT dispone di 20 microlitri per i tag fino a 500 mg di proteine. Questo metodo utilizza il 100 pg di proteina, quindi usiamo un quinto del tag.). Lasciare la reazione per 2 ore a temperatura ambiente.
- I campioni sono stati combinati con tampone per campioni di Laemmli (Bio-Rad) in un rapporto 1:1.
5. Eliminazione delle materie non ha reagito Frazionamento Tag Campione
- I campioni sono stati bolliti per 5 minuti e separato su un pre-cast gel di poliacrilammide 12% (Bio-Rad). Il gel è stato lavato tre volte in intervalli di 5 minuti con filtrato H 2 O e colorato con Coomassie Blue (Bio-Rad) per 1 ora. Il gel è stato de-macchiato durante la notte con H 2 O.
- Dodici frazioni sono state raccolte da ogni corsia di gel. Le frazioni sono state determinate le concentrazioni di banda di proteina. Le frazioni sono state tagliate in pezzi 1mm e raccolto in una provetta da 1,5 ml.
- Pezzi di gel sono state de-tinto con 50% acetonitrile e di bicarbonato di ammonio 0,1 M in H 2 O. Questa operazione deve essere ripetuta fino colore blu viene rimosso da pezzi gel (circa 3-4 volte, 15 minuti ogni volta).
- De-gel macchiati pezzi sono stati essiccati utilizzando il 100% acetonitrile (coprire parti gel) per 15 min, surnatante eliminato, e le parti secche con una SpeedVac.
- Sciogliere tripsina (Promega) a 1:10 (tripsina: proteina) il rapporto del volume stesso (come pool campione marcato) di 25 mM di bicarbonato di ammonio. Ad esempio, 600 pg di proteina campione con un volume di 500 pl bisogno di 60 ug tripsina sciolti in 500 pl di 25 mM di bicarbonato di ammonio.
- Aggiungere la soluzione di tripsina ai pezzi gel e impostare sul ghiaccio per reidratare. Se i pezzi non sono coperti aggiungere 50 mM di tampone di bicarbonato di ammonio. Dopo la reidratazione, incubare a 37 ° C per 12-16 ore.
- Rimuovere il liquido estratto dai campioni di proteine incubate.
- Bloccare la reazione e rimuovere qualsiasi residuo digest proteina coprendo pezzi gel con 5% acido formico, 50% acetonitrile .. Agitare pezzi gel per 20 min a temperatura TEmperature. Trasferire il surnatante ai tubi di proteine estratte dal campione 5.7. Ripetere la procedura di estrazione due volte. Asciugare i peptidi estratti utilizzando il SpeedVac.
6. Arricchimento del campione di cysTMT etichetta-peptidi
- Aggiungere un gradiente di anti-TMT resina per provette da 0,5 ml per un impasto al 50%. (Gradiente è stata determinata dalla concentrazione di banda in gel frazionamento. Se una raccolta frazione costituita da una fascia scura, più resina è necessaria. Frazioni con deboli bande richiedono meno resina come c'è meno proteina.)
- La resina viene poi lavata tre volte con un volume di colonna 1x soluzione salina tamponata Tris (TBS) (25 mM Tris, 0,15 M NaCl, pH 7,2) (Pierce Thermo Scientific Research Products Protein).
- Aggiungere 200 ul 1xTBS a ciascun campione. I campioni vengono quindi aggiunti anti-TMT resina (Thermo Scientific Products Pierce ricerca sulle proteine) e agitato a temperatura ambiente per 2 ore seguita da oscillare notte a 4 ° C.
- Aggiungere il campione alla colonna (TheRMO scientifici Pierce prodotti proteici di ricerca).
- Lavare ogni colonna tre volte con TBS 1x 200 pl. Segue poi con lavaggio tre volte con 0,05% CHAPS (disciolto in 1x TBS).
- La colonna viene poi lavata tre volte con 4 M urea in 1x TBS. Duecento microlitri di H 2 O viene poi utilizzato per lavare la colonna tre volte.
- Ogni campione viene eluito tre volte con 200 microlitri tampone di eluizione 50% (50% acetonitrile, 0,4% TFA).
- I campioni vengono poi essiccati in un concentratore a vuoto.
7. Analisi di Spettrometria di Massa
- Risospendere i campioni in 12 pl del 3% acetonitrile con 0,1% di acido formico e iniettare 5 microlitri direttamente su un Eksigent nanoLC-1D colonna cromatografia liquida ad alta pressione (AB SCIEX, USA).
- Peptidi sarà separata su una colonna C18 II Proteopep ID 75 um x 20 cm (Nuovo obiettivo, USA) utilizzando un gradiente 4-60% (A: acetonitrile 3%, 0,1% di acido formico; B: 97% acetonitrile, 0,1% formico acido)a 3 pl / min per 60 minuti.
- A Thermo Scientific LTQ Orbitrap XL spettrometro di massa è stata utilizzata per rilevare peptidi utilizzando un top 2 x 3 esperimento costituito da stage MS singolo seguito da acquisizione di 3 MS / MS con una maggiore energia C-trap dissociazione (HCD) la frammentazione seguita da 3 MS / MS con la dissociazione indotta da collisione (CID) per l'identificazione delle proteine. I parametri in questa modalità sono: larghezza Isolamento: 3,0 m / z; energia di collisione: 50% (10% con due passaggi). Peptidi Solo doppiamente e triplamente-caricate sono stati selezionati per la frammentazione. Parametri dinamici di esclusione sono stati fissati per: Ripetere count = 1; durata della ripetizione = 60; list size esclusione = 500; Esclusione durata = 28. Valori target sono i seguenti: MS = 5 e 5; MS / MS (HCD) = 1 e 5. Tempi di trasferimento di ioni sono stati fissati a 500 e 300 per FTMS per MS / MS (HCD). Due microscans sono stati richiesti per spettri HCD.
8. Ricerca nel database e quantificazione
- I dati acquisiti CID e HCD eranonalyzed con Thermo Scientific software Discoverer Proteome 1.2 (Thermo Scientific Pierce Prodotti ricerca sulle proteine) attraverso un flusso di lavoro ramificata che ha attuato uno Reporter Ion quantizer (20 ppm tolleranza di massa di ioni frammento) per quantificare il rapporto. Un segmento separato elaborato il MS 2 spettri attraverso la Spectrum Selector, Normalizer Spectrum, ei nodi Grouper Spectrum.
- I dati sono stati poi confrontati con il motore di ricerca SEQUEST. Una tolleranza 20 ppm di massa è stato utilizzato. Modifiche statici sono stati fissati per il reagente cysTMT (304,18 Da) e modifiche dinamiche inclusi fosforilazione e l'ossidazione della metionina (Da 15,99). Un database personalizzato per Solanum lycopersicum è stata composta utilizzando RNA dati (con circa 350.000 voci dalla Harvard University) 13 è stato utilizzato in entrambi i casi.
9. Risultati rappresentativi
Una immagine rappresentativa di una foglia di impianto di controllo di pomodoro e una foglia di Pseudomonas è inoculatomostrato nella Figura 1. Una differenza tra controllo trattati e le foglie di Pseudomonas trattati si osserva. Dopo che le foglie vengono rimossi e colorati con DAB, la de-colorazione processo permette la colorazione istochimica a mostrare segni di ROS nel tessuto fogliare (Figura 2). Figura 2A è rappresentativo di una foglia di controllo senza colorazione. Figura 2B è rappresentativo di una foglia trattata con colorazione positiva per Pseudomonas e H 2 O 2 produzione. Un esempio di uscita Proteome Discover dati di una proteina differenzialmente regolati redox viene mostrato nella Figura 3. Questa proteina è un noto ferredossina-1 redox regolata proteina 14 e ha dimostrato di giocare un ruolo nella difesa contro pomodoro Pseudomonas syringae pv 15. Intensità di picco tra il controllo e campioni inoculati viene utilizzato per ottenere la quantificazione relativa, che mostrava cambiamenti significativi nella ferredoxin-1 regolazione redox (p <0,05). Picchi ad alta intensità suggeriscono che questa proteina è ossidato in risposta al trattamento patogeno. Figura 4 è un esempio di uscita Proteome Discover dati di una proteina che ha simile regolazione redox di una proteina tra un campione e controllo inoculato. Picchi di intensità simile suggeriscono la presenza di legami disolfuro non regolata da un cambiamento di trattamento. Il metodo rivoluzionerà il modo di rilevare gli scienziati redox cisteine reattivi e disolfuri 10.

Figura 1. Un'immagine rappresentativa di pomodoro lascia inoculato con soluzione di controllo (A) e Pseudomonas (B).

Figura 2. Un'immagine rappresentativa di colorazione DAB di pomodoro lascia inoculato con soluzione di controllo (A) e Pseudomonas (B). Le foglie sono state colorate con 3'-3 'diaminobenzidina. Clorofilla è stato rimosso dalle foglie mediante bollitura in etanolo al 95%. Colorazione scura indica la presenza di H 2 O 2. Lascia solo inoculato con coltura batterica ha mostrato una colorazione scura.
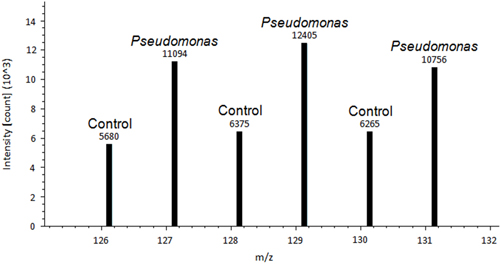
Figura 3. Un esempio di output Proteome Discover dati di ferredossina-1, differenzialmente regolati redox proteina 14. Intensità del picco di sopra di ogni picco viene utilizzato per la quantificazione assoluta. Picco di intensità tra controllo e campioni inoculati viene utilizzatoper eseguire la quantificazione relativa.

Figura 4. Un esempio di uscita Proteome Discover dati di una proteina che ha intensità simile picco tra un campione e controllo inoculato. Picchi di intensità simile suggeriscono la presenza di legami disolfuro non regolata da un cambiamento di trattamento.
Discussione
Questo protocollo fornisce informazioni su come eseguire la colorazione DAB così come cysTMT quantificazione etichettato cisteina redox. Queste procedure sono utili in esame produzione di ROS nonché l'effetto sulla regolazione proteina quando Solanum lycopersicum viene inoculato con Pseudomonas syringae. I metodi presentati in questo protocollo forniscono un modo per esaminare ROS in campioni di foglie intere in un modo che fa sì che la minor quantità di danno al tessuto fogliare. La procedura di etichettatura fornisce un metodo per esaminare le proteine regolate potenzialmente redox utilizzando un metodo di etichettatura cisteina. Questo è utile quando l'esame di una fase precoce della risposta allo stress.
Metodi quali isotopo codificato marcatore per affinità (ICAT) e cysTMT può essere utilizzato in esame potenziali redox proteine regolamentati in campioni biologici. ICAT permette l'etichettatura e il confronto di due campioni 12. Entrambi i metodi etichettare cisteine libere e possono essere utilizzati per la proteina quantificzione 10,12. Tuttavia, il metodo cysTMT consente una riduzione nella variazione sperimentale e multiplazione 10. Il numero di tag a disposizione permette ai ricercatori di comprendere repliche o più campioni nel loro design sperimentale. Avendo più campioni fornisce il potenziale per un numero superiore di proteine identificate. Uno dei principali svantaggi della tecnica cysTMT è che compromette la qualità complessiva di identificazione di proteine a causa delle fasi di arricchimento selettivo per cysTMT etichettati-peptidi (6,5-6,6). Il numero di peptidi per l'identificazione di proteine dipende in larga misura il numero di residui di cisteina nella sequenza della proteina. Questo problema può essere superato presentando una parte del campione triptico prima di arricchimento per l'identificazione spettrometria di massa proteica.
A causa della natura del disegno sperimentale così come il meccanismo di etichettatura che utilizza il metodo cysTMT, certi passi sono critici. Durante l'esecuzione di proteine Precipitation e pellet lavaggi (3,9) è importante mantenere i campioni raffreddata su ghiaccio per ridurre la degradazione delle proteine. Durante etichettatura cysTMT, rimozione del reagente riduzione (4,6) è importante perché i campioni possono subire etichettatura inversa. Etichettatura inversa è possibile ridurre se reagente rimane nel campione. Se i campioni vengono ridotte dopo l'etichettatura, il tag cysTMT può essere rimosso. Quando le etichette vengono aggiunti ai campioni, il pH deve essere controllata (4,7) in modo da avere un'efficienza ottimale etichettatura. Inoltre, l'analisi dei dati dipende da ciò che è richiesto del ricercatore e l'obiettivo finale in utilizzando il protocollo. È anche dipendente dal software utilizzato come ogni software deve algoritmi differenti.
Questo esperimento utilizza patogeno come stimolatore per la maggiore produzione di specie reattive ossidativi nel pomodoro, ma altre reazioni redox regolamentati può essere misurato di conseguenza. Questo progetto sperimentale è adattabile ad altri impianti e sistemi animali.
Divulgazioni
Non ci sono conflitti di interesse dichiarati.
Riconoscimenti
Gli autori desiderano ringraziare il Dr. Greg Martin (Cornell University) e il suo gruppo per aver fornito il ceppo DC3000, semi di pomodoro, e consiglio. Si desidera inoltre ringraziare il Dr. Zhonglin Mou per un aiuto con il protocollo DAB e la Divisione Proteomics UF presso il Centro Interdisciplinare per la Ricerca in Biotecnologie per l'assistenza allo sviluppo del metodo. Il protocollo per l'estrazione delle proteine è stato modificato dal Hurkman e Tanaka 16. Il protocollo sulla cysTMT etichettatura, a pochi passi da 4 a 6 è stato adattato in base al prodotto originale Thermo Fisher Scientific manuale Pierce 17. Questo lavoro è stato finanziato dalla National Science Foundation (MCB 0.818.051 a S Chen).
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reattivo | Azienda | Numero di catalogo | |
| Metromix 500 | BWI Aziende | TX-500 | |
| 3,3 '-diaminobenzidina | Sigma-Aldrich | D8001 | |
| ReadyPrep sequenziale kit del reagente di estrazione 3 | Bio-Rad | 163-2104 | |
| CB-X proteina saggio | Geno Tecnologia | 786-12x | |
| cysTMT reagenti | Thermo Scientific Protein Pierce Prodotti di ricerca | 90071 | |
| Laemmli Sample Buffer | Bio-Rad | 161-0737 | |
| Bio-Safe Comassie (G-250 macchia) | Bio-Rad | 161-0786 | |
| Microcon 3KD colonna | Millipore | 42403 | |
| Immobilizzato anti-TMT resina | Thermo Scientific Protein Pierce Prodotti di ricerca | 90076 | |
| Centrifuga colonna | Thermo Scientific Protein Pierce Prodotti di ricerca | 89896 | |
| Proteopep II colonna C18 | Nuovo obiettivo | PFC7515-PP2-10 | |
| NanoLC-1D HPLC | AB Sciex | 90389 | |
| LTQ Orbitrap XL | Thermo Scientific | 0020137580 | |
| SpeedVac | Labconco | 7812013 | |
| Proteome Discoverer 1,2 software | Thermo Scientific Protein Pierce Prodotti di ricerca | ||
| Tripsina | Promega | V5111 | |
| Oakridge provetta da centrifuga | Thermo Scientific Nalgene Società | 3139-0050 | |
| Microcentrifuga tubo (2 ml) | USA Scientific | 1620-2700 | |
| 12% Mini-PROTEAN TGX Precast Gel | Bio-Rad | 456-1043 | |
| Top of Form > Bio-Safe Coomassie StainBottom della Forma | Bio-Rad | 161-0786 | |
| TMT arricchimento kit | Thermo Scientific Protein Pierce Prodotti di ricerca | 90077 |
Riferimenti
- Almeida, N. F. A draft genome sequence of Pseudomonas syringae pv. tomato T1 reveals a type III effector repertoire significantly divergent from that of Pseudomonas syringae pv. tomato DC3000. Mol. Plant Microbe Interact. 22, 52-62 (2009).
- Preiter, K. Novel virulence gene of Pseudomonas syringae pv. tomato strain DC3000. J. Bacteriol. 187, 7805-7814 (2005).
- Apostol, I., Heinstein, P. F., Low, P. S. Rapid stimulation of an oxidative burst during elicitation of cultured plant cells: role in defense and signal transduction. Plant Physiol. 90, 109-116 (1989).
- Orozco-Cardenas, M., Ryan, C. A. Hydrogen peroxide is generated systemically in plant leaves by wounding and systemin via the octadecanoid pathway. Proc. Natl. Acad. Sci. U.S.A. 96, 6553-6557 (1999).
- Tonks, N. K. Redox redux: revisiting PTPs and the control of cell signaling. Cell. 121, 667-670 (2005).
- Alvarez, S., Wilson, G. H., Chen, S. Determination of in vivo disulfide-bonded proteins in Arabidopsis. J. Chromatogr. B. 877, 101-104 (2009).
- Apel, K., Hirt, H. Reactive oxygen species: metabolism, oxidative stress, and signal transduction. Annu. Rev. Plant Biol. 55, 373-399 (2004).
- Dayon, L. Relative quantification of proteins in human cerebrospinal fluids by MS/MS using 6- plex isobaric tags. Anal. Chem. 80, 2921-2931 (2008).
- Thompson, A. Tandem mass tags: a novel quantification strategy for comparative analysis of complex protein mixtures by MS/MS. Anal. Chem. 75, 1895-1904 (2003).
- Rosenblatt, M. A novel cysteine-reactive tandem mass tag reagent for subproteome labeling, enrichment and quantitation. , TP169 (2010).
- Clark, J. Phenotypic analysis of Arabidopsis mutants: diaminobenzidine stain for hydrogen peroxide. Cold Spring Harb. Protoc. , (2009).
- Sethuraman, M. Isotope-Coded affinity tag (ICAT) approach to redox proteomics: identification and quantification of oxidant-sensitive cysteine thiols in complex protein mixtures. J. Proteome Res. 3, 1228-1233 (2004).
- Asso, M. EPR and redox characterization of ferredoxins I and II from Desulfovibrio vulgaris Miyazaki. Biochem. Biophys. Res. Commun. 211, 198-204 (1995).
- Huang, H. e. Disease resistance to bacterial pathogens affected by the amount of ferredoxin-I proteins in plants. Mol. Plant Pathol. 8, 129-137 (2007).
- Hurkman, W., Tanaka, C. Solubilization of plant membrane proteins for analysis by two-dimensional gel electrophoresis. Plant Physiol. 81, 802-806 (1986).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon