È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Isolamento dei ribosomi Polipeptidi Bound nascenti
In questo articolo
Riepilogo
Una tecnica per identificare i siti di pausa traslabile su mRNA viene descritto. Questa procedura è basata su isolamento di polipeptidi nascenti accumulano sui ribosomi durante traduzione in vitro di un mRNA bersaglio, seguita dall'analisi dimensione delle catene nascenti elettroforesi utilizzando un gel denaturante.
Abstract
Il tasso di allungamento traslazionale è non uniforme. mRNA struttura secondaria, l'uso codone mRNA e proteine associate possono alterare il movimento ribosoma sul messaggio per una rassegna vedi 1. Tuttavia, è ormai ampiamente riconosciuto che l'utilizzo codone sinonimo è la causa primaria di non-uniformi tassi di allungamento traslazionali 1. Codoni sinonimo non vengono utilizzati con frequenza identica. Una polarizzazione esiste l'uso di codoni sinonimo con alcuni codoni usati più frequentemente di altri 2. Codone bias è organismo così come tessuto specifico 2,3. Inoltre, la frequenza di uso dei codoni è direttamente proporzionale alla concentrazione delle affine tRNA 4. Così, un codone frequentemente usato avrà maggiore moltitudine di tRNA corrispondenti, implicando che un codone di frequente si tradurrà più velocemente di un infrequente uno. Così, regioni mRNA arricchito in codoni rari (siti pausa potenziali) saranno di regola rallentare il movimento del ribosoma Message e causa l'accumulo di peptidi nascenti delle rispettive dimensioni 5-8. Questi siti di pausa può avere un impatto funzionale l'espressione della proteina, la stabilità mRNA e folding delle proteine per una rassegna vedi 9. Infatti, è stato dimostrato che la riduzione di tali siti di pausa può alterare il movimento ribosoma mRNA e successivamente possono influenzare l'efficienza di co-traduzionale (in vivo) 1,7,10,11 ripiegamento delle proteine. Per comprendere il processo di ripiegamento delle proteine in vivo, nella cella, che è accoppiato alla fine al processo di sintesi proteica è essenziale di acquisire informazioni complete sull'impatto codone di utilizzo / tRNA contenuto sul movimento dei ribosomi lungo mRNA durante l'allungamento traslazionale .
Qui si descrive una tecnica semplice che può essere utilizzata per localizzare siti di traduzione principali pausa per un dato mRNA tradotto in vari sistemi acellulari 6-8. Tale procedura si basa sull'isolamento del nascente polipeptidi accumulating sui ribosomi durante la traduzione in vitro di un mRNA bersaglio. La ragione è che a bassa frequenza codoni, l'aumento del tempo di residenza dei risultati ribosomi in quantità maggiore di peptidi nascenti delle dimensioni corrispondenti. In vitro mRNA trascritti viene utilizzato per reazioni di traslazione in vitro in presenza di amminoacidi marcati radioattivamente per consentire il rilevamento delle catene nascenti. Al fine di isolare ribosoma associazione complessi polipeptide nascente la reazione di traduzione è sovrapposto di soluzione di glicerolo 30% seguito da centrifugazione. Polipeptidi nascenti in polysomal pellet sono ulteriormente trattati con ribonucleasi A e risolti mediante SDS PAGE. Questa tecnica può essere potenzialmente utilizzati per ogni proteina e permette l'analisi di movimento ribosoma lungo mRNA e la rilevazione dei siti di pausa principali. Inoltre, questo protocollo può essere adattato per studiare fattori e condizioni che possono alterare il movimento ribosoma e quindi potenzialmente possono anche alterare the funzione / conformazione della proteina.
Protocollo
1. Preparazione del DNA template e trascrizione in vitro
- Il gene di interesse viene clonato sotto T7 e / o ad esempio SP6 promotore di trascrizione.
- Per la trascrizione in vitro del DNA stampo è linearizzato con un enzima di restrizione appropriato taglio a valle del codone di stop ORF e / o mRNA 3 '. Si deve verificare linearizzazione completa del DNA plasmide eseguendo il prodotto della digestione di restrizione elettroforesi su gel di agarosio.
- Il plasmide linearizzato viene utilizzato per la reazione di trascrizione in vitro. Diversa concentrazione di DNA stampo può essere testato per identificare concentrazione ottimale di DNA necessarie per la trascrizione in vitro. Generalmente, con Kit MEGAscript Ambion Trascrizione di alto rendimento (Ambion / Life Technologies, Grand Island, NY), 1 mg DNA linearizzato cede 40-60 ug di mRNA. Trascrizione in vitro viene effettuata seguendo le istruzioni del fabbricante (Ambion / Life Technologies, Grand Island, NY).
- Dopo la trascrizione in vitro l'mRNA viene purificato mediante precipitazione litio cloruro secondo le istruzioni del produttore (Ambion di MEGAscript alta Kit Trascrizione resa).
- integrità mRNA è ulteriormente verificato mediante elettroforesi su gel di acrilammide o agarosio.
2. Traduzione cellulare in vitro gratuito
Per la traduzione in vitro utilizzando R abbit R eticulocyte lisato, RRL (Promega, Madison, WI), attenersi alla seguente procedura:
- Preparare 100 pl in istruzioni del produttore traduzione in vitro reazione successivo. In breve, in acqua priva di nucleasi aggiungere 2 microlitri miscela di aminoacidi meno metionina (1 mM), 10 unità di inibitore della RNasi (Invitrogen / Life Technologies, Grand Island, NY), 20 uCi di sostanze radioattive [35 S]-metionina (Biomedicals MP, Solon, OH), 70 microlitri di lisato di reticolociti di coniglio (Promega, Madison, WI).
- Incubare °e reazione a 30 ° C per 5 min a caldo pre la reazione di traduzione. Aggiungere 2 ug di mRNA alla reazione di traduzione e incubare a 30 ° C per 5 min.
- Arrestare la reazione ponendo la reazione di traduzione in ghiaccio.
3. Isolamento di polipeptidi nascenti dal Reaction traduzione in vitro
- Per isolare polipeptide nascente associato a ribosomi (polisomi), reazione di traduzione è sovrapposto di 4,5 ml di glicerolo 30% in 10 mM Tris-HCl pH 7,6, contenente 100 mM KCl, 10 mM MgCl 2, e centrifugata a 100.000 xg in un rotore-110 TLA (Beckman Coulter, Inc., Brea, CA) per 1 ora a 4 ° C.
- Il supernatante viene rimosso attentamente ulteriormente.
- Il pellet contenente polysomal polipeptidi nascenti viene risospeso in un piccolo volume (10-15 pl) di 1 mM Tris-HCl pH 7,6, contenente 0,5 mg / ml ribonucleasi A (Invitrogen / Life Technologies, Grand Island, NY), e incubate per 30 min a 37 ° C. Perper migliorare l'idrolisi del peptidil-tRNA legame estere, NaOH viene aggiunto ad una concentrazione finale di 10 mM, e l'incubazione è continuata per ulteriori 30 min.
In vitro reazione traduzione assemblato senza mRNA e eseguite alle stesse condizioni (seguita da isolamento di peptidi nascenti come è stato descritto) servirebbe come controllo principale. Trattamento della reazione di traduzione (s) con puromicina prima di sottoporre l'estratto di centrifugazione in gradiente di densità può servire come un controllo aggiuntivo.
4. Risolvere il polipeptide nascente sulla Tris tricina SDS-PAGE
- I polipeptidi isolati nascenti vengono risolti e analizzati in Tris-tricina SDS-PAGE. Si noti che la quantità del materiale caricato sul gel dipende dalla efficienza di etichettatura proteine, l'efficienza di traduzione di proteine e molti altri parametri. La quantità di materiale caricata deve essere regolato empiricamente per consentire ai BESt visualizzazione e separazione delle singole catene polipeptidiche nascenti.
- Dopo gel elettroforesi sono fissati, essiccati sotto vuoto usando gel Dyer e sottoposti ad autoradiografia. La distribuzione dei polipeptidi nascenti sono osservate usando phosphoimaging.
5. Risultati rappresentativi
Bovina Gamma-B sonoro superiore cristallino è stato tradotto nel lisato di reticolociti di coniglio così come in E. coli S30 Sistemi estratto (Promega, Madison, WI) seguito da isolamento del nascenti catene polipeptidiche. figura 1 illustra fasi coinvolte nella isolamento di ribosoma catene legate nascenti. In questo studio lo scopo era quello di testare, se modificando l'ambiente del repertorio cioè traduzione di tRNA (noto per essere sostanzialmente diverso nei mammiferi (RRL) e procarioti (E. coli) sistemi a causa delle differenze di polarizzazione tra gli organismi codone) può alterare il movimento ribosoma lungo mRNA e effettuare la distribuzione dei siti di pausa di traduzione.Altered movimento ribosoma dovrebbe portare a cambiamenti nella distribuzione delle dimensioni di polipeptidi nascenti accumulano durante la traslazione, nonché la loro intensità relative. Intensità relativa delle bande riflettono la durata della pausa. I polipeptidi sono stati isolati e nascenti risolte 16,5% T, 6% C Tris-tricina SDS PAGE (Figura 2). Gamma-B sonoro superiore cristallino è una proteina di 20 kDa. L'osservazione di polipeptidi nascenti di non identici lunghezze e intensità in RRL ed E. sistemi coli indica che il movimento dei ribosomi lungo mRNA in questi due sistemi segue cinetiche traduzione differenti. Per esempio, abbiamo osservato un gruppo importante di 17,7 kDa in E. coli lisato e non nel sistema RRL, suggerendo che vi sia un ulteriore sito pausa traslazionale nel terminale 3 'della regione di mRNA (codifica regione C-terminale di Gamma sonoro superiore cristallino B), quando si traduce in E. coli sistema. Prendiamo atto che Gamma-B sonoro superiore cristallino porti tandem Arg codoni (AGG 153 e AGA 154) che sono frequenti nei sistemi di mammifero, ma sono noti per essere estremamente raro e lentamente tradotto in E. coli. un nuovo peptide nascente accumulando in E. coli del sistema (~ 17,7 kDa Figura 2.2) è probabilmente causato da una pausa ribosomiale a questi codoni tandem rari. Si nota che nessuna bande possono essere osservati in assenza di mRNA (Figura 2.3) e che il trattamento puromicina rimuove la maggior parte delle catene nascenti da polisomi (Figura 2.3). Il trattamento prolungato con puromicina (> 15 min) rimuove tutte le catene nascenti (dati non mostrati). Ciò conferma che i prodotti osservati polipeptidiche sono ribosoma nascenti catene di associati, piuttosto che mRNPs cosedimenting con polisomi.
Come menzionato sopra, è polarizzazione codone dell'organismo come pure il tessuto specifico. L'esempio rappresentativo qui descritto, indica chiaramente che modificando l'ambiente del repertorio cioè traduzione di tRNA / codonepolarizzazione può portare a modificare il movimento del ribosoma lungo l'mRNA. Evidentemente, questa semplice tecnica non può soltanto permettere l'identificazione dei siti di pausa traslazione lungo mRNA, ma permette anche rapido confronto dei siti di pausa la traduzione tra diversi sistemi.
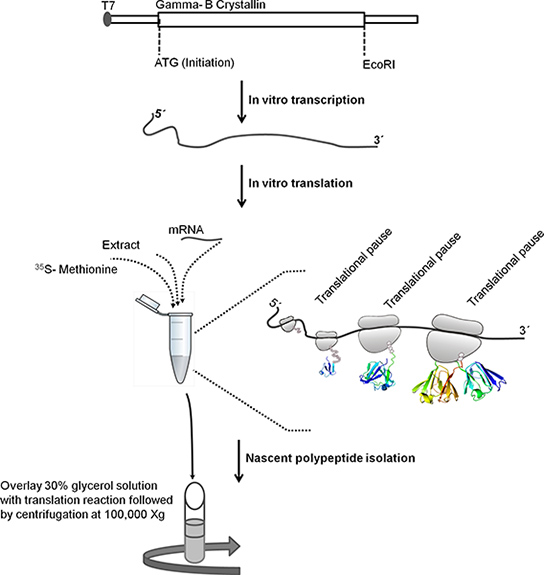
Figura 1. Schema che spiega i passaggi necessari per isolare ribosoma polipeptidi legati nascenti. Bovina Gamma-B sonoro superiore cristallino sequenza di ORF (528 paia di basi) è stato clonato a valle del promotore T7 in pBSKS +. Per la trascrizione in vitro del DNA stampo è stato linearizzato trattandolo con EcoRI. Modello linearizzato è stato utilizzato per la trascrizione in vitro. Promotore T7 nel modello del DNA è riconosciuta da T7 RNA polimerasi che trascrive la valle Gamma-B gene sonoro superiore cristallino. mRNA viene purificato utilizzando cloruro di litio metodo di precipitazione. L'mRNA purificato viene utilizzato per la traduzione in vitro. Seguitoincubazione, la reazione di traduzione è sovrapposto di soluzione di glicerolo 30% e centrifugato per 1 ora a 4 ° C a 100.000 X g per isolare ribosoma polipeptidi legati nascenti.
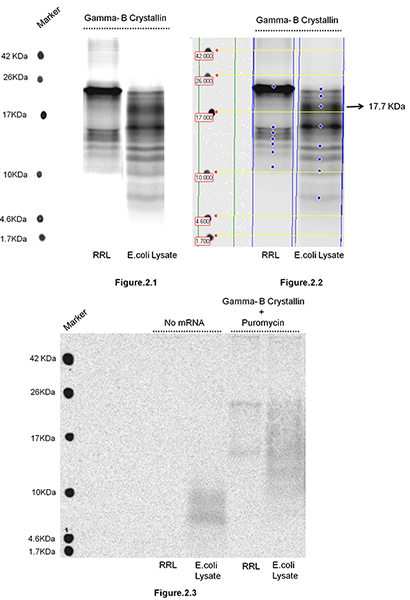
Figura 2. Isolamento dei bovini Gamma-B cristallina polipeptidi nascenti tradotti in Coniglio reticolociti lisato (RRL) e E. Lysate coli. 2 mRNA mcg è stato miscelato in 100 microlitri di reazione contenente RRL o E. coli S30 estratto System (Promega, Madison, WI) secondo le istruzioni del produttore con 20 uCi [35 S]-metionina e incubate a 30 ° C per 5 min. Dopo incubazione la reazione di traduzione è stato stratificato sopra di 4,5 ml di glicerolo 30% in 10 mM Tris-HCl pH 7,6, contenente 100 mM KCl, 10 mM MgCl 2, e centrifugato per 1 ora a 4 ° C e 100.000 g in X A-110 TLA rotore (Beckman Coulter, Inc., Brea, CA). Il pellet è stato isolato polyribosome Resutrascorsa in 20 pl di 1 mM Tris-HCl pH 7,6, contenente 0,5 mg / ml ribonucleasi A (Invitrogen / Life Technologies, Grand Island, NY) seguita da trattamento con NaOH (come descritto nella sezione protocollo). Polipeptidi nascenti sono stati risolti sul 16,5% T, C 6% Tris-Tricina gel SDS PAGE. Il gel è stato essiccato ed esposto per autoradiografia. Dopo l'esposizione il gel è stato analizzato utilizzando scanner Typhoon Imaging di GE Healthcare. La figura 2.1 mostra il gel con polipeptidi nascenti isolati e risolti dopo le reazioni di traduzione effettuato in due diversi sistemi (RRL ed Escherichia coli). Figura 2.2 Il gel è stato analizzato da Image Quant TL ., software v2005 e qui utilizzato come esempio per mostrare che il peso molecolare e l'intensità dei polipeptidi nascenti che si accumulano nei due sistemi possono essere facilmente analizzati e confrontati La figura 2.3 mostra due serie di controlli: polipeptidi nascenti isolati e risolti sulla PAGE SDS dopo la traduzione Le reazioni sono state assemblatesenza mRNA e / o dopo le reazioni di traduzione sono stati trattati con puromicina (5 min, conc finale. 1 mM) prima della reazione è stata sottoposta a centrifugazione.
Discussione
Per risultati riproducibili, la qualità e la concentrazione dei componenti utilizzati per la trascrizione in vitro e le reazioni di traduzione sono critiche. In questo studio abbiamo usato i kit disponibili in commercio e gli estratti che forniscono dati altamente riproducibili, se maneggiato con cura. Tuttavia, traduzione-competenti estratti possono essere preparati dalla cella di propria scelta, se necessario. Qualità di mRNA può influenzare la traduzione, quindi è della massima importanza per testare l&#...
Divulgazioni
Non ci sono conflitti di interesse dichiarati.
Riconoscimenti
Questo lavoro è stato finanziato dal programma Human Frontier Science concessione RGP0024.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente / Kit | Azienda | Numero di catalogo | |
| MEGAscript T7 alta Kit Trascrizione rendimento | Ambion | AM1333 | |
| Ribonucleasi Inhibitor | Invitrogen | 15518012 | |
| Trans [35 S]-Label | MP Biomedicals | 0151006 | |
| Ribonucleasi-A | Invitrogen | 12091 | |
| Coniglio reticolociti sistema Lysate, nucleasi trattato | Promega | L4960 | |
| Sistema di E. coli per i modelli S30 estratto lineari | Promega | L1030 | |
| Centrifugazione | Beckman Coulter | Optima TLX ultracentrifuga | |
| Stoccaggio fosforo autoradiography | GE Healthcare | Typhoon 9410 imager modalità variabile | |
| Software per l'analisi nascente polipeptide | GE Healthcare | Immagine Quant TL, v2005 |
Riferimenti
- Komar, A. A. A pause for thought along the co-translational folding pathway. Trends Biochem. Sci. 34, 16-24 (2009).
- Sharp, P. M., Cowe, E., Higgins, D. G., Shields, D. C. Codon usage patterns in Escherichia coli, Bacillus subtilis, Saccharomyces cerevisiae, Schizosaccharomyces pombe, Drosophila melanogaster and Homo sapiens; a review of the considerable within-species diversity. Nucleic Acids Res. 16, 8207-8210 (1988).
- Dittmar, K. A., Goodenbour, J. M., Pan, T. Tissue-specific differences in human transfer RNA expression. PLoS Genet. 2, e221 (2006).
- Ikemura, T. Codon usage and tRNA content in unicellular and multicellular organisms. Mol. Biol. Evol. 2, 13-34 (1985).
- Wolin, S. L., Walter, P. Ribosome pausing and stacking during translation of a eukaryotic mRNA. EMBO J. 7, 3559-3569 (1998).
- Krasheninnikov, I. A., Komar, A. A., Adzhubei, I. A. Nonuniform size distribution of nascent globin peptides, evidence for pause localization sites, and a cotranslational protein-folding model. J. Protein Chem. 10, 445-454 (1991).
- Komar, A. A., Lesnik, T., Reiss, C. Synonymous codon substitutions affect ribosome traffic and protein folding during in vitro translation. FEBS Lett. 462, 387-391 (1999).
- Komar, A. A., Jaenicke, R. Kinetics of translation of γ B crystallin and its circularly permutated variant in an in vitro cell-free system: possible relations to codon distribution and protein folding. FEBS Lett. 376, 195-198 (1995).
- Jha, S., Komar, A. A. Birth, life and death of nascent polypeptide chains. Biotechnol. J. 6, 623-640 (2011).
- Thanaraj, T. A., Argos, P. Ribosome-mediated translational pause and protein domain organization. Protein Sci. 5, 1594-1612 (1996).
- Kimchi-Sarfaty, C., Oh, J. M., Kim, I. W., Sauna, Z. E. A "silent" polymorphism in the MDR1 gene changes substrate specificity. Science. 315, 525-528 (2007).
- Schägger, H., von Jagow, G. Tricine-sodium dodecyl sulfate-polyacrylamide gel electrophoresis for the separation of proteins in the range from 1 to 100 kDa. Anal. Biochem. 166, 368-379 (1987).
- Shirole, N., Balasubramanian, S., Yanofsky, C., Cruz-Vera, L. Isolation of Translating Ribosomes Containing Peptidyl-tRNAs for Functional and Structural Analyses. J. Vis. Exp. (48), e2498 (2011).
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R. S., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324, 218-223 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon