Method Article
Reverse Genetics recupero mediato del Infettive Norovirus Murine
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
I Norovirus sono una delle principali cause di gastroenterite ancora tecniche molecolari per la loro caratterizzazione sono ancora relativamente nuovi. Riportiamo due diversi approcci di genetica inversa per il recupero efficiente dei norovirus murino (MNV), l'unico membro di questo genere che possono essere propagate in coltura cellulare.
Abstract
Norovirus umani sono responsabili della maggior parte dei casi di gastroenterite umana (GE) a livello mondiale e sono problema ricorrente in ambienti dove vicino da persona a persona di contatto non possono essere evitati 1, 2. Negli ultimi anni un aumento nell'incidenza di focolai negli ospedali è stata riportata, causando interruzioni significative per la loro capacità operativa nonché grandi perdite economiche. L'identificazione di nuovi approcci antivirali è stato limitato a causa dell'incapacità di norovirus umani per completare una infezione produttiva in coltura cellulare 3. L'isolamento recente di un norovirus murino (MNV), strettamente legato al norovirus umana 4, ma che può essere propagato in cellule 5 ha aperto nuove strade per lo studio di questi patogeni 6, 7.
Risultati replica MNV nella sintesi di nuovo genomico senso positivo e subgenomici molecole di RNA, l'ultimo dei quali corrisponde all'ultimo THIRd del genoma virale (Figura 1). MNV contiene quattro diversi telai di lettura aperti (ORF), di cui ORF1 occupa la maggior parte del genoma e codifica sette proteine non strutturali (NS1-7) rilasciati da un precursore poliproteina. ORF2 e ORF3 sono contenuti all'interno della regione subgenomico RNA e codificano le proteine del capside (VP1 e VP2, rispettivamente) (Figura 1). Recentemente, abbiamo identificato che ORF4 ulteriore sovrapposizione ORF2, ma in una cornice diversa lettura è funzionale e codifica per un fattore di virulenza mitocondriale localizzata (VF1) 8.
Replica di virus RNA senso positivi, compresi norovirus, avviene nel citoplasma risultante nella sintesi di nuovi livellate genomi di RNA. Per promuovere traduzione virale, i virus sfruttano diverse strategie volte al reclutamento del macchinario cellulare sintesi proteica 9-11. È interessante notare, traduzione norovirus è guidato dalla proteina virale multifunzionale-primer VPGlegata covalentemente al 5 'di entrambi RNA genomici e subgenomico 12-14. Questo sofisticato meccanismo di traduzione è probabile che sia un fattore importante nella scarsa efficienza di recupero virale tradizionali approcci di genetica inversa.
Riportiamo due diverse strategie basate sulla generazione di norovirus murino-1 (indicato come MNV qui allegata) trascritti limitato al 5 '. Uno dei metodi coinvolge sia sintesi in vitro e tappatura di RNA virale, mentre il secondo approccio implica la trascrizione di MNV cDNA in cellule esprimenti T7 RNA polimerasi. La disponibilità di questi sistemi genetica inversa per lo studio di MNV e un modello animale piccolo ha fornito una capacità senza precedenti per sezionare il ruolo di sequenze virali di replicazione e patogenesi 15-17.
Protocollo
1. RNA Trascrizione e tappatura per il recupero delle Infettive MNV
Questo protocollo è progettato per consentire un efficace recupero del MNV infettiva da cDNA tramite la trascrizione in vitro e la sua successiva tappatura in vitro (punto 1.1). Le trascrizioni risultanti vengono poi ricoperte trasfettati in cellule di recuperare infettiva MNV (punti 1.2 e 1.3). Questo approccio fornisce il metodo più sensibile per il recupero di MNV con rese tipiche in eccesso di 10 5 unità infettive per 35 mm di diametro)-piatto di cellule per MNV. Il protocollo è descritto di seguito:
Sintesi di 1,1 infettive trascritti MNV ridotte:
- Digerire il plasmide contenente il cDNA di tipo selvatico MNV (pT7: MNV 3'Rz) con Nhe I per ottenere DNA lineare Nhe I riconosce un sito unico di restrizione dopo l'estremità 3 'polyA coda MNV del genoma (Figura 2).. Plasmidi linearizzati sono tipicamente purificaticon l'utilizzo di colonne di silice (ad esempio GFX PCR Kit Gel DNA Banda Purificazione da GE Healthcare) ed eluito in H 2 O.
- In vitro trascrivere il vettore linearizzato usando T7 RNA polimerasi come descritto in precedenza 17. Molti kit commerciali sono disponibili per questo scopo e fornire un metodo riproducibile di grandi quantità di sintesi dell'RNA, come MEGAScript (Life Technologies) e RiboMAX (Promega). Le reazioni di trascrizione sono tipicamente DNAse digerito prima ulteriori analisi, ma in molti casi non è richiesto come purificazioni cloruro di litio come descritto qui di seguito non precipitare il DNA in modo efficiente.
- Analizzare una piccola aliquota della reazione di trascrizione di RNA, tipicamente 0,5 microlitri o meno, mediante elettroforesi su gel di agarosio per garantire la reazione di trascrizione ha funzionato in modo efficiente e RNA è full-length. Mentre molti utenti potrebbero voler eseguire denaturazione gel per dimensionare correttamente l'RNA, si utilizzano in genere non-denaturante su gel di agarosio electrophorESIS come un metodo rapido per analizzare l'integrità dell'RNA. Il genoma MNV come prodotta dal clone cDNA infettivo pT7: MNV 3'Rz funzionerà a circa 3 Kbp rispetto ad una scala in un dsDNA non denaturante gel di agarosio (Figura 3).
- Si consideri che altri metodi sono disponibili come alternativa per ottenere una rapida analisi di integrità dell'RNA, come utilizzando un bioanalyser Agilent (Figura 3).
- Si noti che la risoluzione gel poveri si possono incontrare se troppo RNA viene caricato. Fare molta attenzione per garantire il gel di agarosio viene preparato utilizzando RNasi-free i reagenti per evitare il degrado RNA durante l'elettroforesi che può influenzare la risoluzione band. Riscaldamento RNA a 65 ° C seguito da raffreddamento in ghiaccio può anche aiutare in alcuni casi.
- Purifica campione di RNA per rimuovere i nucleotidi prive di personalità giuridica. Molti metodi sono disponibili per questo approcci basati colonna di silice compreso tuttavia in questo protocollo si utilizzano in genere cloruro di litio come alternativa conveniente. PerA tal fine, aggiungere H 2 O a raggiungere un volume finale di 100 ul e quindi aggiungere 40 ul di soluzione di precipitazione cloruro di litio (7,5 M LiCl, 50 mM EDTA, pH 8,0, Ambion) e conservare il campione a -20 ° C per almeno 30 min.
- Pellet l'RNA mediante centrifugazione a 12.000 g X a 4 ° C per 15 min.
- Rimuovere il surnatante, facendo attenzione a non disturbare il pellet di RNA traslucido e lavarlo in 150 microlitri di etanolo al 70%. Centrifugare la provetta a 12.000 g X a 4 ° C per 15 min.
- Rimuovere l'etanolo e l'aria secca degli RNA, evitando il pellet si asciughi completamente come questo renderà difficile la risospensione.
- Poi, risospendere le trascrizioni MNV in 50-100 ml di soluzione di archiviazione RNA (Ambion). Si deve prestare attenzione per garantire che tutti gli RNA si è dissolta in modo corretto. Qualora le RNA sembrano difficile da sciogliere completamente, riscaldamento del campione a 60 ° C può aiutare a risospendere esso. Qualsiasi materiale insolubile deve quindi essere rimossa by centrifugazione prima di RNA quantificazione. Le trascrizioni sono purificati cappuccio e richiedono un ulteriore passo in vitro tappatura di essere infettiva (Sistema m7G ScriptCap Capping, Biotecnologie Epicentre).
- Quantificare l'RNA mediante spettrofotometria. A seconda della natura e l'entità della reazione di trascrizione, rese tipiche variano dal 50-150 pg di RNA per 100 pl reazione di trascrizione. L'integrità dell'RNA devono essere analizzate prima della reazione tappatura corto 100-300 ng di campione in un gel di agarosio 1% (Figura 3).
- Per migliorare l'efficienza di RNA tappatura, calore 60-70 ug di MNV trascritti di RNA a 65 ° C per 10 minuti e poi posizionare il tubo immediatamente in ghiaccio. Questo passaggio può ridurre qualsiasi effetto inibitorio della struttura RNA relativa al limite massimo. Pulse il tubo in una microcentrifuga refrigerati per raccogliere gocce formate durante la fase di riscaldamento.
- Preparare una miscela di reazione di tappatura, come suggerito dal produttore (ScriptCap m7GSistema di copertura, le Biotecnologie Epicentre). Brevemente, aggiungere 60-70 pg di RNA MNV ad un volume di reazione finale di 100 ul. La miscela di reazione incappucciamento può contenere 10 pl di 10 x tampone di tappatura (500 mM TrisHCl pH 8,0, 60 mM KCl, 12,5 mM MgCl2), 10 pl di 10 mM GTP, 0,5 pl di 20 mM S-adenosil metionina, 2,5 pl di Scriptguard (100 unità), e 4 pl di enzima Scriptcap (40 unità).
- Durante la reazione istituire, mantenere RNA trascritti sul ghiaccio per evitare il degrado. Mescolare bene la miscela di reazione e quindi incubare a 37 ° C per 1 h. Notare la dimensione di reazione può essere ridimensionato secondo la quantità di trascrizione capped richiesto.
- Purificare il RNA mediante precipitazione LiCl come spiegato in precedenza (vedi punto 1.1.6). Sciogliere il pellet in 50-100 pl di soluzione di conservazione dell'RNA (Ambion) e quantificare la quantità di RNA. Tipicamente, i campioni di RNA sono successivamente normalizzato a 1 pg / pl. Anche in questo caso, controllare tutte le RNA si è dissolto in modo corretto. Se non è stato correttamente dissolto, calore tegli campione a 60 ° C per consentire la dissoluzione. Rimuovere mediante centrifugazione eventuali materiale insolubile prima quantificazione RNA.
- Controllare l'integrità di RNA di nuovo prima di procedere con la fase di trasfezione. Per questo scopo, eseguire una quantità di 100-300 ng di campione in un gel di agarosio 1% (Figura 3).
1,2 Recovery di Neon-mediata trasfezione di RNA nelle cellule Raw264.7:
Per il recupero dei virioni infettivi MNV in una linea cellulare permissiva è possibile elettroporare la trascrizione capped MNV in Raw264.7 cellule usando Neon sistema di transfezione (Invitrogen). Raw264.7 sono cellule suscettibili all'infezione MNV, supportando vari cicli di replicazione del virus e la successiva re-infezione. Come risultato, rese tipiche si avvicinerà superiore a 10 5 unità infettive per ml a 24 ore dopo la trasfezione, ma picco a> 10 7 unità infettive dopo 48 ore.
- Un giorno prima transfection, semi Raw264.7 cellule a circa il 50% di confluenza. Tipicamente due palloni T75 di cellule sono necessari per 3 trasfezioni.
- Il giorno della transfezione, raschiare i monostrati cellulari in Eagle modificato da Dulbecco (DMEM) contenente 10% siero di vitello fetale (FCS), assicurando si genera una sospensione di cellule singole pipettando ripetute.
- Determinare la concentrazione di cellule vitali in un emocitometro con esclusione trypan blu per etichettare le cellule non vitali.
- Agglomerare le cellule a 1.200 X g per 5 min e risospendere in DMEM contenente 10% FCS ad una concentrazione finale di 8 x 10 6 cellule / ml.
- Poco prima della trasfezione, aliquota 1 ml di cellule per trasfezione e pellet loro a 1.200 X g per 2 min. Togliere la carta e lavare le cellule in 500 microlitri di PBS (senza Mg 2 + / Ca 2 +). Centrifugare le cellule nuovamente a 1200 X g per 2 min. Nota è consigliabile mantenere le cellule in DMEM più a lungo possibilecome memoria in PBS per lunghi periodi di tempo può compromettere la vitalità cellulare e il tasso di trasfezione.
- Rimuovere PBS da tubi e aggiungere 130 microlitri di soluzione di risospensione (Neon kit sistema di transfezione, Invitrogen) ad una concentrazione finale di 6 x 10 7 cellule / ml. Si deve prestare attenzione a sospendere nuovamente le cellule evitando la formazione di bolle che provocano scintille durante la trasfezione e cellula di sopravvivenza compromesso.
- Aggiungere la quantità appropriata di trascrizione ridotta MNV alle cellule (Figura 2), generalmente 1,3 pg di RNA ridotta viene aggiunto a 130 pl di cellule risospese e agitarli. Quindi, raccolgono 100 pl della miscela nella punta 100 pl trasfezione Neon. Particolare attenzione deve essere adottate per garantire che non si formano bolle nella cella elettroporazione (Neon kit sistema di transfezione punta di 100 pl) in quanto ciò potrebbe provocare il guasto di questo esperimento.
- Elettroporare le cellule utilizzando un singolo impulso a 1700 V per 25 msec, enSuring assenza di scintille durante il pulsante che indica la presenza di bolle nel campione. Nel caso scintille dovrebbe avvenire, scartare il campione e ripetere la trasfezione. Rilasciare le cellule in una provetta Eppendorf contenente 1 ml di antibiotici libero DMEM contenente 10% FCS. Nota ogni punta può essere riutilizzato fino a tre volte con lo stesso campione di RNA se un maggior numero di cellule trasfettate sono richiesti.
- Successivamente, distribuire le cellule del tubo in pozzetti indipendenti contenenti una quantità appropriata di preriscaldato antibiotici libero DMEM contenente 10% FCS. Come guida generale, 150 microlitri della sospensione cellulare generato durante la fase 1.2.8 sono sufficienti per un singolo pozzetto di una piastra 24-piatto contenente 0,5 ml di DMEM preriscaldato, mentre 300 microlitri sono appropriate per un pozzetto di una piastra 12-piatto contenente 1 ml di DMEM preriscaldato.
- Incubare le cellule a 37 ° C e 10% di CO 2 per 24 a 72 ore. Quindi, rilasciare virioni infettivi da cellule da uno (o più)cicli di gelo e disgelo e determinare titolo virus nel campione utilizzando saggio di placca o TCID50. Si noti che i lisati possono essere chiariti mediante centrifugazione per 1-2 minuti a velocità massima o con la loro filtrazione attraverso un filtro 0,22 micron pori prima titolazione. Tipicamente, MNV raggiunge titoli di circa 1 x 10 6 TCID50/ml a 24 ore dopo la trasfezione e fino a 1 x 10 9 a 72 ore dopo la trasfezione.
- La presenza e la stabilità di mutazioni introdotte in pT7: 3'Rz MNV sono tipicamente determinata mediante sequenziamento dei virus recuperati dopo 2 a 5 passaggi aggiuntivi in Raw264.7 cellule.
1,3 Recovery da lipofezione in BHK-21 celle:
Un metodo più diretto e spesso più conveniente per il recupero di MNV infettiva da trascritti tappo è tramite lipofezione (Lipofectamine 2000, Invitrogen). Dato che le cellule Raw264.7 sono difficili per trasfettare utilizzando approcci basati su lipidi che normalmente fanno uso di OTHer facile per trasfettare linee cellulari, quali BHK-21, che è una linea immortalizzata derivata da bambino fibroblasti renali di criceto. Come approccio standard nel nostro laboratorio usiamo BSR-T7 cellule, un derivato della linea di cellule BHK-21, come mentre queste cellule sono facili da transfettare e sostenere la replicazione MNV, mancano di un recettore adatto a consentire vari cicli di reinfezione . Come risultato, la resa virus generata da questo sistema è un'indicazione di un singolo ciclo di replicazione del virus. Questo approccio è particolarmente utile quando esamina l'effetto della mutazione sul recupero virus quanto consente trasfezioni multipli da eseguire a costi notevolmente ridotti rispetto al Neon trasfezione mediata e inoltre non richiede apparecchiature speciali. Vale la pena notare che le altre linee cellulari già disponibili come embrionali umane cellule renali 293T supporta anche un efficace recupero del MNV comunque in condizioni di trasfezione deve prima essere ottimizzato per garantire un'efficace erogazione RNA.
- TripsinaISE un monostrato di cellule BHK-21 (o BSR-T7 cellule), i semi di 7,5 x 10 5 cellule in un piatto di diametro 35 mm in antibiotico mezzi privi di crescita e incubare le cellule a 37 ° C con 10% di CO 2 durante la notte. Raddoppiare la quantità di cellule in ciascuna piastra se le trasfezioni sono previste per lo stesso giorno della semina, e permettere alle cellule di aderire alla piastra per 2-3 ore a 37 ° C con 10% CO 2. Si noti che altre cellule che sono adatti a questo approccio includono cellule 293T, umane carcinoma epatocellulare Huh7 cellule di scimmia verde africana e COS7 cellule.
- Rimuovere il supporto dalle cellule e sostituire con 3 ml di terreno fresco senza antibiotici per garantire la massima efficienza di trasfezione.
- Preparare una miscela di 1-2 ug di trascrizione ridotta MNV in 100 pl di Opti-MEM (Invitrogen) e miscelare con 4 pl di Lipofectamine 2000 precedentemente miscelata in 100 pl di Opti-MEM. Mescolare il campione accuratamente pipettando su e giù 15 volte. Lasciarela miscela a temperatura ambiente per 20 minuti.
- Aggiungere i complessi di transfezione contenenti trascrizioni MNV innevate in una goccia a goccia di moda per il monostrato cellulare e scuotete delicatamente la piastra in direzioni perpendicolari.
- Incubare le cellule a 37 ° C e 10% di CO 2 per 24 a 72 ore. In seguito, rilasciare virioni infettivi dalle cellule dal congelamento e lo scongelamento e determinare titolo virus mediante saggio di placca o TCID50. Le rese di circa 1 x 10 6 TCID50/ml sono raggiunti.
2. Recupero diretto di MNV infettiva da cDNA in cellule che esprimono T7 RNA polimerasi
Questo protocollo è progettato per consentire il recupero di MNV nelle cellule mediante la trascrizione di un plasmide infettiva ospitare la sequenza genomica completa cDNA da una polimerasi T7 espresso nelle cellule. Linee cellulari differenti possono essere utilizzati per ripristinare MNV infettiva da questo approccio, anche se tipicamente si ottengono i massimi rendimenti con BHK-21 e BSR-T7 ceLLS 15. Usiamo tipicamente BSR-T7 in quanto le cellule che crescono più velocemente del clone parentale linea BHK. Le cellule sono infettate con fowlpox (FPV) codifica per RNA polimerasi T7 (FPV-T7) 18 che funziona come un virus helper per guidare l'espressione di RNA virale e successivo recupero del virus infettivo (Figura 4). Sebbene BSR-T7 cellule esprimono costitutivamente T7 RNA polimerasi, questa espressione non è sufficiente a recuperare MNV infettiva dopo la trasfezione di pT7: MNV 3'Rz in assenza di helper FPV-T7. Mentre le rese tipiche di questo sistema sono almeno 10 volte inferiore a quelle sopra descritte, questo approccio fornisce un metodo rapido di mutanti di screening per consentire l'identificazione di mutazioni debilitanti. In genere questo metodo viene utilizzato prima per valutare la fattibilità di un costrutto cDNA. In caso di costruire o non riescono a produrre virus infettivo o sembrano cedere virus a livelli inferiori rispetto a quella del clone wild type infettiva, allora la base di RNA avvicinamench sopra descritto è intrapresa.
- Trypsinise un monostrato di cellule BHK-21 (o cellule BSR-T7) e sementi di 7,5 x 10 5 cellule in un piatto da 35 mm di antibiotici terreni di crescita liberi e incubare le cellule a 37 ° C e 10% CO 2 durante la notte. Aggiungere il doppio della quantità di cellule in ciascuna piastra se le trasfezioni sono previste per lo stesso giorno della semina, e permettere alle cellule di aderire alla piastra per 2-3 ore a 37 ° C e 10% CO 2.
- Rimuovere terreni di coltura cellulare e aggiungono 700 pl di FPV-T7 a ciascun pozzetto (Figura 4). Una molteplicità di infezione (MOI) di ~ 0,5 PFU per cellula, sulla base di titolazioni su fibroblasti primari embrionali di pollo, viene generalmente usato. Tuttavia è importante notare che nuove preparazioni di virus helper coltivati in fibroblasti primari sono funzionalmente titolati per determinare la dose necessaria per il recupero del virus efficiente. Protocolli per la propagazione e la titolazione di FPV-T7 sono stati precedentemente descritti 18.
- Incubate a 37 ° C e 10% di CO 2 per 1 ora per consentire FPV-T7 per infettare le cellule. Quindi, aggiungere 2 ml di antibiotici libero DMEM contenente 10% FCS e incubare le cellule per un'ora aggiuntiva a 37 ° C e 10% di CO 2 per consentire l'espressione T7 RNA polimerasi.
- Per procedere con la trasfezione del plasmide infettiva, in primo luogo rimuovere il supporto dalle cellule infettate, lavare con 2 ml di supporto (10% FCS in DMEM privo di antibiotici), e infine coprire il monostrato cellulare con 3 ml di mezzo. Gli antibiotici non dovrebbero essere aggiunti ai mezzi perché può interferire con l'efficacia di Lipofectamine 2000 (Invitrogen).
- Preparare una miscela di 1 ug di tipo selvatico MNV cDNA infettiva plasmide (es pT7: MNV 3'Rz) in 100 pl di Opti-MEM (Invitrogen) e miscelare con 4 pl di Lipofectamine 2000 (Invitrogen), precedentemente miscelata con 100 pl di Opti-MEM (Invitrogen). Mescolare accuratamente la reazione pipettando su e giù 15 volte e mantenere la miscela a temperatura ambienteratura per 20 minuti.
- La miscela di trasfezione risultante dovrebbe essere poi aggiunta goccia a goccia per il monostrato di cellule e la piastra deve essere delicatamente agitata in direzioni perpendicolari.
- Incubare la FPV-T7 infetto, MNV plasmide transfettate cellule a 37 ° C e 10% di CO 2 per 24 a 72 ore. Cellule trasfettate con infettiva plasmide pT7: MNV 3'Rz normalmente rendering titoli da 1 x 10 4 a 5 x 10 4 TCID50/ml.
3. Risultati rappresentativi
Entrambi gli approcci genetica inversa sono altamente efficace per il recupero di MNV infettivo nelle cellule in coltura, come mostrato nella Figura 5. MNV infettiva con titoli superiori a 10 5 TCID50/ml vengono recuperati a 24 ore dopo la trasfezione di RNA in capped MNV Raw264.7 cellule. Analogamente, la trasfezione di malattie infettive plasmide pT7: MNV 3'Rz in BSR-T7 cellule precedentemente infettate con helper FPV esprimere T7 (FPV-T7) ha portato a titoli virali in gran parte eccedenti gliDing 10 4 TCID50/ml (Figura 5). Questi valori titolo virale ottenuti con RNA sintetici e molecole di DNA sono simili a quelli ottenuti in trasfezioni coinvolgono naturali VPG-linked RNA isolato da virioni infettivi nelle stesse cellule (Figura 5). Questi risultati evidenziano l'elevata efficienza degli approcci di genetica inversa descritte qui per recuperare geneticamente definito varianti MNV in coltura cellulare.
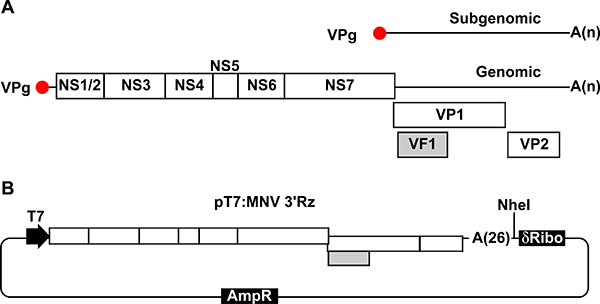
Figura 1. Illustrazione del genoma MNV e plasmide per il recupero del virus infettivo. A, Rappresentazione schematica di MNV genoma dell'organizzazione. Ogni proteina regione codificante è illustrato come un unico bianco. ORF1 viene tradotto in 7 differenti proteine non strutturali (NS1 / 2 a NS7) che vengono rilasciate da poliproteina precursore dopo auto-proteolitica elaborazione. ORF 2 codifica la principale proteina capside VP1, ORF 3 codifica il tappo minoresid proteine VP2 e ORF4 sovrapposizione con ORF2 regione codificante codifica fattore di virulenza VF1. RNA genomici e subgenomico contengono una coda polyA a loro estremità 3 'di lunghezza variabile. B, plasmide contenente MNV cDNA usato nei nostri approcci di genetica inversa (pT7: MNV 3'Rz). CDNA MNV è fusa ad una coda polyA di 26 residui nella sua estremità 3 '. La sequenza di cDNA MNV si trova immediatamente a valle di un tronco di sequenza del promotore T7, per consentire T7-driven trascrizione, ed a monte di un unico Nhe I sito e una sequenza di DNA codificante per un auto-scissione ribozima dopo. Queste sequenze sono strumentali per garantire terminazione della trascrizione di RNA genomico subito dopo la presente coda polyA alla estremità 3 '.
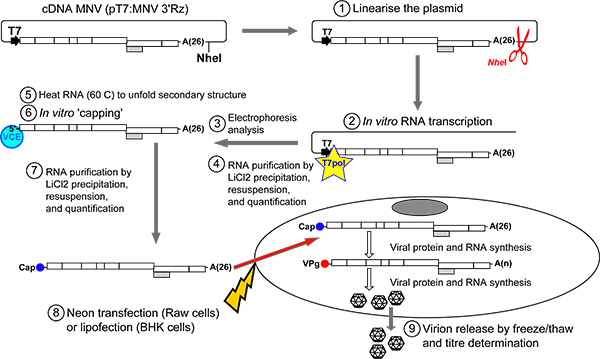
Figura 2. Panoramica del protocollo per il recupero delle malattie infettive MNV da RNA trascritto e tappati in vitro Il plasmide pT7:. MNV 3'Rz è linearizzata immediatamente a valledella sequenza genomica MNV usando enzima di restrizione Nhe I (passo 1). Dopo purificazione del DNA, MNV trascritti di RNA sono generati in vitro usando T7 RNA polimerasi (step 2). Prodotti di trascrizione di solito eseguita con una mobilità apparente di 2,5-3Kb su un non denaturante 1% gel di agarosio (passaggio 3, Figura 3). Il DNA stampo viene eliminata l'impiego di una DNAsi RNAsi-free. RNA viene poi purificato da nucleotidi liberi da LiCl precipitazione (passaggio 4). Il prodotto di RNA può quindi essere limitato in vitro dopo essere stato precedentemente riscaldata a 65 ° C a svolgersi secondari strutture di RNA (passaggi 5-6). Dopo purificazione da LiCl precipitazione, RNA è transfettato in sia Raw264.7 cellule (sistema di transfezione Neon, Invitrogen) o BSR-T7 cellule (Lipofectamine 2000, Invitrogen) (passi 7-8). Una volta all'interno della cellula, capped trascritti di RNA saranno tradotte in proteine virali che catalizzano la replicazione virale in nuove trascrizioni MNV molecole di RNAcontenente una molecola corretta VPG alla fine il loro 5 '. Successivi cicli di replicazione virale accompagnata di traduzione genererebbe un gran numero di genomi virali che saranno incapsidati per generare virioni infettivi. Per facilitare la liberazione del virus dalle cellule, uno o più cicli di congelamento e scongelamento sono eseguite (passo 9). Rese virali possono essere quindi determinato dalla TCID50 o procedure di analisi della placca.
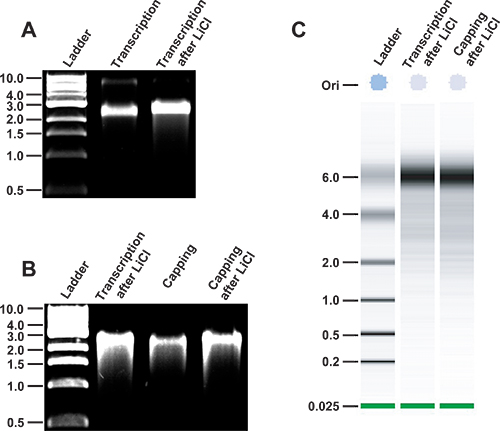
Figura 3. L'analisi di integrità MNV RNA trascritti lungo il protocollo. A, integrità di MNV RNA sintetizzato in vitro. Il plasmide pT7: MNV 3'Rz viene dapprima linearizzato usando enzima di restrizione Nhe I. Dopo purificazione del DNA, MNV trascritti di RNA sono generati in vitro usando T7 RNA polimerasi (corsia 2). RNA è quindi purifi ed da nucleotidi liberi da LiCl precipitazione (corsia 3). I prodotti della trascrizione sono gestiti in modo non-denaturante gel di agarosio all'1% in parallelo a 1-Kb DNA ladder (New England Biolabs, corsia 1). Mobilità relativa di trascritti virali condizioni non denaturanti condizioni è simile a un prodotto di dsDNA 2,5-3 Kb. B, Integrità del MNV trascritti di RNA dopo la riduzione. Trascritti MNV purificati precedentemente da LiCl precipitazione (corsia 2) sono sottoposti a tappatura enzimatica (corsia 3) e purificazione mediante precipitazione LiCl (corsia 4). C,, analisi in un chip Agilent RNA 6000 Nano di trascrizioni MNV (seconda corsia) e sigillato trascrizioni MNV (terza corsia), che sono stati precedentemente precipitati in LiCl. Una scala ssRNA viene eseguito in parallelo.
= "Pdflinebreak"> 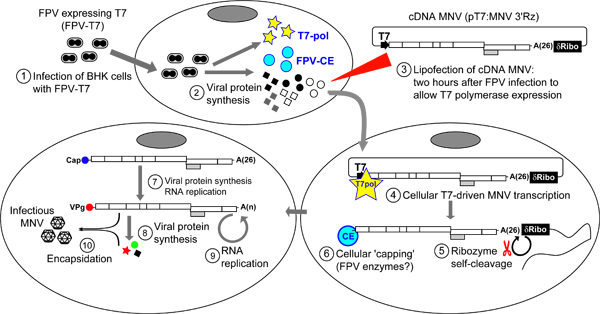
Figura 4. Panoramica del protocollo per il recupero di MNV infettiva da cDNA. Inizialmente, BSR-T7 (o BHK) le cellule sono infettate con un virus ricombinante fowlpox (FPV) che esprime il batteriofago T7 RNA polimerasi (FPV-T7) (fase 1). Le cellule infette sono incubate per 2 ore prima di ulteriore trattamento per consentire l'espressione di proteine FPV ricombinante che comprende la T7 RNA polimerasi (step 2). Successivamente, pT7: MNV 3'Rz è trasfettato nelle cellule usando Lipofectamine 2000 (Invitrogen) (fase 3). Una volta all'interno della cellula, pT7: MNV 3'Rz è riconosciuta dalla polimerasi T7 RNA che sintetizza MNV trascritti di RNA (passaggio 4). La presenza di un auto-congiunzione δ-Ribozyme sequenza al 3 'del genoma garantisce la trascrizione 3' terminus si trova subito dopo la coda polyA (fase 5). Alcuni trascritti virali sono intracellulare ricoperto da un enzima FPV tappatura (fase 6). Le trascrizioni risultanti MNV innevate sarà tradotto per generare le proteine che catalizzano MNV MNV replica trascrizioni. Neosintetizzate MNV molecole di RNA contenenti una molecola corretta VPG alla loro estremità 5 'subirebbe successivi cicli di replicazione virale accompagnata da traduzione che possono in definitiva portare alla generazione di virus infettivo incapsidati. Per facilitare la liberazione del virus dalle cellule, uno o più cicli di congelamento e scongelamento sono eseguite (fase 7). Rese virali possono essere quindi determinato dalla TCID50 o procedure di analisi della placca.
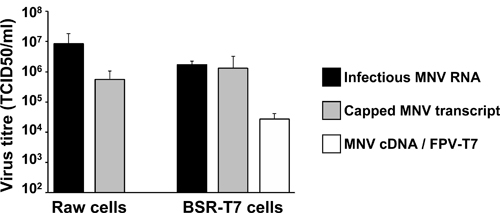
Figura 5. Risultati rappresentativi di titoli virali ottenuti da diverse genetica inversa avvicina descritto nel testo. Barre grigie rappresentano i titoli virali ottenuti a 24 ore dopo Neon-Transfezione di 2 x 10 6 cellule Raw264.7, o dopo lipo-trasfezione di 2 x 10 6 BSR-T7 cellule con trascritti in vitro e tappati MNV RNA. Barre bianche rappresentano il titolo virale tipicamente ottenuta dopo lipofezione di pT7: MNV 3'Rz (MNV cDNA) in 2 x 10 6 cellule BSR-T7 precedentemente infettati per 2 ore con virus ricombinante che esprimono fowlpox polimerasi T7 (FPV-T7). Come controllo positivo per la transfezione in Raw e BSR di T7cells, si utilizzano in genere 2 ug di RNA estratto da cellule infettate con MNV che contengono alti livelli di VPG-linked MNV RNA. I controlli negativi sono state effettuate con uno o RNA MNV pT7: MNV 3'Rz codificante una mutazione frameshift (F / S) che abolisce la replica, con conseguente nessun virus rilevabile (dati non mostrati).
Discussione
Qui abbiamo illustrato due differenti approcci inverse genetici che permettono il recupero di MNV infettiva in coltura cellulare. Sia avvicina efficacemente bypassare il requisito assoluto per il collegamento covalente del VPG alla estremità 5 'del genoma di RNA virale attraverso la generazione del ridotte trascritti MNV che vengono poi riconosciuti dai ribosomi cellulari. Trascrizione in vitro seguita da enzimaticamente tappatura è più efficiente in il recupero di MNV virulenta della trascrizione di plasmidi infettive in cellule esprimenti T7 RNA polimerasi, in cui i trascritti può essere chiuso da gli enzimi FPV tappatura. Titoli virali recuperati con questi sistemi genetica inversa sono simili a quelle ottenute mediante trasfezione di virali VPG-linked RNA purificati da colture di cellule infette 17 (Figura 5). La transfezione di capped MNV RNA in cellule permissive Raw264.7 rende un titolo virus solo l'1-log inferiore esperimenti di transfection di RNA totale da cellule infette contenenti virale VPG-linked RNA (Figura 5). Questo fatto incoraggia ulteriori indagini per determinare se l'aggiunta di una molecola VPG alla estremità 5 'di trascritti generati da questi sistemi può comportare un aumento rese virali che possono rivelare aspetti funzionali sottostanti infettività MNV in cellule associate a VPG. Tuttavia, consideriamo questo sistema genetica inversa come una altamente efficiente comparabile ad altri virus RNA genetica inversa sistemi attualmente utilizzati in cui l'RNA trascritto in vitro permette il recupero dei titoli solo 10-100 inferiori nelle infezioni reali con virioni 19, 20.
Nel complesso, le metodologie attuali costituiscono un significativo passo avanti nel campo della biologia molecolare e norovirus fornirci gli strumenti per indagare i ruoli funzionali di proteine e RNA motivi conservati nei genomi di norovirus. Questi approcci sono già stati combinati con cmodello di topo ATTUALE a disposizione e hanno dimostrato che MNV recuperato da cDNA infettiva è in grado di causare l'infezione letale di> 80% STAT1-/ - topi in meno di 10 giorni 4, 21. Facendo uso di questo sistema abbiamo recuperato mutanti vitali norovirus murini della proteina del capside e di un tratto polypyrimidine coinvolto nel legame di fattori diversi host (PTB e PCBP) che mostrano un fenotipo alquanto attenuati in vivo 21, 22. Inoltre, abbiamo recentemente dimostrato che i virus che mancano della capacità di esprimere la proteina da VF1 ORF4 efficiente replicarsi nelle cellule in coltura, ma ancora una volta hanno ridotto la virulenza nei topi rispetto al WT MNV 8. Questi studi ci incoraggiano a progettare versioni attenuate di norovirus umani sulla base di studi MNV che potrebbero essere indagati come potenziali candidati vaccini.
Divulgazioni
Non abbiamo nulla da rivelare.
Riconoscimenti
Questa ricerca è stata finanziata da una borsa di studio Wellcome Trust senior assegnato a Ian Goodfellow, e Marie Curie Intra European Fellowship (7 ° PQ Consiglio europeo della ricerca) ha assegnato ad Armando Arias. Vorremmo ringraziare Io Hong Cheong, il dottor Rebecca Robey e il dottor Mike Skinner per averci dato il permesso di utilizzare il loro bioanalyser Agilent e contribuendo con l'esecuzione campioni di RNA.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reattivo | Azienda | Numero di catalogo | |
| Soluzione di cloruro di litio precipitazioni | Ambion | AM9480 | |
| RNA soluzione di storage | Ambion | AM7000 | |
| MEGAscript T7 sceneggiatura | Ambion | AM1333 | |
| ScriptCap m7G Capping sistema | Epicentre Biotecnologie | SCCE0610 | |
| Trasfezione sistema Neon | Invitrogen | MPK5000 | |
| Neon trasfezione kit sistema | Invitrogen | MPK1025 | |
| Opti-MEM I | Invitrogen | 31985070 | |
| Lipofectamine 2000 Transfection Reagent | Invitrogen | 11668-027 | |
| Agilent RNA 600 Nano kit | Agilent | 5067-1511 | |
| Agilent 2100 bioanalyzer | Agilent | G2939AA | |
| Illustra GFX PCR DNA e Kit Banda Gel Purificazione | GE Healthcare | 28-9034-70 | |
| RiboMAX Large Scale Production System-T7 RNA | Promega | P1300 |
Riferimenti
- Outbreaks of gastroenteritis associated with noroviruses on cruise ships--United States. MMWR Morb. Mortal. Wkly. Rep. 51, 1112-1115 (2002).
- Lopman, B. A. Epidemiology and cost of nosocomial gastroenteritis, Avon, England, 2002-2003. Emerg. Infect. Dis. 10, 2002-2003 (2004).
- Duizer, E. Laboratory efforts to cultivate noroviruses. J. Gen. Virol. 85, 79-87 (2004).
- Karst, S. M., Wobus, C. E., Lay, M., Davidson, J., Virgin, H. W. T. STAT1-dependent innate immunity to a Norwalk-like virus. Science. 299, 1575-1575 (2003).
- Wobus, C. E. Replication of Norovirus in cell culture reveals a tropism for dendritic cells and macrophages. PLoS Biol. 2, e432(2004).
- Bok, K. Inhibition of norovirus replication by morpholino oligomers targeting the 5'-end of the genome. Virology. 380, 328-337 (2008).
- Kim, Y., Thapa, M., Hua, D. H., Chang, K. O. Biodegradable nanogels for oral delivery of interferon for norovirus infection. Antiviral Res. 89, 165-173 (2011).
- McFadden, N. Norovirus Regulation of the Innate Immune Response and Apoptosis Occurs via the Product of the Alternative Open Reading Frame 4. PLoS Pathog. 7, e1002413(2011).
- Kormelink, R., van Poelwijk, F., Peters, D., Goldbach, R. Non-viral heterogeneous sequences at the 5' ends of tomato spotted wilt virus mRNAs. J. Gen. Virol. 73, 2125-2128 (1992).
- Shuman, S., Hurwitz, J. Mechanism of mRNA capping by vaccinia virus guanylyltransferase: characterization of an enzyme--guanylate intermediate. Proc. Natl. Acad. Sci. U.S.A. 78, 187-191 (1981).
- Jang, S. K., Pestova, T. V., Hellen, C. U., Witherell, G. W., Wimmer, E. Cap-independent translation of picornavirus RNAs: structure and function of the internal ribosomal entry site. Enzyme. 44, 292-309 (1990).
- Chaudhry, Y. Caliciviruses differ in their functional requirements for eIF4F components. J. Biol. Chem. 281, 25315-25325 (2006).
- Goodfellow, I. Calicivirus translation initiation requires an interaction between VPg and eIF 4. E. EMBO Rep. 6, 968-972 (2005).
- Herbert, T. P., Brierley, I., Brown, T. D. Identification of a protein linked to the genomic and subgenomic mRNAs of feline calicivirus and its role in translation. J. Gen. Virol. 78 ( Pt5 ), 1033-1040 (1997).
- Chaudhry, Y., Skinner, M. A., Goodfellow, I. G. Recovery of genetically defined murine norovirus in tissue culture by using a fowlpox virus expressing T7 RNA polymerase. J. Gen. Virol. 88, 2091-20100 (2007).
- Ward, V. K. Recovery of infectious murine norovirus using pol II-driven expression of full-length cDNA. Proc. Natl. Acad. Sci. U.S.A. 104, 11050-11055 (2007).
- Yunus, M. A., Chung, L. M., Chaudhry, Y., Bailey, D., Goodfellow, I. Development of an optimized RNA-based murine norovirus reverse genetics system. J. Virol. Methods. 169, 112-118 (2010).
- Arias, A., Perales, C., Escarmis, C., Domingo, E. Deletion mutants of VPg reveal new cytopathology determinants in a picornavirus. PLoS One. 5, e10735(2010).
- Werf, S. vander, Bradley, J., Wimmer, E., Studier, F. W., Dunn, J. J. Synthesis of infectious poliovirus RNA by purified T7 RNA polymerase. Proc. Natl. Acad. Sci. U.S.A. 83, 2330-2334 (1986).
- Bailey, D., Thackray, L. B., Goodfellow, I. G. A single amino acid substitution in the murine norovirus capsid protein is sufficient for attenuation in vivo. J. Virol. 82, 7725-7728 (2008).
- Bailey, D. Functional analysis of RNA structures present at the 3' extremity of the murine norovirus genome: the variable polypyrimidine tract plays a role in viral virulence. J. Virol. 84, 2859-2870 (2010).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon