È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Realizzazione e utilizzo di microarrays microambiente (MEArrays)
In questo articolo
Riepilogo
Un metodo combinatorio screening funzionale per ottenere approfondimenti sugli impatti della composizione molecolare di microambienti su funzioni cellulari è descritto. Il metodo si avvale di microarray esistenti basate su tecnologie per generare matrici di microambienti definite combinatorie che supportano l'adesione cellulare e analisi funzionale.
Abstract
Le interazioni tra le cellule e il loro micro circostante avere conseguenze funzionali per comportamento cellulare. A livello di singola cellula, microambienti distinti possono imporre differenziazione, migrazione e proliferazione fenotipi, e sul livello dei tessuti del microambiente processi complessi come morfogenesi e tumorigenesi 1. Non solo la cella e il contenuto molecolare di impatto microambienti delle cellule all'interno, ma lo fanno anche l'elasticità 2 e geometria 3 del tessuto. Definito come la somma totale di cellula-cellula,-ECM, e-solubili interazioni di fattori, oltre alle caratteristiche fisiche, il microambiente è complessa. I fenotipi di cellule all'interno di un tessuto sono in parte a causa del loro contenuto di genomica e in parte a causa delle interazioni con il microambiente combinatorie. Una delle maggiori sfide è quello di collegare specifiche combinazioni di componenti microambientali con comportamenti distintivi. ent "> Qui, vi presentiamo il microambiente microarray (MEArray) piattaforma per il cell-based screening funzionale di interazioni con microambienti combinatorie 4. Il metodo consente il controllo simultaneo della composizione molecolare e il modulo elastico, e combina l'uso di microarray ampiamente disponibili e tecnologie micropatterning. schermi MEArray richiedono da un minimo di 10.000 cellule per matrice, che facilita gli studi funzionali di tipi di cellule rare, come le cellule progenitrici adulte. Un limite di questa tecnologia è che microambienti tessuto intere che non può essere completamente ricapitolato su MEArrays. Tuttavia, il confronto di risposte nel tipo di cellula stessa a numerosi microambienti correlati, ad esempio le combinazioni a coppie delle proteine ECM che caratterizzano un determinato tessuto, fornirà approfondimenti come i componenti microambientali suscitare fenotipi funzionali tessuto-specifiche.
MEArrays può essere stampato usando una varietà di ricombinante GROfattori wth, citochine e proteine purificate ECM, e loro combinazioni. La piattaforma è limitato solo dalla disponibilità di reagenti specifici. MEArrays sono suscettibili di analisi e ritardi, ma più spesso vengono utilizzati per l'analisi del punto finale di funzioni cellulari che sono misurabili con sonde fluorescenti. Per esempio, la sintesi del DNA, apoptosi, acquisizione di stati differenziati, o di prodotti genici specifici sono comunemente misurata. Brevemente, il flusso di base di un esperimento MEArray è preparare vetrini rivestiti con substrati di stampa e di preparare la piastra master di proteine che devono essere stampati. Quindi gli array sono stampati con un robot microarray, le cellule sono fatte attaccare, crescere in coltura, e quindi sono chimicamente fissato al raggiungimento del punto finale sperimentale. Saggi fluorescenti o colorimetrico, ripreso con microscopi tradizionali o scanner microarray, sono utilizzati per rivelare importanti fenotipi molecolari e cellulari (Figura 1).
Protocollo
1. Preparazione di stampa Substrati
La decisione di utilizzare polidimetilsilossano (PDMS) rivestita o poliacrilammide (PA) rivestite vetrini dipende dai parametri importanti del disegno sperimentale. Il modulo elastico dei due polimeri possono essere sintonizzati per simulare le rigidezze dei diversi tessuti, alterando la base / cura rapporto PDMS, e l'acrilammide / bis-acrilamide rapporto PA. PDMS può imitare tessuti rigidi nel range di 1-10MPa (cartilagine esempio, cornea, e pareti arteriose), e PA può imitare più morbidi tessuti nell'intervallo 100Pa-100kPa (es. mammella, cervello, fegato e prostata) 5. PDMS è poco costoso, facile da preparare, e la geometria delle caratteristiche stampate sarà identico alla testa dei perni di stampa. Così la dimensione e la forma delle caratteristiche può essere facilmente controllata mediante perni con geometrie diverse. PDMS è più idrofobico di PA, che provoca alcuni problemi durante la manipolazione cellulare e immunoscontenenti passi, e può essere incompatibile con alcune linee cellulari. Perché PA è un idrogel e un nativo non-fouling superficie, le cellule solo allegare al punti in cui ci sono le proteine che le cellule adesione al supporto. La geometria delle caratteristiche stampati su gel PA non seguire con precisione la geometria della testa di spillo, di solito diventano circoli a causa della diffusione, indipendentemente dalla geometria pinhead utilizzato. Stampa di contatto parametri di tempo e di diametro pin può essere empiricamente determinato per dimensione del tratto ottimale su gel PA.
Polidimetilsilossano (PDMS)
- In una tazza di plastica monouso combinare Sylgard 184 Base elastomero siliconico con il catalizzatore nel rapporto 10:1, mescolare energicamente con una linguetta di legno o plastica depressore quindi degassare in una campana sottovuoto temperatura ambiente per 30 min.
- Centro di un vetrino standard del mandrino azionato a vuoto di un rivestitore a rotazione, quindi filo 0,5 ml del polimero elastomero mista sul centro della superficie di scorrimento. Spin a6000 rpm per 60 sec.
- Curare i PDMS rivestite con diapositive di una 70 ° C forno o su una piastra calda digitale (protetto dalla polvere) per 4 ore a tutta la notte.
- Diapositive induriti possono essere utilizzati immediatamente, o conservati per diversi mesi in una scatola diapositiva che è sigillato all'interno di un sacchetto di plastica Ziploc e conservato in un cassetto. Il PDMS attira la polvere, quindi deve essere ben protetto dalla circolazione dell'aria ambiente.
- Nota: nitrile o altri non-lattice guanti devono essere indossati quando si lavora con il kit di elastomero PDMS. Contatto accidentale con i guanti in lattice inibisce la polimerizzazione PDMS.
Poliacrilammide (PA)
- NaOH incisione: Collocare i vetrini in blocco termico a 80 ° C. Aggiungere 1 ml di 0,1 N NaOH in ogni diapositiva, facendo attenzione a coprire l'intera superficie diapositiva. Lasciar evaporare NaOH (una pellicola bianca dovrebbe formare sulla superficie del vetrino). Dal momento che il gel PA può solo fissare saldamente il NaOH superfici incise, il gel PA si staccano durante la fase di essiccazione, se il surf intera diapositivaasso non è coperto da NaOH. Se la superficie di scorrimento non è stato coperto completamente, ripetere aggiungendo 1 ml di 0,1 N NaOH. Vetrini possono essere conservati a temperatura ambiente (RT) per diversi giorni. Nota: Un'alternativa alla incisione NaOH è di ozono o plasma-pulire i vetrini.
- 3-Aminopropyltriethoxysilane (APES) rivestimento: in una cappa aspirante, porre i vetrini in un piatto da 15 ml e aggiungere 300 ul APES su ogni diapositiva NaOH acidato. Lasciate che i APES reagire con la NaOH vetrini per 5 min. Il superamento di questo tempo causare difficoltà a lavare che non ha reagito reagente APES. APES lavare accuratamente con acqua deionizzata due a tre volte su entrambi i lati delle diapositive. Se il lavaggio non è completa, APES sarà ossidato da glutaraldeide per formare un deposito marrone su vetrini a passo 1,8.
- Ossidazione glutaraldeide: Aspirare tutte le soluzioni delle superfici di scorrimento. In ogni piatto 15 cm, aggiungere 25 ml di glutaraldeide 0,5% in PBS. Reagire per 30 minuti in una zona scura. Dopo 30 minuti, aspirare tutta la glutaraldeide e l'uso non-lint lavorosalviette stre Kimwipes (ad esempio) ad asciugare con cura le diapositive. I vetrini possono essere conservati a temperatura ambiente per un massimo di un giorno.
- Preparazione gel: Dopo preparazione di miscele PA inclusi acrilammide, bis-acrilammide e DDH 2 O in accordo con la seguente tabella, degas per 30 min, e quindi posizionare miscele PA sul ghiaccio per rallentare polimerizzazione. Aggiungi APS e TEMED e mescolare bene a destra prima di fare i gel. Pipettare miscele PA sulle superfici di scorrimento e posto 24 mm x 50 mm, numero 1 coprioggetto in cima alla PA. Evitare di premere coprioggetto e vetrino insieme e di evitare la formazione di bolle. Per gel> 40.000 Pa utilizzare 100 pl, per altri gel con 350 microlitri.
| Modulo desiderato (Pa) | Acrilamide% | Bis-acrilamide% | L'acrilamide dal 40% magazzino (ml) | Bis-acrilamide dal 2% magazzino (ml) | Acqua deionizzata (ml) | APS (pl) | TEMED (pl) |
| 480 ± 160 | 3 | 0,06 | 0,75 | 0,3 | 8,95 | 100 | 10 |
| 4470 ± 1190 | 5 | 0,15 | 1,25 | 0,75 | 8 | 100 | 10 |
| 40400 ± 2390 | 8 | 0,48 | 2 | 2,4 | 5,6 | 100 | 10 |
Adattato da 6,7.
- Lasciate che il gel PA polimerizzare per 2 ore, quindi rimuovere coprioggetti in acqua deionizzata.
- Lavare i vetrini gel PA in grandi vasi Coplan in acqua durante la notte (~ 8 ore) per rimuovere acrilamidi che non hanno reagito.
- Diapositive a secco in forno a 37 ° C per 2-4 ore o fino a quando PA gel indurisce completamente.
- PA gel vetrini possono essere conservati a 4 ° C per un mese in una scatola sigillata.
2. Proteine Maestro Preparazione Piatto
- Tutte le proteine should di aliquotare scorte di soluzioni 10X nei buffer raccomandate dal fornitore e conservato a -80 ° C. La maggior parte delle proteine ECM sono solubili in acqua deionizzata, ma il pH può essere necessario regolare con gocce di acido acetico. La maggior parte dei fattori di crescita, citochine, e domini extracellulari dei recettori ricombinanti sono preparati con BSA in PBS, ma quelle condizioni variano. Filtrare le aliquote delle proteine attraverso un micron 0,45 da 4 mm nylon siringa filtro (Nalgene) prima di riporlo.
- Progettare una lastra secondo combinazioni proteina desiderata e diluizioni. Cellule aderenti solito contare sulla presenza di almeno un ECM compatibile di mediare l'adesione cellulare, ma anticorpi epitopi superficie cellulare possono anche mediare attaccamento volte. E 'una buona idea aggiungere gratuitamente colorante FITC o fluorphore coniugato proteina ad almeno un bene in modo che gli array possono essere facilmente orientata in seguito.
- Preparare la piastra master diluendo le combinazioni di proteine con buffer di stampa composto da 100 mMTris-acetate/20% glycerol/0.05% Triton-100X pH 5,2. Tipicamente ciascun pozzetto di una piastra a 384 pozzetti contiene non più di 10 pl.
- Registrare il contenuto di ogni bene in ogni piatto principale in un file di scheda base di dati delimitati e fornire ogni piatto principale con un numero di identificazione univoco. A sei cifre data seguito dall'iniziale del designer serve spesso a tal fine (MMDDYYinitial). Poiché i volumi e sono piccole, aliquote di proteine può essere utilizzato in modo efficiente per generare un gran numero di piastre replicate. Si raccomanda che le piastre di master sono conservati a -80 ° C e ciascuna piastra master deve sottoporsi non più di due cicli di congelamento-scongelamento.
3. MEArray stampa
- MEArrays può essere stampato con robot microarray più convenzionali di stampa. Stampanti pin Quill che utilizzano silicone o perni in acciaio inox funzionano bene, ma la viscosità proteina può essere problematico. Stampanti capillari sono ideali robot microarray di stampa per questa applicazione, mentre lavoranoBene con soluzioni di proteine viscosi.
- Per ottenere una buona potenza statistica all'interno di una matrice, 10 a 12 punti replicati di ogni microambiente è raccomandato. Un tale progetto da consentire il confronto di attività in uno microambiente relativo ad un altro nello stesso array utilizzando semplici T-test statistiche. Dunnette prova può essere usato per confrontare l'attività in un ambiente di controllo con microambienti altri. Questo design funziona meglio quando un fenotipo funzionale è stato associato con il microambiente di controllo prima di eseguire gli esperimenti MEArray.
- L'umidità deve essere mantenuta intorno al 50%. Controllo dell'umidità è importante perché una bassa umidità può asciugare la soluzione all'interno dei perni o nei pozzetti della piastra master causando inefficiente deposizione sui substrati di stampa. L'umidità può essere controllato in modo efficace da drappeggi il robot con non poroso teli di plastica e utilizzando sia un umidificatore e un deumidificatore destinata a mantenere il 50% di umidità. Plattens stampa Cooled può essere Useful per la conservazione di alcune proteine, ma l'attenzione deve essere presa per evitare la formazione di condensa sulle diapositive.
- Ogni array stampato deve essere etichettati con congelatore a prova di etichette dei vetrini codificati con un numero di serie che si compone di identificatore della piastra master seguito da un numero a tre cifre (MMDDYYinitial-nnn). Come ogni array viene utilizzato o distribuito, dettagli dei loro trattamenti sperimentali devono essere mantenuti in un database. Monitoraggio delle date di stampa e il numero di cicli gelo-disgelo delle piastre maestri aiuterà ad individuare le condizioni ottimali per il mantenimento della riproducibilità.
- MEArrays stampati devono essere conservati in scatole sigillate scorrimento a -20 ° C per non più di un mese. Riproducibilità declina notevolmente in seguito.
4. Coltura di cellule di mammifero su MEArrays per l'analisi funzionale
- Allega camere di coltura: Per limitare il volume dei media e il numero di colture cellulari necessarie sui MEArrays, una camera di plastica è fitted circondare la matrice stampata. Per molti array, una sola camera da un 2-camera di scorrimento (Nunc) che contiene una superficie di 4,2 cm 2 può essere utilizzato. Rimuovere le camere dalla diapositiva camera fabbricato e tagliare le camere a metà con una lama di rasoio. Utilizzare una siringa 3 ml di applicare uno strato sottile di silicone acquario (DAP) al bordo di una camera e premere sulla superficie di un MEArray. Evitare di collocare l'applicazione camera di silicone acquario sulle caratteristiche di matrice.
- Blocco e risciacquo: MEArrays devono essere ben sciacquati per rimuovere i monomeri che non hanno reagito, che può essere tossico per le cellule. Se PDMS rivestite vetrini sono stati usati, quindi le regioni tra le caratteristiche stampate prima devono essere bloccati con un rivestimento non-fouling per impedire l'adesione cellulare; incubare le matrici in 1% Pluronic F108 (BASF) in acqua o 2% BSA in acqua per 15 minuti sotto vuoto. PA diapositive gel non richiedono una fase di bloccaggio. In tutti i casi, risciacquare array con mezzo di coltura tre volte per cinque minuti (scelta supporto dipendesulle cellule utilizzate, ma l'uso di antibiotici è raccomandato indipendentemente dal supporto o cellule). Gel PA richiedono un ulteriore incubazione 30 minuti in media per reidratare il gel.
- Cella attaccamento: Quattro o cinque gli array possono adattarsi all'interno di un unico 15 centimetri piatto Petri sterile. Coprire il piatto Petri con un coperchio per mantenere le matrici sterile. Aggiungere metà del volume finale media al MEArray aggiungendo le cellule in media a una concentrazione finale di 10.000 a 1.000.000 cellule / ml. Cellule si attaccano alle caratteristiche stampate a tassi diversi a seconda della composizione del microambiente stampata. Controllare che non uniforme visualizzando le matrici attraverso un microscopio invertito tappa in 15 intervalli di 20 minuti. Agitando delicatamente la MEArrays avanti e indietro, le cellule di fissaggio in modo modellato può essere distinto dai galleggianti, cellule separati.
- Rimozione delle cellule non legate: On PA rivestite MEArrays, le cellule non legate possono essere aspirato e il supporto può essere sostituito con un volume adeguato. Il PDMS rivestite MEArrays, i media non può essere completamente rimosso dal pozzetto perché le cellule seccano e muoiono quasi immediatamente. Così il PDMS rivestite MEArrays, le cellule non legate devono essere rimossi mediante un processo di scambio successivi di metà del volume del supporto finché le cellule non legate vengono rimosse, come determinato mediante ispezione microscopica. La de-bagnante effetto di PDMS è meno evidente quando il siero contenenti supporto è utilizzato rispetto al supporto di stampa definita, e quando la BSA è utilizzato per bloccare le aree non stampate rispetto al Pluronics F108.
- Le cellule possono essere coltivate su MEArrays posti all'interno di 15 cm piastre Petri per molti giorni con normali cambiamenti dei media. Cambiamenti dei media sulle diapositive PDMS deve essere fatto con successive modifiche della metà del volume del supporto.
- Fissativi comuni, come paraformaldeide e metanolo / acetone, sono compatibili con i sistemi MEArray. Quando colorazione delle cellule su PA rivestite MEArrays, fissativi possono essere aggiunte e spazzato via proprio come sarebbe in una procedura di colorazione convenzionale.Tuttavia quando colorazione delle cellule su PDMS rivestite MEArrays, la superficie deve rimanere umido anche durante il fissaggio. Aspirare la metà dei mezzi di comunicazione e di sostituirlo con un fissativo. Ripetere l'operazione alcune volte fino a quando il bene è riempito con una maggioranza di fissativo. Dopo il fissaggio, il fissativo è gradualmente sostituito nello stesso modo con un tampone di bloccaggio che è appropriato per la fase successiva di analisi.
- Immunocolorazione è comunemente utilizzato per analizzare le funzioni cellulari. Routine di colorazione può variare, ma quando si lavora sulle MEArrays PDMS, si ha la necessità di eseguire ogni lavaggio e fase di aspirazione come sopra, cambiando gradualmente le soluzioni e non permettere mai che la superficie de-bagnato. De-bagnatura provoca artefatti di colorazione.
- Le celle possono essere rimossi con l'aiuto di una lama di rasoio. Coprioggetto può essere montato in cima MEArrays colorarle con Fluoromount-G (Southern Biotech). Rilevazione può essere effettuata con la maggior parte multicolore fluorescenza microarray scanner o su microscopi confocali con motorizzatapiastrelle modalità di acquisizione di immagini.
5. Risultati rappresentativi
Un esempio di deposizione di proteine modellato su un circuito stampato PDMS-costeggiato MEArray utilizzando una squadra punta sugli silicio su un perno penna microarray-stampa robot è mostrato in Figura 2. Deposizione di proteine diverse che vengono stampate può essere verificato immunofluorescenza usando anticorpi (Figura 2A). Diluizioni delle soluzioni proteiche nella piastra master sono riflettenti della quantità (intensità fluorescente) che si deposita sulla superficie di substrati di stampa (Figura 2B). Cellule devono allegare alle caratteristiche stampate in maniera evidente modellata (Figura 2C).
Un esempio di un esperimento che mostra MEArray diluizioni inversi di due proteine microambiente suscitato specifici profili di espressione di cheratina in modo dipendente dalla concentrazione proteica in un umano p multipotenti epiteliali mammarielinea cellulare rogenitor (D920 cellule), è mostrato in Figura 3. Trame Bubble sono utili per determinare se specifici fenotipi sono imposti sulle cellule sulle funzioni multiple su una serie di diluizioni. Per esempio, se una molecola particolare in un microambiente provoca un fenotipo distinto, una volta che il componente è stato diluito istruttivo abbastanza in uno sfondo di un ECM neutro il fenotipo deve cambiare o scomparire. Rilevamento Immunofluorescenza di cheratina cheratina 8 e 14 proteine filamenti intermedi è stato eseguito con un 4200A Axon (Molecular Devices) scanner per microarray. Dodici serie di diluizioni replicati sono stati stampati su ogni MEArray, e il 2 rapporto registro di cheratina cheratina da 8 a 14 intensità media di fluorescenza è stata rappresentata come un complotto bolla per dare un'idea realistica di variazione e la riproducibilità del segnale. È mostrato dati da un MEArray che è stato fissato dopo aver attaccato cellule e le cellule non legate sono state lavate via (Figura 3A), e dopo 24 ore di culre (Figura 3B). Per questa analisi relativamente piccola, una ANOVA è stato utilizzato per determinare la varianza media dal segnale in ogni punto, e raggruppate a due code T-test sono stati utilizzati per determinare se le diverse diluizioni di collagene di tipo I e P-ricombinante umana caderina causato cambiamenti nell'espressione cheratina. Non c'era nessuna variazione rispetto al valore medio tra le cellule sulle caratteristiche appena dopo attaccamento, tuttavia, ci sono state differenze significative nell'espressione cheratina tra le cellule dopo 24 ore di esposizione ai diversi microambienti. T-test di verifica che tipo ad alta concentrazione di collagene ho suscitato maggiore cheratina 8 espressione, mentre alte concentrazioni di P-caderina suscitato un forte cheratina 14 Segnale dopo 24 ore. Questo risultato è stato coerente con le precedenti relazioni che la P-caderina contenenti microambienti imporrà di K14 che esprimono il fenotipo mioepiteliali bi-potenti cellule progenitrici mammarie 4.
Un esempio di scanner a tuttad MEArray stampato su 40.000 Pa PA gel è mostrato in Figura 4.

Figura 1. Un diagramma di flusso della procedura MEArray. Primo, i substrati di stampa sono preparati sia con PDMS o PA. In secondo luogo, le piastre master sono preparati e annotati in un database. Terzo, le MEArrays sono stampati e codificati con numeri di serie. In quarto luogo, camere di coltura sono attaccati, le superfici sono blocchi e / o risciacquato, quindi le cellule sono consentito per il collegamento e le cellule non legate vengono lavate via. Quinto, le cellule possono essere trattati con colorazione o saggio biologico dopo un periodo di incubazione su disegno sperimentale. Infine, Immagini di MEArray possono essere ottenuti e analizzati con scanner e software adeguati.
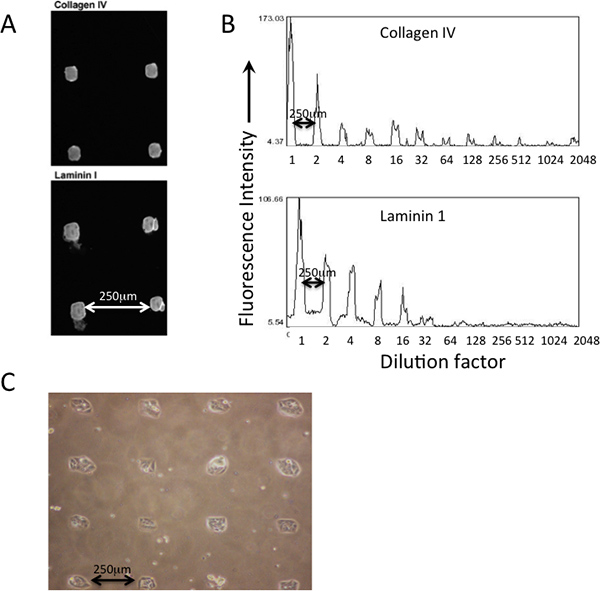
Figura 2. Deposabbondanza ition e relativa di proteine stampati possono essere verificati con immunocolorazione prima di adesione delle cellule. A) Gli anticorpi che hanno riconosciuto collagene di tipo IV e laminina-111 sono stati utilizzati per verificare la loro presenza nelle caratteristiche stampate di un MEArray. B) Utilizzo di una media di intensità dei pixel di analisi in funzione NIH ImageJ software, l'abbondanza relativa delle due proteine attraverso una serie di diluizioni, partendo da una soluzione proteica 200 mcg / ml, può essere valutata qualitativamente. C) Fase di Micrografia D920 cellule attaccate alla forma quadrata caratteristiche di un PDMS stampato rivestito MEArray.
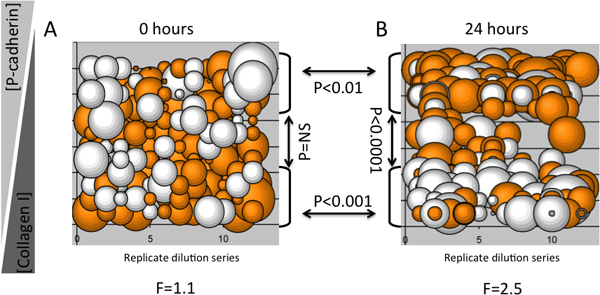
Figura 3. Un esempio di analisi MEArray utilizzando cambiamenti nell'espressione cheratina in una linea di cellule progenitrici multipotenti come funzioni del tempo e microambiente. Ogni bolla rappresenta i rapporti di cheratina cheratina 8 e 14 livelli di proteine 10-15 cellule attaccate ad un featura in un MEArray. L'espressione è stata determinata con sonde immunofluorescenza. A) mostra i rapporti di cheratina in cellule appena dopo attaccamento, e B) mostra i rapporti di cheratina dopo 24 ore su un array che è stata placcata in parallelo. La concentrazione massima di entrambe le proteine era 200 pg / ml e diluita due volte. Il diametro di una bolla rappresenta il valore del rapporto log 2 di cheratina cheratina 8 e 14 intensità media, e la codifica colore arancione e bianco indica valori> 0 e <0, rispettivamente. F-value per one-way ANOVA e P-valori di T-test, e le staffe con le frecce che identificano le popolazioni a confronto, sono mostrati.

Figura 4. Un esempio di scansione MEArray acquisito utilizzando una modalità piastrelle di acquisizione su un microscopio confocale a scansione laser. HCC1569 cellule ci hanno permesso di incorporare il DNA analogico EdU per 4 ore prima della fissazione.DAPI (blu) e Edu (rosso) sono mostrati.
Discussione
Il metodo qui presentato MEArray consente analisi funzionali della cellula e interazioni microambiente combinatori 4. Analisi MEArray combina l'uso di tecnologie di micropatterning di base, biologia cellulare, e robot di stampa microarray e dispositivi di analisi che sono disponibili in molti impianti multiutente. MEArray schermi sono compatibili con più tipi di cellule aderenti, sebbene senza siero formulazioni supporti possono essere necessario regolare in alcuni casi per includere BSA o siero <1%, ...
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
ML è sostenuto dalla NIA (R00AG033176 e R01AG040081) e dal Laboratorio di ricerca e sviluppo Diretto, US Department of Energy # contratto DE-AC02-05CH11231.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente | Azienda | Numero di catalogo | Commenti (opzionale) |
| Vetrini 25 mm x 75 millimetri | VWR | 48311-600 | |
| Vetro coprioggetti (n ° 1) 24 mm x 50 mm | VWR | 48393-241 | |
| Colorazione piatto (o vaso Coplan) | VWR | 25461-003 | |
| Piastre di Petri (15 cm) | BD Falcon | 351058 | |
| NaOH (1.0N) | Sigma-Aldrich | S2567 | |
| APES (> 98% (3-Aminopropyl) trietossisilano) | Sigma-Aldrich | A3648 | |
| Glutaraldeide | Sigma-Aldrich | G7651 | 50% in acqua |
| APS (> persolfato di ammonio 98%) | SIGMA-Aldrich | A3678 | Preparare soluzione al 10% di lavoro con DDH 2 O |
| TEMED (N, N, N ', N'-tetrametiletilendiammina) | Sigma-Aldrich | T9281 | |
| Acrilamide (40%) | Sigma-Aldrich | A4058 | |
| Bis-acrilamide (2% w / v) | Fisher BioReagents | BP1404-250 | |
| 0,45 micron filtro siringa da 4 mm nylon | Nalgene | 176-0045 | |
| FITC | Sigma-Aldrich | F4274 | |
| PDMS (polidimetilsilossano) | Dow Corning | 3097358-1004 | Sylgard 184 kit elastomero via Adesivi Ellsworth |
| 2-camera di diapositive | NUNC | 177380 | |
| Pluronic F108 | BASF | 30089186 | |
| Aquarium sigillante | Dow Corning | DAP 00688 | |
| Fluormount-G | Southern Biotech | 0100-01 | |
| Bicchieri di plastica usa e getta | |||
| Abbassalingua | |||
| Guanti in nitrile | |||
| Vetrino da microscopio scatole di plastica | |||
| Spin verniciatore | WS-400B-6NPP/LITE | Laurell Technologies Corporation | |
| Forno | |||
| Piastra digitale | |||
| 384 pozzetti | Un marchio appropriato per il robot microarray | ||
| Microarray stampa robot | |||
| Inversione di fase e microscopio a fluorescenza | |||
| Axon microarray scanner | Molecular Devices | Configurazioni multiple esistono |
Riferimenti
- Bissell, M. J., Labarge, M. A. Context, tissue plasticity, and cancer: are tumor stem cells also regulated by the microenvironment. Cancer Cell. 7, 17-23 (2005).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126, 677-689 (2006).
- McBeath, R., Pirone, D. M., Nelson, C. M., Bhadriraju, K., Chen, C. S. Cell shape, cytoskeletal tension, and RhoA regulate stem cell lineage commitment. Dev. Cell. 6, 483-495 (2004).
- LaBarge, M. A. Human mammary progenitor cell fate decsions are products of interactions with combinatorial microenvironments. Integrative Biology. 1, 70-79 (2009).
- Kim, H. N. Patterning Methods for Polymers in Cell and Tissue Engineering. Annals of biomedical engineering. , (2012).
- Boudou, T., Ohayon, J., Picart, C., Pettigrew, R. I., Tracqui, P. Nonlinear elastic properties of polyacrylamide gels: implications for quantification of cellular forces. Biorheology. 46, 191-205 (2009).
- Tse, J. R., Engler, A. J. Preparation of hydrogel substrates with tunable mechanical properties. Current protocols in cell biology. Chapter 10, Unit 10 (2010).
- Flaim, C. J., Chien, S., Bhatia, S. N. An extracellular matrix microarray for probing cellular differentiation. Nat. Methods. 2, 119-125 (2005).
- Soen, Y., Mori, A., Palmer, T. D., Brown, P. O. Exploring the regulation of human neural precursor cell differentiation using arrays of signaling microenvironments. Mol. Syst. Biol. 2, 37 (2006).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon