È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Uno strumento di analisi che quantifica Modifiche morfologia cellulare dalle immagini a fluorescenza tridimensionali
In questo articolo
Riepilogo
Abbiamo sviluppato una piattaforma software che utilizza Imaris Neuroscienze, ImarisXT e MATLAB per misurare i cambiamenti nella morfologia di una forma indefinita preso da tridimensionale fluorescenza confocale di singole cellule. Questo nuovo approccio può essere usato per quantificare i cambiamenti nella forma cellulare dopo l'attivazione del recettore e rappresenta quindi un possibile strumento aggiuntivo per la scoperta di farmaci.
Abstract
I più comuni strumenti di analisi software disponibili per la misurazione della fluorescenza sono le immagini per i dati bidimensionali (2D) che si basano su impostazioni manuali per l'inclusione e l'esclusione dei punti di dati e computer-aided pattern recognition per rafforzare l'interpretazione ei risultati delle analisi. È diventato sempre più importante essere in grado di misurare immagini di fluorescenza costruiti dataset tridimensionali (3D) per essere in grado di catturare la complessità delle dinamiche cellulari e capire la base della plasticità cellulare all'interno di sistemi biologici. Strumenti sofisticati di microscopia hanno permesso la visualizzazione di immagini 3D di fluorescenza attraverso l'acquisizione di immagini multispettrali di fluorescenza e potente software di analisi che ricostruisce le immagini da pile confocale che poi forniscono una rappresentazione 3D delle immagini raccolte 2D. Advanced Design metodi basati Stereology sono progrediti dal ravvicinamento e le ipotesi di original model-based stereology 1, anche in sezioni di tessuto complesse 2. Nonostante questi progressi scientifici in microscopia, rimane una necessità per un metodo analitico automatizzato che sfrutta appieno i dati intrinseci 3D per permettere l'analisi e la quantificazione delle variazioni complesse della morfologia cellulare, la localizzazione delle proteine e traffico recettore.
Le attuali tecniche disponibili per quantificare immagini di fluorescenza sono Meta-Morph (Molecular Devices, Sunnyvale, CA) e Image J (NIH) che forniscono l'analisi manuale. Imaris (Andor Technology, Belfast, Irlanda del Nord), il software fornisce la funzione MeasurementPro, che consente la creazione manuale di punti di misura che possono essere inseriti in un volume o un'immagine disegnata su una serie di sezioni 2D per creare un oggetto 3D. Questo metodo è utile per singolo clic misurazioni di punti per misurare una distanza di linea tra due oggetti o per creare un poligono che racchiude una regione di interesse, ma è difficile da applicare a complex strutture di rete cellulare. Filament Tracer (Andor) consente il rilevamento automatico del neuronale 3D filamento simile tuttavia, questo modulo è stato sviluppato per misurare strutture definite come i neuroni, che sono costituite da assoni, dendriti e spine (struttura ad albero). Questo modulo è stato ingegnosamente utilizzato per effettuare misurazioni morfologiche cellule non neuronali 3, tuttavia, i dati di uscita di fornire informazioni di un'estesa rete cellulare utilizzando un software che dipende da una forma cella definita piuttosto che essere una forma amorfa modello cellulare. Per superare il problema di analizzare amorfo a forma di cellule e rendere il software più adatto a una domanda di biologico, Imaris sviluppato cella Imaris. Questo è stato un progetto scientifico con la Eidgenössische Technische Hochschule, che è stato sviluppato per calcolare il rapporto tra cellule e organelli. Mentre il software consente l'individuazione di vincoli biologici, forzando un nucleo per cellamediante membrane cellulari di cellule segmento, esso non può essere utilizzato per analizzare dati di fluorescenza non sono continuativi perché idealmente costruisce superficie cellulare senza spazi vuoti. A nostra conoscenza, per il momento non modificabile dall'utente approccio automatizzato che fornisce informazioni morfometrica 3D da immagini di fluorescenza è stato sviluppato che consente di ottenere informazioni spaziali cellulare di forma indefinita (Figura 1).
Abbiamo sviluppato una piattaforma di analisi utilizzando il Imaris modulo software di base e Imaris XT interfacciato con MATLAB (Mat Works, Inc.). Questi strumenti consentono la misurazione 3D di cellule senza una predefinita forma e incoerenti con componenti di rete fluorescenza. Inoltre, questo metodo consentirà ai ricercatori che hanno esteso le competenze nei sistemi biologici, ma non familiarità per applicazioni informatiche, effettuare quantificazione dei cambiamenti morfologici nelle dinamiche cellulari.
Protocollo
1. Tridimensionale analisi morfometrica delle variazioni fenotipiche cella singola
- Umane embrionali renali (HEK293) cellule sono state trasfettate con emoagglutinina (HA)-tagged fattore di rilascio della corticotropina recettore-2 (CRF-R2), una proteina recettore accoppiato G (GPCR) come precedentemente descritto 4, 5.
- Le cellule sono state lasciate non trattate (nessun trattamento, NT), stimolate con il CRF-R2 ligando endogeno, fattore di rilascio della corticotropina, CRF (1 pM, 30 min), o pretrattato con un selettivo CRF-R2 antagonista, anti-sauvagine 30 (AS -30, 1 pM, 30 min) prima del trattamento agonista.
- Le cellule sono state poi fissate, permeabilizzate e trattati con anti-HA. CRF-R2 è stata visualizzata con Alexa 594 nm coniugato anti-topo (IgG 1) anticorpo. DAPI è stato usato per visualizzare la fase mitotica nuclei.
- Per limitare la soggettività sperimentatore, le condizioni sperimentali non erano noti solo dopo le immagini sono state acquisite ed analizzate.
- Abbiamo acquisito immagines da fisso HEK293 cellule usando un piano-apocromatico 63x/1.4 obiettivo olio DIC e Zeiss LSM 510 META microscopio confocale collegato ad un coerente integrato a due fotoni sistema laser composto da un Verdi-V5 laser e un Mira 900-F sistema laser.
- Durante il processo di acquisizione dei dati, le cellule sono state compartimenti sia dal sezionamento multispettrale, 488 nm e 790 nm (~ 350 nm Ex 2PH.) E z-partizionamento (incrementi di 0,5 micron) per includere dati dalla membrana nucleare del recettore esterno extracellulare estremità.
- I dati di fluorescenza sono stati elaborati utilizzando Imaris, che permette la visualizzazione e segmentazione di dataset microscopia 3D, e un modello 3D composto voxel cubici è stato creato per l'analisi morfometrica.
Poi, Imaris XT modulo è stato utilizzato per Imaris interfaccia con il linguaggio MATLAB programma per computer per determinare il punto coordinate delle estensioni GPCR.
Per tener conto della variabilità cellulare, abbiamo ottenuto e analizzato le immagini di fluorescenza taken da 22 celle: nessun trattamento (NT) (n = 7), agonista (CRF) trattamento (n = 8) e di pre-trattamento con antagonista (AS-30) prima del trattamento con agonisti (n = 7) (Figura 2) .- La regione di interesse (ROI) dovrebbe includere una cella che non è in fase di mitosi attiva e non vicino ad altre cellule. In questo modo, l'analisi comprenderà cellule con un solo nucleo e le estensioni recettore non sono disturbati da vicinanza di altre cellule.
- La struttura 3D cellulare è stato ricostruito dai dati multispettrali di fluorescenza utilizzando Imaris (v.7.1.1).
- Dopo l'algoritmo progettato da Imaris, prima il rendering di superficie è stato utilizzato per rappresentare la membrana nucleare. Imaris determinerà se vi è più di un nucleo del ROI.
- Poi la creazione di punti di algoritmo è stato utilizzato per individuare il CRF-R2 estensione. Rilevamento Spot è stato utilizzato in quanto compensa il rumore di fondo e l'intensità irregolare delcomplessa rete di cellule a forma amorfa.
- Per massimizzare l'inclusione di ciascuna unità di rivelazione di fluorescenza del CRF-R2, diametro dei punti 'stato impostato a 0,2 micron, che è la più piccola unità all'interno dell'immagine per estrapolare informazioni distinte nella forma di una intensità misurata utilizzando un filtro gaussiano. Posto per il filtraggio è stata costituita nel processo di creazione automatica punti. Il software, tuttavia, offre all'utente la possibilità di utilizzare i filtri per definire i parametri.
- Per evitare il troncamento dei dati, il set di dati è stato convertito da 8 bit (unsigned) punto fisso, a 32-bit decimale.
- I dati sono stati scambiati voxel intensità di individuare dati di coordinate utilizzando Imaris modulo XT interfacciato con MATLAB e l'esatta posizione spaziale di ogni spot è stato determinato eseguendo una trasformazione distanza utilizzando la membrana nucleare come punto di riferimento (figura 3).
- I dati ottenuti possono essere quantificati e presentati in formato grafico per la sanalisi tatistical. I confronti tra i gruppi sono state eseguite utilizzando ANOVA a due vie e post-test di Bonferroni. I dati sono presentati come media ± DS. Le differenze sono considerati significativi a * p <0,05. Calcoli sono stati fatti con GraphPad Prism 5.02 (Figura 4).
2. Risultati rappresentativi
Per dimostrare la potenza del nostro approccio, abbiamo quantificato i cambiamenti cellulari che derivano dalla interazione di G recettori accoppiati a proteine (GPCR) e del fattore di rilascio della corticotropina recettore-2 (CRF-R2) con il suo ligando endogeno CRF in cellule HEK293 trasfettate.
Mostriamo che CRF-R2 recettori si trovano nella membrana plasmatica e sporgono da regioni finiti della membrana delle cellule (Figura 2A e Movie 1). Utilizzando l'analisi convenzionale 2D, è possibile rilevare questo sottoinsieme di extracellulari CRF-R2 recettori solo se si analizza il recettore di adesione pointeri sul vetro copre. Di conseguenza, si perde qualsiasi altra informazione derivata da z-impilati dati multispettrali (Figura 5).
Quando le cellule vengono trattate con CRF, i recettori extracellulari sono notevolmente ridotti, come mostrato dalla diminuzione della distanza dei punti della membrana plasmatica. Sono anche ridistribuiti da postazioni principalmente finite in un numero di posizioni discrete (Figura 2B e Movie 2).
L'effetto della CRF sulla distribuzione recettore di membrana è impedito dal pretrattamento con il CRF-R2 antagonista specifico, antisavagine 30 (AS-30) e troviamo che i CRF-R2 estensioni non cambiano (Figura 2C e Movie 3).
La distribuzione distale di macchie, i tracciati in 5 micron spettro di colori intervalli codificati, è utilizzata per visualizzare la distanza dei voxel della membrana nucleare. Nessun trattamento e antagonista pretreatment (AS-30, 1 mM, 30 min) prima del trattamento con agonisti (CRF, 1 pM, 30 min) non mostrano alcun significativo (ns) differenza di contrazione GPCR. Trattamento delle cellule con l'agonista (CRF, 1 pM, 30 min) riduce progressivamente il numero di CRF-R2 contenenti voxel rispetto a nessun trattamento, 0-5 micron (ns), 6-15 micron (** p < 0,01) e> 15 micron (*** p <0,005), o rispetto a AS-30 trattamento, 0-10 micron (ns), 11-15 micron (** p <0.01) e> 15 micron (*** p <0,005) (Figura 4).
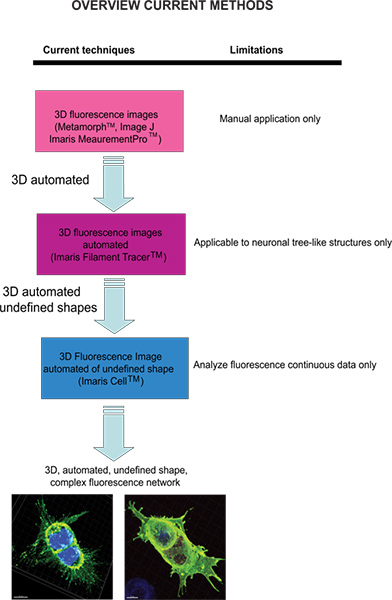
Figura 1. Schema delle tecniche attualmente disponibili e la loro limitazione per analizzare le immagini di fluorescenza. Clicca qui per ingrandire la figura .
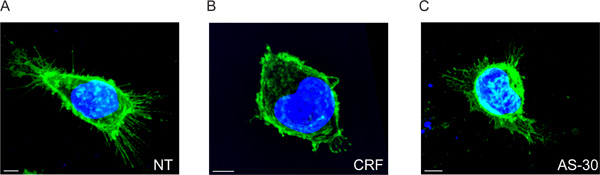
Figura 2. 3D immagini multispettrali di fluorescenza di cellule HEK293 trasfettate con HA-CRF-R2 prima e dopo il trattamento con agonisti e antagonisti. Immagini che mostrano uniti HA-CRF-R2-sondati con anticorpi anti-HA e visualizzati con Alexa 488 coniugato anticorpo anti-topo (IgG 1) secondaria; DAPI è stato utilizzato per visualizzare i nuclei. Le immagini sono state acquisite con scansione laser confocale (CLS) microscopio. Scala bar 5 micron.
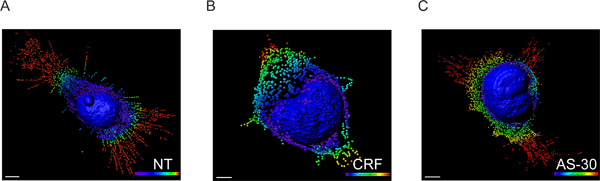
Figura 3. Modello 3D di cellule HEK293 trasfettate con HA-CRF-R2 ricostruita da immagini utilizzando il software Imaris CLS. Il rendering della superficie del nucleo e la creazione di punti di descrizione delle estensioni GPCR convertiti in piccole vescicole. I dati di fluorescenza sono stati elaborati utilizzando Imaris che permette la visualizzazione e la segmentazione del set di dati 3D microscopia. Poi, Imaris XT è stato utilizzato per Imaris interfaccia con MATLAB. Le intensità voxel sono stati scambiati inposto le coordinate. Le macchie spettro codifica a colori (blu, verde 0-5 micron, 6-10 micron micron, giallo, rosso, 11-15 e> 15 micron) rappresentano il modulo di distanza la membrana nucleare. Scala 5 micron.
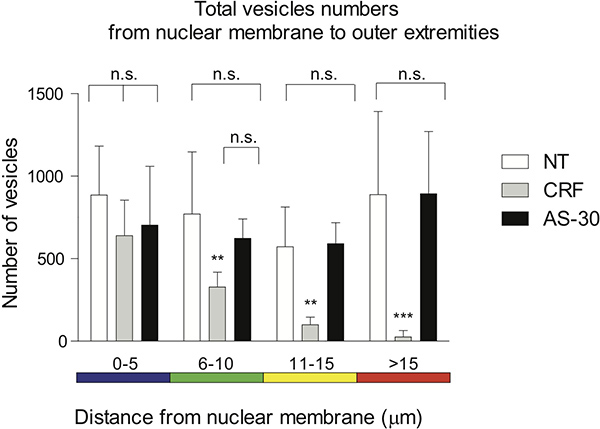
Figura 4. Rappresentazione grafica della distribuzione dei punti tracciati distale nello spettro dei colori codificati a intervalli di 5 micron è utilizzata per visualizzare la distanza dei voxel della membrana nucleare. Nessun trattamento e pretrattamento con un antagonista (AS-30, 1 mM, 30 min) prima del trattamento con agonisti (CRF, 1 pM, 30 min) non mostrano alcun significativo (ns) differenza di contrazione GPCR. Trattamento delle cellule con agonisti (CRF, 1 pM, 30 min) riduce progressivamente la distanza del numero di CRF-R2 contenenti voxel rispetto a nessun trattamento 0-5 micron (ns), 6-15 micron (** p <0.01) e> 15 micron (*** p <0,005), o AS-30 di trattamento 0-10 micron (ns), 11-15 micron (** p <0.01) e> 15 micron (*** p <0,005).
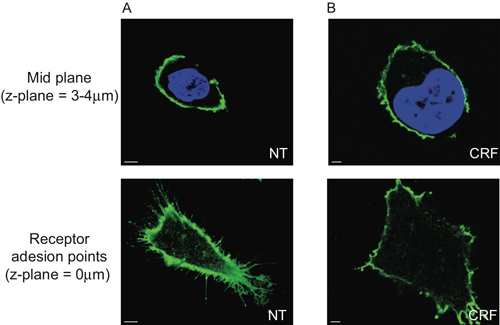
Figura 5. Limitazione dell'analisi 2D morfometrica di cellule HEK 293 trasfettate con HA-CRF-R2. Il piano intermedio sezione di celle (3-4 _m sopra il vetrino coprioggetto) che mostra il centro dei nuclei visualizzata con DAPI e HA-CRF-R2 sondati con anticorpi anti-HA e visualizzati utilizzando Alexa 488nm coniugato anti-topo (IgG 1) secondaria anticorpo e acquisite con CLS non mostra alcuna differenza tra (A) nessun trattamento (NT) e (B) agonista (CRF, 1 pM, 30 min), mentre i punti recettori di adesione sono drammaticamente diversi.
Movie 1. Rotazione libera modello 3D in modalità "Surpass" di cellule HEK 293 trasfettate con HA-CRF-R2, nessun trattamento, per valutare le differenze tra fenotipo cellulare proteine recettoriali. Bar Scala 5-20 micron./ "Target =" _blank files/ftp_upload/4233/4233movie1.avi "> Clicca qui per visualizzare filmati.
Movie 2. Rotazione libera modello 3D in modalità "Surpass" di cellule HEK 293 trasfettate con HA-CRF-R2, trattamento con l'agonista, CRF (Centro Ricerche Fiat, 1 pM, 30 min), per valutare le differenze tra fenotipo cellulare proteine recettoriali. Bar Scala 5-20 micron. Clicca qui per visualizzare filmati.
Movie 3. Rotazione libera 3D in modalità "Surpass" di cellule HEK 293 trasfettate con HA-CRF-R2, il pretrattamento con un antagonista (AS-30, 1 mM, 30 min) prima del trattamento con agonisti (CRF, 1 pM, 30 min ) per valutare le differenze fenotipo cellulare tra proteine recettoriali. Bar Scala 5-20 micron. Clicca qui per visualizzare filmati.
Discussione
Abbiamo mostrato che il trattamento CRF indotto un significativo cambiamento nella morfologia e localizzazione del CRF-R2. La variazione CRF-R2 è stata inibita mediante trattamento antagonista selettivo. Abbiamo dimostrato che modifiche recettore non sono stati rilevati e non può essere misurata utilizzando le tecniche standard 2D multispettrali. La capacità di studiare complesse immagini 3D è fondamentale per incorporare la complessità dei parametri biologici per l'analisi morfometrica. Siamo stati in grado di...
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Ringraziamo il Biological Imaging Development Center (BIDC) dell'Università della California, a San Francisco per l'uso del Imaris, Imaris XT e Matlab. Ringraziamo V. Kharazia per l'assistenza tecnica e AT Henry, LK Floren, L. Daitch per il loro contributo alla redazione del manoscritto. Questo lavoro è stato sostenuto da un finanziamento da parte dello Stato della California Medical Research su abuso di alcool e di sostanze attraverso la UCSF di SEB, il National Institutes of Health: 1R21DA029966-01 e di aggiudicazione NIH pista veloce per schermare la raccolta MLSMR di SEB, UCSF Facoltà di Farmacia ( Ufficio di Presidenza e Farmacia Clinica) e la Scuola di Medicina (Clinical Pharmacology & Therapeutics sperimentali) a CLHK.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente | Azienda | Numero di catalogo | Commenti (opzionale) |
| Di rene embrionale umano (HEK293) | Tipo americano Culture Collection | CRL-1573 | |
| Eagle modificato da Dulbecco (DMEM) | Invitrogen | 11965118 | |
| Siero bovino fetale (FBS) | Invitrogen | SH30070.03 | |
| AlexaFluor-488 (IgG2b) | Invitrogen | A-11001 | |
| monoclonale anti-HA.11 (IgG 1) | Covance | 16B12 | |
| DAPI | Vector Laboratories | H-1200 | ; |
| CRF | Sigma | C2917 | |
| Antisauvagine-30 (AS-30) | Sigma | A4727 |
Riferimenti
- West, M. J. Design-based stereological methods for counting neurons. Prog, Brain Res. 135, 43-51 (2002).
- Burke, M., Zangenehpour, S., Mouton, P. R., Ptito, M. Knowing what counts: unbiased stereology in the non-human primate brain. J. Vis. Exp. (27), e1262 (2009).
- Sugawara, Y., Ando, R., Kamioka, H., Ishihara, Y., Honjo, T., Kawanabe, N., Kurosaka, H., Takano-Yamamoto, T., Yamashiro, T. The three-dimensional morphometry and cell-cell communication of the osteocyte network in chick and mouse embryonic calvaria. Calcif. Tissue Int. 88, 416-424 (2011).
- Vickery, R. G., von Zastrow, M. Distinct dynamin-dependent and -independent mechanisms target structurally homologous dopamine receptors to different endocytic membranes. J. Cell Biol. 144, 31-43 (1999).
- Bartlett, S. E., Enquist, J., Hopf, F. W., Lee, J. H., Gladher, F., Kharazia, V., Waldhoer, M., Mailliard, W. S., Armstrong, R., Bonci, A. Dopamine responsiveness is regulated by targeted sorting of D2 receptors. Pro.c Natl. Acad. Sci. U.S.A. 102, 11521-11526 (2005).
- Gordon, A., Colman-Lerner, A., Chin, T. E., Benjamin, K. R., Yu, R. C., Brent, R. Single-cell quantification of molecules and rates using open-source microscope-based cytometry. Nat. Methods. 4, 175-181 (2007).
- Schock, F., Perrimon, N. Molecular mechanisms of epithelial morphogenesis. Annu. Rev. Cell Dev. Biol. 18, 463-493 (2002).
- Pincus, Z., Theriot, J. A. Comparison of quantitative methods for cell-shape analysis. J. Microsc. 227, 140-156 (2007).
- Spiller, D. G., Wood, C. D., Rand, D. A., White, M. R. Measurement of single-cell dynamics. Nature. 465, 736-745 (2010).
- Loo, L. H., Wu, L. F., Altschuler, S. J. Image-based multivariate profiling of drug responses from single cells. Nat Methods. 4, 445-453 (2007).
- Yarrow, J. C., Totsukawa, G., Charras, G. T., Mitchison, T. J. Screening for cell migration inhibitors via automated microscopy reveals a Rho-kinase inhibitor. Chem. Biol. 12, 385-395 (2005).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon