È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
RNA Secondaria previsione della struttura utilizzando la forma di alta velocità
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
High-throughput selettivo 2 'acilazione idrossile analizzato da primer extension (SHAPE) utilizza un romanzo chemical probing tecnologia, la trascrizione inversa, elettroforesi capillare e software di predizione della struttura secondaria per determinare le strutture di RNA da diverse centinaia a diverse migliaia di nucleotidi a singolo nucleotide risoluzione.
Abstract
Comprendere la funzione di RNA coinvolti in processi biologici richiede una conoscenza approfondita della struttura dell'RNA. A questo fine, la metodologia denominata "2 selettivo high-throughput 'acilazione idrossile analizzato da primer extension", o la forma, permette la previsione di RNA struttura secondaria con risoluzione singolo nucleotide. Questo approccio utilizza agenti chimici che probing acilato preferenzialmente singole regioni o flessibile filamento di RNA in soluzione acquosa. Siti di modificazione chimica vengono rilevati mediante trascrizione inversa del RNA modificati, ed i prodotti di questa reazione sono frazionati mediante elettroforesi capillare automatizzato (CE). Dal momento che la transcriptasi inversa si ferma a quei nucleotidi di RNA modificati dai reagenti SHAPE, la libreria di cDNA risultante indirettamente associa quei ribonucleotidi che sono a singolo filamento nel contesto delle RNA piegati. Utilizzando il software ShapeFinder, i elettroferogrammi prodotti dalla automatizzato CE vengono elaborati e convertiti in nutavoli reattività cleotide che sono a loro volta convertiti in vincoli pseudo-energetiche utilizzate nel RNAStructure (V5.3) algoritmo di predizione. Le strutture di RNA bidimensionali ottenute combinando SHAPE sondando con in silico RNA predizione della struttura secondaria sono stati trovati per essere molto più preciso di strutture ottenute utilizzando il metodo da solo.
Introduzione
Per capire le funzioni di RNA catalitici e non codificanti coinvolti nella regolazione dello splicing, la traduzione, la replicazione del virus e il cancro, una conoscenza dettagliata della struttura dell'RNA è richiesto 1,2. Purtroppo, la previsione accurata di RNA folding rappresenta una sfida formidabile. Agenti di sondaggio classici soffrono molti svantaggi come la tossicità, incompleta copertura dei nucleotidi e / o velocità limitata a 100-150 nucleotidi per esperimento. Senza aiuto secondarie algoritmi di predizione di struttura sono allo stesso modo svantaggioso, a causa di imprecisioni derivanti dalla loro incapacità di distinguere efficacemente tra strutture energeticamente simili. Grandi RNA in particolare sono spesso refrattari ai metodi di determinazione della struttura 3D come cristallografia a raggi X e risonanza magnetica nucleare (NMR), grazie alla loro flessibilità conformazionale e grandi quantità di campioni altamente puri richiesti per queste tecniche.
High-capacità SHAPE risolve molti di questi problemi fornendo un efficace, semplice approccio per sondare le strutture di grandi RNA a singolo nucleotide risoluzione. Inoltre, i reagenti utilizzati per la forma sono sicuro, facile da maneggiare e, a differenza della maggior parte dei reagenti chimici probing, reagiscono con tutti e quattro i ribonucleotidi. Questi reagenti possono anche penetrare le membrane cellulari, rendendo possibile sondare RNA nel loro contesto in vivo (s) 3. Originariamente sviluppato nel laboratorio 4 settimane, SHAPE è stato utilizzato per analizzare una grande varietà di RNA, l'esempio più notevole è determinazione della struttura secondaria completa del ~ 9 kb HIV-1 RNA genoma 5. Altri risultati di rilievo utilizzando FORMA includono delucidazione delle strutture di viroidi infettive 6, RNA umani lunghi non codificanti 7, ribosomi lievito 8 e riboswitches 9, nonché per identificare i siti di legame delle proteine nel virione-associata HIV-1 RNA 3. While le varianti originali e high-throughput del protocollo SHAPE sono stati pubblicati altrove 10-12, il presente lavoro fornisce una descrizione dettagliata della determinazione della struttura secondaria dell'RNA da SHAPE high-throughput utilizzando oligonucleotidi fluorescenti, la Beckman Coulter CEQ 8000 Genetic Analyzer, e (v5.3) software SHAPEfinder e RNAStructure. Sono inclusi anche inediti dettagli tecnici e consigli sulla risoluzione dei problemi.
Variazioni di forma
L'essenza di SHAPE e sue variazioni è l'esposizione di RNA in soluzione acquosa a anidridi elettrofili che acilato selettivamente 2'-idrossile (2'-OH) gruppi ribosio, producendo addotti ingombranti nei siti di modificazione. Questa reazione chimica serve come mezzo di interrogare dinamica strutturale RNA locali, come nucleotidi a singolo filamento sono più inclini ad adottare conformazioni favoriscano attacco elettrofilo da questi reagenti, mentre la base accoppiato o architettonico fabbrnucleotidi ained sono meno o non reattivo 10. Siti di formazione di addotti vengono rilevati mediante trascrizione inversa avviando da fluorescente o radioattivo primer ibridati ad un sito specifico sulla RNA modificato (il "(+)" reazione di innesco di estensione). Quando trascrittasi inversa (RT) non riesce ad attraversare i ribonucleotidi acil derivati, un pool di cDNA prodotti viene prodotto il cui lunghezze coincidono con i siti di modificazione. Un controllo, "(-)" primer estensione reazione utilizzando RNA che non è stato esposto a reagente viene eseguita anche in modo che la chiusura prematura della sintesi del DNA (vale a dire "stop") a causa di RNA struttura, non specifico filamento di RNA rotture, ecc, maggio. essere distinta da facendo una pausa prodotto mediante modificazione chimica. Infine, due reazioni di sequenziamento dideossi-avvio dagli stessi primer sono utilizzati come marcatori per correlare nucleotidi reattivi con la sequenza primaria di RNA dopo elettroforesi.
In applicazione originale di SHAPE, lo stesso 32 P-end-marcato primer viene utilizzato per il (+), (-), e due reazioni di sequenziamento. Prodotti di queste reazioni sono caricati in pozzetti adiacenti in un gel di poliacrilammide lastra 5-8%, e frazionato mediante elettroforesi su gel denaturante di poliacrilammide (PAGE; Figura 1). Analisi quantitativa delle immagini gel prodotte da SHAPE convenzionale può essere eseguita utilizzando SAFA, un software semi-automatico di analisi footprinting 13.
In contrasto, la forma ad alta velocità impiega primer fluorescente ed elettroforesi capillare automatizzata. Specificamente, per ogni regione di RNA in esame, una serie di quattro primer DNA avente una sequenza comune ma diverso 5 'etichette fluorescenti devono essere sintetizzati o acquistati. Questi oligonucleotidi diversamente etichettati servono a primi due reazioni forma e le due reazioni di sequenziamento, i cui prodotti sono raggruppati e frazionato / rilevati mediante elettroforesi capillare automatizzato (CE). Whereas il profilo di reattività 100-150 nt di RNA può essere ottenuto da una serie di quattro reazioni usando l'approccio originale, SHAPE alta produttività consente risoluzione del 300-600 nt da un singolo campione riunito 3. Fino a 8 serie di reazioni può essere frazionata simultaneamente, mentre ben 96 campioni possono preparato per frazionamento nel corso di 12 consecutivi piste CE (Figura 2). Inoltre, il software SHAPEfinder, sviluppato per elaborare e analizzare i dati che emergono dal QVC e altri analizzatori genetici, è più automatizzato e richiede meno interventi da parte dell'utente di SAFA 13 o altri pacchetti di gel di analisi.
Metodologie high-throughput più avanzati hanno recentemente emerso come PARS (analisi in parallelo di struttura RNA) 14 e Frag-Seq (frammento-sequencing) 15, che utilizzano enzimi struttura-specifiche, piuttosto che reattivi alchilazione in combinazione con tecniche di sequenziamento di nuova generazione per ottenere information su la struttura dell'RNA. Nonostante l'attrattiva di queste tecniche, le molte limitazioni inerenti alla nucleasi tastatura rimangono ancora 16. Questi problemi possono essere aggirate nel sequenziamento SHAPE (SHAPE-Seq) protocollo 17, dove sequenziamento di nuova generazione è preceduta dalla modificazione chimica e trascrizione inversa di RNA in una maniera simile a quella effettuata per SHAPE convenzionale. Mentre questi metodi possono rappresentare il futuro della determinazione della struttura dell'RNA, è importante ricordare che sequenziamento di nuova generazione è molto costoso, e rimane non disponibile per molti laboratori.
FORMA Data Analysis
Dati prodotti nell'analizzatore genetico è presentato nella forma di un elettroferogramma, in cui l'intensità di fluorescenza del campione (s) che fluisce attraverso il rivelatore capillare è tracciata contro un indice di tempo di migrazione. Questa trama prende la forma di tracce sovrapposte corrispondenti ai quattro canali di fluorescenzas utilizzato per rilevare i diversi fluorofori, e dove ogni traccia è composta da picchi corrispondenti ai singoli prodotti di cDNA o sequenziamento. Dati elettroferogramma vengono esportati dalla analizzatore genetico come file di testo delimitato da tabulazioni e importati in trasformazione ShapeFinder e software di analisi 18.
ShapeFinder è inizialmente utilizzato per eseguire una serie di trasformazioni matematiche sui dati per garantire che i tempi di migrazione e di volumi di picco rispecchiano fedelmente le identità e le quantità dei prodotti di reazione, rispettivamente. Picchi sono poi allineati e integrati, ed i risultati tabulati insieme alla sequenza di RNA primario. Un "profilo reattività" per il segmento pertinente del RNA è ottenuto sottraendo i valori di controllo della (+) valori associati a ogni RNA nucleotide, e normalizzare i dati come descritto di seguito. Questo profilo viene importato in RNAstructure (v5.3) 19,20 software che converte la reattività val normalizzatoUES in vincoli pseudo-energia che sono incorporati nella algoritmo pieghevole struttura secondaria dell'RNA. Combinando chemical probing e piegatura algoritmi in questo modo migliora significativamente l'accuratezza di previsione strutturale rispetto a uno metodo da solo 12,21. L'uscita di RNAstructure (v5.3) comprende le immagini di più bassa energia RNA strutture secondarie con codice colore Con il profilo SHAPE reattività (s), così come le stesse strutture in testuale notazione dot-staffa. Questi ultimi possono essere successivamente esportati in software dedicato alla visualizzazione grafica di RNA struttura secondaria come Varna 22 e PseudoViewer 23.

Figura 1. Diagramma di flusso della determinazione della struttura dell'RNA tramite SHAPE 4,10. (A) RNA may essere ottenuto da campioni biologici o di trascrizione in vitro. (B) A seconda della sorgente, l'RNA è piegato o altrimenti elaborati e modificati con il reattivo di SHAPE. (C) trascrizione inversa utilizzando fluorescente o radioattiva primer marcati. (d) i prodotti di cDNA sono frazionato sia tramite elettroforesi su gel a base capillare o lastra. (E) Analisi Fragment. (F) previsione della struttura dell'RNA. Clicca qui per ingrandire la figura.
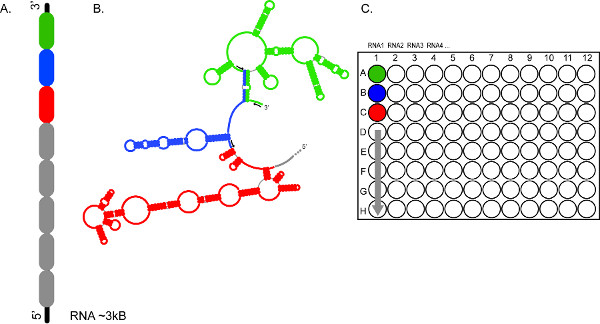
Figura 2. Il carattere high-throughput di SHAPE CE-based permette una rapida analisi di molteplici RNA, e / o più segmenti dello stesso RNA. (A) Rappresenta come un RNA può essere diviso in sezioni 300-600 nt (codice colore in verde, blu e rosso) (B) Sezioni di RNA vengono sondati in modo indipendente utilizzando diversi set di primer fluorescenti (frecce nere) (C) imposta di Le reazioni sono raggruppati e caricati nei pozzetti A1, B1, C1, ecc, rispettivamente, fornisce una copertura completa per il ~ 3 kb RNA1. Prodotti di reazione di RNA 2, 3, 4, ecc possono essere preparati in modo simile per frazionamento in corse elettroforetiche consecutivi. Clicca qui per ingrandire la figura.
Protocollo
Progettazione Primer e l'estensione del capolinea del RNA 3 '
Per analizzare lunghi RNA da SHAPE ad alta produttività, una serie di siti di ibridazione del primer deve essere selezionata in modo tale che (i) sono separati da ~ 300 nt, (ii) sono 20-30 nt di lunghezza, e (iii) che il RNA / ibridi DNA prodotto da DNA ricottura a questi siti hanno una temperatura di fusione previsto> 50 ° C. Inoltre, i segmenti di RNA che si prevede di essere prevalentemente strutturate dovrebbero essere evitati, anche se procedere a tale constatazione richiede alcune preconoscenza della struttura di RNA, che spesso non è disponibile. Primer di DNA che ibridano a questi siti devono quindi essere progettati, avendo cura di garantire che essi non sarebbero tenuti a formare dimeri stabili o strutture secondarie intraelicoidali.
Una volta progettato, set di primer devono essere sia acquistati (ad esempio da Integrated DNA Technologies, Ames, Iowa) o sintetizzati 24,25. Primer 5'-etichettato con Cy5, Cy5.5,WellRedD2 (Beckman Coulter) e IRDye800 (Lycor) / WellRedD1 (Beckman Coulter) sono più adatti per il Beckman Coulter CEQ 8000, fornendo una buona intensità del segnale, riducendo al minimo la diafonia. Oligonucleotidi etichettati, possono essere conservate a tempo indeterminato, in piccole aliquote da 10 mM a -20 ° C; evitare il congelamento / scongelamento ripetuti cicli.
Utilizzando primer disegnati in questo modo, è possibile avere dati di forma praticamente per un intero RNA di qualsiasi lunghezza. Tuttavia, la sequenza in corrispondenza o vicino all'estremità 3 'terminale di un RNA è sempre inaccessibile a SHAPE, a meno che l'RNA è progettato per contenere una 3' estensione terminale (ad esempio un "cassette struttura") a cui un primer può essere ibridato 4.
RNA Preparazione tramite elettroforesi capillare
Sebbene RNA da campioni biologici possono essere utilizzati per SHAPE ad alta produttività, il protocollo qui proposta è ottimizzato per RNA prodotta mediante trascrizione in vitro. Commerciale TRAkit nscription quali MegaShortScript (Ambion) utilizzato in combinazione con MegaClear RNA colonne di purificazione (Ambion) sono adatti a generare grandi quantità di RNA puri. RNA devono essere conservati in tampone TE tra -20 ° C e -80 ° C. Per ottenere i migliori risultati, RNA dovrebbero apparire omogeneo sia da elettroforesi su gel di poliacrilammide denaturante e non denaturante.
1. RNA pieghevole
- In una provetta da 0,5 ml, diluire 12 pmol di RNA a 18 ml con acqua e aggiungere 2 ml di tampone di rinaturazione 10X. Mescolare bene.
- Riscaldare a 85 ° C per 1 minuto, poi raffreddare a 4 ° C ad una velocità di 0,1 ° C / sec.
- Aggiungere 100 ml di acqua e 30 ml di tampone 5X pieghevole.
- Incubare a 37 ° C per 30-60 min, a seconda delle RNA essendo piegate. In generale, Mg 2 +-dipendente pieghevole di lunghi e più strutturato RNA richiedono tempi di incubazione più lunghi.
- Trasferire una aliquota microlitri 72 per ciascuna delle due provette da 0,5 ml microcentrifuga: modified (+) e di controllo (-).
2. Modificazione chimica dell'RNA
Ben caratterizzato, reagenti elettrofili FORMA includono anidride isatoica (IA), anidride N-methylisatoic (NMIA), anidride 1-metil-7-nitro-isatoica (1M7) 26, e cianuro di benzoile (BzCN) 27. Di questi, il più comunemente usato per SHAPE high-throughput sono 1M7 e NMIA, e solo il secondo è disponibile in commercio (Life Technologies). La concentrazione finale di modificare reagente deve essere ottimizzato per ciascun RNA avere "single-hit" modifica cinetica, cioè la condizione in cui la maggior parte RNA in soluzione vengono modificati una volta nella regione di RNA da analizzare 11. Questa concentrazione ottimale può essere determinato eseguendo più reazioni in cui la concentrazione del reagente viene variata attraverso la gamma (s) indicato nella tabella qui di seguito nella sezione 2.1. Utilizzare la concentrazione di reagente che produce un segnale facilmente rilevabile durante minimizing la differenza di intensità di segnale tra prodotti di sintesi di DNA lunghi e brevi (ad esempio, Figura 3).
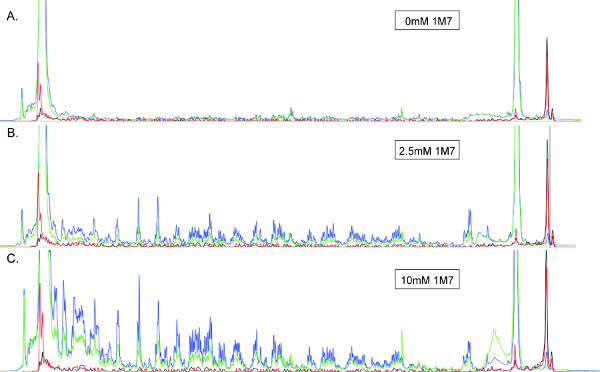
Figura 3. Elettroferogrammi SHAPE prodotte da un ~ 360 nt RNA trattato con (A) 0 (B) 2,5 mm o (C) 10 mM 1M7. Tutti elettroferogrammi vengono visualizzati sulla stessa scala. Blu, tracce verdi, rosso e nero corrispondono a (+) prodotti di reazione (Cy5), (-) prodotti di reazione (Cy5.5), e le due scale di sequenziamento (D2 WellRed e IRDye800), rispettivamente. Gli RNA utilizzati per produrre un'immagine (B) è stato trattato con la quantità ottimale di 1M7, dimostrando una buona risoluzione di picco e intensità, con il minimo decadimento del segnale in tutta la traccia (a sinistra). Leggi lunghezza è massimo in queste condizioni. In contrasto, l'assenza di int medioensità, picchi ben risolti in (A) suggerisce una concentrazione non ottimale delle 1M7. Al contrario, il decadimento evidente segnale in (C) indica che i singoli hit cinetica non viene osservata, e l'RNA è finita-modificato. In tali casi, specialmente quando RT non ci si aspetterebbe di incontrare il 5 'terminale della stampo di RNA, legga lunghezza sarà subottimale.
- Preparare il brodo 10X di reagente SHAPE (NMIA o 1M7). Ciò si ottiene con l'aggiunta di una piccola quantità di reagente ad una provetta da microcentrifuga 1,5 ml, quindi aggiungendo DMSO per ottenere la concentrazione desiderata. Attenzione: soluzioni reagenti forma deve rimanere anidro fino mescolato con RNA. Conservare DMSO in essiccatore a temperatura ambiente, e preparare soluzioni madre immediatamente prima dell'uso per ridurre al minimo l'esposizione al vapore acqueo ambiente.
Reattivo Optimum concentrazione 10 volte (in DMSO) Tempo dicompleta degradazione del reagente 27 NMIA 10-100 mM ~ 20 min 1M7 10-50 mM 70 sec
Tabella 1. Reagenti elettrofili utilizzati per la modifica RNA. - Aggiungere 8 ml 10X NMIA/1M7 o DMSO anidro a modified (+) e di controllo (-) si mescola, rispettivamente Note:. 2,5 mm ha dimostrato di essere una concentrazione di partenza efficace sia NMIA e 1M7, indipendentemente dalla RNA da analizzare.
- Incubare a 37 ° C per 50 min (NMIA) o 5 min (1M7), come appropriato.
- RNA precipitato per aggiunta di 8 microlitri (0,1 vol) di 3 M NaOAc (pH 5,2), 8 microlitri 100 mM EDTA, 240 microlitri (3 vol) di etanolo freddo ed 1 ml 10 mg / ml di glicogeno. Mettete in frigorifero per 2 ore e poi centrifugare a 14000 g per 30 min a 4 ° C. Lavare il pellet due volte con etanolo al 70% freddo. Attenzione: È importanteminimizzare il tempo di refrigerazione, il tempo e la velocità di centrifugazione, per minimizzare co-precipitazione del sale, poiché ciò può influenzare negativamente risoluzione picco durante l'elettroforesi.
- Rimuovere il surnatante con una micropipetta, e aria secca pellet per 5 min a temperatura ambiente.
- Sciogliere RNA precipitati in 10 microlitri di buffer TE e incubare 5 minuti a temperatura ambiente. Questo è abbastanza sciolta RNA per due reazioni di trascrizione inversa. Conservare la parte non utilizzata a -20 ° C. Attenzione: risospensione meccanica del pellet non è di solito necessario, e può danneggiare l'RNA.
3. Trascrizione inversa
Questo passaggio genera i prodotti cDNA fluorescenza marcata che vengono utilizzati per identificare indirettamente il grado di nucleotidi di RNA sono stati modificati da un reagente SHAPE. Per la forma, le prestazioni di Superscript III (Invitrogen) RT era superiore a tutti gli altri RT testato, ed è l'enzima scelto per l'uso con questoprotocollo. Oligonucleotidi marcati con Cy5 e Cy5.5 sono usati per adescare la (+) e (-) reazioni, rispettivamente. Per RNAs più brevi, primer sono ibridate ad un 3 'estensione terminale del RNA nativo (ad esempio, un "cassette struttura") al fine di ottenere informazioni sul terminale 3' 4. Attenzione: Da questo punto attraverso CE, i campioni devono essere protetti da luce.
- Preparare (+) e (-) i campioni per la trascrizione inversa in provette da 0,5 ml microcentrifuga. Per la reazione (+) RT, miscelare 5 ml di RNA modificati (+), 6 microlitri di acqua e 1 ml Primer Cy5-marcato (10 pM), per la (-) reazione di RT, mescolare 5 microlitri controllo RNA (-), . 6 microlitri di acqua e 1 ml Cy5 marcato Primer (10 micron) Attenzione: Sarstedt provette PCR (REF 72.735.002) sono raccomandati per questa applicazione.
- Porre le provette in un termociclatore e temprare innesco a RNA e prepararsi per la trascrizione inversa, applicando il seguente programma: 85 ° C, 1 min, 60 ° C, 5 min;35 ° C, 5 min, 50 ° C, tenere premuto.
- Durante la fase di ricottura, preparare abbastanza 2.5X mix RT per il numero di reazioni da eseguire, più il 50% (ad esempio per due (+) e due (-) reazioni, scala 4,5 volte). Una reazione richiede 8 microlitri, come segue: 4 microlitri tampone 5x RT, 1 ml 100 mM DTT, 1,5 microlitri di acqua, 1 microlitri 10 mM dNTPs, 0,5 microlitri SuperScript III RT. Tenere su ghiaccio. Attenzione: tampone RT 5X e 100 mM DTT sono forniti con il SuperScript III RT.
- Una volta che la temperatura delle miscele ricottura raggiunge i 50 ° C, aggiungere 8 ml di 2.5X RT miscela per il (+) e (-) reazioni Raccomandazione:. Warm il mix RT a 37 ° C per 5 minuti prima di aggiungere le reazioni .
- Incubare per 50 min a 50 ° C, poi raffreddare a 4 ° C e / o posto sul ghiaccio. Nota: L'incubazione delle reazioni RT per più di 50 min può comportare prodotti cDNA aberranti.
- Idrolizzare RNA aggiungendo 1 ml 4 M NaOH e riscaldamento a 95 ° C per 3 min. Reazioni raffreddare su ghiaccio e poi li neutralizzano con l'aggiunta di 2 ml di 2 M HCl. Attenzione: L'omissione di questo passo si traduce in separazione cattiva qualità dei prodotti di cDNA.
- Combinare (+) e (-) e reazioni precipitare il cDNA aggiungendo 0,1 vol di NaOAc 3 M, 0,1 vol di 100 mM EDTA, 1,5 vol di etanolo freddo ed 1 ml di 10 mg / ml di glicogeno. Mettete in frigo per 2 ore, poi centrifugare a 14000 g per 30 min a 4 ° C. Lavare il pellet due volte con il freddo il 70% di etanolo. Attenzione: centrifugazione a tassi più elevati o per un più lungo periodo di risultati in difficoltà risospendere il pellet (s).
- Risospendere pellettato cDNA in 40 ml di formammide deionizzata mediante riscaldamento a 65 ° C per 10 minuti, seguita da vortex vigorosa per più di 30 min. Attenzione: Pellet può essere invisibile. Mancanza di segnale o segnale debole elettroforesi seguente può essere il risultato di guasto di sciogliere adeguatamente il pellet in questa fase.
Scale di sequenziamento servono come marcatori per determinare la posizione dei nucleotidi durante l'elaborazione dei dati. Questi vengono generati utilizzando un kit Cycle Sequencing USB (# 78500), DNA avente la stessa sequenza delle RNA in fase di studio, e primer marcati con D2 WellRed o D1/Lycor 800. Tipicamente, il DNA impiegato in questa reazione è la stessa utilizzata come stampo per la trascrizione degli RNA in questione. Anche se il protocollo di reazione presentata qui da vicino assomiglia a quella raccomandata dal costruttore del kit, la reazione è calcificato diverse volte. Mentre DDA e ddT sono utilizzati come catena terminatori nelle reazioni descritte di seguito, qualsiasi coppia di terminatori può essere utilizzato per generare le scale sequenziamento.
- Miscelare 40 ml di miscela di terminazione Dda, 5 pmol di DNA stampo, 4,6 ml di tampone Sequenase 10X, 10 ml di WellRed D2 primer marcato, 4,6 ml di Sequenase e acqua per portare il volume complessivo a 82 microlitri. Aggiungere il Sequenase scorso. Prepare una seconda reazione di sequenziamento nello stesso modo, utilizzando ddT e Licor IR800 primer marcato invece.
- Procedere alla amplificazione usando USB condizioni raccomandate. Attenzione: non è richiesta l'aggiunta di olio minerale né consigliata per i protocolli / PCR che utilizzano un coperchio riscaldato.
- Unire la Dda e DDT reazioni di sequenziamento in una provetta da microcentrifuga 1,5 ml (~ 164 ml totale).
- DNA precipitato come segue: Aggiungere 16 microlitri NaOAc 3 M (pH 5,2), 16 microlitri 100 mM EDTA, 1 ml 10 mg / ml di glicogeno, e 480 microlitri 95% di etanolo. Mescolare bene, incubare a 4 ° C per 30 min e centrifugare a 14000 xg per 30 min a 4 ° C.
- Risospendere pellettato cDNA in 100 ml di formammide deionizzata mediante riscaldamento a 65 ° C per 10 minuti, seguita da vortex vigorosa per almeno 30 min.
5. Frazionamento dei prodotti di reazione mediante elettroforesi capillare
Elettroforesi capillare permette simultaneaseparazione dei prodotti di sintesi di cDNA provenienti da quattro reazioni raggruppati in un unico campione. Otto campioni possono essere frazionata simultaneamente, mentre ben 96 campioni possono essere frazionata durante una singola corsa (figura 2).
- Miscelare 40 ml di campioni di SHAPE in pool con 10 microlitri delle scale di sequenziamento in pool, e trasferimento a piatti per campioni da 96 pozzetti. Attenzione: E 'indispensabile che i reagenti Beckman Coulter e piastre (compresi LPA-I gel, tampone di corsa, olio minerale, campione carico e soluzione del campione e le piastre di buffer) essere utilizzati con la Beckman-Coulter CEQ 8000 Genetic Analyzer.
- Programma e preparare strumento elettroforesi capillare e di avviare l'esecuzione come da istruzioni del produttore. Nota: Per una migliore risoluzione di campioni, utilizzare i parametri del metodo CAFA pubblicati in precedenza 28.
Idealmente, al di fuori di primer e picchi forte-stop, segnali per ogni picco in tutti e quattro dell'elettroferogramma tgare dovrebbero essere nel range lineare; un graduale calo dei segnali è accettabile. A volte, tuttavia, grandi picchi (fermate) sono evidenti anche nella reazione di controllo, e questi possono interferire con il conseguente trattamento dei dati. CDNA troncati che danno origine a questi picchi possono essere il risultato di un ostacolo naturale durante la trascrizione inversa (ad esempio struttura secondaria dell'RNA), o la degradazione dell'RNA. Nel primo caso, additivi come betaina potrebbe migliorare RT processività e ridurre fermandosi terminazione RT / prematuro.
Elaborazione dei dati
Software ShapeFinder permette all'utente di visualizzare e trasformare tracce CE e li converte in profili di reattività SHAPE 18. Una volta che i valori di reattività sono tabulati, si sono normalizzati e importati in RNAStructure (v5.3) per generare e perfezionare i modelli strutturali secondari.
6. ShapeFinder Software
Un'estensione del BaseFinder traccia elaborazione platmodulo 29, la versione pubblicata ShapeFinder è disponibile gratuitamente per uso non commerciale 18. Istruzioni dettagliate per la movimentazione in ShapeFinder dati sono forniti con la documentazione del software.
- Elettroferogrammi sono importate dal CEQ in ShapeFinder, dove si sono adeguati per correggere (i) di fondo fluorescente, (ii) la sovrapposizione spettrale tra i canali fluorescenti, (iii) la mobilità turni impartita dal primer in modo diverso con tag, (iv) le differenze di intensità di fluorescenza comuni prodotti etichettati con fluorofori diversi, e (v) decadimento del segnale derivante dalla cessazione anticipata di trascrizione inversa.
- La funzione "Setup" del "Allineare e integrare" strumento in ShapeFinder assegna automaticamente le identità dei singoli picchi e correla questa la sequenza di RNA come definito da input dell'utente e le due scale di sequenziamento. Anche se le assegnazioni iniziali sono generalmente imperfetto, gli errori possono essere corretti manualmente usando la funzione "Modifica" dilo stesso utensile. Infine, la funzione "Fit" calcola le aree sotto il allineati (+) e (-) i picchi di reazione, e cataloga questi valori di reattività con il corrispondente numero di nucleotidi in un file di testo delimitato da tabulazioni.
Nota: L'analisi dei dati è essenziale per l'accuratezza di SHAPE, e alcune considerazioni sono molto importante durante questa analisi, tra cui:
- Segnale-rumore: Il rapporto segnale-rumore deve essere tale che i singoli picchi dovrebbero essere facilmente identificabili anche per le posizioni con bassa reattività. Sebbene ShapeFinder fornisce un'opzione smoothing dei dati; questa alternativa dovrebbe essere usato con cautela estrema, come può la successiva analisi skew.
- Regione di analisi: in genere, dati attendibili possono essere ottenuti da cDNA 300-600 nt a lungo, a partire da una regione di 40-80 nt rimosso da innesco 3 'terminale e termina come cade il segnale a livelli difficili da distinguere dal rumore di fondo. L'uso di piùset di primer ple dovranno analizzare più tratti di RNA. In questo caso, si raccomanda che la sovrapposizione del segnale affidabile tra serie di primer è nel range di 30-50 nt. Su RNA più brevi, dove trascrittasi inversa raggiunge frequentemente la fine del template di RNA, bisogna fare attenzione ad escludere quei picchi le cui rapporto segnale-rumore è interessato dal DNA sintesi forte arresto.
- Decadimento del segnale: Il decadimento del segnale è legata alla misura del cambiamento RNA durante l'esperimento e la processività imperfetto della RT. Idealmente, cinetica single-hit relativi alla regione di RNA in analisi devono essere realizzati in modo da massimizzare leggere lunghezza. Shapefinder contiene uno strumento che è efficace nel correggere per decadimento del segnale, ma, poiché questo tende a introdurre errore nell'analisi - soprattutto quando non sono osservati singoli hit cinetica, è meglio usato quando decadimento del segnale è minimo (cioè, quando la distribuzione di picchi è coerente con il singolo hit vacchetic). Recentemente, migliorati gli algoritmi per trasformare decadimento del segnale del segnale sono stati pubblicati il 30 e dovrebbe essere indagato se il decadimento del segnale è di particolare interesse in un particolare esperimento.
- Segnale di scala. Probabilmente il passo più arbitraria nel trattamento dei dati SHAPE, il profilo di controllo deve essere adattato in modo che l'intensità dei picchi tra minimamente reattiva (+) e (-) tracce sono uguali. Scalare la traccia di controllo per troppo grande misura si tradurrà in una grande varietà di valori di reattività negativi nel primo quartile (vedi normalizzazione dei dati di seguito). In questo caso, il fattore di scala dovrebbe essere ridotto di conseguenza e dati reintegrato.
- Assegnazione Peaks. In generale, la versione automatizzata dell'assegnazione picco funziona bene. Quando il processo non riesce, tuttavia, è imperativo che l'utente di assicurarsi che tutti i picchi sono stati riconosciuti dal software, in particolare quando il rapporto segnale-rumore è basso. Picchi di spalla, per esempio, non sono sempre identificabili, e G-ricchi SEguenze sono spesso compressi.
7. Normalizzazione dei dati
Per incorporare i profili di reattività nucleotide nella struttura secondaria algoritmo usato da RNAStructure (V5.3) del software, e / o per confrontare i profili di RNA strettamente correlate, dati di forma devono essere normalizzati in modo standardizzato 12. Ciò comporta (i) esclusione di quelli fuori dai calcoli successivi, (ii) determinare la reattività "effettiva massima" (cioè, la media del massimo 8% del valore reattività, esclusione di quelli fuori), e (iii) normalizzazione dividendo tutti i valori di reattività la "massima efficacia", come segue:
- Aprire il file di testo delimitato da tabulazioni generato dopo l'allineamento e l'integrazione e copiare il suo contenuto in un foglio Excel. La colonna più a destra di questo file (RX.area-BG.area) contiene i valori assoluti reattività SHAPE calcolati per ogni nucleotidica del RNA. Le colonne più a sinistra riguardano questa reattività all'RNA paillettesrenza.
- Calcolare e memorizzare il primo e terzi i valori per (RX.area-BG.area) utilizzando la funzione di Excel "= quartile (array, quarto)" quartile (cioè, il 25 ° percentile e 75 °)
- Calcolare e memorizzare la differenza interquartile "= quartile (array, 3)-quartile (array, 1)"
- Calcolare e memorizzare il "valore di cutoff outlier" con la formula "= (quartile, array, 3) +1,5 * ((quartile (array, 3)-quartile (array, 1))". Tutti reattività valori superiori a questo valore sono ad essere esclusi dai calcoli successivi.
- Copiare i valori di reattività (RX.area-BG.area) e incollarle, colonna vuota adiacente, quindi ordinare questi valori tali che il più grandi sono nella parte superiore della colonna.
- Nella colonna "valori di ordinata" appena creato, eliminare i valori maggiori del valore di cutoff outlier.
- Calcolare e memorizzare la media del massimo 8% dei valori reattività rimanenti nel "ordinata valori column ". Questo valore è la" effettiva reattività massima ".
- Dividere il indifferenziati (RX.area-BG.area) di ciascun nucleotide (compresi valori anomali) dal "effettivo massimo" valore reattività per ottenere i "valori di reattività normalizzati". Memorizzare questi in una colonna vuota, lasciando una colonna vuota a sinistra. Quindi, copiare il numero di nucleotidi a sinistra della tabella e incollarli nella colonna vuota direttamente alla sinistra dei "valori di reattività normalizzati".
- Copiare e incollare le coppie di valori di reattività posizione-normalizzati nucleotide in un editor di testo.
- Eliminare i valori inferiori a -0.09 (ossia, lasciare spazi in bianco), in quanto questi sono probabilmente il risultato di RT pausa durante la sintesi del DNA per motivi diversi modificazione chimica del modello. Inoltre, i valori di reattività dei nucleotidi a cui si osserva una forte pausa sul modello non modificato (come determinato dal controllo visivo del profilo "Allineare e integrare" ShapeFinder), devono essere esclusi.
- Save il file con estensione. forma per l'uso in analisi strutturale con RNAstructure (v5.3) del software.
8. Data Modeling
RNAstructure (v5.3) il software è usato per predire sperimentalmente supportata struttura secondaria dell'RNA (s) con i vincoli energetici pseudo-liberi derivati da SHAPE analisi 19. Il software fornisce rappresentazioni grafiche delle strutture più bassi di energia 2D RNA così come rappresentazione testuale di queste strutture in notazione dot-staffa. Quest'ultimo può essere importato in un visualizzatore di struttura dell'RNA di preferenze dell'utente, ad esempio Pseudoviewer 23 o Varna 22, per produrre immagini di qualità pubblicazione.
Nota: Si deve prestare attenzione nel considerare le strutture prodotte dalla (V5.3) software RNAstructure. Ad esempio, il software non può risolvere le interazioni terziarie come pseudoknots e loop che si baciano, né può distinguere se la mancanza direattività in una determinata regione è dovuto alla protezione basepairing o sterico da proteine legate. Come conseguenza, questi fattori, insieme con le energie per le singole strutture, devono essere considerati nel presentare un modello strutturale definitivo.
Risultati
RNA contenente il virus HIV-1 rev risposta elemento (RRE) ea 3 'terminale cassetta struttura 4 è stato preparato da un plasmide linearizzato mediante trascrizione in vitro, dopo di che è stato piegato da riscaldamento, raffreddamento e incubazione a 37 ° C in presenza di MgCl 2. RNA è stato esposto a NMIA e poi invertire trascritto da un primer di DNA 5'-end-marcato ibridato a cassetta struttura terminale 3 '. La libreria di cDNA risultante SHAPE, insieme a reazioni di cont...
Discussione
Presentiamo qui un protocollo dettagliato per la forma ad alta velocità, una tecnica che permette la determinazione della struttura secondaria a risoluzione singolo nucleotide di RNA di qualsiasi dimensione. Inoltre, abbinando i dati di forme sperimentali con secondari algoritmi di predizione di struttura facilita la generazione di modelli di RNA in 2D con un maggior grado di precisione di quanto sia possibile con uno dei due metodi da solo. La combinazione di primer fluorescenza marcata e automatizzato CE offre signif...
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
S. Lusvarghi, J. Sztuba-Solinska, KJ Purzycka, JW Rausch e SFJ Le Grice sono supportate dal programma di ricerca intramurale del National Cancer Institute, National Institutes of Health, USA.
Materiali
| Name | Company | Catalog Number | Comments |
| REAGENTS | |||
| N-methylisatoic anhydride (NMIA) | Life technologies | M25 | Dissolve in anhydrous DMSO |
| 1-methyl-t-nitroisatoic anhydride (1M7) | see ref. 22 | ||
| Superscript III Reverse Transcriptase | Life technologies | 18080044 | 10,000 units |
| Thermo sequenase cycle sequencing kit | Affymetrix | 78500 | |
| Materials provided by the user | |||
| RNA of interest | 6 pmol per reaction (the limit of detection will be determined by the instrument) | ||
| Sets of four 5' labeled primers (Cy5, Cy5.5, WellRed D2 and WellRed D1/Licor IR800) | Primers are complementary to the RNA and are used in reverse transcription and sequencing reactions. The listed fluorophores are optimal for the Beckman Coulter 8000 CEQ. Primers may be purchased or synthesized in house. | ||
| DNA template | DNA is used for sequencing reactions, and must contain the sequence of the RNA being studied - including any 3'terminal extension, if present. Where applicable, it is often convenient to use the RNA transcription template. | ||
| Buffers | |||
| 10x RNA renaturation buffer | 100 mM Tris-HCl pH 8.0, 1 M KCl, 1 mM EDTA | ||
| 5X RNA folding buffer | 200 mM Tris-HCl pH 8.0, 25 mM MgCl2, 2.5 mM EDTA, 650 mM KCl. (This buffer might be changed depending on the case (e.g. pH, EDTA, Mg, RNase inhibitor) | ||
| 2.5X RT mix | 4 μl 5X buffer, 1 μl 100 mM DTT, 1.5 μl water,1 μl 10 mM dNTPs, 0.5 μl SuperScript III. Note that the 5X buffer and 100 mM DTT are provided with purchase of SuperScript III (Invitrogen). | ||
| GenomeLab Sample Loading Solution (Beckman Coulter) | Attention: Avoid multiple freeze-thaw cycles | ||
| EQUIPMENT | |||
| Capillary electrophoresis | Beckman | CEQ8000 | |
| Thermocycler | varies | ||
Riferimenti
- Scott, W. G., Martick, M., Chi, Y. I. Structure and function of regulatory RNA elements: ribozymes that regulate gene expression. Biochim. Biophys. Acta. 1789, 634-641 (2009).
- Moore, P. B., Steitz, T. A. The roles of RNA in the synthesis of protein. Cold Spring Harb. Perspect. Biol. 3, a003780 (2011).
- Wilkinson, K. A., et al. High-throughput SHAPE analysis reveals structures in HIV-1 genomic RNA strongly conserved across distinct biological states. Plos Biol. 6, 883-899 (2008).
- Merino, E. J., Wilkinson, K. A., Coughlan, J. L., Weeks, K. M. RNA structure analysis at single nucleotide resolution by selective 2 '-hydroxyl acylation and primer extension (SHAPE). J. Am. Chem. Soc. 127, 4223-4231 (2005).
- Watts, J. M., et al. Architecture and secondary structure of an entire HIV-1 RNA genome. Nature. 460, 711-716 (2009).
- Xu, W., Bolduc, F., Hong, N., Perreault, J. P. The use of a combination of computer-assisted structure prediction and SHAPE probing to elucidate the secondary structures of five viroids. Mol. Plant Pathol. , (2012).
- Novikova, I. V., Hennelly, S. P., Sanbonmatsu, K. Y. Structural architecture of the human long non-coding RNA, steroid receptor RNA activator. Nucleic Acids Res. 40, 5034-5051 (2012).
- Leshin, J. A., Heselpoth, R., Belew, A. T., Dinman, J. High-throughput structural analysis of yeast ribosomes using hSHAPE. RNA Biol. 8, 478-487 (2011).
- Souliere, M. F., Haller, A., Rieder, R., Micura, R. A powerful approach for the selection of 2-aminopurine substitution sites to investigate RNA folding. J. Am. Chem. Soc. 133, 16161-16167 (2011).
- Wilkinson, K. A., Merino, E. J., Weeks, K. M. Selective 2 '-hydroxyl acylation analyzed by primer extension (SHAPE): quantitative RNA structure analysis at single nucleotide resolution. Nat. Protoc. 1, 1610-1616 (2006).
- McGinnis, J. L., Duncan, C. D. S., Weeks, K. M. High-Throughput Shape and Hydroxyl Radical Analysis of Rna Structure and Ribonucleoprotein Assembly. Method Enzymol. 468, 67-89 (2009).
- Low, J. T., Weeks, K. M. SHAPE-directed RNA secondary structure prediction. Methods. 52, 150-158 (2010).
- Das, R., Laederach, A., Pearlman, S. M., Herschlag, D., Altman, R. B. S. A. F. A. Semi-automated footprinting analysis software for high-throughput quantification of nucleic acid footprinting experiments. Rna-a Publication of the Rna Society. 11, 344-354 (2005).
- Kertesz, M., et al. Genome-wide measurement of RNA secondary structure in yeast. Nature. 467, 103-107 (2010).
- Underwood, J. G., et al. FragSeq: transcriptome-wide RNA structure probing using high-throughput sequencing. Nat. Methods. 7, 995-1001 (2010).
- Mauger, D. M., Weeks, K. M. Toward global RNA structure analysis. Nat. Biotechnol. 28, 1178-1179 (2010).
- Lucks, J. B., et al. Multiplexed RNA structure characterization with selective 2'-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Proc. Natl. Acad. Sci. USA. 108, 11063-11068 (2011).
- Vasa, S. M., Guex, N., Wilkinson, K. A., Weeks, K. M., Giddings, M. C. ShapeFinder: a software system for high-throughput quantitative analysis of nucleic acid reactivity information resolved by capillary electrophoresis. RNA. 14, 1979-1990 (2008).
- Reuter, J. S., Mathews, D. H. RNAstructure: software for RNA secondary structure prediction and analysis. BMC Bioinformatics. 11, 129 (2010).
- Pang, P. S., Elazar, M., Pham, E. A., Glenn, J. S. Simplified RNA secondary structure mapping by automation of SHAPE data analysis. Nucleic Acids Res. 39, e151 (2011).
- Deigan, K. E., Li, T. W., Mathews, D. H., Weeks, K. M. Accurate SHAPE-directed RNA structure determination. Proc. Natl. Acad. Sci. USA. 106, 97-102 (2009).
- Darty, K., Denise, A., Ponty, Y. VARNA: Interactive drawing and editing of the RNA secondary structure. Bioinformatics. 25, 1974-1975 (2009).
- Byun, Y., Han, K. PseudoViewer: web application and web service for visualizing RNA pseudoknots and secondary structures. Nucleic Acids Res. 34, 416-422 (2006).
- Brown, T., Brown, D. J. S., Eckstein, F. . Oligonucleotides and Analogues - A Practical Approach. , 20 (1990).
- Legiewicz, M., et al. The RNA Transport Element of the Murine musD Retrotransposon Requires Long-range Intramolecular Interactions for Function. J. Biol. Chem. 285, 42097-42104 (2010).
- Steen, K., Siegfried, N. A., Weeks, K. M. Syntheis of 1-methyl-8-nitroisatoic anhydride (1M7). Protocol Exchange. , (2011).
- Mortimer, S. A., Weeks, K. M. A fast-acting reagent for accurate analysis of RNA secondary and tertiary structure by SHAPE chemistry. J. Am. Chem. Soc. 129, 4144-4145 (2007).
- Mitra, S., Shcherbakova, I. V., Altman, R. B., Brenowitz, M., Laederach, A. High-throughput single-nucleotide structural mapping by capillary automated footprinting analysis. Nucleic Acids Res. 36, e63 (2008).
- Giddings, M. C., Severin, J., Westphall, M., Wu, J., Smith, L. M. A software system for data analysis in automated DNA sequencing. Genome Res. 8, 644-665 (1998).
- Aviran, S., et al. Modeling and automation of sequencing-based characterization of RNA structure. Proc. Natl. Acad. Sci. USA. 108, 11069-11074 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon