Method Article
Isolamento di precursori delle cellule B di sottoinsiemi di sangue del cordone ombelicale
In questo articolo
Riepilogo
Qui si descrive un protocollo per isolare sottoinsiemi di precursore cellule B dal sangue del cordone ombelicale. Una sufficiente quantità e qualità di acidi nucleici può essere estratto dalle cellule e utilizzati in saggi successivi utilizzando DNA o RNA.
Abstract
Il sangue del cordone ombelicale è altamente arricchito per le cellule progenitrici ematopoietiche in fasi diverse di impegno lignaggio. Abbiamo sviluppato un protocollo per isolare precursori delle cellule B in quattro diversi stadi di differenziazione. Poiché i geni sono espressi e modificazioni epigenetiche verificarsi in modo tessuto-specifico, è fondamentale per discriminare tra tessuti e tipi di cellule, al fine di essere in grado di identificare le alterazioni nel genoma e dell'epigenoma che può portare allo sviluppo della malattia. Questo metodo può essere adattata a qualsiasi tipo di cellula presente nel sangue del cordone ombelicale in qualsiasi fase di differenziazione.
Questo metodo si compone di 4 fasi principali. Primo, le cellule mononucleari sono separate mediante centrifugazione densità. Secondo, le cellule B sono arricchiti con anticorpi coniugati biotina che riconoscono e rimuovere le cellule B non dalle cellule mononucleari. Terzo le cellule B sono fluorescenti marcate con anticorpi di superficie cellulare di proteine specifiche per singole fasidi sviluppo delle cellule B. Infine, le cellule sono ordinati contrassegnati in modo fluorescente e singole popolazioni sono recuperati. Le cellule recuperate sono in quantità e qualità sufficiente per essere utilizzato in saggi di acido nucleico a valle.
Introduzione
Al fine di identificare aberrazioni che sono presenti nella malattia, è di fondamentale importanza che usiamo tessuti sani o cellule che corrispondono al tipo di tessuto o cellule colpite dalla malattia. Una ragione di ciò è che la variazione epigenetica tra tipi di tessuto è responsabile della regolazione dell'espressione genica ed è fondamentale per la differenziazione cellulare durante il normale 1,2 sviluppo umano. Una seconda ragione è che aberrante regolazione genica tessuto specifica può avere conseguenze disastrose sullo sviluppo normale ed è noto per contribuire a una moltitudine di stati patologici tra cui il cancro. Pertanto, una migliore comprensione di una malattia che coinvolge le cellule ematopoietiche richiede la conoscenza delle cellule ematopoietiche sane.
Lo sviluppo di cellule ematopoietiche in procede midollo osseo attraverso un ordine sistematico di eventi caratterizzati da cambiamenti nell'espressione di marcatori di superficie cellulare 3. Gli studi che coinvolgono i partecipanti adulti hannodimostrato che il midollo osseo contiene di solito un basso numero di cellule B precursori 4,5; che coinvolgono studi pediatrici partecipanti hanno mostrato che la percentuale di precursore cellule B è relativamente alta in individui meno di 5 anni di età 6. Il sangue del cordone ombelicale viene utilizzato come fonte di cellule staminali ematopoietiche nel trattamento di malattie del sangue e tumori correlati, è disponibile attraverso banche del sangue del cordone e si arricchisce di B e cellule T immature 7, che sono le cellule bersaglio di disturbi multipli compresa leucemia e linfomi.
Precursore cellule B nel midollo osseo sono stati ampiamente fenotipizzati 8,9 e può essere definita dalla presenza di marcatori specifici della superficie cellulare che possono essere utilizzati per ordinare queste cellule in sottoinsiemi distinti. Normali cellule B differenziazione procede attraverso una serie di fasi nel midollo osseo a partire con le prime pro-B cellule e che si conclude con immaturi o ingenuo B-cellule. Secondoa van Zelm e colleghi 10, pro-cellule B sono caratterizzate dalla presenza di CD34 e nel passaggio alla fase 2 (Pre-BI) CD19 viene acquisita. Cellule Fase 3 (Pre-BII) non è più espresso CD34 e cominciare a esprimere IgM citoplasmatica. Infine, una caratteristica della fase 4 (immature cellule B) è l'espressione di IgM di superficie. La strategia di ordinamento descritto in questo protocollo è stato descritto da Caldwell e colleghi 6 e include l'uso di marcatori cellulari soli 3 superficie che riduce notevolmente la complessità e il costo di esecuzione di esperimenti selezione cellulare. Nel loro lavoro, una relazione tra CD45 e le fasi di B-differenziazione delle cellule è stato stabilito. Essi hanno osservato che cellule B nel midollo osseo visualizzare diversi livelli di espressione di CD45. Specificamente, le cellule che esprimono alti livelli di CD45 corrispondeva a cellule che esprimono IgM superficie immature (cellule B), quelli che esprime un livello intermedio di CD45 corrispondeva a cellule che esprimevano cytoplasmic IgM (pre-BII cellule), e quelli che esprimono livelli bassi di CD45 corrispondeva a cellule che non esprimono IgM citoplasmatica (pre-cellule BI). Questo protocollo utilizza la strategia sviluppata da Caldwell e colleghi 6 isolare sottoinsiemi di precursori delle cellule B di sangue del cordone ombelicale (Figura 1) che possono essere utilizzati in saggi a valle richiedono elevate qualità acidi nucleici come il CpG metilata-assay isola recupero (MIRA ) 11 e quantitative real time PCR. Il metodo impiega una separazione iniziale con biglie magnetiche ad esaurire tutti i non-B cellule da sangue del cordone ombelicale e richiede solo 3 colorazione con anticorpi (CD34, CD19 e CD45). Le cellule che sono state recuperate rappresentano 4 stadi di B-differenziazione delle cellule: 1) CD34 +, CD19 + (fine) pro-B e l'inizio del pre-BI, 2) CD34 -, CD19 +, CD45 bassa (fine pre-BI); 3) CD34 -, CD19 +, CD45 med (pre-BII) e 4) CD34 -; CD19 +, CD45 alta (immature cellule B).
Protocollo
1. Isolamento di cellule mononucleate da sangue del cordone ombelicale
- Preparare EDTA-tampone PBS-aggiungere 5 ml di albumina di siero bovino (BSA) soluzione madre a 95 ml di tampone di lavaggio (diluizione 1:20). Degas il buffer e mantenere il buffer su ghiaccio. IMPORTANTE: La mancata degassare il buffer può causare risultati non ottimali quando isolare CD19 + cellule B perché le bolle possono bloccare la colonna di isolamento.
- Preparare 50 ml provette coniche per la centrifugazione in gradiente di densità. Determinare il numero di tubi necessari per l'elaborazione del sangue del cordone ombelicale (1 tubo in grado di elaborare 8 ml di sangue) e aggiungere 15 ml di Ficoll-Paque PLUS ad ogni provetta.
- Diluire 8 ml di sangue di cordone con 24 ml di DPBS (1X) e accuratamente la miscela diluita strato sangue del cordone sopra il Ficoll-Paque PLUS in ciascuno dei 50 ml provette coniche. Non mescolare il sangue e Ficoll-Paque PLUS. IMPORTANTE: Per evitare la miscelazione del sangue del cordone ombelicale e Ficoll-Paque PLUS, tenere il tubo ad angolo di 45 gradi estrato della miscela sangue lentamente.
- Centrifugare a 400 xg per 40 min a 20 ° C. Cellule mononucleate (MNC) rimarrà al plasma-Ficoll-Paque PLUS interfaccia che granulociti ed eritrociti sedimenti grazie alla maggiore densità alla pressione osmotica di Ficoll-Paque PLUS. Etichetta sette 5 ml a fondo tondo tubi da utilizzare nella parte 3 con il campione ID, la data e il seguente:
| Tubo | Etichetta | Scopo |
| 1 | Senza macchia | Cellule non colorati per la normalizzazione durante citometria a flusso |
| 2 | 7AAD | Per determinare la vitalità durante la citometria a flusso |
| 3 | + + + | Contiene le cellule che verrà ordinata |
| 4 | 34 + | Per raccogliere CD34 + / CD19 +(Fine pro-B - inizio pre-BI), le cellule |
| 5 | Bassa 45 | Per raccogliere CD34 - / CD19 + / CD45 bassa (pre-BI), le cellule |
| 6 | 45 med | Per raccogliere CD34 - / CD19 + / CD45 med (pre-BII), le cellule |
| 7 | Alta 45 | Per raccogliere CD34 - / CD19 + / CD45 alta (B) le cellule immature |
Coat tubi 4, 5, 6, e 7 con 2% FBS e collocare tutte le provette in ghiaccio.
- Aspirare lo strato superiore di plasma con attenzione ed evitare il contatto con lo strato di cellule mononucleate. Usando una pipetta di vetro da 10 ml, trasferire accuratamente lo strato di cellule mononucleate a un nuovo tubo da 50 ml. Unire le cellule mononucleate da tre tubi insieme in un unico tubo da 50 ml.
- Riempire il tubo con PBS, mescolare delicatamente e centrifugare a 300 xg per 10 min a20 ° C. Aspirare accuratamente il sopranatante senza disturbare il pellet di cellule. Ripetere 1x. Dopo il primo lavaggio, risospendere il pellet e trasferire in un unico tubo da 50 ml.
- Risospendere delicatamente il pellet di cellule in 200 microlitri di PBS. Rimuovere 1 ml di sospensione cellulare, aggiungere e 1 ml di PBS in una provetta da 1,5 ml microcentrifuga e ritirate conteggio (conteggio celle dopo l'immissione 50 ml di sospensione cellulare nella centrifuga). Riempire la provetta con PBS e centrifugare a 200 xg per 15 minuti per rimuovere le piastrine. Rimuovere il surnatante completamente senza disturbare il pellet.
2. Modificato B-cella di isolamento da cellule mononucleate mediante separazione MACS
- Risospendere il pellet dal punto 1.7 con 160 microlitri freddo (4 ° C)-EDTA PBS.
- Aggiungere 40 microlitri B-CLL cocktail di anticorpi biotina. Pipettare per amalgamare bene e incubare per 10 minuti a 4 ° C.
- Lavare le cellule - aggiungere 1 ml a freddo (4 ° C)-EDTA PBS per 10 milioni di cellule (cellule number determinata nella fase 1.7) e centrifugare a 300 xg per 10 min. Aspirare il supernatante completamente e risospendere il pellet cellulare in 320 microlitri freddo (4 ° C)-EDTA PBS.
- Aggiungere 80 ul anti-biotina microsfere, mescolare bene e incubare per 15 minuti a 4 ° C.
- Lavare le cellule - aggiungere 1 ml a freddo (4 ° C)-EDTA PBS per 10 milioni di cellule e centrifugare a 300 xg per 10 min. Risospendere fino a 100 milioni di cellule in 500 microlitri di freddo (4 ° C)-EDTA PBS. Tampone del volume Scala in base al numero delle cellule.
- Preparare colonne MACS e separatori MACS durante la centrifugazione (fase 2.5). Posizionare una colonna LS nel campo magnetico del separatore MACS, aggiungere 3 ml di freddo (4 ° C)-EDTA PBS sulla colonna e permettono il buffer di gocciolare attraverso la colonna. Utilizzare un contenitore per catturare il flusso attraverso. Eliminare il flusso attraverso.
- Posizionare un tubo da 50 ml sotto la colonna e pipettare la sospensione cellulare sulla colonna. Lasciare le cellule senza etichetta a gocciolare through la colonna e nel tubo da 50 ml. IMPORTANTE: Queste sono le cellule che verranno utilizzate per l'etichettatura degli anticorpi e cell sorting. Non gettare il flusso attraverso. Per recuperare tutte le celle da 2,5 passo, aggiungere un 1 ml di freddo (4 ° C)-EDTA PBS alle pareti del tubo conico. Mescolare delicatamente e pipettare la sospensione rimanente cella sulla colonna. Ripetere 1x. Procedere al passo 2,8 usando le cellule senza etichetta raccolto nel tubo conico dopo il passaggio attraverso la colonna.
- Riempire il tubo conico contenente cellule non marcate con PBS e centrifugare a 300 xg per 10 min. Rimuovere con attenzione il sopranatante senza disturbare il pellet di cellule. Aggiungere 200 pl di tampone PBS-EDTA e Risospendere delicatamente le cellule.
3. Anticorpo etichettatura e la preparazione per la selezione delle cellule
- Aspirare 1 ml di sospensione cellulare e riporre in provetta "senza macchia" (Passo 1.4). Aggiungere 500 microlitri tampone PBS-EDTA e memorizzare la provetta in ghiaccio.
- Aggiungere 20pl di ciascun anticorpo (CD19, CD34 e CD45) per milione di cellule nella sospensione cellulare rimanente. Mescolare bene e porre il tubo al buio per 30 minuti a RT. Anticorpi CD sono sensibili alla luce, eseguire i passaggi da 2-4 con le luci spente.
- Dopo 30 minuti di incubazione con gli anticorpi, aspirare 40 ml di sospensione cellulare e inserirlo nel tubo etichetta "7AAD". Aggiungere 1 ml di PBS nella provetta e centrifugare a 500 xg per 2 minuti. Rimuovere il surnatante e risospendere le cellule in 100 pl di tampone di legame. Aggiungere 7 ml di 7AAD (7-Aminoactinomycin D) e incubare al buio per 10 minuti a RT. Durante la fase di incubazione 10 min completa 3,4.
- Aggiungere PBS alla sommità del tubo contenente le cellule rimanenti marcate con anticorpi CD. Centrifugare la provetta a 500 xg per 3 min. Rimuovere il surnatante, aggiungere 500 microlitri EDTA PBS e risospendere il pellet cellulare. Trasferire l'intero volume della sospensione cellulare nella provetta "+ + +". Per recuperare tutti della sospensione cellulare add un ulteriore 1 ml di EDTA PBS al tubo da 50 ml e il trasferimento nel tubo con "+ + +".
- Dopo l'incubazione (Passo 3.3), aggiungere 300 microlitri di buffer vincolante nel tubo 7AAD. La "macchia", "7AAD" e "+ + +" campioni e il "34 +", "45 basso", "45 med" e "45" ad alta tubi sono pronti per la citometria a flusso.
4. Di separazione delle cellule utilizzando il flusso MoFlo XDP Citometro
- Set-up MoFlo per l'ordinamento: Allineare laser; stabilizzare flusso goccia; determinare ritardo goccia.
- Preparare singoli controlli compensazione del colore utilizzando Invitrogen AbC mouse tallone kit (o simile), secondo le istruzioni del produttore. Eseguire controlli a singolo colore, regolando le tensioni dei canali di fluorescenza per la separazione ottimale delle popolazioni positive e negative. Impostare coefficienti di compensazione e di applicare il parametro compensata protocollo di raccolta. Ristabilire coefficienti di compensazione ogni volta che si ottiene un nuovo lotto di anticorpi.
- Creare protocollo incluso il grafico come mostrato in Figura 2.
- Esegui campione senza macchia, impostare la tensione e guadagno per lo scatter in avanti e laterale, e di identificare le popolazioni negative fluorescenza. Poiché recupero del DNA è l'obiettivo del genere, la soglia dovrebbe essere impostata bassa (≤ 1%) in modo che il DNA contenente detriti non contaminare i campioni recuperati. * Verificare che il sistema di evacuazione Aerosol è in esecuzione in ogni momento che vivono le cellule umane sono in corso sul MoFlo.
- Eseguire una piccola aliquota di + + + di esempio (~ 50.000 eventi) per impostare la strategia di gating (Figura 2). Poiché B-cellule progenitori non disperdono in modo identico a maturare cellule B, il backgate ungated CD19 + / CD34 + popolazione sulla trama SSC vs FSC per garantire il gate linfocitario include potenziali Pro-B cellule. Da questo esempio impostare anche la popolazione negativa fluorescenza per 7AAD.
- Eseguire l'esempio 7AAD per determinare la vitalità del campione. Solo le celle di ordinamento di un campione con alta vitalità aldall'inizio (≥ 95%), come le cellule morte si macchia indiscriminatamente e può contaminare le popolazioni di ordinamento.
- Impostare le decisioni in ordine di raccogliere quattro popolazioni: CD19 + / CD34 +, CD19 + / CD34 - / CD45 basso; CD19 + / CD34 - / med CD45 e CD19 + / CD34 - / alta CD45. Includere il linfociti e cancelli discriminazione doppietto nelle decisioni di ordinamento di tutte le popolazioni.
- Per impedire inceppamenti nella punta di ordinamento, filtro + + + campione attraverso un filtro a 40 micron cella immediatamente prima selezione e ri-filtro eventuale aggregazione avviene nel campione durante l'ordinamento.
- Ordinare le cellule in provette di raccolta (Punto 1.4) rivestiti con 2% FBS in PBS in un supporto tubo di raffreddamento o ghiaccio ricco.
- Subito dopo ordinamento è completato, estrarre il DNA.
Risultati
Tra campione variazione gioca un ruolo nel successo del genere cella (Tabella 1). I campioni con buone percentuali di successo hanno bassi livelli di contaminazione detriti (Figura 3A) ed i campioni con tassi di successo povere hanno livelli elevati di contaminazione detriti (Figura 3B). Tra campione variazione può essere in qualche modo controllato se il campione di sangue del cordone è stato raccolto entro 24 ore d'imbarco (O / N priorità).
Le cellule di flusso filtrate da ciascun sottogruppo dei precursori delle cellule B sono in quantità e qualità sufficienti per eseguire isolamenti di acido nucleico. Il DNA isolato è di alta qualità e può essere utilizzato nelle analisi a valle. Abbiamo regolarmente utilizzare questo DNA in MIRA 11 per arricchire per DNA metilato (Figura 4).
| Cord Blood Data Facility | Ordina Scarso | Ordina Buono |
| Il sangue del volume Lavorare con anticoagulante (ml) | 110 | 104 |
| Globuli bianchi [10 3 / pl] | 8,68 | 11,19 |
| Linfociti [10 3 / pl] | 3,88 | 3,76 |
| Linfociti (%) | 44,70 | 33,6 |
| Globuli rossi [10 6 / ul] | 3,65 | 3,05 |
| In-house dei dati | ||
| Numero cellulare dopo Ficoll-Paque | 206 M | 309 M |
| Numero cellulare dopo la separazione MACS | 22 M | 16,5 M |
| Totale Eventi Count (MoFlo XDP) | 30,57 M | 19,97 M |
| La vitalità (%) | 98,00 | 98,00 |
| Le cellule in gate linfocitario (%) | 17,00 | 50,00 |
| CD19 + / CD34 + | ||
| Cella Numero totale | 10959 | 48316 |
| % Degli eventi totali | 0,04 | 0,24 |
| Efficienza | 88% | 88% |
| CD19 + / CD34 - / basso CD45 | ||
| Cella Numero totale | 16619 | 26941 |
| % Degli eventi totali | 0,05 | 0,13 |
| Efficienza | 89% | 87% |
| CD19 + / CD34 - / CD45 med | ||
| Cella Numero totale | 469745 | 507540 |
| % Degli eventi totali | 1,54 | 2,54 |
| Efficienza | 89% | 89% |
| CD19 + / CD34 - / alta CD45 | ||
| Cella Numero totale | 1896062 | 2047142 |
| % Degli eventi totali | 6,2 | 10,25 |
| Efficienza | 91% | 90% |
Tabella 1. Rappresentante statistiche di ordinamento delle celle. Righe 1-5 sono conteggi forniti dalla struttura di sangue del cordone ombelicale. Le righe rimanenti sono dati raccolti nel nostro laboratorio. L'ordinamento poveri conteneva alti livelli di detriti e una bassa percentuale di cellule nel gate linfocitario.
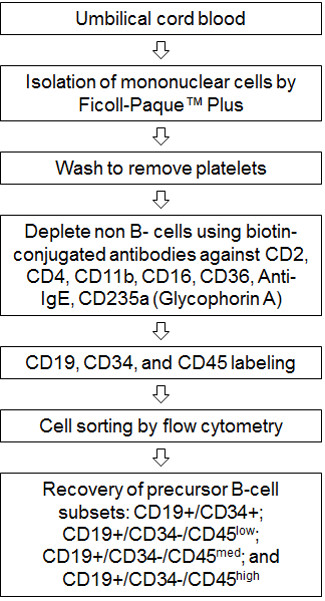
Figure 1. Diagramma di flusso della procedura. Sangue del cordone ombelicale viene elaborato e cellule mononucleate sono isolate utilizzando Ficoll-Paque plus. Una ulteriore fase di lavaggio è incluso per rimuovere le piastrine contaminanti. Cellule B vengono isolati da cellule mononucleate mediante l'umano MACS cellule B kit di isolamento (B-CLL). Questo passo utilizza anticorpi monoclonali coniugati con biotina (CD2, CD4, CD11b, CD36, anti-IgE e CD235a) per rimuovere tutti i non-B cellule. Le recuperati cellule B sono contrassegnati con CD19-APC, CD34-PE e CD45-FITC anticorpi quindi ordinati utilizzando il flusso MoFlo XDP citometro di recuperare precursori delle cellule B sottoinsiemi: CD19 + / CD34 +, CD19 + / CD34 - / CD45 bassa , CD19 + / CD34 - / med CD45 e CD19 + / CD34 - / alta CD45.
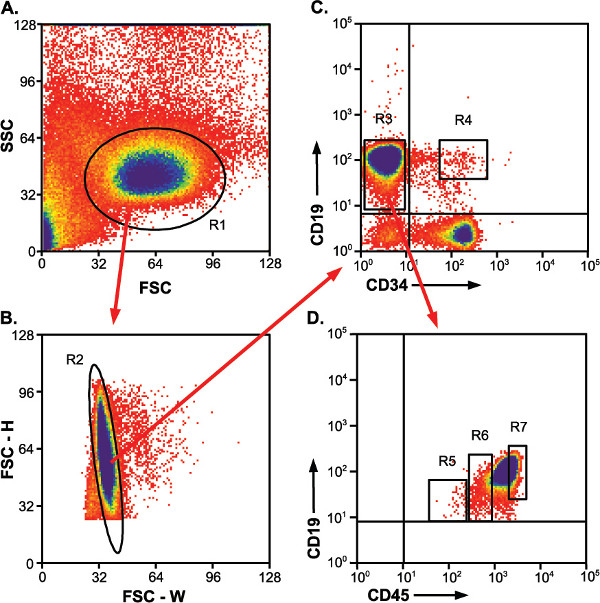
Figura 2. Strategia di gating per l'identificazione e l'ordinamento popmentari. frecce rosse indicano la porta che viene applicato al grafico successivo. Cancelli R4, R5, R6 e R7 indicano popolazioni ordinati. A) Forward scatter plot vs laterale che mostra popolazione linfocitaria arricchito. R1, gate linfocitario. B) Altezza vs larghezza del grafico a dispersione in avanti per identificare singole cellule contro doppiette. R2, porta singola cella. C) CD19-APC cellule positive rientrano nella popolazione CD34-PE negativi e positivi. R3, CD19 + / CD34 - cancello, R4, CD19 + / CD34 + popolazione cancello indica ordinare i canali d) CD19 + / CD34 - le cellule si dividono in tre in qualche modo distinti CD45-FITC popolazioni.. R5 e R6, CD19 + / CD34 - / CD45 e CD19 bassa + / CD34 - / CD45 popolazioni med, rispettivamente. R7, a causa del numero di cellule alte nel CD19 + / CD34 - / CD45 di popolazione, questa porta genere comprende solouna porzione della popolazione. Tutte le cellule di questa popolazione non hanno bisogno di essere raccolti per analisi a valle.
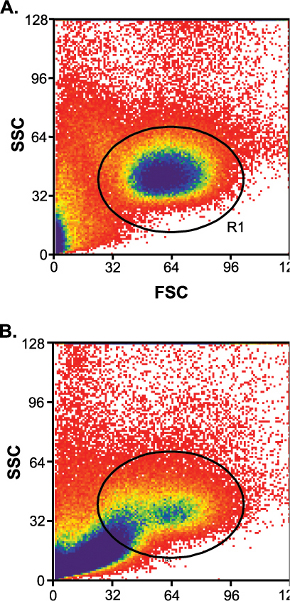
Figura 3. A) Bassi livelli di contaminanti residui dopo arricchimento colonna. R1, gate linfocitario. B) alti livelli di contaminanti residui dopo arricchimento colonna.
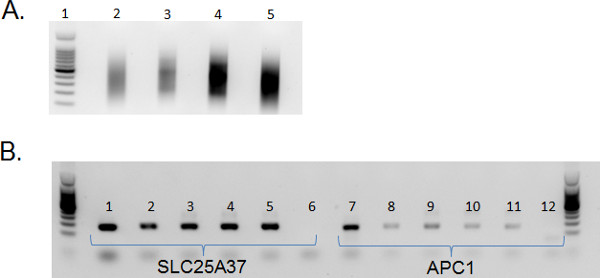
Figura 4. Arricchimento di DNA metilato da parti di precursori cellule B. A) DNA sonicato isolato da precursori delle cellule B sottoinsiemi è di alta qualità. Per DNA biblioteca costruzione è sonicata a una media di 200-600 bp. Corsia 1: Promega 100 bp ladder (catalogo # G2101), Corsia 2: DNA totale isolato da CD19 + / CD34 + cellule; Corsia 3: DNA totale isolato da CD19 + / CD34 - / CD45 delle cellule basse; Lane 4: 100 ng di DNA isolato da CD19 + / CD34 - / CD45 delle cellule med; Lane 5: 100 ng di DNA isolato da CD19 + / CD34 - CD45 / celle ad alta efficienza. DNA è stato sonicato utilizzando il Bioruptor Diagenode in alto per un totale di 9 min (30 sec ON; 30 sec OFF). DNA è stato purificato colonna e visualizzati su un gel di agarosio 1% con SYBR gel verde acido nucleico macchia. B) Dopo MIRA utilizzando il ActivMotif MethylCollector Ultra kit, PCR con SLC25A37 (1-6) e APC1 (7-12) viene effettuata per confermare arricchimento di DNA metilato. 1 e 7: DNA genomico con ultrasuoni (non MIRA), 2 e 8: MIRA-CD19 + / CD34 + DNA, 3 e 9: MIRA-CD19 + / CD34 - DNA / CD45 basso; 4 & 10: MIRA-CD19 + / CD34 - / CD45 med DNA, 5 e 11: MIRA -CD19 + / CD34 - / CD45 DNA alta, 6 e 12: Controllo delle acque. Maggiore amplificazione del SLC25A37 conferma l'arricchimento del DNA metilatonei sottoinsiemi di cellule B precursori.
Discussione
Il fattore con il maggiore impatto sul successo del protocollo è la presenza di contaminanti residui. Se la richiesta di sangue da una banca di sangue del cordone è importante avere il sangue spediti appena dopo la raccolta possibile. Inoltre, i campioni che sono classificati come linfocitosi più alto numero di linfociti, tuttavia, questi campioni non hanno un numero sufficiente di cellule B precursori e non deve essere utilizzato. Per aumentare la probabilità di ottenere un numero sufficiente di cellule per ciascuno dei sottoinsiemi precursori si consiglia iniziando con almeno 85 ml di sangue del cordone ombelicale.
È importante notare che, a differenza degli adulti separazioni sangue periferico, quando frazionamento cordone ombelicale lo strato mononucleare è spesso contaminata con globuli rossi 12. Questo protocollo include un ulteriore passaggio di lavaggio per rimuovere le piastrine contaminanti. Protocolli che descrivono separazioni mononucleate da sangue del cordone ombelicale suggeriscono di inserire una t fase di lisio rimuovere le indesiderate globuli rossi. Si sconsiglia di questo passo perché produce residui inquinanti e ha un impatto negativo sul successo del genere cella.
Al fine di ridurre il numero di cellule che devono essere distinti mediante citometria di flusso è necessario eseguire una B-cell arricchimento prima selezione cellulare. Il protocollo fornito da Miltenyi Biotec, che accompagna il B-Cell Isolation Kit (B-CLL), è stato ottimizzato per il sangue periferico e raccomanda l'utilizzo di 10 microlitri B-CLL cocktail di anticorpi biotina per 10 milioni di cellule. Questo passaggio utilizza anticorpi contro CD2 (cellule T, cellule NK), CD4 (cellule T), CD11b (granulociti, monociti, macrofagi), CD16 (cellule NK, macrofagi, mastociti), CD36 (piastrine), CD235a (cellule eritroidi) ad esaurire non-B cellule del sangue del cordone ombelicale. È importante utilizzare il kit descritto e non le cellule B Kit di isolamento II perché il kit primo contiene un anticorpo contro CD43, presente su cellule pro-B. Durante il nostro optimization di questo protocollo, abbiamo scoperto che un totale di 40 ml di B-CLL cocktail di anticorpi biotina è sufficiente a produrre un risultato positivo sorta cella. Inoltre abbiamo trovato che l'uso di non più di 5 pl più delle B-CLL cocktail di anticorpi biotina ha avuto effetti negativi per l'ordinamento delle cellule. Pertanto, si consiglia di utilizzare 40 ml di B-CLL cocktail di anticorpi biotina per un totale di numero di cellule mononucleate tra 175-250000000. Se si inizia con numeri inferiori o superiori di cellule può essere necessario per scalare i reagenti di conseguenza.
Ci sono numerose pubblicazioni contrastanti descrivono i marcatori che possono essere utilizzati per distinguere sottopopolazioni di cellule B. La maggior parte del discrepanza può essere spiegata dal fatto che la differenziazione è un processo continuo e che la presenza o l'assenza di marcatori di superficie cellulare avviene in modo graduale anziché in modo tutto o niente. In questo protocollo CD34 - / CD19 + e il livello di intensità di CD45 + (bassa, espressione media, alta) è stato utilizzato per distinguere pre-BI, pre-BII e immature cellule B con un aumento del livello di espressione di CD45 corrispondente alla progressione della differenziazione 6. Pro-cellule B sono stati descritti da alcuni come CD19 + / CD34 + 13 mentre altri hanno dimostrato che pro-B sono cellule CD19 - / CD34 + e pre-BI cellule CD19 + / CD34 + 9,10. Sulla base di queste discrepanze che hanno designato le CD19 + / CD34 + cellule più tardi pro-cellule B / primi pre-BI cellule. È importante notare che questa strategia è stata sviluppata per isolare sottoinsiemi di precursore cellule B che corrispondono alle cellule colpite dalla malattia leucemia linfoblastica acuta. Tuttavia, ci sono molteplici strategie di ordinamento che possono essere impiegati a seconda del sottotipo cella di interesse.
Anticorpo-fluorocromi combinazioni dovrebbero essere scelti in base alle capacità del cell sorter disponibili. Come generil regola, la più brillante fluorocromo nel vostro gruppo dovrebbe essere usato per etichettare l'antigene meno popoloso e viceversa. Nel rilevamento doppio macchiato popolazioni, in particolare gli eventi rari come questi, la discriminazione doppietto (Figura 2B) è una parte di vitale importanza della strategia di gating. Questo assicura che i due eventi positivi sono veramente singole cellule colorate con anticorpi sia e non solo due singole cellule colorate aderenti l'uno all'altro.
Ad alta velocità, a 4 vie separazione delle cellule crea necessariamente un ambiente controllato aerosol. Pertanto, dal vivo l'ordinamento delle cellule umane deve essere eseguita con grande cura. Pubblicato le linee guida di sicurezza e di decontaminazione sono disponibili e devono essere verificate e attuate prima del genere 14. Approvazione da comitati istituzionali biosicurezza o loro equivalenti può essere richiesta prima di ordinamento. Per disinfettarli dopo la cernita, candeggina dovrebbe essere aggiunto al contenitore rifiuti ad una concentrazione finale del 10%. Similarly, tutti i tubi di campioni così come tutte le superfici nelle immediate vicinanze devono essere accuratamente decontaminate con una soluzione appena fatto candeggina al 10%.
In sintesi, questo protocollo prevede una strategia per ottenere popolazioni rare di precursori delle cellule B e può essere modificato per isolare qualsiasi popolazione rare presenti nel sangue del cordone incluse le cellule staminali ematopoietiche e cellule T immature. Recentemente, cellule B immature sono stati identificati nel sangue periferico di individui con HIV avanzata 15. Pertanto, l'utilità di questo metodo si estende oltre lo studio di tumori del sangue. Infine, non abbiamo ancora eseguito isolamenti RNA sulle celle di flusso ordinati, tuttavia, questo metodo dovrebbe essere adattabile con l'avvertenza che, nel corso di separazione delle cellule le cellule devono essere ordinati direttamente in Trizol o una soluzione equivalente RNA compatibili come RLT dal kit RNeasy disponibile attraverso Qiagen.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto dal National Institutes of Health (NCI R00 CA132784) a KT
Materiali
| Name | Company | Catalog Number | Comments |
| DPBS (1X) | Gibco by Life Technologies | 14190-144 | |
| Ficoll-Paque PLUS | GE Healthcare Bio-Sciences AB | 17-1440-03 | |
| LS Column | MACS Miltenyi Biotec | 130-042-401 | |
| MACS Multi Stand | MACS Miltenyi Biotec | 130-042-303 | |
| MidiMACS Separator | MACS Miltenyi Biotec | 130-042-302 | |
| B-Cell Isolation Kit (B CLL) | MACS Miltenyi Biotec | 130-093-660 | |
| Fetal Bovine Serum | ATLANTA Biologicals | S11195 | |
| Microcentrifuge tube (1.5 ml) | MIDSCI | SS1500 | |
| BD Pharmingen 7-AAD (7-Aminoactinomycin D) | BD Biosciences | 559763 | |
| DB Pharmingen APC Mouse Anti-Human CD19 | BD Biosciences | 555415 | |
| DB Pharmingen PE Mouse Anti-Human CD34 | BD Biosciences | 560941 | |
| CD45 FITC | BD Biosciences | 347463 | |
| autoMACS Rinsing Solution | MACS Miltenyi Biotec | 130-091-222 | |
| MACS BSA Stock Solution | MACS Miltenyi Biotec | 130-091-376 | |
| BD Falcon 5 ml Polystyrene Round-Bottom Tube | BD Biosciences | 352058 | |
| BD Falcon 50 ml Tube | BD Biosciences | 352098 | |
| PuraFlow Sheath Fluid, 8X | Beckman Coulter | CY30230 | |
| FlowCheck Alignment beads | Beckman Coulter | 6605359 | |
| Ultra Rainbow Alignment beads | Spherotech | URFP-30-2 | |
| ViroSafe Aerosol Evacuation Filter | Beckman Coulter | ML01330 | |
| ABC Mouse bead kit | Invitrogen | A-10344 | |
| 40 μm cell strainer | Fisher Scientific | 22363547 | |
| Fisher Scientific Hemocytometer | Fisher Scientific | 267110 | |
| Microscope | |||
| accuSpin Model 3R Benchtop Centrifuge | Fisher Scientific | 13-100-516 | |
| MoFlo XDP flow Cytometer | Beckman Coulter | ML99030 | |
| Aerosol Evacuation Unit | Beckman Coulter |
Riferimenti
- Song, F., et al. Association of tissue-specific differentially methylated regions (TDMs) with differential gene expression. PNAS. 102 (9), 3336-3341 (2005).
- Ohgane, J., Yagi, S., Shiota, K. Epigenetics: The DNA methylation profile of tissue-dependent and differentially methylated regions in cells. Placenta. 29 (S), 29-35 (2008).
- Brown, G., et al. The sequential determination model of hematopoiesis. Trends Immunol. 28 (10), 442-448 (2007).
- Clark, P., et al. Lymphocyte subsets in normal bone marrow. Blood. 67 (6), 1600-1606 (1986).
- Loken, M. R., et al. Flow cytometric analysis of human bone marrow. II. Normal B lymphocyte development. Blood. 70 (5), 1316-1324 (1987).
- Caldwell, C. W., Poje, E., Helikson, M. A. B-cell precursors in normal pediatric bone marrow. American Journal of Clinical Pathology. 95 (6), 816-823 (1991).
- Tucci, A., et al. Are cord blood B cells functionally mature? Clin. Exp. Immunol. 84 (3), 389-394 (1991).
- Ghia, P., et al. Ordering of human bone marrow B lymphocyte precursors by single-cell polymerase chain reaction analyses of the rearrangement status of the immunoglobulin H and L chain gene loci. J. Exp. Med. 184, 2217-2219 (1996).
- Noordzij, J. G., et al. Composition of precursor B-cell compartment in bone marrow from patients with X-linked agammaglobulinemia compared with healthy children. Pediatric Research. 51 (2), 159-168 (2002).
- van Zelm, M. C., et al. Ig gene rearrangement steps are initiated in early human precursor B cell subsets and correlate with specific transcription factor expression. J. Immunol. 175 (9), 5912-5922 (2005).
- Rauch, T. A., Pfeifer, G. P. The MIRA method for DNA methylation analysis. Methods Mol. Biol. 507, 65-75 (2009).
- Kanof, E. M., et al. Isolation of whole mononuclear cells from peripheral blood and cord blood. Current Protocols in Immunology. , Suppl 19. 7.1.1-7.1.7 (1996).
- LeBien, T. W. Fates of human B-cell precursors. Blood. 96 (1), 9-23 (2000).
- Schmid, I., et al. International society for analytical cytology biosafety standard for sorting of unfixed cells. Cytometry A. 71 (6), 414-437 (2007).
- Malaspina, A., et al. Appearance of immature/transitional B cells in HIV-infected individuals with advanced disease: correlation with increased IL-7. PNAS. 103 (7), 2262-2267 (2006).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon