Method Article
Un Materiali guidata approccio di screening per lo sviluppo di sol-gel quantitative derivate microarray di proteine
In questo articolo
Riepilogo
Un approccio guidato screening di materiale per sviluppare sol-gel derivati proteici microarrays drogati utilizzando un metodo di pin-stampa emergente di fabbricazione è descritto. Questa metodologia è dimostrato attraverso lo sviluppo di acetilcolinesterasi e multikinase microarrays, che sono utilizzati per lo screening conveniente piccola molecola.
Abstract
Microarrays hanno trovato impiego nello sviluppo di saggi high-throughput per i nuovi materiali e la scoperta di piccole molecole lead droga. Qui si descrive un approccio guidato di screening per identificare materiale materiali a base sol-gel che sono adatti per la produzione di microarray di proteine tridimensionali. L'approccio identifica in primo luogo i materiali che possono essere stampati come microarrays, restringe il numero di materiali, identificando quelli che sono compatibili con un determinato dosaggio enzimatico, e poi affina in su materiali ottimali sulla base di ritenzione di massima attività enzimatica. Questo approccio è applicato per sviluppare microarray adatto per due differenti saggi enzimatici, uno che utilizza acetilcolinesterasi e l'altro utilizzando un set di quattro chinasi chiave coinvolti nel cancro. In ogni caso, è stato possibile produrre microarray che potrebbero essere utilizzati per quantitativi piccole molecole saggi di screening e produzione di dose dipendenti curve di risposta inibitore. È importante sottolineare che la capacità di schermare molti compagnoriali prodotte informazioni sui tipi di materiali che meglio si adattano sia la produzione di microarray e la ritenzione di attività enzimatica. I dati materiali forniscono informazioni fabbisogni di materiali di base necessari per la personalizzazione ottimale, ad alta densità di sol-gel microarrays derivati.
Introduzione
Microarrays hanno guadagnato popolarità all'interno della comunità scientifica come metodo per aumentare il throughput test. Poiché lo sviluppo della tecnologia microarray per valutare l'espressione genica 1 a metà degli anni 1990, microarrays hanno trovato impiego nello sviluppo di saggi ad alta produttività per identificare le interazioni proteina-proteina molecola proteica e-piccole, e di trovare nuovi materiali con proprietà uniche. 2-5 Più recentemente, microarrays sono stati sviluppati materiali specifici in cui sono utilizzati per immobilizzare proteine funzionali, producendo un elemento microarray tridimensionale entro cui le proteine sono intrappolate, consentendo facile misurazione dell'attività enzimatica e di inibizione sul microarray stesso utilizzando una fluorescenza idoneo saggio che è accoppiato al fatturato substrato. 5-10 È importante sottolineare che tali microarray può essere progettato per includere tutti i componenti necessari per i campioni dello schermo e controlli insieme in un modo altamente parallelo. 5,8
I primi esempi di microarray di proteine erano tipicamente preparate usando metodi standard per l'immobilizzazione di proteine su supporti solidi, come attacco covalente, 11 affinità catturare 3 e l'adsorbimento fisico. 12 Mentre questi metodi di bio-immobilizzazione consentono maggiore concentrazione del campione e cinetica di reazione richiesto per accelerati saggio di miniaturizzazione, ognuno di loro soffrono svantaggi. In generale, tutti causare una riduzione della funzionalità biomolecola nativa causa di modificazione chimica della superficie, impedito l'accesso ai siti attivi, o orientamento improprio dovuto alla immobilizzazione non specifica. Così, tutti i metodi di tradursi in una diminuzione della sensibilità del test, nonostante un aumento della deposizione di biomolecole, una risposta che probabilmente è dovuta alla necessità di legare artificialmente biomolecole ad una superficie.
Un approccio emergente per la produzione di microarrays biomolecole funzionali è attraverso pin-stampa di proteine drogatosol di silice su supporti solidi, che sono tipicamente vetrini o funzionalizzati o singoli pozzetti di micropiastre. Il processo sol-gel si svolge in un ambiente acquoso a temperatura ambiente, ed è il precursore liquido che viene stampato e poi i gel per biomolecole intrappolare all'interno della matrice 3D, consentendo per alta proteina 13 di carico, nonché intrappolamento di molteplici componenti all'interno lo stesso elemento microarray. 13,14 confezionamento del materiale derivato sol-gel può essere fatto attraverso un'attenta selezione di diversi precursori silice, nonché alterando la componente acquosa attraverso l'uso di tamponi differenti (pH, forza ionica), e l'inclusione di vari additivi (polimeri, piccole molecole) per ottenere un materiale ottimale, la specificità del quale dipende la biomolecola che viene intrappolato. 10
Una limitazione potenziale associato con lo sviluppo di sol-gel microarray di proteine derivate tramite pin-stampa èla necessità di individuare sol-gel di materiali compositi a base che possono essere stampati senza gelificazione nel perno o mostrando proprietà indesiderabili (dimensioni irriproducibili spot, cracking, scarsa adesione, incompatibilità con componenti del saggio, scarsa attività della proteina), una volta stampati su una superficie. 5 simultanea ottimizzazione di tutti questi parametri preclude un approccio materiali in cui possono essere progettati de novo, o esaminato lentamente in modo seriale. D'altra parte, lo screening casuale di molte migliaia o decine di migliaia di materiali è né tempo né conveniente.
In questo articolo, si descrive un approccio di screening diretto che permette la rapida identificazione di materiali adatti per la produzione di microarray di proteine senza la necessità di schermare in modo casuale un gran numero di materiali. Utilizzando un approccio guidato, materiali adatti per la stampa microarray vengono prima identificati, seguito da una serie di schermate di piccole dimensioni per identificare ottimale sol-gel derivato materialiAl combinazioni che possono essere stampate in modo riproducibile, senza screpolature e sono compatibili con un determinato dosaggio. Infine, ottimi materiali sono identificati in base ritenzione dell'attività enzimatica e prestazioni in un saggio di screening finale piccole molecole. In questo modo, i materiali ottimali possono essere identificati da molte migliaia di candidati utilizzando solo poche centinaia di passaggi del test. Dimostriamo questo approccio per la fabbricazione di acetilcolinesterasi sia ad alta densità e microarrays multichinasi e l'uso di tali microarrays per lo screening di piccola molecola.
Protocollo
1. Preparazione di additivi Solutions
- Preparare 25 e 50 mM Tris (idrossimetil) aminometano (Tris base, M r 121,14 g / mol) e HEPES (M r 238,3 g / mol) a pH 7.0, 7.2, 7.4, 7.6, 7.8, 8.0 e 8.2 in 50 ml centrifuga tubi. Regolare il pH con 1 N HCl e NaOH, rispettivamente. Conservare a temperatura ambiente e sostituire dopo 3 mesi.
- Preparare 24% (w / v) poli (vinil alcool) (PVA): pesare 2,4 g di PVA (M w 9.000 - 10.000, 80% idrolizzato) e aggiungere a 10 ml di acqua distillata deionizzata-(DDH 2 O), sciogliere i pellet polimero mediante vortex. Utilizzare la soluzione al 24% (w / v) preparare volumi uguali di 16% (w / v), 12% (w / v) e 8% (w / v) soluzioni PVA. Degas soluzioni mediante ultrasuoni prima dell'uso. Conservare a 4 ° C per un massimo di 1 mese.
- Preparare 24% (w / v) polietilenimmina (PEI): aggiungere 4,8 ml di PEI (M w 1.300, il 50% (w / w) in H 2 O) a 5,2 ml di DDH 2 O. Utilizzare la soluzione al 24% (w / v) preparare volumi ugualidel 16% (w / v), 12% (w / v) e 8% (w / v) soluzioni PEI. Degas soluzioni mediante ultrasuoni prima dell'uso. Conservare a 4 ° C per un massimo di 1 mese.
- Preparare 60% (w / v) di polietilenglicole (PEG): pesare 6 g di PEG (M w 600) ed aggiungere a 10 ml di DDH 2 O, dissolvere i pellet polimerici nel vortex. Utilizzare diluizioni seriali per preparare un uguale volume di 30% (w / v) di PEG. Degas soluzioni mediante ultrasuoni prima dell'uso. Conservare a 4 ° C per un massimo di 1 mese.
- Preparare 24% (v / v) N-(3-trietossisililpropil) gluconamide (GLS): aggiungere 480 ml di GLS (50% in etanolo - magazzino GLS possono richiedere l'uso di un bagno d'acqua a scaldare e sciogliere soluzione se cristallino) a 520 ml di 95% (v / v) di etanolo e mescolare per 20 min per sonicazione. Utilizzare la soluzione al 24% (v / v) per fare un volume uguale del 16% (v / v) GLS. Fai soluzioni fresche di utilizzo al giorno.
- Preparare il 24% (v / v) metiltrimetossisilano (MTMS): aggiungere 240 ml di MTMS a 760 microlitri di esistere acidificati DDH 2 O (pH 2.0, 1 N HCl) e mescolare per 20 minuti da sonicazione. Utilizzare la soluzione al 24% (v / v) per fare un volume uguale del 16% (v / v) MTMS. Fai soluzioni fresche di utilizzo al giorno.
- Preparare 24% (v / v) bis [(3-methyldimethoxylsilyl) propil] ossido di polipropilene (MDSPPO): aggiungere 240 ml di MDSPPO a 760 pl di esistere acidificati DDH 2 O (pH 2.0, 1 N HCl) e miscelare per 20 min da sonicazione. Utilizzare la soluzione al 24% (v / v) per fare un volume uguale del 16% (v / v) MDSPPO. Fai soluzioni fresche di utilizzo al giorno.
- Preparare 24% (v / v) 3 - (amminopropil)-triethoxysilane (APTES): aggiungere 240 ml di APTES a 760 pl di esistere acidificati DDH 2 O (pH 2.0, 1 N HCl) e miscelare per 20 min in sonificazione. Utilizzare la soluzione al 24% (v / v) per fare un volume uguale del 16% (v / v) APTES soluzione. Fai soluzioni fresche di utilizzo al giorno.
- Preparare 24% (v / v) bis (triethoxysily) etano (bis-TEOS): aggiungere 240 ml di bis-TEOS a 760 pl di esistere acidificato DDH 2 O (pH 2.0, 1 N HCl) E mescolare per 20 minuti da sonicazione. Utilizzare la soluzione al 24% (v / v) per fare un volume uguale del 16% (v / v) di bis-TEOS. Fai soluzioni fresche di utilizzo al giorno.
- Preparare 24% (v / v) carboxyethylsilanetriol (Si-COOH): aggiungere 960 ml di Si-COOH (25% in DDH 2 O) a 40 microlitri di esistere acidificati DDH 2 O (pH 2.0, 1 N HCl) e miscelare per 20 min da sonificazione. Utilizzare la soluzione al 24% (v / v) per fare un volume uguale del 16% (v / v) Si-COOH. Fai soluzioni fresche di utilizzo al giorno.
- Preparare a 3 mM ε N - acetil-L-lisina, D-sorbitolo, α-α-trealosio (M r 188,22 g / mol, 182.17 g / mol, e 378,33 g / mol, rispettivamente) e 1,5 mM di Triton X-100 (media M w 625 g / mol) in DDH 2 O. I volumi possono variare a seconda di quanto è richiesto, fare nuove soluzioni per l'utilizzo al giorno.
- Preparare il 60% (w / w) di glicerolo: aggiungere 2,4 g di glicerolo anidro a 1,6 g di DDH 2 O, mix di vortice. Degas soluzionemediante sonicazione prima dell'uso. Conservare a 4 ° C per un massimo di 1 mese.
- Preparare il 30% (w / w) di glicerolo: aggiungere 1,6 g di glicerolo anidro a 2,4 g di DDH 2 O, mix di vortice. Soluzione degassare per sonicazione prima dell'uso. Conservare a 4 ° C per un massimo di 1 mese.
2. Preparazione di Sols silice
Seguendo le procedure riportate di seguito, i rispettivi sol, se tenuta in ghiaccio, possono essere utilizzati fino a 1 ora dopo l'aggiunta di acqua. Sol utilizzati oltre 1 risultato h in diminuzione / incoerenti volte gelificazione del materiale.
- Preparazione di una sodio-silicato (SS) a base sol
- Pesare 120 g di resina a scambio ionico in un becher di plastica da 500 ml.
- Aggiungere 150 ml di 0,1 N HCl e mescolare per 1 ora con un 2 pollici ancoretta magnetica.
- Filtrare la soluzione su Buchner collegata al vuoto.
- Aggiungere lentamente DDH 2 O per lavare la resina filtrata fino a quando il filtrato risultante è chiaro. Questo richiede circa 100ml di DDH 2 O.
- Raccogliere e conservare la resina preparata in un contenitore di plastica a temperatura ambiente per un massimo di 1 mese.
- Pesare 2,59 g di silicato di sodio (SS, 27% (w / w) SiO 2, 10% (w / w) NaOH) in un becher di plastica da 50 ml.
- Aggiungere 10 ml di DDH 2 O per il SS. Agitare delicatamente a mano per miscelare la soluzione.
- Pesare 5,60 g di resina preparata in un bicchiere di plastica separati da 50 ml. Aggiungere alla soluzione di silicato di sodio e mescolare per 2 minuti utilizzando una pollice ancoretta magnetica.
- Filtrare la soluzione miscela con un imbuto Buchner collegato ad un aspiratore collegato ad un rubinetto d'acqua.
- Utilizzare una siringa di plastica da 10 ml, munito di 0,2 micron membrana filtro per filtrare la soluzione sol. Questo produce un sol con il 5,6% (w / w) silice indicato come SS nel testo seguente. Mantenere il sol in ghiaccio quando non in uso.
- ½ SS: aggiungere 1 ml di preparato SS sol a 1 ml di DDH 2 O, mix di vortice. Tenere il sol sul ghiaccio quando non inutilizzare.
- Preparazione di un diglycerylsilane (DGS) basata sol
- Sintetizzare DGS come descritto altrove. 15,16 Conservare DGS cristallini in un essiccatore a temperatura ambiente per un massimo di 6 mesi.
- Pesare circa 1 g di DGS cristallini.
- Utilizzare un mortaio e pestello per macinare i DGS ad una polvere fine. Completare questo passaggio il più rapidamente possibile per evitare l'assorbimento di umidità presente nell'aria.
- Trasferire con cautela i DGS finemente macinate per una fiala di scintillazione da 20 ml vuota, registrare la massa (al millesimo di grammo).
- Aggiungi DDH 2 O per produrre un 0,5 g / ml DGS.
- Sonicare i DGS idrolizzate in ghiaccio per 20 minuti, mescolare con vortex per 5 secondi ogni 5 min. Durante i giorni umidi, completa dissoluzione dei DGS può richiedere l'aggiunta di 10 ml di 1 N HCl. Questo dovrebbe essere aggiunto alla soluzione prima sonicazione.
- Utilizzare una siringa di plastica da 3 ml dotata di un filtro per filtrare la soluzione sol-membrana 0,2 micron, rimuovendo particelle fini. Questo produce un sol con il 5,0% (w / w) di silice indicato come DGS in ulteriore testo. Mantenere il sol in ghiaccio quando non in uso.
- ½ DGS: aggiungere 1 ml di preparato DGS sol a 1 ml di DDH 2 O, mix di vortice. Mantenere il sol in ghiaccio quando non in uso.
3. Pre-screening per identificare i materiali stampabili
Uno schema generalizzato mostra le varie fasi dell'analisi fattore guidata utilizzato per identificare i materiali stampabili è mostrato in Figura 1. Si raccomanda un volume minimo di materiale totale di 100 microlitri. L'uso di volumi più piccoli rende la visualizzazione gelificazione materiale difficile.
- Fase 1: tampone e silano
- Aggiungere 50 ml di tampone (25 e 50 mM Tris o HEPES a pH 7.0, 7.2, 7.4, 7.6, 7.8, 8.0, 8.2) ad una fiala di scintillazione 2 ml di vetro.
- Aggiungere 50 ml di sol appropriato (DGS, ½ DGS, SS, ½ SS) e la miscela in un vortice di moda on-and-off per 10 secondi dallaspostando il flaconcino e fuori dal vortex.
- Impostare materiali a riposare a temperatura ambiente, il monitoraggio del tempo di gelificazione materiale. Il tempo di gelificazione è definito come il punto in cui il materiale smette di fluire liberamente rotante sul flacone in un angolo di 45 °.
- Escludi combinazioni di materiali con tempi di gelificazione a meno di 2,5 ore dalle successive fasi di screening dei materiali.
- Fase 2: tampone, silani e polimeri
- In una fiala di scintillazione in vetro da 2 ml, aggiungere 3,13 ml di uno (w / v) PVA / PEI soluzione di 16% (w / v) o 8% o 6,3 ml di 30% (w / v) o 60% (w / v) di PEG.
- Aggiungere HEPES 50 mM (pH 8,0) per portare il volume combinato di 50 pl, miscelare la soluzione di vortice.
- Aggiungere 50 ml di sol appropriato (DGS, ½ DGS, SS, ½ SS), miscela nel modo descritto al punto 3.1.2.
- Escludi combinazioni di materiali con tempi di gelificazione (come definito al punto 3.1.3) a meno di 2,5 ore dalle successive fasi di screening dei materiali.
- Fase 3: tampone, silano, polimeri e organosilano
- In una fiala di scintillazione in vetro da 2 ml, aggiungere 3,13 ml di soluzione di PVA o 16% (w / v) o 8% (w / v) o 6,3 ml di 30% (w / v) o 60% (w / v) soluzione di PEG.
- Aggiungere 3,13 ml di 16% (w / v) organosilano (GLS, MTMS, MDSPPO, bis-TEOS, Si-COOH o APTES).
- Aggiungere HEPES 50 mM (pH 8,0) per portare il volume combinato di 50 pl, miscelare la soluzione di vortice.
- Aggiungere 50 ml di sol appropriato (DGS, ½ DGS, SS, ½ ß) e miscelare nel modo descritto al punto 3.1.2. Escludi combinazioni di materiali con tempi di gelificazione (come definito al punto 3.1.3) a meno di 2,5 ore dalle successive fasi di screening dei materiali.
- Fase 4: tampone, silano, additivo polimerico, organosilano e piccole molecole
- In una fiala di scintillazione in vetro da 2 ml, aggiungere 2,08 ml di soluzione di PVA o 24% (w / v) o 12% (w / v) o 6,3 ml di 30% (w / v) o 60% (w / v) soluzione di PEG.
- Aggiungi2,08 ml di 24% (w / v) organosilano (GLS, Si-COOH).
- Aggiungere 2,08 ml della soluzione di piccole molecole 3 mM (N ε - acetil-L-lisina, D-sorbitolo, α-α-trealosio) o 1,5 mM (Triton X-100).
- Aggiungere HEPES 50 mM (pH 8,0) per portare il volume combinato di 50 pl, miscelare la soluzione di vortice.
- Aggiungere 50 ml di sol appropriato (DGS, ½ DGS, SS, ½ ß) e miscelare nel modo descritto al punto 3.1.2.
- Escludi combinazioni di materiali con tempi di gelificazione (come definito al punto 3.1.3) a meno di 2,5 ore dalle successive fasi di stampa di materiale.
4. Preparazione di materiali stampabili Protein drogati
Materiali identificati al termine della fase 3.4.6 sono riportati in studi stampabilità, con l'enzima appropriato ora essere incluso nel sol. In tutti i casi di seguito delineato, soluzione acquosa contenente tutti i componenti eccetto il silanoviene preparato in un micropozzetti, seguito dall'aggiunta del sol appena prima della stampa. Per accogliere l'uso di una piastra di microtitolazione in 384, un volume totale di 50 microlitri soluzione è usato invece di 100 microlitri.
- Acetilcolinesterasi (AChE) microarray
- Preparare una kU / ml soluzione di AChE 2 in 50 HEPES (pH 8.0) a seguito del lotto attività enzimatica specifica (unità di attività per mg) le informazioni fornite dal fabbricante.
- Aliquota 2 kU / ml di enzima soluzione madre in 5 frazioni microlitri utilizzando provette da microcentrifuga da 500 microlitri e conservare a -20 ° C. 2 KU / ml soluzione madre AChE rimangono stabili fino a 4 mesi se conservato a -20 ° C.
- Preparare materiali di base, combinando la metà dei volumi di polimeri corrispondenti, organosilani e additivi piccole molecole individuate al passo 3.4.6 in micropiastre da 384 pozzetti.
- Aggiungere 5 ml di 2 kU / ml AChE in 50 HEPES (pH 8,0).
- Usando 50 mM HEPES (pH 8,0), portare l'insiemevolume perfettamente a 25 microlitri della miscela la soluzione pipettando su e giù diverse volte.
- Multikinase microarray
- Preparare 100 soluzioni chinasi ng / ml (p38α, MAPK2, EGFR, GSK-3β) in DDH 2 O dopo l'attività (U / ml e U / mg) e di dati sulle quantità fornite dal fabbricante. Conservare le soluzioni chinasi in aliquote di 2 ml a -80 ° C.
- Preparare chinasi substrati (MBP, p (E 4 Y), GSM), 5 mg / ml, 50 mg / ml o 300 mM in DDH 2 O, seguendo l'informazione quantità fornite dal produttore. Conservare substrati in aliquote da 2,5 ml a -80 ° C.
- Preparare materiali di base, combinando la metà dei volumi di polimeri corrispondenti, organosilani e additivi piccole molecole individuate al passo 3.4.6 in micropiastre da 384 pozzetti.
- Per ogni pozzetto contenente i materiali di base, aggiungere 2,5 microlitri chinasi e 2 ml di rispettivo substrato (p38α e MBP, MAPK2 e MBP, EGFR e p (E 4 Y), GSK-3β e GSM) come preparati in fasi 4.2.1 e 4.2.2, rispettivamente.
- Usando HEPES 50 mM (pH 7.4), portare il volume ben combinato a 25 pl. Mescolare pipettaggio.
5. Microarray Formazione
Questa sezione spiega la procedura dettagliata per materiali di stampa su una superficie singola diapositiva. Per stampare su più piani di scorrimento modificati (ammina, epossidiche, aldeide e PMMA), questa procedura viene ripetuta 4 volte.
- Formare microarray utilizzando un contatto pin-stampa robot dotato di un palcoscenico XYZ. Utilizzare i pin guaina maglie di 100 micron di diametro per depositare le sol proteine drogati.
- Impostare umidità all'interno della camera di stampa al 80-90%. Umidità <80% può provocare l'evaporazione del campione e incongruenze nella stampa, a causa dei piccoli volumi di deposizione.
- Sonicazione perni in DDH 2 O per 15 min prima della stampa e secco sotto corrente di azoto. Utilizzare un tubo cleaner per rimuovere l'umidità residua all'interno del supporto del perno e posizionare accuratamente il perno nella testina di stampa. La mancata rimozione di umidità residua può ostacolare il perno di muoversi liberamente con il titolare, con conseguente punti mancati.
- Modelli di matrice di controllo attraverso il programma Writer Pro Chip (CWP). Per ogni campione ben all'interno della piastra sorgente 384 pozzetti, 200 posti (massimo numero di punti per assorbimento utilizzando il perno guaina SMP3) sono stampati su una superficie modificata con vetrino.
- Iniziare il processo di stampa mediante il software. Set da viaggio in direzione XY a 10 mm / s e velocità di avvicinamento del campione (direzione Z) a 2 mm / s con un tempo di campionamento 2,5 sec caricamento.
- Mettere in pausa la corsa attraverso CWP. Abbassare il perno nel campione e aggiungere 25 ml di rispettiva sol (DGS, ½ DGS, SS, ½ SS) per il bene di pipetta. Mescolare la soluzione con un movimento su-e-giù pipettaggio ripetute 50x. Nel mescolare pipettando, minimizzare la quantità di aria compresa nella soluzione. Air bubbles prevenzionet completare il caricamento del campione all'interno del perno.
- Iniziare il processo di stampa attraverso CWP, e stampare il campione su una superficie di scorrimento. Mettere in pausa il processo di stampa prima del caricamento del campione dal piatto di origine successiva bene.
- Rimuovere il perno di stampa usando un magnete e posizionare un scovolino nella testina di stampa per evitare l'accumulo di umidità nella testina di stampa.
- Sciacquare il perno di stampa con DDH 2 O, e sonicare in pulito DDH 2 O per 30 sec.
- Essiccare il perno stampa sotto corrente di azoto e rimuovere il tubo pulito, posizionando il perno nuovamente dentro la testina di stampa.
- Iniziare la stampa seguendo i passaggi 5,6-5,10, per tutti i campioni rimanenti all'interno della piastra fonte. Fino a 12.000 punti di 100 micron di diametro può essere depositato su una singola diapositiva.
- Questo metodo può essere applicato a materiali di stampa sul fondo di pozzetti in una micropiastra a 96 pozzetti. Dopo il file di calibrazione CWP, ricalibrare il perno di stampa per garantire la distance viaggiato up tra spot deposizione è sopra l'altezza della micropiastra. Questo permette al perno di stampare coerentemente da pozzetto a pozzetto in modo lineare senza danneggiare il perno.
- Età l'array per un minimo di 30 minuti e fino a 24 ore all'interno della camera di stampa a 80-90% di umidità dopo il completamento della stampa intero esperimento.
6. Acetylcholinesterase Activity Assay
- Preparazione di controllo (PC) campioni positivi
- Preparare 1 mM acetiltiocolina ioduro (ATCH: M r 289,18 g / mol) nel 4% glicerolo, 25 mM Tris (pH 7.0) in una provetta da 1,5 ml. ATCH deve essere preparato fresco prima di ogni utilizzo e con un tampone a pH 7,0 come tamponi pH più elevati causano una rapida autohydrolysis, producendo falsi positivi. Il volume finale può essere variata a seconda del numero di campioni (25 microlitri per campione).
- Aggiungere 0,14 ml di 5 mM BODIPY-Fl-L-cistina a 25 pl di 1 mM Atch nel pozzetto di una micropiastra 384-plaTE.
- Aggiungere 24,86 ml 4% glicerolo, 25 mM Tris (pH 7.0), mescolare pipettando con attenzione per evitare la formazione di bolle nella soluzione. Potenziali inibitori enzimatici possono essere incorporati nella soluzione PC prima di portare il volume a 50 ml con tampone.
- Preparazione di controllo (NC) campioni negativi
- Aggiungere 0,14 ml di 5 mM BODIPY-Fl-L-cistina a 49,86 ml di 4% glicerolo, 25 mM Tris (pH 6,5) nel pozzetto di una piastra a 384 microtitolo. Mescolare pipettaggio.
- Sovrastampa PC e soluzioni NC
- Sovrastampa dei microarrays età seguenti passaggi 5,1-5,10 (ignorando passo 5.6) del protocollo. Utilizzare i pin guaina maglie di 235 micron di diametro a depositare la soluzioni sovrastampa NC PC. Questo assicura la soluzione copre il punto precedentemente depositato interamente.
- Array Età al 80-90% di umidità per 1 ora a temperatura ambiente. A causa della autohydrolysis di ATCH, tempi di incubazione più lunghi possono causare falsi positivi o aumento itZYME attività.
7. Attività chinasi Assay
- Preparare 500 mM di soluzione di adenosina-5'-trifosfato (ATP) con magazzino ATP (100 mm) conservati a -20 ° C. Fai la soluzione fresca per ogni esperimento e regolare il volume di necessità sperimentale: ogni soluzione di stampa necessita di 5 ml.
- Preparare una mM di cloruro di 100 magnesio (MgCl2: M r 95.21 g / mol) soluzione madre in DDH 2 O.
- Controllo positivo (PC)
- Aggiungere 2,5 ml di 100 mM MgCl 2 e 5 microlitri 500 mM ATP al pozzetto di una piastra a 384 microtitolo.
- Portare il volume totale di ben 50 ml con 50 HEPES (pH 7.4), mescolare pipettando. Potenziali inibitori enzimatici possono essere incorporati nella soluzione PC prima di portare il volume a 50 ml con tampone.
- Controllo negativo (NC)
- Aggiungere 2,5 ml di 100 mM MgCl 2 e 5 microlitri DDH 2 O al pozzo di un 384-mPiastra icrotiter.
- Portare il volume totale di ben 50 ml con 50 HEPES (pH 7.4) e mescolare pipettaggio.
- Sovrastampa PC e NC cofattori test
- Seguire il punto 6.3.1 del protocollo.
- Array Età al 80-90% di umidità per 2 ore a temperatura ambiente.
- La colorazione del vetrino
- Luogo microarray stampati scivola individualmente in una capsula di Petri con abbastanza colorante (da kit) per coprire l'intera diapositiva. Agitare moderatamente (~ 200 rpm) per 45 minuti con un agitatore.
- Rimuovere il vetrino con pinze e posto in una capsula di Petri pulita con tampone di lavaggio (da kit). Agitare moderatamente (~ 200 rpm) per 45 minuti con un agitatore.
- Rimuovere il vetrino con pinze e far girare a secco utilizzando una tradizionale desktop di microarray microcentrifuga dotato di un supporto per diapositive.
8. Microarray Imaging e analisi
- Imaging
Notare che il metodo di imaging sarà specific al tipo di lettore di matrice disponibili. Per questi studi, un Alpha Innotech NovaRay imager con una sorgente di luce bianca e sistema di rilevamento CCD dotato 478 ± 17 nm di eccitazione e 538 ± 21 nm filtro di emissione per i microarrays AChE, 530 ± 40 nm di eccitazione e 614 ± 62 nm di emissione filtrare per stato utilizzato microarrays chinasi. Sistemi di scansione laser confocale possono anche essere utilizzati per leggere gli array, se le impostazioni saranno specifiche per lo strumento.
- Mettere diapositiva nel supporto diapositive con le macchie rivolti verso l'alto.
- Impostare il numero di sezioni di anteprima (è consigliato un minimo di 2, uno su ciascuna estremità del vetrino da microscopio), la risoluzione (si consiglia un minimo di 4 micron) e l'esposizione (si consiglia auto) all'interno del software di imaging.
- Acquisire immagini diapositive e salvare come file. "Tiff".
- Analisi
- Aprire l'immagine diapositiva acquisita in ImageJ64.
- Fare clic sullo strumento di selezione ovale e misurare lasegnale di intensità di ogni spot utilizzando l'opzione di misura nella scheda analizzare. Quando si seleziona la zona da misurare, selezionare una regione leggermente più grande della macchia osservata e di dimensione coerente tra macchie per ridurre ImageJ soggettività.
- L'intensità media di 25 macchie PC simili, divisa per l'intensità media di 25 macchie simili NC avere PC / NC rapporti delle singole composizioni di materiali.
Risultati
Effettuando un'analisi fattoriale guidata per lo schermo del materiale, siamo stati in grado di ridurre al minimo il numero di materiali testati da ~ 20.000 a poche centinaia che aveva volte gelificazione adatte per la stampa. Applicando una linea guida rigorosa che richiede materiale tempo di gelificazione di 2,5 ore o superiore, materiali che possono intasare i perni di stampa o di produrre matrici riproducibili non sono mai stati stampati. I materiali stampabili individuate per avere sufficienti (> 2,5 ore) volte gelificazione sono stati stampati su 4 diversi piani di scorrimento in vetro funzionalizzate. Per essere considerato "stampabile", il numero massimo di punti per volume captazione del perno doveva essere stampato (SMP3 = 200). Spot sono stati valutati anche per posto morfologia garantire alcuna separazione di fase incrinature o indesiderabili era verificato utilizzando semplici microscopia brightfield come mostrato in Figura 2.
Da questo stadio di materiali stampabili identificati, microarrays sono stati prodotti con AChE echinasi incorporati nel componente acquosa tamponata. I materiali che erano compatibili con la procedura di dosaggio (incluso il potenziale sovrastampa e il lavaggio o la colorazione gradini) sono stati individuati osservando ritenzione di macchie di microarray (nessuna fessurazione, la perdita di macchie o insoliti pattern di fluorescenza) e un controllo positivo (PC) per il controllo negativo (NC ) rapporto superiore a 1 come osservata attraverso dell'immagine. Come questo era circa il 50% dei materiali, un maggior rapporto PC / NC di 3 è stato utilizzato per definire i materiali ottimali con ritenzione dell'attività di proteina. Attraverso questo metodo, 26 sol-gel materiali derivati contenenti materiali AChE e 2 contenenti chinasi soddisfatto i criteri> 3 PC / NC. Figure 3 e 4 mostrano una ripartizione grafica dei 5 passi guidati schermo materiali per l'individuazione di AChE e chinasi ottimale microarrays, rispettivamente.
Le analisi possono anche essere validati attraverso la generazione di un punteggio Z '17. Questo è stato fatto usando il materiale che ha prodotto il più alto rapporto PC / NC. Figura 5 mostra il diagramma Z 'ottenuta confrontando il segnale generato da 200 posti, PC 100 e 100 NC dopo sovrastampa il colorante indicatore e substrato sull'array AChE. Il dolore e le matrici chinasi hanno provocato i rispettivi punteggi Z 'di 0,60 e 0,67, indicativi di un eccellente saggio. Tuttavia, occorre notare che, prima convalida saggio, enzima, colorante, substrato e cofattore concentrazioni on-array dovevano essere ottimizzato con sovrastampa un intervallo di concentrazioni di ogni componente e selezionando la concentrazione che ha prodotto il segnale più elevato, come descritto in dettaglio altrove . 5
Per convalidare le analisi, i dati quantitativi di inibizione sono stati ottenuti utilizzando noti e sconosciuti AChE, con risultati eseguiti in duplicato e utilizzato per produrre trame duplicate (Figura 6A) e curve di inibizione (Figure 6B e 6C). sono stati stampigliati con miscele di inibitori noti piccole molecole biologicamente attive, poi con la tintura e il substrato, e le miscele di controllo contenenti sia gli inibitori noti o senza inibitori sono stati inclusi. Doppi appezzamenti sono stati generati per valutare l'attività enzimatica e qualunque miscela che hanno comportato meno del 25% dell'attività enzimatica sono stati considerati positivi per l'inibizione. Singoli composti da tali miscele sono state poi testate in duplicato per identificare la piccola molecola specifica (s) responsabile di inibizione. Una volta identificate, queste piccole molecole sono stati usati per generare curve di inibizione quantitative per determinare valori di IC 50 e le costanti di inibizione.
Risultati qualitativi simili sono stati ottenuti utilizzando la matrice multikinase con un inibitore della chinasi comune, staurosporina. Figure 7A e 7B mostrano l'immagine di microarray e indicano che le intensità di segnale dopo la sovrastampa e colorare il multikinasegamma sono come previsto per un controllo negativo (- ATP), controllo positivo (+ ATP) e noto inibitore (+ ATP + ab). Per dimostrare la capacità di ottenere dati quantitativi inibizione dai microarrays, un saggio di inibizione dipendente dalla concentrazione è stato fatto per un singolo chinasi. Come mostrato nelle Figure 8A e 8B, intensità del segnale diminuisce all'aumentare della concentrazione dell'inibitore, e la risposta segue la curva concentrazione di inibizione dipendente previsto per il sistema chinasi / substrato p38α/MBP.
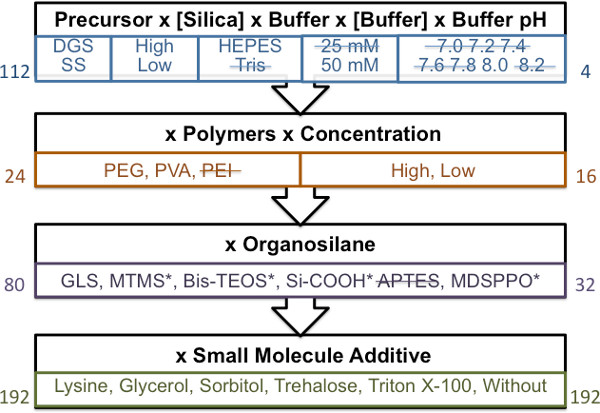
Figura 1. Schematico generale di materiali guidato approccio di screening. Ogni blocco rappresenta una fase dello schermo in ordine sequenziale. Numeri sulla sinistra rappresentano il numero totale dei materiali preparati per l'analisi. Utilizzando un tempo di gelificazione maggiore di 2,5 hr (materiali con tempi di gelificazione inferiore a 2,5 hr are indicato dalla barrato), il numero di materiali che hanno superato ogni stadio e riportati durante la schermata materiale sono indicati dal numero a destra. * Rappresenta materiali con meno di separazione ottimale di fase.

Figura 2. Immagini ottiche mostrano varie modalità di guasto dei materiali al passo stampabilità dello schermo. L'immagine di un materiale "buono" (seconda riga, terza colonna) vengono visualizzati anche per il confronto. Ristampato con il permesso di riferimento 8, copyright 2013 American Chemical Society.
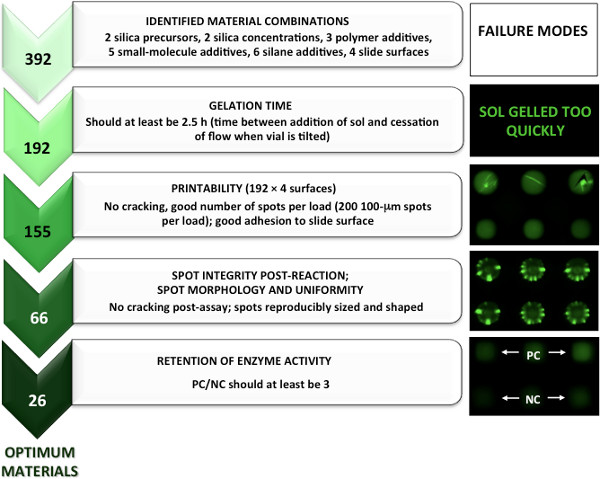
Figura 3. Un approccio di screening materiale diretto per l'identificazione di materiali ottimali per la realizzazione di sol-gel-derivati microarrays AChE. Ristampato con permissione di riferimento 5, copyright 2013 American Chemical Society.

Figura 4. Un materiale diretto approccio di screening per l'identificazione di materiali ottimali per la realizzazione di microarray chinasi sol-gel-derivati. Ristampato con il permesso di riferimento 8, copyright 2013 American Chemical Society.
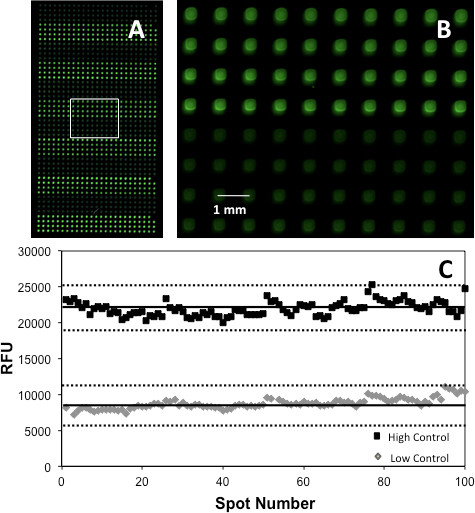
Figura 5. (A) Una sezione di microarray AChE mostrando HC (verde brillante) e LC (verde chiaro) spot (una tavolozza nero-verde è stato applicato come falsi colori per chiarezza di presentazione); (B) una vista ingrandita della zona in scatola per evidenziare posto morfologia e l'allineamento, e. (C) trama un Z 'Le linee continue indicano la media della replicati, mentre le linee tratteggiate corrispondono a 3SD. Ristampato con il permesso di riferimento 5, copyright 2013 American Chemical Society.
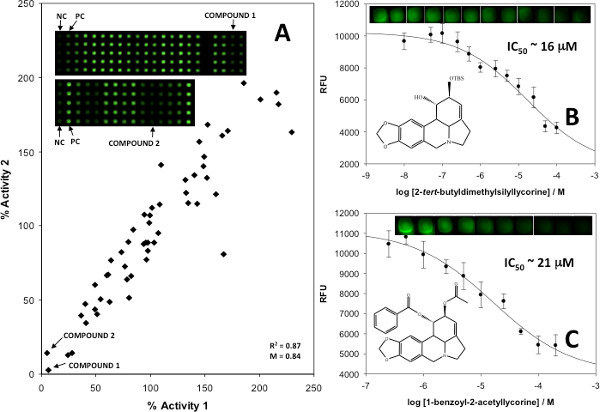
Figura 6. (A) Duplicate trama per lo screening on-serie di analoghi sintetici di alcaloidi Amaryllidaceae; (B) IC 50 appezzamenti di potenziali inibitori identificati contrassegnati come composti 1 e (C) composti 2, con barre di errore rappresentano una deviazione standard della media di 25 replicati. punti rappresentativi sono riportati per illustrare le differenze di segnale proporzionale alla concentrazione di inibitori. Ristampato con il permesso di riferimento 5, copyright 2013 American Chemical Society. Clicca qui per ingrandire la figura .
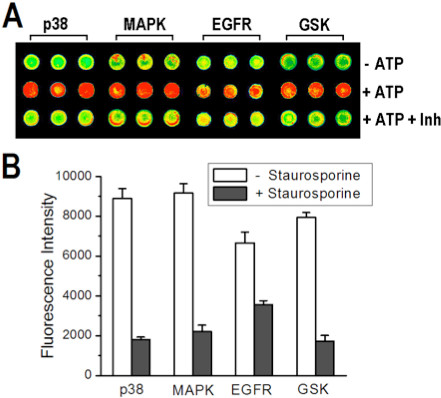
Figura 7. Assay on-array di quattro chinasi utilizzando 1.4SS/1.0PVA per intrappolamento e stampato su un vetrino ammina-derivatizzato. (A) Una immagine di una sezione di microarray in cui macchie con co-chinasi intrappolate con i rispettivi substrati sono stati sovrastampati con tampone (NC, riga superiore), o soluzioni contenenti ATP (PC, riga centrale) o ATP + staurosporina (fila in basso). (B) Istogrammi confronto intensità di segnale tra inibiti e disinibiti reazioni, dopo la sottrazione dei segnali di fondo e barre di errore rappresentano una deviazione standard della media di 25 repliche. Ristampato con il permesso di riferimento 8, copyright 2013 American Chemical Society.
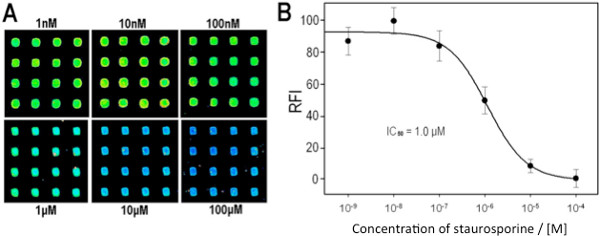
Figura 8. Inibi. sul saggio su un microarray p38a/MBP (A) Sezioni di microarray mostrano macchie rappresentativi overspotted con concentrazioni variabili di staurosporine, come indicato (le immagini sono state ottenute da una singola scansione dello stesso vetrino; immagine composita è mostrata per chiarezza). (B) IC 50 curva generata dalle immagini matrice analizzati. L'intensità ottenuti a 100 mM è stata sottratta da tutti immagini; tutte altre intensità sono stati normalizzati impostando l'intensità raggiunta a 10 nM a un valore di attività 100%. Ristampato con il permesso di riferimento 8, copyright 2013 American Chemical Society.
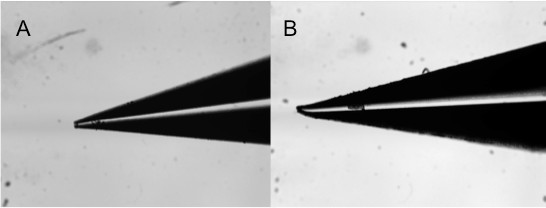
Figura 9. Immagini di pin furtività microscopico utilizzato per il contatto pin-stampa che mostra varie imperfezioni: (A) intasato, (B) piegati.
Discussione
La metodologia descritta qui è stato scelto come il più adatto per identificare stampabili sol-gel derivati con una stampante a contatto, producendo un-tempo di procedura e conveniente per identificare rapidamente materiali ottimali senza dover screening di un gran numero di materiali. Da un totale di ~ 20.000 potenziali materiali, è stato possibile individuare ~ 200 materiali che erano adatte per la stampa sulla base del tempo di gelificazione da solo. Ciò ha ridotto significativamente il numero dei materiali necessari per essere preparati per le successive prove di stampa. Questi materiali stampabili sono state poi stampate su 4 superfici di scorrimento per un totale di 768 combinazioni di materiale-scivolo. In media, 50 punti / replicati di un materiale possono essere stampati in ~ 3 min, compreso il caricamento del campione, luogo di deposizione e la pulizia pin. Di questi, 155 materiali, o circa il 20%, hanno permesso di stampare il numero massimo di punti per la soluzione di assorbimento e prodotte dimensioni piatte riproducibili. Va notato che il 4 slsuperfici ide testati, materiali stampati meglio nell'ordine: ammina> epossidica> aldeide> PMMA, PMMA diapositive non hanno prodotto gli array utili per qualsiasi materiale. Questo è stato probabilmente attribuito alla polarità del rivestimento superficiale. Confrontando le suddette superfici di scorrimento, l'ammina più polare ed epossidiche erano più adatti per i sol acquosi rispetto ai vetrini PMMA. Inoltre, le superfici testate, i vetrini rivestiti di ammine forniscono una superficie potenziale carica positiva per il depositati anionico sol di legame. Sospettiamo, la silice nanoparticelle all'interfaccia tra la slitta ed il sol interagire lungo la superficie. Sia la resina epossidica e le superfici di scorrimento aldeide non hanno la stessa carica iniziale interazione basata. Per assicurare il massimo di deposizione posto è altamente raccomandato di usare vetrini pre-rivestite da un fornitore come Arrayit. Rivestimento in house produce superfici incoerenti che portano alla scarsa riproducibilità punto 13 e, in alcuni casi, può portare alla quantificazione probblemi. 18 Di pari importanza, la temperatura e l'umidità influenzano la "stampabilità" dei materiali. Mentre non esistono studi dettagliati sugli effetti relativi alla temperatura sono state effettuate, la stampa è stata sempre effettuata a temperatura ambiente (23 ± 3 ° C). Umidità (superiore al 80%) è stato inoltre controllata all'interno della camera di stampa per impedire irregolare deposizione forma a causa di piccoli volumi di deposizione (0,7-2,3 nl) ed evaporazione.
Schermo mentre il materiale è stato guidato verso l'identificazione ottimali sol-gel materiali derivati specificamente per la stampa di AChE e chinasi, un piccolo insieme di materiali che sono stati identificati lavorato per entrambi i tipi di proteine. Infatti, sia dei materiali che sono stati identificati per la fabbricazione di microarray chinasi erano basati su SS + PVA + glicerolo, e sia i materiali sono stati identificati anche all'interno dei 26 materiali selezionati per microarray AChE. Questi materiali "ottimali" possono offrire un punto di partenza generico per sviluppare ulteriormente la protein drogati microarrays basati sol-gel, e piccoli schermi incentrati queste composizioni possono identificare i materiali ancora migliori per la fabbricazione di microarray. Un secondo punto da notare è l'importanza dell'enzima utilizzato. Nel caso di AChE (un enzima piuttosto robusto), 26 (o approssimativamente 40%) degli originali 66 materiali identificati come saggio compatibile mantenuto l'attività di AChE intrappolata. Tuttavia, per le chinasi più delicate, solo 2 delle 69 composizioni saggio-compatibili, o circa il 3% dei materiali, sono stati in grado di mantenere l'attività di tutte le chinasi. Mentre un numero sufficiente di enzimi diversi non sono stati studiati per rendere dichiarazioni conclusive, sembra che la fabbricazione gamma di ottimizzazione con enzimi relativamente instabili potrebbero portare all'identificazione di materiali che possono intrappolare una vasta gamma di proteine per permettere mutliplexed microarray fabbricazione.
Indipendente della proteina prescelta, il principale fattore di cut-off per l'identificazione di materiali stampabili stata la necessità di un lungotempi di gelificazione del materiale (> 2,5 ore). Nello sviluppo di materiali basati sulla SS sol-gel, è molto importante garantire che, dopo lo scambio ionico e filtrazione, il sol è a circa pH 4. Sol con un pH iniziale più bassa possono causare materiali con un pH inferiore al neutro, che può influenzare l'attività enzimatica. 19 Regolazione della quantità di Dowex (resina a scambio ionico) a SS può alterare il pH finale del sol. Quando si prepara un nuovo lotto della resina del rapporto di resina di SS deve essere regolata in modo da produrre sol a circa pH 4 seguendo la procedura nella sezione 2 del protocollo.
Analogamente, la preparazione di DGS cristallini è spesso fonte di errore associato a rottura del materiale quando si usa DGS sols based per l'intrappolamento biomolecola. Sebbene non riportata in dettaglio qui, grande cura deve essere presa durante la sintesi di DGS cristallini, in particolare la necessità di evitare la presenza di acqua durante la sintesi, che può produrre silice polyglyceratedTES piuttosto che DGS monomeriche. Inoltre, a causa della natura igroscopica del DGS, il campione cristallino deve essere conservato essiccato e utilizzato entro 6 mesi dopo la sintesi. DGS cristallini di età superiore ai 6 mesi non possono dissolversi completamente (a causa in parte condensato materiale silicato poligliceril) anche con sonicazione in un ambiente acido. DGS incompleta dissoluzione produce sol con un contenuto di silice sconosciuto e incontrollabile e, quindi, i materiali meno robusti.
Un punto importante da notare con la stampa a contatto è la qualità dei perni. Pin danneggiati o maltrattato (Figura 9) non potranno mai produrre matrici riproducibili indipendenti del materiale in fase di stampa. Si raccomanda di controllare la qualità del perno utilizzando un microscopio dissezione per garantire rotti o intasati non sono utilizzati. Gestione attenta assicura una lunga durata per i perni. Libera circolazione del perno è importante. Nei casi in cui l'umidità è intrappolato nella testina di stampa tra la testa e il perno, il pernonon potrà essere installato correttamente e quindi non renderà corretto contatto con la superficie, con conseguente mancanza di deposizione di materiale.
In conclusione, abbiamo fornito un approccio dettagliato lo screening a più fasi per lo sviluppo ad alta densità di proteine drogati microarrays pin-stampati. Lo screening comporta ottimizzazione delle proprietà dei materiali (tempo di gelificazione e stampabilità) per permettere la stampa di materiali, seguita da screening più concentrata per identificare materiali compatibili con un dato saggio ed in grado di mantenere l'attività enzimatica. Questo approccio di screening materiale guidata può essere applicata a formati di microarray supplementari per ridurre tempi e costi associati con la produzione di microarrays ad alta densità efficienti.
Divulgazioni
Non abbiamo nulla da rivelare.
Riconoscimenti
Gli autori ringraziano Maria Monton, Julie Lebert, Jessamyn Little, Xin Ge e Laura Lautens per l'assistenza nello sviluppo di microarray di proteine. Gli autori ringraziano anche le scienze naturali e ingegneria Research Council del Canada (NSERC) per il finanziamento di questo lavoro. Gli autori hanno anche ringraziare la Fondazione canadese per l'innovazione e l'Ontario Innovazione Fiducia per il supporto di questo lavoro. JDB detiene il Canada Research Chair in Chimica Bioanalytical e biointerfacce.
Materiali
| Name | Company | Catalog Number | Comments |
| Reagent/Material | |||
| Poly(vinyl alcohol) (PVA) | Sigma-Alderich | 360627 | 80% hydrolozyed, Mw 600 |
| Polyethylene glycol 600 (PEG) | Sigma-Alderich | 87333 | |
| Polyethylenimine (PEI) | Sigma-Alderich | 482595 | 50 wt% solution in water |
| Carboxyethylsilanetriol (Si-COOH) | Gelest, Inc. | SIC2263.0 | 25% in water |
| N-(3-triethoxysilylpropyl) gluconamide(GLS) | Gelest, Inc. | SIT8189.0 | 50% in ethanol |
| bis[(3-methyldimethoxysilyl)propyl]polypropylene oxide (MDSPPO) | Gelest, Inc. | SIB1660.0 | |
| Methyltrimethoxysilane (MTMS) | Gelest, Inc. | SIM6560.1 | |
| Bis(triethoxysiyly)ethane (Bis-TEOS) | Gelest, Inc. | SIB1817.0 | |
| 3-Aminopropyltriethoxysilane (APTES) | Gelest, Inc. | SIA0610.0 | |
| Glycerol | Sigma-Alderich | 49767 | |
| D-Sorbitol | Sigma-Alderich | 240850 | |
| D-(+)-Trehalose dihydrate | Sigma-Alderich | T9531 | |
| Triton X-100 | Sigma-Alderich | X-100 | |
| Nε-Acetyl-L-lysine | Sigma-Alderich | A4021 | |
| Tris(hydroxymethyl)aminomethane | Sigma-Alderich | 154563 | |
| HEPES | Sigma-Alderich | H3375 | |
| Sodium hydroxide, 1.0 N | LabChem Inc. | LC24350-2 | |
| Hydrochloric Acid, 1.0 N/0.1 N | LabChem Inc. | LC15300-2/LC152220-2 | |
| Magnesium chloride | Sigma-Alderich | M8266 | |
| Diglycerolsilane (DGS) | Prepared in laboratory | ||
| Sodium silicate solution | Fisher Scientific | SS338-1 | |
| Dowex 50WX8-100 ion exchance resin | Sigma-Alderich | 217492 | |
| Acetylthiocholine iodide | Sigma-Alderich | 1480 | |
| Acetylcholinesterase from Electrophorus electricus (electric eel) | Sigma-Alderich | C2888 | |
| BODIPY FL L-Cystine | Invitrogen | B-20340 | |
| Pro-Q Diamond Phosphoprotein/Phosphopeptide Microarray Stain Kit | Invitrogen | P33706 | |
| Adenosine 5'triphosphate disodium salt (ATP) solution | Sigma-Alderich | A6559 | |
| MAP Kinase 2 (MAPK2) | EMD Millipore | 454850 | |
| p38α/SAPK2a (T106M), active | EMD Millipore | 14-687M | |
| Epidermal growth factor (EGFR) | EMD Millipore | Donated by Millipore | |
| Glycogen synthase kinase 3β (GSK-3β) | EMD Millipore | 14-306 | |
| Myelin basic protein (MBP) | EMD Millipore | Substrate for MAPK2 and p38α, Donated by Millipore | |
| GSM | EMD Millipore | 12-533 | Substrate for GSK-3β |
| Poly-glu-tyr polypeptide p(E4Y) | EMD Millipore | 12-440 | Substrate for EGFR |
| Stealth pin | ArrayIt | SMP3 | |
| Stealth pin | ArrayIt | SMP7 | |
| Amine coated slides | ArrayIt | SMM2 | |
| Aldehyde coated slides | ArrayIt | SMA2 | |
| Exposy coated slides | ArrayIt | SME2 | |
| Poly(methylmethacrylate) (PMMA) coated slides | Exakt Technologies Inc. | 41500 | |
| 0.2-μm syringe filter | PALL Life Sciences | 4612 | |
| Equipment | |||
| Virtek Contact Printer | BioRad | ||
| Novaray Fluorescence Slide Imager | Alpha Innotech Corporation | ||
| Desktop microarray centrifuge | ArrayIt | MHC110V | |
| MilliQ Synthesis A10 | Millipore | Used to filter all water required for experiments |
Riferimenti
- Schena, M. M., Shalon, D. D., Davis, R. W. R., Brown, P. O. P. Quantitative monitoring of gene expression patterns with a complementary DNA microarray. Science (New York, N.Y.). 270 (5235), 467-470 (1995).
- MacBeath, G., Schreiber, S. L. Printing proteins as microarrays for high-throughput function determination. Science (New York, N.Y.). 289 (5485), 1760-1763 (2000).
- Zhu, H. H., Bilgin, M. M., et al. Global analysis of protein activities using proteome chips. Science (New York, N.Y.). 293 (5537), 2101-2105 (2001).
- Hook, A. L., Chang, C. -. Y., et al. Combinatorial discovery of polymers resistant to bacterial attachment. Nature Biotechnology. , (2012).
- Monton, M. R. N., Lebert, J. M., Little, J. R. L., Nair, J. J., McNulty, J., Brennan, J. D. A Sol-Gel-Derived Acetylcholinesterase Microarray for Nanovolume Small-Molecule Screening. Analytical Chemistry. 82 (22), 9365-9373 (2010).
- Cho, E. J., Tao, Z., Tehan, E. C., Bright, F. V. Multianalyte pin-printed biosensor arrays based on protein-doped xerogels. Analytical Chemistry. 74 (24), 6177-6184 (2002).
- Cho, E. J., Tao, Z., et al. Tools to Rapidly Produce and Screen Biodegradable Polymer and Sol-Gel-Derived Xerogel Formulations. Applied Spectroscopy. Society for Applied Spectroscopy. 56 (11), 1385-1389 (2002).
- Ge, X., Lebert, J. M., Monton, M. R. N., Lautens, L. L., Brennan, J. D. Materials Screening for Sol-Gel-Derived High-Density Multi-Kinase Microarrays. Chemistry of Materials. 23 (16), 3685-3691 (2011).
- Rupcich, N., Green, J. R. A., Brennan, J. D. Nanovolume Kinase Inhibition Assay Using a Sol-Gel-Derived Multicomponent Microarray. Analytical Chemistry. 77 (24), 8013-8019 (2005).
- Rupcich, N. Coupled enzyme reaction microarrays based on pin-printing of sol-gel derived biomaterials. Analytica Chimica Acta. 500 (1-2), 3-12 (2003).
- MacBeath, G. G., Schreiber, S. L. S. Printing proteins as microarrays for high-throughput function determination. Science (New York, N.Y.). 289 (5485), 1760-1763 (2000).
- Stillman, B. A. B., Tonkinson, J. L. J. FAST slides: a novel surface for microarrays. BioTechniques. 29 (3), 630-635 (2000).
- Rupcich, N., Goldstein, A., Brennan, J. D. Optimization of sol-gel formulations and surface treatments for the development of pin-printed protein microarrays. Chemistry of Materials. 15 (9), 1803-1811 (2003).
- Gill, I., Ballesteros, A. Encapsulation of biologicals within silicate, siloxane, and hybrid sol-gel polymers: An efficient and generic approach. Journal of the American Chemical Society. 120 (34), 8587-8598 (1998).
- Brook, M. A., Chen, Y., et al. Proteins entrapped in silica monoliths prepared from glyceroxysilanes. Journal of Sol-gel Science and Technology. 31 (1), 343-348 (2004).
- Brook, M. A., Chen, Y., Guo, K., Zhang, Z., Brennan, J. D. Sugar-modified silanes: precursors for silica monoliths. Journal of Materials Chemistry. 14 (9), 1469 (2004).
- Zhang, J., Chung, T., Oldenburg, K. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).
- . Next generation of protein microarray support materials: Evaluation for protein and antibody microarray applications. Journal of Chromatography A. , 1-8 (2013).
- Bhatia, R. B., Brinker, C. J., Gupta, A. K., Singh, A. K. Aqueous Sol-Gel Process for Protein Encapsulation. Chemistry of Materials. 12 (8), 2434-2441 (2000).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon