Method Article
Protocolli per l'implementazione di un
* Questi autori hanno contribuito in egual misura
In questo articolo
Erratum Notice
Riepilogo
Questo protocollo di cinque giorni delinea tutte le fasi, attrezzature e software supplementare necessario per la creazione e l'esecuzione di un sistema di espressione TX-TL cell-free da zero efficiente Escherichia coli endogeno basato. Con i reagenti, il protocollo dura 8 ore o meno per impostare una reazione, raccogliere e elaborare i dati.
Abstract
Sistemi di espressione acellulari ideale può teoricamente emulare un ambiente cellulare in vivo in una piattaforma controllata in vitro. 1 Questo è utile per esprimere proteine e circuiti genetici in modo controllato e per fornire un ambiente di prototipazione per biologia sintetica. 2,3 Per raggiungere il secondo obiettivo, i sistemi di espressione acellulari che conservano endogena Escherichia coli meccanismi di trascrizione-traduzione sono in grado di riflettere più accuratamente dinamiche cellulari in vivo rispetto a quelle a base di T7 RNA polimerasi trascrizione. Descriviamo la preparazione e l'esecuzione di un efficiente E. endogeno basato coli trascrizione-traduzione (TX-TL) sistema di espressione senza cellula in grado di produrre quantità equivalenti di proteine come sistemi basati T7 a una riduzione dei costi del 98% a sistemi commerciali simili. 4,5 La preparazione di tamponi e estratto cellulare grezzo sono descritto, così come l'esecuzione di untre tubo di reazione TX-TL. L'intero protocollo richiede cinque giorni di tempo per preparare e produce abbastanza materiale per un massimo di 3000 singole reazioni in una preparazione. Una volta preparato, ogni reazione prende meno di 8 ore dal setup per la raccolta e l'analisi dei dati. Meccanismi di regolazione e trascrizione esogeno di E. coli, come repressori lac / Tet e T7 RNA polimerasi, potrà essere integrata. 6 immobili endogeni, come l'mRNA e la velocità di degradazione del DNA, può anche essere adeguato. 7 Il sistema di libera espressione di cellule TX-TL è stata dimostrata per la grande montaggio scala circuito, esplorando fenomeni biologici, e l'espressione di proteine sotto le due promotori-T7 e endogeni. 6,8 modelli matematici di accompagnamento sono disponibili. 9,10 Il sistema risultante ha applicazioni uniche nella biologia sintetica come un ambiente di prototipazione, o "TX- TL biomolecolare breadboard. "
Introduzione
La tecnologia libera espressione-Cell ha iniziato nel 1950 come puramente traslazionale, avanzando anni più tardi a comprendere i meccanismi di trascrizione-traduzione accoppiati con T7 DNA del batteriofago. 11,12 Da allora, sono stati compiuti numerosi sforzi per ottimizzare la creazione di estratto cellulare grezzo (o E . estratto coli S30). 13,14 Queste ottimizzazioni includono prolungare cell-free sintesi proteica attraverso la rigenerazione dell'ATP o modifiche di deformazione, e riducendo i tempi ei costi di protocollo. 15-17 esistono sistemi di espressione cell-free alternativi che uso ricostituito componenti in sostituzione di greggio estratto di cellule per l'espressione. 5 Sia estratto cellulare grezzo e metodi di ricostituzione sono state sviluppate per uso commerciale.
Con l'avvento della biologia sintetica, vi è una maggiore necessità di una piattaforma ben caratterizzato per provare ed esprimere i moduli biologici ingegnerizzati e circuiti. 18,19 Questa piattaforma deve essereversatile, ben caratterizzato, semplice da manipolare, e focalizzato sui componenti forniti dall'utente. Nonostante sia sviluppato mezzo secolo prima, sistemi acellulari basano su E. coli intrinsecamente condividere questi requisiti, in quanto sono una rappresentazione semplificata in vitro dei processi cellulari senza la complessità di crescita e metabolismo. Inoltre, tutte le conoscenze fondamentali da lavoro in vivo su E. coli si applica facilmente a E. sistemi cell-free coli.
Anche se i sistemi di espressione cell-free possono avere applicazioni in biologia sintetica, ad oggi l'obiettivo della maggior parte dei sistemi di espressione cell-free è stata la massimizzazione delle proteine e la resa metabolita. Questo viene ottenuto usando batteriofago T7 trascrizione di sequenze guidati dai promotori T7. 20 Anche se l'espressione è efficiente e robusto, questi sistemi hanno uno scopo altamente specializzato. Metodi di regolazione delle cellule sono limitate, i modelli di DNA target deve essereriprogettato per includere i promotori T7, e alcune sequenze come i complessi ribosomali non possono essere trascritti e assemblati. 21,22 attuali sistemi di espressione cell-free non sono in grado di mantenere rendimenti elevati preservando meccanismi regolatori endogeni, la versatilità necessaria per la biologia sintetica.
Abbiamo sviluppato un E. endogeno coli sistema di espressione cell-free che conserva l'efficienza della espressione proteica dimostrata dai sistemi precedenti, ma aggiunge ulteriore versatilità, consentendo espressione e la regolazione basato su entrambi (T7 o altro) meccanismi endogeni ed esogeni. Il protocollo qui descritto si basa inizialmente su Kigawa et al. (2004) e Liu et al. (2005), ma ha modifiche significative. Utilizza Mg e K-glutammato over-Mg e K-acetato per una maggiore efficienza, rimuove 2-mercaptoetanolo, e lisa le cellule utilizzando un tallone-beater. 17,23,24 Bead-pestaggio è scelto sopra omogeneizzazione,metodi basati pressione, o sonicazione per il suo costo inferiore e rendimenti paragonabili ai sistemi concorrenti. 23 acido 3-fosfoglicerico (3-PGA) è usato come fonte di energia come è stato trovato che invia rese proteiche superiori rispetto alla creatina fosfato e fosfoenolpiruvato. 4,25 Il nostro sistema può produrre fino a 0,75 mg / ml di proteina reporter utilizzando un promotore a base sigma70-con gli operatori lambda-fago o un promotore T7-driven, simile a rese da altri sistemi commerciali. 4,6 Cinque giorni sono tenuti a produrre tutti i reagenti necessari (Figura 1). Inoltre, fornisce una riduzione dei costi del 98% rispetto ai sistemi comparabili commerciali cell-free - Costi per materie sono $ 0,11 per 10 microlitri di reazione, che sale a 0,26 dollari, con manodopera inclusa (Figura 2).
Protocollo
1. Estratto cellulare Crude Preparazione
Preparazione estratto cellulare greggio nell'arco di tre giorni richiede due persone a condurre in modo efficiente. Il protocollo è composto funzionalmente da tre parti: la crescita della cultura (passo 1.1 al passo 1.11), lisi cellulare (punto 1.12 al punto 1.37), ed estrarre chiarificazione (passo 1.38 a passo 1.52). Si presenta diviso in giorni per convenienza. Estratto ideale può produrre 0,75 mg / ml di deGFP dal plasmide pBEST-OR2-OR1-Pr-UTR1-deGFP-T500 (Addgene # 40019), ed ha una concentrazione di estratto cellulare grezzo tra 27-30 mg / ml di proteina. 4 Tuttavia , caratteristiche estratto variano da lotto a lotto. La ricetta che segue fornisce sufficiente per circa 3.000 reazioni singole (6 ml di estratto di cellule greggio). Se ridimensionamento, si consiglia di utilizzare non meno di 1/6 dei valori qui indicati. A causa di vincoli di tempo, scaling up non è raccomandato.
Giorno 1
- Preparare terreni di coltura batterica, culpiastra tura, e dei media integratori come descritto nella tabella 1. Vedi supplementare Materiale 1 per le ricette.
- . Consecutive BL21-Rosetta2 ceppo da -80 ° C su una piastra di agar 2xYT + P + Cm e incubare per almeno 15 ore a 37 ° C o fino colonie sono facilmente visibili Nota: Cloramfenicolo (Cm) viene usato per selezionare un plasmide codifica tRNA rare nel ceppo BL21-Rosetta2.
Day 2
- Preparare buffer e supplementi come descritto nella Tabella 2. Vedi supplementare Materiale 1 per le ricette.
- Preparare e sterilizzare materiali necessari per il giorno 3, tra cui: 6 x 4 palloni L Erlenmeyer con la copertura di alluminio (autoclave), 4 x 1 L bottiglie centrifuga sterili, imbuto (autoclave), 100 g di 0,1 mm perle di vetro (autoclave), 2 mescolare-bar (autoclave), 1 L e 500 ml cilindro graduato (autoclave), 2 x 1 L bicchieri (autoclave), 3 ml siringa con 18 aghi sterili (G), 2-3 flboe avena, 2-3 10k MWCO cassette di dialisi (sterile), cuvette.
- Preparare mini-cultura 1. Aggiungere 4 ml di 2xYT + P media e 4 ml di Cm per un 12 ml sterile provetta di coltura e pre-riscaldare a 37 ° C per 30 min.
- Seminare mini-cultura 1 con una colonia dal + P + piastra Cm agar 2xYT. Incubare a 220 rpm, 37 ° C per 8 ore.
- 7 ore e 30 minuti più tardi, preparare mini-cultura 2. Aggiungere 50 ml di 2xYT + P media e 50 ml di Cm in un pallone sterile da 250 ml e pre-riscaldare a 37 ° C per 30 min.
- Seminare mini-cultura 2 con 100 ml di mini-cultura 1 e incubare a 220 rpm, 37 ° C per 8 ore.
Day 3
- Pesare quattro sterili da 50 ml provette Falcon vuoti e di massa record nella tabella 3. Raffreddare provette Falcon su ghiaccio, che saranno successivamente utilizzati nella fase 1.18.
- 7 ore e 30 minuti dopo la fase 1.8, preparano finale terreni di coltura batterica. Usando una sterile 1 L cilindro graduato, trasferimento 660 ml di 2xYT + P supporti into ognuno dei sei 4 palloni L Erlenmeyer e pre-riscaldare a 37 ° C per 30 min. Nota: 4 L o superiore beute sono consigliati per una corretta aerazione.
- Aggiungere 6,6 ml di mini-cultura 2 in ciascuna beuta 4 L. Incubare a 220 rpm, 37 ° C fino a quando la coltura raggiunge un diametro di 1,5-2,0 a 600 nm (corrispondente alla fase di crescita medio-log). Verificare OD periodicamente con una diluizione 1:10 cultura per la precisione Questo passaggio non dovrebbe richiedere più di 3 ore - 3 ore 45 min;. Rapida crescita e la raccolta durante la fase di mid-log è fondamentale per la qualità dell'estratto.
- Subito dopo la crescita, trasferire tutte le culture uniformemente in quattro bottiglie da centrifuga 1 L e centrifugare a 5000 xg per 12 min a 4 ° C per sedimentare le cellule batteriche.
- Mentre centrifugazione, completa preparazione tampone S30A aggiungendo 4 ml di 1 M DTT a 2 L di S30A precedentemente preparato. Mescolare e mantenere tampone sul ghiaccio.
- Quando centrifugazione, rimuovere completamente il surnatante dal punto 1.12 da decanting e tamponando le bottiglie centrifuga su un tovagliolo di carta sterile.
- Aggiungere 200 ml di tampone S30A a 4 ° C per ciascuno dei quattro bottiglie da centrifuga, e agitare le bottiglie vigorosamente fino a pellet è completamente solubilizzato senza rimanenti grumi. Centrifugare le quattro bottiglie a 5.000 g per 12 min a 4 ° C.
- Rimuovere completamente il surnatante dal passo precedente decantando e tamponando le bottiglie centrifuga su un tovagliolo di carta sterile.
- Ripetere i punti 1.15 e 1.16.
- Aggiungere 40 ml di tampone S30A a 4 ° C per ogni bottiglia centrifuga. . Trasferire ciascun pellet e la combinazione S30A in un tubo Falcon refrigerata da 1.9) Nota: Questo passo è quello di trasferire il pellet in un contenitore più piccolo.
- Centrifugare le provette Falcon a 2,000 g per 8 minuti a 4 ° C. Rimuovere il surnatante per decantazione.
- Re-centrifugare le provette Falcon a 2.000 g per 2 min a 4 ° C. Rimuovere completamente il surnatante residuo pipetta. Tenere su ghiaccio.
- Pesare fi nostri tubi Falcon con pellet e messa record nella tabella 3. Calcolo della massa pellet, S30A polmone necessario, e la massa di perline necessari sulla base delle formule specifiche in Tabella 3.
- Carico e tallone corretto battito del pellet è fondamentale per rendere estratto di qualità, ed è il passo più difficile. Si consiglia di rivedere il video prima di tentare. Il mancato evitare bolle d'aria e distribuire uniformemente perline si tradurrà in estratto inefficiente.
- Aggiungere la quantità di tampone S30A calcolato in Tabella 3 per ogni provetta Falcon, vortex fino omogeneo, e ritorno in ghiaccio.
- Pur mantenendo le altre provette Falcon su ghiaccio, aggiungere perline intermittenza per un unico tubo Falcon in tre aliquote, ciascuno utilizzando 1/3 del totale delle perline. Dopo l'aggiunta di ciascuna aliquota di perline, vortex per 30 sec. Posizionare il tubo Falcon su ghiaccio tra gradini vortex e dopo vortice finale. Dopo aver aggiunto l'ultima aliquota, garantireperle sono uniformemente distribuite. Una pasta spessa dovrebbe essere formato.
- Preparare un 5 ml (volume) punta della pipetta tagliando la fine con una lama di rasoio sterile per creare un'apertura 3-4 mm. Comporre pipetta 2 ml Nota: Diversi set pipetta e suggerimenti forniscono diverse quantità di aspirazione, che non può essere sufficiente per tirare e rilasciare soluzioni perline celle di spessore; una pipetta da 1 ml con estremità rimosso può essere utilizzato in sostituzione..
- Mettere 20 tubi bead-battendo sul ghiaccio.
- Verificare alta viscosità della soluzione in cella stallonatore usando pipetta modificato. Dovrebbe essere viscoso al punto di appena esce dalla punta della pipetta durante l'espulsione. Se troppo viscoso, regolare puntale in base al punto 1.25. Se non abbastanza viscoso, sfere possono essere aggiunti in incrementi di (massa pellet * 0,05), ad una massa massima di (massa pellet * 5.1). Dopo ogni aggiunta di perline, vortex per 30 sec e ritorno al ghiaccio. Vedere la Figura 3a per una dimostrazione di viscosità.
- Rimuovere la soluzione bead-cella dal tubo Falcon con pipetta modificata, e trasferire in un tubo tallone battendo sterile, riempiendolo per tre quarti con la soluzione tallone-cell. Spin estremamente breve (1 sec) su un contatore mini-centrifuga per eliminare le bolle d'aria, senza ridistribuire perline. Vedere le figure 3b-d per le immagini fisse di tubo tallone-pestaggio carico.
- Finire aggiungendo soluzione tallone-cell per formare un menisco concavo.
- Aggiungere una piccola goccia di soluzione bead-cellule sulla parte interna di un tappo tubo tallone battere, facendo attenzione a non impedire il labbro esterno del tappo, altrimenti il tubo tallone battendo non si chiude sufficientl y. Toccare il tappo su una superficie piana e verificare che non vi siano bolle d'aria sul fondo del tappo.
- Tappare il tubo tallone battendo con il tappo-tallone battere dal passaggio precedente. Mano per assistente cordone pestaggio. Se fatto correttamente, il coperchio deve essere chiuso ermeticamente, senza bolle d'aria should essere visibile, e poco (se presente) la soluzione tallone-cell dovrebbe traboccare. Ripeti il processo di caricamento se bolle d'aria sono visibili o il tappo non fa chiudere completamente.
- Vortex tubo Falcon dal punto 1.24 con il restante soluzione di sfere celle per assicurare una distribuzione uniforme di perline. Ripetere i passaggi 1,28-1,31 fino a quando il tubo Falcon è vuota, quindi ripetere i passaggi 1,24-1,31 per ogni provetta Falcon supplementare.
- Condotta passi 1,33-1,38 contemporaneamente. Hanno assistente take compilato tallone battendo tubi da 1.31 e posto sul ghiaccio. Dopo due tubi-tallone battendo pieni sono stati raccolti e sono stati in ghiaccio per almeno un minuto, cominciare tallone pestaggio.
- Batti un tubo per 30 sec a 46 giri al minuto. Mettere a testa in giù sul ghiaccio per 30 secondi mentre battendo l'altro tubo.
- Ripetere passaggio precedente tale che ogni tubo tallone battendo pieno è stato battuto per 1 min totale.
- Ripetere i passaggi 1,33-1,35 fino a 8 tubi bead-battendo pieni (o l'importo massimo della centrifuga può contenere) have stati elaborati. Poi, costruire apparecchio filtrante da 15 ml Falcon (figura 3e). Aggiungere un nuovo tappo tallone battere, TV a parte a faccia in su, al fondo di un 15 ml Falcon. Quindi, rimuovere il tappo dal tubo tallone battere elaborati e premere colonna micro-cromatografia saldamente sulla fine del tubo tallone battere elaborati fino completamente sigillato. Snap off fine eluizione della colonna micro-cromatografia, e posizionare colonna micro-cromatografia, eluizione finisce giù, in tubo tallone-bordare vuoto. Inserire questo complesso in 15 ml Falcon. Ripetere l'operazione per tutte le 8 tubi-perline battendo pieni; tenere su ghiaccio al termine.
- Centrifuga 8 apparati di filtro, tubo Falcon livellata, a 6.000 g per 5 minuti a 4 ° C per separare estratto e pellet da perline.
- Verificare che ogni tubo tallone-pestaggio ha prodotto estratto praticabile. Estratto Correttamente battere non sarà torbido, e il pellet avrà due strati distinti. Eliminare tutte le provette torbidi, e trasferire il surnatante dai tubi non-torbide into singoli 1,75 ml provette micro-centrifuga, prendendo come poco pellet possibile. Tenere su ghiaccio finché non sono stati elaborati tutti i tubi-perline battere. Vedere la Figura 3f confrontando un tubo tallone-pestaggio correttamente vs correttamente elaborato.
- Provette a micro-centrifuga dal punto precedente a 12.000 g per 10 minuti a 4 ° C.
- Trasferire il surnatante privo di pellet in tubi bead-battendo vuoti utilizzando una pipetta, consolidando 500 microlitri in un nuovo tubo tallone battere.
- Incubare passaggio precedente, con tappi-perline battendo rimossi, a 220 rpm, 37 ° C per 80 min. Questo passaggio digerisce restante acidi nucleici utilizzando esonuleasi endogeni rilasciati durante il processo tallone-bordare, e può essere fatto in piedi tubo tallone battendo in una provetta di coltura tissutale.
- Preparare i materiali per dialisi. Completa S30B preparazione tampone aggiungendo 2 ml di 1 M DTT a 2 L di S30B precedentemente preparato. Mescolare e aggiungere 900 ml in ciascuna delle due sterile 1 L bicchieri. Aggiungi sterile agitatore magnetico in ogni bicchiere, conservare a 4 ° C.
- Dopo il punto 1.41, estratto dovrebbe apparire torbida. Consolidare estratto in 1,5 ml aliquote a 1,75 ml provette micro-centrifuga e centrifugare a 12000 g per 10 minuti a 4 ° C.
- Usando una pipetta, consolidare surnatante privo di pellet in 15 ml provette Falcon su ghiaccio, e mescolare bene dalla tappatura tubo e inversione. Save 10 ml di surnatante in ghiaccio per passo 1.47.
- Determinare la quantità totale di estratto prodotto, e idratare il numero necessario di 10k MWCO cassette dialisi mediante submersing in S30B per 2 min, assumendo 2,5 ml di estratto per cassette.
- Caricare cassette con 2,5 ml di estratto. Ogni bicchiere può richiedere fino a 2 cassetti, Dializzare, mescolando, a 4 ° C per 3 ore Nota: il carico parziale di cassette è accettabile.. Dialisi aumenta proteina resa di produzione.
- Durante il passaggio precedente, caratterizzare proteine estrattoconcentrazione con un saggio di Bradford, utilizzando estratto salvato nel passo 1,44. Vedi supplementare Materiale 2 per i dettagli.
- Dopo la dialisi è completa, estratto aliquota di 1,5 ml a 1,75 ml provette micro-centrifuga. Centrifugare a 12.000 g per 10 min a 4 ° C. Un pellet formerà sul fondo della provetta.
- Consolidare surnatante limpido dal punto precedente pipettando in una provetta Falcon 15 ml sul ghiaccio. Omogeneizzare capovolgendo 5-10x.
- Sulla base di concentrazione determinato dalla Bradford nel passaggio 1.47, determinare quantità di estratto di aliquota in singoli 1,75 ml provette. Ogni singolo tubo deve avere un volume di 810-900 mg di proteina totale. Estratto dovrebbe avere una concentrazione totale di proteine superiore a 27 mg / ml. Questo passaggio richiede assistenza per condurre speditamente Nota:. Estratto Aliquotare inferiore a 30 mg / ml in 30 microlitri aliquote, e la scala se la concentrazione è più elevata, ad esempio, estratto aliquota a 28 mg / ml da 30 microlitri, ed estratto aliquota al 32mg / ml del 28,1 microlitri.
- Estratto Aliquota passo successivo 1.50, avendo cura di evitare bolle. Estratto Flash-congelamento in azoto liquido Nota:. Aliquote con bolle possono essere rimosse per centrifugazione a 10.000 xg per 30 sec a 4 ° C.
- Rimuovere i tubi di azoto liquido con un colino e memorizzare immediatamente a -80 ° C. Sicurezza: Indossare occhiali protettivi, i tappi di estrarre tubi possono venire fuori a causa della differenza di temperatura tra l'azoto liquido e temperatura ambiente.
2. Amino Acid Solution Preparazione
Amino Acid soluzione deve essere preparata in massa. La ricetta che segue utilizza un kit completo di RTS Amino Acid Sampler, fornendo sufficienti per circa 11.000 singole reazioni. Se ridimensionamento, si consiglia di utilizzare non meno della metà di un kit. Ogni amminoacido nella stock è alimentato a 1,5 ml, 168 mm, ad eccezione di leucina a 140 mm. La composizione finale del Amino Acid Solution è:leucina, 5 mM, tutti gli altri amminoacidi, 6 mM. Questo è 4x concentrazione di lavoro.
- Rimuovere tutti i 20 aminoacidi tra -20 ° C e scongelare a temperatura ambiente. Una volta scongelate, vortex fino aminoacidi dissolvono, incubando a 37 ° C, se necessario. Dopo amminoacidi vengono sciolti, mettere tutti gli amminoacidi in ghiaccio tranne Asn, Phe, e Cys, che vengono mantenute a temperatura ambiente. Cys potrebbero non dissolversi completamente.
- Su ghiaccio, aggiungere 12 ml di acqua sterile per una sterile 50 ml tubo Falcon.
- Aggiungere 1,5 ml di ciascun amminoacido nel seguente ordine, avendo cura di agitare la provetta Falcon dopo ogni aggiunta e di mantenere la soluzione sul ghiaccio: Ala, Arg, Asn, Asp, Gln, Glu, Gly, His, Ile, Lys, Met, Phe, Pro, Ser, Thr, Val, Trp, Tyr, Leu, Cys. Cys possono essere aggiunti come una sospensione. Dopo l'aggiunta, vortice finché soluzione è relativamente chiaro, incubando a 37 ° C, se necessario. Cys potrebbero non dissolversi completamente.
- Aliquota Amino Acid Soluzione in 50 tubi in26 ml ciascuno su ghiaccio. Aliquota del resto a 500 ml per provetta su ghiaccio. I 26 microlitri aliquote saranno utilizzati per calibrare estratto, mentre verranno utilizzate le aliquote microlitri 500 per la preparazione di buffer. Mentre la suddivisione in aliquote, vortice lo stock principale frequentemente per evitare la distribuzione ineguale della sospensione.
- Aliquote freeze flash in azoto liquido e conservare a -80 ° C. Sicurezza: Indossare occhiali protettivi, i tappi di estrarre tubi possono venire fuori a causa della differenza di temperatura tra l'azoto liquido e temperatura ambiente.
- Optional: Condurre un saggio di attività della neo-made Amino Acid Solution contro precedentemente effettuate Solutions aminoacidi.
3. Soluzione Preparazione Energy
Energy Solution è utilizzato sia per la calibrazione estratto cellulare grezzo e per la creazione di buffer, e dovrebbe essere preparato in massa. La ricetta che segue fornisce sufficiente per circa 10.000 singole reazioni. Se il ridimensionamento, è recommeNDED utilizzare non meno di 1/24 dei valori qui indicati. Come l'Energy Solution è un costo monetario significativo, gli utenti prima volta può decidere di preparare a scala 1/24. La composizione finale di Energy Solution è: HEPES pH 8 700 mm, ATP 21 mm, 21 mm GTP, CTP 12,6 millimetri, UTP 12,6 millimetri, tRNA 2,8 mg / ml, 3,64 mM CoA, NAD 4,62 mM, cAMP 10,5 mm, acido folico 0,95 mm, spermidina 14 mm, 3-PGA 420 mm. Questo è 14x concentrazione di lavoro. Se lo si desidera, ogni elemento individuale nella tabella 4 può essere conservato a -80 ° C per un uso successivo.
- Rimuovere tutte le sostanze chimiche in Tabella 4 da -80 ° C, -20 ° C, o 4 ° C a temperatura ambiente per 30 min.
- Preparare soluzioni madre come descritto nella Tabella 4. Vedi supplementare Materiale 1 per le ricette. Mettere tutte le soluzioni sul ghiaccio dopo la preparazione.
- In un tubo Falcon da 15 ml, aggiungere nel seguente ordine, avendo cura di agitare la provetta Falcon dopo ogni aggiunta e per mantenere le soluzioni on ghiaccio: 3,6 ml 2 M HEPES, 144 microlitri di acqua, 1,39 ml nucleotide mix, 576 microlitri 50 mg / ml tRNA, 576 microlitri 65 CoA mM, 276 microlitri NAD 175 mm, 170 ml 650 cAMP mM, 288 ml 33,9 di acido folinico mM , 144 ml 1 M spermidina, e 3,09 ml di 1,4 M 3-PGA.
- Aliquota Energy Solution in 50 tubi in 7 ml ciascuno su ghiaccio. Aliquota del resto a 150 ml per provetta su ghiaccio. I 7 aliquote microlitri saranno utilizzati per calibrare estratto, mentre verranno utilizzate le aliquote di 150 microlitri per la preparazione di buffer. Mentre la suddivisione in aliquote, vortice il titolo principale di frequente.
- Aliquote freeze flash in azoto liquido e conservare a -80 ° C. Sicurezza: Indossare occhiali protettivi, i tappi di estrarre tubi possono venire fuori a causa della differenza di temperatura tra l'azoto liquido e temperatura ambiente.
- Optional: Condurre un saggio di attività di recente fatto Energy Solution contro precedentemente fatte Energy Solutions.
4. Buffer Preparazione
Buffer Preparazione richiede il completamento di Cell greggio Estrarre Preparazione, Amino Acid Solution preparazione, e la soluzione Preparazione energia. Ogni buffer è unico per una partita di estratto cellulare grezzo. Mg-glutammato, K-glutammato e DTT (in questo ordine) sono ottimizzati in questa sezione per produrre reazioni con i massimi livelli di espressione. Il protocollo che segue utilizza un modello di pre-scritta, TXTL_e (template) _calibration_JoVE.xlsx (Supplemental materiale 3), per calibrare estratto di cellule greggio pre-preparati e preparare buffer. Tuttavia, si può anche calibrare estratto cellulare grezzo e preparare tampone senza il modello ottimizzando Mg-glutammato, K-glutammato e DTT manualmente e la creazione di buffer tale che insieme con l'estratto, è il 75% di un volume totale di reazione. Se la calibrazione manualmente, condizioni di reazione finali possono essere trovate al punto 5.
- Completa il modulo "Dati Generali".
- Scongelare su ghiaccio 100 mM Mg-glutammato (4 ° C), 3 M K-glutammato (4 ° C), 6mM Amino Acid Solution (26 microlitri, -80 ° C), Energy Solution (7 ml, -80 ° C), DTT 100 mM (-20 ° C), DNA di controllo positivo (-20 ° C), 40% PEG- . 8000 (4 ° C), estratto di cellule greggio (-80 ° C) e acqua (4 ° C) Nota: Utilizzare 1 nM concentrazione di lavoro pBEST-OR2-OR1-Pr-UTR1-deGFP-T500 (Addgene plasmide 40019) per il controllo positivo (eccitazione 485 nm, emissione 525 nm), o un altro riferimento che produce intensità di segnale. 4
- Preparare sette 10.5 reazioni microlitri, testando una serie di 4-10 mm Altre Mg-glutammato, aliquotandolo set quantità di magazzino Mg-glutammato in tubi micro-centrifuga individuali. Nota: Sebbene 10.5 reazioni microlitri sono inizialmente preparate, la reazione finale è di 10 microlitri.
- Preparare master mix come indicato nel modello sotto "calibrazione Mg-glutammato", aggiungendo un extra di 80 mm di K-glutammato. Tenere su ghiaccio e vortex dopo l'aggiunta di ciascun elemento Nota:. I valori qui e nel modello sono riportatioltre alle quantità di Mg-glutammato, K-glutammato, e TDT presenti nel buffer S30B usato per fare estratto cellulare grezzo.
- Aggiungi mix master campioni contenenti Mg-glutammato e preparare le reazioni. Vedere i passaggi 5,10-5,13 per le istruzioni dettagliate.
- Eseguire reazione a 29 ° C, sia in un incubatore o un lettore di piastre.
- . Determinare la concentrazione ottimale Mg-glutammato per livello di end-espressione e la velocità massima di espressione della proteina (Figura 4a) Nota: Tempi di esecuzione variano a seconda dell'esperimento, ma in genere l'ultima in 8 ore.
- Ripetere i passaggi 4,2-4,7 per K-glutammato nella sezione "calibrazione K-glutammato", che definisce i tenori Mg-glutammato a quelli trovati nella fase 4.7.
- Ripetere i passaggi 4,2-4,7 per il digitale terrestre in "calibrazione DTT", che definisce i tenori Mg-glutammato a quelli trovati nel passo 4.7 e livelli di K-glutammato a quelli trovati nel passaggio 4.8. Nota: Abbiamo scoperto che ha aggiunto DTT non influisce significativamente end- livelli di espressione.
- Usavalori trovati in calibrazione sotto "composizione Buffer" per determinare la composizione di tampone per essere preparati. In base alla quantità di estratto cellulare greggio prodotto, una ricetta master mix è prodotto per una determinata quantità di buffer.
- Scongelare aliquote elencate in master ricetta mix su ghiaccio. Una volta scongelato, preparare master mix, mantenendo su ghiaccio e vortex dopo l'aggiunta di ogni elemento.
- Aliquota per importo indicato alla voce "la composizione Buffer". Tubi tampone Flash-freeze in azoto liquido. Mentre la suddivisione in aliquote, vortice lo stock principale di frequente.
- Rimuovere i tubi di azoto liquido con un colino e memorizzare immediatamente a -80 ° C. Sicurezza: Indossare occhiali protettivi, i tappi di estrarre tubi possono venire fuori a causa della differenza di temperatura tra l'azoto liquido e temperatura ambiente.
5. Esecuzione sperimentale di una reazione TX-TL
Condizioni di reazione finali sono: 8,9-9,9 mg / ml prin (da estratto grezzo), 4,5 MM-10.5 mM Mg-glutammato, 40-160 mm K-glutammato, 0,33-3,33 mM DTT, 1.5 mM ogni amminoacido tranne leucina, leucina mM 1,25, 50 HEPES mM, ATP 1,5 mM e GTP, CTP 0.9 mM e UTP, 0,2 mg / ml tRNA, 0,26 CoA mM, 0,33 mM NAD, 0.75 cAMP mM, 0,068 mM acido folico, 1 spermidina mM, 30 mM 3-PGA, 2% PEG-8000. A TX base -TL reazione ha 3 parti (tubi): estratto di cellule greggio, tampone, e DNA. Il rapporto è: 75% tampone ed estratto, il 25% del DNA Le reazioni possono variare di volume, e usiamo 10 microlitri per convenzione per minimizzare il volume di reazione e consentire l'esecuzione in una piastra a 384 pozzetti.. Volumi più grandi richiedono agitazione per una corretta ossigenazione. Il protocollo che segue utilizza un modello di pre-scritta, TXTL_JoVE.xlsx (Supplemental Material 4), per condurre una reazione di 10 microlitri. Articoli in viola indicano i valori di input utente, e gli elementi in blu indicano i reagenti supplementari da aggiungere alla reazione. Tuttavia, si può anche effettuare uno spirito di reazioneHout il modello seguendo condizioni di reazione descritte sopra.
- Completa il modulo "Dati Generali".
- Sotto "Mix Preparazione Maestro", inserire il valore percentuale estratto dal punto 4.1 nella scatola viola.
- Progetta il tuo esperimento in silico utilizzando il "Mix preparazione Master" (righe 10-17) e "Preparazione del DNA" (righe 19-50) sezioni. In generale, le costanti possono essere messi nella sezione "Mix Preparazione Master", mentre le variabili possono essere messi nella sezione "Preparazione del DNA". Riduci campioni per esperimento per evitare l'evaporazione del campione e sperimentale, tempo di polarizzazione di inizio. Vedere la Figura 6 per una configurazione di esempio.
- Sotto "Mix Preparazione Maestro", aggiungere i reagenti, quali induttori o proteine, che andranno in tutti i campioni ad una concentrazione costante. Partendo con la riga 14, compilare le aree ombreggiate blu, mantenendo un reagente per ogni riga. Le unità sono rapporti relativi.
- In "Preparazione del DNA", DNA aggiungere che sarà specif campioneic. ID campione # 1 e # 2, corrispondenti a controlli positivi e negativi, rispettivamente. ID campione # 3 e soprattutto sono modificabili dall'utente per il DNA, la concentrazione magazzino in ng / ml, la lunghezza in paia di basi, la concentrazione finale desiderata in Nm, e ripetizioni (di 10 reazioni microlitri). La quantità di azioni DNA per raggiungere la concentrazione finale desiderata viene calcolato automaticamente. . Il totale di tutti i somme delle righe di 10.5 * n, dove n è il numero di ripetizioni Nota: Sebbene il volume finale di reazione è 10 microlitri, i calcoli assumono un volume totale di 10,5 microlitri per reazione, per tenere conto di volume perso durante il pipettaggio.
- In "Preparazione del DNA," aggiungere reagenti o DNA complementare che sarà specifico campione colonne blu. Concentrazioni di DNA Archivio in Nm possono essere calcolati in "Preparazione del DNA", mentre reagenti specifici esempio richiedono il calcolo manuale basato su un volume totale di reazione di 10.5 * n. I volumi immessi vengono sottratti dal volume d'acqua della stessa riga.
- Rimuovere numbe necessariar di tubi di tampone, estratto di cellule greggio, e il controllo positivo sotto "Tubes per scongelare," di -20 ° C o -80 ° C e scongelare il ghiaccio.
- Preparare campioni di DNA. Per ogni ID esempio, un'aliquota il DNA indicata, l'acqua e gli elementi forniti dall'utente per la sezione "Preparazione del DNA" in un tubo micro-centrifuga, a temperatura ambiente. Nota: per evitare la perdita di campione, di recente tarati e pipette basso-stick Si raccomanda puntali per pipette e provette micro-centrifuga.
- Quando i tubi dal punto 5.7 vengono scongelati, preparare il master mix costituito da tampone, estratto, e tutti gli elementi forniti dall'utente globali basate sulle caselle ombreggiate arancio, mantenendo su ghiaccio e vortex dopo l'aggiunta di ogni elemento. Nota: Extract è estremamente viscoso. Aliquote con bolle possono essere rimosse per centrifugazione a 10.000 xg per 30 sec a 4 ° C.
- Aggiungere la quantità di master mix indicato nelle cellule arancione in "Preparazione del DNA" (colonna O) per ogni campione di DNA, e mantenere a temperaturature. Trattare questo come l'ora di inizio di reazione.
- Vortex ogni campione, e centrifugare a 10.000 xg per 30 secondi a temperatura ambiente per abbattere qualsiasi campione residuo e per ridurre bolle.
- Se conducendo la reazione in tubi micro-centrifuga, incubare direttamente a 29 ° C. . Altrimenti, pipetta 10 ml di campione in una piastra da 384 pozzetti Nota: Le reazioni in volumi superiori a 10 ml possono richiedere l'agitazione per l'ossigenazione.
- Piastra centrifugare a 4000 xg per 30 secondi a temperatura ambiente per abbattere qualsiasi campione residuo e per ridurre bolle. Sigillare piastra poi per evitare l'evaporazione.
- Eseguire reazione a 29 ° C. Nota: Tempi di esecuzione variano a seconda dell'esperimento, ma in genere durano meno di 8 ore.
Risultati
Abbiamo presentato un protocollo di cinque giorni per la preparazione di un Escherichia coli endogeno basato sistema di espressione acellulare TX-TL. Una cronologia esempio per la creazione dei reagenti - estratto di cellule greggio e tampone - può essere trovato in Figura 1. Una volta creati, questi possono essere conservati a -80 ° C per un massimo di un anno. Dopo i reagenti sono creati, setup sperimentale e l'esecuzione può essere fatto in meno di 8 ore.
Inoltre, abbiamo ottimizzato le condizioni di espressione del sistema di espressione acellulare TX-TL. Altre aggiunte forniti dall'utente, come tamponi o soluzioni di DNA, devono essere calibrati per la tossicità anticipo. Ad esempio, diversi metodi di elaborazione dei risultati plasmidi di espressione differente dovuto contenuto di sale. Abbiamo anche testato l'effetto di tampone di eluizione Tris-Cl sull'efficienza reazione (Figura 5).
Un esempio di calibrazione grezzo estratto cellulare, riferendosi passo 4,1-4,9, è mostratoin figura 4a. In generale, i nostri esperimenti mostrano che l'estratto cellulare grezzo è più sensibile ai livelli di Mg-glutammato, seguiti da livelli di K-glutammato. Per dimostrare il sistema di libera espressione di cellule, abbiamo costruito e testato un ciclo di feedback negativo in base a tet repressione. 26 (Figura 6). Nel sistema di espressione cell-free, la stessa corsa circuito con e senza ATC mostra un end-point cambiamento di espressione di 7 volte di deGFP giornalista dopo otto ore di espressione. Anche se questo esperimento non richiede induttori globali o repressori, se necessario, possono essere aggiunti in "Mix Preparazione Maestro".
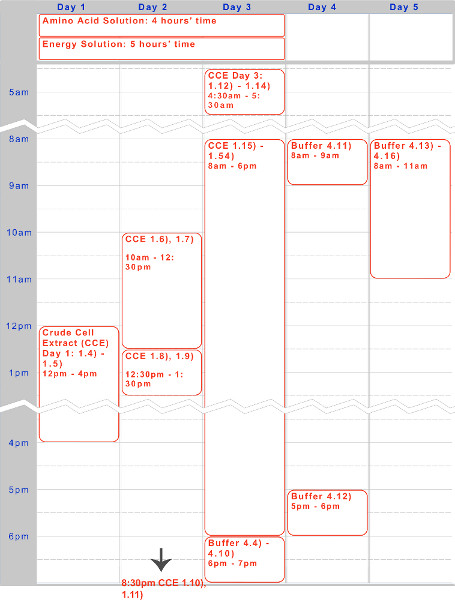
Figura 1. Timeline di estratto di cellule greggio, soluzione di aminoacidi, e la preparazione soluzione energetica. Timelin A cinque giorni e per l'esecuzione tipica del protocollo è dato sopra, ottimizzato per incubazioni overnight e fasi di lavoro durante il giorno.
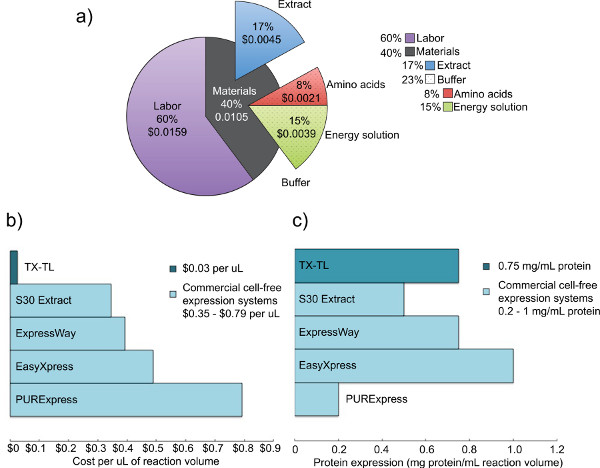
Figura 2. Costi e analisi di espressione di competere estratti cellulari greggio. a) Ripartizione dei costi di manodopera e dei materiali del sistema di libera espressione di cellule TX-TL. Sulla base dei costi dei reagenti come di Dicembre 2012, e il costo del lavoro di $ 14 all'ora. B) Confronto dei costi di sistema libera espressione di cellule TX-TL vs altri sistemi commerciali. I costi sono ripartiti per ml, anche se i volumi di reazione possono variare per kit. C) Confronto tra TX-TL cell-free sistema di espressione del rendimento rispetto ad altri sistemi commerciali. Resa espressione proteica determinata dalle norme produttore./ 50762fig2large.jpg "target =" _blank "> Clicca qui per ingrandire la figura.

Figura 3. Caricamento e lavorazione di un tubo tallone-pestaggio. a) Dimostrazione della corretta viscosità della soluzione di cellule-tallone. Soluzione Cell-tallone avrà una viscosità dipende da molti fattori, tra cui quantità di tampone S30A aggiunto, la quantità di perline aggiunti, e il tempo trascorso sul ghiaccio. B) Caricamento di tubo tallone battendo prima breve centrifugazione tavolo. La centrifugazione rimuove le bolle accumulate durante il caricamento. C) Bolle emersione dopo tavolo centrifugazione. La dimensione delle bolle varia, possono essere spuntato o rimosso utilizzando un puntale d) completamente riempito tubo tallone battere prima di tappatura.. Un menisco si forma nel tallone battendo tube, e il tappo ha abbastanza per coprire e causare piccole quantità per riempire troppo. e) apparecchio filtrante correttamente caricato. Questi possono essere riutilizzati. F) Confronto correttamente vs tubo tallone battere correttamente elaborato. Il tubo sulla sinistra è un tubo ben-beat - è dotato di un piccolo e ben delineato strato superiore, e molto chiaro surnatante. Il tubo a destra è ottimale, in base al formato grande, secondo strato nebuloso e il surnatante nebuloso. Tubi che sono subottimale non dovrebbero essere sottoposti a ulteriori elaborazioni.
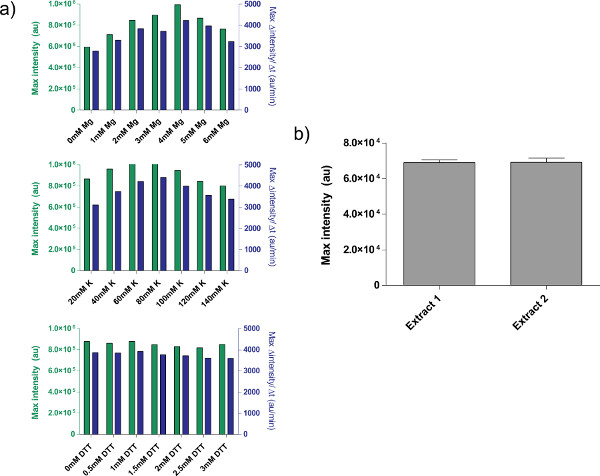
Figura 4. Proprietà di preparati estratto grezzo. a) curva di taratura tipici per estratto cellulare grezzo. Estratto grezzo è calibrato per ulteriori Mg-glutammato, K-glutammato, e livelli DTT, in questo ordine. Viene mostrato endpoint fluorescence dopo 8 ore, così come velocità massima di produzione di proteine sulla base di una media mobile a 12 minuti. Sulla base di queste trame, un intervallo accettabile di ulteriori Mg-glutammato è di 4 mm, K-glutammato è 60-80 mm, DTT è 0-3 mm. Si noti che ogni estratto grezzo deve essere calibrato in modo indipendente per queste tre variabili. B) Variazione dalle preparazioni estratto. È mostrato Endpoint fluorescenza di due estratti grezzi preparati in date diverse, le barre di errore sono 1 deviazione standard da tre piste indipendenti in giorni diversi. Clicca qui per ingrandire la figura .
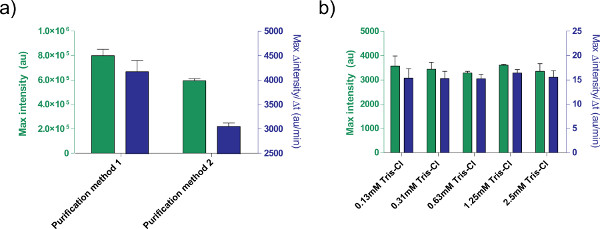
Figura 5. Effetti della soluzione di DNA sull'efficienza espressione. a) Confronto tra due diversi depurazionemodalità di trattamento dei plasmidi. 1 nM di pBEST-OR2-OR1-Pr-UTR1-deGFP-T500 viene preparata utilizzando solo un kit QIAprep Spin Miniprep (metodo di purificazione 1) o post-elaborata con un kit di purificazione QIAquick PCR (metodo di purificazione 2). Puo 'endpoint fluorescenza dopo 8 ore, così come velocità massima di produzione di proteine sulla base di una media mobile a 12 minuti. Le barre di errore sono 1 deviazione standard da quattro piste indipendenti in giorni diversi. B) Effetto del tampone di eluizione (Tris-Cl). Diverse concentrazioni di Tris-Cl sono confrontati in una reazione espressione cell-free base all'espressione di 1 nM di pBEST-OR2-OR1-Pr-UTR1-deGFP-T500. Le concentrazioni indicate sono concentrazioni finali di Tris-Cl nella reazione; tampone di eluizione utilizzato è 10 mM Tris-Cl. Le barre di errore sono 1 deviazione standard da tre piste indipendenti in giorni diversi. Clicca qui per ingrandire la figura .
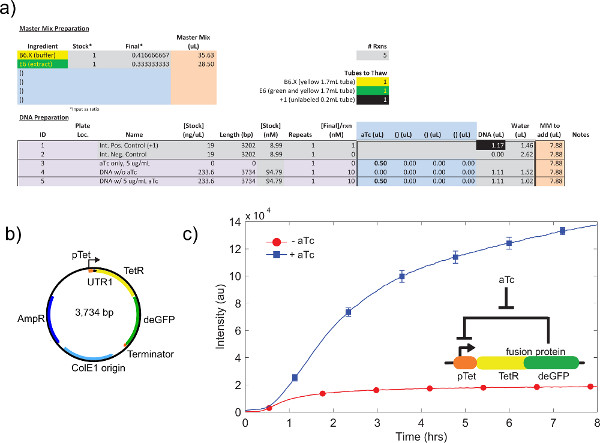
Figura 6. Esempio di run TX-TL di un ciclo di feedback negativo. a) la configurazione di esempio di una reazione esecuzione cell-free. Prove "a" "off" stato del ciclo di feedback negativo rispetto, con controlli positivi e negativi. B) plasmide mappa di feedback negativo. C) I risultati rappresentativi. Dati riflette esperimento in a) e b), con controllo negativo sottratto dal segnale. Circuito genetico mostrato in insert. Le barre di errore sono 1 deviazione standard da tre piste indipendenti in giorni diversi. Clicca qui per ingrandire la figura .
| Nome | Concentrazione | Importo | Sterilizzazione | Note |
| Cloramfenicolo (Cm) | 34 mg / ml in etanolo | 1 ml | Filtro sterilizzare (0,22 micron) | Può essere fatto in grandi volumi conservati a -20 ° C per un uso successivo. |
| 2xYT + P + piastra Cm agar | 31 g / L 2xYT, 40 mM potassio fosfato bibasico, 22 mM fosfato di potassio monobasico, 34 mg / ml di cloramfenicolo | 1 piastra | Autoclave | |
| 2xYT + P supporti | 31 g / L 2xYT, 40 mM potassio fosfato bibasico, 22 mM fosfato di potassio monobasico | 4 L | Autoclave |
Tabella 1. Reagenti per giorno 1 del protocollo estratto cellulare grezzo.
| Nome | Concentrazione | Importo | Sterilizzazione | Note |
| Tris base | 2 M | 250 ml | Sterilizzare Filter (0,22 micron) o in autoclave | Può essere conservato a temperatura ambiente. |
| DTT | 1 M | 6 ml | Filtro sterilizzare (0,22 micron) | Può essere fatto in grandi volumi e conservati a -20 ° C per un uso successivo. |
| Tampone S30A | 14 mM Mg-glutammato, 60 mM K-glutammato, 50 mM Tris, pH 7.7 | 2 L | Autoclave | Per raggiungere pH 7.7, titolare con acido acetico. Aggiungi DTT a 2 mm di concentramento finale appena prima dell'uso. Conservare a 4 ° C. |
| Tampone S30B | 14 mM Mg-glutammato, 60 mm K-glutammato, ~ Tris 5 mM, pH 8,2 | 2 L | Autoclave | Per raggiungere pH 8,2, titolare con 2MTris. Aggiungi DTT a 1 mM concentrazione finale appena prima dell'uso. Conservare a 4 ° C. |
Tabella 2. Reagenti per il giorno 2 del protocollo estratto cellulare grezzo.
| Falco | ||||
| 1 | 2 | 3 | 4 | |
| Vuoto 50 ml Falcon (g) | ||||
| 50 ml Falcon con pellet (g) | ||||
| Massa Pellet (50 ml Falcon con pellet - vuoto 50 ml Falcon) (g) | ||||
| S30A volume di tampone di aggiungere (massa pellet * 0,9) (ml) | ||||
| Massa totale di perline per aggiungere (massa pellet * 5.0) (g) | ||||
Tabella 3. Tampone S30A e calcolatrice massa tallone, per il giorno 3 del protocollo estratto cellulare grezzo.
| Nome | Concentrazione | Importo | Sterilizzazione | Note |
| HEPES | 2 M, pH 8 | 4 ml | Nessuno | Per raggiungere pH 8, titolare con KOH. |
| Nucleotide Mix | , 94 mM CTP 156 mM ATP e GTP e UTP, pH 7.5 | 1,5 ml | Nessuno | Per raggiungere pH 7.5, titolare con KOH. |
| tRNA | 50 mg / ml | 600 microlitri | Nessuno | |
| CoA | 65 mm | 600 microlitri | Nessuno | |
| NAD | 175 mM, pH 7,5-8 | 300 ml | Nessuno | Per raggiungere pH 7.5-8, titolare con Tris a 2 M. |
| cAMP | 650 mM, pH 8 | 200 ml | Nessuno | Per raggiungere pH 8, titolare con Tris a 2 M. |
| Acido folinico | 33.9 mM | 300 ml | Nessuno | Anche se sono necessari solo 300 ml, ricetta supplementare è di 1.15 ml. |
| Spermidina | 1 M | 150 microlitri | Nessuno | Conservare a 4 ° C, calore a 37 ° C per fondere. |
| 3-PGA | 1.4 M, pH 7.5 | 3.2 ml | Nessuno | Per raggiungere pH 7.5, titolare con Tris a 2 M. |
Tabella 4. Reagenti per prepararsi per il protocollo Energy Solution.
Supplemental Materiale 1. Ricette per Articoli.
Cloramfenicolo, 34 mg / ml: Preparare 0,51 g cloramfenicolo e aggiungere etanolo a 15 ml. Sterilizzare Filter (0,22 micron), aliquota da 1 ml tubi, conservare a -20 ° C per un uso successivo.
2xYT + P + piastra Cm agar: Preparare 1,24 g 2xYT, 1,6 ml di soluzione di potassio fosfato bibasico @ 1 M, 0,88 ml di potassio fosfato monobasico @ 1 M, 0,6 g di agar, e l'acqua a 40 ml. Autoclave. Lasciate raffreddare a 50 ° C e aggiungere 40 microlitri Cm. Aliquota 25 ml in una piastra di Petri di 100 x 15 mm, e lasciate raffreddare per un'ora.
2xYT + P supporti: Preparare 124 g 2xYT, 160 ml di fosfato di potassio dibasico @ 1 M, soluzione di 88 ml di potassio fosfato monobasico @ 1 M, e acqua per 4 L. Aliquot fuori in 2 x 1,88 L e 0,24 L. Autoclave.
Tris base, 2 M: Preparare 60,57 g di base Tris e acqua a 250 ml. Sterilizzare, conservare a temperatura ambiente per un uso successivo.
DTT, 1 M: Preparare 2.31 g DTT e acqua a 15 ml. Sterilizzare Filter (0,22 micron), aliquota da 1 ml tubi, conservare a -20 ° C per un uso successivo.
Tampone S30A: Preparare 10.88 g Mg-glutammato e 24,39 g K-glutammato, 50 ml Tris a 2M, acido acetico (a pH 7,7), e acqua per 2 L. Autoclave, conservare a 4 ° C, aggiungere 4 ml di 1 M DTT prima dell'uso.
Tampone S30B: Preparare 10.88 g Mg-glutammato e 24,39 g K-glutammato, Tris a 2 M (pH 8.2), e acqua per 2 L. Autoclave, conservare a 4 ° C, aggiungere 2 ml di 1 M DTT prima dell'uso.
HEPES: Preparare 1.91 g HEPES (MW 238,21), KOH (a pH 8), e acqua a 4 ml.
tRNA: Preparare 30 mg di tRNA e acqua a 600 ml.
CoA: Preparare 30 mg di CoA (MW 767,53) e l'acqua a 600 ml.
NAD: Aggiungere 34.83 mg di NAD (MW 663,43), Tris a 2 M (a pH 7,5-8), e acqua a 300 ml. (Aggiungere 27 ml di Tris a 2 M per portare la soluzione a pH 7,5-8).
cAMP: Aggiungere 42.80 mg di cAMP (MW 329,22), Tris a 2 M (a pH 8), e acqua di 200 microlitri. (Aggiungere 73 ml di Tris a 2 M per portare la soluzione a pH 8).
Acido folinico (33,9 mm): A 20 mg di solido sale di calcio di acido folico (511,5 MW), aggiungere 1,15 ml di acqua.
Spermidina: Preparare 23.55 ml di spermidina (MW 145,25) e acqua a 150 ml. Preparare a temperatura ambiente dopo la fusione brevemente a 37 ° C.
3-PGA: Aggiungere 1,03 g di 3-PGA (MW 230,02), Tris a 2 M (pH 7,5), e acqua a 3,2 ml. (Aggiungere 1,73 ml di Tris a 2 M per portare la soluzione a pH 7,5).
Nucleotide Mix: Aggiungere 145 mg di ATP dipotassio diidrato sale (619,4 MW), 133 mg di GTP sale disodico (MW 567,14), 79,4 mg di CTP diidrato sale disodico (MW 563,16), 82,6 mg di diidrato UTP sale trisodico (MW 586,12) , KOH al 15% di diluizione (a pH 7,5), e acqua a 1,5 ml. (Aggiungere 353 ml di KOH al 15% diluizione per portare la soluzione a pH 7,5).
Supplemental materiale 2. Bradford Assay.
- Rimuovere agente Bradford da 4 ° C e impostare a temperatura ambiente.
- Preparare 50 ml BSA standard a 1 mg / ml ed a 0.1 mg / ml.
- Preparare 40 ml 20x diluizione dell'estratto dal punto 1.47.
- Aggiungere 800 microlitri di acqua a 7 cuvette.
- Preparare cuvette standard per la 0 mg / ml, 1 mg / ml (10 ml 0,1 mg / ml BSA), 2 mg / ml (20 ml 0,1 mg / ml BSA), 4 mg / ml (4 microlitri 1 mg / ml BSA) , 6 mg / ml (6 ml 1 mg / ml BSA).
- Preparare cuvette sperimentali per 2 ml di campione e 4 ml di campione.
- Aggiungere 200 ml di agente Bradford ad ogni provetta e mescolare bene by pipettaggio. Incubare a temperatura ambiente per almeno 10 min.
- Produrre curva standard a OD 595 nm utilizzando cuvette dal punto 6.5. Rifiuta curva standard se r 2 <0,95.
- Determinare la concentrazione di estratto di OD a 595 nm utilizzando cuvette dal punto 6.6.
Materiale supplementare 3. Buffer foglio di calibrazione.
Vedere TXTL_e (template) _calibration_JoVE.xlsx .
Materiale supplementare 4. Libera espressione-Cell eseguito foglio di calcolo.
Discussione
Sistema di espressione cell-free TX-TL basata Il Escherichia coli endogena qui descritto è una reazione di facile conduzione tre tubo che può richiedere meno di otto ore da impostare alla raccolta dei dati. Il processo di creazione di tutti i reagenti richiede tempo totale di cinque giorni (con notevoli esigenze di lavoro in un solo giorno), ma produce estratto grezzo di 3.000 reazioni e buffer-making reagenti per 10.000 reazioni (Figura 1). Inoltre, estratto grezzo e buffer facendo reagenti sono stabili per almeno 1 anno a -80 ° C, consentendo molteplici usi di una preparazione di 4. A $ 0,11 per 10 reazione microlitri (0,26 dollari compreso il lavoro), i costi sono inferiori del 98% rispetto comparabile sistemi commerciali (Figura 2).
Ci sono alcune limitazioni irrisolti, tuttavia, al sistema. L'efficienza fine di ciascun preparato estratto cellulare grezzo può variare sulla base di competenza degli utenti e delle condizioni ambientali, sebbene tvariazione di rendimento ypical è compresa tra il 5-10% (Fig. 4b). Come risultato, lotto a lotto variabilità sia espressione end-point e nelle dinamiche di espressione sono da aspettarsi. Queste variazioni saranno probabilmente rimarrà fino estratto è completamente caratterizzata o fino a quando la creazione estratto è completamente automatizzato. Se il sistema di libera espressione-cella viene utilizzata per condurre esperimenti quantitativi sensibili, si consiglia di eseguire tutti gli esperimenti con la stessa produzione di estratto cellulare grezzo. La resa da un unico lotto estratto cellulare greggio, circa 3000 reazioni, dovrebbe essere sufficiente per i corsi sperimentali tipici. Anche se abbiamo il sospetto variazione può essere rimosso da scalare e automatizzare la procedura, tali tentativi comporterebbe un investimento di risorse notevole.
Inoltre, anche se i livelli di espressione end-point sono ragionevolmente facile da determinare, più lavoro deve essere fatto in comprensione dinamica intrinseca al sistema cell-free. È noto che sia CONCORRENZ risorsan risorse e limitazione può influenzare le dinamiche di espressione. Ad esempio, limitato sigma endogeno 70 può risultare in un regime saturare con maggiore DNA stampo produrre un profilo di espressione analoga a quella del nucleotide o amminoacido esaurimento. 9,27 Tuttavia, dinamiche non devono essere pienamente compreso per utilizzare il sistema. Per gli aumenti puri di rendimento, ottimizzazione può essere fatto da approcci di machine-learning. 28 Questioni di competizione per le risorse e di limitazione possono essere affrontate da modelli matematici verificata utilizzando dati sperimentali.
Il protocollo qui presentato è ottimizzato per un ceppo BL21-Rosetta2, ma è generalizzabile ad altri E. coli. Modifiche in BL21-Rosetta2, quali la rimozione del gene codificante lon proteasi e l'aggiunta di geni codificanti tRNA rari, permettono la produzione di proteine massima. Abbiamo tentato il protocollo con altri due ceppi di estratto - BL21 solo e BL21 trxA ko-and ha trovato il 50% in meno di rendimento di proteine. Noi ipotizziamo che i rendimenti allo stesso modo diminuiscono quando si utilizzano altri ceppi. Altre variazioni di parametri, come il passaggio terreno di coltura 2xYT per LB ed altri brodi ricchi, hanno portato a una diminuzione dei rendimenti delle proteine.
Sistemi di espressione cell-free che utilizzano i macchinari di trascrizione-traduzione endogena ed esogena e meccanismi di regolazione hanno ampie applicazioni in proteine e metaboliti di espressione e di biologia sintetica. 3,29 Invece di essere limitato a circuiti T7-regolato, si può immaginare la produzione di biomolecole complesse in un ambiente user-controllabile tramite un mix di nativo E. promotori coli e meccanismi di trascrizione e regolazione esogena in dotazione. Senza limitazioni di divisione cellulare e il metabolismo, variabilità nei circuiti sintetici come il repressilator o in multistrato vie metaboliche come quelle che producono artemisinina può essere ridotta o meglio compresa. 30,31 Noi have utilizzato questi vantaggi per implementare interruttori genetici, nonché per capire sigma fattore sequestro 9,32 Tale tecnologia può anche costituire la spina dorsale del "minimal" o le celle "artificiali" -. piccola, ben caratterizzato e autosufficienti unità incarnata estratto. 33,34
In definitiva, ci aspettiamo usi immediati di questo sistema di libera espressione di cellule endogene come un ambiente di prototipazione per la biologia sintetica. Soprannominato il "TX-TL tagliere biomolecolare", il sistema di espressione cell-free offre un ambiente controllabile in cui in ultima analisi, circuiti sintetici destinati in vivo l'espressione può subire turni di prototipazione - cicli di test sul plasmide di base, lineare, o DNA sintetizzati chimicamente, seguita mediante analisi e la modifica rapida. Turni prototipazione possono essere aiutati da modelli matematici predittivi attualmente in fase di sviluppo. Rimuovendo la clonazione e manipolazione vivo per i circuiti non definitive, prevediamo ingegneritempi di ciclo gneria per essere ridotto a 1-3 giorni invece dello standard attuali settimane.
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari in competizione.
Riconoscimenti
Ringraziamo Jongmin Kim, Dan Siegal-Gaskins, Anu Thubagere, e Enoch Yeung per l'assistenza razionalizzare il protocollo, e Chiara Chen e Barclay Lee per l'assistenza nelle prime fasi del progetto. Questo materiale si basa su lavoro sostenuto in parte dalla Defense Advanced Research Projects Agency (DARPA / OMT) del programma Living Fonderie, numero di contratto HR0011-12-C-0065 (DARPA / CMO.ZZS è supportato anche da un UCLA / Caltech Medical Scientist Fellowship Program Formazione e da un DoD, Air Force Office of Scientific Research, Science National Defense and Engineering Graduate (NDSEG) Fellowship, 32 CFR 168. Le opinioni e le conclusioni contenute in questo documento sono quelle degli autori e non deve essere interpretato come rappresentazione ufficialmente politiche, esplicitamente o implicita, della Defense Advanced Research Projects Agency o il governo degli Stati Uniti.
Materiali
| Name | Company | Catalog Number | Comments |
| 2xYT | MP biomedicals | 3012-032 | |
| 3-PGA | Sigma-Aldrich | P8877 | |
| ATP | Sigma-Aldrich | A8937 | |
| Bacto-agar | BD Diagnostics | 214010 | |
| Bead-beating tubes (polypropylene microvials) | BioSpec | 522S | |
| Beads, 0.1mm dia. | BioSpec | 11079101 | |
| BL21 Rosetta 2 E. coli strain | Novagen | 71402 | |
| Bradford BSA Protein Assay Kit | Bio-rad | 500-0201 | |
| cAMP | Sigma-Aldrich | A9501 | |
| Chloramphenicol | Sigma-Aldrich | C1919 | |
| CoA | Sigma-Aldrich | C4282 | |
| CTP | USB | 14121 | |
| Cuvettes, 1.5ml | Fisher | 14-955-127 | |
| DTT | Sigma-Aldrich | D0632 | |
| Folinic acid | Sigma-Aldrich | F7878 | |
| GTP | USB | 16800 | |
| HEPES | Sigma-Aldrich | H6147 | |
| K-glutamate | Sigma-Aldrich | G1149 | |
| Mg-glutamate | Sigma-Aldrich | 49605 | |
| Micro Bio-Spin Chromatography Columns | Bio-Rad | 732-6204 | |
| NAD | Sigma-Aldrich | N6522 | |
| Nunc 384-well optical bottom plates | Thermo-Scientific | 142761 | |
| Nunc sealing tape | Thermo-Scientific | 232701 | |
| PEG-8000 | Promega | V3011 | |
| Potassium phosphate dibasic solution | Sigma-Aldrich | P8584 | |
| Potassium phosphate monobasic solution | Sigma-Aldrich | P8709 | |
| RTS Amino Acid Sampler | 5 Prime | 2401530 | |
| Slide-A-Lyzer Dialysis Cassettes, 10k MWCO (Kit) | Thermo-Scientific | 66382 | |
| Spermidine | Sigma-Aldrich | 85558 | |
| Tris base | Fischer | BP1521 | |
| tRNA (from E. coli) | Roche Applied Science | MRE600 | |
| UTP | USB | 23160 | |
| 1L Centrifuge Bottle | Beckman-Coulter | A98813 | This is specific for Avanti J-series; obtain equivalent size for centrifuge in use. |
| 4L Erlenmeyer Flask | Kimble Chase | 26500-4000 | |
| Avanti J-26XP Centrifuge | Beckman-Coulter | 393127 | Or 1L-capable centrifuge equivalent. |
| Forma 480 Orbital Shaker | Thermo Scientific | 480 | Or chest-size 6x4L shaker equivalent. |
| JLA-8.1000 Rotor | Beckman-Coulter | 363688 | Or 1L-capable, 5000 x g rotor equivalent for centrifuge. |
| Mini-Beadbeater-1 | BioSpec | 3110BX | |
| Supplemental Material 1. Recipes for Items. Chloramphenicol, 34 mg/ml: Prepare 0.51 g chloramphenicol and add ethanol to 15 ml. Filter sterilize (0.22 μM), aliquot to 1 ml tubes, store at -20 °C for later use. 2xYT+P+Cm agar plate: Prepare 1.24 g 2xYT, 1.6 ml potassium phosphate dibasic solution @ 1 M, 0.88 ml potassium phosphate monobasic solution @ 1 M, 0.6 g agar, and water to 40 ml. Autoclave. Let cool to 50 °C and add 40 μl Cm. Aliquot 25 ml into a 100x15 mm petri dish, and let cool for an hour. 2xYT+P media: Prepare 124 g 2xYT, 160 ml potassium phosphate dibasic solution @1 M, 88 ml potassium phosphate monobasic solution @ 1 M, and water to 4 L. Aliquot out into 2x1.88 L and 0.24 L. Autoclave. Tris base, 2 M: Prepare 60.57 g Tris base and water to 250 ml. Sterilize, store at RT for later use. DTT, 1 M: Prepare 2.31 g DTT and water to 15 ml. Filter sterilize (0.22 μM), aliquot to 1 ml tubes, store at -20 °C for later use. S30A buffer: Prepare 10.88 g Mg-glutamate and 24.39 g K-glutamate, 50 ml Tris at 2M, acetic acid (to pH 7.7), and water to 2 L. Autoclave, store at 4 °C, add 4 ml 1 M DTT before use. S30B buffer: Prepare 10.88 g Mg-glutamate and 24.39 g K-glutamate, Tris at 2 M (to pH 8.2), and water to 2 L. Autoclave, store at 4 °C, add 2 ml 1 M DTT before use. HEPES: Prepare 1.91 g HEPES (MW 238.21), KOH (to pH 8), and water to 4 ml. tRNA: Prepare 30 mg of tRNA and water to 600 μl. CoA: Prepare 30 mg of CoA (MW 767.53) and water to 600 μl. NAD: Add 34.83 mg of NAD (MW 663.43), Tris at 2 M (to pH 7.5-8), and water to 300 μl. (Add 27 μl of Tris at 2 M to bring the solution to pH 7.5-8). cAMP: Add 42.80 mg of cAMP (MW 329.22), Tris at 2 M (to pH 8), and water to 200 μl. (Add 73 μl of Tris at 2 M to bring the solution to pH 8). Folinic Acid (33.9 mM): To 20 mg of solid folinic acid calcium salt (MW 511.5), add 1.15 ml water. Spermidine: Prepare 23.55 μl of spermidine (MW 145.25) and water to 150 μl. Prepare at room temperature after melting briefly at 37 °C. 3-PGA: Add 1.03 g of 3-PGA (MW 230.02), Tris at 2 M (to pH 7.5), and water to 3.2 ml. (Add 1.73 ml of Tris at 2 M to bring the solution to pH 7.5). Nucleotide Mix: Add 145 mg of ATP dipotassium salt dihydrate (MW 619.4), 133 mg of GTP disodium salt (MW 567.14), 79.4 mg of CTP disodium salt dihydrate (MW 563.16), 82.6 mg of UTP trisodium salt dihydrate (MW 586.12), KOH at 15% dilution (to pH 7.5), and water to 1.5 ml. (Add 353 μl of KOH at 15% dilution to bring the solution to pH 7.5). Supplemental Material 2. Bradford Assay.
See TXTL_e(template)_calibration_JoVE.xlsx. Supplemental Material 4. Cell-free expression run spreadsheet. See TXTL _JoVE.xlsx. | |||
Riferimenti
- Noireaux, V., Bar-Ziv, R., Libchaber, A. Principles of cell-free genetic circuit assembly. Proceedings of the National Academy of Sciences of the United States of America. 100, 12672-12677 (2003).
- He, M. Y., He, Y. Z., Luo, Q., Wang, M. R. From DNA to protein: No living cells required. Process Biochem. 46, 615-620 (2011).
- Forster, A. C., Church, G. M. Synthetic biology projects in vitro. Genome Res. 17, 1-6 (1101).
- Shin, J., Noireaux, V. Efficient cell-free expression with the endogenous E. Coli RNA polymerase and sigma factor 70. Journal of biological engineering. 4, 8(2010).
- Shimizu, Y., et al. Cell-free translation reconstituted with purified components. Nat Biotechnol. 19, 751-755 (2001).
- Shin, J., Noireaux, V. An E. coli Cell-Free Expression Toolbox: Application to Synthetic Gene Circuits and Artificial Cells. Acs Synth Biol. 1, 29-41 (2012).
- Shin, J., Noireaux, V. Study of messenger RNA inactivation and protein degradation in an Escherichia coli cell-free expression system. Journal of biological engineering. 4, 9(2010).
- Shin, J., Jardine, P., Noireaux, V. Genome Replication, Synthesis, and Assembly of the Bacteriophage T7 in a Single Cell-Free Reaction. Acs Synth Biol. 1, 408-413 (2012).
- Siegal-Gaskins, D., Noireaux, V., Murray, R. M. Biomolecular resource utilization in elementary cell-free gene circuits. Pao, L., Abramovitch, D. American Control Conference, Jun 17-19, Washington, DC, , AACC. 1531-1536 (2013).
- Karzbrun, E., Shin, J., Bar-Ziv, R. H., Noireaux, V. Coarse-grained dynamics of protein synthesis in a cell-free system. Physical review letters. 106, 048104(2011).
- Hoagland, M. B., Stephenson, M. L., Scott, J. F., Hecht, L. I., Zamecnik, P. C. Soluble Ribonucleic Acid Intermediate in Protein Synthesis. J Biol Chem. 231, 241-257 (1958).
- Wood, W. B., Berg, P. Effect of Enzymatically Synthesized Ribonucleic Acid on Amino Acid Incorporation by a Soluble Protein-Ribosome System from Escherichia Coli. Proceedings of the National Academy of Sciences of the United States of America. 48, 94(1962).
- Zubay, G. In-Vitro Synthesis of Protein in Microbial Systems. Annu Rev Genet. 7, 267-287 (1973).
- Pratt, J. M. Transcription and Translation: A Practical Approach. Hames, B. D., Higgins, S. J. , IRL Press. 179-209 (1984).
- Kim, H. C., Kim, D. M. Methods for energizing cell-free protein synthesis. Journal of bioscience and bioengineering. 108, 1-4 (2009).
- Michel-Reydellet, N., Calhoun, K., Swartz, J. Amino acid stabilization for cell-free protein synthesis by modification of the Escherichia coli genome. Metabolic engineering. 6, 197-203 (2004).
- Liu, D. V., Zawada, J. F., Swartz, J. R. Streamlining Escherichia coli S30 extract preparation for economical cell-free protein synthesis. Biotechnology progress. 21, 460-465 (2005).
- Andrianantoandro, E., Basu, S., Karig, D. K., Weiss, R. Synthetic biology: new engineering rules for an emerging discipline. Molecular systems biology. 2, 2006.0028(2006).
- Kwok, R. Five hard truths for synthetic biology. Nature. 463, 288-290 (2010).
- Tabor, S., Richardson, C. C. A bacteriophage T7 RNA polymerase/promoter system for controlled exclusive expression of specific genes. Proceedings of the National Academy of Sciences of the United States of America. 82, 1074-1078 (1985).
- Lewicki, B. T., Margus, T., Remme, J., Nierhaus, K. H. Coupling of rRNA transcription and ribosomal assembly in vivo. Formation of active ribosomal subunits in Escherichia coli requires transcription of rRNA genes by host RNA polymerase which cannot be replaced by bacteriophage T7 RNA polymerase. Journal of molecular biology. 231, 581-593 (1993).
- Iskakova, M. B., Szaflarski, W., Dreyfus, M., Remme, J., Nierhaus, K. H. Troubleshooting coupled in vitro transcription-translation system derived from Escherichia coli cells: synthesis of high-yield fully active proteins. Nucleic acids research. 34, e135(2006).
- Kigawa, T., et al. Preparation of Escherichia coli cell extract for highly productive cell-free protein expression. Journal of structural and. 5, 63-68 (2004).
- Matsuda, T., et al. Improving cell-free protein synthesis for stable-isotope labeling. Journal of biomolecular. NMR. 37, 225-229 (2007).
- Sitaraman, K., et al. A novel cell-free protein synthesis system. Journal of biotechnology. 110, 257-263 (2004).
- Becskei, A., Serrano, L. Engineering stability in gene networks by autoregulation. Nature. 405, 590-593 (2000).
- Maeda, H., Fujita, N., Ishihama, A. Competition among seven Escherichia coli sigma subunits: relative binding affinities to the core RNA polymerase. Nucleic acids research. 28, 3497-3503 (2000).
- Caschera, F., et al. Coping with complexity: machine learning optimization of cell-free protein synthesis. Biotechnology and bioengineering. 108, 2218-2228 (2011).
- Hodgman, C. E., Jewett, M. C. Cell-free synthetic biology: thinking outside the cell. Metabolic engineering. 14, 261-269 (2012).
- Elowitz, M. B., Leibler, S. A synthetic oscillatory network of transcriptional regulators. Nature. 403, 335-338 (2000).
- Tsuruta, H., et al. High-level production of amorpha-4,11-diene, a precursor of the antimalarial agent artemisinin, in Escherichia coli. Plos One. 4, e4489(2009).
- Gardner, T. S., Cantor, C. R., Collins, J. J. Construction of a genetic toggle switch in Escherichia coli. Nature. 403, 339-342 (2000).
- Jewett, M. C., Forster, A. C. Update on designing and building minimal cells. Current opinion in biotechnology. 21, 697-703 (2010).
- Noireaux, V., Libchaber, A. A vesicle bioreactor as a step toward an artificial cell assembly. Proceedings of the National Academy of Sciences of the United States of America. 101, 17669-17674 (2004).
Erratum
Formal Correction: Erratum: Protocols for Implementing an Escherichia coli Based TX-TL Cell-Free Expression System for Synthetic Biology
Posted by JoVE Editors on 1/01/1970. Citeable Link.
A correction to Figure 5's legend has been made for the article Protocols for Implementing an Escherichia coli Based TX-TL Cell-Free Expression System for Synthetic Biology. Method 1 and method 2 have been switched.Z
The figure legend was update from:
Figure 5. Effects of DNA solution on expression efficiency. a) Comparison of two different purification methods for processing plasmids. 1 nM of pBEST-OR2-OR1-Pr-UTR1-deGFP-T500 is prepared using only a QiaPrep Spin Miniprep Kit (Purification method 1) or post-processed with a QiaQuick PCR purification kit (Purification method 2).
to
Figure 5. Effects of DNA solution on expression efficiency. a) Comparison of two different purification methods for processing plasmids. 1 nM of pBEST-OR2-OR1-Pr-UTR1-deGFP-T500 is prepared using only a QiaPrep Spin Miniprep Kit (Purification method 2) or post-processed with a QiaQuick PCR purification kit (Purification method 1).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon