È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Ad alta risoluzione Spatiotemporal Analisi del recettore Dynamics per singola molecola di microscopia a fluorescenza
In questo articolo
Riepilogo
This protocol describes how to use total internal reflection fluorescence microscopy to visualize and track single receptors on the surface of living cells and thereby analyze receptor lateral mobility, size of receptor complexes as well as to visualize transient receptor-receptor interactions. This protocol can be extended to other membrane proteins.
Abstract
Microscopia a singola molecola sta emergendo come un potente approccio per analizzare il comportamento delle molecole di segnalazione, in particolare per quanto riguarda quelli di aspetto (ad es., Cinetica, la coesistenza di diversi stati e popolazioni, interazioni transienti), che sono in genere nascosti nelle misure Ensemble, come quelli ottenuti con metodi biochimici o microscopia standard. Così, eventi dinamici, quali le interazioni recettore-recettore, possono essere seguite in tempo reale in una cellula vivente con alta risoluzione spazio-temporale. Questo protocollo descrive un metodo basato sulla marcatura con fluorofori organici piccoli e luminosi e riflessione interna totale fluorescenza (TIRF) microscopia di visualizzare direttamente singoli recettori sulla superficie delle cellule viventi. Questo approccio permette di localizzare con precisione i recettori, misurare la dimensione dei complessi recettoriali e catturare gli eventi dinamici come le interazioni recettore-recettore transitori. Il protocollo fornisce una descrizione dettagliata di come perform un esperimento singola molecola, compresa la preparazione del campione, l'acquisizione delle immagini e l'analisi delle immagini. Come esempio, l'applicazione di questo metodo per analizzare due proteine G recettori, cioè., Β 2-adrenergici e γ-aminobutirrico tipo B acido (GABA B) recettore, viene segnalato. Il protocollo può essere adattato ad altre proteine di membrana e modelli cellulari diversi, metodi di trasfezione e strategie di etichettatura.
Introduzione
Recettori situati sulla superficie cellulare percepire l'ambiente extracellulare e rispondono a una varietà di stimoli, come odoranti, ioni, piccole e grandi neurotrasmettitori ormoni proteici. La fluidità delle membrane cellulari consente movimenti di recettori e altre proteine di membrana. Ciò è essenziale per la formazione di complessi proteici e il verificarsi di interazioni proteina-proteina transitori, come quelli utilizzati da recettori per assemblare in unità funzionali e trasdurre segnali nell'interno delle cellule. Ad esempio, G-proteine-recettori accoppiati (GPCR), che costituiscono la più grande famiglia di recettori di superficie cellulare 1, sono stati proposti per formare di-/oligomers, che sembra essere coinvolto nella regolazione messa a punto di trasduzione del segnale e potrebbe avere importanti conseguenze fisiologiche e farmacologiche 2-5.
Microscopia singola molecola ha il grande potenziale di visualizzare direttamente con l'alta spatiotemprisoluzione orale il comportamento dinamico dei singoli recettori situati sulla superficie delle cellule viventi, compresi loro associazione per formare dimeri e complessi molecolari ordine superiore 6-10. Questo offre diversi vantaggi rispetto ai metodi biochimici e microscopia standard, che di solito riportano il comportamento medio di migliaia o milioni di molecole.
Etichettatura proteina con un fluoroforo sufficientemente luminoso e fotostabili è essenziale per microscopia singola molecola. Questo protocollo sfrutta il tag SNAP recentemente introdotto 11 per collegare in modo covalente fluorofori organici piccole e luminose ai recettori della superficie cellulare. SNAP è un kD tag 20 proteine derivate da l'enzima O alchiltransferasi umano 6-alkylguanine-DNA riparazione del DNA, che può essere irreversibile etichettato con fluoroforo-coniugato benzylguanine (fluoroforo-BG) derivati. CLIP, un tag ulteriormente ingegnerizzato derivato da SNAP, può essere invece etichettato con fluoroforo-cDerivati benzylcytosine onjugated 12.
Il protocollo riportato in questo manoscritto spiega come transfettare e l'etichetta SNAP-tagged 11 recettori con piccole fluorofori organici e utilizzare riflessione interna totale in fluorescenza (TIRF) microscopia a visualizzare singoli recettori o complessi recettore sulla superficie delle cellule viventi 10. I risultati del protocollo riportati in> 90% di efficienza etichettatura di un extracellulare SNAP-tagged proteine della superficie cellulare 10. Ulteriori informazioni su come utilizzare dati singola molecola di analizzare la dimensione e la mobilità dei complessi recettoriali, nonché per catturare le interazioni recettore-recettore transitori, è fornito. Un flusso di lavoro schematicamente l'intero protocollo è data in Figura 1. Come esempio, la trasfezione di ovaio di criceto cinese (CHO) con-G recettori accoppiati alla proteina SNAP-tag (GPCR) seguita da marcatura con un derivato fluoroforo-BG come nonché la sua applicazione alla quantify e il monitor del recettore di-/oligomerization sono descritti. Questo protocollo può essere estesa ad altre proteine della superficie cellulare e tag fluorescenti (ad es., CLIP), così come ad altri metodi di trasfezione e di etichettatura.
Protocollo
1. Preparazione del campione
- Pulizia Coverslip
NOTA: Lavorare sotto una cappa aspirante.- Utilizzare pinzette pulite per posizionare vetrini (diametro 24 mm) in un vetrino porta che separa le singole lamelle.
- Mettere il supporto con vetrini in un bicchiere e aggiungere cloroformio fino a quando i coprioggetti sono coperti. Coprire il becher con foglio di alluminio per ridurre l'evaporazione e ultrasuoni in un bagno sonicatore per 1 ora a RT. Prendete il titolare vetrino fuori del bicchiere e lasciare i coprioggetti asciutto.
- Ripetere passo 1.1.2 con la soluzione di NaOH 5M invece di cloroformio.
- Estrarre il porta vetrino in una nuova becher e lavare tre volte con acqua distillata. Mettere coprioggetti puliti in una piastra di coltura cellulare di vetro riempito con 100% di etanolo.
- Preparazione dei campioni di taratura
- Sciogliere colorante fluorescente in solvente appropriato.
- Preparare una diluizione seriale 1:10 del colorante fluorescente che vannoda 1:00 a 1 nM filtro (0,22 micron) di acqua sterilizzata.
- Prendere coprioggetti puliti memorizzati in 100% etanolo e lavare con acqua sterilizzata per filtrazione. Spot 20 ml di ogni diluizione colorante fluorescente su un vetrino pulito separato. Lasciate che i coprioggetti asciugare sotto una cappa sterile. Proteggere i coprioggetti dalla luce e dalla polvere fino al momento dell'uso. Utilizzare questi esempi per stimare l'intensità di molecole fluorescenti singoli (vedi punto 3).
- Transfezione
- Cellule CHO cultura in miscela 1:1 medio / nutriente Dulbecco Modified Eagle F-12 (DMEM/F12) supplementato con 10% siero bovino fetale (FBS), 100 U / ml di penicillina e 100 pg / ml di streptomicina a 37 ° C, in . 5% di CO 2 NOTA: Usare mezzi di comunicazione liberi fenolo-rosso tutto l'esperimento per ridurre al minimo autofluorescenza.
- Prendere coprioggetti puliti dalla soluzione di etanolo al 100%, lavarli con sterile tampone fosfato salino (PBS), e mettere un vetrino in ciascun pozzetto di un 6 celle ben cultura plmangiato.
- Trypsinize, contare e cellule CHO seme con una densità di 3 x 10 5 cellule / pozzetto nella piastra di coltura cellulare 6-pozzetti contenenti le lamelle. Lasciare che le cellule crescono in un incubatore (37 ° C, 5% CO 2) per 24 ore al fine di raggiungere ca. 80% di confluenza, che è la densità cellulare ottimale per la trasfezione.
- Per ogni bene, diluire 2 mg di DNA plasmide desiderato (ad es., SNAP-tag β del recettore 2-adrenergici) e 6 ml Lipofectamine 2000 a due tubi separati contenenti 500 microlitri di media OptiMEM. Incubare a temperatura ambiente per 5 min.
- Unire le soluzioni dal punto 1.3.4 in una provetta e miscelare per ottenere una miscela di trasfezione. Incubare la miscela di trasfezione a temperatura ambiente per 20 min.
- Durante l'incubazione (1.3.5), prendere le cellule CHO e lavare due volte con pre-riscaldato (37 ° C) PBS. Sostituire PBS con 1 ml / pozzetto di fenolo mezzo DMEM/F12 aneritro supplementato con 10% FBS ma senza antibiotici.
- Aggiungere l'intero transfectmiscela di ioni (1 ml) dal punto 1.3.5 goccia a goccia in ogni pozzetto, e delicatamente Rock il piatto avanti e indietro per garantire la completa miscelazione.
- Incubare per 2 a 4 ore a 37 ° C, in 5% CO 2 e procedere subito dopo il passaggio successivo. NOTA: Queste condizioni di trasfezione sono stati ottimizzati per raggiungere densità dei recettori <0,45 particelle / 2 micron, che sono adatti per singola l'imaging molecola. Le regolazioni possono essere necessari quando si utilizzano diverse celle, costrutti o reagenti.
- Etichettatura Protein
- Diluire 1 ml di soluzione madre fluoroforo-BG in 1 ml di mezzo DMEM/F12 supplementato con 10% FBS per ottenere una concentrazione finale di 1 mM. Prendere le cellule trasfettate dall'incubatore e lavare due volte con preriscaldata (37 ° C) PBS. Sostituire PBS con 1 ml di soluzione 1 mM fluoroforo-BG e incubare per 20 min a 37 ° C 5% CO 2 in un incubatore.
- Dopo l'incubazione, lavare le cellule trevolte con DMEM/F12 mezzo supplementato con 10% FBS, ogni volta seguiti da 5 minuti di incubazione a 37 ° C. Prendete un vetrino (con cellule marcate) con le pinzette e mettetelo in una camera di imaging.
- Lavare due volte con 300 microlitri di buffer di imaging. Aggiungere 300 ml di buffer di immagini fresche e procedere immediatamente alla diagnostica per immagini (parte 2).
2. Acquisizione Immagine
NOTA: Usare un totale di fluorescenza di riflessione interna (TIRF) microscopio, dotato di un obiettivo ad immersione in olio ad alta apertura numerica (ad esempio, apertura numerica 100X magnification/1.46.), I laser adatti (ad esempio, 405 nm, 488 nm, 561 nm e 645 laser a diodi nm), un dispositivo ad accoppiamento di carica dell'elettrone moltiplicando (EMCCD) macchina fotografica, un incubatore e un controllo della temperatura per visualizzare molecole fluorescenti singoli.
- Impostare i parametri desiderati microscopio, cioè., Linea laser, l'angolo TIRF (questo parametro controlla la penetrazionezione profondità del campo evanescente), tempo di esposizione, il frame rate e il numero di immagini per film 10. Mantenere il controllo del riscaldamento / incubatore e la temperatura sempre per evitare derive di temperatura e umidità di condensa.
- Mettere una goccia di olio di immersione sull'obiettivo 100X del microscopio. Posizionare la camera di imaging con le cellule marcate sul supporto esemplare di microscopio, e portare le cellule a fuoco in illuminazione campo chiaro.
- Passare alla illuminazione TIRF. Mantenere potenza del laser più basso possibile per consentire una ricerca per la cella desiderata ma allo stesso tempo minimizzando fotoscolorimento.
- Selezionare la cella desiderata e fine regolare la messa a fuoco. Impostare la potenza del laser ad un livello che permette la visualizzazione di singoli fluorofori. Acquisire sequenza di immagini e salvare file di sequenza di immagini crudo. Tiff.
3. Calibration (Fluorofori singoli su vetro e monomeriche / dimerica controlli Receptor)
- Assemblare ogni sampl taraturae preparato come descritto in 1.2 nella camera di imaging. Collocare ogni campione sul microscopio e scegliere il campione contenente macchie diffrazione limitata ben separati che candeggina in un unico passaggio. NOTA: Questi punti rappresentano singole molecole del colorante fluorescente.
- Acquisire TIRF sequenze di immagini come descritto al punto 2. Importante: gli stessi parametri di imaging devono essere utilizzati per tutti gli esperimenti, compresi quelli per la calibrazione.
- Eseguire la rilevazione e l'analisi di monitoraggio come indicato in 4,1-4,2. Estrarre l'intensità di ciascuna particella come descritto al punto 4.2.6. Da questi dati, calcolare la media (μ) e la deviazione standard (σ) dell'intensità dei singoli fluorofori.
- Facoltativo: eseguire la stessa analisi su cellule trasfettate con un recettore sulla superficie cellulare monomerica (. Esempio, CD86), N-terminale con etichetta una o due copie di SNAP 10 ed etichettato con il derivato fluoroforo-BG. Follow la procedura sopra descritta per il singolo fluoroforo su vetro. Stimare l'efficienza di etichettatura come descritto in Calebiro, D. et al. 10
4. Image Analysis
- Preparazione sequenza di immagini
- Utilizzare un software di elaborazione delle immagini (ad es., ImageJ) per ritagliare le immagini.
- Salvate singoli fotogrammi come separato. Immagini TIFF in una nuova cartella, indicando su ogni immagine il numero di telaio.
- Misurare la superficie cellulare disegnando una regione di interesse (ROI) lungo il contorno di una cella mediante lo strumento di misura in ImageJ o uno strumento simile in un altro software. Utilizzare questo valore per calcolare la densità delle particelle dividendo il numero totale di particelle all'inizio del film dalla superficie cellulare.
- Rivelazione di particelle e di monitoraggio
NOTA: Utilizzare un software non commerciali come u-track 13, lavorando in Matlabambiente, per rilevare automaticamente e tenere traccia di particelle recettore singoli.
NOTA: L'algoritmo u-track si basa su un approccio di monitoraggio a più ipotesi. Questo approccio unisce le particelle tra i fotogrammi con la costruzione di matrici di costo, in cui vengono assegnati i singoli probabilità che una data particella in un fotogramma corrisponde a una data particella nel frame successivo, appare, scompare o si fonde / divide con / da altre particelle. La soluzione che minimizza globalmente i costi, cioè., Quello con la più alta probabilità, è finalmente selezionato. Questo permette anche di inseguimento una particella scomparire temporaneamente, un fenomeno tipico causato da fluoroforo lampeggiante. L'ultima versione di u-track (2.1.0) dispone di interfacce utente grafiche che facilitano l'esecuzione di queste analisi.- Dal prompt dei comandi Matlab, digitare "movieSelectorGUI" per aprire l'interfaccia di selezione di film. Seguire le istruzioni per creare unnuovo database di film a partire dalle immagini separate salvate in precedenza (vedi 4.1.2).
- Fornire la dimensione dei pixel in nm, intervallo di tempo in secondi, apertura numerica, profondità di bit fotocamera e lunghezza d'onda di emissione del fluoroforo, richiesta per il rilevamento di particelle e di inseguimento. Salvare il database di film.
- Dall'interfaccia selezione di film, eseguire l'analisi, la scelta di "particelle singole" come tipo di oggetti. Verrà visualizzata una nuova finestra in cui i parametri utilizzati per il rilevamento di particelle e di monitoraggio possono essere definiti. Inizia con i parametri di default. Più tardi, regolare questi parametri se la qualità della rilevazione e / o di monitoraggio non è soddisfacente (ad esempio, alcune particelle non vengono rilevati o tracce sono frammentati) Facoltativo:. Sotto le impostazioni di rilevamento, selezionare "i risultati di monitoraggio Esportare in formato matrice" per memorizzare le coordinate e le ampiezze delle particelle in una singola matrice (campo denominato "trackedFeaturedInfo"). Per una descrizione dettagliata di questi parametri, fare riferimento alla documentazione u-track.
- Eseguire l'algoritmo di rilevamento. Questo algoritmo determinare automaticamente la posizione e l'intensità sopra sfondo di ogni spot diffrazione limitata (ie., Complessi singoli recettori / recettore), calibrando una funzione gaussiana bidimensionale con deviazione standard pari alla funzione punto diffusione del microscopio intorno massimi di intensità locale . Quindi, eseguire l'algoritmo di tracking. Memorizzare i risultati delle analisi in un file mat..
- Utilizzare la routine "movieViewer", contenuto nel pacchetto u-track, o quelli personalizzati simili a visualizzare le tracce e controllare la qualità della rilevazione e di monitoraggio.
- Aprire il file mat. Vedere la posizione e l'ampiezza (cioè., Intensità) delle particelle rilevate ad ogni frame. Dati generati nel passaggio 4.2.4 sono contenuti nel campo tracksCoordAmpCG della ar tracksFinalray e / o in trackedFeaturedInfo. Dal numero totale di particelle rilevato calcolare la densità delle particelle dividendo questo valore per l'area superficiale delle cellule misurata a 4.1.3.
- Facoltativo: utilizzare le coordinate della particella nel tempo (vedere 4.2.6) analizzare il moto delle particelle recettore. Calcolare gli spostamenti medi quadrati (MSD) e coefficienti di diffusione (D) utilizzando Matlab o software simili. Per ogni particella e ogni intervallo di tempo (Δ t) considerato, calcolare lo spostamento quadratico medio (MSD) utilizzando la seguente formula:
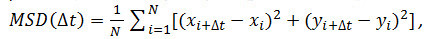
dove Δ t è l'intervallo di tempo in fotogrammi, N è il numero di passi analizzati, x ed y sono x della particella e coordinate Y al telaio indicata dall'indice. Utilizzare il MSD su appezzamenti di tempo per valutare il tipo di moto di una data particella: relazioni lineariindicare diffusione gratuita (ie., moto browniano), una curvatura positiva (cioè., la curva appare come una parabola) suggerisce movimento diretto, una curvatura negativa è indicativo del movimento confinato 14. Nel caso di particelle diffondenti banche, calcolare il coefficiente di diffusione (D) di ciascuna particella inserendo i dati MSD ottenuti con la seguente equazione: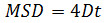
- Calcolo della dimensione delle particelle con il metodo di montaggio gaussiana
NOTA: Una volta che la distribuzione di intensità di campioni di taratura (singoli fluorofori su vetro e / o recettori monomerici fluorescente) è noto, eseguire una misura gaussiana mista sulla distribuzione delle intensità delle particelle all'inizio di una sequenza di immagini per determinare la dimensione di complessi recettoriali (cioè., il numero di recettori per particella) 10. Eseguire queste analisi utilizzando Matlab o ssoftware analoghe sui.- Calcolare l'intensità di ciascuna particella facendo la media dell'intensità della particella dal primo fotogramma per fotogramma prima della prima variazione di intensità verificato (nella maggior parte dei casi decremento per photobleaching).
- Eseguire un raccordo gaussiana misto secondo la seguente equazione:
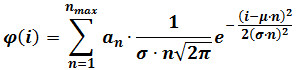
dove φ (i) è la frequenza di particelle aventi intensità I, n è il numero dei componenti, α n è un parametro che contribuisce alla altezza del componente n, μ e σ sono la media e la deviazione standard della intensità di riferimento singoli fluorofori . (calcolato come descritto al punto 3) NOTE: Determinare tegli massimo numero di componenti (n max) per ogni sequenza immagine aumentando progressivamente n max finché l'aggiunta di un componente non più produrre un migliore raccordo statisticamente, come giudicato da un F-test. - Optional: (. Esempio, gli ultimi 60 fotogrammi di una sequenza di 400 telaio) Eseguire un fit gaussiano mista sulla distribuzione dell'intensità ottenuto sugli ultimi fotogrammi del filmato. Sostituire μ e σ con i valori ottenuti dopo il montaggio, che forniscono stime raffinate per tali parametri e ripetere il punto 4.3.2.
- Calcolare l'area sotto la curva (AUC) di ciascun componente del fit gaussiano mista. Calcolare l'abbondanza relativa di particelle recettore di differenti dimensioni (cioè, monomero, dimero, trimero, ecc) dividendo il valore AUC di ogni componente l'AUC dell'intera distribuzione.
- Facoltativo: utilizzare dati provenienti da diverse cellule e le corrispondenti densità di particelle (calcolati come descritto in 4.2.6) per generare trame dove la distribuzione di particelle con dimensioni diverse è correlata con la densità delle particelle 10.
- Calcolo della dimensione delle particelle con metodo sagomata passo
NOTA: Utilizzare un analisi passo-montaggio come metodo alternativo per determinare la dimensione dei complessi recettoriali 10. La base per questa analisi è che la distruzione indotta dalla luce (photobleaching) di una singola risultati fluoroforo in sua scomparsa istantanea - quindi particelle contenenti n fluorofori dovrebbero progressivamente candeggina produrre un profilo di intensità con n passi.- Estrarre i profili di intensità di ogni particella dal file. Mat generato da u-track o simile software di rilevamento / tracking (vedi 4.2.6).
- Utilizzare un algoritmo passo-montaggio, come quello presentatoin rif. 10, per contare il numero di passi sbianca per ogni particella.
- Facoltativo: utilizzare i risultati per generare distribuzioni mostrano l'abbondanza relativa di particelle recettori di diverse dimensioni e correlare con densità di particelle come precedentemente descritto per i risultati del raccordo gaussiana mista (vedi 4.3).
Risultati
Il protocollo descritto può essere applicato ad una varietà di differenti proteine di membrana. Come esempio, risultati rappresentativi ottenuti con β 2-adrenergici e GABA B sono riportati 10. Poiché i segnali fluorescenti da singole molecole sono deboli, minimizzazione di fluorescenza di fondo è il primo passo fondamentale per ottenere buoni risultati. Quindi, è importante utilizzare coprioggetti ampiamente puliti (Figura 2A) così da minimizzare autofluores...
Discussione
Il protocollo descritto permette l'analisi della disposizione spaziale, mobilità e dimensioni dei complessi recettore della superficie cellulare a livello di singola molecola. Rispetto all'uso di proteine fluorescenti, etichettatura con piccole fluorofori organici, che sono più luminoso e fotostabile, ha il vantaggio di permettere la visualizzazione estesa del recettore particelle singole. Poiché i livelli di espressione estremamente bassi sono raggiunti (<0,45 particelle recettore / micron 2),<...
Divulgazioni
The authors declare that they have no competing financial interests.
Riconoscimenti
The development of this protocol was supported by grants from the European Research Council (Advanced Grant TOPAS to M.J.L.) and the Deutsche Forschungsgemeinschaft (Grants CA 1014/1-1 to D.C. and SFB487 to M.J.L.). T.S. was supported by the Alexander von Humboldt Foundation.
Materiali
| Name | Company | Catalog Number | Comments |
| Chloroform | AppliChem GmbH | A1585 | CAUTION: toxic and irritating substance as well as a possible carcinogen |
| NaOH | Sigma-Aldrich | S8045 | CAUTION: strong base and highly corrosive reagent |
| Absolute ethanol | Sigma-Aldrich | 32205 | |
| Glass coverslip | Marienfeld-Superior | 111640 | 24 mm diameter, 0.13-0.16 mm thickness |
| 0.2 mm sterile filter | Sarstedt | 83.1826.001 | |
| CHO cells | ATCC, USA | ATCC CCL-61 | Chinese hamster ovary cell line |
| 6-well cell culture plate | Nunc | 140675 | |
| DMEM/F-12 medium | GIBCO, Life Technologies | 11039-021 | Phenol-red free medium |
| Fetal bovine serum | Biochrom | S 0115 | |
| Penicillin - streptomycin | Pan Biotech GmbH | P06-07 100 | |
| Trypsin-EDTA | Pan Biotech GmbH | P10-23100 | |
| Lipofectamine 2000 | Invitrogen, Life Technologies | 11668-019 | |
| Opti-MEM I Reduced Serum Medium | Invitrogen, Life Technologies | 31985-047 | |
| Fluorophore-conjugated benzylguanine | New England BioLabs | S9136S | SNAP-Surface Alexa Fluor 647. Make a 1 mM stock solution in DMSO. Store at -20°C. |
| DMSO | AppliChem GmbH | A1584 | |
| Imaging buffer: | 137 mM NaCl, 5.4 mM KCl, 2 mM CaCl2, 1 mM MgCl2, 10 mM HEPES, pH 7.3, sterile-filtered | ||
| NaCl | AppliChem GmbH | A1371 | |
| KCl | AppliChem GmbH | A3582 | |
| CaCl2 | AppliChem GmbH | A2303 | |
| MgCl2 | AppliChem GmbH | A3618 | |
| HEPES | AppliChem GmbH | A3724 | |
| Imaging chamber | Molecular Probes, Life Technologies | A-7816 | Attofluor Cell Chamber, for microscopy |
| TIRF-M | Leica | Model: DMI6000B | |
| TIRF objective | Leica | 11 506 249 | HCX PL Apo 100x/1.46 Oil CORR |
| EM-CCD camera | Roper Scientific | Photometrics Cascade 512B | |
| Temperature controller | Pecon | Tempcontrol 37-2 digital | |
| ImageJ software | NIH, USA | http://rsbweb.nih.gov/ij | |
| u-track software | Laboratory for computational cell biology, Dept. of Cell Biology, Harvard Medical School, USA | http://lccb.hms.harvard.edu/software.html | |
| Matlab software | The MathWorks, USA |
Riferimenti
- Pierce, K. L., Premont, R. T., Lefkowitz, R. J. Seven-transmembrane receptors. Nat Rev Mol Cell Biol. 3, 639-650 (2002).
- Angers, S., Salahpour, A., Bouvier, M. Dimerization: an emerging concept for G protein-coupled receptor ontogeny and function. Annu Rev Pharmacol Toxicol. 42, 409-435 (2002).
- Ferré, S., et al. Building a new conceptual framework for receptor heteromers. Nat Chem Biol. 5, 131-134 (2009).
- Milligan, G. G. protein-coupled receptor hetero-dimerization: contribution to pharmacology and function. Br J Pharmacol. 158, 5-14 (2009).
- Lohse, M. J. Dimerization in GPCR mobility and signaling. Curr Opin Pharmacol. 10, 53-58 (2010).
- Ulbrich, M. H., Isacoff, E. Y. Subunit counting in membrane-bound proteins. Nat Methods. 4, 319-321 (2007).
- Triller, A., Choquet, D. New concepts in synaptic biology derived from single-molecule imaging. Neuron. 59, 359-374 (2008).
- Hern, J. A., et al. Formation and dissociation of M1 muscarinic receptor dimers seen by total internal reflection fluorescence imaging of single molecules. Proc Natl Acad Sci U S A. 107, 2693-2698 (2010).
- Kasai, R. S., et al. Full characterization of GPCR monomer-dimer dynamic equilibrium by single molecule imaging. J Cell Biol. 192, 463-480 (2011).
- Calebiro, D., et al. Single-molecule analysis of fluorescently labeled G-protein-coupled receptors reveals complexes with distinct dynamics and organization. Proc Natl Acad Sci U S A. 110, 743-748 (2013).
- Keppler, A., et al. A general method for the covalent labeling of fusion proteins with small molecules in vivo. Nat Biotechnol. 21, 86-89 (2003).
- Gautier, A., et al. An engineered protein tag for multiprotein labeling in living cells. Chem Biol. 15, 128-136 (2008).
- Jaqaman, K., et al. Robust single-particle tracking in live-cell time-lapse sequences. Nat Methods. 5, 695-702 (2008).
- Saxton, M. J., Jacobson, K. Single-particle tracking: applications to membrane dynamics. Annu Rev Biophys Biomol Struct. 26, 373-399 (1997).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon