Method Article
Flusso di lavoro per High-contenuti, Quantificazione singola cella di fluorescenti Marcatori da Universal Microscopio dati, supportati da Open Source Software
In questo articolo
Riepilogo
Presentato è un flusso di lavoro flessibile, l'informatica che consente l'analisi di immagini basata multiplex di cellule fluorescente. Il flusso di lavoro quantifica marcatori nucleari e citoplasmatici e calcola marcatore traslocazione tra questi compartimenti. Le procedure sono previste per la perturbazione di cellule utilizzando siRNA e metodologia affidabile per il rilevamento marcatore mediante immunofluorescenza indiretta in formati da 96 pozzetti.
Abstract
I progressi nella comprensione dei meccanismi di controllo che disciplinano il comportamento delle cellule in coltura di tessuti aderenti modelli mammiferi stanno diventando sempre più dipendenti dalle modalità di analisi singola cellula. Metodi che forniscono i dati compositi che riflettono i valori medi dei biomarcatori di popolazioni cellulari rischiano di perdere dinamiche sottopopolazione che riflettono l'eterogeneità del sistema biologico studiato. In linea con questo, gli approcci tradizionali vengono sostituiti da, o supportati con forme più sofisticate di test cellulari sviluppati per consentire la valutazione al microscopio ad alto contenuto. Questi test potenzialmente generare un gran numero di immagini di biomarcatori fluorescenti, che hanno permesso accompagnando pacchetti di software proprietario, consente per le misure multi-parametriche per cella. Tuttavia, i costi relativamente elevati di capitale e superspecializzazione di molti di questi dispositivi hanno impedito la loro accessibilità a molti ricercatori.
Descritto qui è unworkflow universalmente applicabili per la quantificazione di più intensità marcatori fluorescenti da specifiche regioni subcellulare di cellule singole adatti per l'uso con le immagini dalla maggior parte microscopi a fluorescenza. La chiave di questo flusso di lavoro è l'implementazione del software Profiler cellulare liberamente disponibile da 1 a distinguere le cellule individuali in queste immagini, li segmento in regioni subcellulari definite e fornire intensità marcatore di fluorescenza valori specifici di queste regioni. L'estrazione dei singoli valori di intensità di cellule dai dati immagine è lo scopo centrale di questo flusso di lavoro e verrà illustrato con l'analisi dei dati di controllo da una schermata di siRNA per G1 regolatori checkpoint in cellule umane aderenti. Tuttavia, il flusso di lavoro qui presentata può essere applicato all'analisi dei dati da altri mezzi perturbatori cellulare (per esempio, schermi composti) e altre forme di marcatori cellulari fluorescenza base e quindi dovrebbe essere utile per una vasta gamma di laboratori.
Introduzione
Il lavoro qui presentato descrive l'uso del software Profiler cella liberamente disponibili per eseguire ripartizione algoritmo guidata di immagini di microscopia a fluorescenza di cellule aderenti per identificare le singole cellule e regioni subcellulari definite. Questo approccio, denominato segmentazione di immagini, consente la successiva analisi multi-parametrica delle cellule immaginati quantificando marcatori fluorescente localizzate ad ogni cella o regione subcellulare (denominato oggetti segmentate). Questo flusso di lavoro costituisce la base per consentire l'analisi ad alta contenuto e lo scopo di servire come uno strumento che può essere ulteriormente sviluppato e modificato per soddisfare multi-parametrica, analisi singola cellula in laboratorio senza accesso agli strumenti ad alto contenuto specializzati o software proprietario. I file forniti con questo manoscritto includono una serie di test di importanti dati di immagine grezzi, le impostazioni di algoritmo e script di supporto per generare l'analisi descritto. La condizione impostazioni algoritmo fo Profiler cellulare sono ottimizzati per i set di dati di esempio ei dettagli sezione Discussioni quanto possono essere necessari aggiustamenti per consentire l'uso dei dati di immagine provenienti da altri studi.
Una volta dati quantitativi è stato estratto utilizzando Profiler cellulare, laboratori diversi possono avere esigenze diverse per come utilizzare le informazioni presentate dai valori delle celle individuali nei dati grezzi; mostrato qui è un approccio con cui porte vengono applicati ai dati grezzi per ogni test. Usando questi cancelli, i dati sono trasformati in termini binarie di risposta, consentendo la visualizzazione delle tendenze collegano diversi trattamenti con le sottopopolazioni di cellule in risposta definito dalle porte. Le porte sono impostati sulla base di osservazioni delle distribuzioni di dati ottenuti appropriati controlli negativi e positivi per ogni misura rilevante. L'uso dei cancelli è solo un esempio di come gestire le misure, basati su celle prime. Anche qui mostrato è l'uso di intensità DNA nucleare measurements nella loro forma grezza come un intervallo continuo di valori in combinazione con i dati gated. Altri approcci per la gestione dei dati di analisi di immagine devono essere considerati, a seconda della natura dello studio; alternative statistici all'utilizzo cancelli per l'assegnazione di celle a sottopopolazioni sono stati segnalati 2 e sistematiche confronto tra le strategie per riassumere i dati ad alta contenuto attraverso un gran numero di parametri sono stati segnalati 3.
Analisi-alto contenuto di dati immagine hanno trovato impiego in studi cellulari di droga-risposta, invertire la genetica e stress ambientale segnalazione 4-6. Il merito di analisi ad alto contenuto di deriva dal fatto che l'analisi algoritmica dei dati microscopia a fluorescenza permette parametri quantitativi e spaziali da considerare simultaneamente in singole celle 7. In questo modo, esiti cellulari per analisi multiple possono essere, comportamento differenziale incrociato dei test-dsottopopolazioni cellulari efined possono essere monitorati all'interno di condizioni sperimentali e test possono includere la considerazione di variabili morfologiche. Il flusso di lavoro strategie e analisi discusso qui, come per altri approcci ad alto contenuto, sono in grado di fornire dati multiplex che sono riferimenti incrociati alle singole celle. Studi ad alto contenuto di metodi tuta che generano immagini al microscopio a fluorescenza e sono applicabili all'analisi dei dati che vanno da decine di immagini prodotte in basso erogato microscopia convenzionale basato sulla fluorescenza attraverso le migliaia di immagini prodotte utilizzando piattaforme ad alto contenuto di screening automatizzati.
Il flusso di lavoro è illustrata qui con i dati di esempio da cui test separati sono misurate in termini di intensità sia marcatori fluorescenti nucleari o traslocazione citoplasmatica / nucleare di una proteina reporter fluorescente, rispettivamente. Il flusso di lavoro è flessibile in quanto questi saggi possono essere considerati separatamente o in combinazione seconda each dato domanda di ricerca da diversi ricercatori. I dati di esempio sono prodotti come parte di una interferenza dell'RNA (RNAi) esperimento (Figura 1). Piccoli oligonucleotidi RNA interferenti (siRNA) vengono utilizzati per atterramento specifiche proteine in cellule di carcinoma colorettale umano HCT116 che danno luogo a cambiamenti per due reporter fluorescenti di chinasi (CDK) attività di ciclina-dipendente. La fosforilazione CDK6-dipendente della proteina retinoblastoma nucleare di serina 780 (P-S780 RB1) è valutata con anticorpi colorazione. Nelle stesse cellule, una proteina fluorescente-tag giornalista verde di attività CDK2 (GFP-CDK2 giornalista) è valutata dal nucleare rapporto citoplasmatica dove in assenza di attività CDK2 reporter risiede nel nucleo e su navette attivazione CDK2 nel citoplasma 8. Inoltre, il DNA nucleare di ciascuna cella viene tinto con un colorante-DNA intercalanti, Bisbenzimide, che serve come mezzo per identificare le cellule e definire i confini nuclei nelle immagini così unmisura sa dell'abbondanza DNA fornire informazioni sulla posizione del ciclo di cella della cella (Figura 2).
Le attività di CDK2 e CDK6 sono rilevabili come cellule transito da G1 alla fase S del ciclo cellulare 5 e succedono 9,10 e, come tale, stretta concordanza tra i due reporter in cellule singole è previsto. L'insieme di dati di dimostrazione qui utilizzato analizza come esempio l'effetto di siRNA obiettivi CDK6, proteina retinoblastoma (RB1) e un controllo negativo non-targeting (Tabella 1). Knockdown di CDK6 deve provocare sia una diminuzione della RB1 epitopo P-S780 e un accumulo di cellule in fase G1 del ciclo cellulare. Il knockdown RB1 serve da controllo reattivo per la specificità dell'anticorpo fosfo-S780. Immagini al microscopio a fluorescenza da formalina fisso 11, colture cellulari HCT116 fluorescenti colorate vengono utilizzati per l'analisi delle immagini algoritmica. I dati numerici risultante viene quindi utilizzato perriferimento incrociato i giornalisti e misurare l'impatto dei diversi stati atterramento.
La dimensione potenziale dei dati prodotti da questo tipo di analisi può presentare una sfida per normali strumenti di analisi. Ad esempio, i dati delle celle individuali possono essere più grandi di alcuni software foglio ospiterà. Incluso sono script Perl che eseguono semplici, altamente ripetitivo, lavorazione sotto la supervisione dei dati per aiutare l'analisi di grandi set di dati. Gli script Perl sono scritti appositamente per i file di output prodotti da cellule Profiler, durante l'elaborazione di file di immagini con una specifica convenzione di denominazione dei file (Figura 3), e consentono un numero variabile di campi per bene da utilizzare nell'analisi. Spesso è importante dati del saggio cancello singola cella per monitorare tendenze sottopopolazioni di cellule 5 e qui illustrato è l'uso di uno script Perl per contrassegnare ogni cella basata su un cancello insieme predeterminato per ogni tipo di saggio. Sono inclusi anche gli script opzionali Perlche riassumono l'esito dei dati per i singoli pozzi (o condizioni), offrendo: percentuale di cellule all'interno del cancello set ed i valori medi dei punteggi del test grezzi. Quest'ultimo modo più omogenea visualizzazione dati, vale dove risposte influenzano tutti o la maggior parte delle cellule all'interno di un pozzo. Come discusso sopra, tale valutazione è meno utile di quello previsto dalla persona gating dati cella in cui la risposta si limita a un sottogruppo di cellule all'interno di una popolazione.
L'utilità del flusso di lavoro descritto non è limitato alle perturbazioni da siRNA o saggi marcatori descritti. Gli studi hanno utilizzato questo approccio per saggiare le risposte in tessuto esperimenti di coltura utilizzando combinazioni di siRNA, inibitori chimici e trattamento con radiazioni e per la valutazione dei marcatori diversi CDK6 e l'attività CDK2 5.
Concettualmente, la strategia sperimentale permette una varietà di regioni subcellulari biologicamente utili da registrare automaticamentein singole cellule presenti nelle immagini al microscopio a fluorescenza. Come tale, questo approccio può produrre, dati multiplex rivelare informazioni biologiche quantitative che possono perdere attraverso tecniche che si concentrano sulle popolazioni piuttosto che singole celle. Con piccole modifiche, il flusso di lavoro di avvicinamento e di analisi descritto può produrre, i dati delle singole celle quantitativi per le uscite di analisi basate sulla fluorescenza e le risposte delle cellule-biologica, in cui la valutazione quantitativa del contenuto di DNA, la quantificazione della fluorescenza nucleare o citoplasmatica o la spola di marcatori tra queste due scomparti singolarmente o in modo multiplex è di interesse. Come requisiti editrici tendono sempre più verso la presentazione dei dati grezzi apertamente accessibili, l'accesso e la familiarità con strumenti gratuiti per l'analisi di immagini di microscopia, come quelle descritte qui sarà anche di diretto interesse per i laboratori in cerca di rianalizzare i dati pubblicati.
Protocollo
1. sperimentale Perturbazione e etichettatura delle cellule per la risposta Markers (Schermo Reverse Transfection siRNA)
- In una sterile cultura del tessuto cappa pipetta 70 ml di 200 Nm siRNA in 1x tampone siRNA in pozzetti di una pianura 96 piastra sterile, bene. Diluire transfezione lipidi in 40 volumi di supporti privi di siero DMEM ed erogare 105 ml in ciascun pozzetto contenente siRNA.
NOTA: La diluizione 262,5 ml di lipidi in 10,5 ml di DMEM senza siero produce un master mix adatto per un intero 96 pozzetti di siRNA, offrendo 2,6 ml di lipidi per pozzetto. L'utilizzo di 200 Nm a partire siRNA concentrazione in questa fase fornirà una soluzione di lavoro di 20 Nm nella fase 1.3, ma le procedure di lavoro per le concentrazioni di lavorare fino a 5 Nm, con la concentrazione di partenza adeguato di conseguenza (cioè 50 Nm). Concentrazione di lavoro inferiore può ridurre punteggi positivi falsi fuori bersaglio, anche se possono ridurre l'entità della risposta on-target, che porta ad aumentare di on-target percentuali di falsi negativi. - Mescolare la piastra dolce vibrazione per dieci minuti a temperatura ambiente. Suddividere i risultanti 175 ml in tre 50 microlitri repliche per bersaglio su un, colture di tessuti opachi trattati, piastra a 96 pozzetti con una base trasparente.
- Transfect Reverse erogando 8.000 cellule per pozzetto in 150 microlitri DMEM contenente 10% di siero direttamente sui complessi lipidi-siRNA 50 microlitri. Cellule colorettali umani Usa HCT116 esprimono stabilmente un marcatore GFP-tagged segnalazione dell'attività CDK2 5,8. Nessun ulteriore miscelazione è necessario. Sigillare la piastra con una sterile, colla membrana traspirante per controllare l'umidità e prevenire 'edge-effetti' piatto e posizionare la piastra in un incubatore umidificato a 37 ° C, 5% di CO 2 per 48 ore.
- Aspirare il supporto in modo che una piccola quantità residua di supporto rimane nei pozzetti. Fissare le cellule aggiungendo 100 ml di formaldeide al 4% tamponata ad ogni pozzetto ed incubare in una cappa aspirante per 10 min a camera temperature.
- Rimuovere la soluzione di fissaggio aspirando la piastra. A questo punto o interrompere l'esperimento lavando piastra tre volte con 100 ml di soluzione salina tampone fosfato (PBS) e quindi memorizzare sigillato, sotto 100 ml di PBS al buio a 4 ° C per un massimo di una settimana, o procedere con il permeabilizzazione delle cellule.
NOTA: Si consiglia di lastre di lavorazione il più presto possibile dopo la fissazione, e in generale preferiscono lo stoccaggio delle lastre completamente trasformati. Conservanti biocidi, quali il thimerosal, azoturo di sodio, o alternative commerciali possono essere aggiunti per prevenire la crescita micoroganismal. L'aggiunta di inibitori di fosfatasi aiuta a preservare fosfo-epitopi, e altri mezzi per conservare gli stati di modificazione delle proteine può essere utile in importanti contesti di analisi - Rimuovere PBS dalla piastra e permeabilize le cellule aggiungendo 100 ml di soluzione di permeabilizzazione. Incubare per 10 minuti a temperatura ambiente senza agitare. Aspirare la soluzione permeabilizzazione utilizzando un MulticanaleAnnel pipetta. Ripetere questa operazione tre volte.
- Bloccare le cellule aggiungendo 100 microlitri soluzione blocco per pozzetto per 30 minuti a temperatura ambiente. Rimuovere la soluzione blocco aspirando la piastra, poi sondare con 50 ml di anticorpo anti RB1 P-S780 diluito 500 volte nella soluzione di blocco per 2 ore al buio a temperatura ambiente.
- Lavare la piastra tre volte con 100 microlitri di soluzione di lavaggio piastra, lasciando la soluzione sulla piastra per 5 minuti ogni volta. Sonda la piastra una notte al buio a 4 ° C con 50 microlitri fluorescenza-tagged anticorpo secondario diluito 1.000 volte in soluzione blocco integrato con 2 mM del colorante DNA specifico-cromatina Bisbenzimide. Lavare la piastra tre volte prima e deposito chiuso, in 100 microlitri di PBS al buio a 4 ° C. Immagine la piastra entro due settimane.
2. Imaging e segmentazione di immagini
- Utilizzare un microscopio confocale o spinning-disk di fluorescenza con un obiettivo 20X per prendere parte 16-bit, immagini TIFF in scala di grigi a tre canali corrispondenti al colorante DNA, GFP e fluorofori immuno-colorazione. Cattura molti set di immagini non sovrapposte, di cui qui come cornici, per l'immagine di circa 1.000-2.000 cellule per bene.
- Nome i file di immagine in modo sistematico in modo che ogni nome di file è una combinazione unica di 'nome dell'esperimento', 'beh indirizzo', 'numero di telaio' e 'identificatore di canale', in questo ordine (Figura 3). Il set di dati di esempio utilizza "Blue" (cromatina colorazione DNA) o "Green" (GFP) o "Red" (il fluoroforo immuno-tinto) come identificatori di canale. L'indirizzo ben, numero di fotogramma e il canale identificatore sono ulteriormente denominato metadati dell'immagine. Utilizzare il simbolo di sottolineatura per evitare confusione bene e telaio metadati.
- Nome i file con questi elementi di metadati nell'ordine specificato. Ciò è necessario per garantire che i passi successivi software Correset di gruppo ctly di immagini per l'analisi.
- Scaricare e installare il Profiler cella freeware, attivo Perl Community Edition, R ambiente di programmazione statistica e rstudio. Accettare tutte le opzioni di default durante l'installazione; Gli utenti PC che installano attivo Perl dovrebbero consentire a tutti possibilità per PATH, associazione di file di estensione e la mappatura script in cui viene richiesto. Attivo Perl è facoltativo per gli utenti Mac, ma saranno di necessità di eseguire lo script Perl in fase 3.2 dalla riga di comando del terminale piuttosto che utilizzare l'icona clic.
- Aprire il software Profiler cellulare, fare clic su 'File', 'Import Pipeline' poi 'Da file' e selezionare il file 3_channels_pipeline.cppipe (Figure S1A e S1B). Il file contiene le istruzioni necessarie per il software di interpretare l'immagine metadati del file dalla convenzione nome del file descritto. Profiler cellulare riguarda ora le immagini, estrae il DNA nucleare e intensità di anticorpi da these e utilizza il canale GFP per calcolare il rapporto di intensità rispetto nucleare citoplasma per ogni cella rilevati (figure 4 e 5).
- Fare clic sul 'impostazioni di uscita View' pulsante nell'angolo in basso a sinistra della finestra Profiler cella. Nella parte superiore del nuovo schermo sono caselle di testo etichettati 'cartella predefinita Input' e 'cartella di destinazione predefinita'. Uno alla volta, fare clic sulla cartella-icone a destra di queste caselle e selezionare il percorso dei file di immagine per l'analisi e la destinazione dei dati estratti, rispettivamente (Figura S1C).
- Iniziare l'analisi delle immagini, premendo il pulsante 'analizzare le immagini "nell'angolo in basso a sinistra del Profiler Cell. Nella parte inferiore dello schermo osservare il tempo rimanente per l'estrazione dei dati, 'Stop Analysis' e pulsanti "Pausa". Se necessario sospendere l'analisi selezionando il tasto 'Pause' in qualsiasi momento, il che è utilequando si guardano le immagini in fase di analisi (descritto al punto 2.8).
- Facoltativamente, aprire le finestre per una qualsiasi delle fasi di analisi dell'immagine facendo clic oculari icone nel pannello all'estrema sinistra della finestra del programma (Figura S1D). Osservare la finestra 'IdentifyPrimaryObjects' e quelli per 'secondari' e 'Oggetti Terziario' per verificare che le impostazioni attuali Profiler cellulare per effettuare la segmentazione di immagini adatte (vedi Figura 1 e discussione per consigli sulla modifica di queste impostazioni).
- Fare clic su 'Ok' nella finestra di messaggio che appare quando l'analisi è completa. Vai alla posizione 'Default Output Folder', dove tutti i file di dati con i risultati vengono salvati come valori separati da virgole-(.csv) i file (Figura S2A).
3. Estrazione dei dati
- Trovare il nuovo file 'Nuclei.csv', che è inclusa tra iUscita dal Profiler cellulare. Questo file contiene i dati delle singole celle per fluorescenti intensità anticorpo nucleare, intensità DNA nucleare e valori del rapporto giornalista GFP-CDK2 (Figure 6A e S2A).
NOTA: Numerosi laboratori vorranno elaborare questo tipo di dati in base alla natura dei loro saggi. Suggerito per i dati attuali è il gating delle cellule di ogni condizione di trattamento in base ai dati di anticorpi ei valori giornalista GFP-CDK2 utilizzando lo script Perl fornito '2_gate_classifier.pl'. - Copiare il file script Perl fornito '2_gate_classifier.pl' nella stessa cartella del file di dati del 'Nuclei.csv' (Figura S2A). Fare doppio clic sull'icona per lo script Perl e, quando richiesto, digitare il nome completo del file di dati seguito da un '.csv' nome nome del file in cui le cellule devono essere recintato e infine i valori di gate per l'anticorpofluorescenza e dati giornalista GFP-CDK2.
NOTA: Come determinare principalmente impostazioni del gate e applicare questi per l'analisi dei dati sono discussi di seguito nella sezione Dati rappresentativi e figura 6 (per analizzare i dati forniti utilizzo '0.004' e '1.5', rispettivamente). Gli utenti Mac devono eseguire lo script Perl dalla riga di comando del terminale digitando: 'perl 2_gate_classifier.pl'. - Osservare il file appena creato, che unisce i valori grezzi singoli test cellule dai dati originali Profiler cellulare con etichette sub-popolazione che mostrano come ogni cellula da ogni pozzetto esegue contro entrambe le porte (Figura 6c).
- Tracciare i dati per ogni condizione sperimentale utilizzando le singole etichette sottopopolazione di cellule aprendo il software rstudio. Fare clic su 'File' e 'Apri File', quindi selezionare il file fornito 'analysis.r'. Osservare i comandi per tracciare figure 6B, 7 e 8 nella finestra superiore sinistra rstudio (Figura S2B). Nella finestra in alto a sinistra, tra i simboli doppi apici sulle linee 5 e 6, digitare l'indirizzo del computer della cartella contenente i dati gated. Includere la lettera di unità e il nome del file stesso, rispettivamente, (ad esempio, "C: / cartella di Analisi / output di analisi" e "nuclei_gated.csv").
NOTA: Se rstudio viene utilizzato per la prima volta su un determinato computer, il pacchetto grafico R 'ggplot2' dovrà essere installato prima. Questa è una volta solo passo per una nuova installazione di rstudio, dopo che questo passo diventa ridondante. Per installare 'ggplot2', fare clic sulla scheda denominata "pacchetti" sopra la finestra nell'angolo in basso a destra della rstudio, fare clic sul pulsante 'Install Packages "che appare sotto questo. Apparirà una nuova finestra. Tipo 'ggplot2' (omettendo quotazioni) in 'pacchetti' sritmo in questa nuova finestra e, infine, cliccare sul pulsante 'Install' per chiudere la finestra, installare le funzioni ggplot2 necessarie e tornare alla finestra rstudio principale per continuare dal passaggio 3.6. - Evidenziare le linee da 1 a 17 nella finestra in alto a sinistra di rstudio, quindi fare clic sul pulsante 'Run'. Ciò inserire i dati sperimentali, valori soglia e dettagli della posizione e in R (Figura S2C). R ora memorizzare temporaneamente i dati rilevanti per la stampa.
- Evidenziare singoli blocchi di codice rimanente sotto la linea 17 e creare le trame corrispondenti facendo clic sul pulsante 'Run' come prima. Osservare le trame nella finestra in basso a destra della rstudio e salvare il numero di formati facendo clic sul pulsante 'Export' (Figura S2D).
- Mentre la chiusura rstudio, fare clic su 'Non salvare' quando richiesto. Questo impedisce la confusione sul prossimo utilizzo di rstudio, che altrimenti contenere datidalla sessione precedente.
Risultati
L'esempio di immagini generate con il reverse-transfezione siRNA protocollo di screening sono stati preparati e analizzati con il software Profiler cellulare. La risultante dati grezzi numerica è tale che ogni cellula è rappresentato individualmente, riconducibile a sua immagine e ben di origine e misurato per diversi parametri di intensità di fluorescenza (Figura 6A). Per ogni cella individuata l'intensità di fluorescenza nucleare media per la RB1 anticorpi P-S780 e l'intensità del DNA integrato per le maschere del DNA nucleare dye-definiti sono determinati. Valori di intensità GFP media per nucleo e nel citoplasma regioni di ogni cella sono anche registrati consentendo il calcolo del nucleare rispetto a fluorescenza citoplasmatica del reporter GFP-CDK2. A valle di questi algoritmi di fluorescenza misure di intensità si fa uso di questi dati delle celle singole per definire porte per due saggi, colorazione anticorpo nucleare e GFP-CDK2 giornalista. Annotazione successiva delle celle the base del risultato del test e l'uso di queste etichette per consentire sottopopolazioni specifiche per essere ulteriormente caratterizzato da una terza misura (contenuto di DNA nucleare) è descritto.
Istogramma dei dati di intensità di fluorescenza grezzi raccolti per ogni test sono un modo efficace di valutare come sottopopolazioni di cellule si comportano in condizioni diverse. Gli istogrammi in figura 6B mostrano le distribuzioni di popolazione di dati delle singole celle di pozzi tripla per ogni condizione di atterramento RNAi. A sinistra sono i dati di intensità anticorpo nucleare e sulla destra sono i dati corrispondenti per il reporter GFP-CDK2. I dati anticorpi RB1 P-S780 rivela che esistono sostanzialmente le cellule in due popolazioni riguardo a questo modificazione post-traduzionale e che popolazioni di cellule con perdita di RB1 fosforilata su S780 possono essere distinti come un picco sinistro intensità nucleare che si arricchisce quando CDK6 viene abbattuto da siRNA. Questo stesso picco sinistrasi vede quando RB1 è di per sé l'obiettivo RNAi, che riflette la rimozione definitiva della proteina e quindi P-S780 RB1 colorazione. Al contrario, le stesse condizioni sperimentali per le stesse cellule, quando osservato attraverso il saggio giornalista GFP-CDK2, mostrano una dinamica differente nei dati delle singole celle. Una distribuzione continua si osserva, con un unico picco, ma siRNA che disturba il ciclo cellulare (siCDK6) e provoca accumulo nei risultati di fase G1 in un'estensione della spalla destra di tale distribuzione (cioè indicando una maggiore presenza di cellule che mostrano un aumento del rapporto GFP nucleare / citoplasma, rappresentato sullo asse X).
Anche mostrato gli istogrammi della figura 6B sono i valori di gate (barre verticali) che vengono scelti sulla base delle distribuzioni di entrambe le serie di dati del saggio. La regola utilizzata per i dati anticorpi RB1 P-S780 è quello di definire la posizione del cancello come mezza altezza: posizione di massima larghezza sulla spalla sinistra del principale(A destra) di picco quando si considerano i dati della cella di controllo negativi (non-targeting siRNA). I dati evidenziati in rosso sono cellule con ridotta e assente RB1 P-S780, che si identifica con questa porta. Un cancello simile posizionato sulla spalla opposta della distribuzione valore del rapporto è utilizzato per il reporter GFP-CDK2. I risultanti cellule sottopopolazione alta rapporto, che mancano o funzionalità ridotta attività CDK2, vengono visualizzati in verde. Per illustrare l'analisi multiplex di due saggi figura 6C mostra l'implementazione di entrambi i valori di gate utilizzando lo script perl 2_gate_classifier.pl per convertire i dati grezzi (Figura 6A) nel file annotato sotto. Questo nuovo file contiene i dati originali accanto una nuova colonna di etichette di classe per ogni cella ei due valori di gate utilizzati per distinguerli (in questo caso porte di 0,004 per i dati anticorpi e 1.5 per il reporter GFP-CDK2 sono stati utilizzati, rispettivamente) .
Dopo aver classificato le singole celle From ciascuna condizione atterramento sulla base dei due test è ora possibile utilizzare queste etichette di classe per aiutare l'annotazione dei grafici dei dati del test. Figura 7 mostra disperdono grafici dei dati delle celle individuali per il P-S780 RB1 e GFP saggi CDK2 Dall'esempio di dati per tutte e tre le condizioni RNAi. Numeri annotazione quadranti sui dispersione mostrano le percentuali relative di ciascuna sottopopolazione gated all'intera per quel contesto knockdown e si generano in R utilizzando le etichette di classe sopra descritti. Queste trame rivelano che, rispetto alle cellule trasfettate con non-targeting siRNA (Figura 7B), cellule trasfettate con siCDK6 rivelare una rete di distribuzione di dati sia spostata verso il basso sulla Y (che indica assenza di RB1 fosforilazione a serina 780) ea destra sulla asse X (che indica bassa attività CDK2, Figura 7C). Entrambi questi cambiamenti sono attesi per atterramento di questo obiettivo. In contrasto con questo, i dati di SirCellule trasfettate B1 (Figura 7A) mostra una perdita della colorazione anticorpale in linea con perdita dell'epitopo, ma poco effetto nella distribuzione dei dati per il reporter CDK2 rispetto ai controlli trasfettate con non-targeting siRNA, suggerendo nessun grande effetto sulla GFP giornalista -CDK2 nasce da RB1 atterramento.
Per esplorare ulteriormente l'uso dei dati di singole cellule, la classificazione sottopopolazione e test multiplexing Figura 8 mostra la rappresentazione della dispersione dei dati siCDK6 dei profili di istogramma Figura 7C accanto appaiati per intensità DNA integrato. Le coppie di istogrammi riguardano opposte metà di tutta la popolazione, diviso sulla base di una intensità anticorpo (destro della dispersione) o GFP-CDK2 valori del rapporto Reporter (sopra la dispersione). La quantificazione dell'intensità DNA nucleare per queste popolazioni mostra due picchi caratteristici di 2N e 4N contenuto di DNA come picchi sinistro e destro, rispettivamente. La intentions delle porte illustrate nelle figure 6, 7 e 8 sono tali che le cellule identificate partire per P-S780 RB1 (contrassegnato: P-S780-) o con un valore elevato rapporto dal reporter GFP-CDK2 (etichettati: G1) sarà essere in fase G1 del ciclo cellulare. In effetti, gli istogrammi profilo DNA per sottopopolazioni identificate con uno di questi saggi prevalentemente contengono cellule con contenuto di DNA 2N. Profili DNA della popolazione opposta gated (etichettati: P-S780 + o Non-G1) contiene cellule con le distribuzioni che vanno da 2N a 4N, in linea con tali cellule adottando una serie di ciclo cellulare posizioni fase post-G1.
Sebbene il fuoco qui è la generazione e l'analisi dei dati delle singole celle da immagini macchiate modo fluorescente, è anche utile essere in grado di prendere questi dati e riassumere ciascun saggio su base ben by-ben-per monitorare la variabilità tra le repliche e le prestazioni di tutti i pozzetti per un esperimento di un intero piatto di dati. Figura 9 mostra i dati di ogni trattamento siRNA riassunti come i valori medi di pozzi triplicato per la percentuale delle cellule all'interno dei cancelli applicati a) i dati RB1 P-S780 e b) i dati giornalista GFP-CDK2. I valori tracciati in A e B sono prodotti da due ulteriori script Perl forniti con questo manoscritto; 'Antibody_fluorescence_summary.pl' e 'G1assay_summary.pl', rispettivamente. Questi script utilizzano i dati grezzi creati da Cell Profiler (Nuclei.csv) e dati del rapporto per così come i) cellule totali misurate per pozzetto, ii) il numero di cellule all'interno del cancello, iii) le cellule per cento all'interno del cancello e iv) l'aritmetica media dei misurati, dati grezzi per bene. Questo è incluso come opzione adatta per guardare attraverso grandi insiemi di dati di analisi, prima di concentrarsi sui dati di trattamento individuali utilizzando la valutazione multiplex dei dati delle singole celle come illustrato in Figure 7 e 8. I grafici visualizzati qui plot 'iii) le cellule per cento entro il cancello' per entrambi i test, che si adattano alle distribuzioni di dati non normali visti per i dati P-S780 RB1 e GFP-CDK2 nei istogrammi in figura 6B. Questi script anche calcolare 'iv) la media aritmetica dei misurati, dati grezzi per quel bene', che sarebbe adatto analisi dei dati per le risposte popolazione omogenea e la distribuzione dei dati normale, prima e dopo la perturbazione sperimentale.
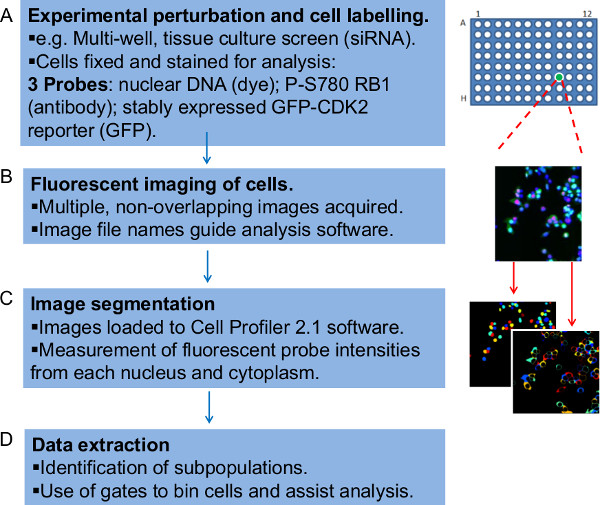
Figura 1:. Panoramica delle fasi del flusso di lavoro per analizzare quantitativamente i dati di immagine al microscopio fluorescente Il flusso di lavoro è qui rappresentato come quattro fasi (A) In primo luogo, è necessario preparare sperimentalmente cellule per l'imaging a fluorescenza.. L'esempio qui descritto è quella di unschermo in cui aderenti cellule tumorali umane siRNA trattati vengono coltivate per 48 ore, fissate e colorate su una piastra di coltura tissutale a 96 pozzetti. Sono presenti in triplice copia in pozzetti separati all'interno della piastra diverse condizioni RNAi. Le cellule sono colorate con un colorante DNA, un anticorpo specifico per RB1 fosforilata sul CDK4 e 6 selettivo sito di destinazione serina-780 (P-S780 RB1) e sono anche stabilmente esprimono un GFP-CDK2-giornalista, riportando l'uscita del ciclo cellulare G1. Collettivamente queste sonde fluorescenti costituiscono due saggi valutati separatamente all'interno del flusso di lavoro. (B) immagini al microscopio parallelo per ciascuna sonda fluorescente (canale) vengono generati e denominate tale da includere i dettagli con cui il software di analisi dell'immagine può organizzare i dati. (C) La file di immagine vengono caricati nel software Profiler Cell, che identifica algoritmicamente singole cellule e le coppie associati di nuclei e citoplasma prima di cedere misure di intensità per le tre sonde fluorescenti rilevanoed in ciascuno di essi. (D) Infine, uno script Perl viene utilizzato per organizzare i dati quantitativi prime prodotte. Questo passaggio si applica cancelli i dati di intensità di fluorescenza per ogni cella, in modo efficace binning le cellule in sottopopolazioni, che può essere inserito, rintracciato e interrogato. Clicca qui per vedere una versione più grande di questa figura.
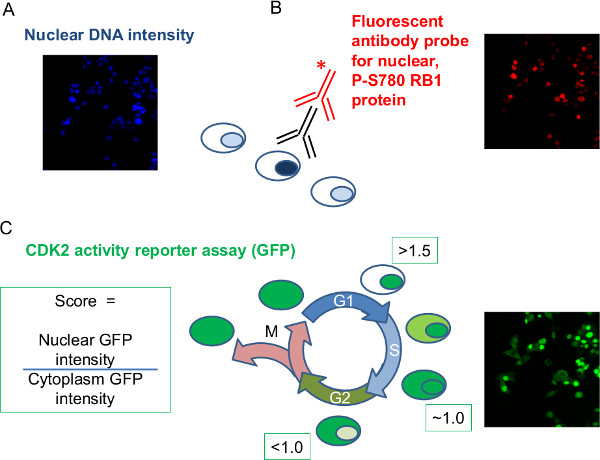
Figura 2: I dati sperimentali a essere ottenuta tramite analisi di immagine I, siRNA-trattati, cellule fluorescente fissate dai dati di esempio sono stati impostati ripreso e misure di intensità corrispondenti prese per cella.. Dati di immagine rappresentativi sono riportati per ciascun parametro registrata durante l'analisi dell'immagine intensità (A) DNA nucleare:. L'intensità della colorazione di colorante DNA nucleare viene utilizzato per produrre una misura del DNA per nucleo (B) Intensità nucleare di fosfo-RB1:.-Immuno colorazione specifica per P-S780 RB1 con un (nero) anticorpo primario e fluorescente contrassegnati anticorpo secondario (rosso) consentire una misurazione dell'intensità RB1 fosforilazione a . S780 per nucleo (C) GFP-CDK2 giornalista: Le cellule utilizzate stabilmente esprimere una proteina reporter GFP-tagged che trasloca tra il nucleo e il citoplasma in un modello di serie con il ciclo cellulare. Doppia misurazione dell'intensità GFP nucleare e citoplasmatica accoppiati per ogni cella permette di calcolare un rapporto per cellula che può essere utilizzata per distinguere fase G1 dal resto del ciclo cellulare. Tre obiettivi siRNA saranno utilizzati per illustrare l'analisi; un non-targeting siRNA controllo negativo; CDK6 siRNA come controllo positivo in perturbando RB1 fosforilazione e il progresso del ciclo cellulare; RB1 siRNA per stabilire specificità anticorpale.k "> Clicca qui per vedere una versione più grande di questa figura.

Figura 3: Organizzazione di file di immagine prima dell'analisi immagini Le immagini prelevate dalla piastra di coltura tissutale sono denominati sistematicamente per consentire al software di analisi di immagine per correlare i dati di immagine sul contesto sperimentale originale.. Questa informazione è collocato all'interno del nome del file per ogni immagine. (A) In ciascun pozzetto della piastra esperimento può corrispondere a diversi target RNAi o trattamenti, la parte forme di indirizzo e del nome del file. (B) Il numero di telaio è parte del nome del file . come ogni pozzetto è ripreso a raccogliere più, non si sovrappongono frames (C) sonde fluorescenti da ciascun frame vengono esposte separatamente; di conseguenza, i nomi dei file devono anche riflettere il canale ogni immagine si riferisce a. (D) i nomi dei file di esempio, in materia di bene (G12), Telaio (2) con l'immagine che rappresenta uno dei canali (blu, rosso, verde). Le linee tratteggiate collegano gli elementi filename alle rappresentazioni schematiche rilevanti per Beh, Frame e Channel, rispettivamente. Cliccate qui per vedere una versione più grande di questa figura.

Figura 4:. L'uso del cellulare Profiler per misurare il DNA nucleare e anticorpi colorazione Con le impostazioni nel file fornito gasdotto (3_channels_pipeline.cppipe), il profiler cellulare misure di software di analisi delle immagini di fluorescenza valori di intensità per il DNA nucleare e anticorpi vincolante relativo alle singole celle . (A) I nuclei sono identificati il 'blu' immagine del canale di DNA tinto. (B) < / Strong> Le posizioni dei DNA macchiato nuclei sono tenuti temporaneamente in un 'maschera Nuclei'. La maschera Nuclei viene quindi sovrapposto (C) le immagini blu e rosso di canale (DNA e anticorpi fluorescenza dati, rispettivamente) ed i valori di fluorescenza di segmenti di immagine che si sovrappongono con la maschera sono registrati contro ogni cella identificata. Individuazione di successo di nuclei vicini, separati può essere valutato visivamente l'aspetto della maschera Nuclei. Per l'illustrazione, mostrata cerchiato in questa immagine maschera, sono esempi in cui le impostazioni scelte per l'algoritmo hanno mis-identificati nuclei vicini come un unico nucleo. Regolazione delle impostazioni algoritmo per ridurre al minimo questi eventi viene introdotti nella sezione di discussione. Cliccate qui per vedere una versione più grande di questa figura.
/ftp_upload/51882/51882fig5highres.jpg "/>
Figura 5: Uso di Profiler cellulare per misurare le intensità GFP nucleari e citoplasmatici Il CDK2 giornalista GFP-tag trasloca tra il nucleo e il citoplasma in relazione alla posizione del ciclo cellulare delle cellule.. Allo stesso tempo, che Profiler cellulare calcola i DNA e anticorpi intensità nucleari per cella (Figura 4), calcola anche il nucleare rapporto citoplasma di intensità GFP per ogni cella. (A) I dati colorante DNA per ogni immagine viene utilizzato per generare una maschera Nuclei. Profiler (B) Cell utilizza la maschera Nuclei congiuntamente dell'immagine GFP dal reporter GFP-CDK2 per inizializzare la posizione di ogni cella con e poi si espande il perimetro di ogni cella per stimare l'intera impronta di ciascuna cella. Questo diventa un nuovo, 'maschera cellulare'. (C) La maschera nuclei viene sottratto dalla maschera cella di cedere una serie ciambella-come citoplasma contorni, che diventanola 'maschera citoplasma'. (D) La maschera e il citoplasma maschera nuclei sono utilizzati da Profiler cellulare per misurare coppie di valori GFP nucleari e citoplasmatici. Questi valori associati vengono quindi utilizzati per Profiler cellulare per calcolare indici, che informano per la posizione di ogni cella nel ciclo cellulare. Cliccate qui per vedere una versione più grande di questa figura.
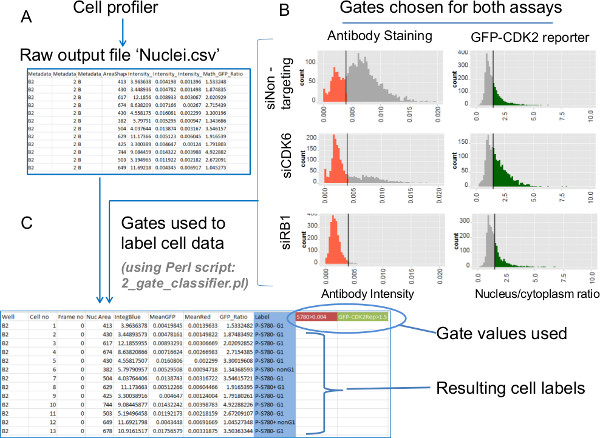
Figura 6:. L'estrazione dei dati - Elaborazione dati delle singole celle grezzi imponendo porte su valori di analisi delle tendenze biologici dai dati delle celle individuali per la colorazione di anticorpi e saggi giornalista GFP-CDK2 vengono estratti utilizzando dati gated. Istogrammi dei dati grezzi consentono l'identificazione dei valori cancello adatti. Questi sono poi imposti con uno script Perl. (A) Laprodotto finale di analizzare i file di immagini con le impostazioni fornite di Profiler cellule sono separate da virgole valore (.csv) file. Questi file c ontain dati delle celle individuali relativi a ciascuno dei diversi segmenti sub-cellulare. Il file 'Nuclei.csv' contiene tutte le misure selezionate relative all'uso della maschera Nuclei. Queste misure comprendono intensità anticorpo nucleare, intensità DNA nucleare e il rapporto di GFP (nucleo / citoplasma). (B) istogrammi dell'intensità anticorpo nucleare (a sinistra) e rapporti di giornalista GFP-CDK2 (destra) tracciata dai dati delle celle individuali per ogni condizione di atterramento siRNA . Le barre sugli istogrammi visualizzati mostrano le posizioni del cancello desiderati per questi test. Dati colorate su istogrammi indicano le sottopopolazioni gated. (C) Le porte per i due saggi illustrati in B vengono applicati ai dati grezzi usando lo script Perl '2_gate_classifier.pl'. Lo scriptcrea una copia modificata del file di output Profiler cellulare originale (Nuclei.csv) assistere successiva tracciato. I due valori cancello vengono registrati nel nuovo file (evidenziato in colore qui) e viene aggiunta una nuova colonna 'Label'. La etichette bin ciascuna cella in una delle quattro possibili sottogruppi basati sui due valori di analisi gated per ogni cella. Queste etichette sono utilizzati in successivi lotti che presentano calcoli dei contributi di ogni sottopopolazione, nonché il controllo incrociato dei parametri aggiuntivi generati in Profiler cellulare. Clicca qui per vedere una versione più grande di questa figura.
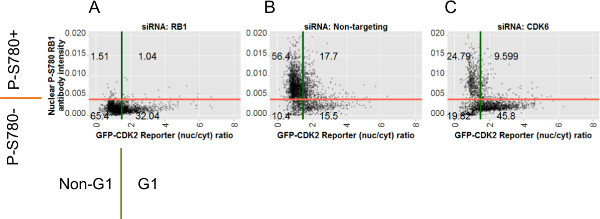
Figura 7:. Diagrammi a dispersione per ogni condizione di siRNA raffigurante i dati grezzi per le singole cellule e le posizioni del cancello Scatter appezzamenti di individati doppi cellulari da tutte le immagini per le condizioni siRNA indicati: (A) siRB1; (B) controllo negativo-Sinon mira; (C) siCDK6. Tramato contro le assi Y sono valori di fluorescenza nucleare da anti-P-S780 RB1 colorazione. Tramato contro le assi X sono i valori del rapporto corrispondenti calcolati dal giornalista GFP-CDK2. Le barre rosse e verdi indicano le posizioni delle porte per il cancello RB1 P-S780 e le porte del reporter GFP-CDK2, rispettivamente. Le due porte dividono le cellule in quattro sottopopolazioni ei numeri sui quadranti risultanti sono il numero percentuale di cellule da ciascuna di esse. Annotazioni intorno agli assi per A indicano le quattro possibili etichette elementi applicati a ciascuna cella dal 2_gate_classifier.pl script Perl. Queste etichette sono mostrati in relazione al rispettivo cancello dosaggio e sono utilizzati in R-script (analysis.r) per generare le trame in figure 6, 7 e 8. Cliccate qui per vedere una versione più grande di questa figura.
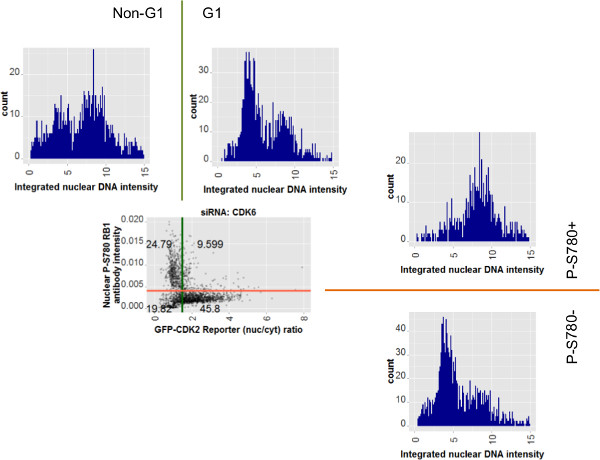
Figura 8: Cellulari sottopopolazioni definite dai due saggi di transito G1 mostrano 2N e 4N DNA profili in linea con il risultato del test La rappresentazione della dispersione dei dati per le celle siCDK6 si ripete dalla figura 7C.. Intorno della dispersione sono istogrammi per intensità DNA nucleare integrata di sottogruppi della popolazione. Quelli sopra la dispersione riguardano il saggio giornalista GFP-CDK2. Quelli a destra della dispersione riguardano nucleare fosfo-RB1 solo misure di anticorpi. Le linee di gate colorati vengono estesi per mostrare il loro rapporto con gli istogrammi. Sono indicate anche le etichette porta per la quale sono stati selezionati i dati della cella per questi complotti supplementari. Le cellule con perdita di RB1 fosforilati su serina 780 (P-S780-) o quelli con un alto GFP-CDK2 giornalista nucleare rapporto citoplasmatica (indicanti un'attività basso CDK2) mostrano profili DNA prevalentemente 2N-like, mentre i loro omologhi opposte per ogni rispettiva analisi mostrano una distribuzione di 2N e 4N, caratteristica di un post-G1 popolazione fase mista di cellule. Cliccate qui per vedere una versione più grande di questa figura.
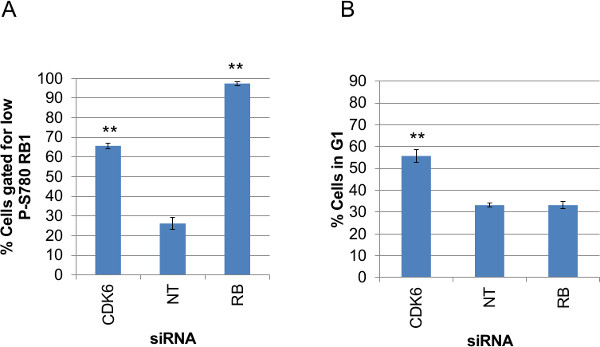
Figura 9: Riepilogo appezzamenti di valori di analisi gated per ogni siRNA condizione trame dati di sintesi del gated (A) P-S780 dati RB1 e dati (B) GFP-CDK2 da pozzi tripla per ogni condizione di knockdown siRNA.. I valori sono stati calcolati dalla uscita Profiler cellulare grezzo (Nuclei.csv) con ilScript Perl, 'antibody_fluorescence_summary.pl' (A) o 'G1assay_summary.pl' (B). I valori tracciati sono mezzi per la percentuale delle cellule all'interno del cancello applicato a ciascun test. Bar indicano errori standard calcolati da pozzi triplice copia. Spaiati, homoscedastic valori T-Test P per ogni condizione di atterramento rispetto ai non-targeting siRNA sono riportati sopra i dati tracciati dove P <0.001 (**) e P <0,05 (*). Cliccate qui per visualizzare una versione più grande di questo figura.
Figura S1. Installazione di software Profiler cellulare per l'analisi delle immagini. (A) Schermata di Profiler cellulare prima di qualsiasi impostazione di analisi delle immagini sono inseriti. (B) Schermata di Profiler cellulare dopo i particolari algoritmi contenuti in '3_channels_pipeline.cppipe' sono stati caricati. L'alto scheda illuminato in alto a sinistra indica che questa schermata mostra i parametri per la fase 'LoadImages' dell'analisi. Cliccando sulle altre parti della lista di sotto di questo rivelerà i dettagli per le fasi successive di analisi. (C) Schermata di Profiler cellulare con dettagli per cartella di input e la cartella di output è entrato. (D) Schermata di Profiler cellulare dopo il 'Analyze Pulsante immagini "è stato cliccato per iniziare l'analisi. Sovrapposto sono tre nuove finestre che illustrano le maschere algoritmicamente prodotte generati dal software dalle immagini sotto analisi. Queste finestre sono accessibili facendo clic sulle icone 'occhio' alla posizione aperta accanto ai passi importanti nell'analisi, nell'angolo in alto a sinistra della finestra principale Profiler cellulare. Questi punti di vista aiutano l'utente a verificare se le impostazioni che generano le maschere coriandoli color d'accordo con gli originali, in scala di grigi, i dati di accompagnamento.
ent "> Figura S2. L'uso di Perl e rstudio al cancello dati delle celle individuali e tracciare le sottopopolazioni di cellule risultanti. (A) Il pannello di destra mostra la cartella scelto di ricevere i file di output .csv (icone verdi) dall'analisi Profiler cella. Gli script Perl forniti con il manoscritto (icone blu) vengono copiati in questa cartella. Evidenziato è lo script Perl '2_gate_classifier.pl', che è stato fatto doppio clic con il mouse per produrre la finestra di dialogo nel pannello di sinistra. Sono indicate le prompt e corrispondenti digitato le risposte necessarie per cancello i dati delle celle singole dal file 'Nuclei.csv'. (B) Schermata di rstudio subito dopo aver caricato lo script 'analysis.R'. Evidenziati sono i comandi per caricare i dati gated da A in il software prima del tracciato (i dettagli della nota nelle linee 5 e 6 dovrà essere regolato in base alla quale i dati gated si trova sul calcolor utilizzato per l'analisi). (C) Schermata di rstudio una volta i dati sono stati caricati. (D) Schermata di rstudio mostra ha evidenziato il blocco di codice necessaria per produrre il grafico mostrato nella finestra in basso a destra. Codici per ciascun lotto sono separati da righe vuote e raggruppati per tipo di trama.| bersaglio siRNA | Indirizzi pozzetti |
| Non-targeting (NT) | E5, F5, G5 |
| Retinoblastoma (RB) | E7, F7, G7 |
| Ciclina chinasi dipendente 6 (CDK6) | B2, C2, D2 |
Tabella 1: Well indirizzi e corrispondenti condizioni siRNA utilizzato nell'esempio set di dati.
Discussione
Il flusso di lavoro descritto costituisce una procedura di perturbazione multipozzetto di cellule utilizzando siRNA, successiva rivelazione marcatore e infine uso di una serie di procedure del software supportato per facilitare l'estrazione dei dati quantitativi risultanti dalle immagini al microscopio a fluorescenza. L'approccio è focalizzato sulla consegna di valori di intensità nucleari e citoplasmatici per singole celle, che ha ampia applicazione pratica in molte applicazioni basati su celle. I dati di esempio utilizzati qui è stato generato in un ambiente schermo siRNA in cui due saggi fluorescenti per il transito ciclo cellulare fase G1 sono testati e correlati tornare a una misura più diretta biofisica del contenuto di DNA nucleare.
L'uso di un colorante fluorescente per DNA immagine DNA nucleare è un passo indispensabile nel processo di segmentazione dell'immagine in quanto consente l'identificazione delle singole cellule e la conseguente 'maschera Nuclei' serve come punto di partenza per identificare corrispondente cytoplasmiregioni c. Il CDK2 giornalista GFP-tag, che è stabilmente espressa nelle cellule, dà una variabile ancora costantemente superiore segnale di fondo nel citoplasma con cui questo vano può essere delineata. La stessa analisi condotta dovrebbe essere applicabile all'analisi degli eventi proteine traslocazione con altri reporter fluorescenza legati adatti e la loro risposta alla perturbazione. Inoltre, sostituendo il reporter GFP-CDK2 con coloranti fluorescenti specifici citoplasma consentirebbe l'uso alternativo di questo algoritmo per misurare le dimensioni del citoplasma e le dimensioni relative delle cellule nelle immagini.
Un'altra considerazione di progetto nella strategia di segmentazione dell'immagine qui descritto è l'uso di Profiler cellulare per fornire valori di intensità integrati per la quantificazione del DNA. Integrazione dell'intensità valori per il DNA nucleare dati colorazione permettono di possibili variazioni nelle dimensioni nucleo, e rappresenta un fiammifero vicino per i profili di quantificazione vistoper propidio ioduro macchiato FACS dati. Tuttavia, l'intensità integrata non può fornire uno strumento adeguato per valutare la funzione della proteina dove la concentrazione media, esemplificato da intensità media di antigene di fluorescenza, è biologicamente più rilevante rispetto alla quantità totale integrata di proteine (e fluorescenza associata) all'interno di un compartimento cellulare. Quindi significa valori di intensità sono stati utilizzati per la P-S780 RB1 e dati GFP. L'opzione per alterare tra le due modalità (media o integrato) di valutazione dei dati si trova sul pannello 'ExportToSpreadsheet' del software Profiler cella.
Le impostazioni di analisi nel file 3_channels_pipeline.cppipe sono ottimizzati per le immagini del set di dati di esempio. Analisi di nuove immagini set con questo protocollo sarà necessario che i nomi dei file adottano la convenzione di denominazione sopra descritto (Figura 3). Inoltre, la sensibilità valori in base alla luminosità della colorazione del DNA nucleare e le soglie per lo sfondointensità dei nuovi set di immagini potrebbe essere necessario regolare all'interno delle impostazioni Profiler cellulare. Dato il ruolo chiave della colorazione DNA contiene per costruire le varie maschere di segmentazione dell'immagine, l'applicazione di impostazioni di sensibilità corrette per questo canale è essenziale all'analisi successo di nuovi dati di immagine con il software Profiler cellulare. Il file di impostazioni Profiler cellulare fornito (3_channels_pipeline.cppipe) contiene note su dei parametri più utili per adattare l'analisi di nuovi dati. Queste note sono nella casella di testo nella parte superiore dello schermo nella finestra principale Profiler cellulare e includono una guida su come modificare le impostazioni di sensibilità e regolazione del numero di canali da analizzare. Come incriminato nella sezione protocollo 2.8, per testare le impostazioni per i nuovi dati di immagine può essere necessario per osservare la segmentazione delle immagini durante l'analisi delle immagini cliccando aprire le icone 'occhio' per ciascuno dei 'Identificare ... Objects fasi di protocollo (Figura S1D ). In particolare, la visualizzazione dei dati di immagine attraverso 'IdentifyPrimaryOjects' mostrerà se la maschera Nuclei è identificato correttamente dalle immagini della colorazione DNA. Nella pagina software Profiler cellulare per il modulo "IdentifyPrimaryOjects 'è il fattore di correzione soglia. Tentativi ed errori regolazione di questo valore sarà risolvere la maggior parte degli errori di riconoscimento nucleari. I valori di equilibrio del canale DNA contro l'intensità di sfondo per ogni immagine. Valori del fattore di correzione Soglia cerniera circa 1, dove più grande di questo è più spinto (buono per le immagini chiare) e meno di 1 è indulgente (adatti per le immagini con meno contrasto tra la colorazione e lo sfondo).
L'uscita dei dati grezzi singola cella da Profiler cellulare può essere analizzata in vari modi per soddisfare le esigenze di altri studi. Qui è illustrato l'uso di uno script Perl per applicare porte a due dei parametri misurati per cella al fine di assistere l'estrazione tendenze biologiche dalla un datod permesso incrociato delle sottopopolazioni individuate con ulteriori misurazioni. Anche se è ugualmente possibile includere elementi di gating nell'ambito della Profiler Cell, il percorso alternativo usato qui fornisce una maggiore flessibilità e velocità, in particolare se grandi insiemi di dati devono essere valutate. La fase più lenta nelle fasi di acquisizione post-immagine del protocollo attuale è la gestione del software Profiler cella. Profiler cellulare qui viene eseguito senza imporre porte per produrre un insieme di dati grezzi non-gated che può essere rianalizzato con conseguente script Perl più rapidamente e, se necessario, iterativamente con diversi valori di gate. Non tutti gli studi conoscere in anticipo i valori di gate adatti come questo può variare con reagenti in un dato insieme di dati, e potenzialmente nel tempo. E ', quindi, consigliabile per generare istogrammi raffiguranti la distribuzione dei dati grezzi ottenuti da Profiler cellulare per i controlli positivi e le cellule mock-perturbate al fine di individuare idonei valu cancelloes per i parametri di interesse.
Sono scritti Gli script Perl ad accettare una struttura della colonna rigidamente definito di dati da Profiler cellulare e può smettere di funzionare se un utente modifica il numero di uscita parametri Profiler cellulare utilizzando le impostazioni della 'ExportToSpreadsheet'. Per contribuire ad attuare la modifica delle impostazioni note sono inclusi all'interno dei file di script Perl. Per vedere questi visualizzare lo script in un editor di testo, preferibilmente editor di testo per programmatori impostata codice colore elementi Perl (ad esempio, http://www.activestate.com/komodo-edit). Queste note indicano dove per regolare lo script di adattarsi ai cambiamenti nel formato dei dati. Simile agli script Perl, il file R-codice fornito (analysis.r), che contiene le istruzioni per tracciare i dati dai dati di analisi di immagine, può essere letto in un editor di testo o software rstudio vedere note aggiuntive sull'uso e l'adattamento. Queste note possono essere completato con i dettagli sulle espressioni regolari e Perl 12 e il pacchetto ggplot2 13 per R, entrambi i quali formano la base per come i dati vengono letti, annotato e tracciati rispettivamente.
Nuovi studi usando la microscopia a fluorescenza, nonché dati grezzi depositati con le pubblicazioni open source sono suscettibili di metodi di analisi, come quelle qui descritte. La natura stessa dei dati ad alta contenuto si presta ad analisi ricorsiva con accentuazioni diverse analitiche a seconda degli interessi di ricerca di ogni osservatore. Anche se le domande che possono essere poste dei dati sono limitati dalle sonde utilizzate originariamente, i dati delle immagini possono essere spesso significato rianalizzati oltre l'ambito degli studi che le hanno generate.
Divulgazioni
Gli autori non hanno nulla da rivelare
Riconoscimenti
Questo lavoro è stato sostenuto da sovvenzioni CRUK 15043 e 14251 CRUK.
Ringraziamo Daniel Wetterskog e Ka Ho Kei per l'assistenza tecnica e la lettura critica del manoscritto.
Materiali
| Name | Company | Catalog Number | Comments |
| AllStars negative control siRNA | Qiagen | 1027280 | Negative control siRNA. |
| CDK6 siRNA | Dharmacon/ Custom synthesis | NA | Antisense sequence: 5' CUCUAGGCCAGUCUUCUUCUU |
| RB1 siRNA | Dharmacon/ Custom synthesis | NA | Antisense sequence: 5' GGUUCAACUACGCGUGUAATT |
| 5x siRNA buffer | Thermo Scientific | B-002000-UB-100 | To be diluted with nuclease-free, non-DEPC treated water. |
| Nulcease-free water (non-DEPC) | Applied Biosystems | AM9937 | For dilution of siRNA buffer. |
| Hiperfect | Qiagen | 301705 | Transfection lipid. |
| DMEM | Life Technologies | 41966052 | Pyruvate and high-glucose supplemented tissue culture media. |
| 96 well tissue culture plate | Falcon | 3072 | Plain, 96 well tissue culture plate for the parallel, compartmentalized mixing of transfection complexes. |
| Packard Viewplate | Perkin Elmer LAS | 6005182 | 96 well TC plate with optical base on which cells are transfected, grown, fixed and eventually imaged. Supplied with opaque, adhesive plate seals, which are used during storage of used plates. |
| Breathable membrane | Alpha Labs | LW2783 | Sterile, gas-permeable, adhesive membrane. |
| Neutral buffered formalin, 10% | Sigma-Aldrich | HT5012-1CS | 10% Formalin (4% formaldehyde) fixative used neat in protocol. |
| Triton X-100 | Sigma-Aldrich | X100PC | Non-ionic detergent |
| [header] | |||
| Tris | Sigma-Aldrich | T1503 | Trizma base (tris(hydroxymethyl)aminomethane) |
| Tween 20 | Sigma-Aldrich | P2287 | For plate wash solution. |
| Anti-P-S780 RB1 antibody | Abcam | ab32513 | Rabbit monoclonal |
| AlexaFluor647 Anti-rabbit | Invitrogen | A21245 | Highly cross-adsorbed, fluorescently labelled secondary antibody. |
| Hoechst 33342 (Bisbenzimide) | Sigma-Aldrich | B2261 | Fluorescent, chromatin-intercalating, DNA dye. |
| Permeabilization solution | NA | NA | 0.1% Triton X-100 in 50 mM Tris-buffered saline, pH 8.0. |
| Plate wash solution | NA | NA | Tris-buffered saline containing 0.1% Tween-20. |
| Block solution | NA | NA | 5% powdered milk in Tris-buffered saline and 0.1% Tween-20. For blocking plate prior to immuno-staining and dilution of antibodies. |
| Cell Profiler 2.1 | Broad Institute | http://www.cellprofiler.org/download.shtml | |
| Active Perl Community Edition | ActiveState | http://www.activestate.com/activeperl/downloads | |
| R programming environment | The R Foundation | http://www.r-project.org | |
| Rstudio | Rstudio | http://www.rstudio.com/ | |
Riferimenti
- Carpenter, A. E., et al. CellProfiler: image analysis software for identifying and quantifying cell phenotypes. Genome Biol. 7 (10), 100 (2006).
- Khan, A., Eldaly, H., Rajpoot, N. A gamma-gaussian mixture model for detection of mitotic cells in breast cancer histopathology images. J Pathol Inform. 4, 11 (2013).
- Selzer, P., Beibel, M., Gubler, H., Parker, C. N., Gabriel, D. Comparison of multivariate data analysis strategies for high-content screening. J Biomol Screen. 16 (3), 338-347 (2011).
- Lyman, S. K., et al. High content, high-throughput analysis of cell cycle perturbations induced by the HSP90 inhibitor XL888. PLoS One. 6 (3), e17692 (2011).
- Richardson, E., Stockwell, S. R., Li, H., Aherne, W., Cuomo, M. E., Mittnacht, S. Mechanism-based screen establishes signalling framework for DNA damage-associated G1 checkpoint response. PLoS One. 7 (2), e17692 (2012).
- Heynen-Genel, S., Pache, L., Chanda, S. K., Rosen, J. Functional genomic and high-content screening for target discovery and deconvolution. Expert Opin Drug Discov. 7 (10), 955-968 (2012).
- Krausz, E. High-content siRNA screening. Mol Biosyst. 3 (4), 232-240 (2007).
- Gu, J., Xia, X., et al. Cell Cycle-dependent Regulation of a Human DNA Helicase That Localizes in. DNA Damage Foci. Mol Biol Cell. 15 (7), 3320-3332 (2004).
- Mittnacht, S. Control of pRB phosphorylation. Curr Opin Genet Dev. 8 (1), 21-27 (1998).
- Mittnacht, S. The retinoblastoma protein--from bench to bedside. Eur J Cell Biol. 84 (2-3), 97-107 (2005).
- Nybo, K. GFP imaging in fixed cells. BioTechniques. 52 (6), 359-360 (2012).
- Schwartz, R. L., Foy, B. D., Phoenix, T. . Learning Perl - Making Easy Things Easy and Hard Things Possible. , 1-363 (2011).
- Wickham, H. . ggplot2: Elegant Graphics for Data Analysis. , 978-970 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon