È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Da Veloce imaging di fluorescenza di Molecular legge Diffusione su membrane cellulare dal vivo in un microscopio commerciale
In questo articolo
Riepilogo
Spatial distribution and temporal dynamics of plasma membrane proteins and lipids is a hot topic in biology. Here this issue is addressed by a spatio-temporal image fluctuation analysis that provides conceptually the same physical quantities of single particle tracking, but it uses small molecular labels and standard microscopy setups.
Abstract
È diventato sempre più evidente che la distribuzione spaziale e il moto dei componenti della membrana, come i lipidi e le proteine sono fattori chiave nella regolazione di molte funzioni cellulari. Tuttavia, a causa della dinamica veloci e le minuscole strutture coinvolte, risoluzione spazio-temporale molto elevata è necessaria per catturare il reale comportamento delle molecole. Qui vi presentiamo il protocollo sperimentale per lo studio della dinamica delle proteine e dei lipidi plasmatici-membrana fluorescenza marcata nelle cellule vive con alta risoluzione spazio-temporale. In particolare, questo approccio non ha bisogno di tenere traccia di ogni molecola, ma calcola comportamento popolazione utilizzando tutte le molecole in una regione della membrana. Il punto di partenza è una immagine veloce di una regione determinata sulla membrana. Successivamente, una completa funzione di autocorrelazione spazio-temporale è calcolato correlando immagini acquisite ad aumentare i ritardi, per esempio ogni 2, 3, n ripetizioni. E 'possibile dimostrare che la larghezzadel picco dei spaziali aumenti funzione di autocorrelazione al crescente temporizzazione in funzione del movimento delle particelle a causa della diffusione. Pertanto, il montaggio della serie di funzioni di autocorrelazione consente di estrarre la proteina effettiva media cilindrata piazza da immagini (IMSD), qui presentato in forma di apparente diffusività vs spostamento medio. Questo produce una visione quantitativa della dinamica medi delle singole molecole con precisione nanometrica. Utilizzando una variante GFP-tagged della transferrina recettore (TFR) e un ATTO488 contrassegnata con 1-palmitoil-2-idrossi-sn -glycero-3-fosfoetanolamina (PPE) è possibile osservare la regolazione spazio-temporale di proteine e lipidi diffusione su micron-sized regioni membrana nell'intervallo di tempo di micro-to-milli-secondi.
Introduzione
Partendo dal modello originale "mosaico fluido" di Singer e Nicolson, l'immagine di membrana plasmatica cellulare è stato costantemente aggiornato nel corso degli ultimi decenni, al fine di comprendere il ruolo emergente del citoscheletro e lipidi domini 1,2.
Le prime osservazioni sono state ottenute da recupero fluorescenza dopo photobleaching (FRAP) svelamento che una frazione significativa di proteine di membrana è immobile 3-5. Questi studi pionieristici, anche se molto informativo, sofferto dalla relativamente scarsa risoluzione spaziale (micron) e di tempo (secondi) di configurazioni FRAP. Inoltre, essendo una misura media insieme, FRAP manca di dare informazioni sul comportamento singola molecola.
In questo contesto, la possibilità di etichettare specificamente una singola molecola di etichetta molto luminosi (permettono lo studio del processo di diffusione una molecola alla volta) ha avuto molto successo. In particolare, spingendo l'risoluzione temporale dell'approccio singola particella di monitoraggio (SPT) per i tempi microsecondi, Kusumi, et al. accesso maturata alle funzioni sconosciute della dinamica dei lipidi e delle proteine che grandemente contribuito al riconoscimento del ruolo di base di actina-membrana scheletro in membrana fisiologia 6 , 7. Questi risultati hanno generato il così chiamato il 'picchetto recinto e' il modello, in cui lipidi e proteine diffusione è regolata da scheletro a base actina-. Tuttavia, al fine di avere accesso alla enorme quantità di informazioni fornite da SPT molte questioni sperimentali devono essere affrontate. In particolare, la procedura di etichettatura è tipicamente composta da molte fasi come la produzione, la purificazione e l'introduzione di specie marcate nel sistema. Inoltre, le grandi etichette, come punti quantici o nanoparticelle metalliche, sono spesso necessari per raggiungere la scala cronologica inferiore al millisecondo e la reticolazione delle molecole bersaglio per l'etichetta non poteva essere evitato in molti casi. Infine, molte traiettoriedevono essere registrati per adattarsi criteri statistici e contemporaneamente è richiesta una bassa densità dell'etichetta per consentire il monitoraggio.
Rispetto a SPT, spettroscopia di fluorescenza di correlazione (FCS), superando molti di questi inconvenienti, rappresenta un approccio molto promettente per studiare la dinamica molecolare. In realtà, FCS funziona bene anche con le etichette dim e densi, consentendo di studiare la dinamica di molecole proteiche-tag fluorescenti nelle cellule trasfettate. Inoltre, permette di raggiungere elevate statistiche in un periodo di tempo limitato. Infine, nonostante la densità "alta" delle etichette FCS fornisce informazioni di singole molecole. Grazie a tutte queste proprietà, FCS rappresenta un approccio molto semplice ed è stato ampiamente applicato a studiare la dinamica dei lipidi e proteine sia in membrane modello e in vivo cellule 8-10. Molti approcci diversi sono stati proposti per aumentare la capacità di FCS per rivelare i dettagli di diffusione molecolare. Per esempio, è stato shproprio che eseguendo FCS sulle aree di osservazione di diverse dimensioni si può definire un "diritto FCS diffusione" caratteristiche illuminanti nascosti del moto molecolare 11,12. Oltre ad essere variato in termini di dimensioni, l'area focale è stato ripetuto anche il 13, si trasferisce in uno spazio lungo le linee 14-20 o coniugati con telecamere veloci 21,22. L'utilizzo di questi correlazione 'spazio-temporale' approcci, parametri biologici rilevanti dei diversi componenti di membrana sono stati quantitativamente descritti su entrambe le membrane modello e quelle biologiche attuali, quindi visione cedimento in membrana organizzazione spaziale.
Tuttavia, in tutti gli FRAP e applicazioni FCS descritto finora la dimensione della zona focale rappresenta un limite di risoluzione spaziale che non può essere superata. Diversi metodi di imaging super-risoluzione sono state recentemente sviluppate per aggirare questo limite. Alcuni sono basati sulla precisione di localizzazione, come ad esempio stocastico la microscopia ottica ricostruzione (STORM) 23,24, la microscopia localizzazione fotoattivazione (PALM) 25, fluorescenza PALM (FPALM) 26, e single-particelle di tracking PALM (sptPALM) 27: relativamente grande quantità di fotoni necessari ad ogni snapshot, tuttavia, limita la risoluzione temporale di questi metodi per almeno diversi millisecondi, ostacolando così la loro applicabilità in vivo.
Al contrario, una promettente alternativa per Super Resolution Imaging sono stati aperti dal spazialmente modulando l'emissione di fluorescenza con i metodi di deplezione emissione stimolata (STED o reversibili saturabili transizioni ottiche di fluorescenza (RESOLFT)) 28,29. Questi approcci combinano la sagomatura del volume osservazione ben al di sotto del limite di diffrazione con la possibilità di utilizzare microscopi a scansione veloce e sistemi di rilevazione. In combinazione con l'analisi di fluorescenza fluttuazione, microscopia STED permesso di sondare direttamente le dinamiche spazio-temporali nanoscala di lipidi e proteins in membrane cellulari dal vivo 30,31.
Le stesse grandezze fisiche di microscopia basata STED possono essere ottenuti da un spettroscopia immagine correlazione spazio-temporale modificato (STICS 32,33) metodo che è adatto per lo studio della dinamica delle proteine fluorescenti-taggato membrana e / o lipidi in cellule vive e da un microscopio commerciale. Il protocollo sperimentale qui presentata è composta da pochi passi. Il primo richiede l'imaging veloce della regione di interesse sulla membrana. Poi, la pila risultante di immagini viene utilizzato per calcolare le funzioni di correlazione medi spazio-temporali. Con il montaggio della serie di funzioni di correlazione, la 'legge di diffusione' molecolare può essere ottenuto direttamente dalle immagini sotto forma di una diffusività apparente (D app) - vs medi di equipaggio grafico di spostamento. Questa trama dipende in modo critico l'ambiente esplorato dalle molecole e permette di riconoscere direttamente le modalità di diffusione attualidel lipide / proteina di interesse.
In più particolari, come precedentemente indicato 34, la funzione di autocorrelazione spazio-temporale della serie dell'immagine acquisita dipende in modo critico dalla dinamica delle molecole si muovono nella serie di immagini raccolte (si ricorda che lo stesso ragionamento può essere applicato in una linea di acquisizione dove solo una dimensione nello spazio è considerato). In particolare, si definisce la funzione di correlazione come:
 (1)
(1)
dove  rappresenta l'intensità di fluorescenza misurata nella posizione x, y, al tempo t,
rappresenta l'intensità di fluorescenza misurata nella posizione x, y, al tempo t, ![]() e
e ![]() rappresenta la distanza in x eindicazioni y rispettivamente,
rappresenta la distanza in x eindicazioni y rispettivamente, ![]() rappresenta l'intervallo di tempo, e
rappresenta l'intervallo di tempo, e ![]() rappresenta la media. Questa funzione può essere espressa come:
rappresenta la media. Questa funzione può essere espressa come:
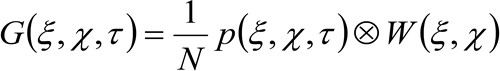 (2)
(2)
dove 'N' rappresenta il numero medio di molecole nella zona di osservazione, ![]() rappresenta l'operazione di convoluzione nello spazio, e
rappresenta l'operazione di convoluzione nello spazio, e 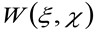 rappresenta l'autocorrelazione della vita strumentale. Quest'ultimo può essere interpretato come una misura di quanto i fotoni di un singolo emettitore sono sparsi nello spazio a causa della configurazione ottica / registrazione (la cosiddetta Point Spread Function, PSF, geneRALLY ben approssimata da una funzione gaussiana). Infine,
rappresenta l'autocorrelazione della vita strumentale. Quest'ultimo può essere interpretato come una misura di quanto i fotoni di un singolo emettitore sono sparsi nello spazio a causa della configurazione ottica / registrazione (la cosiddetta Point Spread Function, PSF, geneRALLY ben approssimata da una funzione gaussiana). Infine, 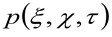 rappresenta la probabilità di trovare una particella a distanza
rappresenta la probabilità di trovare una particella a distanza ![]() e
e ![]() dopo un ritardo
dopo un ritardo ![]() . Se consideriamo una dinamica diffusiva, in cui le particelle si muovono in modo casuale in tutte le direzioni e flussi netti non sono presenti, questa funzione è anche ben approssimata da una funzione gaussiana dove la varianza può essere identificato come il Mean Square Displacement (MSD) della particella in movimento . Così, la vita della funzione di correlazione (indicato anche come
. Se consideriamo una dinamica diffusiva, in cui le particelle si muovono in modo casuale in tutte le direzioni e flussi netti non sono presenti, questa funzione è anche ben approssimata da una funzione gaussiana dove la varianza può essere identificato come il Mean Square Displacement (MSD) della particella in movimento . Così, la vita della funzione di correlazione (indicato anche come ![]() ), Può essere definito come la somma dei DMS particelle e la vita strumentale e può essere misurata con una misura gaussianazione della funzione di correlazione per ogni ritardo. La misurato i MSD può essere usato per calcolare un diffusività apparente delle molecole in movimento
), Può essere definito come la somma dei DMS particelle e la vita strumentale e può essere misurata con una misura gaussianazione della funzione di correlazione per ogni ritardo. La misurato i MSD può essere usato per calcolare un diffusività apparente delle molecole in movimento ![]() e uno spostamento medio
e uno spostamento medio ![]() come:
come:
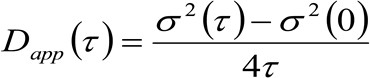 (3)
(3)
 (4)
(4)
Poche considerazioni sul setup sperimentale utilizzato può guidare il lettore attraverso i seguenti sezioni. Al fine di eccitare selettivamente i fluorofori sulla membrana basale delle cellule useremo una riflessione interna (TIR) illuminazione totale vivente, utilizzando una fluorescenza TIR commerciale (TIRF) microscopio (i dettagli si possono trovare nella sezione materiali). Inoltre, al fine di raccogliere the fluorescenza useremo un obiettivo ad alto ingrandimento (100X NA 1,47, alta apertura numerica è necessaria per l'illuminazione TIRF) e una fotocamera EMCCD (dimensione fisica del pixel sul chip 16 micron). Per raggiungere una dimensione di pixel di 100 nm applichiamo una lente di ingrandimento supplementare di 1.6X. Come discusso in seguito, una risoluzione temporale inferiore a 1 msec sarebbe necessario per descrivere correttamente le dinamiche di lipidi di membrana veloci sotto i 100 nm. Per raggiungere questa risoluzione temporale dobbiamo selezionare una regione di interesse (ROI) più piccolo dell'intero circuito integrato della fotocamera (512 x 512). In questo modo, la fotocamera leggerà un numero ridotto di linee aumentando la risoluzione temporale. Tuttavia, in questo regime lettura il lasso di tempo sarebbe limitata dal tempo necessario per spostare le cariche dalla esposizione al chip di lettura sulla fotocamera e di solito dell'ordine di millisecondi per 512 x 512 pixel EMCCD. Per battere questo limite, una tecnologia emergente consente di spostare le ROI linee solo che invece di tutta la struttura, won una riduzione effettiva pratica della dimensione del chip esposto (chiamata modalità ritagliata sensore nel nostro EMCCD). Per questa configurazione sia efficace, il chip di fuori della ROI deve essere coperta da una coppia di feritoie montati nel percorso ottico. Grazie a questa configurazione di risoluzione tempo fino a 10 -4 secondi può essere raggiunto. Si prega di notare, tuttavia, che questo approccio può essere accoppiato con molte diverse configurazioni sperimentali, come spiegato nella sezione 'discussione'.
Dimostrazione del metodo sarà fornito in cellule vive, utilizzando sia un ATTO488 contrassegnata con 1-palmitoil-2-idrossi-sn -glycero-3-fosfoetanolamina (ATTO488-PPE) e una variante di GFP-etichetta della transferrina recettore (GFP TFR). Nel caso di ATTO488-PPE questo approccio può recuperare correttamente un'applicazione D pressoché costante in funzione di spostamento medio indica una diffusione prevalentemente libera, come precedentemente riportato 30,35. Al contrario, TFR-GFP mostra una diminuzione D app in funzione della cilindrata media, suggerendo diffusione parzialmente confinata 6. Inoltre, in quest'ultimo caso è possibile quantificare la costante diffusione locale e la superficie media di confinamento di numerose micron sul piano membrana.
Protocollo
Calibrazione 1 Sistema
- Point Spread Function (PSF) calibrazione
- Diluire 10 ml di soluzione di 30 nm perline fluorescenti (circa 5 mM) in 90 ml di acqua distillata e poi sonicare la soluzione per 20 min. Tagliare un quadrato (1 cm x 1 cm) pezzo di gel di agarosio (3%) e deposito 10 microlitri della soluzione sulla superficie del gel. Capovolgere il pezzo di gel sul vetro fondo di un piatto 2 centimetri Petri e spremere la goccia sul vetro.
- Accendere il setup di acquisizione, mettere il campione nel supporto, impostare l'esposizione della fotocamera e EMgain (100 msec e 1.000 sono buoni parametri ma ottimizzano secondo il sistema) e attendere che la fotocamera si raffreddi.
- Impostare esposizione della fotocamera a 100 msec, fotocamera EMgain a 1.000, modalità di acquisizione al fotogramma Transfer, 100 ripetizione automatica e impostazione di risparmio.
- Usando l'oculare e la messa a fuoco luce trasmessa al confine del gel e quindi spostare l'obiettivo verso il centro del gel, regolare la messa a fuoco e start la procedura di allineamento laser (in LAS AF, selezionare 'installazione TIRF' e seguire la procedura di allineamento automatico).
- Trovare un campo di vista con isolati singoli punti, esattamente concentrarsi sul punto più luminoso (che di solito rappresenta perline aggregato) come riferimento, acquisire 100 fotogrammi e ripetere l'operazione 5-6 volte al fine di acquisire diversi punti singoli.
- Importare la serie acquisita ad un programma di elaborazione dei dati e la media dello stack nel tempo (Figura 1A) e selezionare una singola goccia isolato. Fare attenzione a selezionare i più piccoli per evitare di aggregati di particelle.
- Montare la distribuzione selezionata intensità (un esempio di singolo profilo perline è presentato in Figura 1B) con una funzione gaussiana con il comando "gaussfit" (negli strumenti ICS-Matlab nei Materiali in Matlab). Verificare la bontà del fit ispezionando i residui ottenuti (un esempio di profilo gaussiano attrezzata con i residui corrispondenti viene presentato in Figura 1B).
- Calibrazione della fotocamera
- Accendere la fotocamera e attendere che la fotocamera si raffreddi. Impostare l'impostazione di acquisizione della fotocamera, (vale a dire, per la fotocamera utilizzata impostiamo l'esposizione a 0.5 msec, fotocamera EMgain a 1.000, modalità di acquisizione alla modalità ritagliata, la dimensione del ROI di 32 x 128, 10.000 ripetizioni) e iniziare l'acquisizione del fondo telecamera segnale.
- Import acquisito serie telaio di un programma di elaborazione dei dati. Calcolare e controllare l'intensità media in ogni pixel per verificare che lo sfondo fotocamera circa piatto nella regione selezionata del chip. In modalità crea, rimuovere la prima e le ultime linee orizzontali (da 3 a 10 a seconda delle dimensioni della ROI) per ogni frame perché lo sfondo fotocamera è solitamente polarizzato nelle linee di confine.
- Creare un istogramma dei valori (definito anche livello digitale, DL) nelle immagini acquisite pila (usando il comando 'cron' in Matlab) e tracciare il logaritmodi conseguente frequenza (utilizzando il comando semilogy in Matlab). Un esempio di distribuzione DL per telecamera sfondo è presentata nella Figura 2.
NOTA: Se la fotocamera funziona bene, la trama mostra un picco di circa gaussiano (a profilo parabolico in log-scala) che rappresenta la distribuzione dei valori associati al fotone zero seguito da un decadimento esponenziale (una retta con pendenza negativa in log-scala ) che rappresenta la distribuzione dei valori associati a 1 fotone (Figura 2). In particolare, il centro e la varianza della funzione gaussiana rappresentano la telecamera di offset e l'errore, rispettivamente, mentre la costante di decadimento esponenziale della parte rappresenta una stima della DL assegnato dalla fotocamera per ogni singolo fotone. In Matlab utilizzare la sezione "CalibrateCamera" dello Script in materiali di supporto. - Ripetere l'operazione per tutti la telecamera selezionata EMGain e Gain.
2 etichettatiPreparazione delle cellule
- Per preparare i liposomi richiesti per incorporazione di lipidi 36, sciogliere separatamente 1 mg di DOPE (1,2-dioleoyl- sn -glycero-3-fosfoetanolamina), 1 mg di DOTAP (1,2-dioleoyl-3-trimetilammonio-propano), e 1 mg di PPE-ATTO488 in 1 ml di cloroformio. Mescolare 0,5 ml di soluzione DOPE, 0,5 ml di soluzione di DOTAP, e 25 ml di soluzione di PPE-ATTO488 e secco sotto vuoto per 24 ore. Aggiungere 0,5 ml di tampone di HEPES 20 mM, vortex per 15 minuti e ultrasuoni per 15 min a 40 ° C.
- Per preparare la cella, lavare 3 volte con PBS un piatto p100 di confluenti CHO-K1 (ovaio di criceto cinese), aggiungere 1 ml di tripsina e conservare in incubatrice per 5 min. Sospendere le cellule staccate aggiungendo 9 ml di terreno DMEM / F12 supplementato con 10% di FBS e sementi 150 ml di soluzione di cellule in una capsula di Petri contenente 800 ml dello stesso terreno.
- Conservare in incubatore per 24 ore a 37 ° C e 5% di CO 2. Per incorporazione di lipidi, sostituire medio cella con 500microlitri di terreno privo di siero; dopo 30 min, aggiungere 2 ml di soluzione di liposomi; dopo 15 minuti di lavaggio con PSB e aggiungere nuovo medium DMEM / F12 per l'imaging.
- Per la trasfezione, le cellule trasfezione secondo il protocollo Lipofectamine (istruzioni del produttore) utilizzando TFR-GFP plasmide e conservare 24 ore in incubatrice prima di imaging.
3 Acquisizione Dati
- Preparazione di installazione
- Al fine di termostato al microscopio, 24 ore prima dell'esperimento accendere l'incubatore.
- Al fine di applicare il tempo di acquisizione più veloce realizzabile, opera in modalità ritagliata sensore (vedi introduzione) e utilizzare una prima telecamera per l'imaging (fotocamera 1) e seconda fotocamera per selezionare la cella (2 macchina fotografica). Uno schema di configurazione di setup è presentato in Figura supplementare S1. Poi, per allineare le due telecamere Accendere il microscopio e attendere che le telecamere si raffreddino.
- Situato su entrambe le fotocamere, i parametri per l'imaging luce trasmessa (cioè , 20 msec di tempo di esposizione, 1 per EM Gain) e mettere il microscopio in modalità Luminoso campo.
- Mettere i campioni nel supporto e mettere a fuoco utilizzando oculare, inviare la luce alla telecamera 1 e spingere delicatamente le fessure che consentono la luce solo sul ROI utilizzato per l'imaging cellulare (qui un ROI di 32 x 32 pixel).
- Spostare una cella nella regione selezionata e trasmettere la luce alla telecamera 2, quindi disegnare una ROI nel software che controlla la telecamera 2 per avere un riferimento.
- Imaging (Figura 3A)
- Prima di tutto, allineare il laser TIRF secondo la procedura di configurazione. Nel nostro setup, selezionare il 'impostazione TIRF' e avviare la procedura di allineamento automatico. Quando il laser è allineato set 70 nm di profondità di penetrazione (circa 70 °).
- Impostare il tempo di esposizione di 70 msec e EMGain a 100 su entrambi videocamera 1 e la videocamera 2; quindi, selezionare una cella utilizzando la fotocamera 1, quindi inviare la luce fotocamera da 2 e mettere a fuoco con precisione la membrana cellulare. Impostare l'esposizione minima camera 2, 1000 EMGain, modalità corta del sensore, 10 5 ripetizioni e salvataggio automatico impostato come file FITS (Flexible Image Transport System, un formato che può essere facilmente gestito).
- Avviare l'acquisizione di registrare la serie di immagini. Rilasciare il guadagno e la modalità ritagliata per consentire la stabilizzazione della temperatura prima di acquisire una nuova cella, quindi ripetere gli ultimi due passaggi al fine di acquisire 8-10 cellule.
4 Calcolo del Mean Square Cilindrata da Imaging (i MSD)
NOTA: Il seguente protocollo può essere applicato direttamente ai dati grezzi. Allo stesso tempo, l'intero protocollo è valido per acquisizioni dati simulati sia in Matlab e in SimFCS. Il link per il tutorial corrispondenti si possono trovare nella sezione 'Materials'.
- Calcolo con Matlab
- Importare la serie acquisita Matlab utilizzando ImportImageSeries copione. Calcolare l'intensità media di ogni immagine nel tempo utilizzando il command dire sui primi 2 dimensioni e utilizzare complotto per vedere il vettore risultante.
- Se più del 10% di fotoscolorimento è presente, scartare la serie o rimuovere la prima parte di essi. Se è inferiore, provare a correggere l'effetto sulla funzione di correlazione sottraendo ad ogni immagine la sua intensità media, come mostrato prima 37.
- Calcolare l'intensità media di ciascun pixel mediante medio sulla terza dimensione e visualizzare l'immagine risultante.
NOTA: Particolare attenzione è richiesta al fine di evitare correlazioni artefatti. Infatti, come precedentemente indicato per tecniche simili 38, i bordi delle celle nonché su vescicole fuoco potrebbe introdurre una forte correlazione. Se l'ispezione di immagine media rivela i bordi delle celle o di vescicole fuoco, cercare di escludere la regione coinvolta altrimenti scartare l'acquisizione. Per correggere l'effetto di queste strutture immobili sottrarre l'intensità media temporale da ogni pixel 39. - Calcola tegli correlazione spazio-temporale (G (ξ, χ, τ)) utilizzando la funzione CalculateSTICScorrfunc. Rimuovere G (ξ, χ, 0) in quanto la correlazione a causa del rumore girato in regime di scarsa luminosità domina G (0,0,0); la correlazione dovuta al rivelatore domina la G (± 1,0,0), e il movimento delle particelle durante il tempo di esposizione potrebbe deformare G (ξ, χ, τ) per τ = 0, aumentando la vita misurata (questo effetto scompare per τ > 0) 34.
- Media G (ξ, χ, τ> 0) con un tempo-bin logaritmico per ridurre il rumore utilizzando la funzione "LogBinStack" per il sostegno materiale e poi montare il G risultante (ξ, χ, τ) utilizzando la funzione "gaussfit" di gli strumenti di ICS-Matlab nei Materiali per recuperare l'i MSD (la seconda colonna della matrice risultante).
- Tracciare la σ ottenuta vita (τ) 2 (i MSD) in funzione del tempo. Se i dati sono troppo rumorosi, cercare di aumentare il numero di acqucornici IRED, aumentano la potenza del laser, media più G (ξ, χ, τ) insieme.
- Calcolo da SimFCS
- Aprire i file acquisiti con ImageJ usando BIOFORMAT importatore plug-in e salvare serie acquisite Tiff sequenza.
- Aprire strumento SimFCS e selezionare RICS e selezionare File> Importa immagini multiple (complementare Figura S2).
- Selezionare Fit, inserire i parametri di acquisizione corretti e chiudere la finestra di misura (complementare figura S3).
- Selezionare Display> Intensità Media> CH1 e verificare la presenza di photobleaching (complementare figura S4).
- Se più del 10% di fotoscolorimento è presente scarti serie o se è possibile caricare nuovamente la sequenza di immagini rimuovendo la prima parte della serie.
- Se lo sbiancamento è inferiore del 10% selezionare Strumenti> i MSD> Impostazione dei parametri, controllare 'Usa media mobile', ambientato nel pannello ROI sulla sinistra unumero di telaio per il movimento attenzione media pagatore che il tempo corrispondente è superiore al tempo di diffusione caratteristica (per particella che si muove a 1 micron 2 sec -1 un tempo di 10 secondi è una buona media mobile)
- Selezionare Strumenti> IMSD> Calcola i MSD (Figura complementare S5) e in forma ed esportare la i MSD dalla memo pad (figura complementare S6).
5 Calcolo della Legge Diffusione del I MSD
- Montare i primi punti per estrapolare l'intercetta (σ 0 2) (5 punti sono sufficienti di solito, ma più punti possono essere montati se essi mostrano un comportamento lineare) e confrontare questo valore con il PSF precedentemente misurato 2. Se sono comparabili, sono state seguite le dinamiche di fluorofori isolate. Per contro, se σ 0 2 >> FPF 2 tentativo di acquisire velocemente per garantire chedinamiche non nascosti sono presenti 34.
- Calcolare la diffusività apparente (D app) e lo spostamento medio (R) utilizzando le equazioni 3 e 4 (vedi Introduzione).
- Terreno D app come funzione di R per ottenere una legge di diffusione paragonabile a quello che viene misurata con variazione posto a base FCS 12 (Figura 3D).
Risultati
Per calibrare la vita strumentale, l'immagine di una singola fluorescente nano-tallone può essere misura come descritto nel protocollo punto 1.1. Una tipica immagine fluorescente di queste perle è presentato in Figura 1. Il montaggio della distribuzione dell'intensità da una funzione gaussiana 2D restituisce buoni residui e permette che misura la vita strumentale a 270 nm. Questo valore è in buon accordo con il limite di diffrazione atteso stimato con l'equazione di Rayleigh. Questa tara...
Discussione
Tracciamento singola particella (SPT) rappresenta una delle strategie più comuni per lo studio della dinamica molecolare e ha il grande vantaggio di misurare traiettorie delle particelle. Questo a sua volta permette di sondare il comportamento anche di poche particelle etichettati in un sistema complesso. Tuttavia, per raggiungere questo vantaggio SPT bisogno tipicamente una bassa densità della sonda ed etichette molto luminosi. In particolare, per ottenere l'alta risoluzione temporale (intervallo msec) sonda inor...
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
This work is supported in part by NIH-P41 P41-RRO3155 and NIH P50-GM076516 (grant to EG), and Fondazione Monte dei Paschi di Siena (grant to FB).
Materiali
| Name | Company | Catalog Number | Comments |
| iXon Ultra 897 | Andor | DU-897U-CS0 | |
| Solis | Andor | ||
| CHO-K1 | ATCC | CCL-61 | |
| ATTO 488 labeled PPE | ATTO-TEC GmbH | AD 488-151 | |
| DOPE | Avanti Polar Lipids, Inc. | 850725 | |
| DOTAP | Avanti Polar Lipids, Inc. | 890890 | |
| 100x Penicillin-Streptomycin-Glutamine | Gibco | 10378-016 | |
| DMEM/F-12 | Gibco | 21331 | |
| FBS | Gibco | 10082147 | |
| HEPES | Gibco | 15630-106 | |
| PBS | Gibco | 10010-023 | |
| SimFCS 3.0 | Globals Software | the software can be downloaded here: http://www.lfd.uci.edu/globals/ | |
| DMI6000 with TIRF modulus | Leica | ||
| LAS AF | Leica | ||
| Lipofectamine 2000 | Lipofectamine | 11668019 | |
| Matlab | MathWork | ||
| ImageJ | NIH | ||
| [header] | |||
| C-terminal GFP tagged Tranferrin Receptor | OriGene | RG200980 | |
| Agar | Sigma Aldrich | A5306 | |
| Chloroform | Sigma Aldrich | 528730 | |
| Latex beads, fluorescent yellow-green, 30 nm | Sigma Aldrich | L5155 | |
| SONICA Ultrasonic Cleaners | SOLTEC | ETH S3 | |
| Petri Dishes | Willco | GWSt-3522 | |
| Bio-Format importer for Matlab | http://www.openmicroscopy.org/site/support/bio-formats5/users/matlab/ | ||
| ICS-MatLab Tools | https://www.cellmigration.org/resource/imaging/software/ICSMATLAB_28-02-06.zip | ||
| Simulation by Matlab Tutorial | https://www.cellmigration.org/resource/imaging/icsmatlab/ICSTutorial.html | ||
| Simulation by SimFCS Tutorial | https://www.cellmigration.org/resource/imaging/ppt-pdf/RICS%20Simulations.ppt | ||
Riferimenti
- Engelman, D. M. Membranes are more mosaic than fluid. Nature. 438 (7068), 578-580 (2005).
- Vereb, G., et al. yet structured: The cell membrane three decades after the Singer-Nicolson model. Proc. Natl. Acad. Sci. U. S. A. 100 (14), 8053-8058 (1073).
- Ishihara, A., Hou, Y., Jacobson, K. The Thy-1 antigen exhibits rapid lateral diffusion in the plasma membrane of rodent lymphoid cells and fibroblasts. 84 (5), 1290-1293 (1987).
- Axelrod, D., et al. Lateral motion of fluorescently labeled acetylcholine receptors in membranes of developing muscle fibers. Proc. Natl. Acad. Sci. U. S. A. 73 (12), 4594-4598 (1976).
- Jacobson, K., Derzko, Z., Wu, E. S., Hou, Y., Poste, G. Measurement of the lateral mobility of cell surface components in single, living cells by fluorescence recovery after photobleaching. J. Supramol. Struct. 5 (4), 10-1002 (1976).
- Kusumi, A., et al. Paradigm shift of the plasma membrane concept from the two-dimensional continuum fluid to the partitioned fluid: high-speed single-molecule tracking of membrane molecules. Annu. Rev. Biophys. Biomol. Struct. 34, 351-378 (2005).
- Kusumi, A., Ike, H., Nakada, C., Murase, K., Fujiwara, T. Single-molecule tracking of membrane molecules: plasma membrane compartmentalization and dynamic assembly of raft-philic signaling molecules. Semin. Immunol. 17 (1), 3-21 (2005).
- Schwille, P., Korlach, J., Webb, W. W. Fluorescence correlation spectroscopy with single-molecule sensitivity on cell and model membranes. Cytometry. 36, 176-182 (1999).
- Gielen, E., et al. Diffusion of sphingomyelin and myelin oligodendrocyte glycoprotein in the membrane of OLN-93 oligodendroglial cells studied by fluorescence correlation spectroscopy. C. R. Biol. 328 (12), 1057-1064 (2005).
- Weiss, M., Hashimoto, H., Nilsson, T. Anomalous protein diffusion in living cells as seen by fluorescence correlation spectroscopy. Biophys. J. 84, 4043-4052 (2003).
- Wawrezinieck, L., Rigneault, H., Marguet, D., Lenne, P. F. Fluorescence correlation spectroscopy diffusion laws to probe the submicron cell membrane organization. Biophys. J. 89 (6), 4029-4042 (2005).
- Lenne, P. F., et al. Dynamic molecular confinement in the plasma membrane by microdomains and the cytoskeleton meshwork. EMBO J. 25 (14), 3245-3256 (2006).
- Ries, J., Schwille, P. Studying slow membrane dynamics with continuous wave scanning fluorescence correlation spectroscopy. Biophys. J. 91 (5), 1915-1924 (2006).
- Ruan, Q., Cheng, M. A., Levi, M., Gratton, E., Mantulin, W. W. Spatial-temporal studies of membrane dynamics: scanning fluorescence correlation spectroscopy (SFCS). Biophys. J. 87 (2), 1260-1267 (2004).
- Berland, K. M., So, P. T., Chen, Y., Mantulin, W. W., Gratton, E. Scanning two-photon fluctuation correlation spectroscopy: particle counting measurements for detection of molecular aggregation. Biophys. J. 71, 410-420 (1996).
- Heinemann, F., Betaneli, V., Thomas, F. A., Schwille, P. Quantifying lipid diffusion by fluorescence correlation spectroscopy: a critical treatise. Langmuir. 28 (37), 13395-13404 (2012).
- Cardarelli, F., Lanzano, L., Gratton, E. Capturing directed molecular motion in the nuclear pore complex of live cells. Proc. Natl. Acad. Sci. U. S. A. 109 (25), 9863-9868 (2012).
- Sanchez, S. A., Tricerri, M. A., Gratton, E. Laurdan generalized polarization fluctuations measures membrane packing micro-heterogeneity in vivo. Proc. Natl. Acad. Sci. U. S. A. 109 (19), 7314-7319 (2012).
- Cardarelli, F., Lanzano, L., Gratton, E. Fluorescence correlation spectroscopy of intact nuclear pore complexes. Biophys. J. 101 (4), 27-29 (2012).
- Di Rienzo, C., et al. Unveiling LOX-1 receptor interplay with nanotopography: mechanotransduction and atherosclerosis onset. Sci. Rep. 3, 10-1038 (2013).
- Unruh, J. R., Gratton, E. Analysis of molecular concentration and brightness from fluorescence fluctuation data with an electron multiplied CCD camera. Biophys. J. 95 (11), 5385-5398 (2008).
- Kannan, B., et al. Electron multiplying charge-coupled device camera based fluorescence correlation spectroscopy. Anal. Chem. 78 (10), 3444-3451 (2006).
- Jones, S. A., Shim, S. H., He, J., Fast Zhuang, X. three-dimensional super-resolution imaging of live cells. Nat. Methods. 8 (6), 499-508 (2011).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy. 3 (10), 793-795 (2006).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Hess, S. T., Girirajan, T. P., Mason, M. D. Ultra-high resolution imaging by fluorescence photoactivation localization microscopy. Biophys. J. 91 (11), 4258-4272 (2006).
- Manley, S., et al. High-density mapping of single-molecule trajectories with photoactivated localization microscopy. Nat. Methods. 5 (2), 155-157 (2008).
- Hell, S. W. Far-field optical nanoscopy. Science. 316 (5828), 1153-1158 (2007).
- Klar, T. A., Hell, S. W. Subdiffraction resolution in far-field fluorescence microscopy. Opt. Lett. 24 (14), 954-956 (1999).
- Eggeling, C., et al. Direct observation of the nanoscale dynamics of membrane lipids in a living cell. Nature. 457 (7233), 1159-1162 (2009).
- Hedde, P. N., et al. Stimulated emission depletion-based raster image correlation spectroscopy reveals biomolecular dynamics in live cells. Nat. Commun. 4, .
- Hebert, B., Costantino, S., Wiseman, P. W. Spatiotemporal image correlation spectroscopy (STICS) theory, verification, and application to protein velocity mapping in living CHO cells. Biophys. J. 88 (5), 3601-3614 (2005).
- Brown, C. M., et al. Probing the integrin-actin linkage using high-resolution protein velocity mapping. J. Cell Sci. 119, 5204-5214 (2006).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. Fast spatiotemporal correlation spectroscopy to determine protein lateral diffusion laws in live cell membranes. Proc. Natl. Acad. Sci. U. S. A. 110 (30), 12307-12312 (2013).
- Mueller, V., et al. STED nanoscopy reveals molecular details of cholesterol- and cytoskeleton-modulated lipid interactions in living cells. Biophys. J. 101 (7), 1651-1660 (2011).
- Kleusch, C., Hersch, N., Hoffmann, B., Merkel, R., Csiszar, A. Fluorescent lipids: functional parts of fusogenic liposomes and tools for cell membrane labeling and visualization. Molecules. 17 (1), 1055-1073 (2012).
- Ries, J., Chiantia, S., Schwille, P. Accurate determination of membrane dynamics with line-scan FCS. Biophys. J. 96 (5), 1999-2008 (2009).
- Kolin, D. L., Wiseman, P. W. Advances in image correlation spectroscopy: measuring number densities, aggregation states, and dynamics of fluorescently labeled macromolecules in cells. Cell Biochem. Biophys. 49 (3), 141-164 (2007).
- Digman, M. A., et al. Measuring fast dynamics in solutions and cells with a laser scanning microscope. Biophys. J. 89 (2), 1317-1327 (2005).
- Ritchie, K., et al. Detection of non-Brownian diffusion in the cell membrane in single molecule tracking. Biophys. J. 88 (3), 2266-2277 (2005).
- Voie, A. H., Burns, D. H., Spelman, F. A. Orthogonal-plane fluorescence optical sectioning: three-dimensional imaging of macroscopic biological specimens. J. Microsc. 170, 229-236 (1993).
- Huisken, J., Swoger, J., Del Bene, ., Wittbrodt, F., J, E. H., Stelzer, Optical sectioning deep inside live embryos by selective plane illumination microscopy. Science. 305 (5686), 1007-1009 (2004).
- Wohland, T., Shi, X., Sankaran, J., Stelzer, E. H. Single plane illumination fluorescence correlation spectroscopy (SPIM-FCS) probes inhomogeneous three-dimensional environments. Opt. Express. 18 (10), 10627-10641 (2010).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon