È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Specifici Sequenza Etichettatura degli acidi nucleici e proteine con metiltransferasi e cofattore Analoghi
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
DNA e proteine sono la sequenza-specifica etichettati con affinità o gruppi reporter fluorescente con DNA o proteine metiltransferasi e analoghi cofattore sintetici. A seconda della specificità cofattore di enzimi, aziridina o doppi analoghi cofattore attivati sono impiegati per l'etichettatura di uno o due step.
Abstract
S -Adenosyl-L-metionina (SAM) o AdoMet metiltransferasi-dipendente (MTase) catalizzano il trasferimento del gruppo metilico attivato dal AdoMet a specifiche posizioni in DNA, RNA, proteine e piccole biomolecole. Questa reazione di metilazione naturale può essere espansa a un'ampia varietà di reazioni di alchilazione con analoghi sintetici cofattore. Sostituzione del centro solfonio reattiva di AdoMet con un anello di aziridina conduce cofattori accoppiabili con DNA da vari MTases DNA. Questi cofattori aziridina possono essere equipaggiati con gruppi reporter diverse posizioni della frazione adenina e utilizzati per S-specific equence M ethyltransferase- I nduced L Abel zione del DNA (sorridendo DNA). Come esempio tipico diamo un protocollo per biotinilazione di pBR322 plasmide DNA sequenza 5'-ATCG A T-3 'con il DNA MTase M.BseCI e il cofattore aziridina 6BAz inun passo. Estensione del gruppo metilico attivato con insaturi gruppi alchil risultati in un'altra classe di analoghi AdoMet che sono utilizzati per m ethyltransferase diretto T rasferimento di un ctivated ruppi G (MTAG). Poiché le catene laterali estesi vengono attivati dal centro solfonio e il legame insaturo, questi cofattori sono chiamati analoghi AdoMet doppio attivati. Questi analoghi funzione non solo come cofattori per DNA MTases, come cofattori aziridina, ma anche per RNA, proteine e piccole molecole MTases. Essi sono tipicamente utilizzati per modificazione enzimatica di substrati MTase con gruppi funzionali uniche che sono contrassegnati con gruppi del reporter in una seconda fase chimica. Questo è esemplificato in un protocollo per l'etichettatura di fluorescenza di proteine istone H3. Un piccolo gruppo propargile viene trasferito dal SeAdoYn cofattore analogica alla proteina dalla lisina istone H3 4 (H3K4) MTase Set7 / 9 seguito da etichettatura clic delalkynylated H3 istone con TAMRA azide. Etichettatura MTase-mediata con analoghi cofattore è una tecnologia abilitante per molte applicazioni interessanti, tra cui l'identificazione e lo studio funzionale di substrati MTase e genotipizzazione del DNA e rilevazione metilazione.
Introduzione
Etichettatura specifica degli acidi nucleici e proteine 1,2 3,4 è di grande interesse per le caratterizzazioni funzionali, diagnosi medica e (nano) biotecnologie. Qui vi presentiamo un metodo enzimatico di etichettatura per questi biopolimeri che si basa su S -adenosyl-L-metionina (SAM AdoMet o) metiltransferasi-dipendente (MTases). Questa classe di enzimi (EC 2.1.1.) Rivolge singole posizioni nucleofili (azoto, ossigeno, zolfo e atomi di carbonio) all'interno specifici residui di acidi nucleici e proteine e naturalmente trasferisce il gruppo metilico attivato della AdoMet cofattore (Figura 1A) 5. Inoltre, MTases possono utilizzare analoghi cofattore sintetici per l'etichettatura specifico con tag affinità, fluorofori o altre etichette (Figura 1B) 6. Sono stati sviluppati due classi di analoghi AdoMet: cofattori Aziridine per S equence-specifica M ethyltransferase- I Nduced L abel ing (sorridendo) 7 e doppie analoghi attivati AdoMet per m ethyltransferase diretto T rasferimento di un ruppi G ctivated (MTAG) 8.

Figura 1: reazioni catalizzate da metiltransferasi (MTases) A. metile trasferimento gruppo dal cofattore naturale AdoMet (SAM) per vari substrati, tra cui il DNA, RNA, proteine e piccole biomolecole B. Etichettatura / funzionalizzazione di acidi nucleici e proteine (NNNNN =.. coppie di basi di DNA, RNA nucleotidi per e aminoacidi delle proteine; XXXXX = sequenza di riconoscimento del MTase con residuo di destinazione in verde) con analoghi cofattore sintetici. Cofattori aziridina contenenti un gruppo giornalista (sfera blu)collegata all'anello di adenina sono sequenza specificamente accoppiato con il residuo di destinazione (a sinistra) e analoghi doppio attivato AdoMet comportare un trasferimento del catene alchiliche estese trasportano un giornalista chimica Y (a destra), che può essere etichettato da bioorthogonal reazione scatto in una seconda fase. Clicca qui per vedere una versione più grande di questa figura.
Cofattori aziridina funzionano meglio con DNA MTases. Essi contengono un anello di tre termini con un atomo di azoto 9 (o un N -mustard 10,11) invece del centro solfonio gruppo reattivo. Protonazione di questo atomo di azoto attiva il azirdinico per l'attacco nucleofilo dal nucleotide obiettivo che porta ad accoppiamento covalente di tutta cofattore con DNA. Collegando gruppi giornalista all'anello adenina cofattori aziridina possono essere utilizzati in combinazione con DNA MTases etichettare DNA in un solo passo ( g> Figura 1B, a sinistra) 7,12. Ciò è illustrato nel dettaglio per la biotinilazione di DNA con 6BAz 13 - 15 (cofattore aziridina con biotina attaccato alla posizione 6 dell'anello adenina) e il DNA MTase specifico adenina da Bacillus stearothermophilus (M.BseCI) 16 (figura 2, vedi sezione Protocollo 2: etichettatura One-step del DNA tramite aziridina cofattori). Oltre a M.BseCI ('sequenza di riconoscimento, il MTases DNA da Thermus aquaticus (M.TaqI, 5'-TCG A -3 5'-ATCG A T-3)'), da Haemophilus heamolyticus (M.HhaI, 5 '-G C GC-3') e da Spiroplasma (M.SssI, 5'-C G-3 ') sono stati utilizzati con successo per biotinilato DNA con 6BAz 17. Inoltre, cofattori aziridina possono essere impiegati per un passo etichettatura fluorescenza DNA 18,19.
ONTENUTO "fo: keep-together.within-page =" always ">
Figura 2:. Sequenza specifica biotinilazione un passo del DNA con M.BseCI e 6BAz Il DNA MTase M.BseCI riconosce la doppia elica sequenza di DNA 5'-ATCG A T-3 'e naturalmente metila il gruppo amminico del secondo adenina residuo (verde) con AdoMet. Con il cofattore aziridinico 6BAz il corso della reazione viene modificato e M.BseCI porta alla specifica sequenza biotinilazione DNA accoppiando tutta cofattore inclusi biotina (blu) con l'adenina destinazione. Clicca qui per vedere una versione più grande di questa figura.
Doppia analoghi AdoMet attivati contiene estese catene laterali insature invece di un gruppo metile al centro solfonio (Figura 1B , a destra) 20. Il legame doppio o triplo insaturo in β-position al centro solfonio compensa elettronicamente effetti sterici sfavorevoli all'interno dello stato di transizione di stabilizzazione coniugativo. Dal momento che sia il centro solfonio e il legame insaturo attivare la catena laterale per il trasferimento enzimatico, questi cofattori sono stati nominati analoghi AdoMet doppio attivati. In genere, essi sono utilizzati per trasferire catene laterali con gruppi unici chimici (giornalisti chimici), come gruppi di aminoacidi, alchini e azide, per l'etichettatura chemio-selettivo in una seconda fase 8,21. In generale, analoghi a doppia attivato AdoMet non può funzionare solo come cofattori per DNA MTases 8,20,21 ma anche per RNA MTases 22,23 e MTases proteine 24-28 permettendo etichettatura supplementare di RNA e proteine. Tuttavia, le catene laterali estese sono stericamente più esigenti di un gruppo metile e allargando le MTase siti attivi di proteine di ingegneria è often necessaria per ottenere velocità di trasferimento efficienti. Un'altra soluzione a questo problema è di utilizzare un analogo AdoMet con un piccolo gruppo propargile (tre atomi di carbonio) in cui l'alchino terminale ha due funzioni: 1. Stabilizzazione dello stato di transizione durante il trasferimento enzimatica e 2. maniglia reattiva per seguenti modificazioni chimiche di rame catalizzata cicloaddizione azide-alchino (CuAAC) clicca chimica. Si è scoperto che il propargilico risultante AdoMet analogico 29 è abbastanza instabile in condizioni di neutralità o leggermente di base e di scarsa utilità. Questo inconveniente può essere risolto sostituendo l'atomo di zolfo con selenio. Il cofattore risultante 5 '- [(Se) [(3 S) -3-ammino-3-carbossipropil] prop-2-ynylselenonio] -5'-deossiadenosina (SeAdoYn, figura 3) è accettata da wild-type DNA, RNA e MTases proteine 30-32 che abolisce la necessità di ingegneria proteica in molti casi. Questo è esemplificato da fluorescenza pro etichettatura proteina con la lisina istone H3 4 (H3K4) MTase Set7 / 9 33 (Figura 3, vedere la sezione protocollo 3: etichettatura proteina Two-step con cofattori doppie attivati).
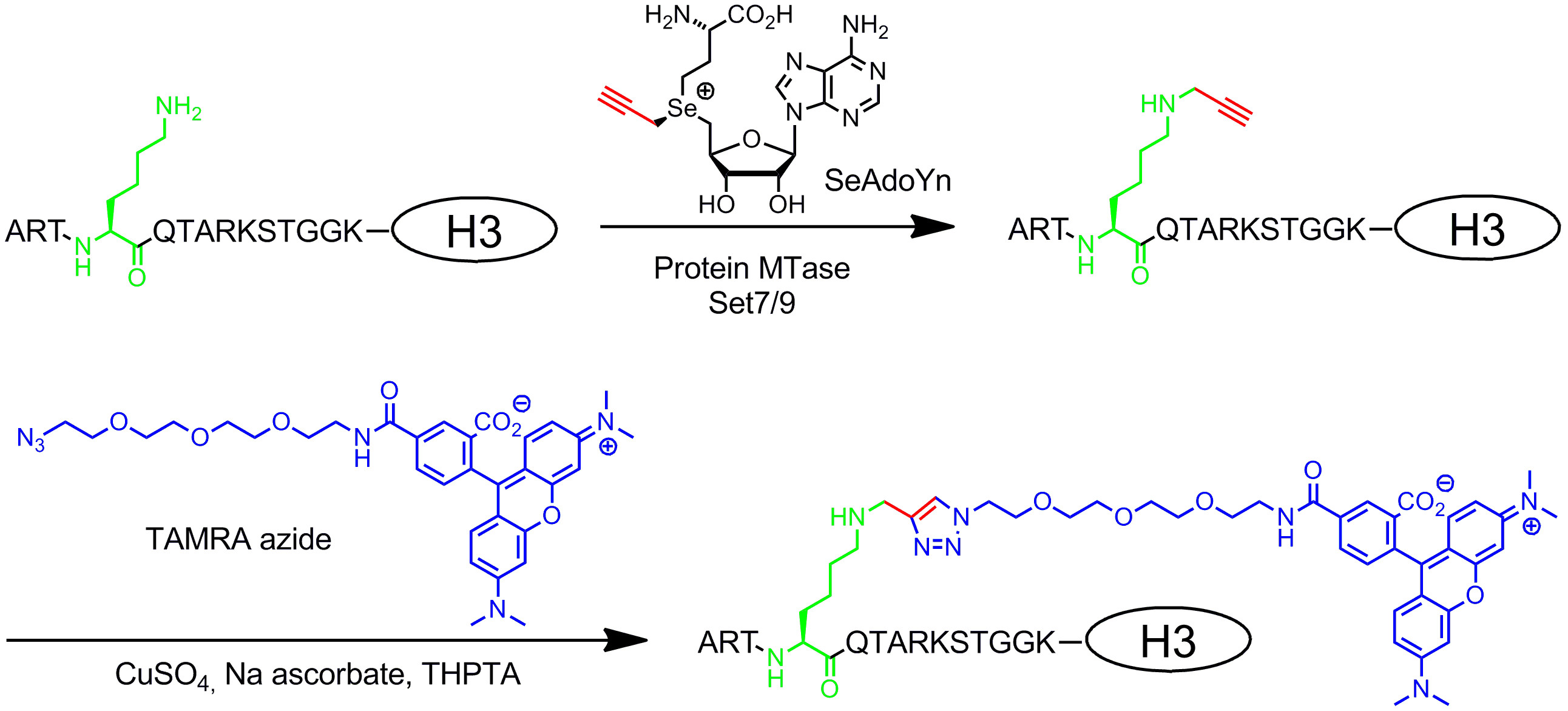
Figura 3:. In due fasi un'etichettatura specifica fluorescenza-Sequenza dell'istone H3 con Set7 / 9, SeAdoYn e TAMRA azide La proteina MTase Set7 / 9 metila naturalmente il gruppo amminico di lisina 4 a dell'istone H3 (H3K4, verde) con AdoMet. Con il cofattore doppio attivato SeAdoYn il MTase trasferisce un piccolo gruppo propargile (rosso) per il residuo di lisina. Il legame terminale collegato tripla è poi selettivamente modificata in una reazione click bioorthogonal (azide-alkyne cicloaddizione di rame-catalizzata, CuAAC) con azide-derivatizzata TAMRA (tetrametilrodamina, blu) fluoroforo.Carico / 52014 / 52014fig3highres.jpg "target =" _ blank "> Clicca qui per vedere una versione più grande di questa figura.
Protocollo
1. Istruzioni generali
- Conservare aziridina cofattore 6BAz (in DMSO) e di proteine MTase Set7 / 9 a -80 ° C e tutti gli altri reagenti tra cui doppio attivato cofattore SeAdoYn e DNA MTase M.BseCI (nel 50% glicerolo) a -20 ° C.
- Determinare la concentrazione di 6BAz e SeAdoYn tramite spettroscopia UV / Vis utilizzando i coefficienti di estinzione ε 269nm (6BAz) = 16000 centimetri -1 m -1 e ε 260nm (SeAdoYn) = 15400 centimetri -1 M -1 in acqua deionizzata. Determinare la concentrazione di MTases dal saggio Bradford o, se il coefficiente di estinzione è disponibile, tramite assorbimento diretto a 280 nm.
- Cercate di evitare di creare bolle pipettando intensivo o vortex per prevenire la perdita di attività enzimatica. Invece, mescolare delicatamente pipettando su e giù.
- Quando si aggiungono cofattori aziridina da soluzioni di riserva in DMSO fare in modo che la concentrazione DMSO finale nel test è menoal 5%. Includere sempre 10 ioni magnesio mm nella tampone per evitare reazioni aspecifiche con DNA.
- Quando si aggiungono cofattori doppie attivati da soluzioni stock acide utilizzare piccoli volumi (soluzioni stock altamente concentrati) per evitare variazioni di pH e di fare in modo che il pH della soluzione test non cambia in modo significativo. Evitare tioli, ad esempio, β-mercaptoetanolo o ditiotreitolo (DTT), in tampone perché possono interferire con la reazione di scatto per complessazione degli ioni rame richiesti.
2. One-step Etichettatura del DNA tramite aziridina Cofattori
- Metiltransferasi-Induced Label ing specifici Sequence (sorridendo) di plasmide DNA con M.BseCI MTase DNA e aziridina cofattore 6BAz.
- Scongelare la soluzione cofattore a 20 ° C e preparare le miscele di reazione in ghiaccio.
- Oltre al dosaggio effettuare un controllo "cofattore", di visualizzare le modifiche specifiche non e un & #8220; enzima "controllo, per assicurarsi che la preparazione MTase è libero di naturale AdoMet cofattore.
- Per il mix dosaggio di 2 ml di 10x tampone di modifica (contenente 100 mM Tris-HCl, 100 mM MgCl 2, 20 mM β-mercaptoetanolo, pH 7,4), 2 ml di pBR322 (0,5 mg / mL), 10 eq. M.BseCI per sequenza di riconoscimento sul DNA (sequenza 1 riconoscimento in pBR322) e il cofattore aziridina 6BAz ad una concentrazione finale di 60 mM in un volume totale di 20 microlitri. Aggiungere cofattore e MTase DNA scorso.
NOTE: β-Mercaptoethanol è tossico, corrosivo e dannosi per l'ambiente. - Per il controllo "cofattore" aggiungere acqua deionizzata invece di M.BseCI e per il controllo "enzima" aggiungere acqua deionizzata al posto di 6BAz.
- Mescolare le soluzioni pipettando gentilmente su e giù.
- Incubare le provette a 55 ° C per 1 ora.
- Centrifugare brevemente per raccogliere tutto il liquido sul fondo delle provette.
- Test restrizione-modificazione per verificare la modifica del DNA.
- Preparare una soluzione mescolando 10 microlitri tampone 10x R.TaqI (contenente 100 mM Tris-HCl, 50 mM MgCl 2, 1 M NaCl, 1 mg / ml di albumina di siero bovino, pH 8,0), 80 microlitri di acqua deionizzata e 3,3 ml di endonucleasi di restrizione (REase) da Thermus aquaticus (R.TaqI, 10 U / ml). Assicurarsi di aggiungere il REase nell'ultimo passaggio.
- Per ogni tubo da 2.1.7 aggiungere 2 ml di 10x tampone R. TaqI e 28 ml di soluzione dall'alto (2.2.1).
- Mescolare le soluzioni pipettando gentilmente su e giù.
- Incubare le provette a 65 ° C per 30 min.
- Centrifugare brevemente per raccogliere tutto il liquido sul fondo delle provette.
- Assay spostamento elettromobilità (EMSA) con streptavidina per verificare la modifica funzionale.
- Rimuovere 25 microlitri da ogni provetta (2.2.5) e aggiungere 2,4 ml di una soluzione di streptavidina (1 mM rispetto alla streptavidina monomer in tampone contenente streptavidina 100 mM Na 2 HPO 4, NaCl 100 mM, pH 7,5; 4 equivalenti di biotina totale). Aggiungere 2,4 ml di tampone streptavidina ai rimanenti tubi.
- Incubare tutte le provette a 37 ° C per 1 ora.
- Analisi tramite elettroforesi su gel di agarosio.
- Aggiungere 5 ml di tampone di caricamento 6x (0,25% blu di bromofenolo, 30% glicerolo) ad ogni provetta.
- Mescolare le soluzioni delicatamente.
- Carico 10 microlitri di ciascun campione nei pozzetti di un gel di agarosio (1% agarosio in tampone TBE 0,5x contenente 1x GelRed da una soluzione 10.000X magazzino).
- Eseguire il gel in tampone TBE 0.5x con 80 V per circa. 1 ora.
- Visualizzare bande di DNA su un tavolo UV (312 nm) con una telecamera CCD dotata di filtro (540 ± 50 nm).
NOTA: la luce UV è dannoso per gli occhi e la pelle.
3. Due-passo Protein Labeling via Cofattori doppie Attivato
- MethyltransfTrasferimento di gruppi attivati (MTAG) con Set7 / 9 e doppio attivato cofattore SeAdoYn per dell'istone H3 lisina 4 Etichettatura (fase di modifica) cancella-Diretto.
- Scongelare i componenti e preparare le miscele di reazione sul ghiaccio. NOTA: Tenere sempre SeAdoYn raffreddato per evitare il degrado.
- Oltre al dosaggio effettuare un controllo "cofattore", di visualizzare le modifiche non specifiche, e un controllo "enzima", di escludere reazioni non specifiche della sonda fluorescente.
- Preparare una soluzione test (20 microlitri) contenente tampone modifica (50 mM Tris-HCl, 5% glicerolo, pH 8,5), 10 micron istone H3, 10 micron Set7 / 9 e 600 micron SeAdoYn (miscela di entrambi epimeri al selenio). Negli ultimi passi aggiungere cofattore e poi MTase.
- Per il controllo "cofattore" preparare una soluzione test come in 3.1.3 e aggiungere 60 AdoMet mM di competere con il cofattore sintetico. Per il controllo "enzima" aggiungere acqua deionizzata al posto di SeAdoYn.
- Mescolare le soluzioni lentamente pipettando su e giù. Controllare il pH aggiungendo 1 ml di ciascuna soluzione di campo superiore di una striscia pH (range pH di 5 - 10).
- Incubare a 37 ° C per 2 ore.
- Nel frattempo preparare un gel SDS gel di poliacrilammide al 12% (in esecuzione: 357 Bis-Tris pH 6,5-6,8 mM, 0,1% (w / v) APS, 0,04% (v / v) TEMED e 12% acrilammide / bisacrilammide 37.5: 1 ; loading gel: 357 Bis-Tris pH 6,5-6,8 mM, 0,1% (w / v) APS, 0,04% (v / v) TEMED e 5% acrilammide / bisacrilammide 37,5: 1).
NOTA: L'acrilamide / bisacrilammide è tossico e la salute pericolosi. Indossare guanti durante questa procedura.
- Etichettatura chimica di lisina alkinylated 4 in H3 via azide-alkyne cicloaddizione (CuAAC) (step etichettatura) di rame-catalizzata.
- Poco prima della fine della reazione modifica preparare una miscela contenente 3 5x click CuSO 4 mM, tris 3 mM (3-idrossipropil-triazolylmethyl) ammina (THPTA), 250 mM ascorbato di sodio e 6 mM TAMRA azide convolume totale di 20 microlitri.
- Aggiungere 5 ml di 5x click mix preparato fresco ogni provetta per avviare il CuAAC e placare la reazione di modifica.
- Mescolare delicatamente pipettando su e giù.
- Proteggere tutti i tubi con un foglio di alluminio dalla luce per evitare fotometabolismo del fluoroforo.
- Incubare a 20 ° C per 1 ora.
- Protein precipitazioni per rimuovere l'eccesso di libera TAMRA fluoroforo.
- Per evitare eclissare della fluorescenza etichettati istone H3 da intensa fluorescenza in gel di libera TAMRA fluoroforo, eliminare l'eccesso di fluoroforo per precipitazione delle proteine (3.3.2 - 3.3.4) 34.
- Aggiungere 75 ml di metanolo, 18,8 ml cloroformio e 50 ml di acqua deionizzata ad ogni provetta e vortex brevemente dopo ogni aggiunta. Centrifugare a 16.000 g per 5 min. Rimuovere la fase superiore senza disturbare lo strato di interfaccia, che contiene la proteina.
- Aggiungere 56,3 ml di metanolo al rimanente fase in ogni provetta, vortex e centrifugare a 16.000 xg per 5 min a pellet proteina. Rimuovere il surnatante. Ripetere questo passaggio per lavare il pellet.
- Coprire i tubi aperti con un panno che non lasci residui e lasciare asciugare per 15-30 min.
- Analisi tramite SDS PAGE.
- Sciogliere proteine precipitate da 3.3.4 in 20 microlitri tampone di SDS carico (50 mM Tris-HCl, 2,5% (w / v) di SDS, 10% (v / v) di glicerolo, 320 mM β-mercaptoetanolo e 0,05% (w / v) blu bromofenolo, pH 6,8). Assicurati di sciogliere completamente il pellet sciacquando le pareti dei tubi con una pipetta.
- Incubare i campioni a 95 ° C per 10 minuti e lasciate raffreddare a 20 ° C.
- Centrifugare brevemente per raccogliere tutto il liquido sul fondo delle provette.
- Caricare l'intero importo di ogni campione nei pozzetti di un gel di poliacrilammide SDS (3.1.7). Utilizzare 50 MOPS mM, 50 mM Tris-X (Tris-base), 5 mM EDTA, 0,1% (w / v) di SDS come tampone di corsa per elettroforesi.
- Eseguire il gel con 120 V per circa. 90 min.
- Visualizzare la fluorescenza in-gel su un tavolo UV (312 nm) con una telecamera CCD dotata di filtro (540 nm ± 50 nm).
NOTA: la luce UV è dannoso per gli occhi e la pelle.
Risultati
One-step Etichettatura del DNA tramite aziridina Cofattori
Questa reazione viene effettuata ad esempio con il DNA MTase M.BseCI, che modifica il secondo residuo di adenina nel doppio filamento 5'-ATCG A T-3 'sequenza ed ha un sito di riconoscimento sul plasmide pBR322 (Figura 4A). Per verificare l'etichettatura plasmide, pBR322 è sfidato con la endonucleasi di restrizione (REase) R.TaqI (5'-TCGA-3 '). R.TaqI ha sette siti pBR322...
Discussione
Etichettatura in un unico passaggio di DNA con MTases DNA e cofattori aziridina (DNA sorridendo) è un metodo robusto ma alcuni aspetti deve essere considerato al momento di pianificare l'esperimento.
Cofattore Aziridine: La concentrazione 6BAz per l'etichettatura del DNA con M.BseCI era di 60 micron. Quando si utilizzano altri MTases DNA la concentrazione cofattore dovrebbe essere ottimizzato, ad esempio concentrazioni partire da 20 micron sono stati impiegati con ...
Divulgazioni
The authors disclose the following competing financial interest: E.W. is inventor on related patents.
Riconoscimenti
The authors thank Kerstin Glensk for preparing the MTases M.BseCI and Set7/9 and gratefully acknowledge funding by the Excellence Initiative of the German Federal and State Governments and RWTH Aachen University. The authors are happy to provide 6BAz and SeAdoYn or other cofactor analogues for collaborative research.
Materiali
| Name | Company | Catalog Number | Comments |
| 6BAz | Synthesized according to Weinhold et al., Patent number US 8,129,106, published March 6, 2012. | ||
| β-Mercaptoethanol | Serva | 28625 | |
| Acetic acid | Fisher Scientific | 10304980 | |
| Acrylamide/Bis Solution, 37.5:1 | Serva | 10688 | |
| UltraPure Agarose | Invitrogen | 16500100 | |
| Ammonium persulfate (APS) | Serva | 13375 | |
| Bis-Tris | Gerbu | 1304 | |
| Boric acid | Gerbu | 1115 | |
| Bromophenol blue Na salt | Serva | 15375 | |
| Copper(II) sulfate | Aldrich | C1297 | |
| Chloroform | Fisher Scientific | 10020090 | |
| Coomassie Brilliant Blue | Serva | 17525 | |
| EDTA disodium salt | Gerbu | 1034 | |
| Ethanol | Merck | 100983 | |
| GelRed (10,000x in water) | Biotium | 41003 | |
| Glycerol (99.5%) | Gerbu | 2006 | |
| FastRuler Low Range DNA Ladder | Thermo Scientific | SM1103 | |
| Histone H3 | Expression plasmid obtained from Dr. Philipp Voigt and Prof. Danny Reinberg; expression and isolation according to T. J. Richmond et al., J. Mol. Biol. 1997, 272, 301-311. | ||
| M.BseCI | Expression plasmid obtained from Dr. Michael Kokkinidis; expression and isolation according to Kapetaniou et al., Acta Cryst. 2006, F63, 12-14. | ||
| Methanol | Fisher Scientific | 10675112 | |
| Magnesiumchloride hexahydrate | J.T. Baker | 4003 | |
| MOPS | Gerbu | 1081 | |
| Sodium chloride | Gerbu | 1112 | |
| pH strip (Neutralit) | Merck | 1,095,330,001 | |
| pBR322 | Thermo Scientific | SD0041 | |
| R.TaqI (10 u/µl) | Thermo Scientific | ER0671 | |
| SeAdoYn | Synthesized according to Willnow et al., ChemBioChem 2012, 13, 1167-1173. | ||
| Set7/9 | Expression plasmid obtained from Prof. Danny Reinberg, expression and isolation according to D. Reinberg et al., Genes Dev.2002, 16, 479-489. | ||
| Streptavidin | Gerbu | 3058 | |
| (+)-Sodium L-ascorbate | Sigma Life Science | A7631 | |
| SDS Granular | Gerbu | 1833 | |
| di-Sodium hydrogenphosphate | Merck | 106,586 | |
| TAMRA azide | Synthesized according to reference 30: Willnow et al., ChemBioChem 2012, 13, 1167-1173. | ||
| TaqI buffer (10x) | Thermo Scientific | B28 | |
| N,N,N',N'-Tetramethylethylenediamine (TEMED) | Acros Organics | 42058 | |
| Tris-HCl | Gerbu | 1028 | |
| Tris-X (TRIS-base) | Gerbu | 1018 | |
| Tris(3-hydroxypropyltriazolyl-methyl)amine (THPTA) | Sigma-Aldrich | 762342 |
Riferimenti
- Gottfried, A., Weinhold, E. Sequence-specific covalent labelling of DNA. Biochem. Soc. Trans. 39, 623-628 (2011).
- Zohar, H., Muller, S. J. Labeling DNA for single-molecule experiments: methods of labeling internal specific sequences on double-stranded DNA. Nanoscale. 3, 3027-3039 (2011).
- Hinner, M. J., Johnsson, K. How to obtain labeled proteins and what to do with them. Curr. Opin. Biotechnol. 21, 766-776 (2010).
- Wua, Y. -. W., Goody, R. S. Probing protein function by chemical modification. J. Pept. Sci. 16, 514-523 (2010).
- Struck, A. -. W., Thompson, M. L., Wong, L. S., Micklefield, J. S-Adenosyl-methionine-dependent methyltransferases: Highly versatile enzymes in biocatalysis, biosynthesis and other biotechnological applications. ChemBioChem. 13, 2642-2655 (2012).
- Klimasauskas, S., Weinhold, E. A new tool for biotechnology: AdoMet-dependent methyltransferases. Trends Biotechnol. 25, 99-104 (2007).
- Pljevaljcic, G., Schmidt, F., Weinhold, E. Sequence-specific Methyltransferase-Induced Labeling of DNA (SMILing DNA). ChemBioChem. 5, 265-269 (2004).
- Lukinavicius, G., Lapiene, V., Stasevskij, Z., Dalhoff, C., Weinhold, E., Klimasauskas, S. Targeted labeling of DNA by methyltransferase-directed Transfer of Activated Groups (mTAG). J. Am. Chem. Soc. 129, 2758-2759 (1021).
- Pignot, M., Siethoff, C., Linscheid, M., Weinhold, E. Coupling of a nucleoside with DNA by a methyltransferase. Angew. Chem. Int. Ed. 37, 2888-2891 (1998).
- Weller, R. L., Rajski, S. R. Design, synthesis, and preliminary biological evaluation of a DNA methyltransferase-directed alkylating agent. ChemBioChem. 7, 243-245 (2006).
- Du, Y., Hendrick, C. E., Frye, K. S., Comstock, L. R. Fluorescent DNA Labeling by N-Mustard Analogues of S-adenosyl-l-methionine. ChemBioChem. 13, 2225-2233 (2012).
- Pljevaljcic, G., Schmidt, F., Scheidig, A. J., Lurz, R., Weinhold, E. Quantitative labeling of long plasmid DNA with nanometer precision. ChemBioChem. 8, 1516-1519 (1002).
- Wilkinson, S., et al. Molecular scale architecture: engineered three- and four-way junctions. Bioconjugate Chem. 19, 470-475 (2008).
- Braun, G., et al. Enzyme-directed positioning of nanoparticles on large DNA templates. Bioconjugate Chem. 19, 476-479 (2008).
- Kim, S., et al. Enzymatically incorporated genomic tags for optical mapping of DNA binding proteins. Chem. Int. Ed. 51, 3578-3581 (2012).
- Rina, M., Bouriotis, V. Cloning purification and characterization of the BseCI DNA methyltransferase from Bacillus stearothermophilus. Gene. 133, 91-94 (1993).
- Weinhold, E., Meier, T., Düfel, H., Markert-Hahn, C., Schmuck, R. Sequence-specific detection of methylation in biomolecules. US Patent. , (2012).
- Pljevaljcic, G., Pignot, M., Weinhold, E. Design of a new fluorescent cofactor for DNA methyltransferases and sequence-specific labeling of DNA. J. Am. Chem. Soc. 125, 3492-3410 (2003).
- Schmidt, F. H. -. G., Hüben, M., Gider, B., Renault, F., Teulade-Fichou, M. -. P., Weinhold, E. Sequence-specific Methyltransferase-Induced Labelling (SMILing) of plasmid DNA for studying cell transfection. Bioorg. Med. Chem. 16, 40-48 (2008).
- Dalhoff, C., Lukinavicius, G., Klimasauskas, S., Weinhold, E. Direct transfer of extended groups from synthetic cofactors by DNA methyltransferases. Nat. Chem. Biol. 2, 31-32 (2006).
- Lukinavicius, G., Tomkuviene, M., Masevicius, V., Klimasauskas, S. Enhanced chemical stability of AdoMet analogues for improved methyltransferase-directed labeling of DNA. ACS Chem. Biol. 8, 1134-1139 (2013).
- Motorin, Y., et al. Expanding the chemical scope of RNA:methyltransferases to site-specific alkynylation of RNA for click labeling. Nucleic Acids Res. 39, 1943-1952 (1943).
- Schulz, D., Holstein, J. M., Rentmeister, A. A chemo-enzymatic approach for site-specific modification of the RNA cap. Angew. Chem. Int. Ed. 52, 7874-7878 (2013).
- Peters, W., et al. Enzymatic site-specific functionalization of protein methyltransferase substrates with alkynes for click labeling. Angew. Chem. Int. Ed. 49, 5170-5173 (2010).
- Islam, K., Zheng, W., Yu, H., Deng, H., Luo, M. Expanding cofactor repertoire of protein lysine methyltransferase for substrate labeling. ACS Chem. Biol. 6, 679-684 (2011).
- Wang, R., Zheng, W., Yu, H., Deng, H., Luo, M. Labeling substrates of protein arginine methyltransferase with engineered enzymes and matched S-adenosyl-l-methionine analogues. J. Am. Chem. Soc. 133, 7648-7651 (2011).
- Islam, K., et al. Bioorthogonal profiling of protein methylation using azido derivative of S-adenosyl-l-methionine. J. Am. Chem. Soc. 134, 5909-5915 (2012).
- Islam, K., et al. Defining efficient enzyme-cofactor pairs for bioorthogonal profiling of protein methylation. Proc. Natl. Acad. Sci. U.S.A. 110, 16778-16783 (2013).
- Binda, O., Boyce, M., Rush, J. S., Palaniappan, K. K., Bertozzi, C. R., Gozani, O. A chemical method for labeling lysine methyltransferase substrates. ChemBioChem. 12, 330-334 (2011).
- Willnow, S., Martin, M., Lüscher, B., Weinhold, E. A selenium-based click AdoMet analogue for versatile substrate labeling with wild-type protein methyltransferases. ChemBioChem. 13, 1167-1173 (2012).
- Bothwell, I. R., et al. Se-Adenosyl-l-selenomethionine cofactor analogue as a reporter of protein methylation. J. Am. Chem. Soc. 134, 14905-14912 (2012).
- Tomkuviene, M., Clouet-d’Orval, B., Cerniauskas, I., Weinhold, E., Klimasauskas, S. Programmable sequence-specific click-labeling of RNA using archaeal box C/D RNP methyltransferases. Nucleic Acids Res. 40, 6765-6773 (2012).
- Nishioka, K., et al. Set9, a novel histone H3 methyltransferase that facilitates transcription by precluding histone tail modifications required for heterochromatin formation. Genes Dev. 16, 479-489 (2002).
- Clark, P. M., et al. Direct in-gel fluorescence detection and cellular imaging of O-GlcNAc-modified proteins. J. Am. Chem. Soc. 130, (2008).
- Lukinavicius, G., Lapinaite, A., Urbanaviciute, G., Gerasimaite, R., Klimasauskas, S. Engineering the DNA cytosine-5 methyltransferase reaction for sequence-specific labeling of DNA. Nucleic Acids Res. 40, 11594-11602 (2012).
- Neely, R. K., Dedecker, P., Hotta, J., Urbanaviciute, G., Klimasauskas, S., Hofkens, J. DNA fluorocode: A single molecule, optical map of DNA with nanometre resolution. Chem. Sci. 1, 453-460 (2010).
- Roberts, R. J., Vincze, T., Posfai, J., Macelis, D. REBASE-a database for DNA restriction and modification: enzymes, genes and genomes. Nucleic Acids Res. 38, 234-236 (2010).
- Petrossian, T. C., Clarke, S. G. Uncovering the human methyltransferasome. Mol. Cell. Proteomics. 10, 1-12 (2011).
- Kriukiene, E., et al. DNA unmethylome profiling by covalent capture of CpG sites. Nat. Commun. 4, 2190 (2013).
- Wang, R., et al. Profiling genome-wide chromatin methylation with engineered posttranslation apparatus within living cells. J. Am. Chem. Soc. 135, 1048-1056 (2013).
- Zhang, C., Weller, R. L., Thorson, J. S., Rajski, S. R. Natural product diversification using a non-natural cofactor analogue of S-adenosyl-l-methionine. J. Am. Chem. Soc. 128, 2760-2761 (2006).
- Stecher, H., et al. Biocatalytic Fiedel-Crafts alkylation using non-natural cofactors. Angew. Chem. Int. Ed. 48, 9546-9548 (2009).
- Lee, B. W. K., Sun, H. G., Zang, T., Kim, B. J., Alfaro, J. F., Zhou, Z. S. Enzyme-catalyzed transfer of a ketone group from an S-adenosylmethionine analogue: A tool for the functional analysis of methyltransferases. J. Am. Chem. Soc. 132, 3642-3643 (2010).
- Winter, J. M., et al. Expanding the structural diversity of polyketides by exploring the cofactor tolerance of an inline methyltransferase domain. Org. Lett. 15, 3774-3777 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon