È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Metodo semplice per DNA fluorescenza
In questo articolo
Riepilogo
Here, we present a simple method for performing fluorescence DNA in situ hybridization (DNA ISH) to visualize repetitive heterochromatic sequences on slide-mounted chromosomes. The method requires minimal reagents and it is versatile for use with short or long probes, different tissues, and detection with fluorescence or non-fluorescence-based signals.
Abstract
DNA ibridazione in situ (ISH DNA) è un metodo comunemente utilizzato per le sequenze di mappatura a specifiche regioni cromosomiche. Questo approccio è particolarmente efficace per la mappatura delle sequenze altamente ripetitive alle regioni eterocromatiche, dove approcci computazionali affrontare sfide proibitive. Qui si descrive un protocollo semplificato per ISH DNA che aggira lavaggi formammide che sono passi standard in altri protocolli ISH DNA. Il nostro protocollo è ottimizzato per l'ibridazione con brevi singole sonde filamento di DNA che portano coloranti fluorescenti, che segnano in modo efficace le sequenze di DNA ripetute all'interno di regioni cromosomiche eterocromatiche tutta una serie di diversi tipi di tessuto insetto. Tuttavia, le applicazioni possono essere estese da usare con sonde più grandi e la visualizzazione di sequenze singola copia (non ripetitivi) DNA. Dimostriamo questo metodo mappando diverse sequenze ripetitive diversi di cromosomi schiacciate da Drosophila melanogaster cellule neurali e Nasoniavitripennis spermatociti. Mostriamo modelli di ibridazione sia per le piccole, le sonde sintetizzati commercialmente e per una sonda più grande per il confronto. Questa procedura utilizza semplici forniture di laboratorio e reagenti, ed è ideale per gli investigatori che hanno poca esperienza con l'esecuzione di ISH DNA.
Introduzione
DNA ibridazione in situ (ISH DNA) è un metodo comunemente utilizzato per le sequenze di mappatura a specifiche regioni cromosomiche. Sonde alle regioni a copia singola all'interno euchromatin possono essere generati attraverso una manciata di approcci, tra cui nick-traduzione o end-etichettatura dei prodotti lunghi di DNA 1,2 e l'incorporazione di deoxygenin (DIG) nucleotidi -attached e il loro riconoscimento attraverso una grande varietà di anticorpi gruppo-coniugato 1-3. Visualizzazione di sequenze eucromatiche in pochi o solo numero copia richiede l'uso di singole, grandi sonde ad alta attività specifica o un cocktail di più, sonde minori che aumentano insieme il segnale.
Al contrario, le sequenze altamente ripetitive trovano in heterochromatin, come DNA satellite, sono bersagli facili per ISH DNA perché normalmente esistono come decine di migliaia di ripetizioni raggruppati in regioni cromosomiche singole note come blocchi. Elementi trasponibili possono anche esseretrovata ad alti numeri di copia presso distinto cromosomica loci 2. In questi casi, singole sonde con bassa attività specifica può effettivamente etichettare sequenze eterocromatiche a causa della loro ibridazione in più siti. Sonde a sequenze ripetitive possono essere sintetizzati oligonucleotidi commercialmente come brevi (30-50 bp) e coniugati chimicamente con qualsiasi più gruppi fluorescenti differenti. Mappatura sequenze ripetitive all'interno heterochromatin utilizzando le tecnologie di sequenziamento del genoma-è difficile a causa di problemi incontrati in impalcature edili all'interno di blocchi satellitari altamente ripetitive 4-6,7. Attualmente, ISH si pone come il modo più efficace di mappare queste sequenze a livello sub-cromosoma. Questa strategia è importante per la mappatura gran numero di sequenze ripetitive che vengono scoperte da studi del genoma e del trascrittoma di sequenziamento in corso.
L'efficienza e la facilità di mappatura sequenze ripetute sui cromosomi montati scorrevoli sarebbero Greatly rafforzata da un protocollo semplificato per ISH DNA. Per esempio, i protocolli esistenti per ISH DNA coinvolgono diversi lavaggi di tessuti ibridizzati in soluzione formammide 2,8, aggiungendo così sostanzialmente il tempo necessario per le sequenze di mappatura e produrre anche grandi quantità di rifiuti chimici per questa costosa reagente. Qui si descrive un metodo di revisione ISH DNA che aggira la necessità di lavaggi formammide e utilizza attrezzature di laboratorio di base e reagenti. Questo metodo è stato originariamente progettato per la rapida mappatura altamente ripetitive sequenze di DNA in regioni eterocromatiche di neuroblasti larvali Drosophila utilizzando oligonucleotidi sintetizzati in commercio che coniugate con coloranti fluorescenti. Tuttavia, questo metodo funziona anche per la mappatura sequenze ripetute utilizzando sonde grandi sintetizzati attraverso altri mezzi e 9,10 per diversi tipi di tessuto e cromosomi diversi. Inoltre, questo metodo può essere utilizzato per mappare le sequenze eucromatiche utilizzando più o multiple sonde brevi all'interno della sequenza euchromatic di interesse.
Protocollo
1. Tissue Dissection e Fixation (60 min)
- Per cervelli Drosophila, 3 ° posto instar larve in una goccia di 1x PBS (tampone fosfato salino). Scegli grande 3 ° instar larve che sono attivamente strisciando da fiale o bottiglie che non sono sovraffollate.
- Utilizzare uno ultrafine paio pinzette per afferrare i ganci bocca e un altro paio pinzette per afferrare 2/3 per tutta la lunghezza del corpo (figura 1A, B). Tirare delicatamente i ganci bocca per esporre il cervello, gangli ventrale, ghiandole salivari e parte del tratto digerente larvale. Utilizzare le pinzette per separare il cervello e gangli ventrale (Figura 1A, B) dalle altre tessuti e posto in una goccia di 1x PBT (tampone fosfato salino con Tween) su un piatto di plastica Petri.
- Per Nasonia dissezioni testicolo, scegliere maschio pupe di 3 giorni di età (corpi giallo con gli occhi rossi). Maschio Nasonia hanno piccole lunghezze pad ala relative a females durante la fase di pupa (Figura 1C).
- Tenere la pupa in alto dell'addome vicino alla regione toracica con una coppia pinzetta, e con l'altra coppia pinzetta, afferrare la punta molto distale dell'addome e tirare fuori i testicoli tear-forma-drop (saranno circondati da grasso corpo che può essere scosso delicatamente; Figura 1D). Staccare tutte le parti del corpo esterno dei testicoli, che impediscono una corretta schiacciamento, e posto in una goccia di 1x PBT su un piatto di plastica Petri.
- Per ogni diapositiva, sezionare un totale di quattro o cinque campioni di tessuto (es., Drosophila cervello larvali o testicoli Nasonia).
NOTA: Più di cinque campioni porteranno a nuclei sovraffollate e cromosomi. - Facoltativamente, per raggiungere una certa separazione dei cromatidi fratelli nelle regioni eucromatiche di cromosomi mitotici, trattare il tessuto con una soluzione ipotonica: trasferire cervelli da 1x PBT a un calo dello 0,5% di sodio citrato per 5-10 (non più di 10) min.
NOTA: Colchicina (un inibitore mitotico) può essere utile per aumentare il numero di figure mitotiche in metafase 11. Tuttavia, il suo utilizzo non è necessaria se si utilizzano molto grande larve sane, e anche indesiderabile perché può influenzare negativamente la risoluzione dei cromosomi, la diffusione e la morfologia. - Inserire una goccia (~ 20 microlitri) di soluzione fissativo (2,5% paraformaldeide in 45% di acido acetico) sulla superficie di un vetrino Sigmacote trattati pulita.
NOTA: Preparare la soluzione fissativa fresca per ogni giorno di utilizzo. Soluzioni fissativo che vanno 1,8-3,7% paraformaldeide nel 45% di acido acetico per ottenere i migliori risultati per i pesci. Utilizzando tessuti diversi cervelli o testicoli, o adattare questo protocollo per immuno-FISH può richiedere la sperimentazione di diversi fissativi (per un elenco di diversi fissativi, vedi 12). - Trasferire accuratamente ciascun campione di tessuto dal buffer dissezione (1x PBT) nella goccia di fissativo con le pinzette ultrafini, Minimizing trasferimento di dissezione tampone nella soluzione fissativo. Posizionare i campioni di tessuto in modo che siano uniformemente distanziati l'uno dall'altro entro la goccia fissativo. Incubare i tessuti in fissativo per 4 minuti a temperatura ambiente.
- Posizionare con cura una diapositiva faccia poli-lisina rivestite giù sul slittamento dei tessuti e la copertura. Non premere a questo punto, ma invece permettono ai due di contattare leggermente in modo che la polizza di copertura attacca sulla parte inferiore della diapositiva. Invertire la diapositiva in modo che il vetrino è in cima.
- Panino lo slittamento scorrevole / tessuti / copertura all'interno di un pezzo di carta piegato filtro. Su una superficie stabile, con il pollice, premere con fermezza verso il basso sulla posizione direttamente sopra il vetrino. Fare attenzione a non scorrimento laterale del vetrino (ciò causerà macchie del tessuto).
- Immergere il vetrino / tessuti / fodera in azoto liquido (vedi apparecchio in figura 1E, F), e lasciate riposare fino a quando il Azoto ferma bollente (è più fine). Rimuovere il vetrino e immediatamente scattare fuori il vetrino con una lametta fresca sfogliando un angolo del vetrino verso l'alto (evitare di graffiare il tessuto fissato con la lama di rasoio).
- Pre-raffreddamento della diapositiva / tessuti / copertura di slittamento su un blocco di ghiaccio secco, copertura scivolare fino, per 1-2 minuti prima sommergendo in azoto liquido contribuisce a prevenire le diapositive di cracking.
- Posizionare immediatamente il vetrino con il tessuto in una vaschetta Coplin piena di etanolo al 100% a temperatura ambiente e lasciare riposare per almeno 5 minuti (questo tempo può essere più lungo, se necessario).
NOTA: Cold 100% etanolo potrebbe anche essere utilizzato. - Rimuovere il vetrino con il tessuto, liberano dal etanolo in eccesso con un Kimwipe (senza toccare il tessuto fisso), e lasciare il vetrino asciugare per 1 ora.
- Passare direttamente al punto 2 o mantenere le diapositive a secco in aria bassa umidità o in una camera di essiccazione per settimane anche mesi prima di eseguire l'ibridazione.
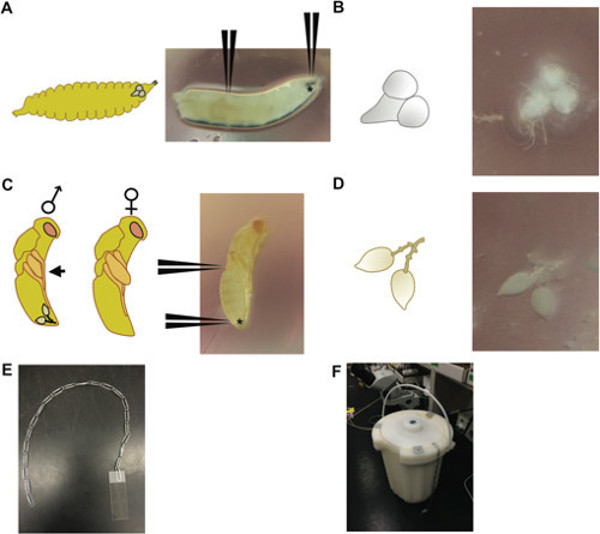
Figura 1: (A) A 3 ° instar Drosophila larva (a destra), con le posizioni indicate per cui per afferrare i ganci bocca (indicato con *) e 2/3 fino in fondo le larve di sezionare il cervello; (Sinistra) uno schema di una larva una stessa fase di sviluppo, raffigurante la posizione relativa del cervello nella testa larvale. (B) Il cervello e gangli ventrale sezionato da un 3 ° instar Drosophila larva (a destra) e uno schema di questo tessuti (a sinistra). (C) 3-giorni-old pupa Nasonia allo stadio giallo occhi corpo-rossi. (D) Una coppia di testicoli sezionato da un vecchio pupa maschio Nasonia 3 giorni (a destra) con le posizioni che indica dove per afferrare la pupa al posteriore dell'addome (indicato con *) ea metà strada sul corpo; (Sinistra) schematico raffigurante maschio e femmina erap pupe; pupe maschio può essere distinto da ali che non si estendono oltre il profilo sagittale (freccia nera), a differenza di femmine che hanno ali che si estendono oltre il profilo; la posizione relativa della coppia testis è mostrato in pupa maschile. (E) Apparecchiatura-una serie di graffette-e metodo (F) utilizzate per immergere i vetrini in un serbatoio di azoto liquido. Stringhe graffetta multipli possono essere utilizzati per immersione simultanea di più diapositive.
2. ibridazione in situ (30 min al giorno 1; 1 h-2,5 ore per lunghi sonde-giorno 2)
- Aggiungere 1 ml (100 ng) di ciascuna sonda a 20 ml di buffer di 1.1x ibridazione. Pipettare Sonda / tampone di ibridazione sulla superficie del tessuto fissa (evitare di toccare il tessuto).
- Posizionare con cura un vetrino direttamente sul tampone di sonde / ibridazione, facendo in modo che la polizza di copertura è centrata direttamente sopra il tessuto. Il buffer dovrebbe migrare a thdi e bordo esterno del vetrino, senza lasciare bolle d'aria.
NOTA: Le piccole bolle che non contattano il tessuto non pongono problemi alla procedura. Rimuovere grandi bolle sollevando attentamente un angolo del vetrino e con attenzione cadere indietro sul vetrino. - Posizionare il vetrino slittamento / tessuto / copertura sulla superficie di un blocco di pre-riscaldato a 95 ° C (coperchio scivolare). Coprire con un grosso pezzo di foglio di alluminio per evitare l'esposizione alla luce. Sia la diapositiva incubare a 95 ° C per 5 min.
NOTA: Un blocco tipico calore con fori per tubi può essere capovolto per fornire una superficie piana su cui posizionare il vetrino / tessuti / copertura di slittamento. - Rimuovere il vetrino, lasciandolo raffreddare leggermente finché è caldo al tatto. Avvolgere attenzione un pezzo di Parafilm allungato intorno vetrino per sigillare il liquido sottostante.
- Posizionare il vetrino sigillato all'interno di una camera di umidità e posizionare la camera in un incubatore preriscaldato a 30 ° C. Incubare a 30 ° C per 4 ore a tutta la notte.
- Creare una camera umida da una scatola di punta vuota o contenitori Tupperware con coperchio con Kimwipes inumidite o tovaglioli di carta posti sul fondo.
NOTA: Le sonde oligo DNA a singolo filamento sono stati progettati per essere 28-33 basi per raggiungere una temperatura di fusione teorica (T m) di 45-47 ° C. Questi lunghezza e T m intervalli riflettono il fatto che molte sequenze ripetitive che abbiamo studiato sono AT-ricco e quindi hanno bassissimo contenuto GC. Sonde più lunghe probabilmente hanno valori più elevati m T; ciò può comportare superiore sfondo ibridazione alla temperatura di ibridazione standard 30 ° C. Così, alcuni di risoluzione dei problemi con le temperature di ibridazione può essere richiesto per ottenere i migliori risultati. Per trovare la migliore temperatura di ibridazione, aumento (o diminuzione) la temperatura di 5 ° C, in modo incrementale.
- Creare una camera umida da una scatola di punta vuota o contenitori Tupperware con coperchio con Kimwipes inumidite o tovaglioli di carta posti sul fondo.
- Rimuovere con attenzione il Parafilm dalla diapositiva e poi rimuovere con attenzione il vetrino lentamente sollevando un angolo. Wash il vetrino tre volte per 15 minuti ogni lavaggio in tampone SSC 0.1x. Coprire la vaschetta Coplin con un foglio di alluminio durante i lavaggi per ridurre al minimo l'esposizione alla luce.
- Se non si usa una lunga sonda biotinilata, passare al punto 2.8.
- Se si utilizza una sonda lunga biotinilato, asciugare la zona attorno il tessuto con un Kimwipe, facendo attenzione a non toccare il tessuto stesso. Mettere 100 ml di soluzione bloccante sopra il tessuto e coprire delicatamente con un coprioggetto, avendo cura di evitare bolle di cattura. Avvolgere il vetrino su vetrino con Parafilm e posto a 37 ° C per 30 min.
- Rimuovere con attenzione il vetrino e asciugare intorno il tessuto con un Kimwipe. Dispensare 100 l di rodamina-avidina diluito 1: 1000 in SBT su tessuto e coprire delicatamente con un coprioggetto, avendo cura di evitare di intrappolare bolle. Avvolgere il vetrino su vetrino con Parafilm e posto a 37 ° C per 30 min.
- Rimuovere con attenzione il vetrino e lavare il vetrino 3 volte per 5 minuti ciascuno in 4x SSCT e poi3 volte per 5 minuti ciascuno in 0.1x SSC.
NOTA: diapositive possono essere lavati per periodi di tempo più lunghi.
- Rimuovere il vetrino e asciugare intorno il tessuto con un Kimwipe asciutto per rimuovere il tampone in eccesso (evitare di toccare il tessuto). Posizionare il lato tessuto diapositiva in un luogo buio per 10-15 minuti o fino a quando l'umidità si dissolve completamente.
- Dispensare 11 ml di mezzo di montaggio Vectashield (con 4 ', 6-diamidino-2-phenylindole-DAPI) sul tessuto. Posizionare accuratamente un coprioggetto pulito (non trattato con Sigmacote) direttamente sopra il centro del mezzo e tessuti di montaggio. Il mezzo di montaggio dovrebbe migrare lentamente verso l'esterno verso i bordi del vetrino.
NOTA: Se il mezzo di montaggio non riesce a raggiungere il bordo del vetrino su tutti i lati, poi un ulteriore 1-2 ml di mezzo di fissaggio può essere applicato a una posizione sul bordo del vetrino per riempire il volume necessario. In questo caso, assicurarsi di asciugare ogni eccesso di terreno dalla superficie di scorrimento primatenuta. - Sigillare i bordi del vetrino con smalto. Evitare la pittura lo smalto sul campione di tessuto.
- Posizionare il vetrino in posizione verticale in un luogo buio e lasciare il chiodo smalto asciutto fino a completa dura (di solito 30 minuti o più). A questo punto, il tessuto immagine o conservare a -20 ° C per 1 settimana per l'imaging tardi.
Buffer / Soluzione Ricette
10x PBS
- 80 g NaCl
- 2,0 g KCl
- 14,4 g Na 2 HPO 4
- 2,4 g KH 2 PO 4
- pH a 7,4, H 2 O a 1 L
1x PBT
- 5 ml 10x PBS
- 45 ml H 2 O
- 0,1% Tween 20
20x SSC
- 175,3 g NaCl
- 88.2 g Na citrato
- in 800 ml H 2 O
- pH 7, H 2 O a 1 L
4x SSCT
- 200 ml 20x SSC
- 799 ml H 2 O
- 0,1% Tween 20
0.1x SSC
- 5 ml 20x SSC
- 995 ml H 2 O
Mix di ibridazione (20 ml; modificato da 11)
- 10 microlitri formammide
- 4 ml 50% destrano solfato
- 2 ml 20x SSC
- 4 microlitri H 2 O
SBT 8 (10 ml)
- 2 ml 20x SSC
- 0,01 g albumina sierica bovina (BSA)
- 10 microlitri Tween 20
- 7,9 ml H 2 O
Soluzione Blocco 8 (10 ml)
- 0,3 g BSA
- 10 microlitri Tween 20
- 2 ml 20x SSC
- 8 ml H 2 O
Soluzione fissativa con paraformaldeide (1 ml)
- 393,75 ml H 2 O (aggiungere acqua prima)
- 450 ml di acido acetico glaciale
- 156.25 microlitri 16% paraformaldeide
Risultati
Per illustrare questo metodo, si ibridati una serie di piccoli oligonucleotidi sintetizzati in commercio che sono stati modificati chimicamente con coniugati fluorescenti (Figura 2) e una sonda più biotinilato (fatta attraverso la traduzione nick di un prodotto di PCR; Figura 2B), ai cromosomi da diversi tessuti differenti tipi (vedi tabella 1). Le sequenze bersaglio inclusi ripetizioni satelliti situati in pericentromeriche (eterocromatiche) regioni dei cromosomi mito...
Discussione
ISH DNA è spesso utilizzato per mappare le sequenze specifiche per i cromosomi. Abbiamo descritto un metodo semplice per ISH DNA ottimizzato per alto numero di copie, sequenze eterocromatiche. Invece di utilizzare lavaggi in una soluzione di formammide, che è un requisito in altri protocolli ISH DNA esistenti, poniamo vetrini montati tissutale direttamente su un blocco di pre-riscaldata per denaturare il DNA. Questo metodo aggira l'uso di grandi quantità di formammide. Un passo fondamentale per la produzione di s...
Divulgazioni
The authors declare that they have no competing financial or any other conflict of interest.
Riconoscimenti
We thank Zhaohua Irene Tang in the W. M. Keck Science Department for the use of her epifluorescence microscope and the Werren lab for donating Nasonia for dissections. This work was supported in part by an NIH-NRSA fellowship (5F32GM105317-02) to AML.
Materiali
| Name | Company | Catalog Number | Comments |
| Poly-L-lysine coated slides (regular slides also can be used) | Sigma Aldrich | ||
| Ultrafine tweezers (5 gauge) | Dumont | ||
| 22 x 22 mm cover slips | Fisher | Sigmacote-treated by immersion for 15 sec, blotting dry, and wiping away all traces of Sigmacote so that cover slip is clear | |
| Sigmacote | Sigma | ||
| Filter paper | 75 - 150 mm | ||
| Paraffin wax paper | |||
| Heat block with thermometer | |||
| Dry incubator | |||
| Razor blades | |||
| Humidity chamber | empty pipette tip box or Tupperware, lined with moistened paper towels or Kimwipes | ||
| Coplin jars | with slide grooves | ||
| Aluminum foil | |||
| Pasteur pipettes | |||
| 1.5 ml microfuge tubes | |||
| Nail polish | clear or colored | ||
| P20 micropipette and plastic tips | |||
| Paperclips | 20 - 25 standard metal paperclips linked to form a chain | ||
| Reagents | |||
| 16% EM grade paraformaldehyde | Electron Microscopy Reagents | ||
| Acetic acid | Sigma | ||
| Liquid nitrogen | |||
| 100% Ethanol, chemical grade | |||
| Commercially synthesized, fluorescently labeled oligos | |||
| Long biotinylated probe | Invitrogen; Alternative steps 2.7.1-2.7.3 | e.g., nick translated and biotinylated with BioNick from Invitrogen | |
| Rhodamine-Avidin | Roche; Alternative steps 2.7.1-2.7.3 | for detection of long biotinylated probe | |
| Hybridization buffer | Recipe above | ||
| 4x SSCT | Recipe above | saline-sodium citrate + Tween | |
| 0.1x SSC | Recipe above | saline-sodium citrate | |
| Blocking solution | Recipe above | ||
| SBT | Recipe above | SSC, bovine serum albumin, Tween | |
| 1x PBT | Recipe above | phosphate-buffered saline + Tween | |
| 1x PBS | phosphate-buffered saline | ||
| Hypotonic solution | 0.5% sodium citrate in H2O | ||
| Formamide | Sigma Aldrich | ||
| Vectashield mounting medium with DAPI | Vector laboratories | ||
Riferimenti
- Blattes, R., Kas, E. Fluorescent in situ hybridization (FISH) on diploid nuclei and mitotic chromosomes from Drosophila melanogaster larval tissues. Cold Spring Harbor Protocols. 2009 (9), (2009).
- Dimitri, P. Fluorescent in situ hybridization with transposable element probes to mitotic chromosomal heterochromatin of Drosophila. Methods in Molecular Biology. 260, 29-39 (2004).
- Pardue, M. L. In situ hybridization to polytene chromosomes in Drosophila using digoxigenin-labeled probes. Cold Spring Harbor Protocols. 2011 (8), 1003-1006 (2011).
- Hoskins, R. A., et al. Heterochromatic sequences in a Drosophila whole-genome shotgun assembly. Genome Biology. 3 (12), (2002).
- Hoskins, R. A., et al. Sequence finishing and mapping of Drosophila melanogaster heterochromatin. Science. 316 (58331), 1625-1628 (2007).
- Treangen, T. J., Salzberg, S. L. Repetitive DNA and next-generation sequencing: computational challenges and solutions. Nature Reviews Genetics. 13 (1), 36-46 (2012).
- He, B., et al. Mapping the pericentric heterochromatin by comparative genomic hybridization analysis and chromosome deletions in Drosophila melanogaster. Genome Research. 22 (12), 2507-2519 (2012).
- Pimpinelli, S., Bonaccorsi, S., Fanti, L., Gatti, M. Fluorescent in situ hybridization (FISH) of mitotic chromosomes from Drosophila larval brain. Cold Spring Harbor Protocols. 2010 (3), (2010).
- Larracuente, A. M., Noor, M. A., Clark, A. G. Translocation of Y-linked genes to the dot chromosome in Drosophila pseudoobscura. Molecular Biology and Evolution. 27 (7), 1612-1620 (2010).
- Ferree, P. M., Barbash, D. A. Species-specific heterochromatin prevents mitotic chromosome segregation to cause hybrid lethality in Drosophila. PLoS Biology. 7 (10), e1000234 (2009).
- Williams, B. C., Karr, T. L., Montgomery, J. M., Goldberg, M. L. The Drosophila l(1)zw10 gene product, required for accurate mitotic chromosome segregation, is redistributed at anaphase onset. The Journal of Cell Biology. 118 (4), 759-773 (1992).
- Gatti, M., Bonaccorsi, S., Pimpinelli, S. Looking at Drosophila Mitotic Chromosomes. Methods in Cell Biology. 44, 371-391 (1994).
- Werren, J. H., Stouthamer, R. PSR (paternal sex ratio) chromosomes: the ultimate selfish genetic elements. Genetica. 117 (1), 85-101 (2003).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon