Method Article
Base-Probe Real-time PCR Approcci per la misurazione quantitativa dei microRNA
In questo articolo
Riepilogo
MicroRNA circolanti sono recentemente emersi come biomarcatori promettenti e innovativi per vari tipi di cancro e di altre malattie. L'obiettivo di questo articolo è quello di discutere di tre diverse piattaforme di PCR real-time sonda-based e metodi che sono disponibili per quantificare e determinare l'abbondanza dei microRNA circolanti.
Abstract
Base-Probe PCR quantitativa (qPCR) è un metodo preferito per misurare trascrizione abbondanza, poiché è uno dei metodi di rilevamento più sensibili che fornisce un'analisi accurata e riproducibile. Chimica a base-Probe offre lo sfondo minimo di fluorescenza rispetto ad altri (di tipo dye) chimiche. Attualmente, ci sono diverse piattaforme disponibili che la chimica a base di probe uso per quantificare trascrizione abbondanza. qPCR in una piastra ben 96 è il metodo più utilizzato di routine, ma solo un massimo di 96 campioni o miRNA può essere testato in un unico passaggio. Questo richiede tempo e noioso se un gran numero di campioni / miRNA sono da analizzare. High-throughput piattaforme sonda-based come microfluidica (es TaqMan carte Array) e gli array nanofluidica (es OpenArray) offrono facilità per rilevare riproducibile ed efficiente l'abbondanza di più microRNA in un gran numero di campioni in breve tempo. Qui, dimostriamo il setup sperimentalend protocollo per miRNA quantificazione da siero o campioni di plasma EDTA, utilizzando la chimica probe-based e tre piattaforme differenti (96 pozzetti, microfluidica e array nanofluidica) offrono livelli crescenti di throughput.
Introduzione
I microRNA (miRNA) sono ~ 22 nucleotidi non codificanti (nc) RNA, funzionante come regolatori dell'espressione genica 1-3. La maggior parte dei miRNA negli animali funzionano attraverso specifiche-sequenza di basi accoppiamento con un mRNA, targeting 3 'UTR, che porta a regolazione negativa dell'espressione genica 2-4. Questo di solito avviene attraverso l'inibizione della traduzione mRNA o ribosomiale drop-off. miRNA in circolazione hanno dimostrato di essere nuovi biomarcatori nella ricerca e campo clinico per una varietà di malattie, come il diabete 5-7, ovaie 8, 9 prostata e della mammella 10,11, l'epatite B 12 e di altre malattie autoimmuni 13. La ricerca è stata condotta per identificare miRNA abbondanti in cellule o tessuti differenti, così come in circolazione dal plasma umano e campioni di siero, che è più accessibile e meno invasivo 9,11-15.
Diversi metodi di quantificazione miRNA sono stati estituito utilizzando più piattaforme, come ad esempio la piattaforma piastra standard 96 pozzetti 4,12,16-18, la piattaforma di carta microfluidica 12,18-23 e la piattaforma nanofluidica matrice 17,24. Quantitativa real-time PCR (qPCR) offre la possibilità di misurare i numeri relativi o assoluti di trascrizioni con multipla (di coloranti o sonda-based) chimiche. Base-Probe real-time PCR chimica offre il vantaggio di sfondo bassa fluorescenza e alta sensibilità per rilevare una singola copia trascrizione. E 'relativamente conveniente, semplice da usare e altamente riproducibile, che lo rende un metodo preferito per quantificare e determinare miRNA 25. Metodo qPCR base-Probe prevede generalmente due fasi: trascrizione inversa (RT) e qPCR 4,26,27. RT è dove il primer stem-loop RT è ibridato ad una molecola di miRNA maturo o primario e convertito complementare (c) del DNA. La quantificazione del prodotto cDNA viene quindi effettuata utilizzando miRNA-specifici primer PCR26-28. Il principio di base di sonda qPCR è basato sul rilevamento di estensione filamento complementare in tempo reale, che comporta l'idrolisi della sonda fluorescente-tag. Queste sonde sono progettate per contenere un reporter fluorescente e un quencher che sono solo parte per consentire FRET (Fluorescence Resonance Energy Transfer). Rilevamento delle emissioni dal giornalista di fluorescenza (emettitore) è mascherato dalla vicinanza della molecola quencher. Quando Taq polimerasi (pol) si estende dal primer monte e raggiunge una forcella (estremità 5 'della sonda), la Taq attività esonucleasi pol idrolizza la sonda, portando ad una fisica di dissociazione / separazione dell'emettitore fluorescente dal quencher. Questa versione di una singola molecola di fluorescenza emettitore è registrato dal rivelatore e presentato come un aumento incrementale nel segnale di fluorescenza da tale bene / reazione. L'aumento della fluorescenza è proporzionale alla quantità di prodotto di PCR generato, permettendo una accurata quantification del 26,28 bersaglio amplificato.
Con l'aumento della domanda in miRNA quantificazione, medio tecnologie high-throughput sono stati sviluppati per consentire a un maggior numero di campioni da elaborare in un breve lasso di tempo. TaqMan Low Density Array (TLDA) è un mezzo throughput microfluidica disegno innovativo basato su sonda basata qPCR chimica offrendo un aumento del numero di miRNA analizzati su una piastra. TLDA comporta l'uso di una piscina predefinito di RT-primers utilizzati per sintetizzare il cDNA. Questi cDNA vengono poi filata in una misura 384 carta e micro-fluidico per determinare l'espressione di più miRNA utilizzando qPCR 22,26,29. Ogni pozzetto della card contiene asciugato primer e sonde per amplificare specifici miRNA (s), quindi fino a 384 reazioni possono essere trasformati in carta singola TLDA 26.
L'array nanofluidics è una piattaforma ad alta produttività che viene utilizzato per il rilevamento di trascritti genici 24 utilizzando la stessa chimica probe-based. Si utilizza una matrice proprietaria offre interazioni idrofobiche-idrofilo per facilitare il caricamento della miscela di reazione 33 nanolitri in una matrice di 3072 fori passanti su uno scivolo 24 in acciaio inossidabile. Questo articolo si concentra su che dimostra come vengono eseguiti questi metodi per quantificare miRNA nel siero / plasma ed i fattori critici che devono essere considerati quando si esegue e interpretare tali dati. Preso di conto, i loro benefici individuali e limitazioni saranno discussi in questo articolo.
Protocollo
RNA totale può essere isolato dal siero utilizzando un protocollo stabilito nel nostro laboratorio 30 o usando altri kit disponibili in commercio.
Sono forniti fogli di calcolo interattivi supplementari per il calcolo dei volumi di reazione in ogni esperimento (con il volume eccedente 5% rappresentato pipettaggio inclusi): NOTE.
1. base Probe-Real-time qPCR utilizzando uno standard 96 pozzetti Piattaforma Piatto
- sintesi del DNA (trascrizione inversa) su campioni di RNA siero / plasma di miRNA
- Calcolare e togliere 10 ng di RNA per ogni reazione sintesi del DNA. Aggiungere acqua priva di nucleasi per portare il volume finale di 10 ng di RNA a 1,67 microlitri (per reazione 5 microlitri). I campioni in ghiaccio.
- Selezionare il miRNA e scongelare i loro primer RT.
- Scongelare i componenti della miscela di reagenti RT: RT Buffer (10X), dNTP (100 mm), RNase Inhibitor (20 U / ml). Non tenere mai posto enzima (Moloney virus della leucemia murina ricombinante(RMoMuLV)) (recombinanat Moloney virus della leucemia murina (rMoMuLV) trascrittasi inversa (50 U / ml) su ghiaccio. Conservare a -20 ° C fino al momento o in un blocco freezer.
- Preparare RT miscela reagente su ghiaccio come descritto nella tabella 1A. Si raccomanda di preparare almeno 5% volume in eccesso per compensare l'errore di pipettamento.
- Aggiungere 2,33 ml di miscela reagente RT ciascuno in un 0,2 ml provetta PCR / piastra.
- Vortex i tubi di primer RT per mescolare, poi brevemente centrifugare a 10.000 g per 10 sec. Aggiungere 1 ml di miRNA specifico fondo RT alle rispettive provette PCR o la piastra. Aggiungere 1,67 ml di RNA diluito nelle rispettive provette PCR o pozzetti nella piastra. Eseguire tutte le aggiunte su ghiaccio.
- Centrifugare la provetta o piastra a 1.950 xg per 5 minuti a 4 ° C.
- Impostare il programma di sintesi miRNA cDNA sul termociclatore con le seguenti condizioni di impostazione: 16 ° C per 30 minuti, 42 ° C per 30 minuti, 85 °C per 5 minuti e 4 ° C in attesa.
- Impostare il volume di reazione di 10 ml. Caricare le provette di reazione o la piastra in cicli termici. Inizia la corsa RT. Conservare le reazioni cDNA (RT) a -20 ° C, se il tempo reale amplificazione PCR non è immediatamente proceduto.
- Probe-based in tempo reale qPCR per la rilevazione dei miRNA maturi
- Scongelare i saggi selezionati sonda-based qPCR (20X) per il rispettivo prodotto RT.
- Mescolare la soluzione esclusiva proprietà di real-time PCR (qPCR mix reattivo) agitando la bottiglia.
- Preparare la miscela reagente qPCR per ogni miRNA da analizzare. Per preparare la qPCR ottenere una provetta sterile da 1,5 ml microcentrifuga per ciascun campione miRNA e aggiungere i componenti in ciascun tubo come descritto in Tabella 1B. Si raccomanda di preparare almeno 5% volume in eccesso per compensare l'errore di pipettamento.
- Aggiungere 4,2 ml di rispettiva miscela reagente qPCR a ciascun pozzetto di una piastra a 96 pozzetti ottico (o tubi). Aggiungere 0,8 microlitridi rispettiva reazione cDNA (sintetizzato nel passo 1.1 per ogni miRNA) al rispettivo bene.
- Sigillare la piastra con adeguata copertura ottica. Centrifugare la piastra (o tubi) a 1.950 xg per 5 minuti a 4 ° C
- Accendere il computer, 96 pozzetti lettore matrice piatto / microfluidica e, infine, avviare il software. Assicurarsi che le macchine sono collegate correttamente e corretta blocco e coperchio riscaldato (per un veloce 96 pozzetti) sono a posto.
- Selezionare esperimento come 96 pozzetti blocco veloce (0,1 ml), curva standard, reagenti TaqMan e modalità veloce. Impostare qPCR sul sistema Real Time PCR usando le seguenti condizioni programma ciclismo: 95 ° C per 20 sec, 50 cicli (95 ° C per 1 sec, 60 ° C per 30 sec).
- Impostare il volume di reazione di 10 ml. Caricare il tubo di reazione o piastra nello strumento. Premere "Start run". Il programma richiede circa 1 ora per completare.
2. Scheda Microfluidics Array basato Probe-(Card A e B)
NOTA: base-Probe panel miRNA si presenta come un insieme di due schede 384 pozzetti microfluidica (scheda Array A e carta Array B). Ogni scheda contiene Primer secchi e sonde per un massimo di 380 miRNA e controlli. prodotto cDNA (con o senza pre-amplificazione) specifico per carta A o B carta viene caricata sulla rispettiva disposizione per PCR in tempo reale.
- Trascrizione inversa (RT)
- Utilizzare questo protocollo per un ingresso RNA totale di 1-1,000 ng. Se l'ingresso è tra 1-350 ng, eseguire una pre-amplificazione (pre-amp) passo. Per gli ingressi superiori a 350 ng, caricare cDNA direttamente sulle schede di matrice senza pre-amp.
NOTA: Per miRNA siero profiling, iniziare con 100 ng di RNA totale. Per un profilo completo miRNA, eseguire due piscine predefiniti di set RT-innesco di reazioni RT (Pool A e Pool B) per campione. - Scongelare i seguenti reagenti sul ghiaccio. RT Primer (10x): Pool A e Pool B, dNTP con dTTP (100 mm), RT Buffer (10x), MgCl 2 (25 mm), RNase Inhibitor (20 U / &# 181; l). Non tenere l'enzima (ricombinante Moloney virus della leucemia murina (rMoMuLV)) trascrittasi inversa (50 U / ml) su ghiaccio. Conservare a -20 ° C fino al momento dell'uso.
- Vortice delicatamente tutti i reagenti, tranne l'enzima, e quindi centrifugare brevemente le provette a 10.000 xg per 10 sec.
- Combinare reagenti, come descritto nella Tabella 2A, in due tubi; un tubo per Gruppo A, l'altra per Pool B. Ciascun tubo contiene solo una piscina predefinito di RT Primer set, sia dal Gruppo A o dal Pool B. Si raccomanda di preparare almeno 5% volume in eccesso per compensare l'errore di pipettamento .
- Miscelare, e brevemente poi centrifugare a 10.000 g per 10 sec. Aliquota 100 ng di ciascun campione di RNA in un nuovo tubo, quindi aggiungere un volume adeguato di acqua priva di nucleasi per un totale di 3 ml.
- Aliquota 4,5 ml di adeguato mix di reagente RT in rispettive provette. Miscelare, e quindi centrifugare brevemente a 10.000 xg per 10 sec. Incubare in ghiaccioper 5 min.
- Posizionare i campioni in un termociclatore e avviare il RT utilizzando le seguenti condizioni: 40 cicli (16 ° C per 2 min, 42 ° C per 1 min, 50 ° C per 1 sec), 85 ° C per 5 minuti, mantenere a 4 ° C. Conservare cDNA generato utilizzando questi piscina predefinito di RT-primer a -15 a -25 ° C o utilizzato immediatamente.
- Utilizzare questo protocollo per un ingresso RNA totale di 1-1,000 ng. Se l'ingresso è tra 1-350 ng, eseguire una pre-amplificazione (pre-amp) passo. Per gli ingressi superiori a 350 ng, caricare cDNA direttamente sulle schede di matrice senza pre-amp.
- Pre-amplificazione
- Scongelare il pool predefinito di primer pre-amp su ghiaccio. Delicatamente primer vortice, e poi brevemente le centrifugare a 10.000 g per 10 sec. Swirl TaqMan PreAmp mix di reagenti (2X) picchiettando leggermente, a mescolare.
- Combinare reagenti, come descritto nella Tabella 2B, in due tubi; un tubo per Gruppo A, l'altro per Pool B.
NOTA: Come descritto in precedenza, Piscina Una pre-amp primer andranno a tubo A e Pool B primer dovrebbero andare a tubo B. Si raccomanda di preparare almeno il 5% del volume in eccesso per compensare l'errore di pipettamento. - Miscelare, e quindi centrifugare brevemente a 10.000 xg per 10 sec.Aliquota 22,5 ml di miscela reagente PreAmp adeguato in nuovi tubi. Aliquotare 2,5 ml di campione cDNA (preparato nella fase RT) nel rispettivo tubo.
- Miscelare, e quindi centrifugare brevemente a 10.000 xg per 10 sec. Incubare in ghiaccio per 5 minuti. Mettere i campioni in un termociclatore e avviare il ciclo di pre-amp. Condizioni bicicletta 95 ° C per 10 min, 55 ° C per 2 min, 72 ° C per 2 min, 12 cicli (95 ° C per 15 sec, 60 ° C per 4 min), 99,9 ° C per 10 min, tenere a 4 ° C.
- Inverti pre-amplificato cDNA per mescolare, e poi brevemente centrifugare a 10.000 g per 10 sec. Aggiungere 75 ml di 0.1x TE Buffer (pH 8,0) pre-amplificato cDNA (1: 4 di diluizione). Invertire diluiti campioni pre-amp per mescolare, e quindi centrifugare brevemente a 10.000 xg per 10 sec. Conservare diluiscono pre-amplificato cDNA a -15 a -25 ° C per un massimo di una settimana, o utilizzato immediatamente.
- Caricamento carte microfluidica e performante qPCR
- Tenere il microfluidicaCarte di miRNA fuori per almeno mezz'ora a temperatura ambiente. Scongelare diluito cDNA pre-amplificata su ghiaccio e mescolare invertendo i tubi seguiti da breve rotazione. Mescolare qPCR mix reagenti agitando la bottiglia.
- Nel nuovo tubo aggiungere 450 ml di qPCR mix di reagente a 9 ml di diluito cDNA pre-amplificata. Per la scheda A, utilizzare pre-amplificata prodotto preparato con fondo Pool A. Aggiungere 441 ml di acqua priva di nucleasi per compensare il volume finale a 900 ml. Capovolgere la provetta per mescolare, e quindi centrifugare brevemente a 10.000 xg per 10 sec.
- Rimuovere la carta TLDA dalla confezione (una volta raggiunta la temperatura ambiente) e posizionarlo su una superficie pulita con il lato foglio verso il basso. Aggiungere 100 pl di miscela di reazione PCR in ciascuna delle 8 porte di riempimento sulla scheda. Ci sono 2 porte su ciascuno dei serbatoio (8 serbatoi in totale su ogni carta).
NOTA: La porta di riempimento è il foro più grande, dove viene aggiunta la miscela di reazione, mentre la porta di sfiato è il foro più piccolo. Ogni carta miRNA TLDA ha 8 serbatoios, ciascuna portando a 48 pozzetti (24 pozzetti in una colonna x 2 colonne), caricando tutti i 384 pozzi nella scheda con la stessa miscela di reazione PCR. Assicurarsi che Pool A e Pool B di ogni campione sono caricati sulle rispettive carte TLDA. Posizionare la carta matrice nelle specializzati matrice microfluidica secchi titolare della carta nella centrifuga. - Per le carte di matrice microfluidica, utilizzare una centrifuga idonea (come Heraeus Multifuge 3SR, 230V centrifuga) e secchi specifici (come ad esempio i titolari di carte "Array TaqMan"). Ogni secchio può contenere fino a 3 schede (caricato / vuoto). Verificare sempre che tutti e 3 gli slot di un secchio sono occupati e il secchio è bilanciato mettendo bucket simile (questo secchio dovrebbe contenere anche 3 carte, vuote o piene) nella fessura opposta della centrifuga. Pur ponendo la carta nel supporto secchio, assicurarsi che gli 8 serbatoi proiettano verso l'alto e pozzetti di reazione di fronte la parete esterna di centrifuga.
- Spin carte a 331 xg per 1 minuto a temperatura ambiente. Dopoprimo giro, aprire la centrifuga e verificare visivamente la miscela di reazione è stato erogato attraverso i 384 pozzi. Ripetere il lancio a stesse impostazioni per 1 più tempo. Rimuovere la carta dal secchio e garantire che il livello della miscela di reazione in ciascuna delle 8 serbatoi è uniforme. Eventuali incoerenze nei volumi di liquido lasciato nei serbatoi rendono la scheda inappropriato utilizzare ulteriormente.
- Carte di matrice microfluidica bisogno di un sigillante specializzata che ha un gruppo di precisione stilo (trasporto) per sigillare i canali di distribuzione del fluido della matrice e distribuire equamente la miscela di reazione in tutti i pozzetti (volume totale di reazione 1 ml / pozzetto).
- Portare il carrello nella posizione di partenza e inserire la scheda caricato nel lato sigillante con pellicola e allineati ai pin stilo sul sigillante. In un colpo uniforme lento, costante e singolo, spingere il carrello attraverso la carta fino a raggiungere il punto finale del sigillante. Rimuovere la carta matrice sigillato e poi tagliare il serbatoios dalla scheda con le forbici.
- Assicurarsi che il vettore blocco corretto, coperchio riscaldato e il campione viene installato in tempo reale matrice microfluidica sistema PCR / macchina.
- Accendere il computer, quindi il sistema di allineamento microfluidica e, infine, avviare il software. Assicurarsi che le macchine siano collegati correttamente. Selezionare esperimento come carta matrice, curva standard, reagenti TaqMan e modalità standard. Importare i file di installazione per una delle carte di A e B. carta Salvare il file.
- Posizionare la carta nel vassoio sigillato strumento pozzetto A1 nell'angolo superiore sinistro e codice a barre verso la parte anteriore dello strumento. Premere il tasto "Start Esegui". Il programma richiede circa 2 ore per completare.
Base-Probe 3. Panel Nanofluidics miRNA umano
- Trascrizione inversa (RT)
- Utilizzare questo protocollo per un ingresso RNA totale di 50-200 ng, ma 100 ng è ottimale per la maggior parte dei campioni. Per un profilo completo miRNA, eseguire due predefipiscine NED di serie RT-innesco di reazioni RT (Pool A e Pool B) per campione.
- Scongelare i reagenti su ghiaccio: piscina predefinito di serie RT Primer (10x), dNTP con dTTP (100 mm), RT Buffer (10x), MgCl 2, (25 mm), RNase Inhibitor (20 U / ml). Non tenere l'enzima (ricombinante Moloney virus della leucemia murina (rMoMuLV) trascrittasi inversa (50 U / ml) su ghiaccio. Conservare a -20 ° C fino al momento dell'uso. Vortex delicatamente tutti i reagenti, tranne l'enzima, e brevemente li centrifugare a 10.000 xg per 10 sec.
- Combinare reagenti, come descritto nella Tabella 3A, in due tubi; un tubo per Gruppo A, l'altra per Pool B. Ciascun tubo contiene solo una piscina predefinito di set RT-primer, sia dal Gruppo A o dal Pool B. Si raccomanda di preparare almeno 5% volume in eccesso per compensare pipettaggio errore.
- Pipetta per mescolare e poi brevemente centrifugare a 10.000 g per 10 sec. Aliquotare 100 ng di ciascun campione di RNA in una nuova provetta, quindi aggiungere un volume appropriatoacqua priva di nucleasi per un totale di 3 microlitri. Aliquota 4,5 ml di adeguato mix di reagente RT nel rispettivo tubo.
- Miscelare, e quindi centrifugare brevemente a 10.000 xg per 10 sec. Incubare in ghiaccio per 5 min. Mettere i campioni in un termociclatore e avviare il programma RT. Condizioni bicicletta 40 cicli (16 ° C per 2 min, 42 ° C per 1 min, 50 ° C per 1 sec), 85 ° C per 5 minuti, mantenere a 4 ° C. Conservare cDNA generato con questi piscina predefinito di RT-primer fissato a -15 a -25 ° C o utilizzare immediatamente.
- Pre-amplificazione (pre-amp)
- Scongelare il pool predefinito di pre-amp set di primer sul ghiaccio. Delicatamente primer vortice, poi brevemente le centrifugare a 10.000 g per 10 sec. Swirl TaqMan PreAmp mix di reagenti (2X) per miscelare.
- Combinare reagenti, come descritto nella Tabella 3B, in due tubi; un tubo per Gruppo A, l'altra per Pool B. Ciascun tubo contiene solo una piscina predefinito di insiemi RT-primers, sia from Pool A o dal Pool B. Si raccomanda di preparare almeno il 5% del volume in eccesso per compensare pipettaggio errore.
- Pipetta per mescolare, e poi brevemente le centrifugare a 10.000 g per 10 sec. Aliquota 22,5 ml di miscela reagente PreAmp adeguato in nuovi tubi. Aliquotare 2,5 ml di campione di cDNA nel rispettivo tubo. Miscelare, e quindi centrifugare brevemente le provette a 10.000 xg per 10 sec. Incubare in ghiaccio per 5 min.
- Mettere i campioni in un termociclatore ed eseguire il pre-amp. Impostare il volume di reazione di 25 microlitri. Condizioni bicicletta 95 ° C per 10 min, 55 ° C per 2 min, 72 ° C per 2 min, 12 cicli (95 ° C per 15 sec, 60 ° C per 4 min), 99,9 ° C per 10 min, tenere a 4 ° C.
- Inverti pre-amplificato cDNA per mescolare, e poi brevemente centrifugare a 10.000 g per 10 sec. In nuovi tubi aggiungere 4 ml di pre-amplificata cDNA a 156 ml di 0.1x TE tampone pH 8.0 (01:40 di diluizione). Invertire diluiti campioni pre-amp per mescolare, e poi brevemente centrifuge a 10.000 xg per 10 sec. Conservare diluito e diluito cDNA pre-amplificato a -15 a -25 ° C per un massimo di una settimana, o usare immediatamente.
- Caricamento nanofluidica Array e Performing qPCR
- Scaricare il file corrispondente piastra (.tpf) dal sito 31 con il numero di serie di diapositive di matrice nanofluidica. Contiene le informazioni di corsa per la specifica diapositiva matrice nanofluidica.
- Scongelare diluito cDNA e TaqMan Real-Time qPCR mix reagente pre-amplificata (se si utilizza per la prima volta) sul ghiaccio. Mescolare qPCR mix reagenti agitando la bottiglia. In nuovi tubi aggiungere 22,5 ml di qPCR mix di reagente a 22,5 ml di diluito cDNA pre-amplificata. Delicatamente vortice di mescolare, e quindi si centrifuga brevemente a 10.000 xg per 10 sec.
- Aliquota 5 ml di ciascun campione in 8 pozzi (2 colonne, 4 righe) della piastra del campione 384 pozzetti workflow nanofluidica array. Assicurarsi che Pool A e Pool B di ogni campione sono in blocchi di 8 pozzetti adiacenti (vedi Figure 1 o complementari excel foglio per il layout).
- Ogni piatto del campione può contenere fino a otto diapositive valore dei campioni. Tuttavia, il sistema per nanofluidica array può elaborare solo 4 array nanofluidica in un'unica seduta. Se più di quattro diapositive vale la pena di campioni devono essere caricati su un piatto del campione, assicurarsi che le restanti sezioni sono sigillati. Sigillare con piastra campione OpenArray sigillante.
NOTA: Si consiglia di ritagli il sigillante nelle sezioni necessarie, quindi le sezioni può essere sigillata / sigillata singolarmente per ridurre l'evaporazione. In alternativa, la piastra può essere sigillato con un sigillante intatta, quindi le sezioni può essere tagliato singolarmente fuori durante il caricamento.
- Ogni piatto del campione può contenere fino a otto diapositive valore dei campioni. Tuttavia, il sistema per nanofluidica array può elaborare solo 4 array nanofluidica in un'unica seduta. Se più di quattro diapositive vale la pena di campioni devono essere caricati su un piatto del campione, assicurarsi che le restanti sezioni sono sigillati. Sigillare con piastra campione OpenArray sigillante.
- Piastra Centrifugare a 490 xg per 1 min a 4 ° C. Caricare i nanofluidica diapositive matrice entro 1 ora. A causa del tempo limitato permesso di sigillare le diapositive, si prega di caricare solo una diapositiva alla volta. Rimuovere il vetrino nanofluidica matrice dal congelatore e lasciarlo venire a temperatura ambienteerature (~ 15 min).
- Assicurarsi che il vettore blocco corretto, coperchio riscaldato e il campione è installato nel sistema nanofluidics array. Accendere il computer e sistema di PCR in tempo reale e il sistema di caricamento. Accedi al rispettivo software e assicurarsi che le macchine sono collegate. Rimuovere i materiali di consumo del sistema di carico (Array coperchio scorrevole, spina e fluidi ad immersione) dalla confezione.
- Tirare delicatamente lo stantuffo della siringa del liquido di immersione per allentare. Togliere il tappo, inserire punta su e aria filo dalla punta. Posizionare le punte del sistema di carico all'interno della macchina e rimuovere il coperchio. Porre piatto del campione all'interno del sistema PCR.
- Mettere guanti. Assicurarsi che siano a tenuta perfetta per ridurre al minimo il rischio di marcatura accidentalmente il coperchio scorrevole. Imballaggi slide aperto con cautela. Lentamente punta scivolare in mano. Non toccare la parte superiore della diapositiva.
- Posizionare diapositiva nel sistema PCR, con il codice a barre sulla sinistra. Rimuovere sigillante dalla porzione della piastra campione destinato carico. Utilizzare il sistema di caricamentosoftware per immettere il codice a barre di scorrimento, spostare la posizione, la posizione del campione e la configurazione punta.
- Quando tutti i controlli necessari siano completati, scivolo di carico premere. Mentre il sistema PCR sta caricando la diapositiva, rimuovere la plastica trasparente e rosso dalla parte inferiore del coperchio scorrevole. Al termine di carico, rimuovere con attenzione e sigillare il vetrino entro 90 secondi.
- Posizionare il vetrino entro il morsetto piatto. Mettere il coperchio vetrino sul vetrino. Pinza per 30 sec. Assicurarsi che il coperchio è posizionato in modo che il codice a barre viene visualizzato correttamente. Rimuovere il gruppo dal morsetto piatto.
- Posizione immersione siringa del liquido all'interno della diapositiva modo che la punta sta premendo contro il coperchio. Lentamente riempire scivolo di liquido ad immersione, garantendo le piste fluide lungo il coperchio. Una volta pieno, sigillare il vetrino con il tappo, girare la vite fino a quando la maniglia si stacca.
- Rimuovere il coperchio di plastica sulla parte superiore del coperchio scorrevole, e poi posizionare accuratamente nel supporto scorrevole del sistema PCR in tempo reale. Assicurarsi che ci sia suPport sul fondo della diapositiva mentre viene abbassato, in modo che non scende improvvisamente, e non toccare la parte superiore della slitta. E 'OK per toccare i lati del sistema / cassette.Initialize PCR diapositiva e avviare il programma per qPCR entro 1 ora.
- Selezionare "OpenArray" all'interno del software di PCR-sistema. Premere "Trovare ID diapositive". Questo richiederà un paio di minuti. Se il software non riesce a trovare l'ID piatto, chiederà che venga inserito manualmente.
- Premere il tasto "Conferma Centri piastra". Ancora una volta, questo richiederà un paio di minuti. Verificare che il punto rosso è all'interno del centro e che non ci siano impronte / segni sulla parte superiore della slitta. Caricare il rispettivo file .tpf per ogni diapositiva e specificare il nome del file risultato e posizione. Premere il tasto "Start Esegui". Il programma richiede circa 2 ore per completare.
Risultati
Il volume consigliato per un saggio basato sonda miRNA reazione qPCR è di 20 ml. NOTA: Abbiamo confermato che un volume di reazione di 5 microlitri può produrre risultati simili a quelli ottenuti usando 20 ml di volume 4,7,30. Abbassando il volume di reazione di 5 microlitri consente una diminuzione del 75% dei costi dei reagenti senza perdita apprezzabile sensibilità. Come illustrato nella figura 2, volumi di reazione di 20 microlitri e 5 microlitri mostrano una forte co-relazione entro 39 cicli (con r 2 di 0,92, p = 0,0002).
Matrice Microfluidics fornisce uno strumento per ottenere dati su 754 miRNA espressi in un campione di circa 5 ore (per la scheda A e Scheda B), che è un modo più efficiente di analizzare più campioni rispetto ai tradizionali 96 PCR pozzetti. Abbiamo confrontato miRNA carte di array microfluidica per lo stesso campione (campione A e campione A ripetizione) Figura 3A -. B mostra un grafico Bland-Altman ( 3A) e Correlazione plot (3B) per tutti i 754 miRNA testati per questi campioni. Ci sono 3 differenti miRNA controllo (U6, RNU44 e RNU48) disposte casualmente su entrambe le schede (scheda A e B) in più posizioni. Quando i valori di soglia ciclo U6 (R) sono confrontati tra le 2 piste, non abbiamo osservato differenze significative tra i valori (Tabella 4). E 'anche importante notare qui che U6 è espresso in maggiore abbondanza (valore Ct inferiore) nel campione valutato. Abbiamo quindi confrontato tutti i miRNA che hanno valori Ct tra 0-19,99 in entrambe le corse (n = 150), che avevano simile espressione dei miRNA nel complesso con un coefficiente di determinazione del 98% (Figura 3C-D). Di tutti i 277 miRNA che hanno valori Ct tra 20 e 29.99 in entrambe le corse, 16 miR differivano significativamente tra l'originale e la ripetizione piste (Figura 3E - F) Il numero di miRNA con differenza significativa betwee.n le corse sono aumentate (89 su 327) in cui sono stati selezionati i valori Ct tra 30-40 per entrambe le corse (Figura 3G - H).
La piattaforma nanofluidics matrice fornisce dati per 754 miRNA da ciascun campione di siero / plasma testati, come rappresentato nella Figura 4A È importante esaminare queste curve di amplificazione -. Come è con tutto qPCR - garantire che il risultato è indicativo della vera amplificazione. Ognuna delle 48 sottoarray (figura 1), contiene anche un saggio per i tre più popolari "pulizia" ncRNAs:. U6, RNU44 e RNU48 figura 4B mostra un raggruppamento tipico di U6 replica da un unico campione. Queste repliche mostrano bassa deviazione standard (SD <0,5) e quindi sono un indicatore di affidabilità. In alternativa, la figura 4C mostra la maggiore variabilità dei replicati U6 (SD> 0,5) in un secondo campione. Questo non nega la validità of rimanenti saggi, sebbene non richiedere una critica più approfondita. U6, come con la maggior parte dei miRNA "pulizia" nei fluidi biologici, può avere una espressione variabile. Va notato che uno dei campioni, illustrate nella Figura 4C, visualizza 4 volte minore contenuto U6 quello illustrato nella figura 4B. Poiché il livello di U6 in campione presentato in 4C è del 75% inferiore a cominciare da quello presentato nel 4B pannello, maggiore variabilità tecnica è previsto a causa della distribuzione di Poisson di trascritti, aggravata dal piccolo volume di reazione 17.
Un altro strumento utile è le immagini di controllo di qualità (QC), disponibili per l'esportazione una volta che la corsa è stata completata. Una lista di questi utilizza la fluorescenza ROX, colorante passivo trovato nella miscela reagente qPCR, per confermare che ciascun foro passante è stato caricato correttamente (Figura 5). Un foro passante, o addirittura un en pneumatici subarray, non può caricare a causa di volume del campione insufficiente, l'evaporazione, bolle presenti nei pozzetti della piastra del campione 384-bene, la mancata rimuovere completamente il sigillo piatto del campione, o difetti nel sistema Accufill o dei suoi suggerimenti. Qualsiasi scaricati fori passanti devono essere identificati per evitare miRNA etichettatura come "non rilevabile", quando in realtà il test è stato mai caricato. Se si verifica questo problema, verificare che almeno 5 ml di campione / mastermix viene caricato in ciascun pozzetto della piastra campione di 384 pozzetti, piatto del campione sia correttamente centrifugato prima del caricamento, il sigillo di alluminio è completamente rimosso, e il OpenArray caricata scivolo è sigillata ed eseguire entro il tempo previsto per tutte le esecuzioni successive. Se i problemi di carico persistono, questi possono essere più probabile di pertinenza di gruppo specifico o sacco di array o di consumo correlati e in seguito si debba sollecitare assistenza attraverso il produttore.
"Src =" / files / ftp_upload / 52586 / 52586fig1.jpg "/>
Figura 1: Layout di campioni per il flusso di lavoro Array Nanofluidics: (A) Ogni piatto del campione da 384 e può contenere i campioni per un massimo di 8 matrici nanofluidica. (B) diluito, pre-amplificato cDNA viene inserito in 8 pozzi (2 colonne di 4 righe), con Pool A e Pool B in gruppi da 8 e adiacenti. Ogni cerchio rappresenta un pozzetto. (C) Ciascun pozzetto della piastra campione verrà caricato in uno sottomatrice dell'array nanofluidics. Ogni piccolo quadrato rappresenta una subarray.
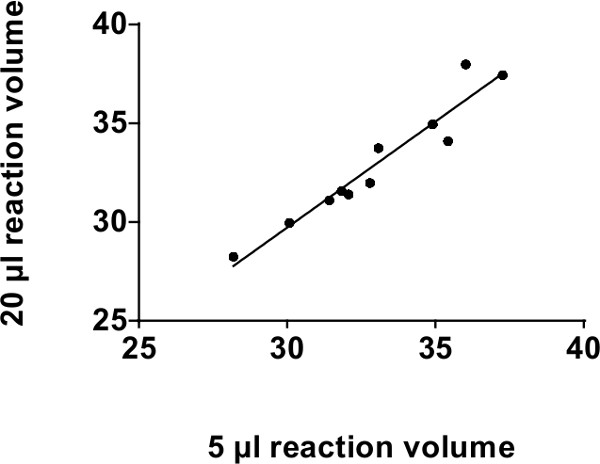
Figura 2: Analisi Co-relazione per convenzionali 96 pozzetti piattaforme PCR: Co-relazione tra 20 ml e 5 volumi di reazione microlitri di TaqMan Real-time qPCR utilizzando una piattaforma standard 96 pozzetti in valori CT (39 cicli). Abbiamo confrontato 4 microRNA differenti (miR-375, miR-30c, miR-30d e miR-7) in 4 diversi campioni di siero e plasma umano. Solo 11 punti dati vengono tracciati dal momento che le altre erano rilevabili. R 2 = 0,92, p = 0,0002.
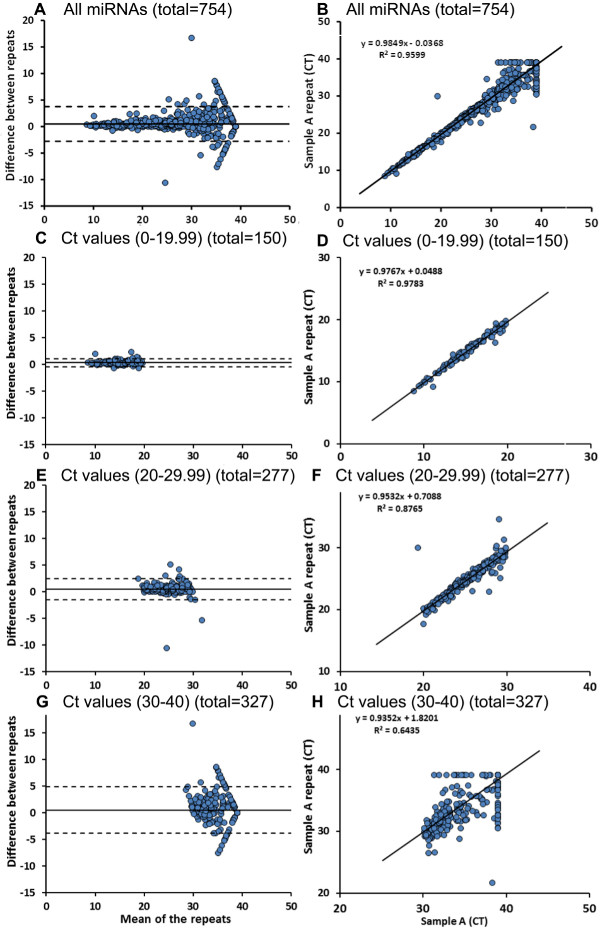
. Figura 3: circolazione miRNA profiling utilizzando carte di microfluidica array utilizzando 2 schede microfluidica (carta-A e carta-B), viene generato un profilo di 754 miRNA (A - B). Come mostrato qui, abbiamo usato lo stesso campione per 2 array di microfluidica corre per verificare la riproducibilità dei risultati delle carte microfluidica. Abbiamo osservato una simile espressione dei miRNA nel complesso, con valori Ct tra 0-19,99 (C - D). Ci sono pochi miRNA (16 su 277), con valori Ct tra 20-29,99 e significative differenze tra piste di ripetizione (E - F). L'ottanta nove di 327microRNA con valori Ct più alti (30-40) hanno mostrato differenze significative tra le due piste (G - H). I dati vengono analizzati utilizzando il test T abbinato. Cliccate qui per vedere una versione più grande di questa figura.
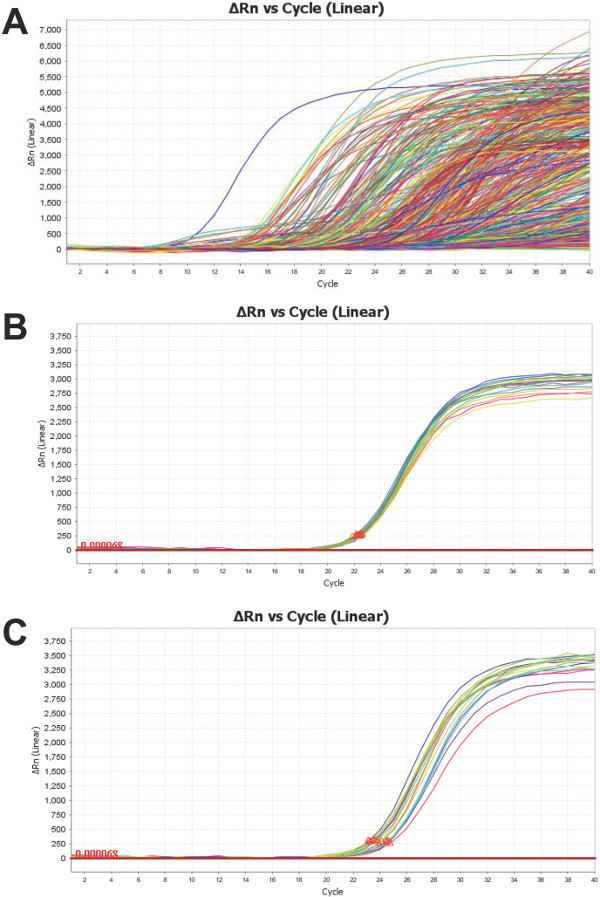
Figura 4: Profilo Rappresentante di curve PCR prodotto di amplificazione: è una figura rappresentativa delle curve (A) di amplificazione combinati di tutti i bersagli miRNA per un campione di plasma umano. Un test per U6 (a comune controllo ncRNA) è posto in ogni subarray. Il campione in (B) dimostra bassa variabilità (SD <0,5), mentre (C) mostra alta deviazione standard (SD> 0.5) entro U6 replica. Entrambi i campioni sono total RNA isolato dal plasma umano. Cliccate qui per vedere una versione più grande di questa figura.

Figura 5: Analisi QC di nanofluidica Array: controllo della qualità (QC) immagini di un nanofluidica matrice caricato correttamente (a sinistra) e un nanofluidica matrice erroneamente caricata (a destra). Il colorante passiva, ROX (presente nella miscela reagente qPCR), fluorescenza per indicare un corretto caricata foro passante. La matrice di destra ha diverse sottoarray / fori passanti che non sono caricati con la miscela dei reagenti qPCR e questi dovrebbero essere identificati come reazioni falsi negativi PCR.
| Componenti | Volume per 5 reazione microlitri (ml) |
| Tampone RT (10x) | 0.5 |
| dNTP (100 mm) | 0.05 |
| RNase inibitore | 0.6 |
| Acqua priva di nucleasi | 1.39 |
| Trascrittasi inversa | 0,33 |
| Volume totale | 2.33 |
Tabella 1A: RT componenti della miscela di reagente in 5 microlitri RT preparazione di reazione per TaqMan tempo reale qPCR utilizzando una piattaforma standard piastra a 96 pozzetti.
| Componenti | Volume per 5 reazione microlitri (ml) | Volume per 20 microlitri di reazione (ml) |
| Veloce Mastermix PCR (2x) | 2.5 | 10 |
| TaqMan qPCR assay (20x) * | 0.25 | 1 |
| Senza Nucleaseacqua | 1.45 | 5.8 |
| Volume totale: | 4.2 | 16.8 |
* Componente saggio basato sulla miRNA selezionato testato.
Tabella 1B: qPCR componenti della miscela di reagente in un 5 o 20 microlitri qPCR preparazione di reazione per TaqMan tempo reale qPCR utilizzando una piattaforma standard piastra a 96 pozzetti.
| Componenti | Volume per reazione (ml) |
| Megaplex RT Primer (10x) | 0.8 |
| dNTP con dTTP (100 mm) | 0.2 |
| MultiScribe trascrittasi inversa (50 U / ml) | 1.5 |
| 10X RT Buffer | 0.8 |
| MgCl 2 (25 mM) | 0.9 |
| RNase Inhibitor & # 160; (20 U / ml) | 0.1 |
| Acqua priva di nucleasi | 0.2 |
| Totale | 4.5 |
Tabella 2A: RT componenti della miscela di reagente in 5 microlitri RT preparazione di reazione per pannello TLDA miRNA.
| Componenti | Volume per reazione (ml) |
| TaqMan PreAmp Mastermix (2x) | 12.5 |
| Megaplex preamplificatore Primer (10x) | 2.5 |
| Acqua priva di nucleasi | 7.5 |
| Totale | 22.5 |
Tabella 2B: preamplificatore componenti della miscela di reagente in un preparato reazione 25 microlitri di pre-amplificazione per il pannello TLDA miRNA.
"Cellspacing =" 0 "> Componenti Volume per reazione (ml) Megaplex RT Primer (10x) 0.75 dNTP con dTTP (100 mm) 0.15 MultiScribe trascrittasi inversa (50 U / ml) 1.5 10X RT Buffer 0.75 MgCl 2 (25 mM) 0.9 RNase Inhibitor (20 U / ml) 0.09 Acqua priva di nucleasi 0.35 Totale 4.5Tabella 3A: RT componenti della miscela di reagente in 5 microlitri RT preparazione di reazione per pannello nanofluidica miRNA probe-based.
| Componentes | Volume per reazione (ml) |
| TaqMan PreAmp Mastermix (2x) | 12.5 |
| Megaplex preamplificatore Primer (10x) | 2.5 |
| Acqua priva di nucleasi | 7.5 |
| Totale | 22.5 |
Tabella 3B:. Preamplificatore componenti della miscela di reagente in un preparato reazione 25 microlitri di pre-amplificazione per il pannello nanofluidica miRNA sonda-base NOTA: Il supplementare interattivo fogli di calcolo Excel previste per le tre piattaforme, rappresenta già l'aggiunta del 5% per compensare pipettaggio errore.
| Campione A | Campione A ripetizione |
| 15,535 | 16,156 |
| 15,471 | 15,652 |
| 15.623 | 16,063 |
| 15,963 | 15,889 |
| 14,006 | 13,993 |
| 14,502 | 14,623 |
| 14,907 | 14,384 |
| 13,732 | 14,946 |
Tabella 4: circolazione miRNA U6 profiling utilizzando microfluidica carta array. Utilizzando due carte di matrice microfluidica (A e B). Ct-valori di miRNA controllo U6 da Campione A (totale = 8). Controllo U6 coerente miRNA abbondanza osservata tra le piste.
Discussione
Le fasi critiche all'interno dei protocolli qPCR sonda-based per ottenere risultati accurati e riproducibili sono per assicurarsi che 1) lo stesso volume e la concentrazione di RT-prodotto viene caricato in ogni reazione qPCR, 2) i rapporti ei volumi corretti dei componenti necessari per reazione qPCR sono preparati e ben mescolato, 3) i volumi corretti e coerenti vengono aggiunti ogni reazione qPCR, e 4) la preparazione e caricamento di ogni campione e reazione mix è completato nel più breve lasso di tempo possibile, mentre ancora memore di i punti critici di cui sopra in precedenza.
Le schede matrice microfluidica bisogno adatto centrifuga e secchi specifici. Ogni secchio può contenere fino a 3 schede (caricato / vuoto). Verificare sempre che tutti e 3 gli slot di un secchio sono occupati e il secchio è bilanciato mettendo bucket simile (questo secchio dovrebbe contenere anche 3 carte, vuote o piene) nella fessura opposta della centrifuga. Pur ponendo la scheda nel buckeSupporto t, accertarsi che le 8 serbatoi proiettano verso l'alto e pozzetti di reazione di fronte la parete esterna di centrifuga. Spin carte a 331 xg per 1 minuto a temperatura ambiente. Dopo il primo giro, aprire la centrifuga e verificare visivamente la miscela di reazione è stato erogato attraverso i 384 pozzi. Ripetere il lancio a stesse impostazioni per 1 più tempo. Rimuovere la carta dal secchio e garantire che il livello della miscela di reazione in ciascuna delle 8 serbatoi è uniforme. Eventuali incoerenze nei volumi di liquido lasciato nei serbatoi rendono la scheda inappropriato utilizzare ulteriormente.
Per avere la stessa concentrazione del RT-prodotto, stessa concentrazione di ingresso di RNA viene aggiunto in ciascuna reazione RT. La concentrazione di RNA totale viene misurata usando un micro-volumespectrophotometer. Il 260/280 rapporto osservato può essere a partire da 1.3 per l'RNA isolato da plasma / siero; questo non sembra avere un effetto sul downstream processo qPCR correlati o dati generati 30. Analogamente, il rapporto di 260/280i campioni di RNA qui testati sono stati tra 1,3-1,7 senza effetti anomali nel qPCR osservati.
Quando si utilizzano campioni a basso contenuto di RNA, come quelli di biofluidi, può essere difficile quantificare RNA prima dell'elaborazione. Si consiglia l'uso del sintetico spike-in presso l'isolamento di RNA e le fasi di trascrizione inversa. Nella nostra esperienza, i candidati Arabidopsis thaliana miRNA (ATH-miR-159A e ath-miR-172 bis) è preferito su Caenorhabditis elegans miRNA (come CEL-miR-39 o cel-miR-54), che nella nostra esperienza può avere maggiore omologia di quelli A. thaliana. L'uso di tale picco-in specifici per fase può spiegare la normalizzazione dei dati miRNA su più campioni analizzati in tempi diversi. Utilizzando un volume d'ingresso fisso di RNA per reazione di sintesi cDNA è anche raccomandato 32,33.
I tre protocolli sonda-based per miRNA quantificazione descritte richiedono quantità variabilidell'ingresso RNA totale, diversi flussi di lavoro e costi. Ciascuno dei flussi di lavoro sono progettati per soddisfare diverse throughput in base al numero di bersagli miRNA e il numero di campioni da analizzare. Con l'aumento della velocità (96 rxn 384 rxn 3072 rxn), il costo per reazione diminuisce con un aumento della quantità di dati ottenuti in una unità di tempo. Poiché tutte queste piattaforme TaqMan utilizzano la chimica, la qualità dei dati ottenuti si può aspettare che sia simile. TaqMan qPCR è un metodo consolidato per identificare l'abbondanza di miRNA in campioni di siero / plasma 9,11,27. Anche se i tre piattaforme discussi qui condividono la stessa chimica, una diminuzione del volume di reazione porta ad una diminuzione della gamma dinamica di rilevamento trascrizione (Farr RJ et al., Dati non pubblicati). La piattaforma qPCR 96-bene è un rendimento più basso, ma la piattaforma alta sensibilità e, a nostro avviso, il "gold standard" per tutti i probe-based (o la tintura a base) piattaforme PCR. Tuttavia, questo puònon essere la piattaforma più economico o efficiente se diverse centinaia o migliaia di campioni vengono analizzati e per più microRNA. Microfluidics (TLDA) e nanofluidica piattaforme (OA) sono piattaforme high-throughput / contenuti progettate per consentire l'acquisizione di dati più grandi in un tempo più breve. Anche se le differenze lotti sono state osservate nelle carte TLDA, questo può essere minimizzato richiedendo carte TLDA dallo stesso lotto. Osserviamo che la piattaforma TLDA (Figura 3) ha mostrato variazioni significative nel 17% dei media - miRNA bassa abbondanza durante il test con lo stesso campione in diversi lotti di carte TLDA. Si consiglia pertanto di utilizzare gli stessi numeri di lotto per l'analisi utilizzando carte TLDA. Questa variazione potrebbe anche essere dovuto alla variabilità tecnica, comprese eventuali potenziali errori di caricamento e di pipettamento. Tuttavia, si consiglia di ordinazione / richiesta dello stesso lotto di carte TLDA. Nessuna variazione lotto significativo è stato osservato sulla piattaforma OA. Nonostante questo, TaqMan speri baseTal qPCR avvicina offerta facilità di misurare l'abbondanza di miRNA in campioni di plasma / siero. Gli approcci di throughput a bassa, media e alta discussi qui offrono la flessibilità necessaria per analizzare una serie di campioni e miRNA con un (a basso rumore di fondo) chimica altamente efficiente, riproducibile e pulita.
Divulgazioni
Articolo costi di trasformazione / pubblicazione sono stati coperti da Life Technologies, dopo l'accettazione di questo manoscritto per la pubblicazione.
Riconoscimenti
Tutti gli autori riconoscono il supporto infrastrutturale del NHMRC CTC, Università di Sydney, la Juvenile Diabetes Research Foundation (JDRF), l'Australia e la Cooper Fondazione Rebecca, Australia. Questa ricerca è stata finanziata attraverso sovvenzioni dal Consiglio australiano Research (FT110100254) e la JDRF, Australia (CRN201314) per AAH WW, RJF e MVJ effettuato tutto bagnato laboratorio di sperimentazione, ASJ effettuata l'analisi dei dati. WW ha scritto la prima bozza. AAH pianificato lo studio e analizzato i dati. Tutti gli autori hanno letto e hanno concordato la versione finale del manoscritto, figure e fogli di lavoro presentati per la pubblicazione.
Materiali
| Name | Company | Catalog Number | Comments |
| 96 well-platform | |||
| For Reverse transcription | |||
| TaqMan MicroRNA Assays INV SM | Applied Biosystems | 4427975 | https://www.lifetechnologies.com/au/en/home/life-science/pcr/real-time-pcr/real-time-pcr-assays/mirna-ncrna-taqman-assays/single-tube-mirna-taqman-assays.html?ICID=search-4427975 The assays comes as a pack of RT primers and PCR primer. |
| TaqMan MicroRNA Reverse Transcription Kit | Applied Biosystems | 4366597 or 4366596 | https://www.lifetechnologies.com/order/catalog/product/4366597?ICID=search-4366597 or http://www.lifetechnologies.com/order/catalog/product/4366596?ICID=search-4366596 The TaqMan MicroRNA Reverse Transcription Kit is the same kit used for reverse transcription in all the threeTaqMan platform- 96-well platform, TLDA and OpenArray. |
| For qPCR run on a 96-well platform | |||
| 2x TaqMan Fast Universal PCR Master Mix, no AmpErase UNG | Applied Biosystems | 4367846 | https://www.lifetechnologies.com/order/catalog/product/4367846?ICID=search-4367846 The 2X TaqMan Fast Universal PCR Master Mix is used for qPCR on the two TaqMan platforms-96-well platform and TLDA. |
| MicroAmp Optical Adhesive Film | Applied Biosystems | 4311971 | https://www.lifetechnologies.com/order/catalog/product/4311971?ICID=search-product |
| MicroAmp Fast Optical 96-Well Reaction Plate, 0.1 ml | Applied Biosystems | 4346907 | https://www.lifetechnologies.com/order/catalog/product/4346907?ICID=search-product |
| Microfluidics platform (TLDA) | |||
| For Reverse transcription | |||
| Refer to 96-well platform TaqMan MicroRNA Reverse Transcription Kit kit (4366597 or 4366596) (as shown above). | |||
| Megaplex RT Primers, Human Pool A v2.1 | Applied Biosystems | 4399966 | https://www.lifetechnologies.com/order/catalog/product/4399966?ICID=search-4399966 The Megaplex RT Primers, Human Pool A v2.1 is used for RT on the two TaqMan platforms- TLDA and OpenArray. |
| Megaplex RT Primers, Human Pool B v3.0 | Applied Biosystems | 4444281 | https://www.lifetechnologies.com/order/catalog/product/4444281?ICID=search-4444281 The Megaplex RT Primers, Human Pool B v3.0 is used for RT on the two TaqMan platforms- TLDA and OpenArray. |
| OR | OR | ||
| Megaplex Primer Pools, Human Pools Set v3.0 | Applied Biosystems | 4444750 | http://www.lifetechnologies.com/order/catalog/product/4444750?ICID=search-4444750 The Megaplex Primer Pools, Human Pools Set v3.0 is used for RT on the two TaqMan platforms- TLDA and OpenArray. |
| For Pre-amplification | |||
| TaqMan PreAmp Master Mix | Applied Biosystems | 4391128 or 4488593 | https://www.lifetechnologies.com/order/catalog/product/4391128?ICID=search-4391128 or http://www.lifetechnologies.com/order/catalog/product/4488593 The TaqMan PreAmp Master Mix is used for pre-amplification of qPCR on the two TaqMan platforms- TLDA and OpenArray. |
| Megaplex PreAmp Primers, Human Pool B v3.0 | Applied Biosystems | 4444303 | https://www.lifetechnologies.com/order/catalog/product/4444303?ICID=search-4444303 The Megaplex PreAmp Primers, Human Pool B v3.0 is used for pre-amplification of qPCR on the two TaqMan platforms- TLDA and OpenArray. |
| Megaplex PreAmp Primers, Human Pool A v2.1 | Applied Biosystems | 4399233 | https://www.lifetechnologies.com/order/catalog/product/4399233?ICID=search-4399233 The Megaplex PreAmp Primers, Human Pool A v2.1 is used for pre-amplification of qPCR on the two TaqMan platforms- TLDA and OpenArray. |
| 1x TE Buffer (100 ml) | Invitrogen | 12090-015 | https://www.lifetechnologies.com/order/catalog/product/12090015?ICID=search-product The 1x TE Buffer is used for pre-amplification of qPCR on the two TaqMan platforms- TLDA and OpenArray. |
| To load and run the 384 well microfluidics (TLDA) card | |||
| TaqMan Array Human MicroRNA A+B Cards Set v3.0 | Applied Biosystems | 4444913 | https://www.lifetechnologies.com/order/catalog/product/4444913 |
| Nanofluidics platform (OpenArray) | |||
| For Reverse transcription | |||
| Refer to 96-well platform TaqMan MicroRNA Reverse Transcription Kit kit (4366597 or 4366596) (as shown above). | |||
| For Pre-amplification | |||
| Refer to TLDA pre-amplification reagents (as shown above) | |||
| To load and run the slides | |||
| TaqMan OpenArray Human MicroRNA Panel, QuantStudio 12K Flex (1 panel) | Applied Biosystems | 4470187 | https://www.lifetechnologies.com/order/catalog/product/4470187?ICID=search-4470187 |
| QuantStudio 12K Flex OpenArray Accessories Kit | Applied Biosystems | 4469576 | https://www.lifetechnologies.com/order/catalog/product/4469576?ICID=search-4469576 |
| OpenArray 384-well Sample Plates | Applied Biosystems | 4406947 | https://www.lifetechnologies.com/order/catalog/product/4406947?ICID=search-4406947 |
| TaqMan OpenArray Real-Time PCR Master Mix | Applied Biosystems | 4462159 | https://www.lifetechnologies.com/order/catalog/product/4462159?ICID=search-4462159 |
| OpenArray AccuFill System Tips | Applied Biosystems | 4457246 | https://www.lifetechnologies.com/order/catalog/product/4457246?ICID=search-4457246 |
| Others | |||
| Nuclease-Free Water | Qiagen | 129117 | http://www.qiagen.com/products/catalog/lab-essentials-and-accessories/nuclease-free-water |
Riferimenti
- Ambros, V. The functions of animal microRNAs. Nature. 431, 350-355 (2004).
- Alvarez-Garcia, I., Miska, E. A. MicroRNA functions in animal development and human disease. Development. 132, 4653-4662 (2005).
- Bartel, D. P. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell. 116, 281-297 (2004).
- Joglekar, M. V., Wei, C., Hardikar, A. A. Quantitative estimation of multiple miRNAs and mRNAs from a single cell. Cold Spring Harb Protoc. 2010, pdb prot5478(2010).
- Joglekar, M. V., Parekh, V. S., Hardikar, A. A. Islet-specific microRNAs in pancreas development, regeneration and diabetes. Indian J Exp Biol. 49, 401-408 (2011).
- Farr, R. J., Joglekar, M. V., Taylor, C. J., Hardikar, A. A. Circulating non-coding RNAs as biomarkers of beta cell death in diabetes. Pediatr Endocrinol Rev. 11, 14-20 (2013).
- Joglekar, M. V., Joglekar, V. M., Hardikar, A. A. Expression of islet-specific microRNAs during human pancreatic development. Gene Expr Patterns. 9, 109-113 (2009).
- Iorio, M. V., Croce, C. M. MicroRNA profiling in ovarian cancer. Methods Mol Biol. 1049, 187-197 (2013).
- Mitchell, P. S., et al. Circulating microRNAs as stable blood-based markers for cancer detection. P Natl Acad Sci USA. 105, 10513-10518 (2008).
- Mattie, M. D., et al. Optimized high-throughput microRNA expression profiling provides novel biomarker assessment of clinical prostate and breast cancer biopsies. Mol Cancer. 5, 24(2006).
- Zhu, W., Qin, W., Atasoy, U., Sauter, E. R. Circulating microRNAs in breast cancer and healthy subjects. BMC Res Notes. 2, 89(2009).
- Zhu, H. T., et al. Identification of suitable reference genes for qRT-PCR analysis of circulating microRNAs in hepatitis B virus-infected patients. Mol Biotechnol. 50, 49-56 (2012).
- Yamada, H., Itoh, M., Hiratsuka, I., Hashimoto, S. Circulating microRNAs in autoimmune thyroid diseases. Clin Endocrinol (Oxf). , (2014).
- Liu, R., et al. Serum MicroRNA Expression Profile as a Biomarker in the Diagnosis and Prognosis of Pancreatic Cancer. Clinical Chemistry. 58, 610-618 (2012).
- Gilad, S., et al. Serum microRNAs are promising novel biomarkers. PLoS One. 3, e3148(2008).
- Joglekar, M. V., Parekh, V. S., Mehta, S., Bhonde, R. R., Hardikar, A. A. MicroRNA profiling of developing and regenerating pancreas reveal post-transcriptional regulation of neurogenin3. Dev Biol. 311, 603-612 (2007).
- Hardikar, A. A., Farr, R. J., Joglekar, M. V. Circulating microRNAs: understanding the limits for quantitative measurement by real-time PCR. J Am Heart Assoc. 3, e000792(2014).
- Wu, C., et al. Diagnostic and Prognostic Implications of a Serum miRNA Panel in Oesophageal Squamous Cell Carcinoma. PLoS One. 9, e92292(2014).
- McDonald, M. K., Capasso, K. E., Ajit, S. K. Purification and microRNA profiling of exosomes derived from blood and culture media. J Vis Exp. , e50294(2013).
- Joglekar, M. V., et al. The miR-30 family microRNAs confer epithelial phenotype to human pancreatic cells. Islets. 1, 137-147 (2009).
- Genda, Y., et al. microRNA changes in the dorsal horn of the spinal cord of rats with chronic constriction injury: A TaqMan(R) Low Density Array study. Int J Mol Med. 31, 129-137 (2013).
- Wang, B., et al. Systematic evaluation of three microRNA profiling platforms: microarray, beads array, and quantitative real-time PCR array. PLoS One. 6, e17167(2011).
- Cuk, K., et al. Circulating microRNAs in plasma as early detection markers for breast cancer. Int J Cancer. 132, 1602-1612 (2013).
- Morrison, T., et al. Nanoliter high throughput quantitative PCR. Nucleic Acids Res. 34, e123(2006).
- Git, A., et al. Systematic comparison of microarray profiling, real-time PCR, and next-generation sequencing technologies for measuring differential microRNA expression. RNA. 16, 991-1006 (2010).
- Benes, V., Castoldi, M. Expression profiling of microRNA using real-time quantitative PCR, how to use it and what is available. Methods. 50, 244-249 (2010).
- Chen, C., et al. Real-time quantification of microRNAs by stem-loop RT-PCR. Nucleic Acids Res. 33, e179(2005).
- Schmittgen, T. D., et al. Real-time PCR quantification of precursor and mature microRNA. Methods. 44, 31-38 (2008).
- Kodani, M., et al. Application of TaqMan low-density arrays for simultaneous detection of multiple respiratory pathogens. J Clin Microbiol. 49, 2175-2182 (2011).
- Taylor, C. J., Satoor, S. N., Ranjan, A. K., Pereirae Cotta,, V, M., Joglekar, M. V. A protocol for measurement of noncoding RNA in human serum. Exp Diabetes Res. 2012, 168368(2012).
- TPF & SPF File Download Options [Internet]. , Available from: http://www.appliedbiosystems.com/absite/us/en/home/products/tpf-spf-download.html (2014).
- Turchinovich, A., Weiz, L., Langheinz, A., Burwinkel, B. Characterization of extracellular circulating microRNA. Nucleic Acids Res. 39, 7223-7233 (2011).
- Li, Y., Kowdley, K. V. Method for microRNA isolation from clinical serum samples. Anal Biochem. 431, 69-75 (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon