Method Article
Correlativo confocale e 3D microscopia elettronica di una cellula sensoriale specifico
In questo articolo
Riepilogo
Here, we introduce a method, cocem3D, to unveil the ultrastructure of a specific cell in its native tissue by bridging confocal and serial block-face scanning electron microscopy.
Abstract
Delimitazione di ultrastruttura di una cellula è importante per capire la sua funzione. Questo può essere un progetto scoraggiante per tipi di cellule rare diffusi in tutta tessuti fatti di tipi cellulari diversi, come le cellule enteroendocrine dell'epitelio intestinale. Questi sensori gastrointestinali di cibo e batteri sono stati difficili da studiare perché la dispersione delle altre cellule epiteliali in un rapporto di 1: 1.000. Recentemente, topi reporter transgenici sono stati generati per identificare le cellule enteroendocrine mediante fluorescenza. Uno di questi è il peptide YY del mouse-GFP. Usando questo mouse, abbiamo sviluppato un metodo per correlare confocale e serial blocco faccia microscopia elettronica a scansione. Abbiamo chiamato il metodo cocem3D e applicato per identificare una cella enteroendocrine specifica nei tessuti e svelare ultrastruttura della cellula in 3D. La risoluzione di cocem3D è sufficiente per identificare organelli piccolo come vescicole secretorie e distinguere membrane cellulari per il volume rendering. Cocem3D può essere facilmente adattato per studiare l'ultrastruttura 3D di altri tipi cellulari specifici nella loro tessuto nativo.
Introduzione
La vita all'interno di una cella si svolge nel tempo e nello spazio. Le modifiche nel corso del tempo sono spesso studiate usando la microscopia time-lapse in combinazione con tecniche di imaging di fluorescenza, come Super-risoluzione microscopia. Spazio, in particolare la disposizione di organelli all'interno di una cellula o cellula-cellula interazioni, può essere ottenuto mediante un quadro completo della struttura fine della cellula. Un resoconto completo di struttura fine di una cella può anche portare chiarezza alla funzione genomica nei casi in cui il genoma è disponibile, come il C. elegans nematode 1 o il placozoa tricoplax piano adherens 2. Serial microscopia sezionamento elettrone è ora riproducibile, tempo efficiente e meno costose attività grazie allo sviluppo di tecnologie automatizzate microscopia elettronica 3D, come seriale blocco faccia microscopia elettronica a scansione 3 (SBEM).
La necessità di informazioni strutturali a chiarire funzione è molto evidente in alcuni celTipi l dove funzione dipende interazioni fisiche cellula-cellula, come i neuroni, glia o cellule epiteliali sensoriali. Siamo particolarmente interessati a chiarire come i segnali sensoriali provenienti da nutrienti nel lume dell'intestino vengono trasdotte in un segnale elettrico che modula in ultima analisi i comportamenti appetitive. Il circuito è complessa ma inizia alla parete intestinale dove i nutrienti vengono a contatto con le cellule epiteliali sensoriali, chiamate cellule enteroendocrine. A differenza di altre cellule epiteliali sensoriali come cellule gustative, cellule enteroendocrine sono dispersi in tutto l'epitelio intestinale con un rapporto di 1-1000 4-7. Di conseguenza, essi sono difficili da identificare e studiare, e per lungo tempo sono stati visti solo come una fonte di ormoni intestinali. Ma con lo sviluppo di topi fluorescenza del reporter specifici delle cellule, la funzione sensoriale complesso di queste cellule sta emergendo. Utilizzando uno di questi topi reporter, un peptide YY-GFP (PyyGFP) topo, abbiamo scoperto che enteroendocrcellule ine hanno una coda citoplasmatica di primo piano che abbiamo chiamato neuropod. La comparsa di neuropods suggerito una funzione conservata in comunicazione cellula-cellula. Così, abbiamo concluso che documentando l'ultrastruttura di una cella enteroendocrine, la funzione di neuropods potrebbe essere derivato.
La necessità di comprendere la struttura di un'appendice in una cella disperso, che è difficile identificare stata la motivazione principale per lo sviluppo di un metodo per combinare la microscopia confocale e SBEM. La cella di interesse è stato identificato utilizzando PyyGFP enteroendocrine specifiche delle celle topi reporter. Il metodo ha permesso di documentare l'intero ultrastruttura di una cellula enteroendocrine e la sua neuropod. All'interno neuropods, abbiamo trovato le caratteristiche strutturali degli assoni neuronali, e al di fuori neuropods, abbiamo trovato un rapporto fisico per glia enterica 8. Infatti, neuropods contengono circa il 70% di tutte le vescicole secretorie suggerendo un ruolo essenziale nella funzione secretoria di queste cellule. Sulla base deldati strutturali, più recentemente abbiamo scoperto che attraverso questi neuropods, cellule enteroendocrine e neuroni che innervano l'intestino formano un circuito neuroepiteliale, simile a quello delle cellule gustative nella lingua 8,9.
Scoprire queste caratteristiche di meccanismi chemosensoriali gastrointestinali derivava da dati strutturali raccolti con questo metodo di microscopia correlativa. Crediamo che questo metodo potrebbe essere utile in altri settori della biologia cellulare, in particolare quando le cellule sono dispersi all'interno dei tessuti in un rapporto molto basso. Abbiamo fatto riferimento al metodo come confocale correlative e seriale blocco faccia microscopia elettronica a scansione in 3D (Cocem3D). Il metodo comprende le seguenti fasi principali: dissezione, microscopia confocale, imaging SBEM, SBEM e immagine confocale di correlazione, e segmentazione manuale. Rispetto ad altri metodi correlativi, il concetto è piuttosto semplice perché la correlazione è fisico piuttosto che chimico.
Protocollo
Tutte le cure degli animali e gli esperimenti sono stati fatti in base a un protocollo approvato dalla Institutional Animal Care and Use Committee Duke University.
Cocem3D derivava da una combinazione di protocolli precedentemente pubblicati 10,11 ed è stato applicato qui per studiare una cella enteroendocrine specifico utilizzando un PyyGFP giornalista del mouse 12. Questo metodo può essere facilmente applicato ad altre cellule di interesse utilizzando commercialmente disponibili topi reporter transgenici. Il metodo è descritto in tre sezioni: microscopia confocale Correlativo, serial blocco faccia microscopia a scansione elettronica (SBEM), e dati di rendering in 3D.
Correlativa Microscopia confocale
L'obiettivo di questa sezione è quello di raccogliere un segmento di tessuto dal colon distale di dimensioni che permettono confocale correlative e di imaging SBEM. Il protocollo è il seguente:
1. La raccolta del Tissue
- Preparare10 ml di soluzione / ml di eparina 50 ug in PBS.
- Preparare xilazina / ketamina anestesia magazzino mescolando 10 ml di ketamina (100 mg / ml) e 1 ml di xilazina (20 mg / ml).
- Diluire xilazina / ketamina disponibile 1: 4 in 0.1M PBS.
- Preparare 100 ml di soluzione fissativa contenente 4% paraformaldeide e 0.1% glutaraldeide in PBS.
- Anestetizzare un mouse PyyGFP, da 6 a 10 settimane, con una dose letale di xilazina / ketamina anestetico. Iniettare l'anestetico per via intraperitoneale a 0,15 ml per 20 g di peso del mouse.
- Verificare il corretto anestesia del mouse pizzicando la coda o le dita dei piedi, e utilizzare pomata sugli occhi per prevenire la secchezza.
- Utilizzare un flusso di pompa peristaltica variabile per perfusione fissativo intracardiaca 10. Utilizzando le forbici chirurgiche (13 cm di lunghezza), taglio aprire la cavità addominale per esporre l'intestino, cuore e polmoni. Tenere il cuore con una pinza stretta modello curvo (lunghezza 12 cm) e inserire l'ago a farfalla (19 G) dalla pompa peristaltica into sinistra ventricolo. Immediatamente, tagliate atrio destro con molla forbici dritto (lunghezza 4 mm). Profumato ad una velocità di 2 ml / min prima con soluzione di eparina per 1 min, e quindi con soluzione fissativo ghiacciata per 15 minuti fino a quando la coda del topo è completamente rigida.
Nota: È molto importante perfusione a bassa velocità per evitare lo scoppio dei piccoli vasi nella mucosa intestinale. Scoppio di navi comprometterà ultrastruttura del tessuto. Se la perfusione è adeguata, il fegato dovrebbe impallidire di colore in 3-5 min. - Utilizzando piccole forbici aprire la cavità addominale e delle accise tutto il colon dalla confluenza con il cieco al retto distale. Collocare il tessuto in PBS ghiacciato. Mentre sommerso in PBS, taglio aperto con piccole forbici a molla i due punti lungo il mesentere.
2. dissezione e fissaggio segmenti del tessuto
- Usando un bisturi, tagliare circa 6 piccoli segmenti di tessuto (2 mm 2) dal colon distale. Effettuare questa operazione su un foglio of impronte dentarie e sommersa in poche gocce di PBS.
- Post-fix i segmenti di tessuto in soluzione fissativa per 3 ore a 4 ° C.
3.-Micro dissezione blocchi di tessuto
- Preparare 5%-basso punto di fusione agarosio in PBS e tenerlo in un bagno di acqua a 45 ° C.
- Incorpora i segmenti di tessuto in 5% basso punto di fusione agarosio con un piccolo contenitore di plastica, come la dimensione standard Tissue-Tek Cryomold.
- Montare le sezioni incluse su una lama microtomo vibrante e riempire la vasca del buffer con PBS ghiacciato.
- Tagliare 300 micron strisce di tessuto a 0,8 ampiezza e 0,04 mm Velocità / sec. Nota: A questo punto, le strisce di tessuto saranno sloggiati dal agarosio.
- Re-incorporare 300 strisce di tessuto micron di agarosio e li montano sul microtomo perpendicolarmente alla lama del vibratome.
- Utilizzando un vibratome, tagliare le strisce di tessuto per uno spessore di 50 micron. Nota: i blocchi di tessuto finali dovrebbero essere di circa 300 micron di larghezza x 50 micron di spessore. Questi dimensioni sono ottimizzati da esperimenti pilota confocale che hanno dimostrato che lo spessore del cellulare enteroendocrine è compresa tra 15-20 micron e lunghezza 30-70 nm del neuropod. Pertanto, l'intera cella potrebbe essere contenuta all'interno di un blocco di tessuto 300 micron di larghezza e 50 micron di spessore, se l'orientamento della cella è parallela alla faccia del blocco tessuti. Variare queste dimensioni a seconda del tipo di tessuto e di cellule di interesse.
- Conservare i blocchi di tessuto in PBS a 4 ° C.
4. confocale Imaging
- Preparare una soluzione colorante nucleare diluendo DAPI in PBS a 1: 4.000.
- Incubare blocchi di tessuto in DAPI nucleare macchia per 5 min. Nota: colorazione DAPI facilita le singole cellule distinguere dai loro nuclei, che è utile per correlare le immagini confocale e SBEM.
- Blocchi di tessuto montaggio su vetrino carica, aggiungere qualche goccia di PBS, e coprire con un vetrino.
- Utilizzando un microscopio confocale, identificare bloccos con villi intatta e cellule PyyGFP di interesse e li ripreso ottenere z-stack sezioni ottiche.
- Utilizzare un obiettivo 20X / 0,8 Zeiss Piano Apochromat ottenere z-pile di 1 micron sezioni ottiche.
- Per il z-stack, utilizzare i canali per 405 nm (DAPI) per determinare la relazione ad altre celle, 488 nm GFP endogena localizzare la cella di interesse, e il contrasto interferenziale differenziale (DIC) per determinare la posizione della relativa cella alla lumen.
- Usa risoluzione di 1.024 pixel o superiore.
5. Incorporare Blocchi in Agarose
- Preparare 10 ml di fissativo contenente 4% paraformaldeide e glutaraldeide al 2,5% in PBS.
- Rimuovere i blocchi di tessuto dal vetrino aggiungendo accuratamente PBS nei bordi del vetrino per far scorrere il vetrino dal vetrino senza danneggiare il blocco di tessuto. Quindi, utilizzare un pennello fine arte di trasferire il blocco del tessuto dalla diapositiva a un 10,5 ml provetta contenente 4% paraformaldeide e 2.5% glutaraldeide fissativo.
- Post-fix blocchi di tessuto O / N a 4 ° C.
- Trasferimento blocchi di tessuto a PBS. Poi, incorporare blocchi appartamento a 5% basso punto di fusione agarosio da loro sandwich tra due vetrini.
Nota: Incorporare i blocchi in un sottile strato di agarosio facilita manipolazione posteriore durante la colorazione. - Tagliare il agarosio in un quadrato e fare una tacca sul lato superiore della mano. Nota: Questo passaggio è necessario per mantenere l'orientamento nei passaggi successivi.
- Conservare blocchi in una provetta da 1,5 ml microcentrifuga con PBS a 4 ° C fino ad ulteriore lavorazione.
Serial Block-faccia microscopia elettronica a scansione (SBEM)
In questa sezione, il blocco dei tessuti è preparato e ripreso con SBEM a basso ingrandimento. L'immagine indagine della faccia blocco viene poi correlata con i dati confocali per identificare la regione contenente la cella di interesse. Una volta che la regioneè identificato, il tessuto viene ripreso con una risoluzione di 7 nm / pixel e fette di 70 nm. Questo è stato sufficiente a risolvere e distinguere grandi denso vescicole secretorie fondamentali dagli altri organelli. Nelle cellule enteroendocrine, questo tipo di vescicole varia tra 100 e 150 nm di diametro 13. Il protocollo è il seguente:
6. sezioni di tessuto colorazione
- Rimuovere il tessuto dal PBS e sciacquare tre volte, 5 minuti ciascuno, in 0.1 M tampone cacodilato.
- Blocchi colorare per 1 h in 0,1% di acido tannico disciolti in 0,1 M tampone cacodilato per aumentare il contrasto delle membrane cellulari 14.
- Effettuare la successiva colorazione e la disidratazione dei tessuti secondo il protocollo pubblicato 11. Dopo Deerinck et al. protocollo, preparare le seguenti soluzioni: 1) 1% (w / v) thiocarbonhydrazide (THC); 2) portare soluzione aspartato; 3) acetato 1% uranile, e 4) ferrocianuro osmio tetraoxyde / potassio (OsO 4 / K ferrocianuro). Mescolare 4% OsO 4 e 2xK Ferrocianuro magazzino [0,3 g K Ferrocianuro e 0.86g Na Cacodylate in 10 ml di H 2 O] in rapporto 1: 1 per rendere OsO 4 / K ferrocianuro. Nota: Avere il tessuto incorporato in agarosio facilita la gestione durante la colorazione. Rimuovere con attenzione agarosio per la successiva infiltrazione della resina.
- Campioni Sciacquare tre volte, 5 min ciascuna, in 0.1 M tampone cacodilato e poi si colorano con la soluzione OsO4 / K Ferrocianuro per 2 ore a 4 ° C.
- Sciacquare in ultra acqua pura per tre volte, 5 minuti ciascuno, e macchia con TCH per 30 minuti a 60 ° C.
- Risciacquare con acqua ultra pura per tre volte, 5 min ciascuno e macchia con 2% OsO 4 (senza K ferrocianuro) per 60 minuti a temperatura ambiente. Sciacquare di nuovo in acqua ultra pura per tre volte, 5 minuti ciascuno.
- Macchia con 1% acetato di uranile O / N a 4 ° C. Risciacquare con acqua ultra pura per tre volte, 5 minuti ciascuno.
- Colorazione con aspartato piombo per 30 minuti a 60 ° C. Risciacquare con acqua ultra pura per tre volte, 5 minuti ciascuno.
- Disidratare i tessuti con il risciacquo in solutions con concentrazioni crescenti di etanolo. Risciacquare 2 volte, 5 min ciascuna, con il 50%, 75%, 85%, 95% e 100% di etanolo assoluto. Al termine, sciacquare sezioni con ossido di propilene due volte, 10 minuti ciascuno.
7. infiltrazione and Embedding sezioni di tessuto in resina
- Infiltrati i blocchi di resina utilizzando l'apposito kit in commercio avaialble. Tessuti Incorpora in resina utilizzando un kit di EPON incorporamento. Preparare una miscela di resina di 48% Epoxy, 20% ml DDSA, 30% ml NMA, e 2% DMP30. Agitare miscela di resina vigorosamente per 5 min e quindi posizionare miscela sotto vuoto per 30 min per permettere bolle di venire in superficie.
- Mescolare resina con ossido di propilene in rapporto 1: 1 e agitare vigorosamente. Rimuovere la disidratazione risciacquo finale da tessuti, e aggiungere resina miscelata con ossido di propilene. Luogo fiala con i campioni in un rotatore O / N e lasciare fiala esordiente. Il giorno dopo, aggiungere appena fatto in resina EPON e mescolare per 90 min a rotatori.
- Incorpora blocchi in resina più piatta possibile da loro sandwich intra le diapositive di vetro che sono stati curati con distaccante liquido per evitare che si attacchino tra loro 15.
- Una volta che i blocchi vengono piatta, curare i blocchi per un'aggiunta 48 ore a 60 ° C.
- Estrarre i vetrini a parte per rilasciare i blocchi.
8. Montaggio e Taglio Block Tissue for Imaging
- Sotto un ambito dissezione, corrispondere all'orientamento dei blocchi tessuto incluse in resina con quella delle micrografie confocale per facilitare l'identificazione delle regioni contenenti le cellule di interesse prima di tagliare il blocco.
- Tagliare il blocco di resina incorporato manualmente fino a un micron blocco faccia ~ 500 x 500.
- Montare il blocco piatto su un perno contenente epossidico conduttivo e asciugare per 30 min. Poi, porre il blocco su una superficie piana e asciutta O / N a 60 ° C.
- Rivestire il blocco con colloidale liquido argento. Mantenere la sezioni di tessuto piano per presentare affettatura del blocco ad angolo retto per facilitare correlating micrografie blocchi viso e confocale seriali.
9. SBEM
- Immagine il blocco utilizzando un microscopio elettronico a scansione dotato di un sistema di SBEM (p .es., 3View).
- Impostare sezione spessore iniziale a ~ 2 micron incrementi e tagliare fino al volto del tessuto emerge dal blocco.
- Acquisire un'immagine Surver di tutto il blocco faccia.
- Utilizzando il software Fiji, prendere le misure sia del blocco faccia seriale e confocale microscopio per generare un denominatore e conto corrente per il campione deformazione durante la fissazione e la colorazione 16.
- Individuare l'area di interesse sulla faccia blocco moltiplicando il denominatore alle coordinate nelle immagini confocali. Utilizzare anche altre caratteristiche strutturali, come la posizione di microvilli, cellule caliciformi, o lamina propria, come riferimento.
- Regione Immagine di interessi ad un 2,25 kV e 7 nm / pixel (o 15,147X ingrandimento) in modalità alto vuoto. Incrementi fetta devono essere impostatia 70 nm o meno.
- Raccogliere i dati grezzi SBEM in formato .dm3 a 16 bit.
10. Ottimizzazione SBEM Immagini per segmentazione Surface
- Convertire immagini SBEM dal formato .dm3 grezzo a 8-bit .tiff.
- Filtro .tiff immagini utilizzando un filtro 0,8 sfocatura nelle isole Figi.
- Ridimensionare i dati impostati al 25% della dimensione originale e salvarla come una pila .tiff per minimizzare la quantità di memoria RAM necessaria per gestire il set.
- Allineare la pila di immagini SBEM utilizzando il plugin Fiji "allineamento lineare stack con SIFT" in modalità di traduzione e ritagliato usando il plugin "crop 3D". Nota: Il ritaglio aiuta a ridurre ulteriormente la quantità di memoria RAM necessaria per la segmentazione e volume rendering.
Rendering dati in 3D
11. Microscopia confocale
- Ricostruire z-stack utilizzando la modalità di funzione "superfici" superano l'automatico. Figura 1D, mostra una recostruzione di ciascun canale utilizzando l'opzione liscio, dettaglio zona superfici a 0,126 micron, e come soglia di intensità assoluta.
12. Serial Block-face SEM
Nota: la segmentazione manuale dei dati è una procedura che richiede molto tempo e in base alle funzioni che devono essere rese la procedura può richiedere diverse settimane. Segmentazione per i video e le cifre presentate in Bohórquez et al. 2014 8 è stato fatto manualmente e ci sono voluti circa 500 ore di lavoro. Si raccomanda di privilegiare le caratteristiche essenziali che necessitano di rendering prima di iniziare il processo di segmentazione. La procedura è come segue:
- Utilizzare lo strumento "superfici" in modalità di disegno e l'opzione "contorno" alla modalità di 50 msec disegno per segmento manualmente il volume reso la cella di interesse.
- Tracciare i contorni su ogni fetta di cella per più agevole il rendering o ogni 5 fette per il rendering più veloce.
- Export im finaleetà utilizzando lo strumento "fotografia" con una risoluzione di almeno 300 dpi.
Risultati
Il metodo presentato qui è stato utilizzato per studiare la ultrastruttura di una cella specifica all'interno di un livello di epitelio. La cella di interesse in questo caso è la cella enteroendocrine sfuggente. La loro identificazione in situ è stato possibile solo negli ultimi anni con lo sviluppo di topi fluorescenza del reporter specifiche delle celle. Nel 2011, abbiamo sviluppato un topo in cui il promotore del PYY ormone guida l'espressione della proteina fluorescente verde 17. In questo mouse PyyGFP, cellule enteroendocrine della parte distale dell'intestino tenue e del colon sono facilmente distinguibili sotto luce UV. Questo modello di topo è stata una base per identificare un blocco di tessuto contenente una cella enteroendocrine ed elaborarla per SBEM. Il metodo di correlazione viene definito qui come cocem3D ed è stato costruito su protocolli precedentemente pubblicati 10,11.
Ottimizzando le dimensioni del blocco di tessuto è critica per la correlazione. In esperimenti preliminari,è stato determinato che in un blocco di tessuto 300 micron di lunghezza x 50 micron di spessore il numero di immagini SBEM è ridotta al minimo e di coprire l'intero corpo della cella di interesse. La figura 1A mostra una trascuranza della dissezione con una lama microtrome vibrante ottenere il blocco di tessuto con le giuste dimensioni. Almeno 100 blocchi sono ottenuti prima smistamento attraverso di loro a scegliere quelli con cellule intatte di interesse. Blocchi selezionati vengono esposte mediante microscopia confocale e immagine z-stacks sono otticamente distanziati ogni 1 micron (Figura 1B e C). Figura 1D mostra una vista volume rendering di una cella enteroendocrine nell'ileo del mouse. La sua neuropod prominente estende sotto cellule epiteliali per ~ 60 micron.
La cella di interesse si trova correlando i confocale z-stack un'immagine SBEM di tutto il blocco viso con. Un esempio è presentato qui di un isolato dal colon distale di un PyyGFPtopo. Dopo aver ottenuto confocali z-stack (Figura 2A), il blocco è incorporato in uno strato sottile di bassofondente agarosio (Figura 2B). Ciò è necessario per la successiva manipolazione del blocco. È importante mantenere l'orientamento del blocco più piatta possibile abbinare le fette ottici con le fette SBEM. Nella figura 2C, un'immagine rappresentativa è mostrato dell'intera faccia del blocco tessuti. Alcune parti di questa figura sono stati precedentemente pubblicati in Bohorquez et al., 2014 8. Questo è stato successivamente correlata con l'immagine confocale tenendo conto dei punti di riferimento del tessuto e le dimensioni. La figura 2D mostra una sovrapposizione della confocale e immagine SBEM del blocco, rivelando la ubicazione precisa della nostra cella di interesse.
Una volta che la cella è stata identificata, imaging SBEM avviene ad una risoluzione sufficiente per identificare vescicole secretorie e altri organelli cellulari. Il set di dati presentati luire è stato ripreso a 7 nm per pixel come le immagini del blocco sono state scattate ogni 70 nm. I set di dati contengono 643 immagini che si estendono su 45 micron di profondità dei tessuti. A pochi micron sono rase durante l'imaging iniziale di tutto il blocco di fronte a basso ingrandimento. Il set di dati SBEM greggio è stato acquisito in formato .dm3 e trasformato per .tiff per il successivo trattamento. Lo stack SBEM è stata ritagliata utilizzando il software Fiji plugin "crop (3D)" blocco contenere solo la regione di interesse. Questo riduce la quantità di memoria RAM necessaria per volume rendering. La cella è stata identificata dalla sua posizione e vescicole secretorie, e segmentato utilizzando il software Imaris. Figura 3 contiene una rappresentazione in 3D della cella enteroendocrine.
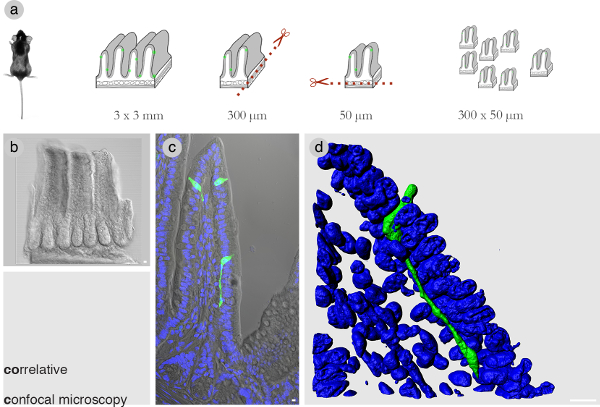
Figura 1:. Microscopia confocale Correlativo (A) del flusso di lavoro a sezionare un pezzo ditessuto intestinale in un tessuto blocco 300 x 50 micron di spessore. (B) Un blocco rappresentante tessuto delle giuste dimensioni. Si noti che un blocco di tessuto di queste dimensioni da piccolo intestino del mouse estende circa 3 villi o, nel caso del colon circa 8 cripte. (C) Un villi nell'intestino tenue contenente una cella di interesse. (D) A confocale z-stack rendering in 3D utilizzando il software Imaris mostra una cella enteroendocrine con un neuropod prominente che corre sotto l'epitelio intestinale. Blu = DAPI macchia nucleare. Bar = 10 micron. Cliccate qui per vedere una versione più grande di questa figura.
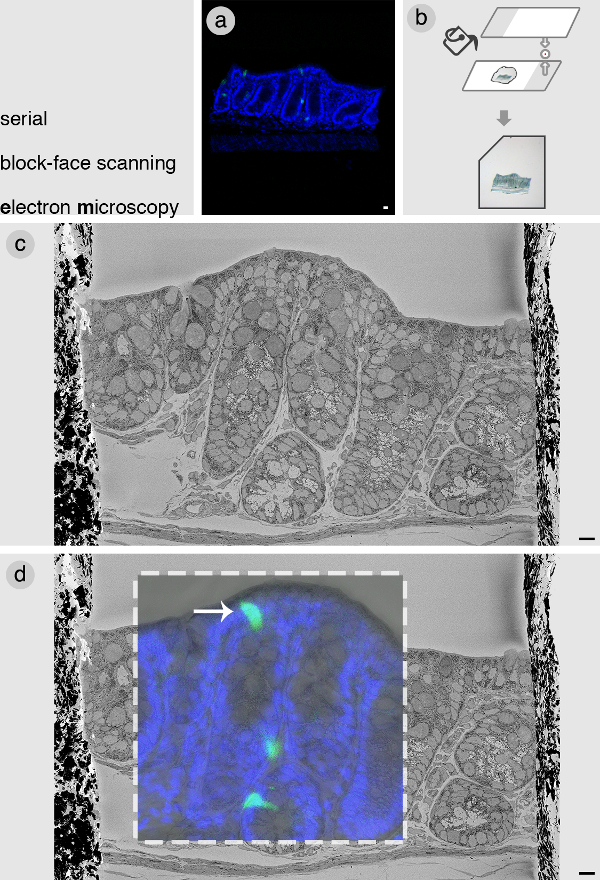
Figura 2: Serial block-faccia microscopia elettronica a scansione (A) immagine confocale di una sele.blocco tessuto CTED dal colon contenente cellule PyyGFP enteroendocrine (verdi) di interesse. (B) Una volta che il blocco viene ripreso al microscopio confocale, viene poi incorporato appartamento a basso punto di fusione agarosio utilizzando vetrini. Incorporare il blocco dei tessuti in agarosio facilita la successiva manipolazione, e la tacca sull'angolo sinistro aiuta a montare il blocco all'interno del SBEM nel giusto orientamento. (C) SBEM immagine del volto del blocco. (D) Correlazione dei dati confocale con SBEM identificare cella di interesse (freccia bianca). Immagini in ced di questa figura sono modificate da Bohorquez et al., 2014 Blu 8. = DAPI macchia nucleare. Bar = 10 micron. Cliccate qui per vedere una versione più grande di questa figura.
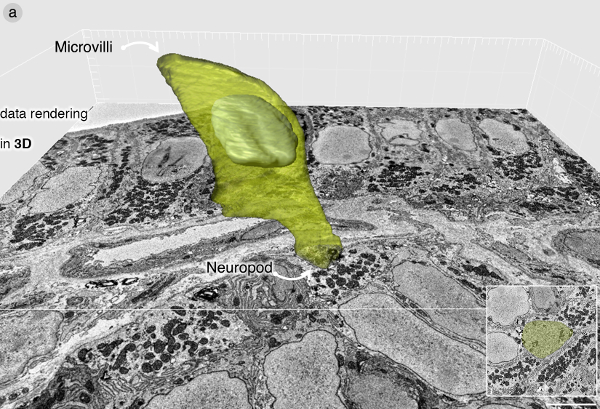
Figura 3:. Rappresentazione dei dati in 3D (A) Rappresentazione volumetrica di cellula enteroendocrine utilizzando il software Imaris. La cella contiene una neuropod piena di vescicole secretorie. Per una descrizione completa della ultrastruttura di una cella enteroendocrine, consultare Bohórquez et al., 2014 8. Cliccate qui per vedere una versione più grande di questa figura.
Discussione
Chemosensation gastrointestinale sta emergendo come un nuovo campo emozionante nella ricerca biomedica. Ciò è in gran parte a causa della scoperta dei recettori del gusto funzionali nelle cellule enteroendocrine 18. Studi successivi hanno dimostrato che le cellule enteroendocrine esprimono i recettori specifici per le sostanze nutrienti, tra cui carboidrati, lipidi e aminoacidi 5,6,19,20. Il fattore catalitico per queste scoperte è stato lo sviluppo di topi reporter, in cui le cellule sono enteroendocrine fluorescente 20. Abbiamo sviluppato uno di questi topi, il mouse PyyGFP, per studiare le cellule enteroendocrine del piccolo intestino e colon distale 17,21. Queste cellule sono interessanti perché secernono PYY e glucagone-like peptide 1, entrambi i quali sono induttori di sazietà 22,23. A quel tempo, un conto ultra-strutturale completa di queste cellule mancava, che abbiamo creduto era essenziale per comprendere i loro meccanismi di segnalazione.
ontent "> Qui, abbiamo descritto un visivamente un metodo per colmare microscopia confocale con SBEM. Il metodo è indicato come Cocem3D, che ci ha permesso di documentare l'ultrastruttura completa delle cellule enteroendocrine. Abbiamo riportato che queste cellule hanno un neuropod che contiene tre quarti di tutte le vescicole secretorie 8. All'interno della neuropod, ci sono neurofilaments e molto simile assoni neuronali, neuropods si nutrono di cellule gliali enteriche 8. Più importante, è se questi neuropods che enteroendocrine cellule connettono fisicamente ai neuroni che innervano l'intestino e colon 9.Uno dei punti di forza di cocem3D è la sua semplicità. Ridurre il campione di tessuto in un blocco che può essere immaginata dal confocale e SBEM facilita l'identificazione di una cella specifica all'interno di un tessuto. Vescicole secretorie e altri organelli possono essere identificati con facilità ad una risoluzione di 7 nm / pixel. Poiché le sezioni in SBEM vengono scartate, ulteriori procescantare di tessuti per identificare specifiche proteine non è un'opzione a questo punto. Tuttavia, lo sviluppo di metodi come ATUM 24, in cui le sezioni dal blocco tessuti sono conservati, sono suscettibili di consentire l'identificazione di proteine specifiche nella cellula. Determinare la posizione specifica di recettori chemiosensoriali sulle cellule enteroendocrine tratta di elementi essenziali per lo sviluppo di terapie farmacologiche per l'obesità, perché le cellule enteroendocrine sono un'interfaccia sensoriale tra il cibo nell'intestino e sazietà nel cervello.
Divulgazioni
Dr. Satish Medicetty è un dipendente di e riceve 100% stipendio da Renovo Neural Inc; tuttavia, questo non altera l'adesione dell'autore alle politiche JoVE alla condivisione dei dati e dei materiali. Nessun conflitto di interessi è dichiarato per rimanenti autori.
Riconoscimenti
Our sincere appreciation is expressed to the following people: Drs. Sam Johnson and Benjamin Carlson of the Duke Light Microscopy Core Facility for their assistance with data visualization software, and Ms. Valerie Lapham and Dr. John M. Mackenzie, Jr. of the Center for Electron Microscopy at North Carolina State University for their advice on electron microscopy. We thank Dr. Elaine B. Bohórquez for her editorial assistance. Authors contributed in the following manner: DVB, SM, and RAL designed experiments and analyzed data. DVB performed experiments and FH performed manual rendering of data. SM is director of Renovo Neural, where SBEM data was acquired. DVB wrote the manuscript and all authors reviewed and edited the final manuscript. This work was supported by NIH grants R01DK091946 and Veterans Affairs grant I01BX002230 to RAL, and F32DK094704, to DVB.
Materiali
| Name | Company | Catalog Number | Comments |
| Phosphate buffered saline | Life technologies | 10010023 | |
| Heparin sodium salt | Sigma | H4784 | |
| Paraformaldehyde | Sigma | 158127 | 4%, freshly made in PBS, final pH 7.4 |
| Glutaraldehyde | Sigma | G5882 | |
| Dental wax | Electron Microscopy Sciences | 72660 | |
| Low-melting agarose | Life technologies | 16520-100 | 5%, freshly made in PBS |
| Standard Tissue-Tek Cryomold | Electron Microscopy Sciences | 62534-25 | |
| DAPI nuclear stain | Life technologies | D1306 | |
| Postively charged glass slides and coverslips | |||
| Fine art paintbrush #1 | |||
| Cacodylate buffer | Electron Microscopy Sciences | 11650 | |
| Tannic acid | Electron Microscopy Sciences | 21700 | |
| EMbed 812 kit | Electron Microscopy Sciences | 14120 | |
| Liquid releasing agent | Electron Microscopy Sciences | 70880 | |
| Liquid silver colloidal | Electron Microscopy Sciences | 12630 | |
| CircuitWorks conductive epoxy | ITW Chemtronics | CW2400 | |
| Variable flow peristaltic pump | VWR | 70730-064 | |
| VT1200S Vibrating blade microtome | Leica | ||
| Zeiss 780i confocal microscope | Carl Zeiss | ||
| Sigma VP Scanning Electron Microscope | Carl Zeiss | ||
| 3view system | Gatan | ||
| Renovo Neural Inc (Cleveland, OH) | http://www.renovoneural.com | Renovo provides 3d EM services | |
| Fiji software | Open access software | ||
| Computer station with 16 GB of RAM or more | |||
| Data visualization software Imaris 7.5 | Bitplane |
Riferimenti
- White, J. G., Southgate, E., Thomson, J. N., Brenner, S. The structure of the nervous system of the nematode Caenorhabditis elegans. Philosophical transactions of the Royal Society of London. Series B, Biological. 314, 1-340 (1986).
- Smith, C. L., et al. Novel cell types, neurosecretory cells, and body plan of the early-diverging metazoan Trichoplax adhaerens. Current biology : CB. 24, 1565-1572 (2014).
- Denk, W., Horstmann, H. Serial block-face scanning electron microscopy to reconstruct three-dimensional tissue nanostructure. PLoS biology. 2, e329 (2004).
- Engelstoft, M. S., et al. Seven transmembrane G protein-coupled receptor repertoire of gastric ghrelin cells. Molecular metabolism. 2, 376-392 (2013).
- Chandra, R., et al. Immunoglobulin-like domain containing receptor 1 mediates fat-stimulated cholecystokinin secretion. The Journal of clinical investigation. 123, 3343-3352 (2013).
- Wang, Y., et al. Amino acids stimulate cholecystokinin release through the Ca2+-sensing receptor. Am J Physiol Gastrointest Liver Physiol. 300, G528-G537 (2011).
- Rogers, G. J., et al. Electrical activity-triggered glucagon-like peptide-1 secretion from primary murine L-cells. The Journal of physiology. 589, 1081-1093 (2011).
- Bohórquez, D. V., et al. An enteroendocrine cell-enteric glia connection revealed by 3D electron microscopy. PloS one. 9, e89881 (2014).
- Bohórquez, D. V., et al. Neuroepithelial circuit formed by innervation of sensory enteroendocrine cells. The Journal of clinical investigation. , (2015).
- Knott, G. W., Holtmaat, A., Trachtenberg, J. T., Svoboda, K., Welker, E. A protocol for preparing GFP-labeled neurons previously imaged in vivo and in slice preparations for light and electron microscopic analysis. Nature protocols. 4, 1145-1156 (2009).
- Deerinck, T., Bushong, E., Thor, A., Ellisman, M. . NCMIR methods for 3D EM: a new protocol for preparation of biological specimens for serial block face scanning electron microscopy. , (2010).
- Bohórquez, D. V., Chandra, R., Samsa, L. A., Vigna, S. R., Liddle, R. A. Characterization of basal pseudopod-like processes in ileal and colonic PYY cells. Journal of molecular histology. 42, 3-13 (2011).
- Nilsson, O., et al. Distribution and immunocytochemical colocalization of peptide YY and enteroglucagon in endocrine cells of the rabbit colon. Endocrinology. 129, 139-148 (1991).
- Mizuhira, V., Futaesaku, Y. New fixation method for biological membranes using tannic acids. Acta Histochem Cytochem. 5, 233-236 (1972).
- Reymond, O. L., Pickett-Heaps, J. D. A routine flat embedding method for electron microscopy of microorganisms allowing selection and precisely orientated sectioning of single cells by light microscopy. Journal of microscopy. 130, 79-84 (1983).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature. 9, 676-682 (2012).
- Bohórquez, D., Chandra, R., Samsa, L., Vigna, S., Liddle, R. Characterization of basal pseudopod-like processes in ileal and colonic PYY cells. J Mol Histol. 42, 3-13 (2011).
- Jang, H. J., et al. Gut-expressed gustducin and taste receptors regulate secretion of glucagon-like peptide-1. Proceedings of the National Academy of Sciences of the United States of America. 104, 15069-15074 (2007).
- Liou, A. P., et al. The G-protein-coupled receptor GPR40 directly mediates long-chain fatty acid-induced secretion of cholecystokinin. Gastroenterology. 140, 903-912 (2011).
- Reimann, F., et al. Glucose sensing in L cells: a primary cell study. Cell metabolism. 8, 532-539 (2008).
- Bohorquez, D. V., Liddle, R. A. Axon-like basal processes in enteroendocrine cells: characteristics and potential targets. Clinical and translational science. 4, 387-391 (2011).
- Batterham, R. L., et al. Gut hormone PYY(3-36) physiologically inhibits food intake. Nature. 418, 650-654 (2002).
- Flint, A., Raben, A., Astrup, A., Holst, J. J. Glucagon-like peptide 1 promotes satiety and suppresses energy intake in humans. The Journal of clinical investigation. 101, 515-520 (1998).
- Hayworth, K. J., et al. Imaging ATUM ultrathin section libraries with WaferMapper: a multi-scale approach to EM reconstruction of neural circuits. Frontiers in neural circuits. 8, 68 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon