È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Planar gradiente di diffusione del sistema per Indagare Chemiotassi in un 3D collagene Matrix
In questo articolo
Riepilogo
Cell migration is an important part of human development and life. In order to understand the mechanisms that can alter cell migration, we present a planar gradient diffusion system to investigate chemotaxis in a 3D collagen matrix, which allows one to overcome modern diffusion chamber limitations of existing assays.
Abstract
The importance of cell migration can be seen through the development of human life. When cells migrate, they generate forces and transfer these forces to their surrounding area, leading to cell movement and migration. In order to understand the mechanisms that can alter and/or affect cell migration, one can study these forces. In theory, understanding the fundamental mechanisms and forces underlying cell migration holds the promise of effective approaches for treating diseases and promoting cellular transplantation. Unfortunately, modern chemotaxis chambers that have been developed are usually restricted to two dimensions (2D) and have complex diffusion gradients that make the experiment difficult to interpret. To this end, we have developed, and describe in this paper, a direct-viewing chamber for chemotaxis studies, which allows one to overcome modern chemotaxis chamber obstacles able to measure cell forces and specific concentration within the chamber in a 3D environment to study cell 3D migration. More compelling, this approach allows one to successfully model diffusion through 3D collagen matrices and calculate the coefficient of diffusion of a chemoattractant through multiple different concentrations of collagen, while keeping the system simple and user friendly for traction force microscopy (TFM) and digital volume correlation (DVC) analysis.
Introduzione
Il movimento preferito di cellule verso un gradiente di concentrazione, noto come chemiotassi, svolge un ruolo importante in processi patologici e fisiologici del corpo. Tali esempi sono la pelle e le mucose guarigione delle ferite 1, morfogenesi 2, infiammazione 3, e la crescita tumorale 4,5. E 'stato anche dimostrato che le cellule tumorali possono migrare attraverso due strategie cellule migratorie individuali e collettive 6. Inoltre, i meccanismi di instabilità diffusionali possono indurre la separazione delle cellule singole o cluster da un corpo / oggetto tumorale e quindi in grado di emigrare verso una fonte di sostanze nutritive e di invadere le zone più ampie e tessuti 7 così.
Inoltre, è stato dimostrato che i meccanismi di migrazione diverse possono essere attivi in 2D e in 3D, a causa di diversi ruoli delle molecole di adesione 8. Pertanto, una mossa per fisiologicamente rilevanti in vitro per indagare motilità cellulare in un measureable e modo semplice è di importanza nella comprensione movimento delle cellule fenomeni 9. Purtroppo, la difficoltà nell'analisi migrazione cellulare, un completo test chemiotassi quantificabile solito richiede un metodo lungo laboriosa, fondata sulla misurazione della motilità cellulare e fenomeni di trasporto modelli imparziale.
Approcci sperimentali passato per indagare chemiotassi cellulari includono la camera di Boyden 10 e il sotto dosaggio agarosio 11. Tuttavia, all'interno di questi primi test, esperimenti di migrazione delle cellule non hanno controllano il movimento in relazione al tempo. Di più, importante, i gradienti di concentrazione usati per gli esperimenti sono stati non ben definiti o completamente compresi mentre solo sostenere la segnalazione per non più di qualche ora. Inoltre, presto chemiotassi tentativi camera limitato la migrazione delle cellule a due dimensioni e non permettono di monitorare la cinetica della migrazione 12. Guardando la camera di Boyden, un saggio finalenon consentirebbe al ricercatore di osservare la migrazione visivamente e non ha potuto differenziare direttamente chemiotassi (movimento direzionale) da chemochinesi (movimento casuale). Inoltre, diverse variabili differenze nella dimensione dei pori e lo spessore delle membrane-reso la camera molto difficile da riprodurre facilmente e nascosti reazione migrante delle cellule di chemochine 13,14.
Con la nuova comprensione di microfluidica, nuove camere e micro-dispositivi sono stati studiati come strumento per indagare locomozione cella in condizioni di flusso interstiziali o chemiotassi 15,16. In questi nuovi dispositivi, sono stati introdotti e studiati, come l'effetto di sollecitazione di taglio su una cella 17,18 nuove metriche cellulari. Purtroppo, le camere chemiotassi microfluidica passati e attuali limitati studi di migrazione cellulare a substrati-an 2D battuta d'arresto importante dal momento che molti processi biologici, tra cui l'invasione delle cellule tumorali e metastasi, e immigrazione cellulare mune, comporta migrazione 3D.
Camere-dove osservazione diretta di una soluzione chemiotattico è in contatto con un gel 3D contenente cellule sono anche stati riportati anche 19,20. Queste camere hanno due scomparti, uno contenente un fattore chemiotattico e una contenente cellule, sono uniti l'uno accanto all'altro in orizzontale 21 o come anelli concentrici 22. Questi sistemi sono puntati nella giusta direzione, ma non mantengono un sistema di chemiotassi per un periodo di tempo prolungato.
Inoltre, i ricercatori hanno esaminato diffusività attraverso membrane di collagene nelle cellule dialisi, nonché la diffusione delle molecole traccianti attraverso campioni di collagene sottoposta a pressione idrostatica 23-25. Alcuni esperimenti di diffusione in gel di collagene affidano a modificazioni fisiche e chimiche del gel utilizzando campi magnetici e incorporazione chimiche 26. Un metodo comune per la modellazione di diffusività in collatessuti azo tati si basa sulla fluorescenza del punto photobleaching continuo. Questo metodo ha rivelato anisotropia nei coefficienti di diffusione di macromolecole nei tessuti collageni orientati. Tuttavia, photobleaching è stato usato nella cartilagine articolare e non collagene matrici. Mentre simile, gli esperimenti di modellazione necessari devono essere effettuati tramite specificamente comprendere il coefficiente di diffusione del gel di collagene. Ancora più importante, i sistemi non utilizzano un metodo per misurare generazione di forza cella.
Sfortunatamente, la maggior parte dei sistemi sembrano mancare uno o due elementi fondamentali per un sistema ideale: il che consente di inseguimento cellulare, una comprensione gradiente di diffusione con un fattore chemiotattico attraverso la matrice, un insieme relativamente semplice con una facilità di riproducibilità, la minimizzazione interazioni cellula-cellula, e la capacità di misurare unità dimensionali per quantificazione (cioè, velocità, forza, concentrazione specifica). Moghe et al. 27 ha proposto un sistema che soddisfaceva la maggior parte di questi requisiti in cui le cellule sono state inizialmente disperse nel gel anziché concentrati sulla superficie del filtro, ma era difficile misurare le forze che la cellula genera.
A questo proposito, vi presentiamo un sistema planare gradiente di diffusione di indagare chemiotassi in una matrice di collagene in 3D, che permette di superare i limiti moderni camera di diffusione dei dosaggi esistenti, che si basa sulla microscopia time-lapse, associato a tecniche di analisi di immagine per la misurazione delle cellule forze in un ambiente 3D. Questo protocollo fornisce un modo semplice, ma innovativo di creazione di una semplice camera di diffusione 3D che può essere usato per studiare chemiotassi 3D in celle diverse.
Protocollo
1. 3D Mold Design e parti di ricambio
- Muffa
- Prima di lavorare, di ottenere un kit di elastomero siliconico, una camera di imaging cellulare dal vivo, un vetrino di vetro di 22 mm e un cubo di metallo alluminio lavorato con dimensioni 10,07 millimetri x 3,95 millimetri x 5,99 millimetri. Preparare la camera di cellule vive per stampaggio ponendo il vetrino nel supporto inferiore e montaggio del resto della camera come indicato dal produttore.
- Successivamente, utilizzando una pinza, posizionare il cubo di metallo alluminio lavorato al centro della custodia camera di imaging cellulare dal vivo e sulla parte superiore del vetrino, e poi mettere da parte.
- Mescolare soluzioni elastomero siliconico secondo il protocollo del produttore per fare 5 ml di elastomero.
- Utilizzando un laboratorio spatola monouso, versare la soluzione elastomero siliconico in configurazione camera di imaging cellulare dal vivo e garantire di non muoversi messo lavorato cubo di metallo alluminio. Posizionare il sistema sul banco di laboratorio, in una posizione sicura O / N per curare.
- La mattina dopo, decostruirela camera di imaging cellulare dal vivo, come raccomandato dal costruttore e tirare fuori la muffa con pinze. Utilizzando pinze, estrarre con attenzione il cubo di metallo alluminio lavorato da muffe. Vai al lavello e lavare lo stampo con acqua deionizzata. Mettere lo stampo su un tovagliolo di carta per asciugarsi.
- Una volta asciutto, di un coltello multiuso mio hobby, tagliare fessure attraverso lo stampo, distanziati 2,34 millimetri di distanza l'uno estremità longitudinale, all'interno dello stampo in silicone. Assicurarsi che i soggiorni di stampo in un luogo sicuro e asciutto fino a quando si è pronti a costruire il sistema per un esperimento.
- Idrofili e idrofobi vetro coprioggetto Preparazione
- Per consentire la matrice di collagene di aderire ad una superficie, creare coprioggetto idrofili:
- Usando una pipetta monouso, misurare 150 ml di 3-amminopropil-trimetossisilano e versare la soluzione in un tubo da 50 ml. Aggiungere 30 ml di etanolo al 100% al tubo 50 ml con una seconda pipetta monouso e chiudere il coperchio. Vortex la soluzione per 2 minuti, assicurando la completa miscelazione. Pla nostra soluzione in una capsula di Petri di vetro e mettere da parte.
- Versare 15 ml di alcol etilico al 100% in un secondo piatto di Petri e mettere da parte (assicuratevi di etichettare i piatti).
- Utilizzando una nuova pipetta monouso, misurare 30 ml di acqua deionizzata e versare la soluzione in una provetta da 50 ml. Successivamente, utilizzando una nuova misura pipetta usa e getta su 1.875 ml di glutaraldeide e versare la soluzione nello stesso tubo da 50 ml e chiudere il coperchio. Vortex la soluzione per 2 minuti, assicurando la completa miscelazione. Versare il composto in terza piastra di Petri, e mettere da parte.
- Utilizzando pinze, togliere un 22 millimetri coprioggetto di vetro rotondo e risciacquare entrambi i lati con 100% di miscela di etanolo con una pipetta monouso.
- Inserire coprioggetto di vetro risciacquati in 3-amminopropil-trimetossisilano con 30 ml di etanolo al 100% piatto soluzione e lasciar riposare in soluzione per 5 min.
- Con una pinza, estrarre coprioggetto di vetro e sciacquare di nuovo con soluzione di etanolo al 100% con la pipetta monouso.
- Goccia coprioggetto di vetro risciacquato in 1.875ml di glutaraldeide e 30 ml di miscela di acqua deionizzata e mettere da parte per 30 min.
- Dopo 30 minuti, rimuovere coprioggetto di vetro con una pinza e sciacquare con acqua deionizzata e collocarlo su asciutto O tessuto / N ad asciugare a temperatura ambiente.
- Ripetere immersione coprioggetto e in movimento per il numero di lamelle, se necessario con le stesse soluzioni generate.
- Fai coprioggetto idrofobiche dal protocollo data.
- Aggiungere 500 pl di tridecafluoro-1,1,2,2-tetrahydrooctyl, 100 ml di acido acetico e 19,4 ml di esano in una provetta da 50 ml e agitare per 2 min. Versare la soluzione in vetro piastra di Petri e mettere da parte.
- Utilizzando pinze pulite, togliere un vetrino di vetro e cadere in soluzione mista preparata per 2 minuti. Dopo 2 minuti sono passati, togliere vetrino di vetro con una pinza e sciacquare con acqua deionizzata con una usa e getta pipetta e luogo coprioggetto su asciutto O tessuto / N ad asciugare a temperatura ambiente. Conservare vetrino in piatti di plastica Petri fino all'uso.
- Ripetere coprioggetto DIPPzione e spostando a molti coprioggetto come necessario con le stesse soluzioni generati.
- Per consentire la matrice di collagene di aderire ad una superficie, creare coprioggetto idrofili:
Assemblea 2. Mold
- Prima di utilizzare stampi, usando una pipetta monouso lavare lo stampo con il 90% di alcool etilico su un contenitore per rifiuti chimici. Quindi, posizionare lo stampo in una capsula di Petri riempita con acqua deionizzata e lasciar riposare O / N.
- Prendete muffa dalla soluzione con pinze e posto su un telo ad asciugare prima di avviare il gruppo di forma.
- Mentre lo stampo è aria di essiccazione, tagliare i coprioggetti idrofili quadrata in due rettangoli leggermente più grandi 3,95 millimetri x 5,99 millimetri utilizzando uno strumento diamante scribing alta precisione. Far scorrere ogni vetrino taglio rettangolo nelle fessure tagliate nello stampo in silicone.
- Vibrazione dello stampo capovolto e applicare grasso per vuoto lungo il fondo dello stampo in silicone con una pipetta monouso. Avanti, capovolgere lo stampo posteriore lato destro in alto e premere il fondo dello stampo sulle coperture in vetro idrofili circolarilabbro per creare una tenuta.
- Con guanti monouso, prendere lo stampo montato e mettere in un piatto e getta Petri e poi in un bio-cappuccio. Accendere lampada UV Bio-cappuccio per 1 ora per sterilizzare lo stampo prima che si verifichi la sperimentazione.
3. Il collagene miscela e 3D Matrix
- Prima di preparare la miscela di collagene, macchia e preparare le cellule per l'imaging secondo protocolli standard 28.
- Utilizzando tecniche di laboratorio standard e protocolli 29,30, in un bio-hood, mescolare carta cellulari e chemiotattico in un tubo da 15 ml alla concentrazione chemoattractant desiderata. Pipettare 5 ml di soluzione specifica cella media-chemiotattico concentrazione in una provetta da 15 ml e spostare la soluzione in un bagno d'acqua riscaldata.
- Usando una pipetta, estrarre 5 ml di supporti cellulari solo in un secondo tubo da 15 ml e posto in un bagno d'acqua riscaldata per riscaldare la soluzione per esperimenti di microscopia.
- In un bio-hood, pipetta 30 ml di 10x tampone fosfatosoluzione ed (PBS) in micro provetta da centrifuga. Aggiungere 6 ml di 1 N di idrossido di sodio (NaOH) in stessa provetta da centrifuga micro e vortex per 15 sec.
- Ottenere soluzione coda di ratto collagene I dal frigo e passare al bio-cappa utilizzando tecniche di laboratorio standard e protocolli 29. Assicurarsi che il collagene è ancora freddo. Pipetta 168,3 ml di collagene nella stessa provetta da centrifuga micro.
- Successivamente, aggiungere 18 ml di microsfere carbossilato modificato fluorescenti giallo-verde con una pipetta alla stessa provetta da centrifuga micro. Vortex provetta micro con tutte le soluzioni per 30 sec.
- Aggiungere 77,7 ml di desideri miscela di cellule media-cell (circa 2 x 10 6 cellule / ml concentrazione) in micro tubo di centrifuga e miscelare con puntale per 20 sec. Se necessario, modificare la densità matrice seguendo la miscela collagene personalizzato adattato per l'aggiunta di perline fluorescenti e tavolo vitalità cellulare (Tabella 1).
- Dispensare 300 & #181; l di miscela di collagene-cellule al centro (pozzetto 2 #) del complesso di stampo preparato. Collocare lo stampo su un piatto Petri monouso e spostarlo in un incubatore standard per 20 min a 37 ° C con 5% di CO 2.
- Togliere sistema da incubatore e mettere di nuovo in bio-cappuccio. Utilizzando pinze, rimuovere entrambi i vetrini idrofobici fissando sulla parte superiore di ogni vetrino e tirando alto e lontano dallo stampo.
- Spostare l'intero sistema alla microscopia e immagine desiderata per assicurare lati dello stampo collagene sono ancora diritto per consentire un gradiente planare diffusione.
- Con il sistema ancora sotto il microscopio, utilizzando un 100 microlitri monocanale pipetta, aggiungere 100 ml di supporti cellule nel pozzetto 1 #. Successivamente, utilizzando una 100 pl monocanale pipetta aggiungere 100 ml di media cella con concentrazione chemoattractant desiderato nel bene # 3.
- Subito dopo l'aggiunta di soluzioni in entrambi i pozzi, iniziare imaging.
4. Imaging e DiffusioneModellismo
- Se si vuole trovare il coefficiente di diffusione per la densità del collagene specifico per calcolare la concentrazione specifica, seguire il protocollo di seguito:
- Seguire il protocollo di cui sopra dal titolo "miscela di collagene e 3D Matrix", invece di generare supporti cella con concentrazione chemoattractant desiderata, fare 5 micron rhodamine in un tubo da 15 ml con calcoli e protocolli 30 standard.
- Immagine matrici collagene 3D con un sistema confocale montato su un microscopio invertito utilizzando un laser ad argon (488 nm) per catturare la fluorescenza. Immagine ogni 2 o 3 secondi per un massimo di 7 ore.
- Per Modeling Diffusione: Usando il modello matematico mostrato di seguito, scrivere un codice software di calcolo per l'elaborazione e l'analisi di questi passaggi generali.
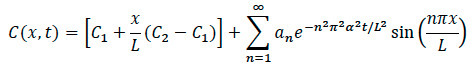
- Importazione di immagini in software di calcolo per ridimensionamento e normalizzazione da parte della massima intensità. Converti immagini in un colore grigio-mappa dell'immagine per selezionare il bordo. Scegliere un asse lungo il centro dell'immagine, a destra del centro e alla sinistra del centro.
- Usando il codice del software di calcolo, tracciare l'intensità normalizzata, insieme con la vera intensità e la differenza tra i due.
- Quindi, immettere i parametri necessari per il montaggio (concentrazione, tempo e lunghezza).
- Tracciare la concentrazione normalizzata in funzione del tempo e il profilo di concentrazione attraverso l'asse x con un'approssimazione polinomiale ai dati.
- Calcolare il coefficiente di diffusione utilizzando il codice software di calcolo.
5. Misure sperimentali
- Riferendosi di nuovo alla equazione di diffusione, riscrivere l'equazione per determinare la concentrazione specifica in qualsiasi posizione all'interno del sistema, come mostrato in seguito.
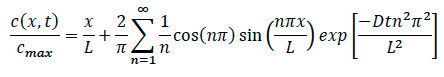
Dove:
L = Lunghezza del the matrice di collagene
x = posizione sotto inchiesta
D = coefficiente di diffusione
t = tempo trascorso - Durante gli esperimenti di chemiotassi, assicurarsi di registrare il tempo specifico che il momento chemochina è stato aggiunto al sistema. Una volta trovato il movimento migrazione delle cellule, assicurarsi di registrare confocale time-lapse, e prendere nota del tempo specifico e la posizione dal bordo dove si è verificato l'imaging.
- Durante l'analisi dei dati, inserire il noto tempo trascorso, la posizione e il coefficiente di diffusione per la cella specifica nell'equazione per trovare la concentrazione specifica normalizzata in qualsiasi punto all'interno del collagene sia per diffusione e esperimenti sulle cellule.
6. Monitoraggio migrazione cellulare Utilizzando TFM
- Per monitorare la migrazione delle cellule utilizzando tecniche TFM / DVC seguire il protocollo di seguito:
- Dopo l'aggiunta di concentrazione chemoattractant desiderato nel pozzetto 3 #, avviare l'imaging delle matrici di collagene 3D con un sistema confocale montato su un micr invertitaoscope per trovare una cella per indagare.
- Una volta che una cellula in movimento viene trovato, posizionare la cella al centro del campo visivo e utilizzare un motore piezoelettrico (all'immagine in asse z), un'immagine ogni 1 o 2 min.
- Per calcolare la generazione di forza cellulare e deformazione, generare un codice di calcolo per trovare gli spostamenti delle perline fluorescenza seguenti protocolli TFM / DVC 31.
Risultati
La capacità di questo test per valutare con precisione la migrazione della cella si basa su una buona configurazione del sistema. Pertanto, è fondamentale per fare in modo di progettare lo stampo sistema di diffusione in modo accurato e fare molta attenzione nel porre coprioggetto sia idrofobe e idrofile, come illustrato nella figura 1. Se il sistema è correttamente progettato e durante la fase di modellazione diffusione garantendo trovare molto buona linea di partenza lineare, si è in grado di ragg...
Discussione
Le fasi più critiche per esperimenti di diffusione di successo con o senza cellule sono: impostare correttamente il gruppo di forma; sviluppare la manualità necessaria per evitare danni durante l'estrazione dei vetrini idrofobici; garantendo di trovare una buona linea di partenza lineare per calcolare correttamente il coefficiente di diffusione; correggere i calcoli sperimentali di collagene e chemiotattico; utilizzare correttamente del sistema di imaging cellulare dal vivo per assicurare matrice non si secca; e m...
Divulgazioni
The authors declare that they have no competing financial interests.
Riconoscimenti
The authors would like to acknowledge Drs. Jonathan Reichner and Angle Byrd for cell experiment insight. The National Science Foundation Graduate Research Fellowship Program (GRFP) supported this work.
Materiali
| Name | Company | Catalog Number | Comments |
| Silicone elastomer kit | Dow Corning Corp | 182 SIL ELAST KIT .5KG | a two-part misture with a 10:1 mix |
| Live cell imaging chamber | Live Cell Instrument | CM-B18-1 | CMB for 18 mm round coverslips |
| 22 mm Glass Coverslip | Fisher Scientific | NC0180281 | Neuvitro Corp. cover slip 22 mm 1.5 |
| Machined aluminum metal cube | |||
| Hobby utility knife | X-Acto | X3201 | |
| 3-(aminopropyl) trimethoxysilane | Sigma-Aldrich | 281778-5ML | |
| Glutaraldehyde | Polysciences, Inc | 00216A-10 | Glutaraldehyde, EM Grade, 8% |
| 50 ml tube | Fisher Scientific | 14-432-22 | Standard floor model and tabletop centrifuges |
| Glass Petri dish | Fisher Scientific | 08-747A | Reusable Petri Dishes: Complete (60 x 15 mm) |
| Forceps | Fisher Scientific | 22-327-379 | Fine Point Forceps |
| Cover glasses | Fisher Scientific | 12-518-105A | Rectangle; 30 x 22 mm; Thickness No. 1 |
| Tridecafluoro-1,1,2,2-tetrahydrooctyl | Gelest | SIT8174.0 | |
| Acetic acid | Sigma-Aldrich | 320099 | Acetic acid ACS reagent, ≥99.7% |
| Hexane | Sigma-Aldrich | 296090 | anhydrous, 95% |
| Ethyl alcohol | Sigma-Aldrich | E7023 | 200 proof, for molecular biology |
| High-precision diamond scribing tool | Lunzer | PV-081-3 | Straight extended tip scribe, .020" (.50 mm) diameter by .200" (5.0 mm) tip length |
| Vacuum grease | Dow Corning | 14-635-5C | High-Vacuum Grease |
| 15 ml tube | Fisher Scientific | 14-959-49D | 15 ml conical centrifuge tubes with hydrophobic, biologically inert surface |
| 10x phosphate buffered solution | Fisher Scientific | BP399-500 | 1.37 M Sodium Chloride, 0.027 M Potassium Chloride, and 0.119 M Phosphate Buffer |
| 1 N sodium hydroxide | Sigma-Aldrich | 38215 | Sodium hydroxide concentrate |
| Collagen I, rat tail | BD Biosciences | 354236 | Rat tail |
| Micro centrifuge tube | Fisher Scientific | 02-681-332 | Volume: 2.0 ml; O.D. x L: 13 x 40 mm; sterile; single-wrapped |
| [header] | |||
| Carboxylate-modified microspheres | Invitrogen | F-8813 | Carboxylate-modified microspheres, 0.5 µm, yellow-green fluorescent (505/515), 2% solids |
| Rhodamine | Sigma-Aldrich | 83689 | Rhodamine B for fluorescence |
Riferimenti
- Adzick, N. S. The Molecular and Cellular Biology of Wound Repair. Ann Surg. 225 (2), 236 (1997).
- Reddi, A. H. Bone morphogenetic proteins: an unconventional approach to isolation of first mammalian morphogens. Cytokine Growth F. R. 8 (1), 11-20 (1997).
- Coussens, , Werb, Z, L. M., Werb, Z. Inflammation and cancer. Nature. 420 (6917), 860-867 (2002).
- Vital-Lopez, F. G., Armaou, A., Hutnik, M., Maranas, C. D. Modeling the effect of chemotaxis on glioblastoma tumor progression. AIChE J. 57 (3), 778-792 (2011).
- Hughes-Alford, S. K., Lauffenburger, D. A. Quantitative analysis of gradient sensing: towards building predictive models of chemotaxis in cancer. Curr. Opin. Cell Biol. 24 (2), 284-291 (2012).
- Friedl, P., Gilmour, D. Collective cell migration in morphogenesis, regeneration and cancer. Nat. Rev. Mol Cell Bio. 10 (7), 445-457 (2009).
- Cristini, V. Morphologic Instability and Cancer Invasion. Clin. Cancer Res. 11 (19), 6772-6779 (2005).
- Lammermann, T., et al. Rapid leukocyte migration by integrin-independent flowing and squeezing. Nature. 453 (7191), 51-55 (2008).
- Parent, C. A., Devreotes, P. N. A cell's sense of direction. Science. 284 (5415), 765-770 (1999).
- Boyden, S. The chemotactic effect of mixtures of antibody and antigen on polymorphonuclear leucocytes. J. Exp. Med. 115, 453-466 (1962).
- Nelson, R. D., Quie, P. G., Simmons, R. L. Chemotaxis under agarose: a new and simple method for measuring chemotaxis and spontaneous migration of human polymorphonuclear leukocytes and monocytes. J. Immunol. 115 (6), 1650-1656 (1975).
- Rosoff, W. J., McAllister, R., Esrick, M. A., Goodhill, G. J., Urbach, J. S. Generating controlled molecular gradients in 3D gels. Biotechnol. Bioeng. 91 (6), 754-759 (2005).
- Wells, A., Kassis, J., Solava, J., Turner, T., Lauffenburger, D. A. Growth factor-induced cell motility in tumor invasion. Acta Oncol. 41 (2), 124-130 (2002).
- Wilkinson, P. C. Assays of leukocyte locomotion and chemotaxis. J. Immunol. Methods. 216 (1-2), 139-153 (1998).
- Bonvin, C., Overney, J., Shieh, A. C., Dixon, J. B., Swartz, M. A. A multichamber fluidic device for 3D cultures under interstitial flow with live imaging: development, characterization, and applications. Biotechnol. Bioeng. 105 (5), 982-991 (2010).
- Noo, L. J., et al. Neutrophil chemotaxis in linear and complex gradients of interleukin-8 formed in a microfabricated device. Nat. Biotechnol. 20 (8), 826-830 (2002).
- Shao, J., et al. Integrated microfluidic chip for endothelial cells culture and analysis exposed to a pulsatile and oscillatory shear stress. Lab Chip. 9 (21), 3118-3125 (2009).
- Haessler, U., Kalinin, Y., Swartz, M. A., Wu, M. An agarose-based microfluidic platform with a gradient buffer for 3D chemotaxis studies. Biomed. Microdevices. 11 (4), 827-835 (2009).
- Friedl, P., Wolf, K. Tumour-cell invasion and migration: diversity and escape mechanisms. Nat. Rev. Cancer. 3 (5), 362-374 (2003).
- Sixt, M., Lammermann, T. In vitro analysis of chemotactic leukocyte migration in 3D environments. Methods Mol. Biol. 769, 149-165 (2011).
- Zigmond, S. H. Ability of polymorphonuclear leukocytes to orient in gradients of chemotactic factors. J. Cell Biol. 75 (2 Pt 1), 606-616 (1977).
- Zicha, D., Dunn, G. A., Brown, A. F. A new direct-viewing chemotaxis chamber. J. Cell. Sci. 99 (Pt 4), 769-775 (1991).
- Gilbert, D. L. Macromolecular diffusion through collagen membranes. Int. J. Pharm. 47 (1-3), 79-88 (1988).
- Ramanujan, S., et al. Diffusion and convection in collagen gels: implications for transport in the tumor interstitium. Biophys. J. 83 (3), 1650-1660 (2002).
- Weadock, K., Silver, F. H., Wolff, D. Diffusivity of 125I-calmodulin through collagen membranes: effect of source concentration and membrane swelling ratio. Biomaterials. 7 (4), 263-267 (1986).
- Erikson, A., Andersen, H. N., Naess, S. N., Sikorski, P., Davies, C. d. L. Physical and chemical modifications of collagen gels: impact on diffusion. Biopolymers. 89 (2), 135-143 (2008).
- Moghe, P. V., Nelson, R. D., Tranquillo, R. T. Cytokine-stimulated chemotaxis of human neutrophils in a 3-D conjoined fibrin gel assay. J. Immunol. Methods. 180 (2), 193-211 (1995).
- Sundd, P., et al. Live cell imaging of paxillin in rolling neutrophils by dual-color quantitative dynamic footprinting. Microcirculation. 18 (5), 361-372 (2011).
- Sanders, E. R. Aseptic Laboratory Techniques: Volume Transfers with Serological Pipettes and Micropipettors. J. Vis. Exp. (63), 2754 (2012).
- Stephenson, F. H. . Calculations for Molecular Biology and Biotechnology: A Guide to Mathematics in the Laboratory 2e. , (2010).
- Bar-Kochba, E., Toyjanova, J., Andrews, E., Kim, K., Franck, C. A Fast Iterative Digital Volume Correlation Algorithm for Large Deformations. Exp. Mech. In press, (2014).
- Shenoy, V., Rosenblatt, J. Diffusion of Macromolecules in Collagen and Hyaluronic Acid, Rigid-Rod-Flexible Polymer Composite Matrixes. Macromolecules. 28 (26), 8751-8758 (1995).
- Leddy, H., Guilak, F. Site-Specific Molecular Diffusion in Articular Cartilage Measured using Fluorescence Recovery after Photobleaching. Ann. Biomed. Eng. 31 (7), 753-760 (2003).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon