È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Un ELISA Based rilegatura e metodo concorrenza di determinare con rapidità interazioni ligando-recettore
In questo articolo
Riepilogo
The presented protocols describe two enzyme-linked immunosorbent assay (ELISA) based techniques for the rapid investigation of ligand-receptor interactions: The first assay allows the determination of dissociation constant between ligand and receptor. The second assay enables a rapid screening of blocking peptides for ligand-receptor interactions.
Abstract
A comprehensive understanding of signaling pathways requires detailed knowledge regarding ligand-receptor interaction. This article describes two fast and reliable point-by-point protocols of enzyme-linked immunosorbent assays (ELISAs) for the investigation of ligand-receptor interactions: the direct ligand-receptor interaction assay (LRA) and the competition LRA. As a case study, the ELISA based analysis of the interaction between different lambda interferons (IFNLs) and the alpha subunit of their receptor (IL28RA) is presented: the direct LRA is used for the determination of dissociation constants (KD values) between receptor and IFN ligands, and the competition LRA for the determination of the inhibitory capacity of an oligopeptide, which was designed to compete with the IFNLs at their receptor binding site. Analytical steps to estimate KD and half maximal inhibitory concentration (IC50) values are described. Finally, the discussion highlights advantages and disadvantages of the presented method and how the results enable a better molecular understanding of ligand-receptor interactions.
Introduzione
Una comprensione completa delle vie di segnalazione richiede una conoscenza dettagliata circa l'interazione ligando-recettore. La maggior parte dei metodi per valutare l'interazione di un particolare ligando con il recettore specifico sono costosi, in termini di tempo, manodopera e richiedono attrezzature e competenze specifiche 1.
Questo articolo descrive due protocolli veloce e affidabile punto per punto per studiare l'interazione ligando-recettore sulla base di un enzima collegato immunosorbent assay (ELISA): il saggio di interazione ligando-recettore diretta (LRA) e l'LRA concorrenza. ELISA è una tecnica molto sensibile, specifico e facilmente disponibile, solitamente usata in quasi ogni laboratorio. ELISA può essere eseguito e adattato in varie mode. I protocolli presentati sono ottimizzati per la ricerca delle interazioni tra i diversi interferoni lambda (INFLs) e la loro recettore.
L'LRA diretta consente una quantificatisu ligando-recettore vincolante rispetto alla concentrazione del ligando e produce pertanto una curva vincolante. Usando un modello appropriato per l'interazione ligando-recettore, i dati possono essere ulteriormente analizzati per stimare la costante di dissociazione (K D).
Nel protocollo presentato, l'equazione Hill comunemente usato è applicata per modellare il legame ligando-recettore. Sebbene altri metodi come la superficie plasmon risonanza a 2,3 consentono la determinazione delle affinità di legame tra due proteine, questa tecnologia è spesso laborioso, costoso e richiede attrezzature speciali di laboratorio.
L'LRA concorrenza consente la proiezione di peptidi inibitori: il legame ligando-recettore viene quantificata rispetto alla concentrazione di peptide. Questo produce una curva dose-risposta che descrive l'effetto inibitorio del peptide. I dati possono essere ulteriormente analizzati per stimare la metà massima concentrazione inibitoria (IC 50 ) del peptide bloccante.
Entrambi protocolli ELISA sono facili da usare e possono essere adattati ad una vasta gamma di domande di ricerca. proteine ricombinanti di qualsiasi tipo possono essere utilizzati per determinare in modo affidabile e veloce le parti di interazione. Inoltre, l'Lra concorrenza può essere utilizzato per determinare siti di interazione critici di ligandi e recettori utilizzando peptidi di blocco, che sono progettati per imitare sia il ligando o il recettore. Se il peptide bloccante mostra l'inibizione efficace e specifico, il peptide occupa un sito critico interazione del ligando (se imita peptidici del recettore) o del ligando (se imita peptide ligando).
Il primo protocollo descrive la determinazione K D valore delle diverse INFLs e la subunità alfa del loro recettore, cioè il recettore dell'interleuchina-28 (IL28RA) usando l'LRA diretta. Successivamente, il secondo protocollo mostra come determinare la capacità di un lungo peptide di 20 aminoacidi diinibire le interazioni INFL-IL28RA. Il peptide è stato progettato per competere con IFNLs al loro recettore sito di legame e consente così una comprensione molecolare dell'interazione. Inoltre, questo peptide può essere utilizzato per bloccare IL28RA in esperimenti in vitro per determinare l'impatto sugli effetti di segnalazione a valle 4.
Protocollo
1. Preparazione dei reagenti
- Per preparare un tampone di rivestimento carbonato, sciogliere 0,36 g di Na 2 CO 3 e 0,84 g NaHCO 3 in 100 ml di acqua distillata; filtro sterile il buffer utilizzando un vuoto spinto 0,22 micron polietersulfone (PES) filtro a membrana e conservare a temperatura ambiente fino a quando l'utilizzo.
- Preparare la soluzione di lavaggio con l'aggiunta di 0,05% v / v Tween 20 in tampone fosfato (PBS).
- Preparare un 5% di sieroalbumina bovina (BSA) (soluzione bloccante) in soluzione PBS sciogliendo 5 g BSA (≥98%) in 100 ml di PBS e conservare a 4 ° C.
- Ricombinante del recettore, leganti e peptidi di blocco
- Reconstitutethe ricombinante subunità umano recettore dell'interleuchina alfa (IL28RA) e ricombinanti ligandi His-tag di IFN umano (IFNL1-3) secondo le istruzioni del produttore e conservare a -80 ° C. Sintetizzare peptidi blocco e utilizzato come precedentemente descritto 4. Utilizzare PBS per preparare differenti concentrazioni di ligandos e peptidi per l'uso nei saggi.
- Per preparare l'anticorpo primario, diluire 6x suo mouse anticorpo monoclonale in PBS con 0,1% BSA a 1: 1000 diluizione. Per preparare l'anticorpo secondario, diluire perossidasi di rafano (HRP) capra coniugato anti-topo IgG (H + L) in PBS con 0,1% BSA a 1: 10.000 diluizione.
- Preparare la soluzione TMB miscelando il reagenti A e B secondo le istruzioni del produttore.
- Preparare la soluzione di arresto con l'aggiunta di 5 N acido solforico (H 2 SO 4) in acqua distillata e conservare a temperatura ambiente.
2. immunoenzimatico Assays (ELISA)
NOTA: Il diretta interazione ligando-recettore ELISA (Lra diretta, Figura 1) può essere utilizzato per misurare la dissociazione recettore-ligando costante (K D), come misura della affinità di legame del recettore-ligando. L'interazione concorrenza ligando-recettore ELISA (LRA concorrenza, figura 2) tuttoflussi di screening di peptidi (e altri composti di blocco), che agiscono di interferire con l'interazione tra ligando e recettore. Il protocollo di base che è stato precedentemente pubblicato 5 è stata ulteriormente ottimizzata.
NOTA: in entrambe ELISA metodi utilizzano pipetta multicanale per l'aggiunta di soluzioni ai pozzetti di 96 pozzetti in ogni passaggio. Nella soluzione decantare o fasi di lavaggio, buttare fuori le soluzioni direttamente nel lavandino.
- Diretto ligando-recettore-Interaction Assay (LRA continua)
NOTA: Per una illustrazione del flusso di lavoro (vedere Figura 1).- Piatto del rivestimento con il recettore ricombinante
- Diluire il recettore ricombinante in tampone carbonato a una concentrazione finale di 100 ng / ml. pozzi mano di 96 pozzetti micropiastra con la concentrazione dei recettori fisso (100 ng / ml) pipettando 100 ul per ogni pozzetto usando una pipetta multicanale. Escludere muri esterni della piastra per evitare così artefatto bordo. Coprire la piastra con un coperchio e incubare latarga a 4 ° CO / N.
- Blocco e aggiunta di leganti
- Il giorno seguente, togliere la soluzione di rivestimento inclinando il piatto contro il lavandino e lavare la piastra 3 volte con soluzione di lavaggio (PBS + 0,05% v / v Tween 20).
- Bloccare i siti recettoriali di legame liberi nella piastra rivestita con 200 ml di soluzione di BSA 5% in ogni pozzetto con una pipetta multicanale e incubare la piastra per 2 ore a temperatura ambiente.
- Eliminare la soluzione di saturazione (vedi punto 2.1.2.1.) E lavare la piastra 3 volte con soluzione di lavaggio.
- Preparare il ricombinante His-tag ligandi a diverse concentrazioni (ad esempio, 8 mg / ml, 4 mg / ml, 2 mg / ml, 1 mg / ml, 0,5 mg / ml, 0,25 mg / ml, 0,125 mg / ml, 0,063 mcg / ml, 0,031 mg / ml, 0,0 mg / ml) in PBS. Aggiungere solo PBS nei pozzetti del bianco.
- Aggiungere 100 ml di ciascuna concentrazione ligando ai pozzetti in duplicato e incubare la piastra per 2 ore a temperatura ambiente permettendo recettore-ligandointerazione.
- L'incubazione con anticorpi
- Dopo l'incubazione con i ligandi, lavare la piastra 3 volte con soluzione di lavaggio.
- Dispensare 100 l di suo anti-soluzione principale del mouse anticorpo monoclonale (1: 1.000) in ogni pozzetto.
- Incubare la piastra a temperatura ambiente per 2 ore; Dopo l'incubazione, scartare la soluzione di anticorpi (vedi punto 2.1.2.1.) e lavare la piastra 3 volte con soluzione di lavaggio.
- Aggiungere 100 ml di HRP accoppiati capra soluzione di anticorpo secondario anti-topo IgG (1: 10.000) in ogni pozzetto. Incubare la piastra per 45 minuti a temperatura ambiente.
- Scartare la soluzione di anticorpi (vedi punto 2.1.2.1.) E lavare la piastra 3 volte con soluzione di lavaggio.
- L'aggiunta di substrato e lo Sviluppo
- Portare le soluzioni del substrato TMB a RT e preparare TMB soluzione di substrato A e B in rapporto 1: 1. Aggiungere 100 ml preparati al momento substrato di ogni bene e mantenere la piastra a temperatura ambiente per 15-30 minuti. Dopo col sufficienti o sviluppo aggiungere la soluzione di arresto 50 ml.
- Leggere la piastra e analisi dei dati
NOTA: Il protocollo descritto si basa sul presupposto che il segnale misurato sale dal legame specifico. Potrebbe essere necessario stimare il contributo di legame aspecifico al segnale ma questo è fuori della portata di questo protocollo.- Leggere l'assorbanza (densità ottica, OD) direttamente a 450 nm.
- Sottrarre il segnale di fondo ai valori della DO misurati e li normalizza. Trasformare tutti i valori della concentrazione di ligando in scala logaritmica (base 10, log 10).
- Tracciare la normalizzata e di fondo corretta valori di OD (asse Y, corrisponde alla frazione di siti di legame del recettore occupati) contro il logaritmo della concentrazione di ligando (asse X, log scala 10).
- Per stimare il valore di K D, inserire i dati per il seguente modulo dell'equazione Hill:
tp_upload / 53575 / 53575eq1.jpg "/>
NOTA: Qui Y indica la frazione dei siti occupati del recettore di legame e Y max il legame massimo; [L] indica la concentrazione di legante libero e il coefficiente di Hill. Se vi è un solo sito di legame per il ligando, il coefficiente di Hill è n = 1. Per sistemi con più di un ligando sito di legame, l'esposizione vincolanti cooperatività positivo se n> 1, cooperatività negativa se n <1 e non cooperatività se n = 1. la costante di dissociazione microscopica è definito e corrisponde alla metà effettiva concentrazione massima EC 50 6. La costante di dissociazione apparente è K d = (K D) n. Nel caso più semplice in cui n = 1, la dissociazione corrisponde costanti a concentrazione ligando in cui la metà del recettore siti di legame sono occupati e K d = K D. Questo modello assume azione di massa vincolante in condizioni di equilibrio, e che soltanto una piccola frazione diil legante aggiunto è legato al recettore, cioè, [L] >> [RL].
- Piatto del rivestimento con il recettore ricombinante
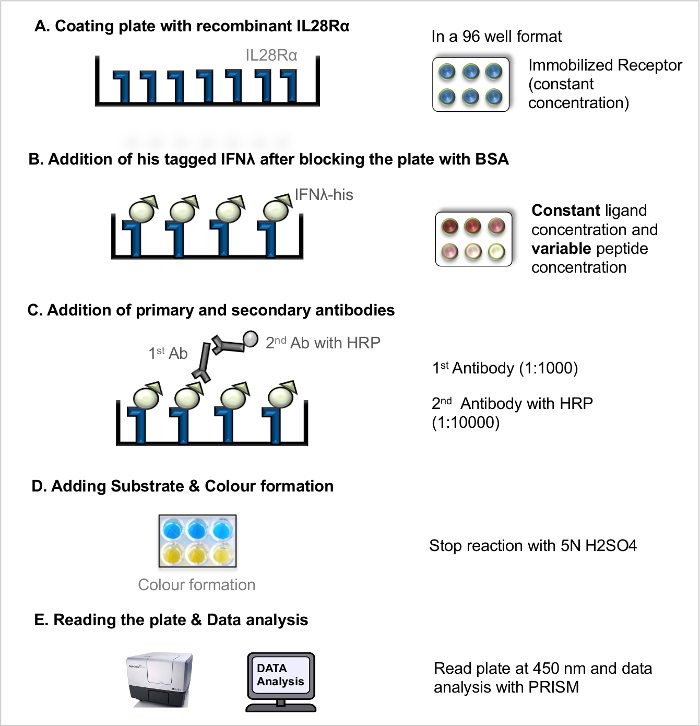
Figura 1. saggio diretto ligando-recettore interazione (LRA diretta). Step-by-step di protocollo per LRA diretta. Si prega di cliccare qui per vedere una versione più grande di questa figura.
- Concorso ligando-recettore-Interaction Assay (competizione LRA)
NOTA:. Per un'illustrazione del flusso di lavoro vedi Figura 2 La procedura di concorso LRA segue la stessa procedura come il LRA diretta (rivestimento della piastra, l'incubazione anticorpo, sviluppo targa) ad eccezione di importanti cambiamenti nel legante e peptidi passo oltre. Regolari controlli negativi sono essenziali per questo test. In uno studio di screening precedente 4, il peptide bloccando strapazzate non ha mostrato effetti antagonisti.- Blocco - aggiunta di leganti e peptidi di blocco
- Il giorno seguente, togliere la soluzione di rivestimento e lavare la piastra (vedi 2.1.2.1).
- Bloccare la piastra rivestita con l'aggiunta di 200 ml di soluzione di BSA 5% in ogni pozzetto ed incubare la piastra per 2 ore a temperatura ambiente.
- Preparare i ligandi His-tag ricombinanti (IFNL1-3) ad una concentrazione fissa (2x-20 ng / ml) in PBS.
- Preparare il peptide bloccante (vedi tabella 3) con differenti concentrazioni comprese tra 10 nM e 100 pM in PBS per garantire una curva dose-risposta.
NOTA: Ciò consente successiva determinazione del valore IC50 per il peptide di blocco. In pozzetti di controllo, aggiungere concentrazione ligando solo fisso senza peptide di ricavare il massimo (100%) vincolante. Nel vuoto, solo aggiungere PBS senza legante o peptide. - Aggiungere 50 ml di ligandi (IFNL1-3) e 50 ml di ogni peconcentrazione ptide ai pozzetti di duplicati.
- Incubare la piastra per 2 ore a temperatura ambiente.
- Leggere la piastra e analisi dei dati
NOTA: Il protocollo descritto si basa sul presupposto che il segnale misurato sale dal legame specifico. Potrebbe essere necessario stimare il contributo di legame aspecifico al segnale ma questo è fuori della portata di questo protocollo.- Leggere l'assorbanza (densità ottica, OD) direttamente a 450 nm.
- Sottrarre il segnale di fondo ai valori della DO misurati e li normalizza. Trasformare tutti i valori della concentrazione di peptide in scala logaritmica (base 10, log 10).
- Tracciare la normalizzata e di fondo corretta valori di OD (asse Y, corrisponde alla frazione di siti di legame del recettore occupati) contro il logaritmo della concentrazione di ligando (asse X, log scala 10).
- Per stimare il valore di IC 50, inserire i dati alla seguente equazione:
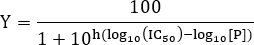
NOTA: Qui [P] è la concentrazione del peptide ed il pendio della collina. La pendenza Hill descrive la pendenza della curva dose-risposta. La IC 50 corrisponde alla concentrazione dell'inibitore alla quale si osserva il 50% di inibizione del legame tra il ligando e recettore.
- Blocco - aggiunta di leganti e peptidi di blocco

Figura 2. Concorrenza ligando-recettore interazione saggio (concorrenza LRA). Step-by-step di protocollo per LRA concorrenza. Cliccate qui per vedere una versione più grande di questa figura.
Risultati
Le costanti di dissociazione tra INFL1-3 e loro recettore subunità alfa IL28RA sono stati determinati utilizzando il LRA diretta. I risultati sono mostrati in Figura 3: La frazione dei siti di legame occupati è tracciata contro il logaritmo della rispettiva concentrazione di IFN. La trama Scatchard dei dati è mostrata nell'angolo in basso a destra. I risultati illustrano che l'Lra diretta produce una curva di legame, che può essere ulteriormente analizzati pe...
Discussione
ELISA è uno standard e il metodo consolidato per molti laboratori. Abbiamo ulteriormente modificato e migliorato un metodo 5,7 precedentemente pubblicato. Il protocollo passo-passo dimostrato mostra come può essere utilizzato in modo semplice per determinare i valori K D di interazioni ligando-recettore. Inoltre, la IC50 di un peptide bloccante che interferisce con l'interazione ligando-recettore può essere determinato.
I principali vantaggi sono la rapida messa ...
Divulgazioni
A.E. was supported by a research grants from the "SNSF Ambizione Score" program (PZ00P3_154709), "Forschungsfond, Förderung strategischer Projekte" University of Basel, Stiftungsinfektionskrankheiten Basel, and Bangeter Rhyner Stiftung. J.L. acknowledges support by an iPhD fellowship of the SystemsX.ch initiative in systems biology program (9th call).
Riconoscimenti
We thank Prof. J. Stelling (Department of Biosystems Science and Engineering, ETH Zurich and Swiss Institute for Bioinformatics, Basel, Switzerland) for his critical review of the manuscript.
Materiali
| Name | Company | Catalog Number | Comments |
| Nunc-Immunoplate (F96 Maxi sorp) | Thermo Scientific | 442404 | ELISA plate |
| Sodium carbonate (Na2CO3) | Merck | 497-19-8 | For ELISA plate coating buffer |
| Sodium hydrogen carbomnate(NaHCO3) | Merck | 144-55-8 | For ELISA plate coating buffer |
| Bovine Serum Albumin (BSA) | Sigma | A7030-100G | 5% BSA in PBS for Blocking |
| rhIL-28Rα/IFNλR1 | R&D systems | 5260-MR | Recombinant human interlukin-28 Receptor alpha |
| rhIL-29/IFNλ1 | R&D systems | 1598-IL/CF | Recombinant human interlukin-29/Carrier free/C-terminal 10-His tag |
| rhIL-28A/IFNλ2 | R&D systems | 1587-IL/CF | Recombinant human interlukin-28A/Carrier free/C-terminal 6-His tag |
| rhIL-28B/IFNλ3 | R&D systems | 5259-IL/CF | Recombinant human interlukin-28B/Carrier free/C-terminal 6-His tag |
| 6x His Monoclonal antibody (Mouse) | Clontech | 631212 | Primary antiboy to capture His tagged Ligands |
| Goat anti-Mouse igG (H+L) | Jackson Immuno Research | 115-035-166 | Horseradish Peroxidase conjucated secondary antibody |
| BDoptEIA TMB reagent set | BD Biosciences | 555214 | ELISA - TMB substrate solution |
| Sulfuric acid (H2SO4) | Fulka | 84720 | 5 N H2SO4 (Enzyme reaction stop solution) |
| Synergy/H1 - Microplate reader | BioTeK | ELISA plate reader |
Riferimenti
- Schneider, P., Willen, L., Smulski, C. R. Tools and techniques to study ligand-receptor interactions and receptor activation by TNF superfamily members. Methods in enzymology. 545, 103-125 (2014).
- Rossi, G., et al. Biosensor analysis of anti-citrullinated protein/peptide antibody affinity. Analytical biochemistry. 465, 96-101 (2014).
- van der Merwe, P. A., Barclay, A. N. Analysis of cell-adhesion molecule interactions using surface plasmon resonance. Curr Opin Immunol. 8, 257-261 (1996).
- Egli, A., et al. IL-28B is a key regulator of B- and T-cell vaccine responses against influenza. PLoS Pathog. 10, e1004556 (2014).
- Rosenbluh, J., et al. Positively charged peptides can interact with each other, as revealed by solid phase binding assays. Analytical biochemistry. 352, 157-168 (2006).
- Goutelle, S., et al. The Hill equation: a review of its capabilities in pharmacological modelling. Fundamental & clinical pharmacology. 22, 633-648 (2008).
- Levin, A., et al. Peptides derived from HIV-1 integrase that bind Rev stimulate viral genome integration. PLoS One. 4, e4155 (2009).
- Egli, A., Santer, M. D., O'Shea, D., Tyrrell, D. L., Houghton, M. The impact of the interferon-lambda family on the innate and adaptive immune response to viral infections. Emerging infectious diseases. , e51 (2014).
- Gad, H. H., Hamming, O. J., Hartmann, R. The structure of human interferon lambda and what it has taught us. J Interferon Cytokine Res. 30, 565-571 (2010).
- Folch, B., Rooman, M., Dehouck, Y. Thermostability of salt bridges versus hydrophobic interactions in proteins probed by statistical potentials. Journal of chemical information and modeling. 48, 119-127 (2008).
- Yuzlenko, O., Lazaridis, T. Interactions between ionizable amino acid side chains at a lipid bilayer-water interface. The journal of physical chemistry. B. 115, 13674-13684 (2011).
- Tissot, A. C., Vuilleumier, S., Fersht, A. R. Importance of two buried salt bridges in the stability and folding pathway of barnase. Biochemistry. 35, 6786-6794 (1996).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon