È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Tutto il corpo Imaging Mass Spectrometry da Infrared Matrix-Assisted Laser desorbimento per ionizzazione elettrospray (IR-Maldesi)
In questo articolo
Riepilogo
A mass spectrometry imaging (MSI) source operated at atmospheric pressure was developed by coupling mid-infrared laser desorption and electrospray post-ionization. Exogenous ice matrix was used as the energy-absorbing matrix to facilitate resonant desorption of tissue-related material. This manuscript provides a step-by-step protocol for performing IR-MALDESI MSI of whole-body neonatal mouse.
Abstract
fonti di ionizzazione ambientali per la spettrometria di massa (MS) sono stati oggetto di molto interesse negli ultimi dieci anni. Matrix-assisted laser desorbimento per ionizzazione elettrospray (Maldesi) è un esempio di tali metodi, in cui le caratteristiche del laser desorbimento / ionizzazione assistita dalla matrice (MALDI) (ad esempio, la natura pulsata di desorbimento) e la ionizzazione elettrospray (ESI) (ad esempio, soft-ionizzazione ) sono combinati. Uno dei principali vantaggi di Maldesi è la sua versatilità intrinseca. In esperimenti Maldesi, un ultravioletta (UV) o laser a infrarossi (IR) possono essere utilizzate per eccitare una matrice di risonanza endogena o esogena. La scelta di matrice non è analita dipendente, e dipende esclusivamente dalla lunghezza d'onda del laser utilizzato per l'eccitazione. In esperimenti IR-Maldesi, un sottile strato di ghiaccio viene depositato sulla superficie del campione come matrice ad assorbimento di energia. La geometria sorgente IR-Maldesi è stato ottimizzato utilizzando il modello statistico degli esperimenti (DOE) per l'analisi di campioni liquidi, nonché Biolcampioni di tessuto ogical. Inoltre, una robusta fonte di imaging IR-Maldesi è stato sviluppato, in cui un laser sintonizzabile mid-IR è sincronizzato con un computer controllato stadio XY traslazionale e uno spettrometro di massa ad alta potere risolutivo. Un'interfaccia utente grafica personalizzata (GUI) consente all'utente di selezionare la frequenza di ripetizione del laser, il numero di scatti per voxel, step-dimensioni del palcoscenico del campione, e il ritardo tra il desorbimento e scansione eventi per la fonte. IR-Maldesi è stato utilizzato in varie applicazioni come l'analisi forense delle fibre e coloranti e MSI di sezioni di tessuto biologico. Distribuzione di diversi analiti che vanno dai metaboliti endogeni a xenobiotici esogene all'interno di sezioni di tessuto può essere misurato e quantificato utilizzando questa tecnica. Il protocollo presentato in questo manoscritto descrive le principali misure necessarie per IR-Maldesi MSI di sezioni di tessuto di tutto il corpo.
Introduzione
imaging con spettrometria di massa (MSI) in modalità microsonda comporta desorbimento del campione da una superficie da una trave (laser o ioni) in posizioni discrete sulla superficie di un campione. Ad ogni punto raster, uno spettro di massa viene generato e gli spettri acquisiti, insieme con la posizione spaziale da cui sono stati raccolti, può essere utilizzato per mappare simultaneamente numerosi analiti nel campione. Questo modo senza etichetta delle immagini accoppiata alla sensibilità e la specificità della spettrometria di massa hanno contribuito MSI diventare uno dei campi più rapida evoluzione in spettrometria di massa 1,2.
Matrix-assisted laser desorbimento / ionizzazione (MALDI) è il metodo di ionizzazione più comune utilizzato per le analisi MSI. Tuttavia, la necessità di una matrice organica e le esigenze aspirapolvere della MALDI pongono limiti significativi sulla riproducibilità, la produttività del campione, ed i tipi di campioni che possono essere analizzati utilizzando il metodo. Un certo numero di pressione atmosferica (AP) iometodi nizzazione sono stati sviluppati negli ultimi anni per aggirare queste restrizioni 3. Questi metodi di ionizzazione ambiente consentono l'analisi di campioni biologici in un ambiente che è molto più vicino al loro stato naturale e semplificare procedura di preparazione del campione prima dell'analisi. Matrix-assisted laser desorbimento per ionizzazione elettrospray (Maldesi) è un esempio di un tale metodo di ionizzazione 4,5.
In esperimenti IR-Maldesi, un sottile strato di ghiaccio è depositato sulla superficie del tessuto come matrice ad assorbimento di energia. Un impulso laser mid-IR viene assorbita dalla matrice di ghiaccio, e facilita desorbimento dei materiali neutri dalla superficie risonante eccitando il OH modalità di acqua di filatura. La neutri partizione desorbita nelle goccioline cariche di un elettrospray ortogonali e sono post-ionizzato in modo ESI-like 4-6. L'aggiunta di matrice ghiaccio esogeno è preferito basandosi unicamente su acqua endogena nel tessuto in quanto contribuisce accontare le variazioni nel contenuto di acqua in differenti compartimenti, e ha dimostrato di migliorare desorbimento 6 e migliorare abbondanza ionico facendo ~ 15 volte 7,8 negli esperimenti di imaging tessuto.
In questo lavoro, utilizziamo IR-Maldesi MSI per suscitare la distribuzione dei metaboliti in diversi organi in tutto il corpo del mouse neonatale. Una panoramica dei parametri modificabili della sorgente IR-Maldesi è dato, e sono dimostrati i passi necessari per l'imaging di successo di sezioni di tessuto.
Protocollo
Nota: Il protocollo seguente descrive tutti i passi necessari per l'esecuzione di esperimenti di IR-Maldesi MSI. Approfondita dettagli circa la geometria ottimizzata della sorgente IR-Maldesi e la sua sincronizzazione con il laser, palco, e spettrometro di massa si possono trovare altrove 5,6. campioni di tessuti animali utilizzati in questo protocollo sono stati ottenuti secondo Institutional Animal Care e Usa Comitato (IACUC) e regolamenti North Carolina State University.
1. Preparazione del tessuto
- Preparare un / bagno di ghiaccio secco isopentano ponendo ~ 200 ml di isopentano in un becher pulito dentro un contenitore secondario di ghiaccio secco in una cappa aspirante. Utilizzare guanti e occhiali di sicurezza in ogni momento durante la manipolazione del bagno di ghiaccio isopentano / secco e il tessuto.
- Euthanize 2-day-old tutta cucciolo di topo neonatale per overdose Avertin (7,5 mg / g di peso corporeo), e poi congelare il tessuto in isopentano / bagno di ghiaccio secco per preservare la struttura del tessuto. Utilizzare un pre-pulitoEd paio di pinze per posizionare e rimuovere il cucciolo mouse cryomold dal isopentano / bagno di ghiaccio secco. Conservare il tessuto flash congelato a -80 ° C fino al momento dell'analisi.
- Utilizzando guanti di protezione, applicare uno strato di temperatura di taglio ottimale (OCT) mezzo di montaggio su un disco criostato esemplare 40mm. Posizionare delicatamente l'intero topo congelato sul supporto del campione ottobre rivestito di aderire mouse per il disco con l'orientamento desiderato per il sezionamento.
Nota: OCT viene utilizzata esclusivamente per aderire il tessuto al disco di preparato per il sezionamento. Non tessuto completamente incorporare in OCT ed evitare l'eccesso come è noto di Office per avere effetti dannosi sulla analisi MSI. Altri media, come la gelatina, possono essere utilizzati per crio-incorporare il tessuto prima di affettarlo 9. - Inserire il disco (con Office e la sezione di tessuto) sul supporto del campione Peltier nel criostato e premere il pulsante di accensione Peltier. Attendere 10 minuti per il campione di venire a equilibrio termico.
- Posizionare il disco campioni (con il tessuto montatoit) all'interno del supporto del disco, e affrontare il tessuto fino al piano desiderato per l'analisi. Tagliate il tessuto in sezioni di spessore desiderato a -20 ° C utilizzando il microtomo rotativo alloggiata all'interno del criostato 10.
Nota: Per corpo intero analizza, tagliare il tessuto in sezioni di 25 micron di spessore per mantenere l'integrità dei tessuti. Per le regioni più piccole (ad esempio, fegato, cervello, rene), tagliare il tessuto in sezioni di 10 micron di spessore.- Utilizzare la lastra di vetro anti-rollio per evitare che la sezione di tessuto fette di rotolare. Utilizzare il tubo di aspirazione criostato e spazzole per rimuovere i detriti dei tessuti indesiderati. Una volta che il tessuto è sezionato, utilizzare la protezione della lama per coprire la lama affilata microtomo per evitare lesioni personali.
- Sezione Mouse Orient sulla piastra campione e scongelare montaggio su di un vetrino di vetro pre-pulizia a portare la slitta più vicino possibile alla sezione di tessuto, senza toccarlo 10.
Nota: Per quantitativa esperimenti MSImenti, rivestire il vetrino con uno standard interno utilizzando uno spruzzatore pneumatico automatizzato prima di montare il tessuto, e scongelare-montare la sezione di tessuto sul vetrino rivestito 11. - Rimuovere la lama microtomo utilizzato per sezionare il tessuto usando l'espulsore, e sicuro eliminare la lama nel contenitore "rifiuti acuminati" appropriata.
- Tenere il vetrino all'interno del criostato fino Passo 3.2 per mantenere la crioconservazione del tessuto.
2. IR-Maldesi Preparazione / Calibration
- Accendere il laser mid-IR e avviare l'applicazione di controllo laser sul computer. Scegliere la lunghezza d'onda desiderata con l'applicazione di controllo laser fornite dal costruttore. esperimenti IR-Maldesi sono in genere eseguite a 2.940 nm.
Nota: Recenti esperimenti hanno indagato la dipendenza delle lunghezze d'onda laser mid-IR e la matrice di ghiaccio con differenze riscontrate, che sembrano essere anticorpi specifici 8. - Accendere il cervoe di controllo, avviare il programma di controllo utente personalizzato (RASTIR), e calibrare la posizione fase utilizzando il tasto home.
- Preparare la soluzione electrospray. Tipicamente, utilizzare un 50:50 (v / v) di metanolo / acqua con acido formico 0,2% per il solvente elettrospray in modalità positiva ioni. Selezionare la composizione del solvente electrospray secondo l'esperimento. Per esempio, usare idrossido di ammonio 5 mm come modificatore elettrospray per applicazioni di imaging in modalità di ioni negativi.
Nota: per la polarità di commutazione IR-Maldesi MSI, utilizzare 1 mM di acido acetico come il modificatore. Questo modificatore è stato trovato ottimale per ottenere segnale stabile e riproducibile in entrambe le modalità comandata e negativo di litio 12. - Riempire una siringa da 1 ml con il solvente elettrospray, e lavare il capillare di silice con nuovo solvente.
- Allineare ESI emettitore in asse con l'ingresso MS utilizzando i parametri di sorgente come la distanza ESI-spot, spot-ingresso distanza, altezza del campione, e la velocità di flusso del solvente che sono stati ottimizzati utilizzando DO statistico E per l'analisi di sezioni di tessuto 6. Nota: Vedere la Figura 1 per una raffigurante schematico questi parametri e valori ottimizzati per MSI di sezioni di tessuto.
- Avviare il electrospray e valutarne la stabilità (> 10 min) controllando la corrente ionica totale (TIC) che, a questo punto, è costituito esclusivamente da composti ambientali. Tipicamente, una variazione TIC <10% oltre 10-15 min è una buona indicazione di stabilità.
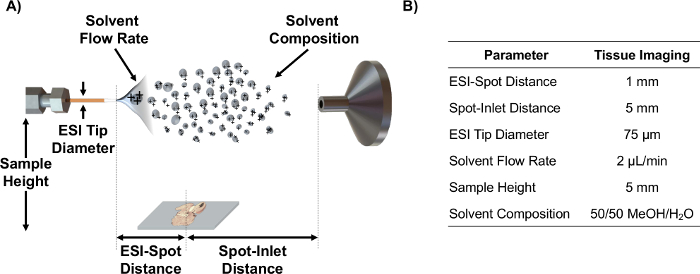
Figura 1. IR-Maldesi schematico e parametri. (A) Schema di configurazione sorgente IR-Maldesi (non in scala) ei parametri regolabili. (B) i valori dei parametri ottimizzati per l'imaging di sezioni di tessuto. Cliccate qui per vedere una versione più grande di questa figura.
- Attenzione: Spegnere il electrospray prima di posizionare campione sul palco.
- Posizionare il tessuto disgelo montato sulla piastra campione di IR-Maldesi all'interno della telecamera.
- Assicurarsi che le mani siano libere dal emettitore ESI e riavviare il electrospray.
- Chiudere lo sportello del contenitore sorgente e spurgo IR-Maldesi con azoto secco per evitare la formazione di condensa sulla superficie del tessuto. Una volta che l'umidità relativa all'interno raggiunge recinzione <3%, attivare l'alimentazione DC (~ 12 V) alla piastra Peltier, che raffreddare il piatto del campione. Nota: Il processo di raffreddamento nella fase di Peltier-raffreddamento è discusso in dettaglio altrove 13.
- Raffreddare la fase a -9 ° C, e consentire il tessuto montato di venire a equilibrio termico (~ 5-10 min).
- Arrestare flusso di azoto ed esporre il tessuto per l'umidità relativa dell'ambiente, aprendo la porta di accesso sorgente. Un sottile strato di ghiaccio depositato sul cosuperficiale ld dello stadio Peltier e montato sezione tissutale causa desublimazione di acqua dall'aria.
Nota: Se l'umidità relativa in laboratorio è inferiore del 10-15%, posizionare un bicchiere di acqua calda all'interno del contenitore per facilitare la formazione dello strato di matrice di ghiaccio. - Dopo che lo strato di matrice di ghiaccio si forma, chiudere lo sportello di accesso e riavviare il flusso di azoto secco per ridurre l'umidità relativa a 10 ± 2%. Regolare la portata di azoto per mantenere l'umidità relativa a questo livello, empiricamente trovato essere l'equilibrio di formazione di ghiaccio e sublimazione per mantenere un livello costante di ghiaccio durante l'esperimento 6.
4. Spettrometria di Massa Imaging Data Acquisition
- Utilizzando il RASTIR GUI (figura 2), spostare il palco per la posizione di analisi facendo clic sul pulsante "posizione del laser". Utilizzare il diodo regolazione del laser per garantire una regione off-tessuto sarà ablazione. Sparate un colpo laser in un ri off-tessutoGion per calibrare l'offset rispetto alla telecamera laser. Fare clic sul pulsante "Laser prova del fuoco" per aggiornare la posizione del laser.
- Ritorno a "posizione CAMERA" e aggiornare la posizione del laser in RASTIR ponendo l'allineamento reticolo laser sul posto ablazione laser (Figura 2-1).
- Istruzioni regione di tasto interesse (ROI) e regolare la dimensione e la posizione della ROI per la sezione di tessuto analizzato (Figura 2-2). Ingresso appropriati parametri sperimentali, come il passo in X e Y (in mm) (Figura 2-3) e il nome del file MSI nella casella "Nome del progetto" (Figura 2-4).
- Quando si disegna una ROI, comprende una porzione di off-tessuto che serve come controllo. Utilizzare questa sezione off-tessuto per il picco di prelievo (punto 5.4).
Nota: Il diametro desorbimento (spot size) del laser sul tessuto è di circa 150 micron; tuttavia, risoluzioni spaziali elevate possono essere ottenute using il metodo di sovracampionamento 14,15.
- Quando si disegna una ROI, comprende una porzione di off-tessuto che serve come controllo. Utilizzare questa sezione off-tessuto per il picco di prelievo (punto 5.4).
- Selezionare la frequenza del laser appropriata dalla casella a discesa, il numero di impulsi laser per voxel (valori interi), e il ritardo tra il grilletto laser e acquisizione dei segnali spettrometro di massa (Figura 2-5). Tipicamente utilizzare due impulsi laser per voxel a 20 Hz a desorbire completamente materiale in un esperimenti IR-Maldesi MSI, con un tempo di ritardo 10 msec per permettere ioni generati per raggiungere l'analizzatore di massa per la misura 5.
Nota: Scegliere il più alto tasso di ripetizione che il laser è in grado di funzionare a. E 'stato recentemente dimostrato che l'uso di frequenza di ripetizione superiore migliorerà analiti rilevabilità 16.
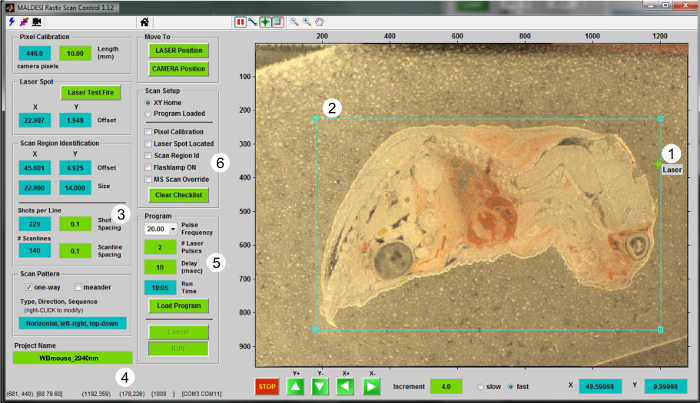
Figura 2. Interfaccia utente per il funzionamento IR-Maldesi MSI. Schermata del programma RASTIR Scan Control è presentato. La procedura per performing un esperimento di MSI sono (1) la localizzazione del punto laser, (2) il disegno di un ROI, (3) la scelta della dimensione del passo fase (in mm), (4) dare un nome al file, (5) la scelta del numero corretto di impulsi per voxel insieme con la velocità desiderata di ripetizione, e (6) il controllo della lista per l'imaging e la configurazione di MS. cliccate qui per vedere una versione più grande di questa figura.
- Scegliere parametri dello spettrometro di massa come modalità di ionizzazione, tensione electrospray, portata solvente, la temperatura capillare, e il tempo di iniezione nel software spettrometro di massa. Fare riferimento alla Tabella 1 per un esempio dei parametri utilizzati in tutto il corpo IR-Maldesi MSI.
Nota: A causa della complessità di campioni biologici e la mancanza di metodi di separazione in analisi MSI, la sorgente IR-Maldesi è accoppiato ad un alto potere di risoluzione ed elevata strumento accuratezza di massa. Un metodo di acquisizione può essere caricato per analysES come il monitoraggio reazione parallela (PRM) 7 o polarità di commutazione MSI 12. - Una volta che sono stati scelti tutti i parametri (laser, palco, e spettrometro di massa), posizionare il master in modalità "stretta di mano" per la sincronizzazione, e iniziare l'acquisizione MS.
- Utilizzando il software di imaging RASTIR, verificare tutti i passaggi in lista di controllo (Figura 2-6) sono completate controllando scatole, e caricare il programma. Una volta che il programma è stato caricato, il pulsante "Esegui" sarà disponibile. Premere Esegui per avviare l'acquisizione del segnale MSI.
- Al termine dell'esperimento di imaging, interrompere l'acquisizione spettrometro di massa e collocare lo strumento in modalità di attesa. Spegnere spurgo di azoto al recinto, spegnere alimentazione alla piastra di Peltier, e spegnere il controller palco e laser.
- Aprire lo sportello di accesso, rimuovere la punta emettitore electrospray dall'interno del contenitore di origine, e rimuovere il metallo riscaldato MS ingresso estesa utilizzando guanti di protezione. Attenzione: L'estesa metallo MS ingresso sarà molto calda.
- Indossare guanti e occhiali protettivi, pulire l'ingresso esteso metallo capillare bagno ad ultrasuoni in acido nitrico 15% per 10 minuti, poi in acqua HPLC per 10 min, ed infine in HPLC metanolo per 10 min. Essiccare l'ingresso metallico capillare sotto flusso di azoto, e reinserire in MS. Nota: Smaltire solvente nei contenitori per i rifiuti idonei dopo.
| Parametro | Valore |
| Modalità di ionizzazione | Positivo |
| elettrospray tensione | |
| Solvente Portata | 2 microlitri / min |
| capillare Temperatura | 275 ° C |
| Intervallo di scansione | m / z 250-1,000 |
| Tipo di scansione | Scansione completa |
| Tempo di iniezione | 110 msec |
| Potere risolutivo |
Tabella 1. Parametri strumento utilizzato in tutto il corpo IR-Maldesi MSI.
Analisi 5. I dati
- Convertire i file di dati grezzi generati dallo strumento di formati di dati, come mzXML 17 o 18 imzML utilizzando software libero, come MSConvert 17 e convertitore imzML 19.
- Inizia MSiReader v1.0, un software open-source sviluppato per l'analisi di alto potere risolvente dati MSI 20, su un computer di elaborazione dedicato. Caricare il file di immagini su MSiReader. Per l'interfaccia utente di MSiReader e alcune delle sue funzionalità integrate, vedi figura 3.
- Generare mappe ioni di analiti di interesse inserendo la loro m / z e scegliere un adeguato finestra m / z in parti per milione (ppm) o Thomson (Th) (Figura 3-1). Per gli strumenti le alte prestazioni (RP)scegliere una finestra nella gamma bassa ppm.
Nota: La finestra m / z appropriata dipende dalla RP e la massa precisione di misura (MMA) dello strumento sorgente IR-Maldesi è accoppiato. - Ulteriori interpretare i dati utilizzando le funzionalità integrate come l'interpolazione (Figura 3-2), la normalizzazione (Figura 3-3), sovrapposizione di immagini ottico (Figura 3-4), e il picco raccolta (Figura 3-5) 20.
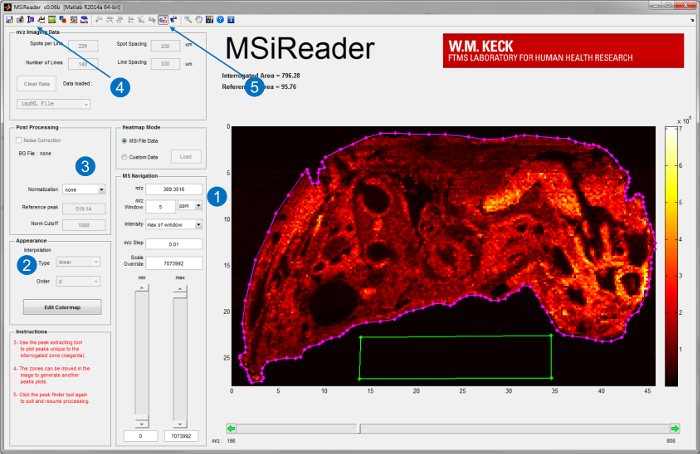
Figura 3. Interfaccia utente di MSiReader; v1.0 20. Una volta che un file viene caricato nel software, mappe di ioni di analiti di interesse vengono visualizzati (1) inserendo la m / z e tolleranza in ppm o Th. Ulteriori analisi come (2) interpolazione o (3) la normalizzazione può essere eseguita. Un'immagine ottica del tessuto può anche essere importato e sovrappostole mappe di ioni (4) per una migliore visualizzazione. Per non mirati analizza il picco di funzione (5) la raccolta può essere utilizzato per estrarre i picchi tessuto-specifiche, scegliendo l'area di tessuto (linea magenta) e una zona di riferimento off-tessuto (riquadro verde). Clicca qui per vedere una versione più grande questa figura.
- Utilizzare la funzione di picco raccolta per generare un elenco di valori m / z tessuto-specifici in base a criteri definiti dall'utente. Nota: L'algoritmo PeakFinder utilizza le differenze di spettro medio di massa di due regioni. Questi valori m / z possono poi essere ricercati nei database di metaboliti quali METLIN 21 o LIPID MAPPE. 22
- Fare riferimento alla Figura 3 per un esempio di scelta di una regione interrogato tessuto (magenta poligono ROI) e della regione di riferimento off-tessuto (verde poligono ROI).
Risultati
Le immagini presentate in Figura 4 mostrano la distribuzione spaziale dei metaboliti in diversi organi nella sezione di tessuto corpo intero. I valori unici m / z a regioni specifiche del corpo sono stati trovati usando MSiReader PeakFinder, seguita dalla elaborazione in batch per la generazione di immagini. Lo strumento di sovrapposizione di immagini (Figura 3-4) è stato utilizzato per allineare l'immagine ottica presa prima deposizione di...
Discussione
Il protocollo di cui sopra sono descritti i passaggi chiave per l'esecuzione di un esperimento IR-Maldesi MSI. Il processo di applicazione della matrice (punto 3) dura circa 20 minuti, che è simile ad un tipico processo di applicazione matrice per esperimenti MALDI MSI per sublimazione o spray-coating utilizzando uno spruzzatore robotico. Inoltre, IR-Maldesi non si basa sulla separazione di analiti in cristalli di matrice 6, e la matrice di ghiaccio può essere usato universalmente per tutti gli analiti ...
Divulgazioni
The authors declare no competing financial interests.
Riconoscimenti
The authors thank Professor H. Troy Ghashghaei from NCSU Department of Molecular Biomedical Sciences for providing the whole mouse tissue. The authors also gratefully acknowledge the financial assistance received from National Institutes of Health (R01GM087964), the W.M. Keck foundation, and North Carolina State University.
Materiali
| Name | Company | Catalog Number | Comments |
| IR-MALDESI Source | Custom-made | N/A | Please refer to references 4 and 12 for an in-depth discussion of IR-MALDESI source development. |
| Q Exactive Plus | Thermo Scientific | Q Exactive Plus Hybrid Quadrupole-Orbitrap Mass Spectrometer | |
| Water, HPLC Grade | Burdick & Jackson | AH365-4 | |
| Methanol, HPLC Grade | Burdick & Jackson | AH230-4 | |
| Formic Acid | Sigma Aldrich | 56302 | |
| Tunable mid-IR Laser | Opotek Inc. | IR Opolette | Tunable 2,700-3,100 nm IR OPO laser |
| Nitrogen Gas | Arc3 Gases | AG S-NI300-5.0 | Grade 5.0 high purity nitrogen gas cylinder (300) |
| Cryostat | Leica Biosystems | CM 1950 | Cryomicrotome |
| High Profile Microtome Blades | Leica Biosystems | 3802123 | Leica DB80HS |
| Mounting Medium (OCT) | Leica Biosystems | 3801480 | Surgipath FSC 22 mounting medium |
| Cryostat Specimen Disc | Leica Biosystems | 14047740045 | 40 mm diameter |
| Glass Microscope Slides | VWR | 48312-003 | Frosted, selected, pre-cleaned |
Riferimenti
- Mcdonnell, L. A., Heeren, R. M. A. Imaging Mass Spectrometry. Mass Spectrom. Rev. 26, 606-643 (2007).
- Chughtai, K., Heeren, R. M. A. Mass spectrometric imaging for biomedical tissue analysis. Chem. Rev. 110 (5), 3237-3277 (2010).
- Robichaud, G., Barry, J. A., Muddiman, D. C. Atmospheric Pressure Mass Spectrometry Imaging. Encycl. Anal. Chem. , (2014).
- Sampson, J. S., Hawkridge, A. M., Muddiman, D. C. Generation and detection of multiply-charged peptides and proteins by matrix-assisted laser desorption electrospray ionization (MALDESI) Fourier transform ion cyclotron resonance mass spectrometry. J. Am. Soc. Mass Spectrom. 17 (12), 1712-1716 (2006).
- Robichaud, G., Barry, J. A., Garrard, K. P., Muddiman, D. C. Infrared matrix-assisted laser desorption electrospray ionization (IR-MALDESI) imaging source coupled to a FT-ICR mass spectrometer. J. Am. Soc. Mass Spectrom. 24 (1), 92-100 (2013).
- Robichaud, G., Barry, J. A., Muddiman, D. C. IR-MALDESI Mass Spectrometry Imaging of Biological Tissue Sections Using Ice as a Matrix. J. Am. Soc. Mass Spectrom. 25 (3), 319-328 (2014).
- Barry, J. A., et al. Mapping Antiretroviral Drugs in Tissue by IR-MALDESI MSI Coupled to the Q Exactive and Comparison with LC-MS/MS SRM Assay. J. Am. Soc. Mass Spectrom. 25 (12), 2038-2047 (2014).
- Rosen, E. P., Bokhart, M. T., Ghashghaei, H. T., Muddiman, D. C. Influence of Desorption Conditions on Analyte Sensitivity and Internal Energy in Discrete Tissue or Whole Body Imaging by IR-MALDESI. J. Am. Soc. Mass Spectrom. 26, 899-910 (2015).
- Nelson, K. A., Daniels, G. J., Fournie, J. W., Hemmer, M. J. Optimization of whole-body zebrafish sectioning methods for mass spectrometry imaging. J. Biomol. Tech. 24 (3), 119-127 (2013).
- Park, J. J., Cunningham, M. G. Thin sectioning of slice preparations for immunohistochemistry. J. Vis. Exp. (3), e194 (2007).
- Bokhart, M. T., Rosen, E., Thompson, C., Sykes, C., Kashuba, A. D. M., Muddiman, D. C. Quantitative mass spectrometry imaging of emtricitabine in cervical tissue model using infrared matrix-assisted laser desorption electrospray ionization. Anal. Bioanal. Chem. 407 (8), 2073-2084 (2015).
- Nazari, M., Muddiman, D. C. Polarity Switching Mass Spectrometry Imaging of Healthy and Cancerous Hen Ovarian Tissue Sections by Infrared Matrix-Assisted Laser Desorption Electrospray Ionization (IR-MALDESI). Analyst. 141, 595-605 (2016).
- Hsu, C. C., et al. Design and Application of a Low-Temperature Peltier-Cooling Microscope. J. Pharm. Sci. 85 (1), 70-74 (1996).
- Jurchen, J. C., Rubakhin, S. S., Sweedler, J. V. MALDI-MS imaging of features smaller than the size of the laser beam. J. Am. Soc.Mass Spectrom. 16 (10), 1654-1659 (2005).
- Nazari, M., Muddiman, D. C. Cellular-level mass spectrometry imaging using infrared matrix-assisted laser desorption electrospray ionization (IR-MALDESI) by oversampling. Anal. Bioanal. Chem. 407 (8), 2265-2271 (2015).
- Rosen, E. P., Bokhart, M. T., Nazari, M., Muddiman, D. C. Influence of C-Trap Ion Accumulation Time on the Detectability of Analytes in IR-MALDESI MSI. Anal. Chem. 87, 10483-10490 (2015).
- Kessner, D., Chambers, M., Burke, R., Agus, D., Mallick, P. ProteoWizard: open source software for rapid proteomics tools development. Bioinformatics. 24 (21), 2534-2536 (2008).
- Schramm, T., et al. ImzML - A common data format for the flexible exchange and processing of mass spectrometry imaging data. J. Proteomics. 75 (16), 5106-5110 (2012).
- Race, A. M., Styles, I. B., Bunch, J. Inclusive sharing of mass spectrometry imaging data requires a converter for all. J. Proteomics. 75 (16), 5111-5112 (2012).
- Robichaud, G., Garrard, K. P., Barry, J. A., Muddiman, D. C. MSiReader: an open-source interface to view and analyze high resolving power MS imaging files on Matlab platform. J. Am. Soc. Mass Spectrom. 24 (5), 718-721 (2013).
- Smith, C. A., O'Maille, G., et al. METLIN: a metabolite mass spectral database. Ther. Drug. Monit. 27 (6), 747-751 (2005).
- Sud, M., et al. LMSD: LIPID MAPS structure database. Nucleic Acids Res. 35, D527-D532 (2007).
- Schwartz, S. A., Reyzer, M. L., Caprioli, R. M. Direct tissue analysis using matrix-assisted laser desorption/ionization mass spectrometry: practical aspects of sample preparation. J. Mass Spectrom. 38 (7), 699-708 (2003).
- Takai, N., Tanaka, Y., Inazawa, K., Saji, H. Quantitative analysis of pharmaceutical drug distribution in multiple organs by imaging mass spectrometry. Rapid Commun. Mass Spectrom. 26 (13), 1549-1556 (2012).
- Liu, J., Gingras, J., Ganley, K. P., Vismeh, R., Teffera, Y., Zhao, Z. Whole-body tissue distribution study of drugs in neonate mice using desorption electrospray ionization mass spectrometry imaging. Rapid Commun. Mass Spectrom. 28 (2), 185-190 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon