È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
La definizione del substrato Specificità per lipasi e fosfolipasi candidati
In questo articolo
Riepilogo
Many predicted (phospho)lipases are poorly characterized with regard to their substrate specificities and physiological functions. Here we provide a protocol to optimize enzyme activities, search for natural substrates, and propose physiological functions for these enzymes.
Abstract
I microrganismi producono un ampio spettro di lipasi (fosfo) che vengono secreti per rendere substrati esterni disponibili per l'organismo. In alternativa, altri (fosfo) lipasi possono essere fisicamente associate con l'organismo producendo causando un turnover di lipidi intrinseche e spesso dando origine ad un rimodellamento delle membrane cellulari. Sebbene potenziali (fosfo) lipasi possono essere previsti con un certo numero di algoritmi quando la sequenza del gene / proteina è disponibile, prova sperimentale delle attività enzimatiche, specificità di substrato e potenziali funzioni fisiologiche è spesso non è stata ottenuta. Questo manoscritto descrive l'ottimizzazione delle condizioni di analisi per i potenziali (fosfo) lipasi con specificità di substrato sconosciute e come impiegare queste condizioni ottimali nella ricerca per il substrato naturale di un rispettivo (fosfo) lipasi. Usando substrati cromogeni artificiali, come i derivati p -nitrophenyl, può aiutare a rilevare un minoreattività enzimatica per un (fosfo) lipasi previsto in condizioni standard. Avendo riscontrato un attività enzimatica tale minore, i parametri distinti di un saggio enzimatico possono essere variate per ottenere un'idrolisi più efficiente del substrato artificiale. Dopo aver determinato le condizioni in cui un enzima funziona bene, una varietà di potenziali substrati naturali devono essere analizzati per la loro degradazione, un processo che può essere seguita con metodi cromatografici distinti. La definizione di specificità di substrato per nuovi enzimi, fornisce spesso ipotesi per un potenziale ruolo fisiologico di questi enzimi, che poi possono essere testati sperimentalmente. Seguendo queste linee guida, siamo stati in grado di identificare una fosfolipasi C (SMc00171) che degrada fosfatidilcolina di phosphocholine e diacylglycerol, in una fase cruciale per il rimodellamento delle membrane nel batterio Sinorhizobium meliloti dalle condizioni di fosforo-limitante della crescita. Per due predetto patatin-come fosfolipasi (SMc00930 e SMc01003) dello stesso organismo, potremmo ridefinire le loro specificità di substrato e chiarire che SMc01003 è una lipasi diacilglicerolo.
Introduzione
Lipidi glicerolo-based come trigliceridi e fosfolipidi (glycero) costituiscono importanti e probabilmente il più noti classi di lipidi 1. Trigliceridi (tag) sono grassi o oli, che di solito fungono da lipidi di stoccaggio, e quindi come potenziali fonti di energia e di carbonio. TAG possono essere degradati dalla lipasi, che sono spesso secrete dall'organismo produzione di digerire variabili esterne e renderli disponibili come fonti di carbonio. Inoltre, lipasi sono stati ampiamente studiati negli anni, grazie alle loro importanti applicazioni biotecnologiche 2.
A causa della loro natura anfifilica e la loro forma quasi cilindrica, proprietà di membrana formante (glycero) fosfolipidi esporre e generalmente costituiscono i principali componenti lipidici di membrana doppo strato 3. In microrganismi semplici, come il batterio Escherichia coli, solo tre varianti principali del gruppo testa, fosfatidilglicerolo (PG), cardiolipina (CL), e phosphatidylethanolamine (PE) è riscontrabile, anche se si deve essere consapevoli che ciascuno di essi può essere sostituito con un considerevole numero di differenti catene grassi acil al sn -1 o sn posizione -2 dando luogo ad un grande numero di differenti specie molecolari 4 . Altri batteri possono avere altri fosfolipidi in aggiunta o al posto. Ad esempio, Sinorhizobium meliloti, un batterio del suolo, che è in grado di formare una radice nodulo simbiosi azotofissatrice con l'erba medica legume (Medicago sativa), contiene in aggiunta al PE un secondo fosfolipide zwitterionico, fosfatidilcolina (PC) 5. Inoltre, i lipidi non contenenti fosforo o glicerolo potrebbe essere parte anfifilico e la forma della membrana cellulare. Ad esempio, dalle condizioni di crescita fosforo limitativo, in S. meliloti, (glycero) fosfolipidi sono in gran parte sostituiti da lipidi di membrana che non contengono fosforo, cioè, sulfolipids, lipidi ornitina, e diacylglyceryl trimethylhomoserine (DGTS) 6. Nei batteri, DGTS è formata da diacilglicerolo (DAG) in un percorso in due fasi 7, ma la fonte per la generazione di DAG non era chiaro. Esperimenti di pulse-chase suggerito che il PC potrebbe essere un precursore per DGTS 8 e utilizzando la metodologia descritta in questo manoscritto abbiamo potuto identificare un fosfolipasi C (PLCP, SMc00171) che si forma in condizioni di fosforo-limitante e che può convertire il PC in DAG e phosphocholine 8.
In uno studio separato, abbiamo scoperto che un sintetasi acil-CoA (FADD) mutante -carente di S. meliloti o di Escherichia coli accumulato acidi grassi liberi entrando fase stazionaria della crescita 9. Sebbene questi acidi grassi sembravano essere derivato da lipidi di membrana, la fonte precisa per gli acidi grassi liberi o l'enzima (s) liberandoli non erano noti. Anche in questo caso, utilizzando la strategia delineata in questo manoscritto, due 10 (fosfo) lipasi Patatin-like (SMc00930 e SMc01003), che ha contribuito alla formazione di acidi grassi liberi a S. meliloti 11 sono stati previsti. Sorprendentemente, SMc01003 utilizzato DAG come substrato convertirlo monoacilglicerolo e infine glicerolo e acidi grassi liberi 11. Pertanto, SMc01003 è una lipasi DAG (DGLA).
Sebbene un certo numero di algoritmi esistenti per prevedere il potenziale (fosfo) lipasi 12,13, la loro funzione precisa e ruolo fisiologico non è generalmente noto. Qui descriviamo un protocollo, per clonare e iperespressione di lipasi (fosfo) previsti o potenziali. Questo manoscritto spiega come saggi enzimatici possono essere sviluppati e ottimizzati per la (fosfo) lipasi sovraespresso utilizzando substrati cromogeni artificiali. Forniamo esempi di come con un metodo enzimatico ottimizzato il vero (fosfo) substrato lipasi può essere incontrato e di come queste scoperte potrebbero arricchire la nostra comprensione della fisiologia microbica.
Protocollo
1. Clone e iperespressione del gene strutturale per lipasi prevista
- Utilizzando la reazione a catena della polimerasi (PCR) 14 e oligonucleotidi specifici (Tabella 1) 15, amplificare il gene di interesse (smc01003, smc00930 o smc00171), previsto per codificare per una lipasi o fosfolipasi, dal DNA genomico dell'organismo ospite (ie , S. meliloti).
- Introdurre siti specifici di restrizione (con la sequenza creazione degli oligonucleotidi). Digerire il frammento di DNA amplificato con gli enzimi di restrizione corrispondenti e clonare in un vettore di espressione, come plasmidi della serie pET 16.
- Dopo aver verificato la sequenza di DNA corretta per il gene clonato, trasformare il vettore ad un ceppo espressioni quali Escherichia coli BL21 (DE3) pLysS 16.
- Preparare una notte pre-cultura dell'espressione ospite E. coli BL21 (DE3) pLYSS, l'ospitare il rispettivo vettore animale domestico con il gene clonato o il vettore vuoto, in 100 ml fiasche di coltura contenente 20 ml di Luria Bertani brodo (LB) 17, più gli antibiotici necessari. Cultura le cellule a 30 ° C (o alla solita temperatura di crescita del batterio da cui la lipasi origine).
- Usando i overnight pre-culture, inoculare 500 ml di terreno LB preriscaldata (più antibiotici richiesti) in 2 fiasche di coltura L per ottenere una densità ottica iniziale a 620 nm (OD 620) = 0,05. Seguire crescita di colture e ad una OD 620 = 0.3, aggiungere isopropil-β-D-tiogalattoside (IPTG) ad una concentrazione finale di 100 mM, e incubare sotto agitazione a 30 ° C per un periodo di 4 ore.
- Al termine del periodo di incubazione, trasferire ogni cultura in una provetta da centrifuga da 500 ml e centrifugare a 5000 xga 4 ° C per 30 min. Risospendere pellet cellulari batteriche in 5 ml di tampone di sospensione (ad esempio, SMc00930- e SMc01003-cellule che esprimono in 50 mM Tris-HCl pH 8.0 e le cellule che esprimono SMc00171-in 50 mm dietanolammina-HCl pH 9.8). Conservare le sospensioni cellulari a -80 ° C fino al momento dell'uso.
2. Preparare estratti proteici privi di cellule e determinare proteine Concentrazione
- Scongelare sospensioni cellulari batteriche e memorizzare sul ghiaccio. Passare sospensioni cellulari tre volte attraverso una cella di pressione a freddo a 20.000 libbre per a 2. Rimuovere le cellule intatte e detriti cellulari mediante centrifugazione a 5000 xg per 30 min a 4 ° C.
- Dopo centrifugazione, preparare aliquote di 100 e 500 microlitri dal supernatante per la successiva analisi e conservarli a -80 ° C fino al momento dell'uso.
- Utilizzare una delle un'aliquota di 100 microlitri per determinare la concentrazione di proteine di estratti privi di cellule distinte da un metodo di scelta o come descritto 18.
3. Utilizzare substrati artificiali per l'ottimizzazione EnzymeAttività di (fosfo) lipasi
- Per una copertura iniziale delle attività enzimatiche distinte, utilizzare substrati artificiali che producono un prodotto colorato per idrolisi, come p -nitrophenol (p -NP).
- Per i saggi enzimatici ottimizzati già con substrati p -nitrophenyl esteri artificiali (delineate per fosfolipasi C PLCP (SMc00171), così come per le fosfolipasi Patatin come previsti SMc00930 e SMc01003), schemi di pipettaggio indicata nella tabella 2.
- Quando esplorando un nuovo potenziale (fosfo) lipasi, preparare un primo dosaggio enzimatico standard contenente 50 mM Tris-HCl, pH 8,5, 100 mM NaCl, 0,05% Triton X-100, 0,5 mM p -nitrophenyl composto contenente (p -nitrophenyl fosfato , bis- p -nitrophenyl fosfato, p -nitrophenyl decanoato, o p -nitrophenyl palmitato), ed estratto proteico cell-free (controllo 1, 3, 10, 30, 100, 300, e 1000 mg) in un volume totale di 1 ml in 1 ml cuve plasticaTTES.
NOTA: Utilizzare pH alcalino (figura 1) quando segue p -nitrophenyl idrolisi in un saggio continuo. In alternativa, utilizzare dosaggi singoli time-point per un intervallo di valori di pH, aggiungendo NaOH al termine del periodo di incubazione per bloccare la reazione enzimatica e per garantire che tutti p -NP è presente nella forma fenolato. - Seguire l'andamento temporale di un aumento di assorbanza a 405 nm, a causa della formazione di p -NP, in uno spettrofotometro a 30 ° C per un periodo di 5 min. Quantificare la formazione inizialmente lineare di p -NP determinando la pendenza iniziale di aumento di assorbanza per volta.
- Calcolare la variazione di concentrazione (Δc) per p -NP utilizzando la legge di Lambert-Beer (ΔA = ε Δc d) 1.
NOTA: ΔA è la variazione lineare di assorbanza determinato, ε è il coefficiente di estinzione molare alla rispettiva lunghezza d'onda (in unità di M -1 cm -1), d è la lunghezza del percorso ottico (1 cm), e Δc è il cambiamento di concentrazione (in unità di M) da determinare.- Considerando che il volume di dosaggio è di 1 ml, calcolare la quantità di p -NP formata.
NOTA: Importo = concentrazione volumetrica x. - Calcolare l'attività enzimatica dividendo la quantità di p -NP formata dal momento in cui è formato. Determinare l'attività enzimatica specifica dividendo attività enzimatica dalla quantità di proteine (in mg) che era responsabile della generazione questa attività.
- Considerando che il volume di dosaggio è di 1 ml, calcolare la quantità di p -NP formata.
- Confronta i cambiamenti di assorbanza provocate da estratti proteici in cui un gene candidato (smc00171, smc00930, o smc01003) era stato espresso con estratti che ospitano solo un vettore vuoto.
NOTA: Per continuare con le seguenti operazioni, le attività specifiche, causate da estratti proteici in cui un gene candidato era stato espresso, dovrebbe essere almeno il doppio o more rispetto ai valori ottenuti per le attività specifiche causate da estratti proteici che ospitano solo un vettore vuoto. - Per ulteriori esperimenti, seleziona quelle condizioni in cui l'idrolisi del composto p -nitrophenyl contenente è minimo con estratti privi di cellule (cioè, vettore vuoto) e per i quali la formazione più pronunciata p -NP e l'anione p -nitrophenolate (Figura 1) possono essere osservati quando estratti proteici sono impiegati, in cui un gene candidato era stato espresso.
- Dopo la determinazione della attività enzimatica iniziale 3.1, ottimizzare le condizioni di analisi per il rispettivo enzima variando pH, tipo di tampone, tampone forza, concentrazioni di NaCl, detergenti come Triton X-100, e l'assenza o la presenza di diversi cationi bivalenti.
- Per diverse concentrazioni di ciascuna variabile, a determinare l'attività enzimatica specifica (vedi 3.1.4.2) (il numero più alto ottenuto definisce la condizionedell'attività enzimatica massima). Utilizzare la combinazione delle condizioni ottimali riscontrate per ciascuna variabile per definire un saggio enzimatico ottimizzato in cui ciascuna variabile è presente nella sua concentrazione ottimale.
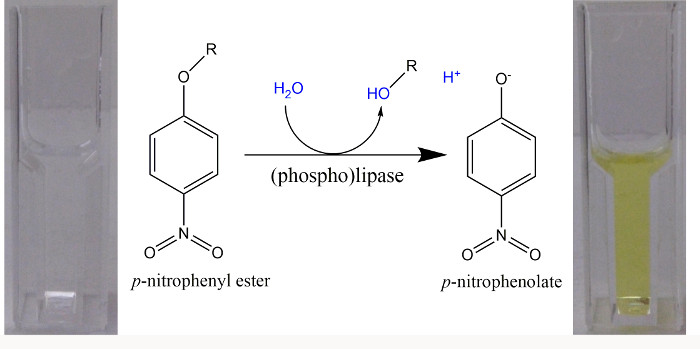
Figura esteri -Nitrophenyl 1. p substrati artificiali per (fosfo) lipasi in un saggio spettrofotometrico. Su idrolisi degli esteri p -nitrophenyl, un acido (R-OH) e p -nitrophenol (p -NP) si formano. Grazie al pK a = 7,2 per la dissociazione del fenolica H + da p -NP, a pH> 9,2 oltre il 99% sono nella luminosa forma p -nitrophenolate giallo e un coefficiente di estinzione molare di 18.000 M -1 cm - 1 può essere utilizzato ad una lunghezza d'onda di 405 nm per la quantificazione del libero p -nitrophenolate 22. Quando sono stati utilizzati i buffer con un pH di 8,5, l'assorbanza è stata determinata a 400 nm e un coefficiente di estinzione molare di 14.500 M -1 cm -1 è stato impiegato 23. Si prega di cliccare qui per vedere una versione più grande di questa figura.
NOTA: Dopo aver definito le condizioni ottimali per l'attività dell'enzima di interesse, intraprendere la ricerca del reale / fisiologica substrato di questo lipasi. In linea di principio, prendere due, spesso complementari, approcci per raggiungere questo obiettivo, un vivo approccio o un approccio in vitro.
4. In Vivo Identificazione della fisiologica Substrato di un Lipasi
ove_content ">. NOTA: In un approccio in vivo, esprimono la lipasi di interesse in un organismo ospite 8,11 al fine di registrare nel corso del tempo se l'espressione della lipasi altera il profilo lipidico host's In un altro approccio in vivo, generare un mutante carente del gene di interesse e di studio 8,11 se il suo profilo lipidico è distinto dal tipo di versione selvaggia 6,8,11. al fine di ottenere una valutazione quantitativa del profilo lipidico di un organismo, un metodo semplice consiste nel radiomarcatura composti cellulari , l'estrazione dei lipidi, separandoli mediante cromatografia, e quantificare i lipidi separati marcati radioattivamente.- Radiomarcatura di lipidi.
- Preparare una notte pre-coltura di un organismo di interesse (E. coli o S. meliloti) in 5 ml di terreno di coltura desiderato (media complessa o terreno minimo definito dall'utente) e crescere a 30 ° C.
- Dalla pre-coltura, inoculare in 20 ml di same mezzo fresco in un pallone di coltura 100 ml per ottenere una OD 620 = 0.3 iniziale per la cultura.
- Prelevare un 'aliquota (1 ml) della coltura in condizioni sterili e trasferire in una provetta a fondo rotondo polistirene 14 ml sterile.
- Aggiungere 1 pCi di acetato [1- 14 C] (60 mCi per mmol) alla coltura 1 ml.
- Incubare la coltura liquida sotto agitazione a 30 ° C per un periodo di 24 ore.
- Al termine del periodo di incubazione, trasferire la coltura in una provetta da 1,5 ml e centrifugare a 12.000 xg a temperatura ambiente per 5 min.
- Risospendere il pellet in 100 ml di acqua. A questo punto, conservare la sospensione cellulare a -20 ° C o immediatamente continuare con l'estrazione di lipidi polari (sezione 4.2).
- Estrazione di lipidi polari.
NOTA: Il metodo descritto qui essenzialmente segue la procedura riportata da Bligh e Dyer 19.- Per i 100 ml di sosp cellulare acquosaensione, aggiungere 375 ml di metanolo: soluzione in cloroformio (2: 1; vol / vol).
- Vortex per 30 sec e incubare per 5 min a temperatura ambiente.
- Centrifugare 5 min a 12.000 xg a temperatura ambiente.
- Trasferire il surnatante in un nuovo 1,5 ml provetta.
- Aggiungere 125 ml di cloroformio e 125 ml di acqua, vortice 30 sec.
- Centrifugare 1 min a 12.000 xg a temperatura ambiente.
- Trasferire la fase cloroformica inferiore per una nuova provetta e asciugare con un flusso di azoto gassoso.
- Sciogliere lipidi essiccati in 100 ml di cloroformio: soluzione di metanolo (1: 1; vol / vol).
NOTA: A questo punto, un'aliquota di 5 microlitri della soluzione lipidica può essere quantificata mediante conteggio a scintillazione liquida. - Per strato sottile analisi cromatografica (TLC), asciugare le restanti 95 microlitri con un flusso di azoto gassoso e ridisciogliere lipidi essiccati in 20 ml di cloroformio: soluzione di metanolo (1: 1; vol / vol). Utilizzare una aliquota di 3 mlper l'analisi TLC.
- Separazione dei lipidi polari mediante cromatografia su strato sottile (TLC).
NOTA: A seconda delle classi di lipidi da analizzare, diverse combinazioni di fasi solide e mobili possono essere impiegati per la separazione. Ecco una separazione tipica di lipidi cariche polari e un altro, più adatti per i lipidi polari neutri, utilizzando ad alte prestazioni cromatografia su strato sottile (HPTLC) fogli di alluminio gel di silice come fase solida, sono delineati.- La separazione dei lipidi polari praticati dalle bidimensionali TLC (2D-TLC).
- Applicare un'aliquota 3 microlitri di campione lipidi in un angolo di un foglio di alluminio HPTLC di silice gel (10 x 10 cm), 2 cm dal bordo della piastra.
- Preparare e miscelare la fase mobile (140 ml di cloroformio, 60 ml di metanolo e 10 ml di acqua) per la separazione nella prima dimensione.
- Coat un TLC in via di sviluppo da camera internamente con carta cromatografia.
NOTA: Questo per garantire che la fase gas della camerasi satura rapidamente (entro 30 minuti) dopo la fase mobile per la prima dimensione è stato aggiunto alla camera e la camera è stata chiusa con una lastra di vetro. - Preparare e miscelare la fase mobile (130 ml di cloroformio, 50 ml di metanolo e 20 ml di acido acetico glaciale) per la separazione nella seconda dimensione e trasferimento in una seconda camera di sviluppo TLC rivestite internamente con cromatografia su carta e lasciare la camera satura.
- Trasferire accuratamente il foglio di alluminio gel di silice HPTLC con il campione lipide essiccato alla prima camera e sviluppare (cioè, esegue cromatografia) la piastra per 60 min in camera chiusa nella prima dimensione 5.
- Rimuovere la placca dalla camera e lasciare asciugare solventi fuori in una cappa a flusso per 30 min.
- Dopo aver piastra di 90 gradi per quanto riguarda la cromatografia precedente, trasferire il foglio di alluminio gel di silice HPTLC, su cui i lipidi sono stati separati in una dimensione, alla secondacamera d e sviluppare la piastra per 60 minuti nella seconda dimensione 5.
- Rimuovere il foglio dalla camera e lasciare che i solventi asciugarsi in una cappa a flusso per almeno 2 ore.
- Separazione dei lipidi polari neutri.
- Applicare 3 aliquote microlitri di campioni lipidi su un foglio di alluminio di gel di silice HPTLC partendo 2 cm dai bordi della lastra. Se più campioni vengono analizzati in cromatografia unidimensionale, mantenere una distanza di almeno 1,5 cm tra i diversi punti di applicazione del campione.
- Preparare e mescolare la fase mobile (140 ml di esano, 60 ml di etere etilico, e 8 ml di acido acetico) e trasferire in una camera di sviluppo TLC rivestiti internamente con carta cromatografia e coperte con una lastra di vetro per lasciare che il satura camera (30 min).
- Trasferire il foglio di alluminio gel di silice HPTLC con i campioni lipidi essiccati nella camera e sviluppare la piastra per 30 min in camera chiusa.
- Rimuovere la placca dalla camerae lasciare che i solventi asciugare fuori in una cappa a flusso per 2 ore.
- La separazione dei lipidi polari praticati dalle bidimensionali TLC (2D-TLC).
- La quantificazione e la visualizzazione dei lipidi polari separati.
- Una volta che la lastra TLC sviluppata è asciutto, incubare con uno schermo luminescenza fotostimolabile (PSL) in una cassetta chiusa per 3 giorni.
- Esporre lo schermo incubate a uno scanner PSL e acquisire un'immagine virtuale dei lipidi radiomarcati separati.
- Eseguire la quantificazione utilizzando il software PSL 20.
- La visualizzazione e l'isolamento di singole classi lipidi polari.
- Incubare foglio TLC sviluppata per 10 min in una camera cromatografia in presenza di 1 g di cristalli di iodio.
NOTA: separata composti lipidici si dissolveranno lo iodio e appaiono come macchie brunastre. - Circle le macchie con una matita, confrontarle con la mobilità relativa (R f) di composti standard (ad esempio, 1,2-dipalmitoyl- sn -glycerol, dipalmitoil-L-α-phosphatidylcholine, DL-α-monopalmitin, o acido palmitico), e di identificare a quale classe di lipidi potrebbero appartenere.
- In una cappa aspirante, lasciare che lo iodio evaporare dal foglio TLC.
- Con l'aiuto di una spatola, raschiare il gel di silice contenente il composto di interesse dal foglio, ed estrarre il composto dal gel di silice con una miscela di 100 ml di acqua e 375 ml di metanolo: soluzione in cloroformio (2: 1; vol / vol).
- Continuare con l'estrazione in base alla Bligh e Dyer, come indicato (4.2.2 in poi).
- Conservare classe di lipidi purificato in 100 ml di cloroformio: soluzione di metanolo (1: 1; vol / vol) a -20 ° C fino al momento dell'uso.
- Incubare foglio TLC sviluppata per 10 min in una camera cromatografia in presenza di 1 g di cristalli di iodio.
5. In Vitro Identificazione della fisiologica Substrato di un Lipasi
NOTA: In un approccio in vitro, lo studio se la lipasi di interesse in grado di convertire una miscela di lipidi isolate o singoli lipidi puri al Hydrol corrispondenteprodotti Ysis alle condizioni definite come ottimale in 3.2.
- Utilizzare sistemi di pipettaggio per saggi enzimatici come da Tabella 3 per specifiche PC fosfolipasi C SMc00171 (vedi 5.2), fosfolipasi A (vedi 5.3), e DAG lipasi SMc01003 (vedi 5.4) attività.
- Determinazione dell'attività fosfolipasi C specifica-PC (Tabella 3).
- Per una provetta da 1,5 ml microcentrifuga, aggiungere 5.000 conteggi per minuto (cpm) del totale 14 PC C-marcato e di una soluzione di Triton X-100.
- Mescolare e secco sotto corrente di azoto.
- Aggiungere dietanolammina-HCl, pH 9,8 tampone, nonché 2 soluzioni di NaCl e MnCl e acqua bidistillata per ottenere un volume finale di 99,5 ml. Vortex per 5 sec.
- Aggiungere 0,5 ml di enzima (5 mg proteina) (ossia, un estratto cellulare in cui overexpressed SMc00171 è presente) per iniziare la reazione. Mescolare brevemente.
- Incubare a 30 ° C per 4 ore.
- Arrestare la reazione da parte delaggiunta di 250 ml di metanolo e 125 ml di cloroformio.
- Estrarre i lipidi come descritto in precedenza (vedi 4.2).
- lipidi separate da unidimensionale (1D) -TLC (vedere 4.3.2 e 4.4), e li analizzano da immagini PSL.
- Determinazione dell'attività fosfolipasi A (Tabella 3).
- Per una provetta da 1,5 ml microcentrifuga, aggiungere 5.000 cpm del totale di 14 fosfolipidi C-marcato ed una soluzione di Triton X-100.
- Mescolare e secco sotto corrente di azoto.
- Per un saggio finale 100 microlitri, aggiungere Tris-HCl, pH 8,5 tampone, soluzione di NaCl e acqua. Vortex per 5 sec.
- Aggiungere 5 ml di enzima (50 mg di proteina) (vale a dire, un estratto cellulare in cui sovraespresso SMc00930 o SMc01003 è presente).
- Incubare a 30 ° C per 5 ore.
- Arrestare la reazione mediante aggiunta di 250 ml di metanolo e 125 ml di cloroformio.
- Estrarre i lipidi come descritto in precedenza (vedi 4.2), separatiloro da 1D-TLC usando 130 ml di cloroformio, 50 ml di metanolo e acido acetico glaciale a 20 ml come fase mobile, e analizzare con immagini PSL.
- Determinazione del diacilglicerolo (DAG) attività della lipasi.
- Preparazione di 14 C-marcato DAG.
- Radiomarcato S. culture meliloti (vedi 4.1) ed estrarre i lipidi polari (vedi 4.2), come descritto. Separato S. meliloti estratti totali lipidi di 1D-TLC in cloroformio: metanolo: acido acetico (130: 50: 20; vol / vol) utilizzando condizioni descritte per la separazione in seconda dimensione al punto 4.3.1.
- Visualizza PC mediante colorazione iodio e usare una matita per segnare la localizzazione di fosfatidilcolina (PC).
- Isolare PC radiomarcato come descritto al punto 4.5.
- Quantificare PC estratti per il conteggio a scintillazione.
NOTA: circa 320.000 cpm PC è previsto. - Trattare PC (250.000 cpm) con 0.1 U di fosfolipasi C da Clostridium perfringens in 50 mM Tris-HCl, pH 7,2, 0,5% Triton X-100 e 10 mM CaCl 2 per 2 ore in un volume totale di 100 microlitri e fermare la reazione aggiungendo 250 ml di metanolo e 125 ml di cloroformio.
- Estrarre i lipidi come descritto in precedenza e separata da loro da 1D-TLC (vedi 4.3.2).
- Isolare diacilglicerolo dalla piastra di silice e quantificare mediante conteggio a scintillazione (come descritto in 4.2)
- Saggio lipasi diacilglicerolo (Tabella 3).
- Per una provetta da 1,5 ml microcentrifuga, aggiungere 5.000 cpm di 14 C-marcato DAG e una soluzione di Triton X-100.
- Mescolare e secco sotto corrente di azoto.
- Per un saggio finale 100 microlitri, aggiungere Tris-HCl (pH 9,0) tampone, una soluzione di NaCl e acqua bidistillata. Vortex per 5 sec.
- Avviare la reazione aggiungendo 5 ml di enzima (50 mg di proteine di estratto cellulare).
- Incubare a 30 ° C per 4 ore.
- Arrestare la reazione mediante aggiunta di 250 ml di metanolo und 125 ml di cloroformio e di estratto di lipidi, come descritto in precedenza (vedi 4.2).
- Analizzare lipidi polari neutri per 1D-TLC (vedi 4.1.3.2) e la successiva di imaging PSL.
- Preparazione di 14 C-marcato DAG.
Risultati
Attività di specifici PC fosfolipasi C SMc00171 con Bis- p -nitrophenyl fosfato
Estratti privi di cellule ottenute da E. coli BL21 (DE3) pLysS x, che avevano smc00171 espressi, sono stati studiati per la loro capacità di idrolizzare bis- p esteri -nitrophenyl fosfato, utilizzando un saggio enzimatico spettrofotometrica, misurando la -NP
Discussione
Nel corso degli ultimi 20 anni, genomi di molti organismi sono stati sequenziati e anche se una ricchezza di dati sequenza del genoma è stato generato, interpretazione funzionale è in ritardo e ostacola quindi la nostra comprensione della funzione del genoma. funzioni geniche in genomi sono spesso assegnati in base alla somiglianza con geni di funzione nota o di insorgenza di motivi conservati. Tuttavia, la precisa funzione di un dato gene spesso non è noto. Soprattutto, previsto geni strutturali per enzimi non posso...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto da sovvenzioni dal Consejo Nacional de Ciencias y Tecnología-Messico (CONACYT-Messico) (82614, 153998, 253549, e 178.359 in Investigación Científica Básica e 118 in Investigación en Fronteras de la Ciencia) e dalla Dirección General de Asuntos de Personal Académico-Universidad Nacional Autónoma de México (UNAM DGAPA-; PAPIIT IN202616, IN203612).
Materiali
| Name | Company | Catalog Number | Comments |
| Chloroform | JT Baker | 9180-03 | TLC analysis & Lipid extraction |
| Methanol | JT Baker | 9070-03 | TLC analysis & Lipid extraction |
| Acetic Acid | JT Baker | 9507-05 | TLC analysis & Lipid extraction |
| Hexanes | JT Baker | 9309-02 | TLC analysis & Lipid extraction |
| Diethylether | Sigma | 32203 | Enzymatic assays |
| bidistilled water | ANY | NA | Enzymatic assays |
| Tris Base | Sigma | T-1503 | Enzymatic assays |
| HCl | Baker | 9535-02 | Enzymatic assays |
| NaCl | Baker | 3624-01 | Enzymatic assays |
| Triton X-100 | Sigma | X-100 | Enzymatic assays |
| LB broth | ANY | NA | Bacterial growth, 10 g tryptone + 5 g yeast extract + 10 g NaCl per liter of bidistilled water |
| tryptone | Becton Dickinson and Company | 211705 | Bacterial growth |
| yeast extract | Becton Dickinson and Company | 212750 | Bacterial growth |
| TY broth | ANY | NA | Bacterial growth, 8 g tryptone + 3 g yeast extract + 66 mg CaCl2·2H2O per liter of bidistilled water |
| CaCl2·2H2O | Baker | 1332-01 | Enzymatic assays |
| isopropyl-β-D-thiogalactoside (IPTG) | Invitrogen | 15529-019 | Bacterial growth |
| Diethanolamine | Sigma | D-8885 | Enzymatic assays |
| MnCl2 | Sigma | 221279 | Enzymatic assays |
| Phospholipase A2 snake venom | Sigma | P0790 | Enzymatic assays |
| Phospholipase C Clostridium perfringens | Sigma | P7633 | Enzymatic assays |
| Bis-p-nitrophenyl phosphate | Sigma | 07422AH | Enzymatic assays |
| p-nitrophenyl stearate | Sigma | N3627 | Enzymatic assays |
| p-nitrophenyl dodecanoate | Sigma | 61716 | Enzymatic assays |
| p-nitrophenyl decanoate | Sigma | N0252 | Enzymatic assays |
| p-nitrophenyl palmitate | Sigma | N2752 | Enzymatic assays |
| p-nitrophenyl butyrate | Sigma | N9876 | Enzymatic assays |
| p-nitrophenyl octanoate | Sigma | 21742 | Enzymatic assays |
| Acetic Acid, sodium salt [1-14C] | Perkin Elmer | NEC084 | Bacterial growth |
| dimethylsulfoxide (DMSO) | JT Baker | 9224-01 | Enzymatic assays |
| Aluminium HPTLC silica gel 60 plates. Silica gel HPTLC plates size 20 x 20 cm, 25 sheets. | Merck | 105547 | TLC analysis & Lipid extraction |
| Spectrometer UV/VIS Lambda 35 | Perkin Elmer | NA | Enzymatic assays |
| Storm 820 Phosphorimager | Molecular Dynamics | NA | Photostimulable Luminescence scanner |
| Multipurpose Scintillation Counter | Beckman Coulter | NA | Radioactivity Quantification |
| French Pressure Cell | ThermoSpectronic | NA | Breakage of cells |
| chromatography paper 3MM Chr | Whatman | 3030917 | TLC analysis |
| Sinorhizobium meliloti 1021our | reference 11 | studied strain | |
| Escherichia coli BL21 (DE3) pLysS Competent cells | Novagen | 69451 | protein expression strain |
| pET9a vector | Novagen | 69431 | protein expression vector |
| pET17b vector | Novagen | 69663 | protein expression vector |
| sterile polystyrene round-bottom tube (14 ml) Falcon | Becton Dickinson | 352057 | radiolabeling of bacterial cultures |
| polypropylene microcentrifuge tubes (1.5 ml) | Eppendorf | 30125.15 | Enzymatic assays |
| 1,2-dipalmitoyl-sn-glycerol | Sigma | D9135 | lipid standard |
| L-α-phosphatidylcholine, dipalmitoyl | Sigma | P6267 | lipid standard |
| DL-α-monopalmitin | Sigma | M1640 | lipid standard |
| palmitic acid | Sigma | P0500 | lipid standard |
Riferimenti
- Nelson, D. L., Cox, M. M. . Lehninger, Principles of Biochemistry. , (2013).
- Jaeger, K. E., Eggert, T. Lipases for biotechnology. Curr. Opin. Biotechnol. 13 (4), 390-397 (2002).
- Dowhan, W., Bogdanov, M., Mileykovskaya, E. Functional roles of lipids in membranes. Biochemistry of Lipids, Lipoproteins and Membranes. , 1-37 (2008).
- Dowhan, W. Molecular basis for membrane phospholipid diversity: why are there so many lipids?. Annu. Rev. Biochem. 66, 199-232 (1997).
- De Rudder, K. E. E., Thomas-Oates, J. E., Geiger, O. Rhizobium meliloti mutants deficient in phospholipid N-methyltransferase still contain phosphatidylcholine. J. Bacteriol. 179, 6921-6928 (1997).
- Geiger, O., Röhrs, V., Weissenmayer, B., Finan, T. M., Thomas-Oates, J. E. The regulator gene phoB mediates phosphate stress-controlled synthesis of the membrane lipid diacylglyceryl-N,N,N-trimethylhomoserine in Rhizobium (Sinorhizobium) meliloti. Mol. Microbiol. 32 (1), 63-73 (1999).
- Klug, R. M., Benning, C. Two enzymes of diacylglyceryl-O-4'-(N,N,N,-trimethyl)homoserine biosynthesis are encoded by btaA and btaB in the purple bacterium Rhodobacter sphaeroides. Proc. Natl. Acad. Sci. USA. 98 (10), 5910-5915 (2001).
- Zavaleta-Pastor, M., et al. Sinorhizobium meliloti phospholipase C required for lipid remodeling duringphosphorus limitation. Proc. Natl. Acad. Sci. USA. 107 (1), 302-307 (2010).
- Pech-Canul, A., et al. FadD is required for utilization of endogenous fatty acids released from membrane lipids. J. Bacteriol. 193 (22), 6295-6304 (2011).
- Banerji, S., Flieger, A. Patatin-like proteins: a new family of lipolytic enzymes present in bacteria?. Microbiology. 150 (Pt 3), 522-525 (2004).
- Sahonero-Canavesi, D. X., et al. Fatty acid-releasing activities in Sinorhizobium meliloti include unusual diacylglycerol lipase. Environ. Microbiol. 17 (9), 3391-3406 (2015).
- Fischer, M., Pleiss, J. The Lipase Engineering Database: a navigation and analysis tool for protein families. Nucl. Acid. Res. 31 (1), 319-321 (2003).
- Sigrist, C. J. A., et al. New and continuing developments at PROSITE. Nucleic Acids Res. 41 (Database issue), D344-D347 (2013).
- Lorenz, T. C. Polymerase Chain Reaction: Basic Protocol Plus Troubleshooting and Optimization Strategies. J. Vis. Exp. (63), e3998 (2012).
- Untergasser, A., et al. Primer3 - new capabilities and interfaces. Nucl. Acids Res. 40 (15), e115 (2012).
- Studier, F. W., Rosenberg, A. H., Dunn, J. J., Dubendorff, J. W. Use of T7 RNA polymerase to direct expression of cloned genes. Methods Enzymol. 185, 60-89 (1990).
- Miller, J. H. . Experiments in Molecular Genetics. , (1972).
- Desjardins, P., Hansen, J. B., Allen, M. Microvolume Protein Concentration Determination using the NanoDrop 2000c Spectrophotometer. J. Vis. Exp. (33), e1610 (2009).
- Bligh, E. G., Dyer, W. J. A rapid method for total lipid extraction and purification. Can. J. Biochem. Physiol. 37 (8), 911-917 (1959).
- Molecular Dynamics. . Phosphorimager SI User´s Guide. , (1994).
- Dixon, M., Webb, E. . Enzymes: Third Edition. , (1979).
- Rudolph, A. E., et al. Expression, characterization, and mutagenesis of the Yersinia pestis murine toxin, a phospholipase D superfamily member. J. Biol. Chem. 274 (17), 11824-11831 (1999).
- Kato, S., Yoshimura, T., Hemmi, H., Moriyama, R. Biochemical analysis of a novel lipolytic enzyme YvdO from Bacillus subtilis. Biosci. Biotechnol. Biochem. 74 (4), 701-706 (2010).
- Peppelenbosch, M. P. Kinome profiling. Scientifica (Cairo). , (2012).
- Manafi, M., Kneifel, W., Bascomb, S. Fluorogenic and chromogenic substrates used in bacterial diagnostics. Microbiol. Rev. 55 (3), 335-348 (1991).
- Kuznetsova, E., et al. Enzyme genomics: Application of general enzymatic screens to discover new enzymes. FEMS Microbiol. Rev. 29 (2), 263-279 (2005).
- Scopes, R. K. . Protein Purification, Principles and Practice. , (2010).
- Geiger, O., Lopez-Lara, I. M., Sohlenkamp, C. Phosphatidylcholine biosynthesis and function in bacteria. Biochim. Biophys. Acta. 1831 (3), 503-513 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon