È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Flusso analisi di citometria di Natural Killer cellulare Lytic attività nel Sangue umano intero
In questo articolo
Riepilogo
This work describes an advanced workflow for the accurate and fast determination of NK (Natural Killer) cell count and NK cell cytotoxicity in human blood samples.
Abstract
citotossicità delle cellule NK è una misura ampiamente utilizzato per determinare l'effetto di un intervento esterno sulla funzione delle cellule NK. Tuttavia, la precisione e la riproducibilità di questo test possono essere considerati instabili, sia a causa di errori dell'utente oa causa della sensibilità delle cellule NK di manipolazione sperimentale. Per eliminare questi problemi, un flusso di lavoro che li riduce al minimo è stato stabilito ed è presentato qui. Per illustrare, abbiamo ottenuto campioni di sangue, in vari momenti, da corridori (n = 4), che sono stati sottoposti ad un intenso periodo di esercizio. In primo luogo, le cellule NK sono contemporaneamente identificati e isolati attraverso CD56 tagging e smistamento magnetico-based, direttamente dal sangue intero e da un minimo di un millilitro. Le cellule NK ordinati vengono rimossi di eventuali anticorpi reagenti o tappatura. Essi possono essere contati per stabilire un accurato conteggio delle cellule NK per millilitro di sangue. In secondo luogo, le cellule NK ordinati (effettori cellule o E) possono essere miscelati con 3,3'-Diotadecyloxacarbocyanine perclorato (DIO) etichettato cellule K562 (cellule bersaglio o T) in un saggio-ottimale 1: 5 T: rapporto E e analizzato utilizzando un flusso-citometria di imaging che permette la visualizzazione di ogni evento e l'eliminazione di falsi positivi o falsi negativi (come doppiette o cellule effettrici). Questo flusso di lavoro può essere completato in circa 4 ore, e consente di ottenere risultati molto stabili anche quando si lavora con campioni umani. Quando disponibile, gruppi di ricerca possono testare diversi interventi sperimentali in soggetti umani, e confrontare le misurazioni attraverso diversi punti di tempo senza compromettere l'integrità dei dati.
Introduzione
cellule killer naturali sono un elemento essenziale del sistema immunitario innato. Mentre sono molto disciplinati, hanno la capacità di riconoscere ed eliminare le cellule anomale attraverso il contatto cellula-cellula e senza attivazione preventiva 1. Come tali, essi formano una linea veloce di difesa contro le infezioni. Esercizio, particolarmente intenso, ha dimostrato di deprimere transitoriamente il sistema immunitario 2, 3, 4, 5. Cellule NK sono particolarmente soggetti a questo effetto 4, 6, 7, creando una finestra di una maggiore sensibilità alla malattia. Pertanto, lo studio di interventi prima, durante o dopo un intenso esercizio con l'obiettivo di ridurre l'impatto sulla funzione delle cellule NK è di particolare interesse per il benessere degli atleti post-concorsi.
Tuttavia, lo studio di tali interventi è complicata da numerosi fattori: 1) le cellule NK sono sparse 8, a circa l'1% del vano globuli bianchi; 2) le cellule NK sono molto sensibili allo stress e si basano su costante esposizione a condizioni fisiologiche di rimanere vitali e stabile durante la sperimentazione; e 3) test standard per determinare la citotossicità delle cellule NK, come Ficoll gradienti 9 e rilasciare dosaggi 10, sono inaffidabili e inconsistenti. La variabilità intrinseca dei campioni umani composti soltanto questi problemi. campioni umani freschi raccolti durante interventi sono abbastanza regolamentato e difficili da procurare, almeno rispetto ai campioni animali o linee cellulari immortalizzate. Questo riduce la possibilità di ripetere gli esperimenti o aggiungere partecipanti allo studio di coorte per raggiungere significativi soglie statistiche. Collettivamente, questi problemi sostengono la necessità di un protocollo semplificato che permette FOR sia high-throughput e l'analisi ad alta affidabilità di NK attività litica delle cellule in campioni umani.
Abbiamo stabilito un flusso di lavoro che riduce il tempo necessario per identificare, isolare e le cellule NK di prova da sangue intero umano, riducendo al minimo l'esposizione a fattori estranei. Il metodo ottimizza l'uso di due strumenti, un sorter magnetico a base cellulare e un flusso di imaging citometro, ed una, T ottimizzata specifico per il dosaggio: rapporto E per consentire il rilevamento di diminuzioni o incrementi di citotossicità delle cellule NK.
Access restricted. Please log in or start a trial to view this content.
Protocollo
NOTA: Tutte le procedure di raccolta del sangue sono state condotte in conformità con le linee guida stabilite dalla Appalachian State University (ASU) Institutional Review Board (IRB).
1. Raccolta Sangue intero
- Avere un flebotomo certificato prelevare il sangue secondo le linee guida dell'Organizzazione Mondiale della Sanità.
- Disegnare il sangue in un 4 ml di tubo di raccolta del sangue contenente acido Di-Potassio etilendiamminotetraacetico (K 2 EDTA). Capovolgere tubo di raccolta sangue secondo le istruzioni del produttore. Mantenere tubo di raccolta di sangue a temperatura ambiente su un agitatore meccanico banco fino separazione.
2. Preparazione delle cellule bersaglio Dio-etichettati
- Crescere K562 cellule in completa di Iscove Modified Dulbecco media (IMDM) supplementato con siero fetale bovino al 10% (FBS) e 1% di penicillina streptomicina per diverse settimane prima del test per garantire la salute delle cellule. Regolare il concentratione delle cellule per 3 x 10 5 cellule / ml al giorno attraverso il completamento di una cella di conteggio e successivo passaggio delle cellule. Eseguire un completo cambiamento dei media ogni 2 o 3 giorni.
- Il giorno del test, eseguire una conta cellulare utilizzando un 1: 1 emocitometro.
- Rimuovere 10 microlitri dal pallone K562 e posto in 1,5 ml di tubo.
- Aggiungere 10 ml di colorante blu trypan nella stessa provetta da 1,5 ml per un fattore di diluizione di 1: 1.
- Delicatamente a scatti la provetta per mescolare K562 cellule e trypan colorante blu.
- Lasciare K562 colorante blu cellula-trypan per incubare per 1 minuto a temperatura ambiente.
- Rimuovere 15 ml di cellule K562 macchiati dal tubo.
- Pipetta su emocitometro per la conta delle cellule.
- Contare le celle nelle quattro piazze d'angolo così come la piazza centrale per un totale di cinque piazze.
- Calcolare conta cellulare utilizzando l'equazione:
Totale cellule / ml = cellule totali contate X (fattore di diluizione / numero di caselle) X 10.000 / mL
- Risospendere le cellule bersaglio K562 con una densità finale di 1 x 10 6 cellule / ml in terreno di coltura libera IMDM siero.
- Per le cellule bersaglio K562 senza macchia, aggiungere 10 ml di cellule bersaglio K562 a uno 15 ml di tubo ad una densità di 1 x 10 6 cellule / ml per un totale di 10 x 10 6 cellule K562. Mettere in un incubatore a 37 ° con 5% di CO 2 fino al momento dell'uso.
- Per le cellule bersaglio DiO macchiato K562, aggiungere 10 ml di K562 obiettivi cellule ad uno 15 ml di tubo ad una densità di 1 x 10 6 cellule / ml per un totale di 10 x 10 6 cellule K562.
- Aggiungere 1 ml di soluzione DiO cella-etichettatura per ml di sospensione cellulare in 15 ml di tubo designato per DiO colorazione e delicatamente vortice. Ad esempio, aggiungere 10 ml di soluzione di cellule-etichettatura DiO a 10 x 10 6 cellule / ml K562 ad un volume di 1 x 10 6 cellule / ml.
- Incubare soluzione K562-DiO per 20 min a 37 ° C con 5% di CO 2 in una provetta da 15 ml.
- Seguiincubazione ing, aggiungere 3 ml di soluzione fisiologica a temperatura ambiente tampone fosfato (PBS) per soluzione K562-dio contenente 15 ml di tubo.
- Centrifugare per 10 min a 135 xg a temperatura ambiente.
- Rimuovere con attenzione il surnatante senza disturbare il pellet cellulare con 1.000 ml di volume regolabile pipetta.
- Aggiungere 10 ml di IMDM fresco alla cella pellet contenente 15 ml di tubo.
- Delicatamente tubo vortex per risospendere le cellule.
- Ripetere i punti 2.7 a 2.10 altre due volte.
- Cellule Conservare in un incubatore a 37 ° con il 5% di CO 2 fino al momento dell'uso.
NOTA: Le celle possono essere memorizzati in incubatrice fino a 24 h, ma è preferibile usarli lo stesso giorno.
3. Preparazione di controlli
- Trasferire il seguente in separati e opportunamente etichettati tubi da 1,5 ml:
- Aggiungere 500 ml di IMDM fresco contenente risospeso DIO-etichettato K562 cellule nel "Double Positive" etichettato 1,5 ml di tubo.
- Aggiungere 500 ml di IMDM fresco contenente risospeso cellule K562 DIO-etichettati in "Dio solo" etichettato 1,5 ml di tubo.
- Aggiungere 500 ml di IMDM fresco contenente cellule K562 senza etichetta risospese nella "ioduro di propidio (PI) solo" etichettati 1,5 ml di tubo.
- Posizionare il doppio positivo e PI solo tubi in un bagno d'acqua C 55 ° per 5 min.
- Dopo la 5 minuti è trascorso, rimuovere i tubi e pulire con il 70% di etanolo.
- Aggiungere 10 L di PI al doppio positivo e PI solo 1,5 mL.
- Mettere tutti i tre controlli K562 cellule bersaglio in incubatore a 37 ° C per 30 min.
- Dopo 30 min di incubazione è trascorso, centrifugare tutti i tre controlli di cellule bersaglio K562 per 2 min a 163 x g.
- Rimuovere con attenzione il surnatante senza disturbare il pellet.
- Risospendere ogni controllo con 20 ml di mezzi di coltura cellulare IMDM fresco, e lasciare nel 37 ° C incubatore con 5% di CO 2 per almeno30 min per il segnale DiO ottimale.
NOTA: I controlli sono ora pronti per essere eseguito attraverso il flusso di imaging citometro.
4. NK cellulare automatizzata Separazione
- Accendere separatore cellulare e consentire il ciclo di start-up alla fine.
- Assicurarsi che tutte le bottiglie di illuminazioni fluidi sono verdi e che la bottiglia è vuota rifiuti.
- Ottenere un ambiente a temperatura 15 ml portaprovette.
NOTA: La selezione è basata su volumi di campione. Ad esempio, per un volume inferiore a 3 ml utilizzano un portaprovette 15 ml e per un volume superiore a 3 mL utilizzano una mL cremagliera 50 del tubo. - tubi campione Etichetta di conseguenza (Ripeti per campione / partecipante): Partecipante 1 campione di sangue intero; Partecipante 1 frazione negativa; Partecipante 1 frazione positiva.
- pipettare delicatamente 1,5 ml di sangue intero dal punto 1.2 in 15 tubo "intero campione di sangue" mL.
- Posizionare il tubo adeguatamente etichettati 15 mL "intero campione di sangue" dal punto 4.5 ed etichettati 15 mL "frazione negativa" etubi "frazione positiva" dal passo 4.4 nel rack tubo. Utilizzare thefollowing cremagliera campione di set-up: Riga (R1) A: Campione di sangue intero, Fila (R2) B: Negativo, frazione senza etichetta, Fila (R4) C: Positivo, frazione magneticamente etichettati.
- Inserire il rack di separazione sul MiniSampler per autolabeling.
- Selezionare "reagente" sul menu ed evidenziare la posizione in cui il flaconcino verrà inserito nel rack separatore.
- Selezionare "Leggi reagente" per attivare il lettore di codici 2D.
- Posizionare la fiala reattivo appropriato davanti al lettore di codici 2D tra 0,5-2,5 cm dal coperchio del lettore di codice.
NOTA: per esempio, il reagente necessario per la separazione delle cellule NK è CD56. - Tenere reagente flacone con un angolo di fronte lettore di codici 2D per una lettura ottimale.
- Posizionare il flacone di reagente in posizione corretta cremagliera separatore.
- Evidenziare le posizioni desiderate nella scheda di separazione sullo schermo.
- Dal sottomenu Etichettatura, assegnare un auprogramma di tolabeling.
- Assegnare le cellule di separazione reagente CD56 microsfere di accumulare posizioni 1, 2, 3, e 4.
- Selezionare il programma di separazione "posselwb".
- Selezionare il programma di lavaggio "risciacquo".
- Inserire un volume di campione di 1.500 ml nel sottomenu "Volume" utilizzando il tastierino numerico.
- Selezionare l'opzione "Invio" sulla tastiera.
- Selezionare "Run" per avviare la separazione delle cellule.
- Selezionare "OK" per confermare tale buffer sufficiente è disponibile per l'elaborazione di tutti i campioni.
5. NK cellulare Conte seguito cellulare Separazione
- Immediatamente dopo separazione cellulare con il separatore cellulare, recuperare la frazione positiva. Lasciare a temperatura ambiente. Questa frazione contiene la popolazione di cellule NK puro desiderato.
- Per ogni singolo campione, eseguire una conta delle cellule utilizzando un emocitometro come da Fase 2.2.
- Dopo i calcoli, registrare la conta delle cellule.
6. citotossicità Assay Preparazione del campione
- Preparare ed etichettare provette da 1,5 ml per ogni campione / partecipante di conseguenza.
- Pipetta rapporto tra cellule NK e K562 DIO-etichettati desiderato in ogni provetta.
NOTA: per esempio, il rapporto desiderato di cellule bersaglio K562 e cellule effettrici NK è 1: 5. - Centrifugare per 5 min a 135 x g.
- Rimuovere con attenzione il surnatante senza disturbare il pellet.
- Risospendere NK-dio etichettato miscela di cellule K562 in 500 ml di mezzi delle cellule NK, senza l'interleuchina-2 (IL-2) e 2-mercaptoetanolo (2-ME) (incompleta NK coltura cellulare media).
NOTA: I terreni di coltura delle cellule NK incompleta è Minimum Essential medio di Eagle con bicarbonato di sodio, senza L-glutammina, ribonucleosides e deoxyribonucleosides. - Aggiungere 5 ml di PI ad ogni provetta.
- Centrifugare per 2 minuti a 163 x g.
- Incubare le cellule a 37 ° C per 2 ore.
- Dopo 2 ore di incubazione, centrifuge per 2 min a 163 x g.
- Rimuovere con attenzione il surnatante senza disturbare il pellet.
- Risospendere le cellule in 25 microlitri incompleta terreni di coltura delle cellule NK.
7. Preparazione di spontanea ( "S") Esempio
- Pipettare 500 ml di cellule K562 DIO-etichettati (concentrazione di 1 x 10 6 cellule / ml) in 1,5 ml di tubo.
- Aggiungere 10 ml di PI ad ogni provetta.
- provetta per centrifuga per 2 min a 163 x g.
- Incubare le cellule a 37 ° C per 2 ore.
- Dopo 2 h di incubazione, centrifugare per 2 min a 163 x g.
- Rimuovere con attenzione il surnatante senza disturbare il pellet.
- Risospendere le cellule in 25 ml incompleta Essential Medium (α-MEM) terreni di coltura delle cellule alfa-minimo.
8. Acquisizione dati con Imaging citofluorimetro
- Premere il tasto verde all'interno dello sportello anteriore della dell'imaging citofluorimetro per accendere lo strumento.
- Attivare all computer associati imaging citofluorimetro.
- Avviare il flusso di imaging citometro software.
- Fare clic sul pulsante "Avvio" per lavare il sistema e preparare la linea del campione.
- Una volta che il "Avvio" è completa, chiudere la finestra "Calibrations".
- Assegnare i canali: sul lato superiore sinistro, cliccare su ogni canale, al fine di assegnare loro.
- Sul lato destro, fare clic sul pulsante grafico a dispersione per creare 4 a dispersione: Raw Max Pixel _MC_6 vs Area_M06, Crudo Max Pixel _MC_2 vs Area_M02, Crudo Max Pixel _MC_5 vs Area_M05 e FieldArea_M01 vs AspectRatio_M01.
- Iniziare l'analisi di campioni in primo luogo usando il comando "Doppio positivo".
- Clicca su "Carica".
- Posizionare il tubo da 1,5 ml con il campione "Double Positive" da Piazza di 3,4 a 3,9 nel caricatore del campione.
- Selezionare l'obiettivo 40X nella scheda "ingrandimento".
- Accendere il 405 mW e642 laser MW.
- Accendere il canale "Brightfield".
- Clicca su "Seleziona intensità."
- Sulla base del campione di controllo "Double positivo", determinare l'intensità desiderata per il laser 405 mW quindi il rivelatore non è sovraccarico.
Nota: Ad esempio, l'intensità ottimale per questo esperimento è stato fissato a 11 mW.
- Dopo il set-up desiderato si ottiene, acquisire i dati.
- Nella scheda "File di acquisizione", inserire il nome del file in un testo personalizzato. Selezionare una cartella in cui salvare il file di dati (s).
- Inserire il numero di cellule di acquisire accanto a "raccogliere". In genere questo numero varia tra 1.000 a 10.000.
- Clicca su "Acquisisci".
NOTA: Una volta che il numero desiderato di cellule viene acquisito, il file di dati viene salvato automaticamente nella cartella precedentemente selezionata.
- Dopo l'acquisizione termina, caricare il campione di controllo successivo - Dio solo il controllo.
- Clicca su "Carica".
- Posizionare il tubo 1,5 ml con l ' "unico Dio" campione nel caricatore di campione.
- Lasciare l'obiettivo 40X nella scheda "ingrandimento" selezionato.
- Lasciare il laser 405 mW acceso.
- Spegnere il laser 642 mW e il canale "Brightfield".
NOTA: Ora che la desiderata set-up è stato raggiunto, i dati possono essere acquisiti. - Nella scheda "File di acquisizione", inserire il nome del file in un testo personalizzato e selezionare una cartella in cui salvare il file di dati (s). Inserire il numero di cellule di acquisire accanto a "raccogliere".. In genere questo numero è 1.000.
- Clicca su "Acquisisci".
NOTA: Una volta che il numero desiderato di cellule viene acquisito il file di dati viene salvato automaticamente nella cartella precedentemente selezionata.
- Ripetere il passaggio 8.10 per il "solo PI" campione di controllo. I campioni sperimentali sono ora pronti per essere raccolti.
- Gestire le remaining campioni sperimentali, tra cui i "Campioni spontanee" S "" come segue:
- Lasciare l'obiettivo 40X nella scheda "ingrandimento" selezionato.
- Accendere il 405 mW e 642 mW laser.
- Attivare il canale "Brightfield".
- Clicca su "Imposta intensità."
- Nella scheda "File di acquisizione", inserire il nome del file in un testo personalizzato e selezionare una cartella in cui salvare il file di dati (s). Inserire in un testo il nome del file personalizzato.
- Inserire il numero di cellule di acquisire accanto a "raccogliere".
- Fare clic sul pulsante "Acquisisci".
NOTA: Una volta che il numero desiderato di cellule viene acquisito il file di dati viene salvato automaticamente nella cartella precedentemente selezionata.
- Ripetere il passaggio 8.12 per tutti i campioni sperimentali.
- Dopo che tutti i dati sperimentali e file sono stati raccolti, fare clic sul pulsante "Shutdown" per sterilizzare il sistema.
9. ImaginAnalisi dei campioni g citofluorimetro
- Aprire il flusso di imaging citometro applicazione software di analisi.
- Sotto "File", aprire un file .RIF sperimentale.
- Costruire una nuova matrice utilizzando i file .RIF singolo colore (Dio-solo il controllo e il controllo PI-only, creato durante fasi 8.10 e 8.11) selezionando "Crea un nuovo Matrix" nella scheda di compensazione nel flusso di immagini citometro software.
NOTA: Il software chiederà per la selezione dei file a colori singoli e unirli per creare un file di matrice (estensione del file .ctm), che deve essere selezionato per applicare la compensazione del canale. - Creare diagrammi di punti utilizzando la funzione di "mattoni" del software.
- Creare un dot-plot (BrightFieldGradient_RMS vs Frequency) per porta le cellule mirate. Chiama il cancello "Focus" (Figura 1A).
- Utilizzando la porta "Focus", creare un diagramma a punti di Bright Campo Area vs campo luminoso Proporzioni di g mangiato le cellule di singoletto. Chiama il cancello "Single" (Figura 1B).
- Utilizzando la porta "Single", creare un diagramma a punti di Intensity_MC_Ch02 vs Intensity_MC_Ch5. Utilizzare questa trama per identificare e porta Dio-positivo solo le cellule (obiettivi, vivo) e PI-dio cellule doppio positive (obiettivi, morte) (Figura 1C).
NOTA: Tutte le trame descritto nei punti 9.4.1, 9.4.2, 9.4.3 e possono essere creati utilizzando la funzione "mattoni" del software.
- Fare clic sulla funzione statistica della trama punto per accedere ai numeri cellulari di ogni porta.
- Calcolare la percentuale di bersagli morti nel campione spontanea e campioni sperimentali utilizzando la seguente formula:
% bersagli morti in campione = (target #dead x 100) / (# vivono obiettivi + #dead obiettivi) - Calcolare citotossicità utilizzando la seguente formula:
% Citotossicità = [(Experimental dead-spontaneo morto) / (morto 100-spontaneo)] x100
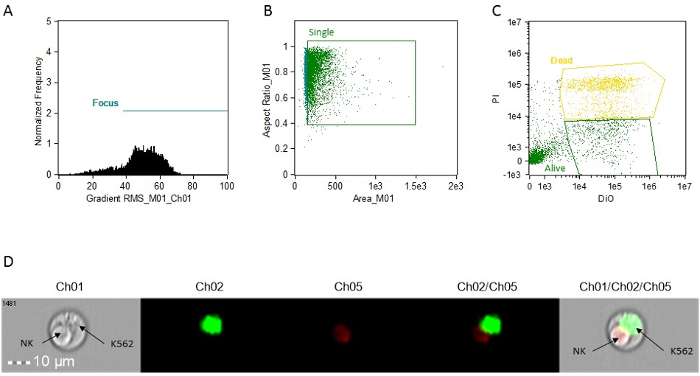
Figura 1: istogrammi rappresentativi, grafici a dispersione e le immagini per l'analisi attività citotossica. Analisi (A) cella di messa a fuoco. (B) analisi singola cella. Analisi (C) bersaglio colorazione delle cellule. Tutte le determinazioni sono realizzati con l'immagine allegata ad ogni evento. Questo può essere letta in software di analisi, semplicemente cliccando sulla manifestazione sui grafici. (D) immagine rappresentativa di un evento doppietto, che mostra un cellulare per apoptosi NK e un obiettivo K562 dal vivo. CH01, Brightfield. Ch02, DIO. CH05, PI. Clicca qui per vedere una versione più grande di questa figura.
Access restricted. Please log in or start a trial to view this content.
Risultati
Determinazione del numero di cellule NK
L'effetto della corsa pesante sul conteggio delle cellule NK nel sangue intero è stato misurato, utilizzando il protocollo di esercizio descritto nella Figura 2. I campioni di sangue sono stati elaborati prima dell'esercizio, immediatamente dopo l'esercizio, 1,5 ore dopo l'esercizio, e, infine, 24 e 48 ore dopo il prelievo di sangue iniziale. La concentrazione di cellule NK per millilitro di sangue intero è...
Access restricted. Please log in or start a trial to view this content.
Discussione
Il metodo descritto in questo studio misura direttamente l'attività specifica di cellule NK di un individuo in risposta a stimoli (in questo caso particolare, esercizio prolungato). Tipicamente, le cellule NK sono isolati dal proprio sangue utilizzando gradienti di densità o di separazione delle cellule utilizzando una combinazione di marcatori. Mentre questi metodi sono ampiamente utilizzati, hanno molti svantaggi: sono in termini di tempo, coinvolgono più manipolazioni, e sono fortemente dipendenti dell'ute...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
This project was supported by Agriculture and Food Research Initiative Competitive Grant no. 2100-68003-30395 from the USDA National Institute of Food and Agriculture.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| K-562 lymphoblasts | ATCC | CCL-243 | |
| Iscove's Modified Dulbecco's Media | ATCC | 30-2005 | High glucose, with L-Glutamine, with HEPES, Sterile-filtered |
| Alpha Minimum Essential medium | ATCC | CRL-2407 | Without ribonucleosides and deoxyribonucleosides but with 2 mM L-glutamine and 1.5 g/L sodium bicarbonate |
| Trypan Blue Solution 0.4% | Amresco | K940-100ML | Tissue culture grade |
| Propridium Iodide Staining Solution | BD Pharmingen | 51-66211E | |
| Vybranto DiO cell-labeling solution | Vybranto | V-22886 | |
| autoMACS Pro Separator | Miltenyi Biotec | 130-092-545 | |
| autoMACS Running Buffer | Miltenyi Biotec | 130-091-221 | |
| autoMACS Washing Buffer | Miltenyi Biotec | 130-092-987 | |
| autoMACS Columns | Miltenyi Biotec | 130-021-101 | |
| Whole Blood CD56 MicroBeads, human | Miltenyi Biotec | 130-090-875 | |
| ImageStream X Mark II Imaging Flow Cytometer | EMD Millipore | ||
| Speedbeads | Amnis Corporation | 400030 | |
| 0.4-0.7% Hypochlorite (Sterilizer) | VWR | JT9416-1 | |
| Coulter Clenz | Beckman Coulter | 8546929 | |
| 70% Isopropanol (Debubbler) | EMD Millipore | 1.3704 | |
| D-PBS (Sheath fluid) | EMD Millipore | BSS-1006-B (1X) | No calcium or magnesium |
| INSPIRE Software | EMD Millipore | Version Mark II, September 2013 | |
| Ideas Application Software | EMD Millipore | Version 6.1, July 2014 |
Riferimenti
- Cerwenka, A., Lanier, L. L. Natural killer cells, viruses and cancer. Nat Rev Immunol. 1 (1), 41-49 (2001).
- Nieman, D. C. Exercise, infection, and immunity. Int J Sports Med. 15, Suppl 3 131-141 (1994).
- Romeo, J., Warnberg, J., Pozo, T., Marcos, A. Physical activity, immunity and infection. Proc Nutr Soc. 69 (3), 390-399 (2010).
- Nieman, D. C. Marathon training and immune function. Sports Med. 37 (4-5), 412-415 (2007).
- Simpson, R. J., Kunz, H., Agha, N., Graff, R. Exercise and the Regulation of Immune Functions. Prog Mol Biol Transl Sci. 135, 355-380 (2015).
- Walsh, N. P., et al. Position statement. Part one: Immune function and exercise. Exerc Immunol Rev. 17, 6-63 (2011).
- Timmons, B. W., Cieslak, T. Human natural killer cell subsets and acute exercise: a brief review. Exerc Immunol Rev. 14, 8-23 (2008).
- Westermann, J., Pabst, R. Distribution of lymphocyte subsets and natural killer cells in the human body. Clin Investig. 70 (7), 539-544 (1992).
- Boyum, A. Isolation of leucocytes from human blood. Further observations. Methylcellulose, dextran, and ficoll as erythrocyteaggregating agents. Scand J Clin Lab Invest Suppl. 97, 31-50 (1968).
- McMillan, R., Scott, J. L. Leukocyte labeling with 51-Chromium. I. Technic and results in normal subjects. Blood. 32 (5), 738-754 (1968).
- Berk, L. S., et al. The effect of long endurance running on natural killer cells in marathoners. Med Sci Sports Exerc. 22 (2), 207-212 (1990).
- McAnulty, L. S., et al. Effect of blueberry ingestion on natural killer cell counts, oxidative stress, and inflammation prior to and after 2.5 h of running. Appl Physiol Nutr Metab. 36 (6), 976-984 (2011).
- Millard, A. L., et al. Brief Exercise Increases Peripheral Blood NK Cell Counts without Immediate Functional Changes, but Impairs their Responses to ex vivo Stimulation. Front Immunol. 4, 125(2013).
- Janeway, C. A., Travers, P., Walport, M., Shlomchik, M. J. Immunobiology: The Immune System in Health and Disease. 5th edn. , Garland Science. (2001).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon