Method Article
Alta risoluzione Quantitative Synaptic Proteome profiling di cervello di topo Regioni Dopo uditiva discriminazione Learning
In questo articolo
Riepilogo
The identification of molecules and pathways controlling synaptic plasticity and memory is still a major challenge in neuroscience. Here, a workflow is described addressing the relative quantification of synaptic proteins supposedly involved in the molecular reorganization of synapses during learning and memory consolidation in an auditory learning paradigm.
Abstract
The molecular synaptic mechanisms underlying auditory learning and memory remain largely unknown. Here, the workflow of a proteomic study on auditory discrimination learning in mice is described. In this learning paradigm, mice are trained in a shuttle box Go/NoGo-task to discriminate between rising and falling frequency-modulated tones in order to avoid a mild electric foot-shock. The protocol involves the enrichment of synaptosomes from four brain areas, namely the auditory cortex, frontal cortex, hippocampus, and striatum, at different stages of training. Synaptic protein expression patterns obtained from trained mice are compared to naïve controls using a proteomic approach. To achieve sufficient analytical depth, samples are fractionated in three different ways prior to mass spectrometry, namely 1D SDS-PAGE/in-gel digestion, in-solution digestion and phospho-peptide enrichment.
High-resolution proteomic analysis on a mass spectrometer and label-free quantification are used to examine synaptic protein profiles in phospho-peptide-depleted and phospho-peptide-enriched fractions of synaptosomal protein samples. A commercial software package is utilized to reveal proteins and phospho-peptides with significantly regulated relative synaptic abundance levels (trained/naïve controls). Common and differential regulation modes for the synaptic proteome in the investigated brain regions of mice after training were observed. Subsequently, meta-analyses utilizing several databases are employed to identify underlying cellular functions and biological pathways.
Introduzione
L'apprendimento si basa sulla formazione di tracce di memoria e la loro manutenzione. E 'ampiamente accettato che un meccanismo di base può rappresentare una formazione attività-dipendente di nuovi e / o riarrangiamento dei contatti sinaptici esistenti tra i neuroni. A livello molecolare, varie modifiche proteine, relocalizations subcellulari e le variazioni del fatturato delle proteine sinaptiche sono stati descritti 1-4 (Lamprecht 2004 # 8). Tuttavia, la maggior parte degli studi finora focalizzati su proteine selezionate piuttosto che sulla composizione proteoma sinaptica globale, ma complessa. L'attuale approccio consente uno screening imparziale per le modifiche proteoma sinaptiche nelle regioni del mouse cervello dopo un esperimento di apprendimento. È adatto a rendere time-point istantanee molecolari dipendenti della riorganizzazione apprendimento indotta dell'architettura sinaptica. Il flusso di lavoro descritto richiede un particolare lavoro di squadra di diversi specialisti in comportamento animale, la biochimica delle proteine, spettrometria di massa e bioinformatics.
Il paradigma dell'apprendimento prescelto, vale a dire il tono discriminazione a modulazione di frequenza (FMTD), è un compito di discriminazione uditiva ben caratterizzato nei roditori 5. Apprendimento e formazione della memoria a lungo termine in questa casella navetta Go / No-Go-task coinvolge meccanismi seconda maggiore segnalazione della dopamina corticale e la sintesi proteica. Di conseguenza, recenti studi di proteomica su gerbilli e topi hanno rivelato dopamina e di apprendimento indotto riarrangiamenti di plastica di componenti sinaptiche in corticale, ma anche nelle regioni del cervello più basali che presumibilmente interagiscono durante FMTD apprendimento e la memoria 6-8. Questo dimostra che la formazione della memoria comporta una complessa interazione di varie regioni del cervello e, quindi, potrebbe essere regolata in modo differenziato all'interno di queste regioni a livello proteoma. Pertanto, la dissezione delle regioni del mouse cerebrali corticali e subcorticali selezionate è incluso nel flusso di lavoro.
Inoltre, il characterizati affidabileacceso anche delle variazioni deboli nella composizione proteica sinaptica richiede un arricchimento di compartimenti pre- e postsinaptici piuttosto che l'analisi di omogenati o frazioni di membrana greggio 9. Protocolli Pertanto, la preparazione di sinaptosomi utilizzanti stabilita prima dell'analisi proteomica è descritto al fine di aumentare il livello di rilevamento e la gamma dinamica per proteine specifiche sinapsi 10,11.
Un prerequisito indispensabile l'utilizzo di spettrometria di massa ad alta risoluzione priva di etichetta per scopi quantitativi è un alto grado di somiglianza dei campioni di proteine. Come ci si aspetta cambiamenti piuttosto sfumature di composizione proteica sinaptica a verificarsi dopo aver appreso, un approccio libero-label sarà opportuno confrontare corrispondenti campioni di proteine ottenute da topi addestrati e ingenuo. In alternativa, le strategie di etichette condizione-specifica di proteine / peptidi utilizzando isotopi stabili (ad esempio TMT, iTRAQ, ICPL e SILAC), così come l'etichetta MS2 a base di qua gratisntification (BANDA) sono utili, ma sono più costosi rispetto all'approccio senza etichetta scelto o ha bisogno di hardware speciale spettrometria di massa.
Dal momento che le proiezioni di proteomica spesso producono insiemi di dati complessi, elaborazione bioinformatica è raccomandato per un'adeguata interpretazione dei dati. Ulteriori meta-analisi possono supportare una migliore comprensione dei potenziali meccanismi molecolari alla base cambiamenti di paradigma legati e l'individuazione di processi cellulari fondamentali coinvolti e vie di segnalazione. metodologie appropriate sono anche descritte di seguito.
Protocollo
Tutte le procedure, tra cui soggetti animali sono stati eseguiti in conformità con le norme della legge federale tedesco, i rispettivi regolamenti UE e le linee guida NIH, e sono stati approvati dal comitato etico della Landesverwaltungsamt Sachsen / Anhalt (42502-2-1102 IFN).
1. Apprendimento uditiva
- Apprendimento discriminazione uditiva nella casella di shuttle (FMTD paradigma) Nota: indossare sempre guanti durante la manipolazione i topi.
- topi Casa C57Bl6 / J in gruppi di tre o quattro, con libero accesso a pellet cibo e l'acqua del rubinetto in gabbie in policarbonato trasparente. Mantenere una luce 12 ore: ciclo scuro nella struttura animali. Se gli animali sono ricevuti da un altro laboratorio o da una società di consentire almeno una settimana di acclimatazione e sistemazione.
- Eseguire una sessione di formazione casella navetta al giorno.
- Prendete il mouse dalla sua gabbia casa nella struttura animale e metterlo in una scatola di navetta poco illuminato all'interno di una camera di prova di suono.
- Utilizzare un programma di apprendimento completamente controllato da computer per l'apprendimento discriminazione uditiva. Inizia con un periodo di adattamento di 3 minuti di silenzio, e quindi avviare la sessione di allenamento.
- Utilizzare sequenze del tono ascendente (4-8 kHz, CS +) come il Go-Go di stimolo durante-prove: L'animale deve attraversare l'ostacolo entro 6 secondi di presentazione tono (risposta corretta, ha colpito). Punire una miss da un lieve piede-shock di 50 - 300 μA, distribuito mediante il pavimento griglia della scatola navetta.
- Utilizzare sequenze del tono discendente (8 - 4 kHz, CS-) come la n-Go-stimolo durante no-go-prove: L'animale deve rimanere nel compartimento corrente della casella shuttle durante il 6 sec di presentazione tono. Punire un falso allarme da un lieve piede-shock di 50 - 300 μA, distribuito mediante il pavimento griglia della scatola navetta.
- Utilizzare gli intervalli tra le prove su 20 5 sec.
- Eseguire 30 Go-prove e 30 no-go-prove per sessione in un ordine pseudo-randomizzato, in modo che uno sefissione si compone di 60 prove e dura circa 25 min.
- Mettere l'animale addestrato di nuovo nella sua gabbia casa nel stabulario.
- cervello dissezione
- Euthanize l'animale al punto di tempo desiderato dopo un numero desiderato di sessioni di formazione mediante dislocazione cervicale (ad esempio 24 ore dopo il completamento della prima sessione). Decapitare l'animale.
- Sezionare rapidamente il cervello attraverso le seguenti fasi: Tagliare prima la pelle e poi il cranio con le forbici dritto lungo la sagittalis Sutura. Eliminare completamente le parti dell'osso che copre il tessuto cerebrale con pinze forti. Estrarre il cervello con una spatola.
- Per la dissezione, posizionare il cervello su una capsula di Petri pieno di ghiaccio. Sezionare la corteccia uditiva, corteccia frontale, lo striato e l'ippocampo allo stereomicroscopio con un bisturi e un ago.
- Localizzare la corteccia uditiva utilizzando punti di riferimento visivi sul cervello surface come i vasi sanguigni e la forma della superficie (Bregma -2.06 a -3.4, dimensioni rostrocaudale 2 mm dorsoventrale 1,3 mm) e bilateralmente sezionare come un blocco di tessuto rettangolare con lo spessore della corteccia.
- Sezionare la corteccia frontale come una fetta cerebrale tra Bregma 3.56 e 1.54 con il Opticum chiasma come punto di riferimento e di esclusione dal tessuto bulbus olfactorius.
- Sezionare lo striato come una fetta cerebrale tra Bregma 1,54 e 0,5 e rimuovere con cautela il tessuto corticale.
- Sezionare l'ippocampo fissando il cervello con l'ago attraverso il cervelletto e srotolare la corteccia partendo dal lobo occipitale.
- Shock-freeze sezionato campioni di cervello in azoto liquido e conservare a -80 ° C.
2. Preparazione di Sinaptosomi o in alternativa un Postsynaptic densità (PSD) Frazione -enriched
NOTA: Durante tutte le procedure, tenere i campioni e tamponi a 0-4 ° C.Buffer contiene appena diluito cocktail di inibitori della proteasi per impedire la degradazione proteolitica delle proteine. Se la fosforilazione della proteina è anche studiato, cocktail di inibitori della fosfatasi devono essere aggiunti. Tutti g-valori indicati sono dati come g (media) durante l'intero protocollo.
- Preparazione di una frazione di membrana grezzo (figura 3A)
- Trasferimento sezionato tessuto cerebrale in un recipiente omogeneizzazione contenente 1 ml ghiacciata tampone A (5 mm HEPES, 320 mm di saccarosio, pH 7.4) e omogeneizzare il tessuto a 900 rpm con 12 colpi.
- Centrifugare i campioni a 1.000 xg per 10 min. Mantenere il surnatante.
- Re-omogeneizzare pellets alle stesse condizioni nello stesso volume di tampone di omogeneizzazione come prima e centrifugare i campioni di nuovo a 1000 xg per 10 min. Combinare surnatanti corrispondenti. Eliminare il P1 pellet, che contengono principalmente nuclei e detriti cellulari.
- Spin i supernatanti combinati per 20 minuti a 12.000 x g. Superna Scartareabi- o l'uso di ulteriore frazionamento 11.
- pellet Risospendere nello stesso volume di tampone di omogeneizzazione come prima usando l'omogeneizzatore con 6 colpi a 900 rpm e centrifuga a 12.000 xg per 20 min. surnatanti scartare. I pellet P2 rappresentano le frazioni di membrana greggio.
- Purificazione di sinaptosomi da frazioni di membrana cerebrali grezzo (figura 3A)
NOTA: frazioni di membrana del cervello greggio possono essere separati in mielina, membrane di luce, sinaptosomi e mitocondri che utilizzano il saccarosio densità passo gradiente di ultracentrifugazione. Per questo pH 5 mM Tris / HCl sono necessari 8,1 tamponi contenenti saccarosio sia a 0,32 M, 1,0 M o 1.2 M concentrazione.- Mentre si esegue la centrifugazione per produrre le frazioni P2, preparare gradienti di saccarosio passo nei tubi ultracentrifuga. Inizia con 2,5 ml di 1,0 M tampone saccarosio e sottolivello con 1,5 ml 1,2 M tampone saccarosio utilizzando una pipetta Pasteur di vetro.
- Re-omogeneizzare frazioni P2in 0,5 ml di tampone 0,32 M di saccarosio manualmente con 6 colpi e il carico sulla parte superiore del gradiente.
- Spin a 85.000 xg per 2 ore in un'ultracentrifuga utilizzando un rotore oscillante.
- Eliminare il strato superiore 0.32 M saccarosio compreso il materiale all'interfaccia al buffer 1.0 M saccarosio (mielina, membrane di luce). Raccogliere sinaptosomi all'interfaccia tampone saccarosio 1.0 / 1.2 M. Il pellet sul fondo della provetta contiene mitocondri.
- Aggiungere tampone di 0,32 M di saccarosio per la frazione sinaptosomi in rapporto 1: 1, mescolare con cura e far girare a 150.000 xg per 1 ora. Sinaptosomi sono in pellet e possono essere risospese in tampone necessario per l'ulteriore elaborazione.
- Preparazione di una frazione PSD arricchita (Figura 3B)
- Omogeneizzare ogni area specifica del cervello da un singolo animale in 100 microlitri tampone di estrazione (5 mM Tris / HCl pH 8,1, 0,5% Triton X-100) in una provetta ultracentrifuga microlitri 200 con (politetrafluoroetilene) pestello PTFEa 2.000 rpm con 12 colpi.
- Aggiungere 100 microlitri di tampone di estrazione, mescolare e incubare per 1 ora a 4 ° C. Spin giù a 100.000 xg per 1 ora e raccogliere con cautela il surnatante S1 con una pipetta 200 ml.
- Re-omogeneizzare pellet P1 nella stessa provetta con 100 microlitri tampone di estrazione di nuovo con un pestello in PTFE a 2.000 rpm con 12 colpi.
- Aggiungere 100 microlitri tampone di estrazione e mescolare bene con una pipetta e far girare a 100.000 xg per 1 ora.
- Unire la S2 surnatante con S1 alla frazione proteica solubile. Questa frazione contiene proteine citosoliche, 0,5% Triton X-100 proteine di membrana solubile e molecole della matrice extracellulare.
- Risospendere il pellet rimanente 50 microlitri 5 mM Tris / HCl pH 8.1. Questa frazione contiene PSD, membrane detergente resistente, elementi del citoscheletro insolubili, mitocondri e detriti cellulari tra cui nuclei. E 'arricchita in PSD che costituiscono il nucleo di strutture post-sinaptici, ma anche parti importanti del presinapticocytomatrix alla zona attiva. Il fattore di arricchimento del PSD è di circa 4 e l'arricchimento dei componenti PSD è stata dimostrata in precedenza. 12
3. Preparazione del campione per la spettrometria di massa
- Lisi e la normalizzazione del campione
NOTA: la normalizzazione del campione per quanto riguarda la concentrazione di proteine è un passo molto importante per raggiungere finalmente dati quantitativi affidabili anche per deboli cambiamenti di espressione delle proteine sinaptiche.- Sciogliere sinaptosomi o preparati PSD-arricchite di ciascuna zona del cervello di un animale in 20 - 50 microlitri (dipendente dalla quantità totale di materiale: per corteccia uditiva con 5 - utilizzo tessuto 15 mg 20 ml) di 8 M urea e incubare su ghiaccio per 1 hr in un bagno a ultrasuoni.
- Per in-gel digerire sciogliere sinaptosomi direttamente nel buffer SDS-campione. di calcolare con attenzione l'importo caricato al fine di evitare il sovraccarico del gel. Si consideri che in questo caso, l'elevato abbondante Protei scaffoldns verranno persi durante l'elettroforesi su gel e in-gel digerire.
- Diluire con 1% di un detergente rimovibile per assicurare una concentrazione finale di 2 M urea. Evitare qualsiasi temperatura superiore a 30 ° C per evitare carbamilazione proteine.
- Eseguire SDS-PAGE con un'aliquota (ad esempio 10 ml) del campione secondo le procedure standard 13,14.
- Colorare il gel con Coomassie Blu secondo il protocollo del produttore. La procedura combina la fase di fissaggio e colorazione con metanolo e acido acetico.
- Determinare la densità ottica di ogni campione per l'intera corsia con uno scanner gel calibrato in trasmissione e calcolare la quantità di proteine relativo.
- Normalizzare i campioni in base a questi calcoli.
- Dividi ogni campione in due parti distinte. Utilizzare un terzo per l'in-gel digest e due terzi per l'in-soluzione di analisi.
- Sciogliere sinaptosomi o preparati PSD-arricchite di ciascuna zona del cervello di un animale in 20 - 50 microlitri (dipendente dalla quantità totale di materiale: per corteccia uditiva con 5 - utilizzo tessuto 15 mg 20 ml) di 8 M urea e incubare su ghiaccio per 1 hr in un bagno a ultrasuoni.
- In-gel digerire
- separazione Gel
- Eseguire una seconda SDS-PAGE utilizzando i campioni di concentrazione aggiustata. Macchia e quantificare i gel per la seconda volta per verificare la qualità di normalizzazione.
- Tagliare ogni corsia di un campione all'interno del gel in diversi settori (8 / corsia) ma escludere l'intervallo di peso molecolare superiore a 170 kDa. Trasferire i pezzi di gel in tubi separati.
- Tagliare le aree in pezzi più piccoli (circa. 1 x 1 mm) con un bisturi per facilitare in gel di efficacia digestione.
- Digest 15
- Lavare i pezzi di gel più volte (a seconda intensità di colorazione) per 10 min con 50 - 150 ml di un tampone costituito da 50% acetonitrile (ACN) e 50 mM di carbonato acido di ammonio (NH 4 HCO 3).
- Rimuovere surnatanti. Coprire i pezzi di gel con ACN e incubare a 20 ° C fino a pezzi di gel diventano bianche e si restringono.
- Rimuovere la ACN e reidratare i pezzi di gel per 5min con 50 ml di 0,1 M NH 4 HCO 3. Aggiungere lo stesso volume di ACN e incubare per altri 15 minuti a 37 ° C.
- Rimuovere ed eliminare completamente il liquido. Essiccare i pezzi di gel in una centrifuga a vuoto.
- Reidratare pezzi di gel in 50 ml di NH 4 HCO 3 contenente 10 mM ditiotreitolo (DTT) e campioni di calore per 45 min a 56 ° C per ridurre residui di cisteina.
- Rimuovere supernatanti e aggiungere 50 ml di NH 4 HCO 3 contenente 55 mm iodoacetamide (IAA) per 30 minuti al buio per carbamidomethylate cisteine ridotte.
- Rimuovere ed eliminare tutto il liquido sopra i pezzi di gel e lavarli due volte con 50 ml di NH 4 HCO 3 e ACN (1: 1) per 10 minuti per rimuovere ogni residuo IAA. campioni secchi in una centrifuga a vuoto.
- Per la digestione limitata di proteine aggiungere 25 mM NH 4 HCO 3 contenente 12,5 ng / ml di tripsina. Il volume richiesto dipende dalle dimensioni e la quantità di gel pieces. Incubare per pochi minuti e verificare se il buffer è assorbito. Aggiungere più di buffer, se necessario, pezzi di gel devono essere completamente coperti. Incubare a 37 ° C durante la notte (min. 12 ore).
- estrazione peptide
- Pezzi di gel di sovrapposizione con 10 - 20 ml di 25 mM NH 4 HCO 3 e aggiungere lo stesso volume di ACN. Incubare per 10 minuti in ghiaccio con bagno a ultrasuoni. Successivamente rimuovere e raccogliere surnatanti che contengono la maggior parte dei peptidi generati.
- Aggiungere 100 ml di tampone di estrazione contenente il 30% / 0,1% di acido trifluoroacetico ACN (TFA) per i pezzi di gel. Ripetere incubazione in un bagno ad ultrasuoni e raccogliere attentamente questo surnatante.
- Ripetere le ultime fasi di estrazione aumentando la concentrazione ACN al 50%. Dopo 10 min di bagno ad ultrasuoni spin down e raccogliere surnatanti.
- Combinare tutti tre surnatanti corrispondenti delle fasi di estrazione e asciugarle in una centrifuga a vuoto. Nota chea seguito della separazione gel 8 aree per corsia / campione vengono combinati per un campione di nuovo in questo passaggio.
- separazione Gel
- In-soluzione digest
- Digerire
- Utilizzare l'importo calcolato (ad esempio 100 ml di lisato 150 microlitri, dipende dalla quantità di materiale e il volume richiesto per risospensione di un campione da una zona specifica del cervello) di campioni normalizzati per ottenere materiale di partenza sufficiente per almeno tre repliche tecnici al eseguire la spettrometria di massa priva di etichetta.
- Aggiungere 2 mM DTT in 25 mM NH 4 HCO 3 e Agitare delicatamente il campione. Ridurre i campioni per 45 min a 20 ° C.
- Aggiungere 10 mM IAA di carbamidomethylate i residui di cisteina. Mescolare e incubare per 30 minuti al buio a 20 ° C.
- Infine, aggiungere 1 ml di una soluzione di tripsina magazzino (1 mg / ml tripsina a 25 mm acido acetico) e incubare a 20 ° C per 12 ore.
- Estrazione in fase solida (SPE) -Purification
- Per rimuovere il detergente cleavable acido, regolare i campioni ad una concentrazione finale di 1% TFA e incubare per 1 ora a 20 ° C.
- Centrifugare i campioni a 16.000 xg per 10 minuti e con attenzione raccogliere surnatanti.
- Porre la colonna SPE in un rack ed equilibrare la matrice con 2 ml di metanolo. Lavare due volte con 2 ml di 0,1% TFA in acqua (tampone B).
- Aggiungere 2 ml di tampone B e caricare il campione. Lavare altre tre volte.
- Eluire i peptidi aggiungendo 200 microlitri 70% ACN / 0.1% TFA. Ripetere questo passaggio.
- Piscina sia eluati e asciugarli giù in una centrifuga a vuoto.
- Digerire
- Fosfo-peptide-arricchimento da TiO 2 cromatografia 16
- Sciogliere peptidi prodotti da in-gel o in soluzione digerire in 150 ml di 80% ACN / 2.5% TFA (tampone C) ed equilibrare ~ 2 mg di TiO 2 perline in 50ml di tampone C.
- Aggiungere perline al campione e incubare in un dispositivo rotante per 1 ora a 20 ° C. In seguito, lo spin perline giù (16.000 xg, 1 min) e raccogliere surnatanti.
- Lavare le perline tre volte con 100 microlitri di tampone C miscelando e centrifugazione dopo 5 min delicatamente. Raccogliere surnatanti. Ripetere questa operazione per tre volte con 100 ml di 80% ACN / 0.1% TFA seguita da tre lavaggi con 100 ml di 0,1% TFA (senza ACN), rispettivamente.
- Unire tutti e dieci i supernatanti, asciugarli in una centrifuga a vuoto e gestirli come frazione fosfo-peptide-impoverito per ulteriore purificazione secondo passo 3.5.
- Eluire i fosfo-peptidi legati con 20 ml di 400 mM NH 4 OH / 30% ACN dalle perline. Ripetere questa operazione tre volte e raccogliere tutti i surnatanti dopo la filatura giù le perline.
- Combinare gli eluati della a-gel digerire e della soluzione in-digest di un campione e gestirli come fosfo-pfrazione eptide arricchita. Asciugarli in una centrifuga sotto vuoto ad un volume finale di 4 - 8 microlitri.
- Concentrare e dissalazione delle frazioni fosfo-peptide-impoverito da micro-SPE
- Sciogliere i peptidi essiccate in 20 ml di 0,1% TFA.
- Equilibrare il fisso C 18 -matrix disegnando 20 l ACN nella punta. Lavare la matrice disegnando 0,1% TFA in acqua nella punta. Ripetere il processo per tre volte.
- Lentamente caricare campione acidificato nella punta (ripetere l'operazione tre volte).
- Lavare i tempi di C 18 -matrix tre con 20 l 0,1% TFA in acqua e scartare la soluzione di lavaggio.
- Eluire peptidi dalla punta della pipetta ripetutamente (3 volte) disegno 20 ml di 70% ACN / 0.1% TFA e raccogliere questa soluzione di eluizione in un tubo separato.
- Combinare gli eluati di un campione e asciugare in una centrifuga a vuoto.
4. analisi del proteoma
NOTA: Proteome analisi viene effettuata su un ibrido a doppia pressione trappola ionica lineare spettrometro di massa / Orbitrap dotato di HPLC ultra. L'HPLC è composto da un autocampionatore raffreddato con un sistema di iniezione 20 microlitri, una pompa binaria carico (campo di portata microlitri), una pompa di separazione binaria flusso nano, un riscaldatore colonna con due micro valvole e un sistema di degasaggio commutazione. I campioni vengono dapprima sottoposti ad una colonna di cattura (ad esempio 100 micron x 2 cm) ad una portata di 7 ml / min seguita dalla separazione su una colonna (ad esempio 75 micron x 25 cm) a 250 nl / min. L'uscita della colonna di separazione è direttamente accoppiato ad un rivestito Pico punta emettitore posizionate a un'interfaccia nano-spruzzo alla fonte spettrometro di massa con ionizzazione.
- Nano-cromatografia liquida e la spettrometria di massa tandem
- Sciogliere campioni peptidici in 12 microlitri 2% ACN / 0.1% TFA per almeno 30 min. Spin down per 15 secondi e trasferire 11 microlitri surnatante al campionatore automatico fiale (conica, ridotto diameter).
- Impostare un regime automatico per applicazione di esempio, separazione cromatografica e spettrometria di massa tandem a software di controllo (ad esempio, Xcalibur) come segue.
- Utilizzare la seguente per Temperatura: Autosampler: 5 ° C; forno a colonna: 45 ° C.
- Utilizzare la seguente per iniezione: Volume: 10 ml; Portata: 7 ml / min (2% ACN, 0,1% TFA); Tempo: 8 min; taratura della valvola: colonna trappola - rifiuti; acquisizione spettrometro di massa: off.
- Utilizzare la seguente per la separazione: Portata: 250 NL / min regolazione della valvola: trappola Colonna separazione; acquisizione spettrometro di massa: il.
0 min - 100 min: 2% ACN, 0,1% di acido formico - 40% ACN, 0,1% di acido formico
100 min - 105 min: 40% ACN, 0,1% di acido formico - 95% ACN, 0,1% di acido formico
105 min - 109 min: 95% ACN, 0,1% di acido formico
109 min - 120 min: 2% ACN, 0,1% di acido formico - Utilizzare le seguenti impostazioni per la spettrometria di massa: Full MS: FTMS; Risoluzione 60.000; m / z gamma 400 - 2.000; SIGNORINA/MS: lineare Iontrap; soglia minima di segnale 500; larghezza isolamento 2 Da; Tempo di esclusione dinamica impostazione 30 sec; ioni singolarmente-caricati sono esclusi dalla selezione; energia di collisione normalizzato viene impostata al 35%, e l'attivazione tempo di 10 msec.
NOTA: Una scansione completa MS è seguita da fino a 15 LTQ MS / MS corre usando collisione indotta dissociazione (CID) degli ioni peptidici più abbondantemente rilevati.
- Eseguire tre repliche tecnici per tutti i campioni.
- L'identificazione delle proteine e l'etichetta quantificazione gratuito
- Processo di massa dati grezzi spettrometria verso l'identificazione delle proteine e la quantificazione priva di etichetta utilizzando una suite commerciale di software (ad esempio, picchi Studio). In contrasto con la maggior parte degli altri pacchetti software proteoma questo particolare software utilizza un algoritmo di de novo -sequencing prima di allineamenti di database di proteine. Tuttavia, questo passaggio può essere facilmente sostituito da altri pacchetti software popolari.
- Usa essimpostazioni prefe- elencati nella tabella 2.
- Fosfo-proteomica
NOTA: l'acquisizione efficiente e affidabile fosfo-peptide richiede alcune modifiche essenziali della configurazione del flusso di lavoro proteomica.- Dopo l'arricchimento fosfo-peptide, campioni mai asciugare completamente. Tenere sempre campioni disciolti.
NOTA: Il legame fosfo-estere di threonines fosforilati o serines è molto fragile. Durante frammentazione collisione indotta nella trappola ionica questo si traduce in una perdita neutra di fosfato. Ciò impedisce ogni ulteriore frammentazione del peptide, che a sua volta è necessaria per l'identificazione. Permesso a banda larga-attivazione nella configurazione spettrometria di massa permette la frammentazione di fosfo-peptidi, anche dopo una perdita neutra del gruppo fosfato. Si esegue un risparmio di tempo "pseudo-MS 3". determinazione fosfo-site nei dati di MS / MS richiede una particolare verifica e valutazione e può essere effettuata da fosfori 3.0.
- Dopo l'arricchimento fosfo-peptide, campioni mai asciugare completamente. Tenere sempre campioni disciolti.
5. Bioinformatica - Meta-Analysis
NOTA: Prima di eseguire annotazione funzionale e analisi di rete, le liste di proteine devono essere pre-elaborato. In primo luogo unire le liste di proteine regolamentati e fosfo-peptidi per ogni regione del cervello separatamente. Poi rimuovere tutti i duplicati UniProt-ID per ogni frazione per evitare errori di interpretazione.
- Analisi di arricchimento singolare GeneCodis 17
- Aprire lo strumento web-based di GeneCodis (http://genecodis.cnb.csic.es)
- Selezionare "Mus musculus" come organismo e "GO processo biologico", come annotazione.
- Incollare un elenco di UniProt-ID di una certa frazione. Invia e attendere fino a quando viene eseguita l'analisi. Clicca su "Analisi arricchimento singolare del processo di andare biologico" e visualizzare i risultati.
- Ripetere il punto 5.1.3 per le altre tre frazioni.
- Per vedere tutte le duplicazioni e le intersezioni tra le liste di risultatoutilizzare un linguaggio di scripting come Perl o Python per filtrare i dati necessari. strumenti simili per l'analisi di arricchimento singolare sono DAVID (https://david.ncifcrf.gov/) e Cytoscape (http://www.cytoscape.org/) con i plugin BINGO (http://apps.cytoscape.org/ apps / bingo) e ClueGO (http://apps.cytoscape.org/apps/cluego).
- Generazione di un grafico basato forza di dati GeneCodis con Gephi (https://gephi.org/)
NOTA: I dati per i grafici deve essere fornito dall'utente, sia in un formato grafico (.gexf, .graphml, dot, .gv, .gml) o inserito manualmente.- Generare i nodi del grafo
- A mano: Open Gephi e clicca su "Laboratorio di dati". Crea nodi. Clicca su "nodi" a sinistra per passare alla tabella "Nodi". Clicca su "Add node". Immettere il nome del termine. Fai clic su "OK" / Premere Invio.
- Alternative: Salva GeneCodis risultano PC. Aprire il txt con un foglio di calcolo. Eliminare tutte le righe except da "Item_Details" (nomi termine). Modifica intestazione "Item_Details" per "definire". Salva foglio di calcolo come ".csv". Ora, in Gephi, cliccare su "Importa foglio di calcolo". Scegliere foglio dal browser di file di Gephi. Fare clic su "Avanti". Fai clic su "Fine".
- Collegamento nodi via bordi.
- Clicca su "bordi" a sinistra per passare alla tabella "bordi". Per ogni nodo (Term): cercare nomi di geni in altri termini. Se uno o più geni sono condivisi -> creare bordo.
- Clicca su "Aggiungi Edge". Selezionare "orientato". Selezionare la sorgente e il nodo di destinazione di elenchi a discesa. Fai clic su "OK" / Premere Invio. Se più di un gene è condivisa, inserire abbondanza in "peso" (tabella).
- Forza basa layout grafico.
- Aprire file di dati del grafico, impostare il tipo di grafico a "orientato", o utilizzare i dati inseriti manualmente, cliccare su "Panoramica" se non è già selected.
- Ridimensionare i nodi a seconda della abbondanza di interconnessioni. Clicca sulle statistiche, eseguire uno "grado medio" (bordi non ponderati) o "Avg. Degree Weighted" (bordi ponderati) sotto "Schema di rete". In "Aspetto", cliccare su "nodi", quindi sul pulsante Formato, successiva, scegliere "attributi" e impostare il parametro Attributi per "AVG. Laurea ponderato" o "grado medio". Fare clic su Applica.
- Infine: Selezionare "Forza Atlas" in "Layout" ed eseguire; cambiare "forza Repulsion", se i nodi sono in collisione.
- Esporta in immagine.
- funzione Screenshot: Cliccare su "Panoramica", il layout grafico cambiamento, spessore del bordo, dimensioni dell'etichetta e scaling con il menu nella parte inferiore della finestra "Grafico". Fare clic sul pulsante della fotocamera a sinistra, e salvare foto.
- funzione di esportazione di "Anteprima": Fare clic su "Anteprima". Modificare preset "default dritto". Cambia impostaziones secondo le preferenze scelti e clicca su "SVG / PDF / PNG" per esportare.
- Generare i nodi del grafo
Risultati
La Figura 1 riassume il flusso di lavoro completo di quantitative proteoma profilazione sinaptica delle regioni del cervello del mouse dopo aver appreso la discriminazione uditiva. Si inizia con la formazione degli animali in una scatola di navetta. Nell'esempio illustrato in figura 2, i topi hanno iniziato a mostrare forte discriminazione tono FM in allenamento 4 °, indicando apprendimento efficace. Gli animali vengono sacrificati in punti tempo selezionato per zona del cervello dissezione. L'arricchimento richiesto sinapsi può sia essere ottenuto mediante la preparazione di sinaptosomi o in alternativa la preparazione di una frazione arricchita PSD, entrambi descritti in dettaglio in figura 3. Il metodo PSD-arricchimento è stato sviluppato per importi tessuto basso, ad esempio 1 - 2 fettine di ippocampo dal cervello di ratto 12, 18. Richiede piccoli tubi, pestelli in PTFE montaggio di questi tubi, e un'unità di foratura laboratorio per alimentare il pestello.
Grazie alla particolare composizione proteica di sinaptosomi, si consiglia vivamente di eseguire la preparazione del campione in due modi diversi, ma complementari. Ponteggi dei PSD sono spesso molto elevato peso molecolare delle proteine che si verificano in alta stechiometria. In-soluzione di analisi è il modo migliore per estrarre in modo efficiente, ma può portare ad un sovracampionamento della miscela peptide generato. La in-gel digest eseguita dello stesso campione in parallelo può escludere tali proteine ad alto peso molecolare e favorire l'analisi delle proteine con medio e basso peso molecolare. Per un'analisi completa sono raccomandati entrambi i tipi di digest proteolitici.
Le diverse quantità di tessuti delle aree cerebrali indagate richiedono un aggiustamento del materiale richiesto miglior confronto. All'interno delle quattro aree cerebrali indagate corteccia uditiva è generalmente fatto limitandoo. Il materiale di tutte le altre aree del cervello deve essere attentamente regolata per l'importo della corteccia uditiva dopo la preparazione di sinaptosomi o frazioni PSD-arricchite (vedi 3.1.1.). pesi tipici di aree del cervello appena preparate da topi sono i seguenti: corteccia uditiva (AC): ~ 50 mg; ippocampo (HIP): ~ 90 mg; striato (STR): ~ 120 mg e corteccia frontale (FC): ~ 100 mg.
Il metodo PSD-arricchimento descritto nella sezione 2.3 ha permesso l'identificazione di circa 1500 proteine diverse e circa 250 differenti fosfo-peptidi per regione del cervello a livello di un singolo animale (Tabella 1). Analisi proteomica 24 ore dopo il primo allenamento ha rivelato che il 7,3% delle proteine identificate e il 5,8% delle fosfo-peptidi hanno mostrato significativa (p <0.05) variazioni quantitative nella loro espressione sinaptica rispetto ai controlli naïve (Tabella 1). Una tendenza evidente per regula giùzione di ponteggi sinaptiche può puntare a un riassetto pronunciato dell'architettura sinaptico durante le prime fasi di apprendimento FMTD. La stragrande maggioranza delle proteine regolate sono stati alterati in maniera specifica regione del cervello, mentre solo il 22% sono risultati essere regolato in due o più aree del cervello. Sei esempi selezionati sono mostrati in Figura 4.
La meta-analisi dei complessi risultati di IPA fornisce la prova per la particolare partecipazione / manipolazione dei seguenti percorsi canonici: "Clathrin-mediata endocitosi di segnalazione", "assonale Guidance Signaling", "Segnalazione di calcio", "RhoA Signaling", "Notch segnalazione "," Rimodellamento di epiteliale giunzioni aderenti "," glutammato Receptor Signaling "," GABA Receptor Signaling "," del recettore della dopamina segnalazione "e" Synaptic lungo termine potenziamento ".
Analisi singolo arricchimento rivelato significativi processi biologici sovrarappresentati nella corteccia frontale in materia di trasporto di proteine, adesione cellulare, la fosforilazione, endocitosi, il trasporto delle vescicole-mediata, lo sviluppo e forebrain axonogenesis (Figura 5). Nei processi biologici uditiva corteccia compreso il trasporto di ioni, di traduzione, di trasporto mRNA, proteine di trasporto e l'apprendimento sono stati evidenti. L'analisi della frazione proteica dell'ippocampo rileva in modo significativo i processi arricchiti connesse al trasporto di ioni, ciclo cellulare, la traduzione, la fosforilazione e lo sviluppo del sistema nervoso. Nello striato, sovrarappresentati processi biologici, compreso il trasporto di mRNA, il trasporto delle vescicole-mediata, axonogenesis, proteolisi, il trasporto di proteine e endocitosi sono stati trovati.
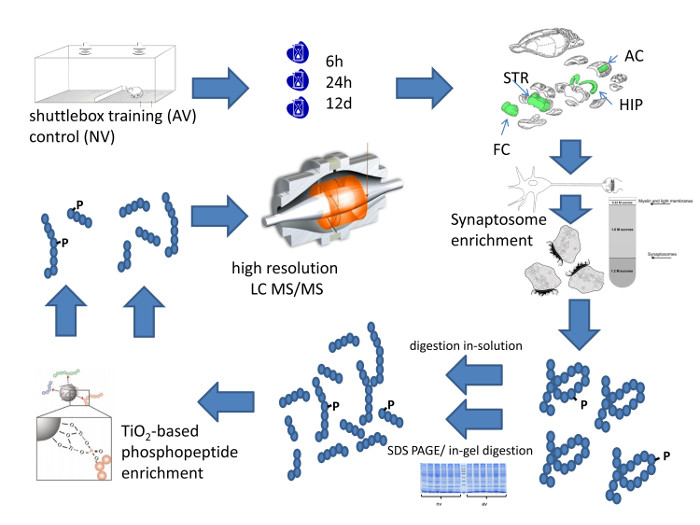
Figura 1: sistematica Workflow dell'approccio metodologico. Questa figura riassume schematicamente il flusso di lavoro di alta risoluzione profiling quantitativa della specifica composizione proteica sinaptica zona del cervello. Clicca qui per vedere una versione più grande di questa figura.

Figura 2: Esempio di prestazioni di topi nel tono FM Discrimination Task. Animali mostrano un tasso crescente di risultati (curva blu) e un tasso decrescente di falsi allarmi (curva nera) nel corso di sessioni di formazione. discriminazione significativo si verifica dalla quarta sessione. Le barre di errore sono forniti come SEM. Clicca qui per visualizzare un larVersione ger di questa figura.

Figura 3: Preparazione del sinaptosomi e la Frazione PSD arricchito. A: Preparazione sinaptosomi. B: Preparazione frazione PSD arricchita. Entrambe le figure spiegano il flusso di lavoro dettagliato di preparazione di sinaptosomi o frazioni in alternativa PSD-arricchiti da tessuti cerebrali. Clicca qui per vedere una versione più grande di questa figura.
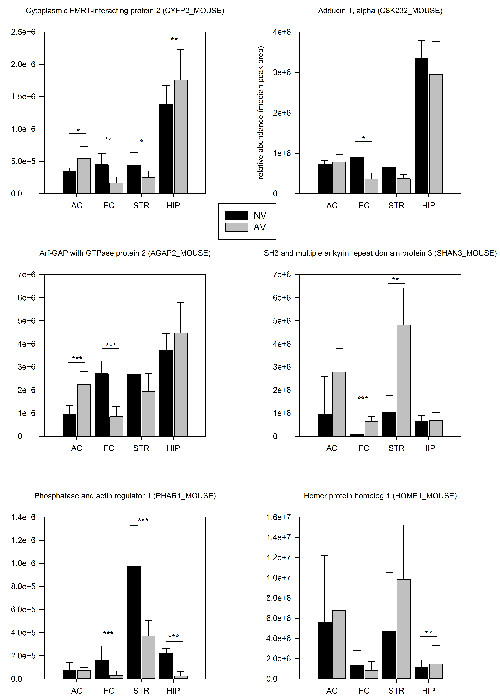
Figura 4: Selected risultati quantitativi proteomica. Le abbondanze relative di sinaptiche proteine selezionati vengono confrontati tra Tra topiined sul compito FMTD (AV, n = 6) e topi di controllo naive (NV, n = 6) 24 ore dopo il primo allenamento. I valori di abbondanza sono stati calcolati come media delle aree dei picchi dei tre più intense peptidi di una proteina. Proteine con cambiamenti significativi abbondanza (AV / NV; t-test) sono contrassegnati all'interno delle piazzole: * p <0.05, ** p <0.01, *** p <0.005. Le barre di errore sono forniti come SD. Clicca qui per vedere una versione più grande di questa figura.

Figura 5: Visualizzazione di percorsi biologici per corteccia frontale da GeneCodis / Gephi. Solo termini significativi del database Gene Ontology (GO) (http://geneontology.org) correlate a "processo biologico" con un numero minimo di proteine di tre sono mostrati qui. Nodi rappresentano termini GO, la dimensione del nodo, la larghezza della linea e il numero di connessioni di un certo nodo rappresentano il numero di proteine che condividono questo termine GO con altri nodi. A causa del metodo "Forza Atlas" di Gephi, nodi correlati si discostano molto attentamente insieme. Clicca qui per vedere una versione più grande di questa figura.
| regione del cervello | AC | FC | HIP | 000 "face =" Calibri "size =" 3 "> STR | Σ |
| proteine identificate | 1435 | 1758 | 1572 | 1507 | 6272 |
| proteine regolamentati (p <0.05) | 59 | 130 | 162 | 108 | face = "Calibri" size = "3"> 459 |
| ↑ AV / NV | 8 | 4 | 76 | 35 | 123 |
| ↓ AV / NV | 51 | 126 | 86 | 73 | 336 |
| phosphomoti identificato fs | 197 | 361 | 273 | 278 | 1109 |
| phosphomotifs regolamentati (p <0.05) | 8 | 22 | 21 | 14 | 65 |
| ↑ AV / NV | 4 | 00000 "face =" Calibri "size =" 3 "> 17 | 5 | 9 | 35 |
| ↓ AV / NV | 4 | 5 | 16 | 5 | 30 |
Tabella 1: Sintesi di un risultato proteomica. Questa tabella riassume un esperimento rappresentativo proteomica di topi addestrati (AV, n = 6) 24 ore dopo la prima sessione di formazione rispetto ai loro controlli naïve (NV, n = 6). Ilsomma di 459 proteine regolate include norme che si sovrappongono. 283 diversi regolamenti sono stati determinati come specifica del cervello. Nel dettaglio, 57 proteine sono regolati in due regioni cerebrali, 18 regolamenti di proteine sono state rilevate in tre regioni del cervello e solo 2 proteine sono regolate in tutte le quattro aree cerebrali indagate.
| tolleranze di errore | |
| massa precursore (trasformata di Fourier spettrometria di massa) | 10 ppm |
| massa frammento di litio (trappola ionica lineare) | 0.6 Da |
| Spaccature massime perse per peptide | 3 |
| modifiche fissi | |
| per in-gel-digerito campioni | Carbamidomethylation di cisteina |
| per i campioni in soluzione digerita | Methylthiolation di cisteina |
| modifiche variabili | Ossidazione di metionina |
| Deamidations di asparagina e / o glutammina | |
| Banca dati | UniProt / Sprot |
| Tassonomia | topo |
| Impostazioni di identificazione-accettazione statistiche | |
| de novo media fiducia locale (ALC) | > 50% |
| Peptide-falso tasso di scoperta (FDR, in base a est. Decoy-fusion) | <1% |
| Proteine significatività (-10logP, basato sul T-test modificato) | > 20 |
| peptidi unici / proteine | ≥ 1 |
| Impostazioni di quantificazione: | |
| Peptidi utilizzati per la quantificazione se: | |
| Peptide significatività (-10logP) | > 30 |
| Identificazione Peptide in | ≥ 50% dei campioni |
| la qualità del segnale peptide | > 1 |
| superficie media Peptide | > 1E5 |
| Peptide tolleranza tempo di ritenzione | <5 min |
| Normalizzazione | dalla corrente totale di ioni (TIC) |
Tabella 2: Impostazioni per la proteina di identificazione (fase 4.2.2).
Discussione
Lo studio presenta un flusso di lavoro metodologico ottimizzato per una accurata profilazione quantitativa di sinaptici cambiamenti di espressione delle proteine durante l'apprendimento e consolidamento della memoria in diverse aree cerebrali di topi. La configurazione offre l'opportunità di studiare l'espressione delle proteine a livello di un singolo animale nonostante l'applicazione richiesta di almeno tre repliche tecniche per campione per l'analisi di spettrometria di massa.
La metodologia tiene conto della particolare composizione proteica del pre e postsynapse costituito da proteine ad alto peso molecolare scaffold ma anche di proteine importanti mediatori recanti pesi molecolari medi o inferiori. Le digerisce in-soluzione di preparati sinaptosomiali determinano una generazione efficiente e, quindi, una sovra-rappresentazione di peptidi scaffold-derivato. Questo, a sua volta, può sopprimere l'analisi di piccole o inferiore proteine abbondanti. La preparazione suggerito di frazioni SDS-PAGE da unaliquota di ciascun campione in combinazione con una procedura di digestione in gel in parallelo facilita l'analisi di proteine medie e basse abbondanza e rappresenta un metodo complementare altamente raccomandato. Dopo l'applicazione spettrometria di massa separata di tutte le frazioni derivanti da un campione (ad esempio in soluzione di analisi, in-gel digeriscono, frazioni fosfo-arricchita combinati) i corrispondenti insiemi di dati MS / MS possono essere combinati e calcolati per l'identificazione delle proteine e quantificazione da picchi maggiori software o pacchetti software alternativi popolari.
In alternativa, l'applicazione individuale di frazioni in gel-digestione-derivato di un campione (trasformati separatamente gel-aree di una corsia di esempio) e frazioni generato del campione preparato in soluzione (ad esempio cromatografia a scambio ionico) alla spettrometria di massa può aumentare la profondità analitica. Tuttavia, questo flusso di lavoro esteso aumenta notevolmente il tempo necessario per LS-MS di acquisizione dati / MS. per generatioè richiesto n di una sequenza molecolare dettagliato di riarrangiamenti di proteine sinaptiche durante l'apprendimento e la formazione della memoria un corso a tempo determinato del profiling proteomica. Questo corso di tempo può iniziare immediatamente dopo o anche durante il primo allenamento e copre un arco di tempo a maglie strette fino a quando le prestazioni degli animali ha raggiunto il livello asintotico della curva di apprendimento dopo circa. 8 - 10 giorni di formazione (vedi figura 2 per i dettagli).
L'analisi delle variazioni di fosforilazione di proteine sinaptiche richiede una particolare attenzione ai tempi selezionati durante l'apprendimento FMTD. Sulle segnalazione cascate da un lato avvio riarrangiamenti di proteine sinaptiche noti per essere innescato da fosforilazioni proteine e dephosphorylations sono attesi a primissime fasi di addestramento di animali. D'altra parte, ci sono lunghi modifiche durata di più proteine sinaptiche fosforilate noti che regolano la connettività e l'assemblaggio entro i sarchitettura ynaptic 19, 20. Tali modifiche post-traduzionali sono attesi anche in momenti successivi di consolidamento della memoria.
I set di dati complessi generati da questo flusso di lavoro proteomica richiedono l'elaborazione bioinformatica per identificare i partecipanti percorsi molecolari e di molecole chiave. La meta-analisi mostra percorsi sovrarappresentati significativi, che svolgono un ruolo nei processi di apprendimento e memoria.
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
We wish to thank Yvonne Ducho and Kathrin Pohlmann for excellent technical assistance. This work was supported by the Deutsche Forschungsgemeinschaft (SFB 779) and by the State Saxony-Anhalt / European Regional Development Fund (ERDF) via the Center for Behavioral Brain Sciences (CBBS).
Materiali
| Name | Company | Catalog Number | Comments |
| 3 M Empore Solid Phase Extraction- Filter | 3M Bioanalytical Technologies | 4245SD | 7 mm/3 ml |
| Acclaim PepMap 100 | Dionex/Thermo Scientific | 164564 | 100 µm x 2 cm, C18 |
| Acclaim PepMap 100 | Dionex/Thermo Scientific | 164569 | 75 µm x 25 cm, C18 |
| Acetic acid | Carl Roth GmbH | 3738.1 | |
| Acetonitrile (ACN) | Carl Roth GmbH | AE70.2 | |
| Acrylamide (30%) | AppliChem | A0951 | |
| Ammonium hydrogen carbonate | Fluka | 9830 | |
| Ammonium hydroxide | Fluka | 44273 | |
| Ammonium persulfate (APS) | AppliChem | A2941 | |
| Biofuge pico | Heraeus GmbH | 75003280 | |
| Blue R-250 | SERVA Electrophoresis GmbH | 17525 | |
| Bromophenol Blue | Pharmacia Biotech | 17132901 | |
| C57BL/6J mice | Charles River | ||
| Cantharidin | Carl Roth GmbH | 3322.1 | |
| Centrifuge tubes for MLS-50 | Beckman Coulter | 344057 | |
| Centrifuge tubes for TLA 100.1 rotor | Beckman Coulter | 343776 | |
| Dithiothreitol (DTT) | AppliChem | A1101 | |
| Eppendorf 5417R centrifuge | VWR | 22636138 | |
| Eppendorf A-8-11 rotor | VWR | 5407000317 | |
| Formic acid | Fluka | 14265 | |
| GeneCodis | http://genecodis.cnb.csic.es/ | ||
| Gephi | https://gephi.org/ | ||
| Glycerol | AppliChem | A1123 | |
| Glycine | AppliChem | A1067 | |
| HALT Phosphatase Inhibitor Cocktail | Pierce /Thermo Scientific | 78420 | |
| HEPES Buffer solution | PAA Laboratories GmbH | S11-001 | |
| Homogenization vessel 2 ml | Sartorius AG | 854 2252 | |
| Hydrochloric acid | Sigma-Aldrich | H1758 | |
| Imidazole | Sigma-Aldrich | I2399 | |
| Ingenuity Pathway Analysis | Qiagen | ||
| Iodoacetamide (IAA) | Sigma-Aldrich | I1149 | |
| Laboratory drilling drive K-ControlTLC 4957 | Kaltenbach & Vogt GmbH | 182997 | |
| LTQ Tune Plus 2.7.0.1112 SP2 | Thermo Scientific | ||
| LTQ Orbitrap Velos Pro | Thermo Scientific | ||
| Macs-mix tube rotator | Miltenyi Biotech | 130-090-753 | |
| Magic Scan 4.71 | UMAX | ||
| Methanol | Carl Roth GmbH | AE71.2 | |
| MLS-50 rotor | Beckman Coulter | 367280 | |
| Optima MAX Ultracentrifuge | Beckman Coulter | 364300 | |
| PageRuler Prestained Protein Ladder | Thermo Scientific | 26616 | |
| PEAKS 7.5 | Bioinformatic Solutions | ||
| Phosphatase Inhibitor Cocktail 3 | Sigma-Aldrich | P0044 | |
| PhosphoRS 3.1 | IMP/IMBA/GMI | ||
| PhosSTOP | Roche | 4906845001 | |
| Plunger/pestle made of PTFE | Sartorius AG | 854 2651 | |
| PotterS homogenizer | Sartorius AG | 853 3024 | |
| Protease Inhibitor complete mini | Roche | 4693159001 | |
| Quantity One 4.5.1 | BioRad | ||
| RapiGest | Waters | 186002122 | |
| Shuttle box | Coulbourne Instruments | ||
| Sodium dodecylsulfate (SDS) | AppliChem | A1112 | |
| Sodium molybdate | Carl Roth GmbH | 274.2 | |
| Sodium tartrate dihydrate | Sigma-Aldrich | 228729 | |
| SONOREX RK 156 Ultrasonic Bath | BANDELIN electronic GmbH & Co. KG | 305 | |
| Soundproof chamber | Industrial Acoustics Company | ||
| Sucrose | Carl Roth GmbH | 4621.2 | |
| Tetramethyl ethylene -1,2-diamine (TEMED) | Sigma-Aldrich | T9281 | |
| Thermomixer basic | CallMedia | 111000 | |
| Titansphere TiO 5µm | GL Sciences Inc. Japan | 502075000 | |
| TLA 100.1 rotor | Beckman Coulter | 343840 | |
| Trifluoro acetic acid (TFA) | Sigma-Aldrich | T6508 | |
| Tris ( hydroxymethyl) aminomethane (TRIS) | AppliChem | A1086 | |
| Triton X-100 | Sigma-Aldrich | T8532 | |
| Trypsin Gold | Promega | V5280 | |
| Ultimate 3000 Ultra HPLC | Dionex/Thermo Scientific | ||
| Ultracentrifuge tube | Beckman Coulter | 343776 | |
| Unijet II Refrigerated Aspirator | Uniequip Laborgeräte- und Vertriebs GmbH | ||
| UNIVAPO 100 H Concentrator Centrifuge | Uniequip Laborgeräte- und Vertriebs GmbH | ||
| Urea | AppliChem | A1049 | |
| Water (high quality purifed) | Resistivity: > 18.2 MΩ*cm at 25 °C Pyrogens: < 0.02 EU/ml TOC: < 10 ppb | ||
| Xcalibur 3.0.63 | Thermo Scientific | ||
| ZipTipC18 Pipette Tips | MILLIPORE | ZTC18S960 |
Riferimenti
- Lamprecht, R., LeDoux, J. Structural plasticity and memory. Nat Rev Neurosci. 5 (1), 45-54 (2004).
- Bingol, B., Schuman, E. M. Synaptic protein degradation by the ubiquitin proteasome system. Curr Opin Neurobiol. 15 (5), 536-541 (2005).
- Richter, J. D., Klann, E. Making synaptic plasticity and memory last: mechanisms of translational regulation. Genes Dev. 23 (1), 1-11 (2009).
- Rosenberg, T., et al. The roles of protein expression in synaptic plasticity and memory consolidation. Front Mol Neurosci. 7, 86 (2014).
- Scheich, H., et al. Behavioral semantics of learning and crossmodal processing in auditory cortex: the semantic processor concept. Hear Res. 271 (1-2), 3-15 (2011).
- Kähne, T., et al. Synaptic proteome changes in mouse brain regions upon auditory discrimination learning. Proteomics. 12 (15-16), 2433-2444 (2012).
- Reichenbach, N., et al. Differential effects of dopamine signalling on long-term memory formation and consolidation in rodent brain. Proteome Sci. 13, 13 (2015).
- Kähne, T., et al. Proteome rearrangements after auditory learning: high-resolution profiling of synapse-enriched protein fractions from mouse brain. J Neurochem. , (2016).
- Li, K., et al. Organelle proteomics of rat synaptic proteins: correlation-profiling by isotope-coded affinity tagging in conjunction with liquid chromatography-tandem mass spectrometry to reveal post-synaptic density specific proteins. J Proteome Res. 4 (3), 725-733 (2005).
- Carlin, R. K., Grab, D. J., Cohen, R. S., Siekevitz, P. Isolation and characterization of postsynaptic densities from various brain regions: enrichment of different types of postsynaptic densities. J Cell Biol. 86 (3), 831-845 (1980).
- Smalla, K. H., Klemmer, P., Wyneken, U., Dermietzel, R. . The Cytoskeleton - Imaging, Isolation, and Interaction. , 265-282 (2012).
- Smalla, K. H., et al. The synaptic glycoprotein neuroplastin is involved in long-term potentiation at hippocampal CA1 synapses. Proc Natl Acad Sci U S A. 97 (8), 4327-4332 (2000).
- Shapiro, A. L., Vinuela, E., Maizel, J. V. Molecular weight estimation of polypeptide chains by electrophoresis in SDS-polyacrylamide gels. Biochem Biophys Res Commun. 28 (5), 815-820 (1967).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Shevchenko, A., Wilm, M., Vorm, O., Mann, M. Mass spectrometric sequencing of proteins silver-stained polyacrylamide gels. Anal Chem. 68 (5), 850-858 (1996).
- Thingholm, T. E., Larsen, M. R., Ingrell, C. R., Kassem, M., Jensen, O. N. TiO(2)-based phosphoproteomic analysis of the plasma membrane and the effects of phosphatase inhibitor treatment. J Proteome Res. 7 (8), 3304-3313 (2008).
- Carmona-Saez, P., Chagoyen, M., Tirado, F., Carazo, J. M., Pascual-Montano, A. GENECODIS: a web-based tool for finding significant concurrent annotations in gene lists. Genome Biol. 8 (1), 3 (2007).
- Bonn, S., Seeburg, P. H., Schwarz, M. K. Combinatorial expression of alpha- and gamma-protocadherins alters their presenilin-dependent processing. Mol Cell Biol. 27 (11), 4121-4132 (2007).
- Tweedie-Cullen, R. Y., Reck, J. M., Mansuy, I. M. Comprehensive mapping of post-translational modifications on synaptic, nuclear, and histone proteins in the adult mouse brain. J Proteome Res. 8 (11), 4966-4982 (2009).
- Li, L., et al. The differential hippocampal phosphoproteome of Apodemus sylvaticus paralleling spatial memory retrieval in the Barnes maze. Behav Brain Res. 264, 126-134 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon