È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Il disegno sperimentale per Microdissezione laser RNA-Seq: Lezioni da una analisi dello sviluppo di mais Leaf
In questo articolo
Riepilogo
Many developmentally important genes have cell- or tissue-specific expression patterns. This paper describes LM RNA-seq experiments to identify genes that are differentially expressed at the maize leaf blade-sheath boundary and in lg1-R mutants compared to wild-type. The experimental considerations discussed here apply to transcriptomic analyses of other developmental phenomena.
Abstract
I geni con ruoli importanti nello sviluppo spesso hanno modelli di espressione spazialmente e / o temporalmente limitati. Spesso questi trascritti genici non vengono rilevate o non sono identificati come differenzialmente espressi (DE) in analisi trascrittomica di organi vegetali integrali. Microdissezione laser RNA-Seq (LM RNA-Seq) è un potente strumento per identificare i geni che sono DE in specifici ambiti di sviluppo. Tuttavia, la scelta dei domini cellulari per microdissect e confrontare, e la precisione dei microdissections sono cruciali per il successo degli esperimenti. Qui, due esempi illustrano considerazioni di progettazione per gli esperimenti trascrittomica; un LM analisi RNA-seq per identificare geni che sono de lungo l'asse prossimale-distale foglia di mais, e un secondo esperimento di identificare geni che sono DE in liguleless1-R (lg1-R) mutanti rispetto al wild-type. Gli elementi chiave che hanno contribuito al successo di questi esperimenti sono stati dettagliati istologica e in SItu ibridazione analisi della regione da analizzare, la selezione della foglia primordi a stadi di sviluppo equivalenti, l'uso di punti di riferimento morfologici per selezionare le regioni per microdissezione e microdissezione dei domini misurato con precisione. Questo documento fornisce un protocollo dettagliato per l'analisi dei domini di sviluppo da LM RNA-Seq. I dati qui presentati illustrano come l'area selezionata per microdissezione influenzerà i risultati ottenuti.
Introduzione
La foglia mais è un modello ideale per studiare la formazione di campi di sviluppo durante morfogenesi, in quanto ha un confine netto tra la lama e guaina che è suscettibile di dissezione genetica (Figura 1A). Durante le prime fasi di sviluppo foglia, una banda lineare di celle più piccole, la band preligule (PLB), suddivide il primordio foglia in domini pre-blade e pre-guaina. A ligule frange-like e atri triangolari sviluppano dalla PLB (Figura 1A, C, D). schermi genetici hanno identificato mutazioni che inficiano il confine lama-guaina. Ad esempio, liguleless1 recessivo (LG1) mutazioni eliminare la ligula e orecchiette 1, 2, 3, 4 (Figura 1B). L'ibridazione in situ ha rivelato che la trascrizione LG1 si accumula al PLB e ligula emergente, che lo rende un ottimo indicatore per lo sviluppo ligula 5, 6 (Figura 1E).
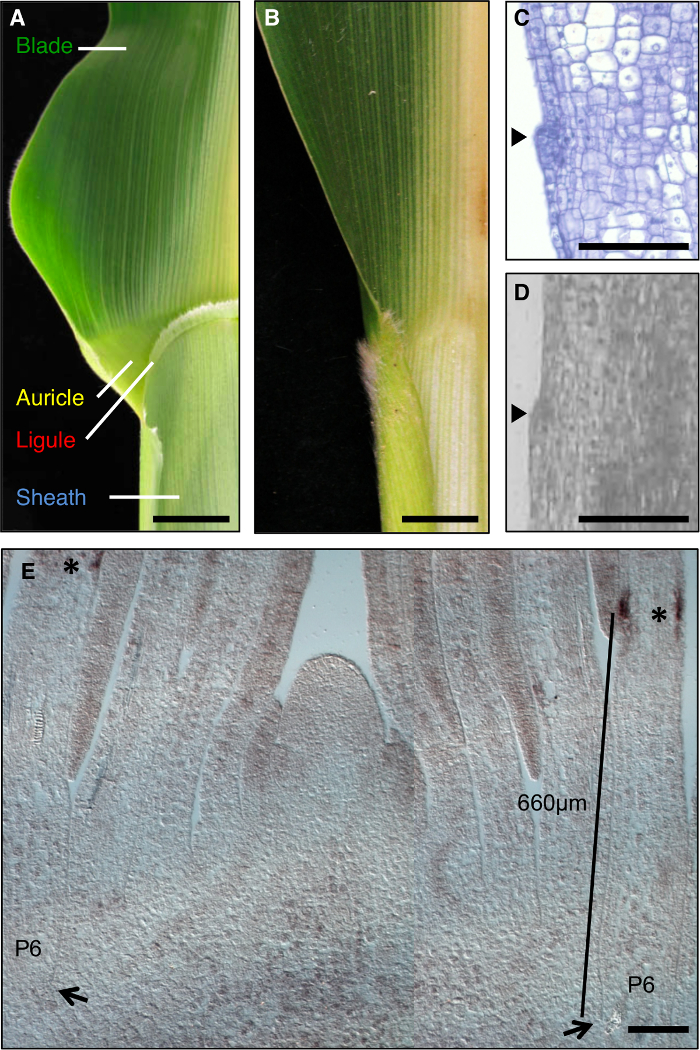
Figura 1: wild-type e foglie di mais liguleless1-R. (A) regione Blade-guaina confine del maturo wild-type foglio che mostra le strutture ligula e padiglione auricolare. (B) regione Blade-guaina confine del maturo liguleless1-R foglio che mostra l'assenza di strutture ligula e padiglione auricolare. Foglie in A e B sono stati tagliati a metà lungo la nervatura centrale. (C) Sezione longitudinale attraverso wild-type foglia primordio. Esempio è stato elaborato e colorati per l'analisi istologica. Il ligule avvio è evidente come un urto che sporge dal piano della foglia (freccia). (D) sez longitudinaleioni attraverso wild-type foglia primordio. Esempio è stato elaborato per LM come descritto nel testo. Arrowhead indica l'avvio ligula. (E) lg1 ibridazione in situ di ripresa apex sezione longitudinale laterale. Gli asterischi indicano l'accumulo di trascrizione LG1 al PLB del primordio foglia P6. Le frecce indicano base del P6 primordio. La barra indica misura dalla base del primordium al PLB. Barre di scala in A e B = 20 mm. Barre di scala nella CE = 100 micron. Questa cifra è stata modificata da riferimento 6 (Copyright dell'American Society of biologi). Clicca qui per vedere una versione più grande di questa figura.
In questo studio, LM RNA-Seq è stato impiegato per identificare una serie di geni che sono differenzialmente espressi (DE) alla lama fodero confine rispetto ad altre parti del primordium foglia e di ide i geni che sono ntify DE in LG1-R mutanti rispetto al wild-type fratelli. LM RNA-Seq è un metodo per quantificare l'accumulo di trascrizione in cellule specifiche o domini cellulari 7. sistemi LM combinano un laser e un microscopio con una fotocamera digitale. tessuto sezionato è montato su guide e vista attraverso il microscopio. Il software LM in genere include strumenti di disegno che consentono all'utente di delineare qualsiasi regione selezionata per microdissezione. I tagli laser lungo la linea, e il tessuto selezionato viene catapultato dal vetrino e in un tubo sospeso sopra la diapositiva. LM consente all'utente di microdissect domini precise, comprese strati cellulari specifici e anche le cellule singole 8, 9. RNA può quindi essere estratta dal tessuto microdissezione. Successivamente, il componente di RNA-Seq utilizza sequenziamento di prossima generazione per sequenziare librerie di cDNA generate dal estratto RNA 10,= "xref"> 11.
I principali vantaggi di LM RNA-Seq sono la capacità di quantificare l'accumulo di trascrizione in domini ben definiti e la capacità di profilare l'intero trascrittoma contemporaneamente 7. La tecnica è particolarmente adatto per sondare eventi precoci di sviluppo in cui la regione di interesse è spesso microscopica. Studi precedenti hanno utilizzato LM combinato con la tecnologia microarray per studiare i processi evolutivi nelle piante 9, 12, 13. RNA-Seq ha il vantaggio di quantificare trascritti attraverso un'ampia gamma dinamica, compresi i geni espressi basso, e le informazioni prima sequenza non è necessaria 10, 11. Inoltre, LM RNA-Seq ha il potenziale per evidenziare evolutivamente importanti geni che possono essere perse nelle schermate di mutagenesi a causa di ridondanza genetica, o per letalità della perdita-di-funzione di mutante.
Geni evolutivamente importanti, come sheath1 stretto (NS1) e a forma di coppa cotyledon2 (cuc2), hanno spesso specifici pattern di espressione di una sola o poche cellule 17, 18, 19, 20. Molti sono espressi solo durante le prime fasi di sviluppo e non nell'organo maturo. Quando interi organi o grandi domini vengono analizzate, queste trascrizioni specifici delle cellule sono diluiti e non possono essere rilevati nelle analisi più convenzionali. Permettendo analisi di domini ben definiti, LM RNA-Seq permette a questi geni tessuto-specifici per essere identificati e quantificati.
fattori cruciali per il successo degli esperimenti descritti qui erano una approfondita analisi istologica che ha guidato la selezione della fase di sviluppo appropriato e dominio per l'analisi, e precisa MeasureMent dei domini delle cellule dei tessuti per LM. Per assicurare che i domini equivalenti sono stati campionati per tutte le repliche, il tessuto è stato raccolto dalla foglia primordi allo stesso stadio di sviluppo ei domini microdissezione stati misurati rispetto a punti di riferimento morfologici come la ligule emergente (Figura 2). E 'noto che alcuni geni sono espressi in un gradiente dalla punta alla base della foglia. Misurando domini precisi, variazione dovuta al campionamento da posizioni diverse lungo l'asse prossimale-distale foglia è stato ridotto al minimo (figura 3A). Con microdissecting domini della stessa dimensione, variazione dovuta differenziale diluizione dei trascritti specifici delle cellule è stato ridotto (Figura 3B). Laterali sezioni longitudinali dell'apice riprese stati usati per tutte microdissections. Questi sono sezioni che sono perpendicolari all'asse nervatura-margine (Figura 4). Usando solo le sezioni che includono il SAM assicura che le regioni laterali equivalenti diprimordi fogliari vengono analizzati.
In campioni trattati e sezionate per LM, il primo segno morfologico escrescenza ligule è un urto sul lato adaxial causa di divisioni cellulari periclinal nell'epidermide adaxial (Figura 1D, Figura 2). È stato stabilito che la ligula emergente potrebbe essere identificate in modo attendibile a plastochron 7 primordi fogliari fase. Eravamo interessati a geni espressi in tutta la regione, tra cui la ligula ligula emergenti e le cellule immediatamente distali che formeranno il padiglione auricolare. Al fine di garantire che le selezioni tessuto equivalenti sono state effettuate, il bump ligula è stato utilizzato come punto di riferimento morfologica e un rettangolo 100 micron centrata su urto ligule stato selezionato per LM (Figura 2A, 2B). rettangoli equivalenti dimensioni di pre-lama e pre-guaina sono stati selezionati dalle stesse primordi fogliari.
Le analisi delle piante mutanti liguleless presentato un Challe diversoESN; LG1-R mutanti non formano un ligule, quindi questa caratteristica morfologica non può essere utilizzato per selezionare la regione per LM. Invece, il dominio di accumulo trascrizione lg1 in wild-type leaf primordia stata determinata, ed una regione che comprenda questo dominio è stato definito. Queste analisi preliminari sono state effettuate su piantine dalla stessa piantagione come sono stati utilizzati per l'analisi finale, dal lavoro precedente ha mostrato che la posizione del PLB varia a seconda delle condizioni di crescita. L'ibridazione in situ ha indicato che le trascrizioni LG1 si accumulano nel PLB di P6 foglia primordi (Figura 1E). Abbiamo scelto un dominio 400-900 micron dalla base della primordia foglia che comprendeva il dominio di espressione lg1 (rettangoli viola, Figura 2A) e catturato queste regioni equivalenti da wild-type e piante lg1-R. Per ridurre al minimo la variazione in background e di crescita genetiche condizioni quando si confrontano transcript accumulo in LG1-R e le piante wild-type, segreganti famiglie di mutanti e wild-type fratelli sono stati utilizzati.
Protocollo
NOTA: Fix tessuto per l'analisi istologica allo stesso tempo che il tessuto è fissato per LM. Esaminare sezioni colorate per caratteristiche morfologiche che guideranno successivamente LM. Quando si confrontano mutante wild-type, eseguire ibridazione in situ o immunolocalizzazione per definire il dominio in cui viene espresso il gene di interesse (in questo caso lg1).
1. Fissazione dei tessuti e la lavorazione
- Crescere appartamenti di piantine di mais a due settimane in condizioni standard 6.
- Sezionare apici sparare per le sezioni laterali (figura 4).
- Excise piantina appena sotto la linea del terreno.
- Utilizzando una lama di rasoio, rimuovere fette sottili dalla base del fusto (tagli 1, Figura 4A) finché un ovale culm circondata da uno o due foglie adulte è visibile (Figura 4B).
- Fare un altro taglio di circa 10 mm sopra la base (tagliare 2, figura 4A).Questo segmento 10 millimetri conterrà il SAM e giovani primordi fogliari.
- Ruotare il segmento di 10 mm, in modo che la base sia rivolto verso l'alto. Fare due tagli paralleli all'asse laterale in modo che una porzione di spessore del tessuto 2-3 mm si ottiene (tagli 3 e 4, Figura 4B). Eliminare le esterne due porzioni e mantenere la fetta centrale per la fissazione e inclusione.
NOTA: foglie esterne possono essere tagliati e scartati.
- Fissare il tessuto e il processo per l'incorporamento.
- Assicurarsi che tutti i materiali da utilizzare nei passaggi successivi sono RNasi gratuito. Trattare soluzioni con dietilpirocarbonato (DEPC) (1 ml DEPC per litro di soluzione. Incubare per una notte con agitazione intermittente, e autoclave). Cuocere vetreria in forno a 200 ° C o superiore per almeno 6 ore e trattare articoli di plastica con una soluzione di decontaminazione RNasi.
- Giorno 1: Immergere le fette di tessuto a ~ 10 ml di soluzione di Farmer (3: 1 Etanolo: Acido acetico) in flacone di vetro sul ghiaccio. Dopo che tutti i campioni sono stati sezionati, applicare vacuum per rimuovere le bolle d'aria e la penetrazione ausilio di fissativo. Tenere sotto vuoto per 10 minuti quindi rilasciare il vuoto lentamente. Sostituire fissativo e incubare a 4 ° C per una notte con un leggero scuotimento.
- 2 ° giorno: incubare nella seguente serie di soluzioni, ~ 10 ml ciascuna, 1 ora ciascuna, tutte con dolce agitazione; 85% di etanolo a 4 ° C, 95% di etanolo a 4 ° C, 100% di etanolo a 4 ° C, 100% di etanolo a 4 ° C, 100% di etanolo a 4 ° C, 1: 1 etanolo: xileni a temperatura ambiente, 100% xileni a temperatura ambiente, 100% xileni a temperatura ambiente.
NOTA: Xileni sono tossici per contatto e l'inalazione. Lavorare in una cappa aspirante e usare guanti appropriati. - Aggiungere tessuto inclusione in paraffina pellet di media a circa la metà del volume di xileni e incubare una notte a temperatura ambiente agitando delicatamente.
- Giorno 3: Trasferire il flacone a 60 ° C forno fino a quando il pellet si sciolgono. Eliminare la soluzione e sostituirlo con fresco tessuto fuso mezzo di inclusione. Modificare le medie altre due voltedurante il giorno.
- 4 ° giorno: Modifica tessuto mezzo di inclusione una volta al mattino. Ritorno a 60 ° C forno fino al pomeriggio.
- blocchi del cast
- Mettere stampi embedding sulla piastra calda della stazione di tessuti incorporamento. Utilizzare pinze per trasferire i campioni di tessuto per gli stampi incorporamento con la superficie di taglio rivolto verso il basso. Riempire lo stampo con paraffina fusa e posizionare l'anello incorporamento sulla parte superiore dello stampo. Trasferire in un piatto freddo fino a quando la paraffina si è solidificato. Memorizzare i blocchi di paraffina a 4 ° C in un contenitore ermetico con gel di silice.
2. Sezioni e scorrere Preparazione
- Tagliare 10 sezioni micron su un microtomo 25.
- Esaminare nastri e scegliere le sezioni mediane. sezioni mediani sono quelli che includono la SAM, che appare come una cupola di cellule circondate da primordi fogliari.
- sezioni Montare su diapositive.
- Collocare i vetrini che sono adatti per LM (sia RNasi gratuito oal forno) su 42 ° C più caldo scorrere e applicare alcune gocce di soluzione di etanolo al 50% per coprire la diapositiva.
- Float sezioni sulla soluzione di etanolo fino a che le sezioni hanno ampliato.
NOTA: le sezioni di galleggiamento sulla soluzione di etanolo piuttosto che l'acqua continua a RNA in uno stato precipitato ridurre la degradazione dell'RNA. - Inclinare scivolo e rimuovere soluzione di etanolo in eccesso mediante aspirazione con una pipetta di trasferimento usa e getta. Utilizzare salviettine privo di lanugine per allontanare qualsiasi soluzione di etanolo aggiuntivo.
- diapositive a secco a 42 ° C per diverse ore o durante la notte. Conservare scivola a 4 ° C in un contenitore ermetico con gel di silice.
- Deparaffinare scorre il giorno di utilizzo.
- Preparare tre vasetti di vetro contenenti Coplin; 100% xileni (xileni I), 100% xileni (xilene II), e 100% di etanolo (~ 50 ml di ciascuna soluzione).
- Utilizzando pinze pulite per trasferire le diapositive, immergere i vetrini in xileni I per 2 minuti, xileni II per 2 minuti, e il 100% di etanolo per 1 min.
- drain scivolatoes su salviettine senza peli e aria secca a temperatura ambiente.
3. Microdissezione di Blade, ligula e guaina campioni di Plastochron 7 Leaf Primordia
- Fissare le diapositive sul palco del microscopio LM. Utilizzare cinque o sei diapositive per ogni replica, utilizzando cinque sezioni per diapositiva.
NOTA: Tissue pool per un singolo replicato è illustrato in figura 5. - Esaminate diapositive e identificare le cinque sezioni più mediani su ciascun vetrino, utilizzando il SAM apice come punto di riferimento centrale.
NOTA: Questo può essere fatto a basso ingrandimento, di solito un obiettivo 5X è sufficiente. - Utilizzando 10X o 20X obiettivo, identificare la posizione del ligule sul plastochron 7 foglio primordium di ogni sezione. Il ligula sarà visibile come un urto che sporge dalla superficie adaxial del primordio foglia. Segnare questa posizione utilizzando lo strumento di disegno del software LM; selezionare l'icona della matita, spostare il cursore sulla positi appropriatasu e fare clic e trascinare il mouse per disegnare.
NOTA: un obiettivo 10X o 20X è adatto per questo e per passi successivi. Quando si utilizzano sezioni laterali, i due lati di ogni foglio primordium saranno presenti in ogni sezione (Figura 2A). - Utilizzando lo strumento righello e lo strumento di disegno rettangolare, misura 100 micron alti rettangoli centrate sulla ligule di ciascuna sezione (rettangoli rossi, Figura 2A, 2B). Questi saranno il campione "Ligula".
- Per utilizzare lo strumento righello; selezionare l'icona righello, spostare il cursore su una estremità dell'oggetto da misurare, fare clic e trascinare per misurare l'oggetto. La lunghezza del righello verrà visualizzato sullo schermo.
- Per disegnare un rettangolo; selezionare l'icona di rettangolo, spostare il cursore su un punto che sarà uno degli angoli del rettangolo, fare clic e trascinare per disegnare un rettangolo di dimensioni adeguate. In alternativa, selezionare lo strumento di disegno linea retta e disegnare quattro linee rette.
- misura 100 micron rettangoli posizionati 50 micron sopra e sotto il rettangolo "Ligula".
NOTA: Questi saranno il "Blade" e campioni "guaina", rispettivamente (verde e blu rettangoli, Figura 2a, 2b). Sulla base dei nostri dati istologici, un rettangolo di 100 micron abbraccia l'intera regione ligula. sono stati scelti porzioni equivalenti dimensioni della lama e guaina per garantire che simili quantità di tessuto sono stati raccolti per ciascuna. Distanziali di 50 micron sono stati usati per garantire che nessun tessuto regione ligula è inavvertitamente incluso nella lama o guaina microdissections.
- Microdissect misurata rettangoli (Figura 2D - 2F) 7, 8, 9, la raccolta di campioni Ligula, Lama e guaina in tubi separati. Utilizzare la funzione di taglio laser per tagliare il sezione di tessuto lungo il contorno del dominio selezionato. Utilizzare il catafunzione pult per spingere il rettangolo di tessuto dal vetrino e nel coperchio del tubo (Figura 2D - 2F).
4. Microdissezione di Blade, ligula e guaina adaxial epidermiche campioni di Plastochron 7 Leaf Primordia
- Selezionare le sezioni e utilizzare lo strumento righello per misurare 100 micron alti segmenti centrati sulla plastochron 7 ligula, come descritto nella sezione 3 (sopra).
- Selezionare solo le cellule epidermiche adaxial di ogni 100 micron alto "Blade" e (selezioni verdi e blu, Figura 2C) "guaina" segmento delineando con lo strumento di disegno. L'epidermide è lo strato cellulare esterno; lato adaxial è quello più vicino al SAM.
- Per il campione "Ligula", selezionare solo le cellule del urto ligula emergente come descritto nella Sezione 3.3 (selezione rosso, Figura 2C).
- Microdissect selezionato regioni, la raccolta di Lama, Ligula e Sheath epidermide campioni in tubi separati, come descritto nella sezione 3.5.
5. Microdissezione di Plastochron 6 Leaf Primordia da Wild-type Fratelli LG1-R e
- Crescere segregare famiglie di mutanti (LG1-R) e le piante wild-type.
- Fissare e sparare processo apici per LM, come descritto nelle sezioni 1,2-1,4. Fissare wild-type e mutanti apici dei germogli in flaconcini separati in modo da tenerli separati. I campioni provenienti dalla stessa semina devono essere fissati e trattati per l'ibridazione in situ.
- Determinare dove LG1 è trascritto nel wild-type fratelli, effettuando LG1 ibridazione in situ 6, 26, 27. Misurare la posizione del accumulo di trascrizione LG1 dalla base della foglia primordio in campioni multipli (Figura 1E).
- Sulla base dei dati in situ ibridazione, scegliere porzione di foglia Primordium che comprende la regione in cui è trascritto LG1. In questo caso, 400-900 micron dalla base di plastochron 6 foglia primordi (rettangoli viola, Figura 2A).
- Microdissect selezionata porzione di foglia primordi, come descritto nella sezione 3.5, raccogliendo LG1-R e campioni wild-type in tubi separati.
6. Applicare RNA tampone di estrazione
- Applicare 50 microlitri tampone di estrazione di RNA al tessuto microdissezione e procedere con l'estrazione di RNA. Continuare con l'estrazione di RNA, l'amplificazione di RNA, la costruzione biblioteca, il sequenziamento e l'analisi bioinformatica, come descritto in riferimento 6.
Risultati
Utilizzando lo schema di LM delineato nella figura 2, a circa 1,000,000-1,500,000 micron 2 di tessuto sono stati raccolti per ogni replica in tutti i-Cell-strati LM (Figura 5), e 200.000 micron 2 per replicare per l'epidermide adaxial LM. Circa 2.500.000 micron 2 di tessuto è stato raccolto per ogni replica nel LM di wild-type primordi foglia LG1-R e. Due turni di amplificazione RNA lineare ha prodotto micro...
Discussione
Disegno sperimentale è un fattore critico in esperimenti di RNA-Seq. Considerazioni chiave sono il dominio precisi (s) e fase di sviluppo (s) da analizzare, e quali i confronti saranno fatti. E 'fondamentale pensare in termini di confronto, poiché l'uscita è in genere un elenco di geni che sono de tra due o più condizioni. Come con tutti gli esperimenti, è importante modificare una sola variabile alla volta. Ad esempio, quando si confrontano diversi domini di foglie, foglie della stessa età e stadio di svi...
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
Gli autori ringraziano S. Nasello per collaborazione in corso e stimolare discussioni sullo sviluppo ligula. Questo lavoro è supportato dalla National Science Foundation Grants MCB 1.052.051 e IOS-1.848.478.
Materiali
| Name | Company | Catalog Number | Comments |
| Diethyl pyrocarbonate | Sigma-Aldrich | 159220 | Used for RNase treatment of solutions |
| Razor blades | Electron Microscopy Sciences | 72000 | |
| RNase Zap | Sigma-Aldrich | R2020-250ML | RNase decontamination solution |
| Ethanol absolute 200 proof | Fisher Scientific | BP28184 | |
| Acetic acid, glacial | Sigma-Aldrich | A6283 | |
| Glass vials - 22 ml | VWR | 470206-384 | |
| Xylenes, histological grade | Sigma-Aldrich | 534056-4L | |
| Paraplast plus | Sigma-Aldrich | P3683-1KG | |
| Disposable base molds, 15 mm x 15 mm x 5 mm | VWR | 15154-072 | |
| Embedding rings | VWR | 15154-303 | |
| Silica gel packets | Electron Microscopy Sciences | 71206-01 | Desiccant for storage of paraffin blocks |
| Oven | Fisher Scientific | 15-103-0503 | Oven must maintain temperature of 60 °C |
| Paraffin embedding station | Leica | EG1160 | |
| Microtome | Leica | RM2235 | |
| Slide warmer | Electron Microscopy Sciences | 71317-10 | |
| Coplin jars | Electron Microscopy Sciences | 70316-02 | |
| Laser microdissector | Zeiss | ||
| KIMWIPES™ Delicate Task Wipers | Kimberly-Clark Professional | 34120 | Lint-free wipes for wicking excess solutions from microscope slides |
| Membrane Slide 1.0 PEN | Zeiss | 415190-9041-000 | Slides for laser microdissection |
| Adhesive Cap 200 opaque | Zeiss | 415190-9181-000 | Tubes for laser microdissection |
| PicoPure RNA Isolation Kit | ThermoFisher Scientific | KIT0204 |
Riferimenti
- Becraft, P. W., Bongard-Pierce, D. K., Sylvester, A. W., Poethig, R. S., Freeling, M. The liguleless-1 gene acts tissue specifically in maize leaf development. Dev Biol. 141 (1), 220-232 (1990).
- Sylvester, A. W., Cande, W. Z., Freeling, M. Division and differentiation during normal and liguleless-1 maize leaf development. Development. 110 (3), 985-1000 (1990).
- Moreno, M. A., Harper, L. C., Krueger, R. W., Dellaporta, S. L., Freeling, M. liguleless1 encodes a nuclear-localized protein required for induction of ligules and auricles during maize leaf organogenesis. Genes Dev. 11 (5), 616-628 (1997).
- Emerson, R. A. The inheritance of the ligule and auricle of corn leaves. Neb. Agr. Exp. Sta. An. Rep. 25, 81-85 (1912).
- Moon, J., Candela, H., Hake, S. The Liguleless narrow mutation affects proximal-distal signaling and leaf growth. Development. 140 (2), 405-412 (2013).
- Johnston, R., et al. Transcriptomic Analyses Indicate That Maize Ligule Development Recapitulates Gene Expression Patterns That Occur during Lateral Organ Initiation. Plant Cell. 26 (12), 4718-4732 (2014).
- Schmid, M. W., et al. A powerful method for transcriptional profiling of specific cell types in eukaryotes: laser-assisted microdissection and RNA sequencing. PLoS One. 7 (1), e29685 (2012).
- Kerk, N. M., Ceserani, T., Tausta, S. L., Sussex, I. M., Nelson, T. M. Laser capture microdissection of cells from plant tissues. Plant Physiol. 132 (1), 27-35 (2003).
- Nakazono, M., Qiu, F., Borsuk, L. A., Schnable, P. S. Laser-capture microdissection, a tool for the global analysis of gene expression in specific plant cell types: identification of genes expressed differentially in epidermal cells or vascular tissues of maize. Plant Cell. 15 (3), 583-596 (2003).
- Marioni, J. C., Mason, C. E., Mane, S. M., Stephens, M., Gilad, Y. RNA-seq: an assessment of technical reproducibility and comparison with gene expression arrays. Genome Res. 18 (9), 1509-1517 (2008).
- Wang, Z., Gerstein, M., Snyder, M. RNA-Seq: a revolutionary tool for transcriptomics. Nat Rev Genet. 10 (1), 57-63 (2009).
- Brooks, L., et al. Microdissection of shoot meristem functional domains. PLoS Genet. 5 (5), e1000476 (2009).
- Cai, S., Lashbrook, C. C. Stamen abscission zone transcriptome profiling reveals new candidates for abscission control: enhanced retention of floral organs in transgenic plants overexpressing Arabidopsis ZINC FINGER PROTEIN2. Plant Physiol. 146 (3), 1305-1321 (2008).
- Li, P., et al. The developmental dynamics of the maize leaf transcriptome. Nat Genet. 42 (12), 1060-1067 (2010).
- Eveland, A. L., et al. Regulatory modules controlling maize inflorescence architecture. Genome Res. 24 (3), 431-443 (2014).
- Takacs, E. M., et al. Ontogeny of the maize shoot apical meristem. Plant Cell. 24 (8), 3219-3234 (2012).
- Aida, M., Ishida, T., Tasaka, M. Shoot apical meristem and cotyledon formation during Arabidopsis embryogenesis: interaction among the CUP-SHAPED COTYLEDON and SHOOT MERISTEMLESS genes. Development. 126 (8), 1563-1570 (1999).
- Ishida, T., Aida, M., Takada, S., Tasaka, M. Involvement of CUP-SHAPED COTYLEDON genes in gynoecium and ovule development in Arabidopsis thaliana. Plant Cell Physiol. 41 (1), 60-67 (2000).
- Takada, S., Hibara, K., Ishida, T., Tasaka, M. The CUP-SHAPED COTYLEDON1 gene of Arabidopsis regulates shoot apical meristem formation. Development. 128 (7), 1127-1135 (2001).
- Nardmann, J., Ji, J., Werr, W., Scanlon, M. J. The maize duplicate genes narrow sheath1 and narrow sheath2 encode a conserved homeobox gene function in a lateral domain of shoot apical meristems. Development. 131 (12), 2827-2839 (2004).
- Bonner, W. A., Hulett, H. R., Sweet, R. G., Herzenberg, L. A. Fluorescence activated cell sorting. Rev Sci Instrum. 43 (3), 404-409 (1972).
- Birnbaum, K., et al. A gene expression map of the Arabidopsis root. Science. 302 (5652), 1956-1960 (2003).
- Brady, S. M., et al. A high-resolution root spatiotemporal map reveals dominant expression patterns. Science. 318 (5851), 801-806 (2007).
- Carter, A. D., Bonyadi, R., Gifford, M. L. The use of fluorescence-activated cell sorting in studying plant development and environmental responses. Int J Dev Biol. 57 (6-8), 545-552 (2013).
- Ruzin, S. E. . Plant microtechnique and microscopy. , (1999).
- Jackson, D., Veit, B., Hake, S. Expression of maize KNOTTED1 related homeobox genes in the shoot apical meristem predicts patterns of morphogenesis in the vegetative shoot. Development. 120, 405-413 (1994).
- Javelle, M., Marco, C. F., Timmermans, M. In situ hybridization for the precise localization of transcripts in plants. J Vis Exp. (57), e3328 (2011).
- Day, R. C., McNoe, L., Macknight, R. C. Evaluation of global RNA amplification and its use for high-throughput transcript analysis of laser-microdissected endosperm. Int J Plant Genomics. , 61028 (2007).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon