È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Produzione, Cristallizzazione e struttura Determinazione
In questo articolo
Riepilogo
Proline-proline endopeptidase-1 (PPEP-1) is a secreted metalloprotease and promising drug-target from the human pathogen Clostridium difficile. Here we describe all methods necessary for the production and structure determination of this protein.
Abstract
New therapies are needed to treat Clostridium difficile infections that are a major threat to human health. The C. difficile metalloprotease PPEP-1 is a target for future development of inhibitors to decrease the virulence of the pathogen. To perform biophysical and structural characterization as well as inhibitor screening, large amounts of pure and active protein will be needed. We have developed a protocol for efficient production and purification of PPEP-1 by the use of E. coli as the expression host yielding sufficient amounts and purity of protein for crystallization and structure determination. Additionally, using microseeding, highly intergrown crystals of PPEP-1 can be grown to well-ordered crystals suitable for X-ray diffraction analysis. The methods could also be used to produce other recombinant proteins and to study the structures of other proteins producing intergrown crystals.
Introduzione
Clostridium difficile è una delle principali cause di infezioni nosocomiali diarrea associata agli antibiotici 1. Questo batterio anaerobico Gram-positivi si trasmette attraverso la sua forma di spore per via fecale-orale. Negli ultimi dieci anni, il nuovo '' epidemia '' o '' ipervirulento '' ceppi (ad es BI / NAP1 / 027) ha causato un drastico aumento delle nuove infezioni e il tasso di mortalità in Nord America e in Europa 2. C. malattia -associated difficile (CDAD) è un pericolo di vita infiammazione del colon con alti tassi di mortalità 3. I sintomi variano da diarrea 4 a colite pseudomembranosa 5 e il megacolon tossico spesso fatale 6.
Il trattamento di CDAD è difficile in quanto i ceppi virulenti sono multi-resistente e il tasso di recidiva è alto 7. Al momento terapia include il metronidazolo antibiotici, fidaxomicina o vancomicina, o in repetitiVely casi ricorrenti di trapianto microbiota fecale. Nuove strategie terapeutiche sono urgentemente necessari 8. Alcuni progressi viene registrato come il anticorpo monoclonale terapeutico Bezlotoxumab, il targeting tossina di C. difficile B 9, ha recentemente superato con successo di fase III di sperimentazione clinica ed è stato depositato per l'approvazione con la FDA ed EMA. Inoltre, nuovi antibiotici sono in fase di sperimentazione in questo momento nelle diverse fasi della sperimentazione clinica 10.
Per sviluppare un trattamento efficace devono essere identificati nuovi bersagli terapeutici. La recente scoperta da C. difficile proteasi endopeptidasi-1 prolina-prolina (PPEP-1; CD2830 / Zmp1; EC 3.4.24.89) è un obiettivo così promettente, come la mancanza di PPEP-1 in un ceppo knock-out diminuisce la virulenza di C . difficile in vivo 11. PPEP-1 è una metalloproteasi secreta 12,13 fendendo due adesine C. difficile al loro C-terminale 13 liberando così il Bacter aderentiia dal epitelio intestinale umano. Pertanto, è coinvolto nel mantenere l'equilibrio tra il fenotipo sessili e motilità di C. difficile. Per sviluppare inibitori selettivi contro PPEP-1 e per capire come riconosce i suoi substrati intima conoscenza della sua struttura tridimensionale è indispensabile. Abbiamo risolto la prima struttura cristallina di PPEP-1 solo e in complesso con un peptide substrato 14. PPEP-1 è la proteasi prima noto che selettivamente scinde i legami peptidici tra due residui di prolina 15. Si lega il substrato in doppio-piegato e stabilizza tramite una rete alifatico-aromatici estesa di residui ubicata nella S-ciclo che copre la proteasi sito attivo 14. Questa modalità substrato vincolante riservate PPEP-1 e non trovato in proteasi umane fino ad oggi. Questo lo rende un bersaglio promettente farmaco, e off-bersaglio effetti degli inibitori molto improbabili.
Per sviluppare e schermo selettivo PPEP-1 inhibitors in futuro è necessario una grande quantità di puro e monodisperse PPEP-1 proteina. Inoltre, per determinare la modalità di legame dei primi inibitori, strutture co-cristalline con PPEP-1 dovrà essere determinato. Nelle nostre mani PPEP-1 produce costantemente cristalli intergrown. Così abbiamo sviluppato una procedura di ottimizzazione per produrre cristalli singoli di diffrazione qualità di PPEP-1. In questo protocollo si descrive in dettaglio la soluzione di produzione, purificazione, cristallizzazione e la struttura di PPEP-1 14. Usiamo espressione intracellulare in Escherichia coli di una variante PPEP-1 privo della sequenza segnale di secrezione, cromatografia di affinità e cromatografia ad esclusione sterica con la rimozione del tag purificazione, seguita da microseeding 16 in uno schermo ottimizzazione e determinazione della struttura tramite zinco singola lunghezza d'onda di dispersione anomala (zinco-SAD) 17. Questo protocollo può essere adattato per la determinazione della struttura produttiva e di altre proteine (ad esempio </ Em> metalloproteasi), in particolare per le proteine che producono cristalli intergrown. Su richiesta, il plasmide DNA del costrutto (pET28a-NHIS-rPPEP-1) dati di diffrazione e può essere fornita per scopi didattici.
Protocollo
1. La clonazione e costruire design
- CLONE la sequenza codone ottimizzato (per E. coli) di C. difficile PPEP-1 senza il segnale peptide [aminoacidi 27-220, di cui in appresso ricombinante PPEP-1 (rPPEP-1) 11] nel vettore pET28a utilizzando Nde I e Xho I siti di restrizione (Figura 1) con un codone di stop al (vettore risultante pET28a-NHIS-rPPEP-1) 3'-end. Questo produce un N-terminale 6xHis-tag proteine (NHIS-rPPEP-1) con un sito di clivaggio trombina permette di rimuovere il tag durante la purificazione (Figura 1). Il plasmide contiene una cassetta di resistenza alla kanamicina per la selezione. I primer utilizzati per la clonazione sono descritte altrove 14.
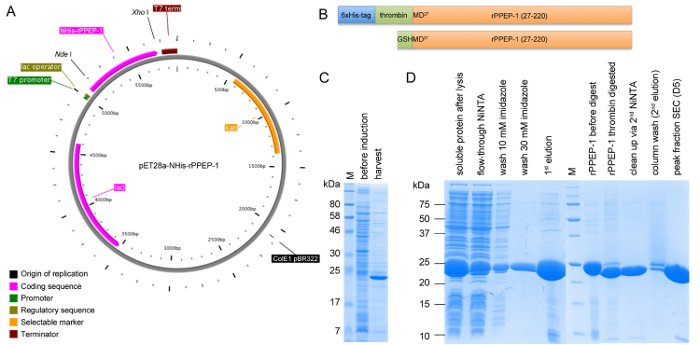
Figura 1: Rappresentazione schematica di costruire pET28a-NHIS-rPPEP-1 e SDS-PAGE analisi di espresfissione e tutte le fasi di purificazione. (A) Programma di vettore di NHIS-rPPEP-1 clonato nel vettore pET28a usando Nde I / Xho ho creato con PlasMapper. (B) Rappresentazione schematica del NHIS-rPPEP-1 costrutto (pannello superiore) e il costrutto finale dopo trombina-scissione del 6xHis-tag con il conseguente ulteriore GSHM-sequenza alla N-terminale (pannello inferiore). Analisi SDS-PAGE (C) dell'espressione in BL21 (DE3) stella a 37 ° C per 4 ore e (D) di campioni da tutti i passaggi di purificazione (M: marcatori di peso molecolare). Clicca qui per vedere una versione più grande di questa figura.
2. Espressione e purificazione di rPPEP-1
- L'espressione di NHIS-rPPEP-1
- Make up e autoclave LB (brodo lisogenia) media (10 g / L triptone, 5 g / l estratto di lievito, 10 g / L di NaCl, regost a pH 7.5 con NaOH). Supplemento con kanamicina solfato (50 mg / ml) al momento dell'uso (terreno LB / Kan).
- Inoculare 200 ml cultura durante la notte da appena trasformato E. coli in terreno LB / Kan. Crescere durante la notte a 37 ° C con agitazione a 220 rpm.
- La mattina successiva, controllare la (densità ottica a 600 nm di lunghezza d'onda) OD 600 della cultura durante la notte. Seminare due 2.8 L sconcertato recipienti contenenti 1 medio L LB / Kan ognuno con la cultura durante la notte ad un OD 600 di 0,1. Supplemento con tre gocce di emulsione acquosa di silicone per evitare la formazione di schiuma in eccesso. Crescere le cellule a 37 ° C agitando a 180 giri al minuto fino a quando la OD 600 raggiunge 0,6.
- Prelevare un campione di pre-induzione per l'analisi SDS-PAGE (equivalente di 1 ml di una cultura a OD 600 = 1); aggiungi IPTG a 0,5 mm concentrazione finale di indurre l'espressione di NHIS-rPPEP-1. Continuare a crescere a 37 ° C / 180 rpm per 4 ore.
- Determinare la 600 OD in un 10x diluzione e prendere un campione raccolto (equivalente di 1 ml di una coltura a OD 600 = 1).
- Raccogliere le cellule per centrifugazione per 20 min a 7000 xg e 4 ° C. Per rimuovere residui pellet cellulari medio risospendere LB da 1 L di cultura in 40 ml di tampone TBS (Tris tamponata salina: 20 mM Tris-HCl, pH 7.5, 200 mM NaCl) e trasferimento in una provetta da centrifuga da 50 ml. Raccogliere le cellule per centrifugazione per 10 min a 10.000 xg e 4 ° C e conservare a -80 ° C fino al momento dell'uso. Analizzare l'espressione (lisati totali e frazioni solubili) tramite SDS-PAGE 18.
- Purificazione di untagged rPPEP-1
- Prendere 50 campioni ml di ogni fase di purificazione per l'analisi SDS-PAGE. Risospendere il pellet cellulare da 1 L di cultura in tampone TBS integrato con 10 mg / ml DNasiI. Utilizzare 5 ml di TBS / DNasiI per g di cellule.
- Lyse le cellule mediante ultrasuoni su ghiaccio / acqua con il 30% di ampiezza per 15 min (2 impulsi sec con pausa di 2 sec). Rimuovere i detriti dal centrifugation per 10 min a 10.000 xg e 4 ° C e trasferimento surnatante in un tubo ultracentrifuga. Eliminare lisato in un'ultracentrifuga per 30 min a 165.000 xg e 4 ° C.
- Lavoro a 4-6 ° C. Utilizzando un sistema di pompa o cromatografia peristaltica equilibrare 2 ml di acido (NiNTA) resina al nichel-nitrilotriacetico in una colonna di vetro con tampone TBS supplementato con 10 mM imidazolo pH 7.5. In alternativa, utilizzare il flusso di gravità.
- Regolare il lisato eliminato con 1 M imidazolo pH 7,5 ad una concentrazione finale di 10 mM. Applicare il lisato alla colonna e lavare stepwise con tampone TBS supplementato con 10 mM e 30 mM imidazolo, rispettivamente fino l'assorbimento UV a 280 nm ha raggiunto la linea di base.
- Eluire la proteina con il tampone TBS più di 250 mm imidazolo. Riequilibrare la colonna di TBS supplementato con 10 mM imidazolo e memorizzare durante la notte.
- Determinare la concentrazione di proteine sia a 280 nm utilizzando il coeffi estinzioneciente di 25.900 M -1 cm -1 o da qualsiasi altro metodo (metodo Bradford esempio 19). Aggiungere 2 unità di trombina per mg di proteine e dializzare la soluzione proteica notte a 4 ° C contro un 50x volume di TBS (50x del volume di eluizione NiNTA).
NOTA: Prendere il vuoto corretta per la determinazione della concentrazione di proteine, come imidazolo assorbe fortemente a 280 nm. - Far passare la soluzione proteica sopra la resina NiNTA equilibrato per rimuovere proteine uncleaved. Successivamente, si applica lo stesso volume di TBS supplementato con 10 mM imidazolo alla colonna di recuperare tutte le proteine scissa. Per pulire la colonna, eluire tutti i restanti proteina con 250 mm imidazolo. Analizzare i campioni tramite SDS-PAGE (Figura 1).
- Concentrare la soluzione proteica a 4 ml in 10 intervalli min a 4000 xg e 4 ° C utilizzando una unità di ultrafiltrazione centrifuga. Mescolare la proteina concentrazione dopo ogni intervallo di evitare la precipitazione e aggregazione. A questo punto occasionalely qualche precipitazione è osservato per rPPEP-1 nonostante il processo di miscelazione.
- Applicare la proteina concentrata ad una colonna di cromatografia ad esclusione sterica pre-equilibrata (in tampone TBS) a 4-6 ° C. Eluire la colonna con tampone TBS, raccogliere 1 ml frazioni e soggetto 5 ml di ogni seconda frazione di analisi SDS-PAGE. rPPEP-1 eluisce con un picco corrispondente ad un monomero (Figura 2). Occasionalmente un picco minore al maggiore peso molecolare è osservata (picco fronting), che corrisponde ad un dimero della proteina. Il rendimento dovrebbe essere di circa 50 mg di pura proteina per L della cultura. Analizzare tutti i campioni tramite SDS-PAGE 18 (Figura 2).
- Prendere 50 campioni ml di ogni fase di purificazione per l'analisi SDS-PAGE. Risospendere il pellet cellulare da 1 L di cultura in tampone TBS integrato con 10 mg / ml DNasiI. Utilizzare 5 ml di TBS / DNasiI per g di cellule.
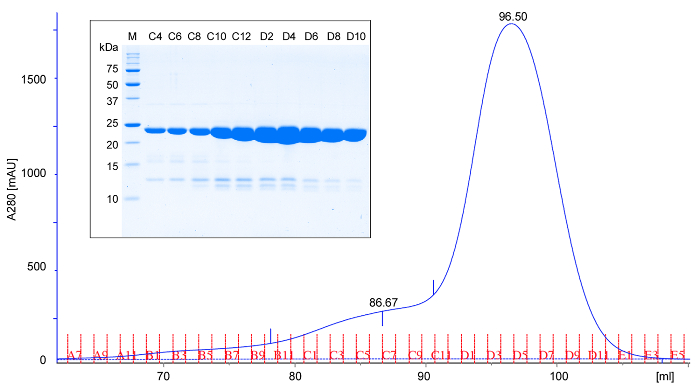
Figura 2: Rappresentante cromatografia dimensione esclusione e SDS-PAGE analisi di rPPEP-1. esclusione dimensione cromatogramma (A280; assorbanza a 280 nm) di purificato etichettato rPPEP-1 utilizzando una) column (16/600 in Tris-HCl, pH 7.5, 200 mM NaCl a 6 ° C. Sulla base del volume di eluizione, rPPEP-1 migra come previsto per una proteina 22 kDa, suggerendo che è prevalentemente monomerica. Raramente un picco fronting minore risulta che corrisponde ad un dimero. (Riquadro) analisi SDS-PAGE delle frazioni da cromatografia dimensione esclusione (M; marcatore di peso molecolare). Ogni seconda frazione viene applicato. Le fasce deboli sotto il principale rPPEP-1 banda corrispondono a che si verificano di tanto in tanto le impurità minori. Clicca qui per vedere una versione più grande di questa figura.
3. Cristallizzazione e Crystal ottimizzazione Utilizzando Microseeding
NOTA: rPPEP-1 cristallizza da condizioni che producono costantemente cristalli altamente intergrown non adatto per l'analisi di diffrazione a raggi X (figura 3). Pertanto, una strategia di ottimizzazione (Figura 4) è stato sviluppato per ottenere cristalli di alta qualità (Figura 5).
- Screening iniziale di rPPEP-1 utilizzando schermi commerciali
NOTA: effettuare prove di cristallizzazione nella seduta formato goccia utilizzando schermi disponibili in commercio standard e un robot cristallizzazione.- Concentrare la proteina purificata a 12 mg / ml usando un dispositivo di ultrafiltrazione centrifuga a intervalli di 5 minuti a 4000 xg e 4 ° C. Mescolare la proteina concentrazione dopo ogni intervallo di evitare la precipitazione e aggregazione. Determinare la concentrazione di proteine sia a 280 nm utilizzando il coefficiente di estinzione di 25.900 M -1 cm -1 o mediante qualsiasi altro metodo (metodo Bradford). Equilibrare la proteina a 20 ° C. Sgombrare tutte le particelle e polvere mediante centrifugazione per 10 min a 16.000 xg e 20 ° C.
- Utilizzare le piastre di cristallizzazione già preriempite SEAled e conservato a 4 ° C o compili pozzetti serbatoio delle piastre con 70 microlitri di ogni condizione di cristallizzazione. Equilibrare tutte le piastre di cristallizzazione a 20 ° C. Lavorare velocemente, come i piccoli volumi si asciugano velocemente. Utilizzare una camera di umidità intorno al molo del robot, se possibile.
NOTA: Utilizzare le seguenti schermate come procedura standard: SaltRx, indice, PEG / Ion, cristallo, Wizard, PACT ++, JCSG ++. - Impostare la schermata pipettando proteine e serbatoio in subwells 2-4. il volume di goccia è di 300 nl e rapporti (proteina: serbatoio) sono 200: 100 (subwell 2), 150: 150 (subwell 3) e 100: 200 (subwell 4) (in nl). Sigillare immediatamente la piastra e posto in una camera a 20 ° C.
- Ispezionare vassoi subito dopo set-up, e poi ispezionare ogni giorno durante la prima settimana seguito da un'ispezione settimanale.
- Co-cristallizzazione di rPPEP-1 con leganti
- Per la co-cristallizzazione di substrato peptide-rPPEP-1 complessi mix rPPEP-1a 24 mg / ml in un rapporto 1: 1 (v / v) con un eccesso molare di 7 volte della soluzione di peptide (Ac-EVNPPVPD-NH 2) polvere liofilizzata solubilizzati in tampone TBS), che darà una concentrazione finale di 12 mg / ml r-PPEP-1 proteine e 7 volte l'eccesso molare di peptide over PPEP-1. Incubare per 30 minuti a 20 ° C e sgombrare tutte le particelle e polvere mediante centrifugazione per 10 min a 16.000 xg e 20 ° C. Procedere cristallizzazione utilizzando la procedura microseeding come descritto per il non legato r-PPEP-1 proteina.

Figura 3: cristalli rappresentativi da schermate iniziali. cristalli Intergrown da rPPEP-1 a 12 mg / ml coltivate in condizioni. (A) schermo a cristalli I / 38 (1,4 M di sodio citrato tribasico disidratano, 0,1 M di HEPES di sodio pH 7,5; 200 nl: 100 nl). / 52 (fosfato dib 2,4 M di ammonio (B) schermo SaltRxASIC, 0,1 M Tris pH 8,5; 100 nl: 200 nl) e (C) (200 nl: 100 nl). (D) schermo SaltRx / 96 (60% v / v Tacsimate pH 7,0, 0,1 M BIS-TRIS propano pH 7,0; 200 nl: 100 nl). La barra di scala = 0.2 mm. rapporti di volume sono sempre proteine: serbatoio. Clicca qui per vedere una versione più grande di questa figura.
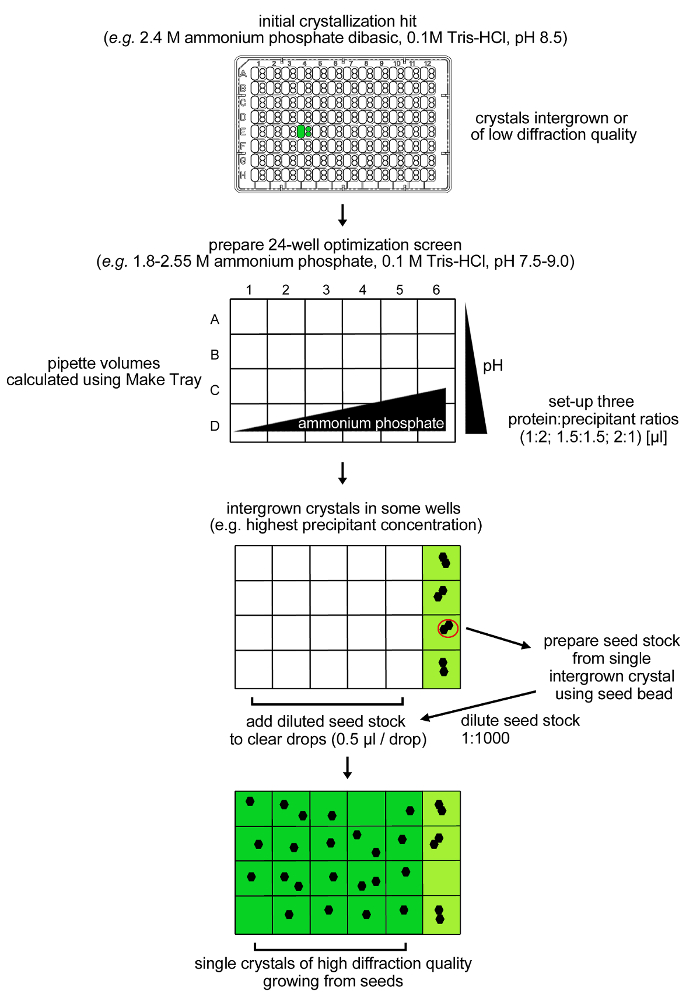
Figura 4: procedura di ottimizzazione per rPPEP-1 cristallizzazione. cristalli iniziali da rPPEP-1 a 12 mg / ml di bassa qualità diffrazione e con più reticoli (intergrown) sono stati riprodotti in una schermata di ottimizzazione 24-condizione. Anche in questo caso, solo i cristalli intergrown sono stati osservati in condizioni contenente 2,55 M di ammonio fosfato bibasico. Uno stock di semi è stato preparato da un singolo cristallo intergrown e diluito 1: 1000 nella stessa Conditione (microseeding). Un volume di 0,5 ml di ceppo diluito è stato aggiunto nelle restanti gocce chiare e cristalli singoli è cresciuto in quasi tutte le condizioni. Clicca qui per vedere una versione più grande di questa figura.
- Ottimizzazione di cristallo usando microseeding
NOTA: cristalli altamente intergrown di rPPEP-1 compaiono dopo due giorni in una condizione contenente 2,4 M di ammonio fosfato bibasico 0.1 M Tris-HCl, pH 8,5 (schermo SaltRx, Stato E4, tutti tre subwells) (Figura 3). Una procedura di ottimizzazione utilizzare uno schermo di griglia attorno alla condizione iniziale combinato con microseeding stato applicato (figura 4).- Preparare una schermata di rete (figura 4) che comprende 24 condizioni con 1,8-2,55 M bibasico ammonio fosfato (con incrementi di 0,15 M) e 0,1 M Tris-HCl pH 7,5-9,0 (a passi di 0,5 pH unità) da approLe soluzioni mangiato azionari (4 M ammonio fosfato e 1 buffer M Tris).
NOTA: Utilizzare l'applet vassoio Marca (http://hamptonresearch.com/make_tray.aspx) per calcolare i volumi e il regime di dispensazione per ottenere 2 ml di ogni condizione che permette di eseguire 10 schermi di ottimizzazione. Il fosfato di soluzione 4 M di ammonio è difficile da preparare. Riscaldare la soluzione durante mescolando per far sciogliere completamente la polvere in acqua. - Pipettare 200 ml di ogni soluzione schermo griglia nei pozzetti di una piastra da 24 pozzetti ed equilibrare a 20 ° C.
- Impostare manualmente il piatto di cristallizzazione. Il volume di goccia è 3 microlitri ei rapporti (proteina: serbatoio) sono 2: 1, 1.5: 1.5 e 1: 2 (in microlitri). Qui, usare una pipetta a spostamento positivo per evitare la formazione di bolle d'aria. Sigillare immediatamente la piastra e posto in una camera a 20 ° C. Evitare la formazione di bolle d'aria.
NOTA: Dopo uno a quattro giorni cristalli altamente intergrown appaiono nelle quattro condizioni che contengono pho 2,55 M di ammoniobibasico sphate e 0,1 M Tris-HCl pH 7.5 - 9.0 (Figura 4). Non cristalli si formano nelle restanti 20 condizioni con concentrazioni di ammonio fosfato sotto 2,55 M. La procedura microseeding viene utilizzato per ottenere cristalli singoli di rPPEP-1 in queste condizioni. - Preparare una microseed magazzino per la raccolta di un singolo cristallo intergrown da una delle due condizioni con 2,55 M di ammonio fosfato bibasico e 0,1 M Tris-HCl pH 8,0 o 8,5. I cristalli possono essere collegati alla superficie di plastica. deformazione attento della plastica circostante con un ago da agopuntura aiuta a staccare i cristalli.
- Trasferimento 50 ml di rispettivo liquido madre in una provetta da 1,5 ml contenente una piccola biglia di vetro lucidato (perline-per-semi). Utilizzando un'ansa nylon montato trasferire il cristallo in 1 ml di soluzione madre poste su un vetrino di copertura in vetro.
- Trasferire il liquido contenente il cristallo nella provetta e vortex ad alta velocità per 30 sec. Fare un 1:1.000 diluizione del magazzino seme in una nuova provetta da 1,5 ml, contenente la stessa condizione appena preparato e vortex accuratamente per 5 secondi.
NOTA: le scorte semi possono essere conservati a -80 ° C per un uso successivo.
- Rimuovere la guarnizione del piatto che copre le 20 condizioni con gocce chiare e pipetta 0,5 ml di brodo di semi (1: 1000 diluizione) nei pozzetti. Sigillare la piastra e posto in una camera a 20 ° C. Cristalli singoli di alta qualità di diffrazione appaiono in 2-7 giorni (Figura 5).
- Preparare una schermata di rete (figura 4) che comprende 24 condizioni con 1,8-2,55 M bibasico ammonio fosfato (con incrementi di 0,15 M) e 0,1 M Tris-HCl pH 7,5-9,0 (a passi di 0,5 pH unità) da approLe soluzioni mangiato azionari (4 M ammonio fosfato e 1 buffer M Tris).
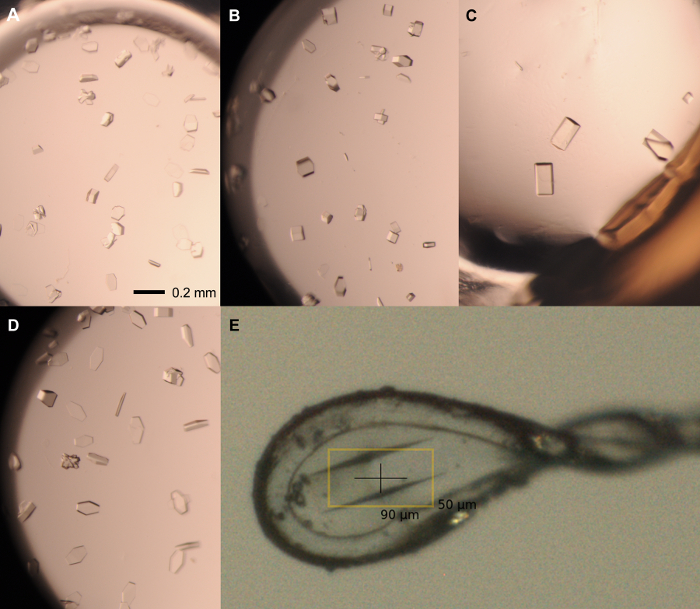
Figura 5: cristalli rappresentativi dallo schermo di ottimizzazione. Cristalli singoli di rPPEP-1 a 12 mg / ml seminato con 1: 1000 diluizione ceppo coltivato nelle seguenti condizioni: (A) 2,1 M di ammonio fosfato bibasico 0.1 M Tris pH 7,5; 1.5 ml: 1,5 microlitri; (B) 2.1 M di ammonio fosfato bibasico 0.1 M Tris pH 7,5; 2 ml: 1 ml; (C) 2,25 M di ammonio fosfato bibasico 0.1 M Tris pH 8; 2 ml: 1 ml; (D) 2,1 M di ammonio fosfato bibasico 0.1 M Tris pH 8; 1 ml: 2 microlitri. (E) Montato cristallo in 0,1-0,2 micron ciclo nylon, coltivato in 2,1 M di ammonio fosfato bibasico 0.1 M Tris pH 8 (2 microlitri: 1 ml) e crio-protetto in 2,1 M di ammonio fosfato bibasico 0.1 M Tris pH 8, 20% glicerolo. La barra di scala = 0.2 mm (AD). Volume razione sono sempre proteine: serbatoio. Clicca qui per vedere una versione più grande di questa figura.
4. Raccolta di montaggio e dati di cristallo
NOTA: Per ottenere la migliore qualità di cristalli di dati di diffrazione deve essere montato al picco della loro qualità e dimensioni. I cristalli possono essere conservati in azoto liquido fino are sottoposto ad analisi di diffrazione ai raggi X a 100 K. Pertanto, la condizione da cui provengono deve essere adattato crio-condizioni. rPPEP-1 cristalli possono essere crio-protetto mediante aggiunta di uno glicerolo al 20% o 30% di saccarosio (sostituzione di acqua nella condizione dalla crio-protettivo).
- montaggio di cristallo
NOTA: Tutte le fasi di manipolazione di cristallo devono essere eseguite con lo stereomicroscopio.- Scegliere la dimensione ottimale di anello in nylon per la lunghezza massima dei cristalli scelti. L'asse più lungo tipico di rPPEP-1 cristalli è di circa 100-200 micron (Figura 5). Preparare un vetrino di copertura e la crio-condizione appropriata (ad esempio 2,1 M di ammonio fosfato bibasico 0.1 M Tris, pH 8,0, 20% glicerolo).
- Riempire i dewar schiuma con azoto liquido, caricare il morsetto flaconcino con una fiala e pre-raffreddare nel liquido 800 ml di schiuma dewar riempito di azoto. Posizionare un manicotto crio e un porta canna crio contrassegnati con un identificativo adattoin azoto liquido-riempita 2 L schiuma Dewar. Caricare la bacchetta magnetica con un ciclo di nylon montato.
NOTA: Indossare indumenti protettivi (Eyeshield / occhiali, guanti) quando si lavora con l'azoto liquido. oggetti caldi immersi in azoto liquido può produrre fuoriuscite.
- Riempire i dewar schiuma con azoto liquido, caricare il morsetto flaconcino con una fiala e pre-raffreddare nel liquido 800 ml di schiuma dewar riempito di azoto. Posizionare un manicotto crio e un porta canna crio contrassegnati con un identificativo adattoin azoto liquido-riempita 2 L schiuma Dewar. Caricare la bacchetta magnetica con un ciclo di nylon montato.
- Tagliare aprire il nastro di tenuta con un bisturi affilato e rimuoverlo con le pinze. Pipettare 1 ml di crio-condizioni sul vetrino di copertura (o alternativamente in un pozzetto vuoto sulla stessa piastra) e rimuovere il cristallo dalla goccia pesca con il ciclo di nylon montata (figura 5). cristalli allegati possono essere facilmente staccati dalla terra deformando la plastica circostante con un ago di agopuntura.
- trasferire rapidamente il cristallo per il calo di crio-condizioni e lasciar riposare e stabilizzare per 1 secondo. Pesce il cristallo il più rapidamente possibile e tuffo-congelare in azoto liquido.
- Quando l'azoto liquido attorno all'anello montato smette bollente, posizionare l'anello nel flacone.Posizionare la fiala sul supporto canna crio e quando caricato con 6 flaconcini posizionare un manicotto di crio intorno al titolare. Memorizzare i cristalli in una vasca riempita con azoto liquido fino al momento dell'uso.
- Scegliere la dimensione ottimale di anello in nylon per la lunghezza massima dei cristalli scelti. L'asse più lungo tipico di rPPEP-1 cristalli è di circa 100-200 micron (Figura 5). Preparare un vetrino di copertura e la crio-condizione appropriata (ad esempio 2,1 M di ammonio fosfato bibasico 0.1 M Tris, pH 8,0, 20% glicerolo).
- Raccolta dati
NOTA: raccolta dei dati può essere effettuato al diffrattometro casa, se disponibile, o in una linea di luce di sincrotrone. Per rPPEP-1 i dati sono stati raccolti presso la linea di luce X06DA della Swiss Light Source, Paul-Scherrer-Institute, Villigen, Svizzera utilizzando un rivelatore di conteggio ibrido fotoni. I dati originali e tutti i file utilizzati nella determinazione della struttura sono disponibili su richiesta.- Impostare la lunghezza d'onda del fascio di 1.282 Å (9.667 keV), che è il x-ray energia bordo di assorbimento caratteristici (picco) dell'elemento zinco. rPPEP-1 è una metalloproteasi che contiene una singola zinco per molecola nel sito attivo.
- Raccogliere dati a 100 K in modalità inversa-beam in 10 ° cunei per un totale di 270 ° in ogni direction. Il tempo di esposizione è di 0,1 sec con 0,1 ° di rotazione per immagine. Impostare la trasmissione al 14% (0.14).
- Per raccogliere un set di dati nativo ad alta risoluzione da un secondo cristallo proveniente dalla stessa condizione di cristallizzazione impostare la lunghezza d'onda del fascio a 1.00 Å (12.398 keV). Raccogliere i dati a 100 K. Il tempo di esposizione è di 0,1 sec con 0,1 ° di rotazione per immagine. Impostare la trasmissione al 70% (0.7).
5. determinazione della struttura tramite zinco-SAD
NOTA: Al fine di determinare la struttura di rPPEP-1 tramite zinco-SAD è necessaria una certa conoscenza cristallografica di base, nonché i pacchetti software XDS 20, Phenix 21 e il programma Folaga 22. Per la visualizzazione delle strutture è necessario il programma PyMOL 23 o 24 Chimera. I dati raccolti alla lunghezza d'onda corrispondente al picco al bordo di assorbimento dell'elemento zinco possono essere utilizzati per singola lunghezza d'onda disp anomalaersion (SAD) 25 per ottenere informazioni di fase che può essere estesa a tutti gli atomi della proteina.
- Elaborazione dati
- Elaborare i due set di dati di picco (normale e inversa) utilizzando il software XDS (in alternativa iMosflm o HKL3000) nel gruppo spaziale P2 1 2 1 2 1 (gruppo spaziale 19) che separa i compagni del Friedel (dati anomali). I parametri di cella unità dovrebbe essere intorno a, b, c (A) = 43.17, 71.68, 117.70 e α = β = γ (°) = 90. Questo dà due HKL-file (file di riflessione).
- Controllare il file CORRECT.LP. Utilizzare dati fino alla risoluzione in cui la CC 1/2 è almeno del 50%. Scala insieme entrambi i set di dati / file di riflessione (HKL-files) usando XSCALE. Controllare il file XSCALE.LP. Verificare quanto il segnale anomalo si estende (SigAno) e nota la risoluzione con una correlazione anomala (anomal Corr) di circa il 30%, che è 2 Å nel caso dei dati utilizzati qui raccolti a 1.67 Å. Questo è ilRisoluzione cut-off per il segnale anomalo usato in Phenix Autosol.
- Convertire il (in scala) HKL-file in un file riflessione CCP4 formato (dal nome, ad esempio, peak_anom.mtz) utilizzando XDSCONV la creazione di un sottoinsieme libero R del 5% e la conservazione dei dati anomali (FRIEDEL'S_LAW = FALSE). Controllare l'MTZ-lima per coerenza con il programma di mtzdmp ispezionare i parametri dell'unità di cella, il gruppo lo spazio e l'esistenza del sottoinsieme libera R (etichetta FreeRflag) ei dati anomali (etichette DANO / SIGDANO). Preparare anche una MTZ-file aggiuntivo con XDSCONV senza estrarre i dati anomali (FRIEDEL'S_LAW = TRUE, chiamato, per esempio, peak_native.mtz) per la raffinatezza in una fase successiva.
- Soluzione sottostruttura (determinazione di fase)
- Eseguire Phenix Autosol utilizzando il file riflessione peak_anom.mtz. Selezionare SAD picco / MAD come tipo di dati e scegliere 2 siti di zinco (come ci sono due molecole per unità asimmetrica). Scegliere l'espe più precisavalori mentale per l'f '/ f' '(parametri determinati in una scansione di fluorescenza alla linea di luce) oi valori theotetical f' = -8,245 e f '' = 3.887. Inoltre caricare il file FASTA contenente la sequenza aminoacidica della proteina cristallizzata.
- Impostare il limite di risoluzione per la risoluzione con una correlazione anomala (anomal Corr) di circa il 30% (determinato a 5.1.2), in questo caso 2 A e selezionare l'opzione "modello autobuild". Utilizzando le fasi dei due siti di zinco trovate da Phenix HySS (parte del gasdotto Phenix Autosol) le fasi per tutta la proteina poteva dedurre e il modello costruito (da Phenix RESOLVE) nel densità elettronica. Il miglior modello si chiama "overall_best.pdb".
- Costruzione del modello, la raffinatezza e la convalida
- Selezionare l'opzione "modello autobuild" per costruire la maggior parte del modello rPPEP-1 automaticamente. Controllare la densità elettronica a 1,0 σ livello di profilo con il program Folaga (Figura 6). Dovrebbe essere connettivo circostante gli atomi del modello. Idealmente anche alcune molecole di acqua dovrebbe essere costruito nel modello (ad una risoluzione migliore di 2,5 A). acqua Bulk (spazio tra le molecole) dovrebbe contenere densità.
- Controllare se l'intero modello è (tutti gli aminoacidi incorporati nel densità elettronica) completi. In caso contrario, costruire manualmente utilizzando gli strumenti forniti da Coot. Affina la struttura eseguendo giri iterative di Phenix Perfeziona con 5 colpi ciascuno raffinatezza utilizzando il file del modello overall_best.pdb, il file peak_native.mtz riflessione e il file di sequenza FASTA; e la costruzione di modello manuale in Coot.
- Convalidare la qualità del modello strutturale con i rispettivi strumenti di Coot.
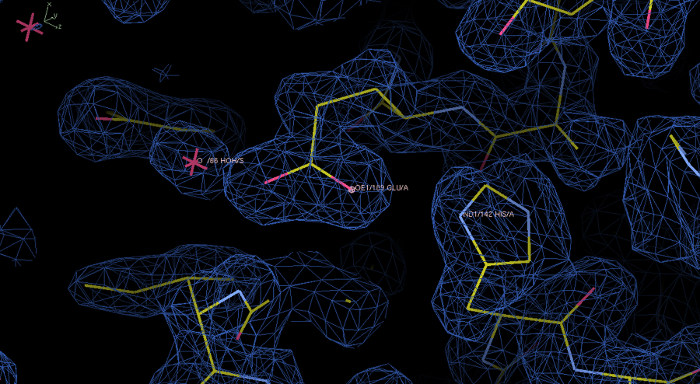
Figura 6: mappa densità elettronica sperimentale e il modello di rPPEP-1 dopo laPhenix Autosol eseguito. densità elettronica in blu ad un livello contorno di 1,0 σ visualizzato nella Folaga programma. In questa mappa iniziale, la densità elettronica è ben risolto e il modello di costruire nella densità elettronica. Il zoom mostra i residui His142 e Glu189, così come una molecola d'acqua. Clicca qui per vedere una versione più grande di questa figura.
6. Struttura Determinazione per alta risoluzione tramite sostituzione molecolare
NOTA: Al fine di ottenere ad alta risoluzione informazioni strutturali su rPPEP-1 un set di dati nativo viene raccolto. Poi una procedura di sostituzione molecolare utilizzando il software 26,27 Phaser (all'interno del pacchetto software Phenix) è impiegato con la struttura determinata tramite zinco-SAD come modello. Questa procedura può essere utilizzata anche in seguito quando la soluzione di strutture rPPEP-1 complessati con piccole molecole.
- Per ottenere una struttura di cristallo con risoluzione superiore (in questo caso fino a 1,4 Å) elaborare l'insieme di dati nativo utilizzando il software XDS (alternativamente iMosflm o HKL3000) in gruppo spaziale P2 1 2 1 2 1 (gruppo spaziale 19). I parametri di cella unità dovrebbe essere intorno a, b, c (A) = 43.17, 71.77, 117.80 e α = β = γ (°) = 90. Questo dà HKL-files (file di riflessione).
- Controllare il file CORRECT.LP. Utilizzare dati fino alla risoluzione in cui la CC 1/2 è almeno del 50%. Convertire il file HKL in un file riflessione CCP4 formato (dal nome, ad esempio, native.mtz) utilizzando XDSCONV la creazione di un sottoinsieme libero R del 5%. Controllare il MTZ-lima per coerenza con il programma di mtzdmp ispezionare i parametri di cella unitaria, il gruppo lo spazio e l'esistenza del sottoinsieme libera R (etichetta FreeRflag).
- Preparare il PDB-file contenente il modello da overall_best.pdb determinato in precedenza e rimuovere tutte le molecole d'acqua e tutti i leganti (vale a dire. l'atomo di zinco). Inoltre caricare il file FASTA contenente la sequenza aminoacidica della proteina cristallizzata. Eseguire Phaser a Phenix utilizzando il file riflessione native.mtz. Ricerca di due molecole per unità asimmetrica.
- Dopo soluzione struttura successo (TFZ-score superiore a 8; qui 10.2) ispezionare il modello (denominato native_phaser.1.pdb) e la mappa densità di elettroni in Coot. Costruire e perfezionare la struttura eseguendo giri iterative di Phenix Perfeziona con 5 colpi ciascuno raffinatezza utilizzando il file del modello native_phaser.1.pdb, il file riflessione native.mtz e il file di sequenza FASTA; e la costruzione di modello manuale in Coot.
- Convalidare la qualità del modello strutturale con i rispettivi strumenti di Coot.
Risultati
rPPEP-1 è sovraespresso in molti ceppi di E. coli, con il più alto rendimento in E. coli BL21 (DE3) stella (Figura 1C). Dopo la prima fase di cromatografia di affinità NiNTA la 6xHis-tag può essere scisso con successo fuori dalla maggior parte delle proteine e nella seconda fase NiNTA proteina non digerita può essere completamente separato dal trombina digerito proteine (Figura 1D). Su un S200 16/600 colonna senza tag rPPEP-1 migra...
Discussione
Cristallografia a raggi X è ancora il metodo più veloce e più accurato per determinare tridimensionali strutture risoluzione quasi atomica delle proteine 28. Tuttavia, esso richiede la crescita di cristalli singoli ben ordinate. Questi sono spesso difficili da ottenere e stato cristallino è artificiale. Tuttavia, un confronto di strutture proteiche determinata mediante cristallografia a raggi X con quelle determinate con altri metodi, in particolare NMR, generalmente mostra un ottimo accordo. Nel caso di ...
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
Ringraziamo il personale del X06DA linea di luce alla sorgente Swiss Light, Paul-Scherrer-Institute, Villigen, Svizzera per il supporto durante la raccolta dei dati di sincrotrone. Siamo grati a Monika Gompert per un eccellente supporto tecnico. Il progetto è stato sostenuto dall'Università di Colonia e concedere INST 216 / 682-1 FUGG dal Consiglio di ricerca tedesco. Una borsa di studio di dottorato presso la Graduate School Internazionale in Sviluppo salute e malattia a CP è riconosciuto. La ricerca che ha portato a questi risultati è stata finanziata dal Settimo Programma Quadro della Comunità Europea (FP7 / 2007-2013), contratto di sovvenzione n ° 283.570 (BioStruct-X).
Materiali
| Name | Company | Catalog Number | Comments |
| Genes / Vectors / cell strains | |||
| pET28a vector | Merck-Millipore | 69864 | Thrombin cleavable N-terminal His-tag |
| E. coli strain BL21 (DE3) Star | ThermoFisher Scientific | C601003 | RNase H deficient |
| Codon-optimized gene (for E. coli) of PPEP-1 (CD630_28300) | Geneart (Thermo Fisher Scientific) | custom | amino acids 27-220 |
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| Yeast extract | any | ||
| Tryptone | any | ||
| Antifoam B | Sigma-Aldrich | A5757 | aqueous-silicone emulsion |
| Agar | any | ||
| Kanamycin | any | ||
| IPTG | AppliChem | A1008 | |
| Tris-HCl | AppliChem | A1087 | Buffer grade |
| NaCl | any | Buffer grade | |
| DNaseI | AppliChem | A3778 | |
| Imidazole | AppliChem | A1073 | Buffer grade |
| Thrombin | Sigma-Aldrich | T4648 | |
| Ammonium phosphate dibasic | Sigma-Aldrich | 215996 | |
| Glycerol 100% | any | purest grade | |
| Sucrose | Sigma-Aldrich | 84097 | |
| Liquid nitrogen | any | for storage and cryocooling of crystals | |
| Name | Company | Catalog Number | Comments |
| Equipment (general) | |||
| Shaking incubator | any | providing temperatures of 20 °C - 37 °C | |
| Glassware | any | baffled Erlenmeyer flasks (50 ml - 2.8 L) | |
| Centrifuge for large culture volumes | any | centrifuge for processing volumes up to 12 L | |
| Sonicator Vibra-Cell VCX500 | Sonics | SO-VCX500 | or any other sonicator / cell disruptor |
| Ultracentrifuge | any | centrifuge providing speeds up to 150,000 x g | |
| NiNTA Superflow resin | Qiagen | ||
| Empty Glass Econo-Column | Bio-Rad | 7371007 | or any other empty glass or plastic column |
| Size exclusion chromatography column HiLoad Superdex 200 16/600 | GE Healthcare | 28989335 | |
| Chromatography system Äkta Purifier | GE Healthcare | 28406264 | or any other chromatography system |
| Dialysis tubing Spectra/Por 3 | Spectrum Labs | 132724 | |
| Dialysis tubing closures | Spectrum Labs | 132738 | |
| Ultrafiltration units (concentrators) 10,000 NWCO | any | ||
| UV-Vis spectrophotometer | any | ||
| Name | Company | Catalog Number | Comments |
| Equipment (crystallography) | |||
| Low volume pipette 0.1-10 µl | any | ||
| Positive displacement pipette Microman M10 | Gilson | F148501 | |
| Crystallization robot | any | ||
| 96-well crystallization plates TTP IQ with three protein wells | TTP | 4150-05810 | or any other 96-well crystallization plate |
| 24-well CombiClover Junior Plate | Jena Bioscience | EB-CJR | |
| Crystal Clear Sealing Tape | Hampton Research | HR3-511 | |
| Siliconized Glass Cover Slides | Hampton Research | HR3-225 | |
| Commercial crystallization screens: SaltRx, Index, PEG/Ion, Crystal | Hampton Research | diverse | |
| Commercial crystallization screens: Wizard, PACT++, JCSG++ | Jena Bioscience | diverse | |
| JBS Beads-for-Seeds | Jena Bioscience | CO-501 | |
| CrystalCap SPINE HT (nylon loops) | Hampton Research | diverse | loop sizes 0.025 mm - 0.5 mm |
| CrystalCap Vial | Hampton Research | HR4-904 | |
| Cryogenic Foam Dewar 800 ml | Hampton Research | HR4-673 | |
| Cryogenic Foam Dewar 2 L | Hampton Research | HR4-675 | |
| Vial Clamp, Straight | Hampton Research | HR4-670 | |
| CrystalWand Magnetic, Straight | Hampton Research | HR4-729 | |
| CryoCane 6 Vial Holder | Hampton Research | HR4-711 | |
| CryoSleeve | Hampton Research | HR4-708 | |
| CryoCane Color Coder - White | Hampton Research | HR4-713 | |
| Scalpel | any | ||
| Straight microforcep | any | for manipulation of sealing tape. etc. | |
| Acupuncture needle | any | e.g. from a pharmacy | |
| Stereo microscope | any | for inspection of crystallization plates and crystal mounting, magnification up to 160X |
Riferimenti
- Bouza, E. Consequences of Clostridium difficile infection: understanding the healthcare burden. Clin Microbiol Infect. 18 (Suppl 6), 5-12 (2012).
- O'Connor, J. R., Johnson, S., Gerding, D. N. Clostridium difficile infection caused by the epidemic BI/NAP1/027 strain. Gastroenterology. 136 (6), 1913-1924 (2009).
- Mitchell, B. G., Gardner, A. Mortality and Clostridium difficile infection: a review. Antimicrob Resist Infect Control. 1 (1), (2012).
- George, W. L., Sutter, V. L., Finegold, S. M. Antimicrobial agent-induced diarrhea--a bacterial disease. J Infect Dis. 136 (6), 822-828 (1977).
- George, R. H., et al. Identification of Clostridium difficile as a cause of pseudomembranous colitis. Br Med J. 1 (6114), 695 (1978).
- Bartlett, J. G. Narrative review: the new epidemic of Clostridium difficile-associated enteric disease. Ann Intern Med. 145 (10), 758-764 (2006).
- Kelly, C. P., LaMont, J. T. Clostridium difficile--more difficult than ever. N Engl J Med. 359 (18), 1932-1940 (2008).
- Ünal, C. M., Steinert, M. Novel therapeutic strategies for Clostridium difficile infections. Expert Opin Ther Targets. 20 (3), 269-285 (2016).
- Kelly, C. P., et al. The Monoclonal Antibody, Bezlotoxumab Targeting C. difficile Toxin B Shows Efficacy in Preventing Recurrent C. difficile Infection (CDI) in Patients at High Risk of Recurrence or of CDI-Related Adverse Outcomes. Gastroenterology. 150 (4), S122 (2016).
- Tsutsumi, L. S., Owusu, Y. B., Hurdle, J. G., Sun, D. Progress in the discovery of treatments for C. difficile infection: A clinical and medicinal chemistry review. Curr Top Med Chem. 14 (1), 152-175 (2014).
- Hensbergen, P. J., et al. Clostridium difficile secreted Pro-Pro endopeptidase PPEP-1 (ZMP1/CD2830) modulates adhesion through cleavage of the collagen binding protein CD2831. FEBS Lett. 589 (24), 3952-3958 (2015).
- Cafardi, V., et al. Identification of a novel zinc metalloprotease through a global analysis of Clostridium difficile extracellular proteins. PLoS One. 8 (11), e81306 (2013).
- Hensbergen, P. J., et al. A novel secreted metalloprotease (CD2830) from Clostridium difficile cleaves specific proline sequences in LPXTG cell surface proteins. Mol Cell Proteomics. 13 (5), 1231-1244 (2014).
- Schacherl, M., Pichlo, C., Neundorf, I., Baumann, U. Structural Basis of Proline-Proline Peptide Bond Specificity of the Metalloprotease Zmp1 Implicated in Motility of Clostridium difficile. Structure. 23 (9), 1632-1642 (2015).
- Rawlings, N. D., Waller, M., Barrett, A. J., Bateman, A. MEROPS: the database of proteolytic enzymes, their substrates and inhibitors. Nucleic Acids Res. 42 (Release 10.0), D503-D509 (2014).
- Bergfors, T. Seeds to crystals. J Struct Biol. 142 (1), 66-76 (2003).
- Dauter, Z., Dauter, M., Dodson, E. Jolly SAD. Acta Crystallogr D Biol Crystallogr. 58 (Pt 3), 494-506 (2002).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Anal Biochem. 72, 248-254 (1976).
- Kabsch, W. XDS. Acta Crystallogr D Biol Crystallogr. 66 (Pt 2), 125-132 (2010).
- Adams, P. D., et al. PHENIX: a comprehensive Python-based system for macromolecular structure solution. Acta Crystallogr D Biol Crystallogr. 66, 213-221 (2010).
- Emsley, P., Lohkamp, B., Scott, W. G., Cowtan, K. Features and development of Coot. Acta Crystallogr D Biol Crystallogr. 66 (Pt 4), 486-501 (2010).
- . . The PyMOL Molecular Graphics System. , (2002).
- Pettersen, E. F., et al. UCSF Chimera--a visualization system for exploratory research and analysis. J Comput Chem. 25 (13), 1605-1612 (2004).
- Wang, B. C. Resolution of phase ambiguity in macromolecular crystallography. Methods Enzymol. 115, 90-112 (1985).
- McCoy, A. J., Grosse-Kunstleve, R. W., Adams, P. D., Winn, M. D., Storoni, L. C., Read, R. J. Phaser crystallographic software. J Appl Crystallogr. 40 (Pt 4), 658-674 (2007).
- Zwart, P. H., et al. Automated structure solution with the PHENIX suite. Methods Mol Biol. 426, 419-435 (2008).
- Zheng, H., Handing, K. B., Zimmerman, M. D., Shabalin, I. G., Almo, S. C., Minor, W. X-ray crystallography over the past decade for novel drug discovery - where are we heading next?. Expert Opin Drug Discov. 10 (9), 975-989 (2015).
- Rubino, J. T., et al. Structural characterization of zinc-bound Zmp1, a zinc-dependent metalloprotease secreted by Clostridium difficile. J Biol Inorg Chem. 21 (2), 185-196 (2016).
- Carson, M., Johnson, D. H., McDonald, H., Brouillette, C., Delucas, L. J. His-tag impact on structure. Acta Crystallogr D Biol Crystallogr. 63 (Pt 3), 295-301 (2007).
- Gasteiger, E., Walker, J. M., et al. Protein Identification and Analysis Tools on the ExPASy Server. The Proteomics Protocols Handbook. , 571-607 (2005).
- Dummler, A., Lawrence, A. M., de Marco, A. Simplified screening for the detection of soluble fusion constructs expressed in E. coli using a modular set of vectors. Microb Cell Fact. 4, 34 (2005).
- Stura, E. A., Wilson, I. A. Applications of the streak seeding technique in protein crystallization. J Crys Growth. 110 (1), 270-282 (1991).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon