È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Il trattamento di droga e
In questo articolo
Riepilogo
Small laboratory fish have become popular models for bone research on the mechanisms underlying human bone disorders and for the screening of bone-modulating drugs. In this report, we describe a protocol to assess the effect of alendronate on bone cells in medaka larvae with osteoporotic lesions.
Abstract
Che formano l'osso osteoblasti interagiscono con osteoclasti osso-riassorbono per coordinare il fatturato di matrice ossea e per controllare l'omeostasi scheletrica. Medaka e larve di pesce zebra sono ampiamente utilizzati per analizzare il comportamento delle cellule ossee durante la formazione ossea, degenerazione, e la riparazione. La loro chiarezza ottica permette la visualizzazione di cellule ossee fluorescenza etichettati e coloranti fluorescenti legati alla matrice mineralizzata scheletrico. Il nostro laboratorio ha generato pesci transgenici Medaka che esprimono il fattore di osteoclasti che induce Receptor Activator of Nuclear factor-kB Ligand (RANKL) sotto il controllo di un promotore di calore shock inducibile. Espressione ectopica di risultati RANKL in eccesso formazione degli osteoclasti attivati, che possono essere visualizzati in linee giornalista con espressione nlGFP sotto il controllo del K (ctsk) promotore catepsina. induzione RANKL e la formazione degli osteoclasti ectopica porta a gravi fenotipi osteoporosi-like. Composto transgenico Medaka Lines che esprimono ctsk: nlGFP in osteoclasti, nonché mCherry sotto il controllo del Osterix (osx) promotore in osteoblasti prematuri, possono essere utilizzati per studiare l'interazione di entrambi i tipi di cellule. Questo facilita l'osservazione in vivo del comportamento cellulare in condizioni di degenerazione e riparazione ossea. Qui, descriviamo l'uso di questo sistema per testare un farmaco comunemente utilizzato nella terapia dell'osteoporosi umana e descrivere un protocollo per l'imaging in tempo reale. Il modello Medaka integra studi in colture cellulari e topi, e offre un nuovo sistema per l'analisi in vivo di azione farmaco nel sistema scheletrico.
Introduzione
Lo scheletro vertebrato fornisce supporto strutturale e protezione per organi, permette la mobilità, e serve come fonte di calcio. Per tutta la vita, la matrice ossea extracellulare viene continuamente girato per mantenere la stabilità ossea e rigidità. Questo processo richiede l'attività strettamente coordinato e interazione degli osteoblasti che formano l'osso e osteoclasti osso-riassorbono. Gli osteoblasti derivano da progenitori mesenchimali multipotenti e producono collagene per formare il osteoide, la parte proteica della matrice ossea 10. Osteoblasti interagiscono con gli osteoclasti per realizzare un'attività equilibrata di entrambi i tipi di cellule, che è richiesto per controllare l'omeostasi ossea 7. A causa di queste interazioni normative complesse, risposte al trattamento farmacologico e l'omeostasi ossea non possono essere completamente esaminati utilizzando studi in vitro. Quindi, vi è una forte richiesta di modelli animali. Rispetto alle impostazioni di coltura cellulare, in modelli in vivo può fornireinformazioni preziose nelle reti pluricellulari all'interno dell'ambiente osso.
Esistono numerosi modelli di mouse per una varietà di malattie delle ossa umane, tra cui l'osteoporosi 16. Tuttavia, la dimensione e l'accessibilità di embrioni di topo rappresentano significative limitazioni per l'imaging dal vivo di processi scheletrici. Piccoli pesci teleostei, invece, servire come un'alternativa interessante per l'imaging in vivo. Zebrafish (Danio rerio) e Medaka (Oryzias latipes) sono diventati popolari modelli animali per la ricerca scheletrico nel corso degli ultimi due decenni, 17, 19, 22, 24. Bone nei pesci teleostei e nei mammiferi è molto simile, sia strutturale che a livello fisiologico, e molti dei geni regolatori chiave e vie di segnalazione sono conservati 3. Come nei mammiferi, pesci teleosteo regolare accuratamente l'attività degli osteoblasti e osteoclasti per bilanciare la formazione dell'osso ed il riassorbimento 26. Ancora più importante, la chiarezza ottica di fish larve permette l'utilizzo di reporter fluorescenti per marcare le cellule ossee e calcificate matrice scheletrico 8, 9, 12, 21, 23, che facilita l'osservazione dei processi cellulari in animale vivente. Inoltre, una serie di strumenti genetici è stato generato per facilitare la ricerca biomedico rilevante nel pesce. Per Medaka in particolare, i metodi per mirata mutazione genetica da CRISPR / Cas9 2, cellule-lineage tracing 6, e transgenesi 14 sono state di recente costituzione e sono ora ampiamente in uso 15 site-specific.
Piccole larve teleosteo sono stati utilizzati con successo per gli schermi chimici, che hanno portato alla scoperta di diversi farmaci farmacologicamente rilevanti 1, 18.
Pesce larve sono tolleranti a basse concentrazioni di DMSO e sono in grado di assorbire composti dal loro ambiente acquatico, sia attraverso la pelle o attraverso il tratto gastrointestinale 1, 5. Il nostro laboratorio in precedenza rappresentanteorted linee Medaka transgenici che esprimono i reporter fluorescenti in cellule ossee sotto il controllo di vari osteoblasti e promotori specifici osteoclasti. Questi includono osteoblasti prematuri (collagene 10A1, col10a1; Osterix, OSX) 20, 21, osteoblasti maturi (osteocalcina, OSC) 27, e osteoclasti (catepsina K, ctsk) 24. Abbiamo anche generato una linea transgenica che esprime il fattore di osteoclasti che induce Receptor Activator of Nuclear factor-kB Ligand (RANKL) sotto il controllo di un shock termico-inducibile promotore 24.
Induzione di RANKL in questo sistema porta alla formazione ectopica di osteoclasti attivi. Questo porta ad un aumento riassorbimento osseo e una grave fenotipo osteoporosi simile, con drasticamente ridotta mineralizzazione nei corpi vertebrali. Abbiamo recentemente dimostrato che l'attività degli osteoclasti in questo modello può essere bloccato dalla bifosfonati etidronato e alendronato, TWO farmaci comunemente utilizzati nella terapia dell'osteoporosi umana, convalidando così Medaka come sistema modello adatto per l'osteoporosi 27.
Grazie alla loro grande prole, rapido sviluppo, e piccole dimensioni di embrioni, transgenici Medaka larve sono particolarmente adatta per lo screening su larga scala di farmaci osteoporosi e per l'analisi in vivo del comportamento delle cellule ossee. Gli studi in Medaka quindi possono integrare in modo efficiente esperimenti in colture cellulari e nei topi che hanno lo scopo di scoprire nuovi bersagli terapeutici e nuove terapie per i disturbi delle ossa umane.
Nel presente studio, si descrive un protocollo per il trattamento di Medaka larve osso-giornalista con il farmaco osteoporosi comuni, l'alendronato. Descriviamo anche in dettaglio come trattate le larve sono montati e preparati per l'imaging dal vivo di matrice ossea e cellule ossee. Questi protocolli possono essere facilmente adattate ad altri composti chimici di piccole dimensioni che sia il lavoro come anabolizzanti osso o droghe riassorbimento osseo.
Access restricted. Please log in or start a trial to view this content.
Protocollo
Tutti gli esperimenti sono stati eseguiti in conformità con i protocolli Istituzionali Animal Care e del Comitato Usa (IACUC) approvato per l'Università Nazionale di Singapore (R14-293).
1. I pesci allevamento e alla raccolta di embrioni
- Sollevare WT, ctsk: nlGFP 24, RANKL: HSE: CFP 24, e OSX: mCherry 21 mono o pesce Medaka compound-transgenici a 26 ° C sotto un ciclo di luce controllata (14 h, 10 h scuro) per indurre la deposizione delle uova.
- la deposizione delle uova quotidiana si svolge durante i primi 30 minuti dopo che la luce è accesa. Le uova si attaccano insieme attraverso filamenti e attribuiscono al ventre della femmina per diverse ore. Utilizzare una rete a maglia fine per la cattura di un adulto di sesso femminile che trasporta un cluster di uovo. Lasciate che il pesce riposare per breve tempo in rete e poi massaggiare delicatamente l'addome del pesce per mettere a nudo con attenzione il cluster ovulo fecondato dall'addome della femmina.
NOTA: Una sana Medaka femminilepuò produrre 10 - 20 uova al giorno per circa 5 mesi. - Mettere le uova in una plastica Petri 60 millimetri. Utilizzare una pipetta di plastica per lavare gli embrioni con 5 - 10 ml di 0.3x Danieau del medium soluzione (pesce; 19,3 mM NaCl, 0,23 mm KCl, 0,13 mm MgSO 4, 0,2 mM Ca (NO 3) 2 e 1,7 mm HEPES, pH 7.0). Aggiungere 1 ml di 0,25% (w / v) di metilene blu stock di 2,5 L di medio pesce per prevenire la crescita fungina.
- rotolare delicatamente grappolo di uova in modo da formare un nodo di filamenti di attacco. Usare pinze per rimuovere con attenzione i filamenti allegato dal cluster ovulo fecondato per ottenere singoli embrioni (Figura 1A).
- Fase gli embrioni in base al Iwamatsu 2004 13.
- Cultura 20 - 30 embrioni per 60 millimetri di plastica Petri in un incubatore a 28 °. Cambiare la media giornaliera di garantire il normale sviluppo degli embrioni.
NOTA: il tempo attorno al palco schiusa (8-9 d postfertilization,DPF) è particolarmente critica per la sopravvivenza. Rimuovere chorions libero di fluttuare per mantenere la media pulito e per garantire buoni tassi di sopravvivenza delle larve.
2. Screening embrioni transgenici
- Utilizzare uno stereomicroscopio dotato di una lampada al mercurio per l'imaging di fluorescenza e GFP, RFP, e filtri CFP per lo screening degli embrioni transgenici per l'espressione reporter fluorescente utilizzando 40X di ingrandimento.
- Visivamente identificare osx: mCherry embrioni di espressione giornalista mCherry nei primi anni di formazione ossa craniche, come il cleithrum, su entrambi i lati della testa posteriore (Figura 1B, freccia), e il parasphenoid, in una posizione centrale nel cranio ventrale ( Figura 1B, punta di freccia).
NOTA: l'espressione Reporter inizia dal 5 in poi DPF 21. - Identificare ctsk: nlGFP embrioni di forte espressione nlGFP nella testa (Figura 1C, freccia) e la coda (Figura 1C, punta di freccia), a partire da6 DPF.
NOTA: osteoclasti endogeni formano solo dopo 21 DPF. nlGFP-cellule che esprimono in questa fase iniziale (6 DPF) non sono osteoclasti, ma altri, finora non caratterizzato, ctsk cellule -positive 24. - Identificare RANKL: HSE: PCP embrioni transgenici di espressione ubiquitaria PCP dopo un breve trattamento termico d'urto per 20 minuti a 39 ° C, condotto presso 2 DPF o poi ai fini dello screening.
NOTA: Il RANKL e CFP transgeni sono sotto il controllo dello stesso bidirezionale Heat Shock Element (HSE). Espressione PCP indica successo RANKL induzione 24. - Eseguire un 1,5-2 h termico trattamento d'urto a 9 DPF o poi per indurre un gran numero di osteoclasti ectopiche nella regione del tronco, che si traduce di conseguenza in un fenotipo osteoporosi simile 24.
NOTA: espressione RANKL transgenico indotta a 9 risultati DPF in una attivazione ectopica di cellule progenitrici degli osteoclasti dormienti, che endogeno non viene attivatoprima del 21 DPF. Usare un bagno d'acqua per ottenere stabili 39 ° C condizioni. Lasciate che i Petri contenenti embrioni Medaka galleggiano sulla superficie dell'acqua. Assicurarsi che il coperchio della scatola di Petri è asciutto per evitare l'affondamento del piatto. - Schermo embrioni da linee di composti, come RANKL: HSE: CFP / ctsk: nlGFP doppio transgenico e OSX: mCherry / RANKL: HSE: CFP / ctsk: nlGFP triplo transgenico, secondo il pattern di espressione di ogni individuo transgene.
NOTA: gli embrioni transgenici omozigoti emizigote e sono stati contraddistinti da diversi livelli di fluorescenza del transgene giornalista. embrioni omozigoti avevano una intensità di fluorescenza che è risultata approssimativamente raddoppiata rispetto a quella dei transgenici hemizygous. Linee di composti che erano omozigoti per entrambi RANKL: HSE: PCP e ctsk: nlGFP sono stati ottenuti incrossing ripetuto per diverse generazioni. Per triplo transgenico OSX: mCherry / RANKL: HSE: CFP / ctsk: nlGFP pesce, RANKL omozigote: HSE: CFP / ctsk: pesce nlGFP sono stati incrociati con OSX omozigote: vettori mCherry. La progenie triple-transgenici eterozigoti risultante sono state sollevate e incrossed di ottenere embrioni omozigoti per RANKL: HSE: CFP. Il RANKL: HSE: PCP transgene deve essere omozigote per ottenere l'induzione efficiente degli osteoclasti ectopiche.
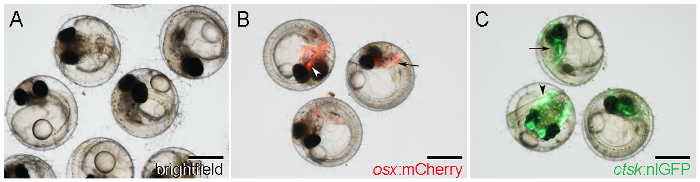
Figura 1: WT e transgenici Medaka embrioni a 7 D Postfertilization (DPF). A. embrioni WT osservati con illuminazione campo chiaro. B. Embrioni transgenici che mostrano OSX: espressione mCherry intorno al cleithrum (freccia) e parasphenoid (punta di freccia). C. Embrioni transgenici che mostrano ctsk: espressione nlGFP nella testa (freccia) e la coda (punta di freccia). Barre di scala: 500 micron.025 / 55025fig1large.jpg "target =" _ blank "> Clicca qui per vedere una versione più grande di questa figura.
3. trattamento con i bifosfonati di Medaka larve
- Preparare soluzioni contenenti diverse concentrazioni dei bifosfonati (BPS) per gli studi dose-risposta.
NOTA: La BP esemplare utilizzato in questo protocollo è l'alendronato.- Sciogliere alendronato in mezzo pesce ad una concentrazione di 100 ug / ml per preparare una soluzione madre.
- Utilizzare un vortex per garantire la completa dissoluzione. Conservare la soluzione di riserva a 4 ° C.
- Preparare diverse soluzioni di lavoro diluendo la soluzione madre con il mezzo pesce per una serie di concentrazioni (cioè, 25, 37,5, 50, 62,5 e 75 mg / mL).
NOTA: Diversi farmaci possono avere diverse assorbimento, distribuzione, metabolismo ed escrezione (ADME parametri), che devono essere considerati durante i test con questo sistema larve Medaka. Inoltre, la solubilità della droga e stabilità possono variare quandoapplicata come soluzione acquosa. Meno composti idrosolubili potrebbero dover prima essere sciolto in solventi organici, come DMSO. In questo caso, una soluzione madre viene preparata in DMSO, che viene poi ulteriormente diluito in terreno pesci. Si noti che le soluzioni di lavoro in acqua (media) pesci possono essere conservati in frigorifero per diversi sett. Tuttavia, le soluzioni contenenti DMSO devono essere conservati a temperatura ambiente per evitare la cristallizzazione.
- Trasferimento Medaka larve in piastre a sei pozzetti (sei larve / e) per il successivo trattamento BP (alendronato).
- Rimuovere il mezzo di pesce con cura utilizzando una pipetta di plastica pulito e aggiungere un piccolo volume (circa. 0,5 mL) di soluzione alendronato in ogni pozzetto.
- Evitare medio pesce rimasto, come la soluzione di BP aggiunto potrebbe essere diluito, il che è particolarmente importante per le soluzioni alendronato meno concentrate.
- Rimuovere un piccolo volume di soluzione di alendronato (fino a 0,5 mL) da ogni pozzetto con una pipetta di plastica pulito e sostituirlo con un volu grandeme (4 mL) di soluzione alendronato.
- Cambiare media quotidianamente per garantire il normale sviluppo degli embrioni.
4. La colorazione dal vivo di matrice mineralizzata con l'osso
- Sciogliere 0.5 g di alizarina complessone (ALC, acido alizarina-3-methyliminodiacetic) o 0,05 g di calceina in 50 ml di terreno di pesce per preparare soluzioni madre 1% e 0,1% rispettivamente. Utilizzare un vortex per garantire la completa dissoluzione.
NOTA: media Pesce senza l'aggiunta di blu di metilene è utilizzato in questa e nelle fasi successive per ridurre autofluorescenza nelle larve. - Utilizzare un filtro siringa e monouso (0,2 micron) per filtrare la soluzione colorante. Conservare la soluzione filtrata al buio a temperatura ambiente.
NOTA: Il colore della, chiara soluzione colorante ALC filtrato è di colore giallo scuro all'arancio. Il colore della soluzione calceina chiaro filtrato è di colore giallo brillante. Le soluzioni possono essere utilizzati per diversi mesi. - Diluire la soluzione filtrata ALC o calceina magazzino 1:10 in media pescee incubare le larve Medaka per 1,5 - 2 ore (0,1% soluzione ALC) o 2 - 2,5 h (0,01% soluzione calceina) in un incubatore a 28 ° se si utilizzano le larve tra il 9 e il 17 DPF. Conservare i campioni al buio.
- Trasferire le larve medio pesce fresco con una pipetta di plastica pulito.
- Rimuovere il supporto di pesce con una pipetta di plastica pulito e aggiungere mezzo di pesce fresco. Ripetere questo passaggio per 3 - 4 volte fino a quando nessuna soluzione rossi o giallo-macchiato (ALC o calceina, rispettivamente) è rimasto sopra. Lasciare le larve in mezzo pesce per 30 - 60 min prima di montaggio per l'imaging di evitare epifluorescenza dal mezzo.
NOTA: 0,1% soluzione ALC colorazione è dannoso per Medaka larve per tempi di esposizione prolungati. I tempi di incubazione di più di 2 h influenzano la sopravvivenza delle larve. Concentrazione e tempo di colorazione devono pertanto essere ottimizzato per diverse fasi, al fine di ottenere risultati ottimali di sopravvivenza degli embrioni e di colorazione.
5. vivo imaging di fluorescenza p>
- Anestetizzare le larve Medaka con 0,01% Tricaine (etil metanosolfonato 3-aminobenzoate) in media del pesce.
NOTA: anestetizzati larve diventano immobilizzato dopo 5 - 10 minuti in soluzione Tricaine e di solito sono distesi o su un fianco o le spalle. - Utilizzare un microloader plastica per orientare le larve in base alla regione di interesse. L'orientamento delle larve utilizzato in questo protocollo è laterale.
- Utilizzare uno stereomicroscopio con illuminazione a fluorescenza per l'imaging. Utilizzare alto ingrandimento quando si scattano immagini, concentrandosi su diverse parti della larve (testa, tronco anteriore, il tronco posteriore, e la coda). Stitch singole immagini insieme alla sovrapposizione delle regioni utilizzando un apposito software di elaborazione delle immagini (inserti in Figura 3G).
NOTA: Ciò contribuisce a migliorare la qualità di immagine di tutte le parti del corpo interessate nel piano focale corretto. - Rientro le larve al mezzo di pesce per il recupero dopo l'imaging.
- Anestetizzare le larve con 0,01% Tricaine in media pesce per 5 - 10 minuti fino a quando diventano immobilizzato.
- Sciogliere bassofondente agarosio all'1,5% in media pesci riscaldandolo in un forno a microonde. Raffreddare la soluzione a circa 30 ° C.
- Aggiungere 0,5-1 ml di liquido 1,5% basso punto di fusione agarosio in media pesce per una capsula di Petri con fondo trasparente. Trasferire le larve anestetizzato nella soluzione con una pipetta di plastica pulito.
NOTA: prendere precauzioni speciali che la temperatura del liquido agarosio bassofondente è abbastanza basso da non danneggiare le larve. - Prima solidifica agarosio, utilizzare un microloader plastica per spingere le larve al fondo del piatto Petri e orientare le larve in base alla regione di interesse. L'orientamento delle larve utilizzato in questo protocollo è laterale.
NOTA: I campioni sono pronti per confocale l'imaging dal vivo dopo l'agarosio solidifica completamente. - Utilizzare un microscopio confocale a ACQimmagini uire.
- Utilizzare una linea laser 543 nm per le analisi mCherry e ALC colorazione. Utilizzare una linea laser 488 nm per nlGFP e analizza calceina colorazione.
- Dopo l'imaging, aggiungi medio pesce alla piastra di Petri e utilizzare un paio di aghi sottili siringa (27 G x 1 ½ ") per rimuovere con attenzione le larve dal agarosio. Trasferire le larve con residuale attaccato agarosio ad una capsula di Petri con il mezzo di pesce per il recupero .
- Elaborare le immagini utilizzando una immagine software di analisi 27.
Access restricted. Please log in or start a trial to view this content.
Risultati
numeri uovo abbondanti, così come le piccole dimensioni delle larve, fanno Medaka un modello eccellente per lo screening farmacologico. Una singola piastra di sei pozzetti è stato usato per coltura fino a 36 larve, sufficiente a fornire dati statisticamente significativi. Un altro grande vantaggio di utilizzare pesce per analisi scheletrica è la possibilità di fare imaging dal vivo. La trasparenza delle larve consente l'uso di proteine fluorescenti per marcare le cellule ossee, nonché l'uso di coloran...
Access restricted. Please log in or start a trial to view this content.
Discussione
I passaggi critici all'interno del protocollo
È essenziale che le condizioni per il trattamento shock termico sono coerenti e stabili quando si confrontano i diversi campioni. Condizioni di temperatura stabili garantiscono livelli simili di RANKL induzione nelle larve transgeniche e, di conseguenza, la formazione di osteoclasti comparabili, che può essere confermata da screening per ctsk: espressione nlGFP. In definitiva, questo porta ad un grado simile di riassorbimento osseo in...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori dichiarano di non avere interessi in competizione o finanziari.
Riconoscimenti
Questo progetto è stato finanziato da sovvenzioni dal Ministero della Pubblica Istruzione di Singapore (MOE, codice di autorizzazione 2013-T2-2-126) e l'Istituto Nazionale della Salute, Stati Uniti d'America (NIH, concedere numero 1R21AT008452-01A1). TY ha ricevuto una borsa di studio di laurea del Dipartimento di Scienze Biologiche NUS. Ringraziamo l'unità confocale del Centro NUS per Bioimmagini Sciences (CBIS) per il loro costante supporto.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Alendronate | Sigma | A4978 | |
| alizarin-3-methyliminodiacetic acid, Alizarin Complexone | Sigma | A3882 | |
| Calcein | Sigma | C0875 | |
| ethyl 3-aminobenzoate methanesulfonate (Tricaine) | Sigma | A5040 | |
| ImageJ (1.4.3.67) | National Institute of Health (NIH) | https://imagej.nih.gov/ij/ | |
| LSM 510 Meta confocal | Zeiss | ||
| LSM Image Browser (4.2.0.121) | Zeiss | http://www.zeiss.com/microscopy/en_de/downloads/lsm-5-series.html | |
| Micro-loader | Eppendorf | 5242956003 | Eppendorf ep T.I.P.S 20 μL |
| NIS-Elements BR 3.0 software | Nikon | ||
| Photoshop CS6 (13.0.0.0) | Adobe | ||
| SMZ1000 stereomicroscope | Nikon |
Riferimenti
- Ablain, J., Zon, L. I. Of fish and men: using zebrafish to fight human diseases. Trends Cell Biol. 23 (12), 584-586 (2013).
- Ansai, S., Kinoshita, M. Targeted mutagenesis using CRISPR/Cas system in medaka. Biol Open. 3 (5), 362-371 (2014).
- Apschner, A., Schulte-Merker, S., Witten, P. E. Not all bones are created equal-using zebrafish and other teleost species in osteogenesis research. Methods Cell Biol. 105, 239-255 (2011).
- Bajoghli, B., Aghaallaei, N., Heimbucher, T., Czerny, T. An artificial promoter construct for heat-inducible misexpression during fish embryogenesis. Dev Biol. 271 (2), 416-430 (2004).
- Barrett, R., Chappell, C., Quick, M., Fleming, A. A rapid, high content, in vivo model of glucocorticoid-induced osteoporosis. Biotechnol J. 1 (6), 651-655 (2006).
- Centanin, L., Ander, J. J., Hoeckendorf, B., Lust, K., Kellner, T., Kraemer, I., Urbany, C., Hasel, E., Harris, W. A., Simons, B. D., et al. Exclusive multipotency and preferential asymmetric divisions in post-embryonic neural stem cells of the fish retina. Development. 141 (18), 3472-3482 (2014).
- Charles, J. F., Aliprantis, A. O. Osteoclasts: more than 'bone eaters. Trends Mol Med. 20 (8), 449-459 (2014).
- DeLaurier, A., Eames, B. F., Blanco-Sanchez, B., Peng, G., He, X., Swartz, M. E., Ullmann, B., Westerfield, M., Kimmel, C. B. Zebrafish sp7:EGFP: a transgenic for studying otic vesicle formation, skeletogenesis, and bone regeneration. Genesis. 48 (8), 505-511 (2010).
- Du, S. J., Frenkel, V., Kindschi, G., Zohar, Y. Visualizing normal and defective bone development in zebrafish embryos using the fluorescent chromophore calcein. Dev Biol. 238 (2), 239-246 (2001).
- Eriksen, E. F. Cellular mechanisms of bone remodeling. Rev Endocr Metab Disord. 11 (4), 219-227 (2010).
- Hockendorf, B., Thumberger, T., Wittbrodt, J. Quantitative analysis of embryogenesis: a perspective for light sheet microscopy. Dev Cell. 23 (6), 1111-1120 (2012).
- Inohaya, K., Takano, Y., Kudo, A. The teleost intervertebral region acts as a growth center of the centrum: in vivo visualization of osteoblasts and their progenitors in transgenic fish. Dev Dyn. 236 (11), 3031-3046 (2007).
- Iwamatsu, T. Stages of normal development in the medaka Oryzias latipes. Mech Dev. 121 (7), 605-618 (2004).
- Kirchmaier, S., Hockendorf, B., Moller, E. K., Bornhorst, D., Spitz, F., Wittbrodt, J. Efficient site-specific transgenesis and enhancer activity tests in medaka using PhiC31 integrase. Development. 140 (20), 4287-4295 (2013).
- Kirchmaier, S., Naruse, K., Wittbrodt, J., Loosli, F. The genomic and genetic toolbox of the teleost medaka (Oryzias latipes). Genetics. 199 (4), 905-918 (2015).
- Komori, T. Animal models for osteoporosis. Eur J Pharmacol. 759, 287-294 (2015).
- Mackay, E. W., Apschner, A., Schulte-Merker, S. A bone to pick with zebrafish. Bonekey Rep. 2, 445(2013).
- MacRae, C. A., Peterson, R. T. Zebrafish as tools for drug discovery. Nat Rev Drug Discov. 14 (10), 721-731 (2015).
- Mitchell, R. E., Huitema, L. F., Skinner, R. E., Brunt, L. H., Severn, C., Schulte-Merker, S., Hammond, C. L. New tools for studying osteoarthritis genetics in zebrafish. Osteoarthritis Cartilage. 21 (2), 269-278 (2013).
- Renn, J., Buttner, A., To, T. T., Chan, S. J., Winkler, C. A col10a1:nlGFP transgenic line displays putative osteoblast precursors at the medaka notochordal sheath prior to mineralization. Dev Biol. 381 (1), 134-143 (2013).
- Renn, J., Winkler, C. Osterix-mCherry transgenic medaka for in vivo imaging of bone formation. Dev Dyn. 238 (1), 241-248 (2009).
- Schilling, T. F., Kimmel, C. B. Segment and cell type lineage restrictions during pharyngeal arch development in the zebrafish embryo. Development. 120 (3), 483-494 (1994).
- Spoorendonk, K. M., Peterson-Maduro, J., Renn, J., Trowe, T., Kranenbarg, S., Winkler, C., Schulte-Merker, S. Retinoic acid and Cyp26b1 are critical regulators of osteogenesis in the axial skeleton. Development. 135 (22), 3765-3774 (2008).
- To, T. T., Witten, P. E., Renn, J., Bhattacharya, D., Huysseune, A., Winkler, C. Rankl-induced osteoclastogenesis leads to loss of mineralization in a medaka osteoporosis model. Development. 139 (1), 141-150 (2012).
- Wakamatsu, Y., Pristyazhnyuk, S., Kinoshita, M., Tanaka, M., Ozato, K. The see-through medaka: a fish model that is transparent throughout life. Proc Natl Acad Sci USA. 98 (18), 10046-10050 (2001).
- Witten, P. E., Huysseune, A. A comparative view on mechanisms and functions of skeletal remodelling in teleost fish, with special emphasis on osteoclasts and their function. Biol Rev Camb Philos Soc. 84 (2), 315-346 (2009).
- Yu, T., Witten, P. E., Huysseune, A., Buettner, A., To, T. T., Winkler, C. Live imaging of osteoclast inhibition by bisphosphonates in a medaka osteoporosis model. Dis Model Mech. 9 (2), 155-163 (2016).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon