Method Article
Una piattaforma cellulare microarray high-throughput per l'analisi correlativa cellulare Differenziazione e forze di trazione
In questo articolo
Riepilogo
differenziazione delle cellule è regolata da una serie di fattori microambientali, tra cui entrambe le proprietà composizione della matrice e materiale del substrato. Descriviamo qui una tecnica che utilizza microarrays di cellule in combinazione con una forza di trazione microscopio per valutare sia la differenziazione cellulare e biomeccaniche interazioni cellula-substrato in funzione del contesto microambientali.
Abstract
microarrays cellulari microfabbricazione, che consistono in combinazioni di contatti stampati di biomolecole su una superficie elastica idrogel, forniscono un elevato throughput di sistema ingegnerizzato strettamente controllato per misurare l'impatto dei segnali biochimici schierati sulla differenziazione delle cellule. I recenti sforzi utilizzando microarray di cellule hanno dimostrato la loro utilità per gli studi combinatorie in cui molti fattori microambientali sono presentati in parallelo. Tuttavia, questi sforzi si sono concentrati principalmente sulla studio degli effetti di segnali biochimici su risposte delle cellule. Qui vi presentiamo una piattaforma microarray cella con le proprietà del materiale regolabili per valutare sia la differenziazione cellulare mediante immunofluorescenza e biomeccanico cellula-substrato interazioni con la forza di trazione microscopia. Per fare questo, abbiamo sviluppato due diversi formati utilizzando idrogel di poliacrilamide di varia il modulo di Young fabbricato su entrambi i vetrini da microscopio o piatti con fondo di vetro Petri. Forniamo best pratiche e risoluzione di problemi per la realizzazione di microarray su questi substrati idrogel, la successiva coltura cellulare su microarray, e l'acquisizione di dati. Questa piattaforma è particolarmente adatto per l'utilizzo nelle indagini di processi biologici per i quali sia biochimica (ad esempio, la composizione della matrice extracellulare) e biofisica (ad esempio, il substrato rigidità) spunti possono svolgere significativo, si intersecano ruoli.
Introduzione
Le interazioni tra cellule e fattori circostanti microambiente mediare una grande varietà di processi biologici durante lo sviluppo, l'omeostasi, e patogenesi della malattia 1, 2, 3, 4. Queste interazioni microambientali includono la consegna di fattori solubili alle cellule, vincolante cellula-matrice, e le interazioni cellula-cellula tramite legame ligando-recettore. In aggiunta alle considerazioni biochimiche precedenti, i parametri biofisici, come le proprietà meccaniche del substrato (ad esempio, il modulo di Young, porosità) e la forma delle cellule, e meccanotrasduzione valle associati hanno sempre ottenuto il riconoscimento come mediatori chiave del differenziamento cellulare 5, 6, 7, 8, 9 , 10. I segnali derivanti da tali interazioni microambientali servono come input per reti geniche e vie di segnalazione. Inoltre, questi componenti cellulari intrinseca anche fornire un feedback al microambiente tramite fattori secreti ed enzimi che degradano matrice, completando un ciclo di coregolamentazione complesso tra i programmi genetici di cellule-intrinseca e dei fattori del microambiente delle cellule-estrinseca 5, 11, 12.
L'utilizzo di sistemi ingegnerizzati per la presentazione controllata di fattori microambientali è dimostrato utile in una varietà di contesti diversi 13, 14, 15. Sistemi di microfabbricazione, in particolare, hanno facilitato preciso patterning spaziale delle proteine e cellule, nonché l'analisi altamente parallelo tramite la miniaturizzazione 13, 16, 17, 18, 19, 20, 21, 22. Microarrays cellulari rappresentano un tale sistema microfabbricazione in cui combinazioni di biomolecole sono contatti stampati su una poliacrilammide idrogel elastica substrato 23, 24, 25. L'inclusione di componenti cellulari-adesivo (cioè proteine della matrice) consente l'adesione sostenuta delle cellule e la cultura su microarray, che è spesso seguita da analisi a valle tramite immunocitochimica e giornalisti fluorescenti. Microarrays cellulari sono stati produttivo orientato verso il raggiungimento di una migliore comprensione del fenotipo delle cellule del fegato 23, 26, neural precursore differenziazione 27, Mammardecisioni y progenitore destino 28, sulle cellule staminali embrionali di manutenzione / differenziazione 23, 29, 30, cancro al polmone metastasi 31, e la risposta terapeutica nel melanoma 32. Abbiamo recentemente dimostrato l'utilizzo di microarray cellulari per definire il ruolo di matrice extracellulare (ECM) composizione proteica nella specifica endoderma 33, fegato progenitore differenziazione 34, 35, e tumori polmonari risposta farmacologica cellule 36. In questi lavori, ci siamo concentrati sulle estendere le capacità combinatorie della piattaforma matrice e ad esplorare le intersezioni di segnalazione cellulare-intrinseco con la composizione della matrice extracellulare e della biomeccanica. Inoltre, abbiamo implementato letture biofisici in questa piattaforma matrice per fornire la capacità di quantitativamente caratterize il ruolo della contrattilità cellulare nel differenziamento elabora 35. Per fare ciò, abbiamo integrato la forza di trazione microscopia (TFM) con microarray cellulari per consentire la valutazione high-throughput di trazione cellule generate. TFM è un metodo ampiamente utilizzato per misurare le forze di trazione cellule generate e fornisce analisi significative circa il coordinamento di singola cellula e la funzione a livello dei tessuti con la composizione e biomeccanica del microambiente locale 37, 38, 39, 40. Così, combinando TFM con microarray cellulari fornisce un sistema high-throughput per la misura chiave, fisiologicamente rilevanti parametri biofisici.
La piattaforma microarray cellule qui descritto si compone di quattro sezioni: la fabbricazione di substrati poliacrilammide, fabbricazione di array, colture cellulari e test di lettura e analisi dei dati. VedereFigura 1 per una sintesi schematica delle prime tre sezioni sperimentali; vedi figura 2 per una sintesi schematica della sezione finale con una particolare attenzione per l'analisi dei dati di immunofluorescenza. Al fine di adattare la piattaforma microarray cella per studi di biomeccanica interazioni cellula-substrato, abbiamo utilizzato substrati poliacrilammide di sintonizzabile modulo di Young ma porosità simili, per Wen et al. 41. Per consentire misurazioni TFM delle forze esercitate da cellule sulla loro substrato, abbiamo implementato un Petri formato piatto fondo di vetro in aggiunta al microscopio in vetro spessore diapositive frequentemente utilizzata da altri gruppi. Così, questa piattaforma microarray cellulare è in grado di misurazioni parallele di differenziazione cellulare tramite immunofluorescenza su vetrini da microscopio e le forze di cellule generate tramite TFM sui piatti con fondo di vetro separate. Abbiamo inoltre applicato diversi miglioramenti per l'approccio analitico comunemente utilizzato con microarrays di cellule. specificalmente, invece di parametrico Z-scoring di intensità complessiva dell'isola, si misura l'intensità cella singola e applicare la normalizzazione quantile al fine di tenere conto di distribuzioni non normali e più accuratamente descrivere il comportamento cellulare. Riteniamo che questi miglioramenti forniscono particolare utilità nei confronti indagini dei processi biologici in cui spunti sia biochimici e biofisici giocano importanti ruoli, che si intersecano. Inoltre, i nostri miglioramenti analitici consentono l'applicazione di microarray cellulari per gli studi di una serie di funzioni cellulari per i quali a cella singola e il comportamento a livello di popolazione divergono.
Protocollo
1. Realizzazione di poliacrilammide Substrati
- substrati di vetro pulito - sia vetrini da microscopio standard per immunofluorescenza endpoint o con fondo di vetro da 35 mm di Petri per TFM - al fine di garantire ottimale poliacrilammide idrogel fabbricazione e l'integrità durante la coltura delle cellule. In alternativa, l'uso di pre-pulizia substrati di vetro.
- Immergere substrati di vetro in 0,25% v / v Triton X-100 in acqua distillata (dH 2 O). Posizionare substrati su un agitatore orbitale per 30 min.
- Rimuovere Triton X-100 soluzione e sciacquare substrati 5 volte con dH 2 O. Lascia substrati immersi nel risciacquo finale e posto su un agitatore orbitale per 30 min.
- Rimuovere dH 2 O e immergere substrati in acetone. Posizionare substrati su un agitatore orbitale per 30 min.
- Rimuovere acetone ed immergere substrati in metanolo. Posizionare substrati su un agitatore orbitale per 30 min.
- Rimuovere metanolo e risciacquare substrati 5 volte con dH 2 substrati O. immergersi in 0,05 N NaOH e posto su un agitatore orbitale per 1 h.
ATTENZIONE: NaOH è molto caustico e può causare gravi ustioni cutanee e gravi lesioni oculari. Indossare guanti, indumenti e occhiali di protezione. - Rimuovere la soluzione NaOH e risciacquare substrati 5 volte con dH 2 O. Utilizzare aria compressa filtrata per asciugare substrati e cuocere in forno a 110 ° C su una piastra calda fino a secco (5 - 15 min). substrati puliti possono essere conservati a temperatura ambiente a tempo indeterminato.
- Silanizzazione substrati di vetro pulito per garantire il fissaggio del idrogel poliacrilammide.
- Immergere substrati di vetro pulite in preparati al 2% v / v 3- (trimetossisilil) propil metacrilato (3-TPM) in etanolo. Posizionare substrati su un agitatore orbitale per 30 min.
ATTENZIONE: 3-TPM è un liquido infiammabile. Tenere lontano da fonti di calore, scintille, fiamme libere e superfici calde e utilizzare solo in una cappa. - Rimuovere la soluzione 3-TPM e immergere substrati in ethanol. Posizionare substrati su un agitatore orbitale per 5 min.
- Utilizzare aria compressa filtrata per asciugare i substrati e cuocere a 110 ° C su una piastra calda fino a secco (5 - 15 min). substrati silanizzata possono essere conservati a temperatura ambiente per un massimo di 1 mese.
- Immergere substrati di vetro pulite in preparati al 2% v / v 3- (trimetossisilil) propil metacrilato (3-TPM) in etanolo. Posizionare substrati su un agitatore orbitale per 30 min.
- Opzione 1: Realizzare idrogel di poliacrilamide su vetrini da microscopio silanizzati per endpoint immunofluorescenza.
- Preparare una soluzione pre-polimero in dH 2 O con l'acrilamide / percentuale bisacrylamide desiderato (w / v) rapporto per fabbricare substrati con moduli di Young di 4 kPa (4% acrilammide, 0,4% bisacrylamide), 13 kPa (6% acrilammide, 0,45 % bisacrylamide), o 30 kPa (8% acrilammide, 0,55% bisacrylamide) e porosità simili, per Wen et al. 41. soluzione Vortex fino chiara e filtro con una siringa di 0,2 micron. Le soluzioni pre-polimero possono essere conservati a 4 ° C per 3 mesi.
ATTENZIONE: L'esposizione a acrilamide o bisacrylamide può causare tossicità acuta, Neurotoxicity, e irritazione. Indossare guanti, indumenti e occhiali di protezione. - Preparare una soluzione fotoiniziatore del 20% w / v Irgacure 2959 in metanolo. Questa soluzione fotoiniziatore non può essere conservata e va preparata di fresco ogni volta.
- Mescolare il pre-polimero e soluzioni fotoiniziatori in un rapporto di 9: 1 (fotoiniziatore pre-polimero). degas eventualmente con una camera a vuoto per 15 minuti per rimuovere le bolle.
- Collocare i vetrini silanizzati in un vassoio di essiccazione di vetro e Dispensare 100 ml di 9: 1 pre-polimero: soluzione fotoiniziatore su ogni diapositiva. coprire delicatamente ogni vetrino con un coprioggetto mm 22 × 60, evitando la creazione di bolle. Si noti che il coprioggetto impedisce l'inibizione della reazione di polimerizzazione dall'ossigeno.
- Vassoio essiccamento posto in un reticolante UV ed esporre i vetrini a 365 nm UV A per 10 min (4 W / m 2). Ottimizzare il tempo di polimerizzazione, se necessario. esposizioni più lunghe rischio difficoltà a rimuovere il vetrino a causa di overpolymerization. esposizioni più brevi rISK underpolymerization e bassa stabilità idrogel.
- Immergere idrogel in DH 2 O per 5 min. Rimuovere coprioggetto con un rasoio, facendo attenzione a non danneggiare le idrogel polimerizzati.
- Lasciare idrogel in dH 2 O a temperatura ambiente per 1 - 3 d, cambiando dH 2 O giornaliero. Disidratare idrogel a 50 ° C su una piastra calda fino a secco (15 - 30 min) e conservare a temperatura ambiente per un massimo di 3 mesi.
- Preparare una soluzione pre-polimero in dH 2 O con l'acrilamide / percentuale bisacrylamide desiderato (w / v) rapporto per fabbricare substrati con moduli di Young di 4 kPa (4% acrilammide, 0,4% bisacrylamide), 13 kPa (6% acrilammide, 0,45 % bisacrylamide), o 30 kPa (8% acrilammide, 0,55% bisacrylamide) e porosità simili, per Wen et al. 41. soluzione Vortex fino chiara e filtro con una siringa di 0,2 micron. Le soluzioni pre-polimero possono essere conservati a 4 ° C per 3 mesi.
- Opzione 2: Realizzare fluorescenti idrogel poliacrilammide tallone contenenti 35 mm piastre di Petri con fondo di vetro silanizzati per la valutazione diretta delle interazioni cellula-substrato utilizzando TFM.
- Sonicare una soluzione stock di 1 micron perline fluorescenti per 15 minuti per disperdere gli aggregati.
- Preparare una soluzione pre-polimero in dH 2 O con l'acrilamide / percentuale bisacrylamide desiderato (w / v) rapporto per fabbricare substrati con moduli di Young di 4 kPa (4% acrilammide, 0,4% bisacrylamide), 13 kPa (6% acrilammide, 0.45% Bisacrylamide), o 30 kPa (8% acrilammide, 0,55% bisacrylamide) e porosità simili, per Wen et al. 41. soluzione Vortex fino chiara e filtro con una siringa di 0,2 micron. Le soluzioni pre-polimero possono essere conservati a 4 ° C per 3 mesi.
ATTENZIONE: L'esposizione a acrilamide o bisacrylamide può causare tossicità acuta, neurotossicità, e irritazione. Indossare guanti, indumenti e occhiali di protezione. - Aggiungere perline fluorescenti alla soluzione pre-polimero ad una concentrazione finale di 0,2% v / v ed agitare per miscelare.
- Preparare una soluzione fotoiniziatore del 20% w / v Irgacure 2959 in metanolo. Questa soluzione fotoiniziatore non può essere conservata e va preparata di fresco ogni volta.
- Mescolare le soluzioni pre-polimero / perline e fotoiniziatori in un 9: 1 (pre-polimero / tallone: fotoiniziatore) rapporto. degas eventualmente con una camera a vuoto per 15 minuti per rimuovere le bolle.
- Posizionare silanizzati mm piastre di Petri 35 con fondo di vetro in un vassoio di essiccazione di vetro e pipet 20 ml di 9: soluzione fotoiniziatore sul centro di ogni piatto: 1 pre-polimero / tallone. coprire delicatamente ogni vetrino con un coprioggetto circolare 12 millimetri, evitando la creazione di bolle. Si noti che il coprioggetto impedisce l'inibizione della reazione di polimerizzazione dall'ossigeno.
- Al fine di distribuire le perline fluorescenti alla superficie dell'idrogel, invertire i piatti e lasciare a temperatura ambiente per 20 min, per Knoll et al. 42.
- Mentre ancora invertito, esporre piatti a 365 nm UV A per 10 min (4 W / m 2). Ottimizzare il tempo di polimerizzazione, se necessario. esposizioni più lunghe rischio difficoltà a rimuovere il vetrino a causa di overpolymerization. Shorter underpolymerization rischio delle esposizioni e bassa stabilità idrogel.
- Immergere idrogel a 0,1 M 4- (2-idrossietil) Acido -1-piperazineethanesulfonic (HEPES) tampone e lasciare a temperatura ambiente in una notte buia. Rimuovere accuratamente coprioggetto con un rasoio, facendo attenzione a non danneggiare il polymeidrogel zato.
- Disidratare idrogel a 50 ° C su una piastra calda fino a secco (15 - 30 min). Idrogeli possono essere conservati a temperatura ambiente al buio per 3 mesi.
2. Realizzazione di Array
- Preparare buffer per stampare biomolecole. Utilizzare il buffer di stampa appropriato alle biomolecole di interesse. Fattore di crescita (GF) buffer di stampa è ampiamente adatto per altre classi di molecole, come ligandi cellula-cellula.
- Per preparare 2 × ECM buffer di stampa proteine, aggiungere 164 mg di acetato di sodio e 37,2 mg di acido etilendiamminotetraacetico (EDTA) per 6 mL dH 2 O. Vortex e incubare a 37 ° C per solubilizzare completamente. Dopo solubilizzazione, aggiungere 50 ml di pre-riscaldato Triton X-100 e 4 ml di glicerolo. Vortex e incubare a 37 ° C ancora per solubilizzare. Aggiungere 40 - 80 ml di acido acetico glaciale, titolazione per regolare il pH a 4,8. 2 × ECM buffer di stampa proteina può essere conservato a 4° C per 1 mese.
ATTENZIONE: L'acido acetico è infiammabile e corrosivo. Indossare guanti, indumenti e occhiali di protezione. - Per preparare 2 × tampone stampa GF, aggiungere acetato di sodio 105,5 mg e 37,2 mg di EDTA a 6 ml di soluzione salina tamponata con fosfato (PBS). Vortex e incubare a 37 ° C per solubilizzare completamente. Dopo solubilizzazione, aggiungere 100 mg 3 - [(3-cholamidopropyl) dimethylammonio] -1-propanesulfonate (CHAPS) e 4 ml di glicerolo. 2 × buffer di stampa proteine GF può essere conservato a 4 ° C per 1 mese.
- Per preparare 2 × ECM buffer di stampa proteine, aggiungere 164 mg di acetato di sodio e 37,2 mg di acido etilendiamminotetraacetico (EDTA) per 6 mL dH 2 O. Vortex e incubare a 37 ° C per solubilizzare completamente. Dopo solubilizzazione, aggiungere 50 ml di pre-riscaldato Triton X-100 e 4 ml di glicerolo. Vortex e incubare a 37 ° C ancora per solubilizzare. Aggiungere 40 - 80 ml di acido acetico glaciale, titolazione per regolare il pH a 4,8. 2 × ECM buffer di stampa proteina può essere conservato a 4° C per 1 mese.
- Preparare piatto fonte.
- In una micropiastra 384 pozzetti V-parte inferiore, unire volumi uguali di 2 × buffer di stampa con ogni soluzione biomolecole al doppio del concentrazione target.
NOTA: Una concentrazione obiettivo appropriato per proteine ECM più comuni è di 250 mg / ml, mentre le concentrazioni target per altri tipi di fattori disposti variano a seconda ritenzione nel idrogel e funzione biologica. Il volume totale in ciascun pozzetto può esserea partire da 5 ml e non deve essere più di 15 ml. In aggiunta alle combinazioni di biomolecole di interesse, includere un marcatore fluorescente Arrayed per facilitare l'analisi delle immagini a valle. Utilizzare destrano rodamina-coniugato (2,5 mg / mL). - Mescolare ogni pozzetto accuratamente pipettando, facendo attenzione a non creare bolle. Centrifugare la micropiastra fonte per 1 min a 100 xg. Realizzare microarray utilizzando piastre di origine preparati lo stesso giorno e conservati a 4 ° C fino microarray fabbricazione.
- In una micropiastra 384 pozzetti V-parte inferiore, unire volumi uguali di 2 × buffer di stampa con ogni soluzione biomolecole al doppio del concentrazione target.
- Pulire perni secondo le istruzioni del produttore prima di ogni esecuzione microarray fabbricazione. Caricare perni pulite direttamente nella testina di stampa della microarrayer.
- Preparare microarrayer e programma utilizzando il software del produttore. Sebbene i passaggi seguenti sono in parte per il particolare microarrayer usato qui, il funzionamento della maggior parte microarrayers è simile.
- Accendere l'umidificatore, regolare il set point al 65% di umidità relativa (non-condensing), e attendere che il reometro corrisponde al set point. Mettere piatto di origine nel adattatore.
- Disidratare substrati idrogel a 50 ° C per 15 minuti e mettere in apposito adattatore. Il microarrayer ha adattatori per entrambi i vetrini da microscopio e micropiastre. Per arraying 35 mm piatti con fondo di vetro Petri, caricare i piatti in un 6-pozzetti micropiastra e posizionare il micropiastra nell'adattatore micropiastra sul Arrayer.
- Regolare i parametri del programma per riflettere esattamente la disposizione della piastra sorgente, gli array, e il formato desiderato (ad esempio, vetrino o micropiastre contenenti 35 mm Petri). Include fasi di lavaggio con acqua e dimetil solfossido (DMSO) tra ciascuna condizione per impedire riporto e la contaminazione incrociata.
- Inizia gamma fabbricazione, il controllo non meno di una volta un'ora che l'umidità non è scesa sotto il 65% di umidità relativa (senza condensa) e che i perni non siano otturati. Se la Humidità è sceso inaspettatamente, mettere in pausa arraying per riempire l'umidificatore e chiaro tubi associati di condensa. Se i perni sono intasati, mettere in pausa arraying per pulire i perni o altrimenti sostituire con perni pre-puliti. Si noti che è possibile matrice più tipi di biomolecole sequenzialmente sui medesimi substrati forniti tempo di asciugatura sufficiente (cioè, 4 h per tutta la notte).
- Una volta che il programma è completo, posizionare gli array fabbricate in una scatola di presentazione o micropiastre coperto con un foglio di alluminio a temperatura ambiente e 65% di umidità relativa (non condensante) durante la notte. Si noti che può essere necessario valutare la qualità matrice e ritenzione con macchie proteiche generale o immunofluorescenza; vedi Brafman et al. per maggiori dettagli 25.
3. cellulare cultura e Assay Lettura
- Il giorno dopo la fabbricazione, posizionare i substrati disposti in 4-camerata piatti (vetrini da microscopio) o 6 e-micropiastra (piastre di Petri) e immergere in 1%v / v di penicillina / streptomicina in PBS; utilizzare 4 ml per diapositive e 3 ml per i piatti. Esporre a UV C per 30 min. soluzione di streptomicina per i terreni di coltura delle cellule scambio di penicillina /.
- Raccogliere e contare le cellule. Seed su array a 500 × Ottobre 3-02 × 10 6 celle / array a 4 ml per vetrino da microscopio e 3 ml per 35 millimetri Petri. Incubare culture di matrice a 37 ° C e 5% CO 2 per 2 - 24 ore o fino a quando la formazione di isole cellulari densamente popolate. Regolare sia la densità e il tempo necessario per le cellule e particolare applicazione semina. Underseeding (vale a dire, a bassa densità o il tempo di semina) potrebbero provocare un calo della popolazione array e gli esiti biologici sghembi. Semina (vale a dire, ad alta densità o il tempo di semina) può causare ridotta integrità serie a causa di isola distacco.
- Dopo consentendo la formazione di isole cellulari, lavare culture di matrice due volte con mezzo di coltura pre-riscaldato; ancora una volta usare 4 ml per diapositive e 3 ml per i piatti. optionally aggiungere controlli e trattamenti adeguati (ad esempio, piccole molecole inibitrici, fattori di crescita, etc.) di interesse per il sistema biologico. Cambiare il supporto degli array ogni 1 - 2 d al fine di mantenere la concentrazione di eventuali trattamenti. Valutare l'espressione marker delle cellule e la funzione delle cellule mediante immunofluorescenza o cellula-substrato interazioni da TFM entro 1 - 5 d di avviare culture matrice - vedere Opzione 1 e Opzione 2 di seguito.
- Opzione 1: Eseguire immunofluorescenza endpoint. Si noti che immunofluorescenza di alcune proteine può richiedere permeabilizzazione più severe utilizzando metanolo, etanolo, o HCl. A causa di potenziali danni per gli array, valutare e ottimizzare ogni protocollo permeabilizzazione prima dell'uso in esperimenti su larga scala.
- Aspirare terreni di coltura cellulare da diapositive matrice in 4-camerata piatti e aggiungere 4 ml / slide di preparati 4% v / v paraformaldeide (PFA) in PBS. Incubare per 15 minuti a temperatura ambiente.
ATTENZIONE: Exposure di PFA può causare tossicità acuta e può irritare o corrode pelle a contatto. Indossare guanti, indumenti e occhiali di protezione e utilizzare solo in una cappa. - soluzione PFA Aspirare e lavare ogni diapositiva 3 volte con 4 ml di PBS. A questo punto, i vetrini fissati possono essere conservati a 4 ° C per 1 settimana. Si consiglia, tuttavia, di continuare attraverso immunomarcatura e montando lo stesso giorno come fissaggio per assicurare l'integrità array.
- Aspirare PBS e aggiungere 4 mL / slitta di 0,25% v / v Triton X-100 in PBS. Incubare per 10 minuti a temperatura ambiente.
- Aspirare Triton X-100 soluzione e lavare ciascun vetrino 3 volte con 4 ml di PBS. Aggiungere 4 mL / slide 5% v / v di siero abbinato alla specie di anticorpo secondario (per esempio, siero asino anticorpi secondari asino) in PBS e incubare a temperatura ambiente per 1 h.
- Rimuovere accuratamente la soluzione di saturazione da ogni diapositiva. Aggiungere 500 ml / slide di anticorpo primario diluito in 5% v / v di siero in PBS. Questo volume è sufficiente a coprire array sia per 1 h di incubazione a temperatura ambiente e incubazione overnight a 4 ° C.
- Lavare ciascuna slitta matrice 3 volte con 4 ml di PBS. Rimuovere accuratamente il lavaggio finale e aggiungere 500 ml / slide di anticorpo secondario appropriato diluito in 5% v / v di siero in PBS.
- Lavare ciascuna slitta matrice 3 volte con 4 ml di PBS. Lavare rapidamente con dH 2 O prima di rimuovere con attenzione le diapositive matrice dalla soluzione con pinze. Utilizzare un tessuto di laboratorio per favorire o secca dH residua 2 O.
- Dispensare 100 ml di soluzione con DAPI di montaggio attraverso la diapositiva pur confermando visivamente una copertura completa di tutta la gamma.
- Posizionare un vetrino mm 22 × 60 sopra la slitta per il montaggio. Sigillare i bordi del coprioggetto con smalto trasparente. Conservare al buio a 4 ° C fino a quando l'imaging, non prima del giorno successivo.
- Camera intero array utilizzando uno scanner microarray o invertita equipp microscopio a fluorescenzaED con una fase di robotica. Scanner microarray forniscono più veloce la lettura, ma possono richiedere Cy3- o compatibili con Cy5 fluorofori e sono spesso di risoluzione limitata al fine di singole cellule (ad esempio, 1 - 10 micron). microscopi fluorescenti offrono la possibilità di usare una varietà di canali fluorescenti ed a risoluzione più elevata (<1 um, ~ 100 × ingrandimento totale) ma forniscono lettura lenta a seconda della qualità della fase robotica e l'ingrandimento / obiettivo.
- Salva le immagini catturate di intere schiere di entrambi i metodi come file TIFF in modo da evitare la compressione dei dati o la perdita associati ad altri formati di file (ad esempio, JPG).
- Aspirare terreni di coltura cellulare da diapositive matrice in 4-camerata piatti e aggiungere 4 ml / slide di preparati 4% v / v paraformaldeide (PFA) in PBS. Incubare per 15 minuti a temperatura ambiente.
- Opzione 2: eseguire la valutazione diretta delle interazioni cellula-substrato utilizzando TFM.
- Preparare una soluzione di 1% v / v di siero albumina bovina (BSA) e 1% v / v di sodio dodecil solfato (SDS) in PBS per dissociare le cellule da substrati durante TFM.
- Spostare 35 mm piastre di Petri contenenti culture matrice diun (CO 37 ° C, 5% 2) incubato, microscopio a fluorescenza invertito con una fase robotizzato per misure TFM.
- In un piatto, segnare le posizioni (coordinata X, Y coordinata) e mettere a fuoco gli aerei (Z coordinata) di singole isole di cellule usando la microscopia a contrasto di fase.
- Passa alla microscopia a fluorescenza lontano rosso per visualizzare le perline. Ritorno a ciascuna delle posizioni salvate nel passaggio precedente e correggere la coordinata Z del piano attivo in modo che solo il primo strato di perline sotto dell'isola cella è a fuoco. Salvare le nuove coordinate e procedere a delle immagini automatizzata di tutte le isole delle cellule di catturare pre-dissociazione fase di contrasto e immagini fluorescenti rossi lontane.
- Aggiungere con cautela 150 ml di soluzione di BSA / SDS al piatto e attendere 5 minuti per consentire la dissociazione delle cellule completa dal substrato; monitorare la dissociazione delle cellule al microscopio a contrasto di fase.
- Dopo le isole di cellule sono state dissociate dal substrato, tornare al tha segnato le posizioni e controllare che il primo strato di perline sono ancora a fuoco. Se queste perle sono fuori dal piano a causa della deformazione indotta dalla trazione cellule generate, quindi correggere la coordinata Z del piano focale in modo che siano più a fuoco. Save the Z-coordinate corrette e ripetere l'imaging automatizzata di tutte le isole per catturare post-dissociazione immagini fluorescenti lontano rossi.
- Ripetere i punti 3.5.1 - 3.5.4 per i piatti rimanenti.
4. Analisi dei dati
- Analisi dei dati immunofluorescenza.
- Processo acquisito le immagini di array. Split immagini composite di matrice in file contenenti singoli canali (cioè, rosso, blu, o verde) e convertire immagini TIFF a 8 bit 43, 44. Applicare binning (ad esempio, 2 × 2 o 4 × 4) per ridurre le dimensioni dell'immagine a ~ 32 megapixel per canale per ridurre i requisiti di memoria durante l'analisi a valle cella singola di Entiri immagini array. Vedere file di codice supplementare dal titolo "array_processing.ijm" per una macro implementazione ImageJ di queste fasi di lavorazione array.
- Nota le coordinate in pixel del alto a sinistra, in basso a sinistra, e inferiore marcatori destrano rodamina coniugato destra o condizioni disposte. Utilizzare queste coordinate per ruotare le immagini a 8-bit TIFF per essere perfettamente verticale e, successivamente, per annotare l'uscita dall'analisi cella singola con condizioni disposte specifiche. Vedere supplementare Codice Files dal titolo "rb_array_rotater.ijm", "rg_array_rotater.ijm", "rgb_array_rotater.ijm", e "array_gridding.ijm" per le implementazioni di questi array di rotazione e gridding passi.
- Effettuare analisi singola cella di binned, ruotare le immagini TIFF a 8 bit in CellProfiler (versione 2.1.1) 45 usando i seguenti moduli: IdentifyPrimaryObjects, IdentifySecondaryObjects, e MeasureObjectIntensity. IdentifyPrimaryObjects identifica nuclei, IdentifySecondaryObjects identifica immunolabels associati a ciascun nucleo cellulare, e fornisce MeasureObjectIntensity quantificazioni per entrambe le etichette nucleari e immunolabels.
- dati monocellulari uscita di tutti e tre i moduli come file CSV dal canale utilizzando il modulo ExportToSpreadsheet per facilitare l'analisi successiva a valle. Vedere supplementare Codice Files dal titolo "b_array_image_analysis.cppipe", "gb_array_image_analysis.cppipe", "rb_array_image_analysis.cppipe", e "rgb_array_image_analysis.cppipe" per tubazioni CellProfiler di attuazione questi passaggi per set di immagini contenenti canali rosso, verde o blu.
- Per trasformare i dati per tenere conto di variabilità sperimentale e distribuzioni unicellulari non gaussiana, applicare la normalizzazione quantile da replicare biologica 46. Questo processo genera una distribuzione condivisa tra repliche e permette il confronto imparziali di cambiamenti di intensità immunolabel. Inoltre, UNLike Z-punteggio e altri metodi parametrici, normalizzazione quantile è non-parametrica e non assume una particolare distribuzione dei dati, consentendo un più rappresentativi analisi del comportamento unicellulare in funzione delle condizioni schierati.
- Tracciare i dati e interpretare. A seconda del sistema biologico e ipotesi, calcolare e tracciare una o più delle seguenti misure d'insieme per ciascuna condizione disposte:
- Calcolare e tracciare cellule per isola come misura combinato di adesione e la sopravvivenza nel corso dell'esperimento.
- Calcolare e tracciare l'intensità immunolabel quantile-normalizzato come misura del destino cellulare o funzione.
- Calcolare e tracciare la percentuale di cellule positive per immunolabel come determinato da intensità superiore ad una soglia coerente, di solito 2 sd sopra l'intensità media del controllo negativo.
- In alternativa, le distribuzioni trama di intensità immunolabel al fine di esaminare e categorizzare il comportamento cella singolain funzione delle condizioni schierati. Queste distribuzioni possono essere ulteriormente caratterizzate mediante misure di tendenza centrale (, mediana, modalità di media) e la variazione (varianza, coefficiente di variazione, fattore di Fano) e metodi di prova ipotesi, come il test di Kolmogorov-Smirnov.
- Analisi dei dati TFM. Qui viene descritto un metodo che incorpora un algoritmo precedentemente sviluppato da Butler et al. e Wang et al. 40, 47.
- Utilizzare ImageJ per convertire in batch le immagini da file TIFF a 8 bit. Applicare binning pixel-media (ad esempio, 2 × 2) per ridurre il costo computazionale e ora analisi valle. Come algoritmi per TFM sono concentrati principalmente sull'analisi singola cellula, l'interfaccia a cellula-substrato delle isole (~ 17,5 × 10 3 micron 2) rispetto all'interfaccia cellula-substrato di una singola cella (75 micron 2) necessitati il passo binning.
- Inserire il contrasto di fase catturato e immagini lontane rosse fluorescenti (sia pre-e post-dissociazione dissociazione) in un ambiente di programmazione scientifica, come MATLAB e processo utilizzando gli algoritmi sviluppati in precedenza di Butler et al. e Wang et al. 40, 47.
- Selezionare tre regioni lontane dall'isola cellulare. Queste regioni sono usati per tenere conto di spostamenti a causa di immagine o di deriva del campione.
- Fornire il fattore convertire da pixel di micrometri (ad esempio, 0.454 pixel / micron), il modulo di Young del substrato (ad esempio, 13 kPa), e il rapporto di Poisson (ad esempio, 0,48 per i gel di poliacrilammide descritti qui).
- Per ogni isola, disegnare un confine intorno alla periferia di definire i vincoli geometrici; tutte le forze al di fuori di questo limite sono azzerati. Questo sistema vincolato è ragionevole data la grande distance (vale a dire, 450 micron) tra le isole.
- Calcola le sollecitazioni di trazione root-mean-square e contrattili momento per ogni isola. Il momento contractile è una misura della tensione residua attraverso l'isola cellulare e ha dimostrato di riflettere la forza delle interazioni cellula-cellula 48. Per ogni condizione schierati, i valori medi di root-mean-square in più isole e repliche biologiche e calcolare associati varianza per la verifica di ipotesi. È anche possibile mediare la distribuzione delle tensioni o momenti di numerose isole per fornire una mappa rappresentativa di entrambe le misure in funzione della geometria, ad esempio, la distanza dal centro dell'isola.
Risultati
Utilizzando questa piattaforma, abbiamo studiato il ruolo di entrambi i segnali biochimici e biofisici nella specifica sorte dei progenitori del fegato 34, 35. Proteina A / G Notch coniugato ligandi hanno mostrato una migliore ritenzione e il clustering in idrogel di poliacrilamide (figura 3A) e sono stati, inoltre, in grado di pilotare la differenziazione dei progenitori del fegato verso un destino delle cellule del dotto biliare (Figura 3B). Utilizzando l'analisi singola cellula, abbiamo quantificato la risposta ai ligandi Notch per le proteine ECM collagene, collagene III, collagene IV, fibronectina e laminina (Figura 3C), trovando che la risposta dei progenitori epatici al ligando dipende anche dalla contesto ECM. Ultimo, abbiamo utilizzato shRNA atterramento di generare progenitori epatici senza i ligandi DLL1 e JAG1. La risposta al ligando Notch schierati variava a seconda del presence di entrambi ligando, confermando che la risposta allo ligando cellulare estrinseco è anche una funzione dell'espressione ligando cellulare intrinseca (Figura 3D). Inoltre, abbiamo osservato una sottopopolazione distinto di doppio-positivo (ALB + / + OPN) cellule nel atterramento DLL1 (Figura 3D). Insieme, questi risultati mostrano rappresentative: (1) le capacità combinatorie del formato array, come esemplificato dalla coppia di molteplici schierati proteine ECM e ligandi Notch con l'atterramento di singoli leganti; (2) la funzionalità non solo schierato proteine ECM ma anche schierato ligando cellula-cellula con Proteina A / coniugazione G-mediata; e (3) l'attuazione della nostra analisi singola cellula e la sua capacità di discernere sottopopolazioni unici.
Abbiamo anche osservato che la differenziazione di progenitori epatici dipende sia la rigidità del substrato e la composizione ECM (Figura 4A ong>), in particolare trovando che il collagene IV è favorevole differenziazione su supporti morbidi e rigidi, mentre fibronectina supporta solo differenziazione su substrati rigidi (Figura 4B). Mappe di calore di misurazioni rappresentative TFM ha suggerito che lo stress prolungato di trazione a bassa rigidità del substrato sul collagene IV provocato differenziazione in cellule del dotto biliare (Figura 4C), un risultato confermato da valori medi di root-mean-square (Figura 4D). Insieme, questi risultati mostrano rappresentative: (1) il successo dell'integrazione di TFM con microarrays di cellule su substrati con una rigidità sintonizzabile per valutare sia il fenotipo cellulare e lo stress di trazione; (2) il coordinamento del destino cellule progenitrici epatiche sia con la composizione della matrice e la rigidità del substrato; e (3) l'attuazione della nostra analisi e tipici profili di stress trazione TFM in microarray cellulari.
e 1 "src =" / files / ftp_upload / 55362 / 55362fig1.jpg "/>
Figura 1: Schema che mostra le prime tre sezioni sperimentali. Nella sezione 1, substrati di vetro vengono puliti e silanizzate per facilitare la realizzazione di idrogel di poliacrilamide. Nella sezione 2, le combinazioni di biomolecole di interesse sono preparati in una micropiastra fonte 384 pozzetti. Un Arrayer robotica viene poi caricato con perni pulito, la micropiastra fonte e gli idrogel di poliacrilamide e inizializzato, fabbricare le matrici sulle idrogel. Nella sezione 3, le cellule vengono seminate su domini Arrayed e ha permesso di aderire, al termine del quale viene eseguito il protocollo di cultura di interesse. Al punto finale, le cellule sono fissate per immunoistochimica / immunofluorescenza o analizzati utilizzando TFM. Barre di scala sono 75 micron. Clicca qui per vedere una versione più grande di questa figura.
entro-page = "1"> 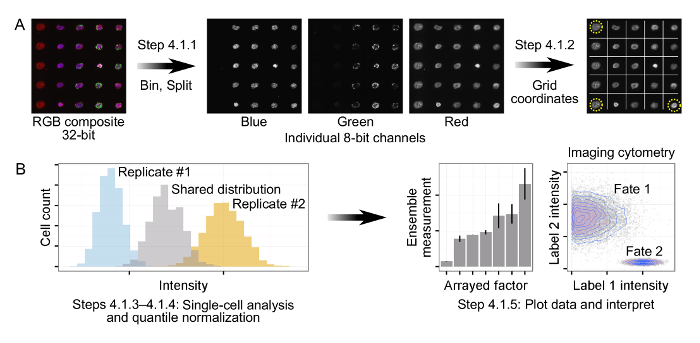
Figura 2: elaborazione ed analisi dei dati da immunofluorescenza Array. (A) piastrella, immagini RGB composito a 32 bit vengono prima cestinate e poi suddiviso in singoli canali a 8 bit. Utilizzando una combinazione di marcatori fluorescenti disposte e isole cellulari, sono identificati tre angoli della matrice per consentire l'orientamento automatico e gridding degli array. (B) di dati Single-cellula è generato per ogni canale delle matrici di ingresso. Al fine di tenere conto di deriva sperimentale, la normalizzazione quantile viene applicata da replicare biologica, la produzione di un singolo di distribuzione condivisa in tutte le repliche. Quantile dati normalizzati viene successivamente tracciata ed interpretato tramite calcolo delle misure Ensemble (ad esempio, cellule / isola, intensità media, le cellule percentuale positiva per un etichetta) o di analisi diretta di distribuzioni unicellulari.m / files / ftp_upload / 55362 / 55362fig2large.jpg "target =" _ blank "> Clicca qui per vedere una versione più grande di questa figura.
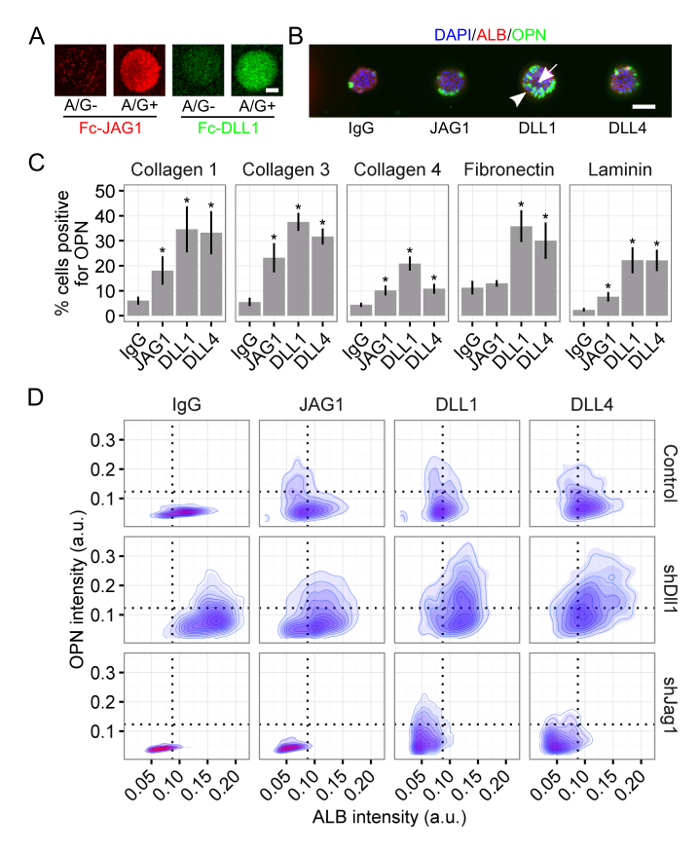
Figura 3: Notch Ligand Presentazione Mediazione di fegato Progenitor differenziazione. (A) ligandi Notch Fc-ricombinante Jagged-1 (JAG1) e Delta-simile 1 (DLL1) espone una migliore ritenzione e il clustering, quando schierato con proteina A / G. barra della scala è di 50 micron. Progenitori (B) del fegato differenziate in cellule del dotto biliare al momento della presentazione con Notch ligando. 4 ', 6-Diamidino-2-phenylindole (DAPI) è un marchio di nucleare, l'albumina (ALB) è un marker delle cellule epatiche, e osteopontina (OPN) è un marker biliare cellule del condotto. barra della scala è di 150 micron. (C) Quantificazione della percentuale di cellule positive per OPN per i ligandi Notch JAG1, DLL1, e Delta-like 4 (Dll4) sulle proteine ECM collagene I, collagen III, IV collagene, fibronectina e laminina. T -test di Student sono state eseguite contro IgG di controllo per ogni ligando Notch disposte all'interno di ogni proteina ECM con valori di p indicato per P <0,05 (*). (D) Imaging citometria di ALB e OPN per le cellule su collagene III presentati con i ligandi Notch JAG1, DLL1, e Dll4. Progenitori fegato senza il Notch ligandi DLL1 e JAG1 (vale a dire, shDll1 e shJag1) sono stati generati utilizzando shRNA atterramento. Dati in (C) presentati come media ± sem Questa figura è stata modificata da Kaylan et al. 34. Clicca qui per vedere una versione più grande di questa figura.
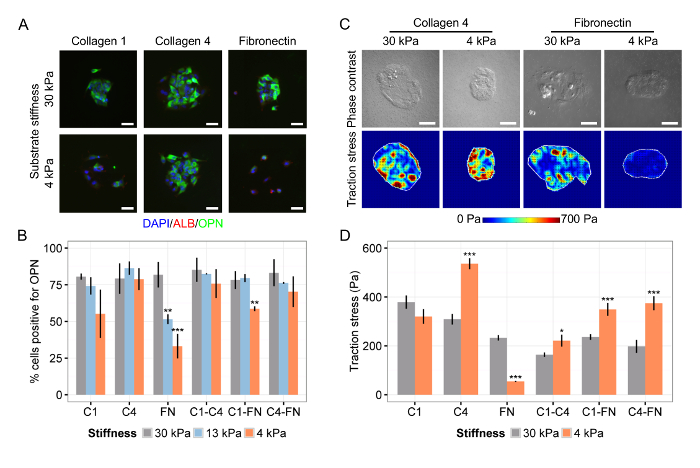
Figura 4: Matrice Composizione e substrato Rigidità Coordinate Fegato Progenitor differenziazione. (A) differenziazione fegato progenitrici biliari cellule del dotto dipende sia composizione ECM e substrato rigidità. DAPI è un'etichetta nucleare, ALB è un marker delle cellule epatiche, e OPN è un marcatore biliare cellule del condotto. (B) Quantificazione della percentuale di cellule positive per OPN su substrati di modulo di Young 30 kPa, 13 kPa, e 4 kPa per il collagene I (C1), collagene IV (C4), fibronectina (FN), e tutti bidirezionali combinazioni di quelle proteine ECM. (C) lo stress cellulare trazione dipende sia rigidità substrato e la composizione ECM. (D) Quantificazione dei valori root-mean-square di stress di trazione su substrati di modulo di Young 30 kPa e 4 kPa per il collagene I (C1), il collagene IV (C4), fibronectina (FN), e tutte le combinazioni a due vie di coloro proteine ECM. In (B) e (D), i dati sono stati presentati come media ± SEM e allievo di t -testsono stati eseguiti contro 30 kPa per ogni combinazione ECM con valori di P indicato per P <0,05 (*), P <0,01 (**), e P <0.001 (***). Barre di scala sono 50 micron. Questa cifra è stata modificata da Kourouklis et al. 35. Clicca qui per vedere una versione più grande di questa figura.
| Sezione | Problema | possibili cause | Soluzione |
| 1. Realizzazione di Polyacrylamide substrato. | Coprioggetti non può essere rimosso da idrogel. | Overpolymerization. | Ridurre il tempo di polimerizzazione a <10 minuti (4 W / m 2). Controllare che crossli UVuscita nker è nel raggio d'azione previsto. |
| Povero polimerizzazione poliacrilammide idrogel. | Underpolymerization. | Aumentare il tempo di polimerizzazione a> 10 minuti (4 W / m 2). Controllare che l'uscita reticolante UV si trova nel raggio previsto. | |
| idrogel poliacrilammide sono danneggiati dopo la rimozione del coprioggetti. | idrogel poliacrilammide morbide sono facili da danneggiare. | Osserviamo diminuendo la resa idrogel fabbricazione (~ 50%) per il più morbido (ad esempio, 4 kPa) idrogeli in particolare. Maneggiare idrogel delicatamente e aumentare il numero di partenza per raggiungere il rendimento desiderato. | |
| 2. Realizzazione di array. | Scarsa o incoerenti morfologia posto. | funzione di umidificatore incoerente. | Controllare che umidificatore e reometro un funzionale nel corso di ogni tiratura e di mantenere il 65% di umidità relativa. |
| Pins bloccato in testina o intasareGED. | Pulire la testina di stampa per consentire il movimento del perno libero. perni Pulire accuratamente prima o dopo ogni tiratura per rimuovere aggregati dai canali pin. | ||
| 3. cellulare cultura e test di esecuzione. | il distacco delle cellule morte o sugli array dopo il collegamento iniziale. | Semina e la proliferazione eccessiva. | Ridurre la densità di semina iniziale e il tempo. Utilizzare "manutenzione" o media "differenziazione" durante la cultura di matrice per ridurre la proliferazione cellulare. |
| Rilascio di monomero acrilammide tossici idrogel. | Immergere idrogel in dH 2 O per almeno 3 d per permettere la diffusione / rilascio di acrilammide monomero e ridurre la tossicità cellulare. | ||
| Le cellule non attribuiscono ad array. | Underseeding. | Aumentare la densità di semina iniziale e il tempo. Utilizzare un tipo di cellula più fortemente aderente. | |
| Scarsa deposizione dimatrix o condizione biomolecole. | Pulire perni di particelle e aggregati, confermano i parametri di stampa, e valutare individuazione di marcatori fluorescenti, ad esempio, rodamina-destrano coniugato. | ||
| Specificità delle interazioni cellula-matrice. | Differenti tipi di cellule aderiscono specificamente per alcuni, ma non altre proteine ECM. Prova più proteine ECM differenti con le vostre cellule. | ||
| Storage Array non ottimale dopo la fabbricazione. | Si consiglia di conservare le matrici fabbricati durante la notte a 65% di umidità relativa e temperatura ambiente, in parte per evitare cambiamenti di fase durante il congelamento. L'adesione cellulare è sensibile sia umidità, temperatura e tempo di conservazione; assicurarsi che questi parametri siano coerenti / ottimizzati per i vostri esperimenti. | ||
| Il distacco di idrogel dal substrato di vetro durante la coltura cellulare. | pulizia scivolo poveri e silanizzazione. | Sostituire le soluzioni di lavoro per la pulizia e la slittasilanizzazione. | |
| idrogel Overdehydrated. | Non lasciare idrogel disidratanti su un piatto caldo per più di 15-30 minuti. | ||
| 4. Analisi dei dati. | Alta variabilità tra i punti replicare e diapositive. | La variabilità della matrice fabbricazione. | Controllare che i perni e le testine di stampa siano puliti. Confermare la funzione umidificatore. Visualizzare e quantificare posto e la matrice qualità utilizzando marcatori fluorescenti. array conservarlo come raccomandato sopra. |
Tabella 1: risoluzione dei problemi.
Discussione
Nei nostri esperimenti, abbiamo trovato che i guasti più comuni sono legati alla qualità di array fabbricati e poco caratterizzati risposta del sistema biologico che interessa. Rimandiamo il lettore alla Tabella 1 per modalità di guasto comuni in esperimenti di microarray cellulare e procedure di risoluzione associati. Per quanto riguarda la qualità delle matrici, in particolare, si raccomanda quanto segue. Confermare la qualità tecnica e la robustezza di arraying programmi, parametri e tamponi utilizzando molecole fluorescente come destrano rodamina-coniugati. perni Pulire accuratamente prima o dopo arraying secondo le istruzioni del produttore e inoltre visivamente controllare che i canali di pin sono chiari di detriti utilizzando un microscopio ottico. Confermare la ritenzione biomolecola Arrayed con macchie di proteine generali o immunomarcatura. Notare che biomolecole con peso molecolare inferiore a 70 kDa spesso non sono mantenute nel idrogel 23, sup> 31. Convalida Arrayed biomolecola cella-funzionalità utilizzando più tipi di cellule. Si noti che solo le cellule aderenti sono compatibili con gli array; Inoltre, l'adesione ad array dipende sia proprietà specifiche delle celle (ad esempio, integrina profilo di espressione) e le proteine ECM selezionati.
A causa di spazio limitato, non abbiamo fornito una vasta trattamento di disegno matrice, il layout e la fabbricazione qui e di rimandare il lettore a lavori precedenti 23, 25. Usiamo generalmente 100 sottoarray Spot (150 micron di diametro posto, 450 micron da centro a centro distanza) composti da 10-20 condizioni di biomolecole unici (cioè, 5-10 punti / condizione). Il numero di sotto-array in un array varia a seconda del numero di condizioni biomolecole di interesse, che può essere scalata comodamente fino a 1.280 su un 25 x 75 mm di traslazione microscopio (~ 6.400 punti in 64 sottoarray)xref "> 25, 31 I parametri sopra ulteriormente variare a seconda delle dimensioni del motivo di interesse;. perni atti a generare i motivi da 75 - 450 micron sono facilmente disponibili.
esperimenti di array sono meglio integrate da validazione di alta punteggio condizioni schierati di interesse utilizzando altri formati di cultura, letture di analisi e sistemi modello biologico. In particolare, si consiglia di un'ulteriore validazione effetti di selezionare le condizioni disposte utilizzando colture di massa in combinazione con tecniche di biologia molecolare standard (ad esempio, qRT-PCR, immunoblotting) o di serie TFM. La manipolazione genetica (ad esempio, l'abbattimento o sovraespressione) del fattore di interesse per un adeguato sistema di modello biologico può anche servire per confermare gli effetti osservati negli array. In vivo modelli animali rappresentano un altro mezzo di convalida e sono stati recentemente utilizzati, per esempio, per confermare il ruolo centrale di galectin-3 e galectin-8il tumore del polmone metastatico nicchia, come inizialmente identificato tramite microarray cella 31, 49.
Un certo numero di altri metodi sono stati usati per sondare regolazione microambientali di funzioni cellulari, compresa una varietà di bidimensionali sistemi microfabbricati 18, 50, 51, 52, 53, 54, 55 e tridimensionali sistemi biomateriale ingegnerizzati 56, 57, 58 , 59, 60, 61. In confronto con altri metodi, i particolari vantaggi della piattaforma microarray cellule descritte qui costituiti da: (1) produzione finocentinaia o migliaia di diverse combinazioni di fattori, consentendo analisi degli effetti di interazione; (2) accessibili, imaging e analisi automatizzata; (3) l'integrazione di entrambe le letture biochimici e biofisici con presentazione controllata di fattori schierati; (4) capacità di variare le proprietà del materiale del substrato; e (5) l'analisi singola cella ad alta contenuto di destino delle cellule e la funzione.
In sintesi, la combinazione di microarrays cellulari con TFM su substrati di sintonizzabile rigidità substrato consente caratterizzazione approfondita dei segnali biochimici e biofisici. Come presentato qui, questa piattaforma è generalizzabile e può essere facilmente applicata ad una varietà di tipi di cellule aderenti e dei contesti di tessuto verso una migliore comprensione della regolazione microambientali combinatoria di differenziazione cellulare e meccanotrasduzione.
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari concorrenti.
Riconoscimenti
Riconosciamo Austin Cyphersmith e Mayandi Sivaguru (Carl R. Woese Istituto di Biologia Genomica, University of Illinois a Urbana-Champaign) per l'assistenza con la microscopia e generosamente accogliere schermo e cattura video al centro di microscopia.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.2 µm syringe filter | Pall Corporation | 4433 | Match with appropriately-sized Luer lock plastic syringes. |
| 100 × penicillin–streptomycin solution | Fisher Scientific | SV30010 | |
| 22 × 60 mm coverglasses | Electron Microscopy Sciences | 63765 | |
| 3-(trimethoxysilyl)propyl methacrylate (3-TPM) | Sigma-Aldrich | 440159 | Store under inert gas per manufacturer's instructions. Exposure of 3-TPM to air could compromise silanization of glass substrates. CAUTION: 3-TPM is a combustible liquid. Keep away from heat, sparks, open flames, and hot surfaces and use only in a chemical fume hood. |
| 3-[(3-Cholamidopropyl)dimethylammonio]-1-propanesulfonate hydrate (CHAPS) | Sigma-Aldrich | C3023 | |
| 35 mm glass-bottom Petri dishes | Cell E&G | GBD00002-200 | 13 mm well consisting of #1.5 coverglass. Enables TFM and live-cell imaging. |
| 384-well polypropylene V-bottom microplate, non-sterile | USA Scientific | 1823-8400 | |
| 6-well polystyrene microplates | Fisher Scientific | 08-772-1B | 35 mm glass-bottom Petri dishes fit into wells of microplate, easing array fabrication. |
| Acetone | Sigma-Aldrich | 179973 | |
| Acrylamide | Sigma-Aldrich | A3553 | CAUTION: Exposure to acrylamide can result in acute toxicity and irritation. Wear protective gloves, clothing, and eye protection. |
| Collagen I, rat tail | EMD Millipore | 08-115MI | |
| Collagen III, human | EMD Millipore | CC054 | |
| Collagen IV, human | EMD Millipore | CC076 | |
| Crosslinker, 365 nm | UVP | CL-1000 | |
| Dextran, rhodamine B-conjugated, 70 kDa | ThermoFisher Scientific | D1841 | Used as a marker for array location. |
| Dimethyl sulfoxide | Fisher Scientific | BP231 | |
| Dulbecco's phosphate-buffered saline (PBS) | Fisher Scientific (HyClone) | SH3001302 | |
| Ethyl alcohol | Decon Labs | 2701 | |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma-Aldrich | ED | |
| Fc-recombinant DLL1, mouse | R&D Systems | 5026-DL-050 | |
| Fc-recombinant DLL4, mouse | AdipoGen | AG-40A-0145-C050 | |
| Fc-recombinant JAG1, rat | R&D Systems | 599-JG-100 | |
| Fibronectin, human | Sigma-Aldrich | F2006 | |
| Fluorescent microscope, inverted | Zeiss | Axiovert 200M | Ensure microscope is equipped with a robotic stage for both automated fluorescent imaging and TFM. Environmental control (i.e., 37 °C and 5% CO2) is highly advisable for TFM. |
| Fluoromount G with DAPI | SouthernBiotech | 0100-20 | |
| Glacial acetic acid | Sigma-Aldrich | 695092 | CAUTION: Acetic acid is flammable and corrosive. Wear protective gloves, clothing, and eye protection. |
| Glycerol | Sigma-Aldrich | M6145 | |
| Irgacure 2959 | BASF Corporation | 55047962 | |
| Laminin, mouse | EMD Millipore | CC095 | |
| Methanol | Sigma-Aldrich | 179957 | |
| Microarray scanner | GenePix | 4000B | Fluorophores must be Cy3- or Cy5-compatible. |
| Microarrayer | Digilab | OmniGrid Micro | Other microarrayers of similar or greater capability can readily be substituted. |
| Microscope slides, 25 × 75 mm | Sigma-Aldrich | CLS294775X25 | ~0.9 – 1.1 mm thickness. |
| N,N′-Methylenebisacrylamide (bisacrylamide) | Sigma-Aldrich | M7279 | CAUTION: Exposure to acrylamide can result in acute toxicity and irritation. Wear protective gloves, clothing, and eye protection. |
| Paraformaldehyde (PFA), 16% v/v | Electron Microscopy Sciences | RT15710 | Prepare PFA fresh (do not store) for optimal fixation. CAUTION: Exposure to PFA can result in acute toxicity and can also irritate or corrode skin on contact. Wear protective gloves, clothing, and eye protection and use only in a chemical fume hood. |
| Protein A/G, recombinant | ThermoFisher Scientific | 21186 | |
| Pyrex drying tray, 2,000 mL | Fisher Scientific | 15-242B | |
| Rectangular 4-chambered culture dish | Fisher Scientific (Nunc) | 12-565-495 | For cell culture on arrayed microscope slides. |
| Sodium acetate | Sigma-Aldrich | S2889 | |
| Sodium hydroxide | Sigma-Aldrich | 415413 | CAUTION: NaOH is highly caustic and can cause severe skin burns and eye damage. Wear protective gloves, clothing, and eye protection. |
| Stealth pin for arraying | ArrayIt | SMP3 | Clean pins after each array run using the instructions of the manufacturer. Produces 150 micron domains; purchase other pin sizes (75–450 microns) as suited to your particular application. |
| Triton X-100 | Sigma-Aldrich | X100 |
Riferimenti
- Joyce, J. A., Pollard, J. W. Microenvironmental regulation of metastasis. Nat Rev Cancer. 9 (4), 239-252 (2009).
- Hsu, Y. C., Fuchs, E. A family business: stem cell progeny join the niche to regulate homeostasis. Nat Rev Mol Cell Biol. 13 (2), 103-114 (2012).
- Whiteside, T. L. The tumor microenvironment and its role in promoting tumor growth. Oncogene. 27 (45), 5904-5912 (2008).
- Jones, D. L., Wagers, A. J. No place like home: anatomy and function of the stem cell niche. Nat Rev Mol Cell Biol. 9 (1), 11-21 (2008).
- Discher, D. E., Mooney, D. J., Zandstra, P. W. Growth factors, matrices, and forces combine and control stem cells. Science. 324 (5935), 1673-1677 (2009).
- Trappmann, B., et al. Extracellular-matrix tethering regulates stem-cell fate. Nat Mater. 11 (7), 642-649 (2012).
- Ivanovska, I. L., Shin, J. W., Swift, J., Discher, D. E. Stem cell mechanobiology: diverse lessons from bone marrow. Trends Cell Biol. 25 (9), 523-532 (2015).
- Engler, A. J., Sen, S., Sweeney, H. L., Discher, D. E. Matrix elasticity directs stem cell lineage specification. Cell. 126 (4), 677-689 (2006).
- Chaudhuri, O., et al. Hydrogels with tunable stress relaxation regulate stem cell fate and activity. Nat Mater. 15 (3), 326-334 (2016).
- McBeath, R., Pirone, D. M., Nelson, C. M., Bhadriraju, K., Chen, C. S. Cell shape, cytoskeletal tension, and RhoA regulate stem cell lineage commitment. Dev Cell. 6 (4), 483-495 (2004).
- Legate, K. R., Wickstrom, S. A., Fassler, R. Genetic and cell biological analysis of integrin outside-in signaling. Genes Dev. 23 (4), 397-418 (2009).
- Kessenbrock, K., Plaks, V., Werb, Z. Matrix metalloproteinases: regulators of the tumor microenvironment. Cell. 141 (1), 52-67 (2010).
- Underhill, G. H. Stem cell bioengineering at the interface of systems-based models and high-throughput platforms. Wiley Interdiscip Rev Syst Biol Med. 4 (6), 525-545 (2012).
- Underhill, G. H., Galie, P., Chen, C. S., Bhatia, S. N. Bioengineering methods for analysis of cells in vitro. Annu Rev Cell Dev Biol. 28, 385-410 (2012).
- Zorlutuna, P., et al. Microfabricated biomaterials for engineering 3D tissues. Adv Mater. 24 (14), 1782-1804 (2012).
- Ruiz, S. A., Chen, C. S. Microcontact printing: A tool to pattern. Soft Matter. 3 (2), 168-177 (2007).
- Guillotin, B., Guillemot, F. Cell patterning technologies for organotypic tissue fabrication. Trends Biotechnol. 29 (4), 183-190 (2011).
- Théry, M. Micropatterning as a tool to decipher cell morphogenesis and functions. J. Cell Sci. 123 (Pt 24), 4201-4213 (2010).
- Ranga, A., Lutolf, M. P. High-throughput approaches for the analysis of extrinsic regulators of stem cell fate. Curr. Opin. Cell Biol. 24 (2), 236-244 (2012).
- Kobel, S., Lutolf, M. High-throughput methods to define complex stem cell niches. Biotechniques. 48 (4), ix-xxii (2010).
- Fernandes, T. G., Diogo, M. M., Clark, D. S., Dordick, J. S., Cabral, J. M. S. High-throughput cellular microarray platforms: applications in drug discovery, toxicology and stem cell research. Trends Biotechnol. 27 (6), 342-349 (2009).
- Montanez-Sauri, S. I., Beebe, D. J., Sung, K. E. Microscale screening systems for 3D cellular microenvironments: platforms, advances, and challenges. Cell Mol Life Sci. 72 (2), 237-249 (2015).
- Flaim, C. J., Chien, S., Bhatia, S. N. An extracellular matrix microarray for probing cellular differentiation. Nat Methods. 2 (2), 119-125 (2005).
- Underhill, G. H., Flaim, C. J., Bhatia, S. N., Parekkadan, B., Yarmush, M. . Methods in Bioengineering: Stem Cell Bioengineering Artech House Methods in Bioengineering. , 63-73 (2009).
- Brafman, D. A., Chien, S., Willert, K. Arrayed cellular microenvironments for identifying culture and differentiation conditions for stem, primary and rare cell populations. Nat Protoc. 7 (4), 703-717 (2012).
- Brafman, D. A., et al. Investigating the role of the extracellular environment in modulating hepatic stellate cell biology with arrayed combinatorial microenvironments. Integr Biol. 1 (8-9), 513-524 (2009).
- Soen, Y., Mori, A., Palmer, T. D., Brown, P. O. Exploring the regulation of human neural precursor cell differentiation using arrays of signaling microenvironments. Mol Syst Biol. 2, 37 (2006).
- LaBarge, M. A., et al. Human mammary progenitor cell fate decisions are products of interactions with combinatorial microenvironments. Integr Biol. 1 (1), 70-79 (2009).
- Anderson, D. G., Levenberg, S., Langer, R. Nanoliter-scale synthesis of arrayed biomaterials and application to human embryonic stem cells. Nat Biotechnol. 22 (7), 863-866 (2004).
- Brafman, D. A., Shah, K. D., Fellner, T., Chien, S., Willert, K. Defining long-term maintenance conditions of human embryonic stem cells with arrayed cellular microenvironment technology. Stem Cells Dev. 18 (8), 1141-1154 (2009).
- Reticker-Flynn, N. E., et al. A combinatorial extracellular matrix platform identifies cell-extracellular matrix interactions that correlate with metastasis. Nat Commun. 3, 1122 (2012).
- Wood, K. C., et al. MicroSCALE screening reveals genetic modifiers of therapeutic response in melanoma. Sci Signal. 5 (224), rs4 (2012).
- Braga Malta, D. F., et al. Extracellular matrix microarrays to study inductive signaling for endoderm specification. Acta Biomater. 34, 30-40 (2016).
- Kaylan, K. B., Ermilova, V., Yada, R. C., Underhill, G. H. Combinatorial microenvironmental regulation of liver progenitor differentiation by Notch ligands, TGFbeta, and extracellular matrix. Sci Rep. 6 (23490), 23490 (2016).
- Kourouklis, A. P., Kaylan, K. B., Underhill, G. H. Substrate stiffness and matrix composition coordinately control the differentiation of liver progenitor cells. Biomaterials. 99, 82-94 (2016).
- Kaylan, K. B., et al. Mapping lung tumor cell drug responses as a function of matrix context and genotype using cell microarrays. Integr. Biol. , (2016).
- Mann, C., Leckband, D. Measuring Traction Forces in Long-Term Cell Cultures. Cellular and Molecular Bioengineering. 3 (1), 40-49 (2010).
- Heisenberg, C. P., Bellaiche, Y. Forces in tissue morphogenesis and patterning. Cell. 153 (5), 948-962 (2013).
- Schwarz, U. S., Soine, J. R. Traction force microscopy on soft elastic substrates: A guide to recent computational advances. Biochim Biophys Acta. 1853 (11 Pt B), 3095-3104 (2015).
- Butler, J. P., Tolic-Norrelykke, I. M., Fabry, B., Fredberg, J. J. Traction fields, moments, and strain energy that cells exert on their surroundings. American Journal of Physiology-Cell Physiology. 282 (3), C595-C605 (2002).
- Wen, J. H., et al. Interplay of matrix stiffness and protein tethering in stem cell differentiation. Nat Mater. 13 (10), 979-987 (2014).
- Knoll, S. G., Ali, M. Y., Saif, M. T. A novel method for localizing reporter fluorescent beads near the cell culture surface for traction force microscopy. J Vis Exp. (91), e51873 (2014).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nat Methods. 9 (7), 671-675 (2012).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Kamentsky, L., et al. Improved structure, function and compatibility for CellProfiler: modular high-throughput image analysis software. Bioinformatics. 27 (8), 1179-1180 (2011).
- Bolstad, B. M., Irizarry, R. A., Astrand, M., Speed, T. P. A comparison of normalization methods for high density oligonucleotide array data based on variance and bias. Bioinformatics. 19 (2), 185-193 (2003).
- Wang, N., et al. Cell prestress. I. Stiffness and prestress are closely associated in adherent contractile cells. Am J Physiol Cell Physiol. 282 (3), C606-C616 (2002).
- Krishnan, R., et al. Substrate stiffening promotes endothelial monolayer disruption through enhanced physical forces. Am J Physiol Cell Physiol. 300 (1), C146-C154 (2011).
- Reticker-Flynn, N. E., Bhatia, S. N. Aberrant glycosylation promotes lung cancer metastasis through adhesion to galectins in the metastatic niche. Cancer Discov. 5 (2), 168-181 (2015).
- Chen, C. S., Mrksich, M., Huang, S., Whitesides, G. M., Ingber, D. E. Geometric control of cell life and death. Science. 276 (5317), 1425-1428 (1997).
- Kilian, K. A., Bugarija, B., Lahn, B. T., Mrksich, M. Geometric cues for directing the differentiation of mesenchymal stem cells. Proc Natl Acad Sci USA. 107 (11), 4872 (2010).
- Nelson, C. M., Chen, C. S. Cell-cell signaling by direct contact increases cell proliferation via a PI3K-dependent signal. FEBS Lett. 514 (2-3), 238-242 (2002).
- Hui, E. E., Bhatia, S. N. Micromechanical control of cell-cell interactions. Proc. Natl. Acad. Sci. U. S. A. 104 (14), 5722-5726 (2007).
- Lutolf, M. P., Blau, H. M. Artificial stem cell niches. Adv Mater. 21 (32-33), 3255-3268 (2009).
- Gobaa, S., et al. Artificial niche microarrays for probing single stem cell fate in high throughput. Nat. Methods. 8 (11), 949-955 (2011).
- DeForest, C. A., Anseth, K. S. Cytocompatible click-based hydrogels with dynamically tunable properties through orthogonal photoconjugation and photocleavage reactions. Nat Chem. 3 (12), 925-931 (2011).
- Nelson, C. M., VanDuijn, M. M., Inman, J. L., Fletcher, D. A., Bissell, M. J. Tissue geometry determines sites of mammary branching morphogenesis in organotypic cultures. Sci. STKE. 314 (5797), 298 (2006).
- Liu Tsang, V., et al. Fabrication of 3D hepatic tissues by additive photopatterning of cellular hydrogels. FASEB J. 21 (3), 790-801 (2007).
- Albrecht, D. R., Underhill, G. H., Mendelson, A., Bhatia, S. N. Multiphase electropatterning of cells and biomaterials. Lab. Chip. 7 (6), 702-709 (2007).
- Chan, V., Zorlutuna, P., Jeong, J. H., Kong, H., Bashir, R. Three-dimensional photopatterning of hydrogels using stereolithography for long-term cell encapsulation. Lab. Chip. 10 (16), 2062-2070 (2010).
- Boghaert, E., et al. Host epithelial geometry regulates breast cancer cell invasiveness. Proc. Natl. Acad. Sci. U. S. A. 109 (48), 19632-19637 (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon