È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Attuatore a base di cellule muscolari cardiache e bioroboto auto-stabilizzante - parte 2
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
In questo studio, un attuatore biologico e un bioroboto di nuoto auto-stabilizzante con bracci elastomerici funzionalizzati sono innalzati con cardiomiociti, coltivati e caratterizzati per le loro proprietà biochimiche e biomeccaniche nel tempo.
Abstract
Negli ultimi anni sono stati sviluppati dispositivi ibridi consistenti in una cellula viva o componente tissutale integrata con una spina dorsale meccanica sintetica. Questi dispositivi, chiamati biorobot, sono alimentati esclusivamente dalla forza generata dall'attività contrattile del componente vivo e, a causa dei loro molti vantaggi inerenti, potrebbe essere un'alternativa ai robot completamente artificiali convenzionali. Qui descriviamo i metodi di semina e caratterizzano un attuatore biologico e un biorobot che è stato progettato, fabbricato e funzionalizzato nella prima parte di questo articolo a due parti. Gli attuatori biologici e dispositivi biorobotici fabbricati costituiti da una base di polidimetilsilossano (PDMS) e da un sottilissimo film sono stati funzionalizzati per l'attaccamento cellulare con fibronectina. Dopo la funzionalizzazione, i cardiomiociti dei topi neonatali sono stati seminati sul braccio di PDMS ad alta densità, con conseguente un foglio di cellule confluente. I dispositivi sono stati fotografati ogni giorno e il movimento del cantiÈ stato analizzato il braccio delle leve. Il secondo giorno dopo la semina abbiamo osservato la flessione delle braccia a sbalzo a causa delle forze esercitate dalle cellule durante le contrazioni spontanee. A seguito di analisi quantitativa della piegatura a cantilever, è stato osservato un graduale aumento dello sforzo di superficie esercitato dalle cellule in maturazione nel tempo. Allo stesso modo, abbiamo osservato il movimento del biorobot a causa dell'attivazione del braccio cantonale PDMS, che ha agito come una pinna. Alla quantificazione dei profili di nuoto dei dispositivi sono stati osservati diversi modi di propulsione, influenzati dall'angolo di riposo della pinna. La direzione del movimento e la frequenza di battitura sono state determinate anche dall'angolo di riposo della pinna e si è osservata una velocità massima di nuoto di 142 μm / s. In questo manoscritto descriviamo la procedura per la popolazione dei dispositivi fabbricati con cardiomiociti, nonché per la valutazione dell'attivazione biologica e dell'attività biorobotica.
Introduzione
I biorobotti sono dispositivi basati su cellule viventi che sono incorporate all'interno di una spina dorsale meccanica che di solito è composta da materiali morbidi e elastici, come PDMS o idrogeli 1 . Le cellule subiscono contrazioni ritmiche, spontaneamente o in risposta a stimoli, e quindi funzionano come attuatori. L'energia generata dalla contrazione delle cellule spinge vari biorobot. Le cellule del cuore dei mammiferi (cardiomiociti) e le cellule muscolari scheletriche vengono spesso utilizzate per l'azionamento biorobotico a causa delle loro proprietà contrattili. Oltre alle cellule muscolo cardiomiociti e scheletriche, sono stati utilizzati altri tipi di cellule, quali i tessuti muscolari degli insetti 2 e i tessuti muscolari esplorati 3 . I tessuti muscolari degli insetti consentono il funzionamento di attuatori biologici a temperatura ambiente.
La funzione e le prestazioni di un biorobot sono principalmente determinate dalla forza e dalla consistenza dell'attuatore biologico ( es. Cellule muscolari), mentre la struttura meccanica della spina dorsale determina principalmente i meccanismi di locomozione, stabilità e potenza. Poiché questi dispositivi sono guidati esclusivamente da forze generate dalle cellule, non ci sono inquinanti chimici o rumori di funzionamento. Pertanto, essi costituiscono un'alternativa energeticamente efficiente ad altri robot convenzionali. Diverse fonti di letteratura hanno discusso i diversi metodi per integrare le cellule viventi ei tessuti in biorobot 1 , 4 , 5 . I progressi nelle tecniche di microfabbricazione e di ingegneria del tessuto hanno permesso lo sviluppo di biotubi che possono camminare, prendere a galla, nuotare o pompare 5 , 6 . In generale, le cellule vengono coltivate direttamente sulla spina dorsale meccanica (polimerica) come un foglio di cellule confluente o sono modellate in strutture di attuazione tridimensionali all'interno di fondi come gli anelli e le strisce. Più spesso, i biorobotti sonoFabbricati utilizzando fogli cardiomiociti 6 , 7 , poiché queste cellule hanno una capacità innata di esporre contrazioni spontanee senza stimoli esterni. D'altro canto, le relazioni sulle fogli delle cellule muscolari scheletriche sono limitate a causa della loro necessità di stimoli per avviare contrazioni in vitro per iniziare la depolarizzazione della membrana 8 .
Questo protocollo descrive innanzitutto come i cardiomiociti di seme su un attuatore biologico funzionalizzato realizzato in un sottile PDMS cantilever. Esso descrive in dettaglio la semina e l'analisi dei profili di nuoto. Il cantilever è funzionalizzato con una proteina adesiva cellulare come la fibronectina e viene seminata confluentemente con cardiomiociti. Poiché le cellule seminate sul contratto di dispositivo, provocano la piegatura del cantilever e quindi agiscono come attuatori. Nel tempo, quando le cellule si maturano, tracciamo i cambiamenti nello stress superficiale sul dispositivo analizzando i video diPiegatura a sbalzo. L'attuatore biologico sviluppato qui può essere utilizzato per determinare le proprietà contrattili di qualsiasi tipo di cellule, come i fibroblasti o le cellule staminali pluripotenti indotte, in quanto subiscono differenziazione.
Gran parte delle precedenti ricerche sui biorobotti è stata incentrata sullo sviluppo di attuatori biologici, mentre l'ottimizzazione dell'architettura biorobotica e delle funzionalità funzionali sono state largamente trascurate. Recentemente, alcuni studi hanno dimostrato l'attuazione di modalità di nuoto in biorobotti ispirati alla natura. Ad esempio, i biorobotti per il nuoto con motion 6 a base flagella, la propulsione delle meduse 9 e i raggi bio-ibridi 4 sono stati progettati. A differenza di altre opere in letteratura, ci concentriamo a variare le proprietà della spina dorsale meccanica per creare una struttura auto-stabilizzatrice. Il biotore sviluppato in questo studio è in grado di mantenere un passo costante, rotolo e imProfondità di mersione mentre nuota. Questi parametri possono essere modificati variando lo spessore di ogni composito base. Le fasi di fabbricazione coinvolte nello sviluppo dell'attuatore PDMS, del bioroboto submergibile e della funzionalizzazione del dispositivo sono descritte in dettaglio nella Parte 1 di questo articolo a due parti, nonché nel nostro lavoro recente 7. La tecnica sviluppata qui può aprire la Modo per lo sviluppo di nuovi biorobotti di grande efficienza per varie applicazioni, come la consegna dei carichi.
Il processo di isolamento seguito in questo studio è simile al processo descritto in un precedente lavoro 10 , così come nel lavoro pubblicato di recente 7 . I metodi di microfabbricazione utilizzati per la fabbricazione degli attuatori PDMS e dei dispositivi biorobot sono descritti in dettaglio nella Parte 1 di questo manoscritto a due parti. La sezione del protocollo di questo manoscritto descrive le fasi di sperimentazione dei cardiomiociti sul PDMS fabbricato aCtuator e il bioroboto dopo la loro funzionalizzazione con proteine adesive cellulari.
Protocollo
Tutte le procedure qui descritte sono state effettuate utilizzando un protocollo approvato e conformemente alle norme del Comitato istituzionale per la cura e l'uso degli animali dell'Università di Notre Dame.
1. Sementi di cellule e cultura
- Prima di iniziare preparare gli elementi necessari: un piccolo imbuto, pipette e caldo Dulbecco's Modified Eagle Medium (DMEM), completato con 10% Fetal Bovine Serum (FBS) e 1% di antibiotico penicillino (completo DMEM).
- Prendete le fiale T-25 con il dispositivo funzionalizzato (attuatore biologico o il bioroboto) all'interno di esso. Fare riferimento alla sezione 4 della parte 1 di questo manoscritto a due parti per i dettagli relativi alla preparazione, alla funzionalizzazione e allo stoccaggio dei dispositivi prima della semina delle cellule.
- Preparare un imbuto, che può essere fatto facendo rotolare un foglio di plastica quadrato. Posizionarlo sopra l'attuatore biologico o il biorobot all'interno della fiala T-25. Regolare il diametro dell'estremità più ampia per adattarlo all'intero dispositivoE l'altezza in modo che si adatta perfettamente quando la parte superiore del pallone viene serrata.
- Per i biorobot, utilizzare un magnete per tenere il dispositivo in posizione sul fondo del pallone durante il processo di semina.
Nota: Qui è stato utilizzato un singolo magnete del disco neodimio (1,26 "di diametro), ma qualsiasi magnete di dimensioni e resistenza simili può anche essere utilizzato per contenere il biorobot con la base magnetica del nichel-PDMS composito. - UV sterilizzare il foglio di plastica per almeno 30 minuti prima di utilizzare.
- Per i biorobot, utilizzare un magnete per tenere il dispositivo in posizione sul fondo del pallone durante il processo di semina.
- Assicurarsi che non vi sia un grande divario tra la base dell'imbuto e il pallone.
- Resuspendere i cardiomiociti in DMEM completati ad una densità di 1,6 x 10 7 cellule / ml e rilasciare lentamente 400 μL di sospensione sul dispositivo attraverso l'imbuto. Utilizzare un emocitometro o qualsiasi altro contatore delle cellule per determinare il numero di cellule ottenute.
- Spostare lentamente il sistema nell'incubatore senza pregiudicare il dispositivo e l'imbutohin. Coltivare per 24 ore a 37 ° C.
- Dopo il periodo di incubazione, rimuovere lentamente l'imbuto, lavare delicatamente il campione con PBS e riempire il pallone con 10 ml di DMEM fresco completo.
Nota: Per i biorobotti, rimuovere il magnete in modo che il dispositivo sia in funzione.
2. Caratterizzazione biochimica
- Saggio del flusso del calcio
Nota: il dosaggio del flusso di calcio è effettuato per valutare l'interconnessione cellulare. La procedura di caricamento delle cellule con il fluorescente colorante specifico per ioni di calcio segue il processo descritto in un protocollo precedentemente stabilito 11 .- In primo luogo, preparare i materiali necessari, l'estere del fluoro-4-acetometil (AM) del calcio, il poliolo non tensioattivo del tensioattivo (vedi tabella dei materiali ) e la soluzione di sale di Tyrode.
- Utilizzando le pinzette lunghe, delicatamente trasferire il dispositivo dal pallone di coltura a un piatto di Petri da 35 mm con 2 mL di solfato di Tyrode's salt zione.
- In un tubo separato di centrifuga, prendere 1 ml di soluzione tirode calda (riscaldata a 37 ° C) e aggiungere 3-5 μl di colorante fluoro-4 AM (concentrazione di lavoro: 3-5 μM) e una parte uguale di tensioattivo non ionico Poliolo (concentrazione di lavoro: 0,2%). Sostituire la soluzione di campione con calda soluzione di Tyrode integrata con il colorante indicatore di calcio, fluo-4 e 0,2% di tensioattivo non ionico. Incubare per 25-30 minuti a 37 ° C.
- Rimuovere la soluzione di tintura e lavare delicatamente il campione con la soluzione fresca di Tyrode. Reincubare il campione in 2 ml di DMEM fresco completato per altri 30 minuti a 37 ° C prima dell'immagine.
Nota: I risultati di questo saggio e video correlati sono forniti nel lavoro pubblicato in precedenza 7 .
- immunofluorescenza
Nota: La doppia immunostaining di tutti i campioni è stata eseguita seguendo protocollo precedentemente stabilito> 12.- Innanzitutto, preparare 10% di siero di capra (GS) in soluzione salina tamponata in fosfato (PBS), 4% di paraformaldeide (PFA) in diH2O, il detergente di cellule di cellule 0,1% (vedere tabella dei materiali ) in PBS, anticorpi primari - anticorpi monoclonali monoclonali troponin-I e anticorpi monoclonali anti-coniglio-43), anticorpi secondari (coniugato Alexa 594 e IgG (H + L) Alexa 488 coniugato per capra) e DAPI.
Attenzione : la paraformaldeide è cancerogena. - Rimuovere il campione di interesse dal pallone e lavare delicatamente due volte con PBS. Fare riferimento alla sezione 4 della Parte 1 di questo manoscritto a due parti per i dettagli relativi alla preparazione e alla funzionalizzazione del campione.
- Aggiungere una gocciolina di PBS su una piccola copertura (diametro 12 mm o 15 mm). Tenete delicatamente la base del dispositivo con le pinzette e tagliate le braccia sottili PDMS (cantilever, figura 1 ) usando le forbici dall'estremità dove si collega alla parte superiore del bassoe. Trasferire le braccia a sbalzo sulla gocciolina con il lato aderito a cellule rivolto verso l'alto. La goccia di PBS impedirà alle cellule di essiccare.
- Fissare i campioni con il 4% di PFA e effettuare la duplice immunostaining dei campioni, come descritto in precedenza 12 .
- Dopo l'immagazzinamento, montare i campioni su una lastra di vetro pulita utilizzando un reagente di montaggio anti-sbiadito e mettere da parte, indisturbati, al buio per 24 ore.
- Ripetere la procedura per tutti i campioni.
Nota: i risultati di questo saggio e le relative immagini sono discusse in dettaglio nel lavoro pubblicato in precedenza 7 .
- Innanzitutto, preparare 10% di siero di capra (GS) in soluzione salina tamponata in fosfato (PBS), 4% di paraformaldeide (PFA) in diH2O, il detergente di cellule di cellule 0,1% (vedere tabella dei materiali ) in PBS, anticorpi primari - anticorpi monoclonali monoclonali troponin-I e anticorpi monoclonali anti-coniglio-43), anticorpi secondari (coniugato Alexa 594 e IgG (H + L) Alexa 488 coniugato per capra) e DAPI.
3. Imaging
- Posizionare la bombola T-25 in posizione verticale in un incubatore di CO 2 e preparare il sistema di imaging all'interno dell'incubatore. Registrare il dispositivo utilizzando una fotocamera (vedere la tabella dei materiali ) con un obiettivo zoom (vedere la tabella dei materiali ). Per una sorgente luminosa,Utilizzare una striscia di luci a LED.
Nota: una striscia di LED a luce bianca è stata utilizzata qui, ma tutti i normali LED funzioneranno anche. - Collegare la fotocamera a un sistema operativo e aprire il software specifico per la fotocamera (vedere la tabella dei materiali ). Fai clic sull'immagine della videocamera sotto la scheda "File" nel pannello superiore per aprire tutte le opzioni della fotocamera e scegliere la videocamera corretta.
- Scegli "live" dall'elenco delle schede del pannello superiore del software.
- Portare manualmente l'immagine regolando la manopola dell'obiettivo. Seleziona "Riduci alla regione di interesse (ROI)" dal pannello superiore. Quindi, disegnare manualmente un rettangolo nel telaio video, racchiudendo l'attuatore biologico e le braccia a sbalzo, per segnare il ROI.
Nota: la scelta di un ROI appropriato minimizza la dimensione dei file di immagine.- Nel caso dei biorobot, catturare l'intero schermo per registrare il movimento nuoto del dispositivo.
NOTA: Non è necessario disegnare un ROI per i biorobot
- Nel caso dei biorobot, catturare l'intero schermo per registrare il movimento nuoto del dispositivo.
- Prima di avviare la registrazione, selezionare "Impostazioni fotocamera" da una delle schede del pannello superiore sullo schermo. Impostare la frequenza fotogrammi regolando il rapporto di esposizione e pixel dell'immagine in diretta facendo scorrere la barra per ciascuno o inserendo manualmente i valori. Impostare i frame rate a circa 30 ± 2 fps.
Nota: la modifica del rapporto di esposizione e pixel cambia la luminosità e il contrasto dell'immagine in diretta. - Fai clic sul pulsante "Registra" dal pannello superiore del software per iniziare a registrare i video degli attuatori con una risoluzione di 1.000 x 1.000 pixel per 30 s. Ripetere il processo per tutti i campioni.
4. Analisi delle immagini degli attuatori biologici su una base stazionaria
- Analizzare le immagini usando un software di programmazione ( ad esempio, Matlab) che esegue lo script personalizzato. Vedere la tabella dei materiali e il Supplemental File per ulteriori dettagli.
Nota: Lo script visualizza ogni fotogramma dei filmati registrati, riceve l'input del mouse dell'utente per registrare il coordinamento dei punti del cantilever sulle immagini, calcola il diametro e il centro del cerchio che passa i punti inseriti tramite il montaggio meno quadrato e Esporta tutti i dati inseriti e calcolati per ulteriore utilizzo.- Aprire il software di programmazione facendo clic sull'icona. Fai clic su "File" -> "Apri" dalla barra dei menu in alto e seleziona il file di script .m per l'analisi delle immagini. Assicurarsi che le immagini TIFF registrate siano nella stessa cartella del file m. Fai clic su "Esegui" per eseguire lo script.
Nota: viene visualizzata una finestra interattiva per l'alterazione. - Premere "play" per avviare il programma effettivo. Fai clic sul pulsante "Apri" e individua il file TIFF che verrà analizzato.
- Fai clic sul pulsante "base & #34; Quindi fai clic sul punto in cui il cantilever si attacca alla base in alto; Premere enter. Questo metterà un indicatore quadrato sull'immagine per ogni telaio per indicare l'ubicazione della base di cantilever.
- Fai clic sul pulsante "Scala" e poi clicca manualmente su un bordo del tallone di vetro. Portare il puntatore del mouse sul lato opposto del tallone di vetro e premere "Invio".
Nota: questo dovrebbe disegnare una linea che misura il diametro del tallone di vetro. Poiché il tallone di vetro è di 3 mm di diametro, ciò si riferirà a 3 mm ai pixel visualizzati. - Fai clic sul pulsante "Analizza". Fare clic lungo il cantilever a breve distanza dal primo marcatore quadrato che rappresenta la base di cantilever.
- Fai clic sul pulsante "Analizza". Quindi, cliccate lungo il cantilever a breve distanza dal primo marcatore quadrato che rappresenta la base a cantilever. Continua a scorrere lungo la sommità, compresa la punta e premere quando entrarefatto. Questo metterà un "x" su ogni punto cliccato sul cantilever.
Nota: Sulla base del coordinamento del marker quadrato e dei marcatori x, il centro e il diametro di un cerchio verranno calcolati utilizzando una funzione di montaggio meno quadrata (fare riferimento al file aggiuntivo allegato per lo script utilizzato). Il cerchio, che passa i marcatori x e il creatore quadrato, verrà sovrapposto all'immagine automaticamente. - Controllare che il cerchio sovrapposto correttamente traccia il profilo a sbalzo.
Nota: quando il cantilever è molto piatto, è difficile valutare se il profilo del cantilever è correttamente tracciato. Fare riferimento alla Figura 3 . - Fare clic sul pulsante "next frame". Questo passerà al fotogramma successivo nel file TIFF. La base e la scala sono già impostati dal passaggio precedente.
- Ripetere i passaggi da 4.1.5 a 4.1.7 finché non sono stati completati tutti i fotogrammi del file TIFF. Una volta che tutti i fotogrammi sono stati proceSsed, fai clic sul pulsante "Esporta".
Nota: creerà un file di foglio di calcolo con il nome del file TIFF per il cantilever appena analizzato. Modificare il nome del file per includere quale lato (sinistra o destra) del cantilever è stato analizzato.
- Aprire il software di programmazione facendo clic sull'icona. Fai clic su "File" -> "Apri" dalla barra dei menu in alto e seleziona il file di script .m per l'analisi delle immagini. Assicurarsi che le immagini TIFF registrate siano nella stessa cartella del file m. Fai clic su "Esegui" per eseguire lo script.
- Calcolo dello stress nel foglio di calcolo.
- Calcolare lo sforzo superficiale, "σ", sul cantilever utilizzando la seguente equazione:
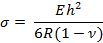
Dove E, R, v , e h sono il modulo Young, il raggio di curvatura, il rapporto di Poisson e lo spessore del cantilever.
Nota: lo spessore del cantilever può essere modificato per modificare la sensibilità. In questo studio i valori erano i seguenti: E = 750 kPa, v = 0.49 e h = 25 μm 13 , 14 . - Calcola lo sforzo di superficie utilizzando l'equazione nel punto 4.2.1. Apri il .Xls foglio di calcolo. Si noti che l'output ha più colonne che mostrano in primo luogo le coordinate x e y della base e del cerchio e quindi del raggio di curvatura. Calcola le coordinate x e y di ogni punto cliccate in base a queste.
Nota: La tracciatura delle sollecitazioni sui telai dell'immagine a tempo indica le variazioni della forza esercitata sul cantilever nel tempo. Le correnti mostrano lo stress sul cantilever durante il rilassamento dei cardiomiociti o lo sforzo statico esercitato sul cantilever a causa delle forze di trazione cellulare. Le cime mostrano lo stress dinamico sulla sommità, esercitata dal battito dei cardiomiociti. Questo valore corrisponde alla quantità massima di forza generata dalla contrazione dei cardiomiociti.
- Calcolare lo sforzo superficiale, "σ", sul cantilever utilizzando la seguente equazione:
5. Analisi dei biorobotti di nuoto
- Registrare la posizione del bioroboto usando il software di analisi delle immagini.
Nota: Consultare l'elenco Materiali per il software utilizzato in thÈ sezione.- Aprire il software di analisi delle immagini ( ad esempio, ImageJ). Premere "File" e "Apri" e selezionare il file video bioroboto per il nuoto. Fare clic su "OK" e lasciare che il programma carichi il file. Aprire il software del foglio di calcolo.
- Nel video biorobot caricato, trovare un riferimento di dimensioni noti ( ad esempio, il tallone di vetro di 3 mm di diametro incorporato nell'attuatore biologico).
Nota: Qualsiasi oggetto con dimensione nota funzionerà. Ciò determinerà i rapporti pixel-a-lunghezza in ciascun video. - Utilizzare lo strumento "Diritto" per disegnare una linea sul tallone di vetro. Fai clic su "Analizza" e seleziona "Imposta scala". Impostare il campo "Distanza nota" a "3.000 μm" e fare clic su "Ok".
Nota: questo imposta le coordinate x e y ai micrometri. - Scegli un punto sul dispositivo che non si avvicina tra i fotogrammi per agire come marcatore.
Nota:Si consiglia di scegliere un angolo della base. - Punto al punto selezionato in 5.1.4 sul primo fotogramma. Registrare le coordinate x e y sul foglio di calcolo.
- Passare alla finestra del software di analisi delle immagini e premere il tasto freccia destra per passare al fotogramma successivo. Punta nuovamente l'indicatore (dal punto 5.1.4) e registrare le coordinate x e y sul foglio di calcolo.
- Ripetere il passo 5.1.6 per tutti i fotogrammi.
- Calcola i parametri nuoto del biorobot utilizzando il foglio di calcolo delle coordinate 7 .
- Calcola il periodo tra i fotogrammi dalla velocità di trasmissione nota di ciascun video.
- Calcolare la variazione delle coordinate x e y tra i fotogrammi per trovare la distanza spostata, compresa la distanza totale.
- Calcolare l'ampiezza della contrazione dalla variazione massima lungo l'asse y. Determinare la frequenza di battitura per ogni biorobot dall'inverso del periodo tra due contrazioni.
- CAlcolare la velocità di nuoto di ogni dispositivo dal tempo totale e dalla distanza spostati nella x-direzione.
- Ripetere la fase 5.2 per ogni video biorobot che è stato analizzato.
- Normalizzare ogni parametro misurato.
Nota: Normalizza tutti i valori per visualizzare meglio le differenze. Questo protocollo dimostra la normalizzazione rispetto a un bioroboto a modalità orizzontale con contrazioni a bassa frequenza (LF orizzontale) ( Figura 4 ) 7 .
6. Analisi dell'espressione di proteine
Nota: i campioni montati preparati nei punti 2.2.4 e 2.2.5 sono stati eseguiti utilizzando un microscopio confocale. Le immagini sono state acquisite a 20X, 40X e 60X in sequenza in tre canali contemporaneamente: 460 nm, 488 nm e 594 nm. Un insieme di 5 immagini è stato catturato a 40x ingrandimento, da diverse posizioni per ogni campione e ogni canale è stato salvato come singolo .TIFFfile. L'impostazione dell'esposizione è stata determinata dall'ingrandimento dell'obiettivo utilizzato ed è stato impostato costante per tutte le catture a tale ingrandimento.
- Aprire il software di analisi delle immagini e selezionare "File" -> "Apri" per caricare le immagini.
- Disegna un poligono rettangolare sul telaio dell'immagine per contrassegnare il ROI. Selezionare "Analizza" -> "Misura" per misurare l'intensità fluorescente media.
- Ripetere il passo 6.2 per raccogliere le misure di intensità da tutti i campioni e calcolare la rispettiva intensità media per ogni condizione.
Nota: qui, una condizione diversa si riferisce a punti temporali diversi, come nel giorno 1, giorno 2 e fino al giorno 6. - Esportare i risultati su un foglio di calcolo per ulteriori analisi statistiche e per la produzione di grafici di dati.
Risultati
L'attuatore biologico realizzato in un sottile PDMS cantilever (25 μm di spessore) e cardiomiociti costituisce il nucleo del bioroboto nuoto, come mostrato nello schema e nella schermata dei dispositivi di Figura 1 . Le cellule iniziano a manifestare contrazioni dopo 24 ore di coltura e la curvatura delle braccia a braccio è stata osservata al giorno 2. Il profilo laterale del dispositivo è stato registrato ogni giorno e lo sforzo di superficie è stato quantifica...
Discussione
La procedura descritta qui descrive un metodo di semina di successo per attuatori e biorobotti basati su PDMS, che facilita l'attaccamento dei cardiomiociti. Inoltre, è stato descritto il processo di acquisizione di immagini e l'analisi successiva che caratterizza il comportamento delle cellule e le prestazioni dei dispositivi.
Abbiamo osservato la contrazione spontanea delle cellule sulle braccia a sbalzo dopo 24 h; L'intensità delle contrazioni ha continuato ad aumentare cost...
Divulgazioni
Gli autori non hanno niente da rivelare.
Riconoscimenti
MT Holley è sostenuto dal programma dei laureati della Louisiana Board of Regents e C. Danielson è sostenuto dal programma Howard Hughes Medical Institute Professors. Questo studio è supportato da NSF Grant No: 1530884.
Materiali
| Name | Company | Catalog Number | Comments |
| Chemicals and reagents | |||
| Cardiomyocytes (primary cardiac cells) | Charles River | NA | Isolated from 2-day old neonatal Sprague Dawley rats |
| Dulbecco’s modified eagle’s media (DMEM) | Hyclone Laboratories | 16750-074 | with 4500 mg/L glucose, 4.0 mM L-glutamine, and 110 mg/L sodium pyruvate |
| Fetalclone III serum | Hyclone industries, GE | 16777-240 | Fetal bovin serum (FBS) |
| Dulbecco’s phosphate buffer (PBS) | Sigma-Aldrich | D1408-100ML | |
| Penicillin-G sodium salt | Sigma-Aldrich | P3032 | |
| Goat serum | Sigma-Aldrich | G9023 | |
| 4,6-diamidino-2-phenylindole dihydrocholride powder (DAPI) | Sigma-Aldrich | D9542 | |
| Fibronectin from bovine plasma | Sigma-Aldrich | F1141 | Solution (1 mg/ml) |
| Calcein-AM and ethidium homodimer-1 kit (Live/Dead Assay) | Molecular Probes | L3224 | |
| Calcium Fluo-4, AM | Molecular Probes | F14217 | calcium indicator dye |
| Tyrodes salt solution | Sigma-Aldrich | T2397 | buffer solution |
| Pluronic F-127 | Molecular Probes | P3000MP | nonionic surfactant-20 % solution in Dimethylsiloxane (DMSO) |
| 16% Parafomaldehyde | Electron microscopy | 15710 | Caution: Irritant and combustible |
| Triton x-100 | Sigma-Aldrich | X-100 100 mL | cell lyses detergent, (4-(1,1,3,3-Tetramethylbutyl)phenyl-polyethylene glycol, t-Octylphenoxypolyethoxyethanol, Polyethylene glycol tert-octylphenyl ether) |
| ProLong gold antifade reagent | Molecular Probes | P10144 | Mounting agent |
| Alexa Fluor 594 Phalloidin | Molecular Probes | A12381 | Actin filament marker |
| Goat anti-rabbit IgG (H+L) secondary antibody, Alexa Fluor 594 conjugate | Molecular Probes | A-11012 | |
| pha | Molecular Probes | A-11001 | |
| Anti-connexin 43 antibody | Abcam | ab11370 | Gap junction marker |
| Anti-cardiac troponin I antibody | Abcam | ab10231 | Contractile protein |
| 16% EM grade paraformaldehyde solution | Electron microscopy | 100503-916 | |
| Polydimethylsiloxane (PDMS) | Elsevier | Sylgard 184 | |
| Materials and Equipment | |||
| Camera | Thor Labs | DCC1545M | |
| LED light strip | NA | NA | Any white LED without spectrum emission |
| Confocal microscope | Nikkon C2 | NA | Confocal microscope with three filter set. |
| Zooming lens | Infinity | Model# 252120 | |
| Software | |||
| Matlab | Mathworks | NA | Used in Section 4) for biological actuator analysis. |
| Image J | National Institute of Health | NA | Java-based image processing software. Used in Section 5) for biorobot analysis. Free Image Processing and Analysis software in java. (https://imagej.nih.gov/ij/) |
| Thor Cam | Thor Labs | NA | Camera operating software |
Riferimenti
- Feinberg, A. W. Biological Soft Robotics. Annu. Rev. Biomed. Eng. 17, 243-265 (2015).
- Akiyama, Y., et al. Room Temperature Operable Autonomously Moving Bio-Microrobot Powered by Insect Dorsal Vessel Tissue. PLOS ONE. 7, 38274 (2012).
- Herr, H., Dennis, R. G. A swimming robot actuated by living muscle tissue. J. NeuroEng Rehabil. 1, 6 (2004).
- Park, S., et al. Phototactic guidance of a tissue-engineered soft-robotic ray. Science. 353 (6295), 158-162 (2016).
- Cvetkovic, C., et al. Three-dimensionally printed biological machines powered by skeletal muscle. Proc. Natl. Acad. Sci. 111, 10125-10130 (2014).
- Williams, B. J., Anand, S. V., Rajagopalan, J., Saif, M. T. A. A self-propelled biohybrid swimmer at low Reynolds number. Nat. Commun. 5, (2014).
- Holley, M. T., Nagarajan, N., Danielson, C., Zorlutuna, P., Park, K. Development and characterization of muscle-based actuators for self-stabilizing swimming biorobots. Lab. Chip. 16, 3473-3484 (2016).
- Hopkins, P. M. Skeletal muscle physiology. Contin Educ Anaesth Crit Care Pain. 6, 1-6 (2006).
- Nawroth, J., et al. A tissue-engineered jellyfish with biomimetic propulsion. Nat Biotechnol. 30 (8), 729-797 (2012).
- Ehler, E., Moore-Morris, T., Lange, S. Isolation and Culture of Neonatal Mouse Cardiomyocytes. J. Vis. Exp. JoVE. (79), e50154 (2013).
- Bers, D. M. Calcium Fluxes Involved in Control of Cardiac Myocyte Contraction. Circ. Res. 87, 275-281 (2000).
- Shin, S. R., et al. Carbon-Nanotube-Embedded Hydrogel Sheets for Engineering Cardiac Constructs and Biological actuators. ACS Nano. 7, 2369-2380 (2013).
- Park, J., et al. Real-Time Measurement of the Contractile Forces of Self-Organized Cardiomyocytes on Hybrid Biopolymer Microcantilevers. Anal. Chem. 77, 6571-6580 (2005).
- Tamayo, J., et al. Quantification of the surface stress in microcantilever biosensors: revisiting Stoney's equation. Nanotechnology. 23, 475702 (2012).
- Nunes, S. S., et al. Biowire: a platform for maturation of human pluripotent stem cell-derived cardiomyocytes. Nat. Methods. 10, 781-787 (2013).
- Louch, W. E., Sheehan, K. A., Wolska, B. M. Methods in Cardiomyocyte Isolation, Culture, and Gene Transfer. J. Mol. Cell. Cardiol. 51, 288-298 (2011).
- Alford, P. W., Feinberg, A. W., Sheehy, S. P., Parker, K. K. Biohybrid thin films for measuring contractility in engineered cardiovascular muscle. Biomaterials. 31, 3613-3621 (2010).
- Sfakiotakis, M., Lane, D. M., Davies, J. B. C. Review of fish swimming modes for aquatic locomotion. IEEE J. Ocean. Eng. 24, 237-252 (1999).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon