Method Article
Determinazione di alta affinità antigene-anticorpo cinetica del legame con quattro piattaforme biosensore
In questo articolo
Riepilogo
Descriviamo qui Protocolli per la misurazione di anticorpo-antigene affinità e cinetica usando quattro piattaforme biosensore comunemente utilizzati vincolante.
Abstract
biosensori ottici privi di Label sono potenti strumenti nella scoperta di nuovi farmaci per la caratterizzazione delle interazioni biomolecolari. In questo studio, si descrive l'utilizzo di quattro piattaforme biosensore comunemente impiegati nel nostro laboratorio per valutare l'affinità di legame e cinetica di dieci anticorpi ad alta affinità monoclonali (mAb) contro proprotein umano convertasi subtilisina tipo KEXIN 9 (PCSK9). Mentre sia Biacore T100 e ProteOn XPR36 sono derivati dalla tecnologia (SPR) consolidata Plasmon superficie risonanza, il primo ha quattro celle di flusso collegati da configurazione flusso seriale, mentre il secondo presenta 36 punti di reazione in parallelo attraverso un improvvisato 6 x 6 incrociato configurazione del canale microfluidico. All'IBIS MX96 opera anche basato sulla tecnologia dei sensori SPR, con una funzione di imaging aggiuntivo che fornisce rilevamento orientamento spaziale. Questa tecnica di rilevazione accoppiato con il flusso continuo Microspotter (CFM) espande il throughput significativamente tramite enabling multiplex stampa array e il rilevamento di 96 sport di reazione contemporaneamente. Al contrario, l'Octet RED384 si basa sul principio ottico BioLayer interferometria (BLI), con sonde fibra ottica che agisce come biosensore per rilevare variazioni configurazione di interferenza dalle interazioni di legame alla superficie punta. A differenza delle piattaforme SPR-base, il sistema BLI non si basa su fluidica flusso continuo; invece, le punte del sensore raccogliere letture mentre sono immersi in soluzioni analita di 384 micropiastra durante l'agitazione orbitale.
Ognuna di queste piattaforme biosensore ha i suoi vantaggi e svantaggi. Per fornire un confronto diretto della capacità di questi strumenti per fornire dati cinetici di qualità, i protocolli descritti illustrano esperimenti che utilizzano lo stesso formato del test e gli stessi reagenti di alta qualità per caratterizzare la cinetica antigene-anticorpo che si adattano alla semplice 1: 1 modello di interazione molecolare .
Introduzione
The acquisition of reliable kinetic parameters for characterizing antibody-antigen interactions is an essential component of the drug discovery and development process1. Optical Surface Plasmon Resonance (SPR) biosensors, the "gold standard" for real-time detection of these interactions, have been used for approximately two decades to enable early selection of criteria-meeting therapeutic antibody candidates2,3. In addition to providing an affinity ranking of antibody candidates by the rapid binding screening of crude supernatants4, and rigorous kinetic constant determinations of purified preparations5, biosensors can further differentiate the functional activity of lead candidates via epitope binning studies6,7.
To meet the growing demand of antibody-based products for various therapeutic indications8, a wide variety of innovative biosensor instruments have been developed in recent years that increase the efficiency of candidate identification and characterization9,10. These instruments differ in microfluidic channel configuration design and/or in the optical principles involved in detecting biomolecular interactions. Specifically, the Octet RED38411, ProteOn XPR3612, and IBIS MX967 have expanded the number of interactions measured in a single binding cycle to 16, 36, and 96 respectively, representing significant throughput improvements over the traditional Biacore platform. Although these various biosensor platforms all provide essential kinetic data for characterizing antibody-antigen interactions, they differ in experimental setup and operational procedures due to variations in instrumental configurations. For example, in the IBIS MX96's SPR imaging array platform, the multiplex ligand immobilization step is performed in an off-line mode in an external printing device separate from the detector7,13; in contrast, the other three biosensor platforms utilize an "all-in-one" setup, where the addition of component onto the sensor surface, whether it is the activation reagent, ligand, or analyte, is recorded in real time and through pre-programmed commands in sequential order. In the Octet RED384, a unique feature of this BioLayer Interferometry (BLI)-based platform is the availability of pre-coated optical fiber biosensors for immediate "dip-and-read" use14, eliminating the need for ligand surface preparation and initiation/conditioning steps, which are often required for the sensor chips in SPR-based platforms. This instrument's fluidic-free design also simplifies the mechanics and avoids clogging and contamination concerns when dealing with crude samples. With the novel 6 x 6 crisscrossing fluidic design in the ProteOn XPR36, a "one-shot" kinetics approach can be implemented by switching the flow channels between horizontal and vertical directions to create 36 interaction spots15. Instead of assessing binding kinetics one ligand or analyte concentration at a time, as is done in the traditional serial flow Biacore T100 platform, this approach offers the ability to monitor up to 36 different interactions simultaneously in a single binding cycle.
Despite their differences, these four biosensor platforms are all widely used by many laboratories worldwide. For new users with little hands-on biosensor experience, deciding which instrument to use can be a challenging task given the differences in instrumental design. To determine the most appropriate instrument for their research purposes, factors such as data quality, performance consistency, throughput, ease of operation, and material consumption need to be considered collectively. While several benchmark studies have explored the variability of kinetic rate constants obtained from multiple laboratories and biosensors5,16, a recently published head-to-head comparison study further addresses the systematic factors that influence data reliability from the instrumental performance point of view17,18. The protocols supplied in this video focus on the experimental setup and procedures in detail, and are accompanied by the research article entitled, "Comparison of biosensor platforms in the evaluation of high affinity antibody-antigen binding kinetics"17. These protocols are intended not only to help new biosensor users implement these instruments for their research purposes, but also to provide additional insights for current biosensor users regarding technical challenges and considerations in experimental designs for evaluating high-affinity antibody-antigen interactions.
Protocollo
1. proteine e anticorpi
- Produrre e purificare la subtilisina proprotein convertasi umano KEXIN tipo 9 (PCSK9) e dieci mAb anti-topo PCSK9-derivato come descritto in precedenza 17.
- Valutare la purezza dei prodotti purificati sequenzialmente iniettando 10 mg di ciascun campione in una colonna analitica esclusione dimensionale (SEC) utilizzando un sistema UPLC 19.
2. Misure cinetica nel T100 Biacore
- Strumento e Preparazione dei reagenti
- Equilibrare la chip di sensore CM5 a temperatura ambiente per 15 min.
- Scongelare due aliquote 200 ml di 200 mM-etil-1 3- (3-dimetilamminopropil) carbodiimmide cloridrato (EDC) e due aliquote di 200 pl di 50 mM N-idrossisuccinimmide (NHS) a temperatura ambiente.
- Preparare una bottiglia 1-L di 1x HBS-EP (10 mM HEPES [pH 7,4], 150 mM NaCl, 3 mM EDTA, e 0,005% v / polisorbato v P20) di tampone di corsa perLUTING una soluzione 10x HBS-EP + magazzino in acqua.
- Preparare 1 ml di proteina A / G a 30 ug / ml in 10 mM acetato di sodio (pH 4,5) e 500 microlitri di ciascun campione mAb a 0,063 ug / ml nel tampone di corsa HBS-EP. Scongelare una fiala congelata di PCSK9 umana sul ghiaccio.
- Nel software di controllo, fai clic su "Strumenti | Eject Chip", posizionare il chip del sensore nella guaina e vicino. Fai clic su "Strumenti | temperatura impostata", inserire "25" ° C la temperatura di analisi e "10" ° C la temperatura del vano.
- L'immobilizzazione superficiale
- Nel software di controllo, fai clic su "File | Apri / Nuovo modello Wizard" e selezionare "immobilizzazione" dalla lista nel pannello di sinistra.
- Fai clic su "Nuovo" per accedere alla finestra di configurazione di immobilizzazione. Scegliere "CM5" come tipo di chip e selezionare "1" cella di flusso per ciclo.
- Seleziona la casella accanto a ogni "cella di flusso immobilizza 1/2/3/4" a activaTe le opzioni. Selezionare "Obiettivo per il livello immobilizzato" e "Amine" come metodo per tutte le celle di flusso.
- Inserire "30 ug / mL ProA / G" come ligando, garantire che il livello di destinazione è impostato su "10000" unità di risposta (RU) per tutte le celle di flusso.
- Controllare "Prime prima di correre", e cliccare su "Avanti". Selezionare "reagente Rack 2", definisce la posizione per i reagenti e pipetta in singole fiale campione di conseguenza. Inserire il rack di nuovo nello strumento, quindi fare clic su "Start" e inserire un nome dell'esperimento per essere salvato per iniziare la corsa.
- Analita Binding e cicli di rigenerazione
- Fai clic su "File | Open / New Method", selezionare "vincolante 20.140.812 hu anti-IgG PCSK9 a Hu PCSK9" e aprire il metodo. Rivedere i parametri nel metodo.
- In "Passi Assay", assicurano ci sono 10 cicli in cui "hu PCSK9" è il nome di "campione" selezionato "Purpose". Il numero di replica è impostata su '1'.
- In "Tipi Cycle", impostare il tempo di contatto a "220" s per la cattura 1 oltre percorso del flusso "2", "110" s per Capture 2 oltre percorso del flusso di "3" e "55" s per la cattura 3 oltre percorso del flusso "4". La portata è "10" microlitri / min per tutti i cicli di cattura.
- Selezionare "Esempio 1", selezionare "soluzione campione" variabile. Impostare il tempo di contatto a "600" s ed il tempo dissociazione a "2700" s oltre percorso di flusso "1, 2, 3, 4". La portata è impostata su "30" microlitri / min.
- Selezionare "Rigenerazione 1", inserire "glicina-HCl pH 1,5" nel campo della soluzione e impostare il tempo di contatto a "20" s oltre percorso del flusso "1, 2, 3, 4".
- In "Impostazioni delle variabili", immettere 2 volte titolazione concentrazioni di "100 nM - 6,25 nM" per Cycle 1-3, "50 nM - 3.12 nM" per Cycle 4-8, e "5 nM - 0,323 nM" fo Ciclo 9-10.
- In "Setup Run", scegliere il percorso del flusso di rilevazione "per 2-1, 3-1, 4-1", fare clic su "Avanti", selezionare "Prime prima di correre", e cliccare su "Avanti".
- Selezionare "campione e di reattivo Rack 1", definire la posizione per i reagenti e pipetta in singole fiale campione di conseguenza. Inserire il rack nello strumento, quindi fare clic su "Start" e inserire un nome dell'esperimento per essere salvato per iniziare la corsa.
- Analisi dei dati mediante BiaEvaluation
- Fai clic su "Cinetica / Affinity | Superficie legato", scegli Fc "2-1", "3-1", o "4-1" a parte e controllare tutte le curve mostrate dalle varie concentrazioni degli analiti così come lo "zero" blank concentrazione.
- "Avanti" per ottenere il buffer vuoto curve sottratti. Quindi fare clic su "Cinetica", scegliere il: modello di "1 1 Rilegatura", e cliccate su "Fit" di ottenere il Parame cinetica montatoTERS.
- Per il montaggio globale di più superfici mAb, cliccare su "Multiple max R" in fondo e aggiungere le curve di legame dagli altri FC per la stessa mAb alla lista.
- "Avanti" per ottenere tutti i buffer di bianco sottratti curve vincolanti. Quindi fare clic su "Cinetica", scegliere "1: 1 Rilegatura" modello, e scegliere "fit locale" per ogni R max nei parametri per ottenere i parametri cinetici montati a livello globale.
3. Misurazioni cinetica nel XPR36 ProteOn
- Strumento e Preparazione dei reagenti
- Equilibrare un chip sensore GLM e una bottiglia da 2 L di PBS-T-EDTA (PBS [pH 7,4], 0,005% Tween-20, e 3 mM EDTA) tampone di corsa a temperatura ambiente per 15 min.
- Nel software di controllo, fai clic su "Eject" e quindi inserire il chip GLM. Impostare la temperatura del chip a "25" ° C e la temperatura autocampionatore a "10" ° C.Fai clic su "Glicerina inizializzazione" e seguire le istruzioni spinto per inizializzare il chip.
- Fai clic su "File | Open" un protocollo di precondizionamento dalla lista, preparare le soluzioni in una piastra da 96 pozzetti. Quindi fare clic su "Esegui", selezionare il protocollo e fare clic su "Start".
- Scongelare un'aliquota 1 ml di 400 mM EDC ed un'aliquota 1 ml di 100 mM N-hydroxysulfosuccinimide (solfo-NHS) a temperatura ambiente.
- Preparare 2 ml di proteina A / G a 60 ug / ml in 10 acetato mM di sodio (pH 4,5) e 500 microlitri di ciascun campione mAb a 0.25 mg / mL, 0,125 mg / ml, e 0,063 mg / ml in PBS-T -EDTA tampone di corsa. Scongelare una fiala congelata di PCSK9 umana sul ghiaccio.
- L'immobilizzazione, Analita Binding e Rigenerazione
- Fai clic su "File | New | Nuovo protocollo". In "Configurazione", inserire il nome dell'esperimento, selezionare chip "GLM", selezionare "Micropiastre", e inserire il volume mL "2.2".
- Selezionare"Protocolli | Campioni", definiscono "EDC / solfo-NHS" come "Activator" in pozzetti H1-H6, "proteina A / G" come "Ligand" in G1-G6, "Etanolamina" come "Disattivatore" in F1-F6 , "Glycine" come "Regenerator" in E1-E6, PBS-T-EDTA "come "vuoto" in D1-D6, "X campione mAb" come "ligando" in C1-C6, e "PCSK9 umana" come" Analita in pozzetti H1-H6 in una seconda piastra a 96 pozzetti.
- Selezionare "Passi", fare doppio clic su "immobilizzazione" nel "Protocollo Editor", i passaggi includono tre successive iniezioni orizzontali di EDC / solfo-NHS, proteina A / G e 1 M etanolammina ciascuno con una portata di "30" microlitri / min e un tempo di contatto di "300" s.
- Istruzioni "Rigenerare", iniettare tre "18" -s impulsi di glicina (pH 1,5) a "100" microlitri / min in entrambe le direzioni orizzontale e verticale, seguito da due "60" -s impulsi di PBS-T-EDTA a "25" microlitri / minanche in entrambe le direzioni.
- Istruzioni "Ligand", iniettare mAb 1 mAb 2 nella direzione verticale utilizzando una portata di "25" microlitri / min e un tempo di contatto di "160" s.
- Clicca "Analita", iniettare cinque concentrazioni di PCSK9 umana (100 nM a 6,25 nM a 2-diluizioni seriali) e un buffer vuoto su 6 canali orizzontali simultaneo, "40" microlitri / min per "600" s di tempo dell'associazione seguiti da "2700" s di tempo di dissociazione.
- Scegliere "Rigenera", iniettare due "18" -s impulsi di glicina (pH 1,5) a "100" microlitri / min in entrambe le direzioni orizzontale e verticale.
- Ripetere i passaggi sopra dopo ogni ciclo di legame per i restanti mAbs.
NOTA: In aggiunta alle 100 nM - 6,25 Nm Serie concentrazione PCSK9 umana, 25 nM - 1.56 nm e 5 nM - vengono utilizzati 0.313 serie di concentramento nM. - Preparare i campioni e reagenti in due piastre da 96 pozzetti secondo le posizioni del reagentedefinito nel passaggio 3.2.2, quindi sulla scheda "Run", selezionare il protocollo / esperimento e fare clic su "Start".
- Analisi dei dati mediante ProteOn Responsabile
- Fai clic su "Tipo di schermo" e selezionare "Analita". Quindi fare clic su "Protocollo Step" e selezionare tutte le curve di legame nella lista.
- Fai clic su "Processo Auto", e fare clic su "Processo | Canale di riferimento | Interspot". Quindi fare clic su "Processo | Doppia Riferimento | Row | A6".
- Fai clic su "Crea set di dati" per salvare i singoli set di dati per ogni mAb in tutte e tre le superfici per l'analisi cinetica.
- Fai clic su "Analisi Dataset", evidenziare ogni set di dati mAb e fare clic su "Analisi | cinetica". Scegli il modello "Langmuir" e "simultanea ka / kd" e fare clic su "Avanti".
- Evidenziare sia l'associazione e la dissociazione gamma adatta, scegliete "Locale" R max, e fare clic su "Avanti" per eseguire la misura per ottenerei parametri cinetici muro.
- Ripetere la stessa operazione, ma scegliere "raggruppato" R max per il montaggio per ottenere i valori sperimentali R max per il calcolo della attività di superficie ligando.
4. Misure cinetica nel Octet RED384
- Reagente e piatto preparazione del campione
- Preparare 50 mL di 1x KB (Kinetic Buffer contenente PBS pH [7,4], 0,02% Tween-20, 0,1% di albumina, e 0,05% di sodio azide) diluendo una soluzione stock 10X in PBS.
- Versare il 1x KB in una piastra a 96 pozzetti in 200 microlitri per pozzetto. Idrato 48 cattura anti-umano (AHC) punte biosensore in questi singoli pozzetti per almeno 10 minuti.
- Preparare ciascun campione mAb a 20 ug / ml, 10 ug / ml, e 5 ug / ml in 1x KB, nonché PCSK9 umana a 7 concentrazioni gradualmente da 100 nM a 1,56 nM a 2-diluizioni seriali.
- Caricare i campioni mAb in una micropiastra a 384 pozzetti inclinato fondo (refeRred come la piastra reagente) e le soluzioni PCSK9 umani in un'altra 384 pozzetti micropiastra (denominato piastra del campione) a 90 microlitri per pozzetto.
- serie di carico 1x KB e glicina (pH 1,5) soluzioni in pozzi all'interno piatto del campione.
NOTA: Questi pozzi KB 1x sono utilizzati per chip di precondizionamento, la stabilizzazione della linea di base, e analiti dissociazione. La soluzione glicina viene utilizzato per la rigenerazione.
- Impostazione metodo sperimentale
- Nel software di acquisizione dei dati, fai clic su "Esperimento | Nuovo Esperimento Wizard | Nuova Cinetica Experiment | Cinetica di base".
- In "Piastra Definizione", attenersi alla seguente procedura per definire i tipi di campione e le posizioni.
- Selezionare i canali "16" e il formato "384-bene".
- Definire le posizioni campione e di reattivo nel display piatto tenendo premuto il tasto Shift mentre sinistro del mouse al pozzo per evidenziare 16 pozzetti alla volta.
- Fare clic destro sullapozzetti contenenti i campioni mAb e selezionare "Load" per significare la fase in cui i mAbs sono caricati (o catturati) sui sensori AHC. Inserire i nomi e le concentrazioni di mAb nella tabella sul lato destro.
- clicca destra nei pozzetti contenenti il PCSK9 umana e selezionare "campione" per indicare la fase in cui i sensori mAb catturati vengono immersi e le interazioni di legame vengono misurati. Immettere i corrispondenti concentrazioni molari nella tabella sul lato destro.
- Definire i pozzetti contenenti 1x KB come "buffer" o "neutralizzazione", ei pozzetti contenenti glicina come "rigenerazione".
- In "Definizione Assay", seguire le istruzioni riportate di seguito per definire il setup sperimentale:
- Fai clic su "Aggiungi" e selezionare "Baseline", "Rigenerazione", "equilibrazione", "Caricamento", "Associazione", e passi "dissociazione" all'elenco con tempi di "30" s, "30" s, "18" s, "200" s, "500" s, e "1800" s, rispettivamente. La velocità shake è "1000" rpm.
- Per aggiungere la procedura per "Lista Assay passi", primo click su un gradino, quindi fare clic sui pozzetti situati sia nel campione o la piastra di reagente. I passi sono i seguenti:
- Equilibrazione-Rigenerazione: precondizione i sensori AHC da tre cicli di "15" -s cali di glicina (pH 1,5), alternate a "15" -s tuffi in 1x KB.
- Caricamento: catturare i campioni mAb sui sensori AHC per "200" s.
- Baseline: stabilire il segnale BLI per "60" s prima della fase di associazione.
- Associazione: immergere i sensori in pozzetti contenenti concentrazioni variabili di PCSK9 umana per un periodo associazione -s "500".
- Dissociazione: immergere i sensori PCSK9-bound in nuovi pozzi di 1x KB per una durata dissociazione -s "1.800".
- Rigenerazione: immergere il mAb-PCSK9 bound sensori nei pozzetti di glicina (pH 1,5) con due "18" -s impulsi tra ogni ciclo vincolante.
- In "Esperimenti Recensione", far scorrere attraverso i passi e apportare le modifiche necessarie per tornare alle schede precedenti.
- In "Esperimenti Run", inserire un tempo "60" S ritardo e scuotere il piatto del campione durante l'attesa. Impostare la temperatura della piastra a "25" ° C e premere "GO".
- Analisi dei dati utilizzando software di analisi dei dati di ForteBio
- Fai clic su "Selezione dei dati | dati caricati | Cinetica | anticorpi PCSK9 vincolante per PCSK9 7.23.14"
- In "Processing", elaborare i dati come segue:
- In Fase 1: fare clic su "Selezione del sensore" a sottolineare sensori in H1-H6, fare clic destro su di essi e fare clic su "Cambia tipo di sensore | sensore di riferimento."
- Nel passaggio 2, selezionare "sottrazione" e selezionare "Sensori medio di riferimento"; sottrarre ciascun sensore campione attivo dalla media dei 6 sensori tampone bianco.
- Nel passaggio 3, scegliere "Align asse Y" e selezionare "Baseline" per allineare tutte le curve di legame alla fase basale prima sezione.
- Al punto 5, selezionare "Savitskij-Golay Filtering" per lisciare le curve di legame, aumentando il rapporto segnale-rumore, e fare clic su "Process Data!".
- Nel passaggio 6, fare clic su "Risultati trasformati" per visualizzare le curve di legame trasformati.
- Nel passaggio 7, rivedere i quattro pannelli a destra che visualizza le curve di legame dopo ogni fase di lavorazione, e premi "Salva trattati dati".
- In "Analisi", selezionare "Associazione e dissociazione" e scegliere: il modello "1 1".
- Evidenziare le curve di legame per "Global (completo)" fit e li raggruppa per "colore". Utilizzare "R max collegato" per eseguire il gruppo di montaggio di sensori rivestiti con lo stesso mConcentrazione ab per ottenere i valori di R max sperimentali per calcolo dell'attività superficie ligando, e "R max scollegato dal sensore" per eseguire raccordo globale su sensori rivestiti con più concentrazioni mAb.
- Fai clic su "Fit Curve" per ottenere i parametri cinetici montati. Salvare i risultati alla fine di ogni analisi raccordo.
5. Misurazioni cinetica nel IBIS MX96
- Strumento e Preparazione dei reagenti
- Equilibrare un chip COOH-G a temperatura ambiente per 15 min.
- Preparare una bottiglia da 1 L di tampone di corsa del sistema (PBS [pH 7,4], 0,01% Tween-20) e il buffer immobilizzazione (acetato di sodio 10 mM [pH 5,0], 0,01% Tween-20).
- Predisporre ogni mAb da 20 ug / ml a 0,16 mg / mL per diluizioni seriali 2-fold sia nel buffer di sistema esecuzione e l'acetato di sodio (pH 5,0) tampone immobilizzazione.
- Dispensare i campioni mAb in due settembrearate 96-pozzetti in cui ogni mAb occupa 8 pozzi verticali nelle concentrazioni titolazione a 200 microlitri per pozzetto.
- aliquote scongelamento di 400 mM e 100 mM EDC sulfo-NHS a temperatura ambiente.
- Preparare 300 ml di proteina A / G a 50 ug / ml in acetato di sodio (pH 5,0) e tampone immobilizzazione pipetta in una fiala.
- Preparare 200 ml di PCSK9 umana da 100 nM a 0,39 nM per diluizioni seriali di 2 volte il sistema tampone di corsa. li pipetta in flaconcini separati.
- Cinetica cicli multipli con array di anticorpi ammine accoppiato
- Mescolare l'EDC / solfo-NHS (400 mM / 100 mM) aliquote ed erogare la miscela nella top 48 pozzetti di una nuova piastra a 96 pozzetti a 200 microlitri per pozzetto.
- Posizionare la piastra mAb sorgente (diluito in acetato di sodio [pH 5,0]) in posizione 2 e la piastra reagente EDC / sulfo-NHS in posizione 1 all'interno della stampante.
- Installare il chip sensore nel CFM. Aprire il "CFM 2.0" software, quindi fare clic su "Carica impostazioni | 96 Amine coppia". La matrice 10 x 8 anticorpo viene creato come segue:
- Consegnare l'EDC / solfo-NHS nella metà superiore della piastra reagente al sensore con 48 microcanali, e li ciclo sopra la superficie del sensore per "5" min.
- Fornire i campioni mAb nella metà superiore della piastra sorgente al sensore e li ciclo attraverso le superfici attivate per min "10".
- Consegnare il buffer immobilizzazione da un tubo conico da 50 mL sulla superficie anticorpo per "5" min.
- Ripetere le procedure sopra per le restanti campioni mAb nella metà inferiore della piastra sorgente.
- Ancorare il chip sensore stampata nello strumento. Nel software di controllo, fai clic su "File | Connetti | Aperto misura | Misura esistente" e selezionare "2014/08/15".
- In "Generale", selezionare "G - Sistema Prime" in "sceneggiatura completa del sistema". Quindi selezionare "G - QuEnch" per disattivare le superfici mAb iniettando 150 microlitri 1 M etanolammina.
- Fai clic su "Camera" per visualizzare le matrici mAb e regolare il contrasto, se necessario. Posizionare le caselle quadrate rosse a circondare le macchie mAb, e spostare le caselle quadrate verdi ai interspots situati tra i punti mAb attivi per riferimento.
- In "Cycle Analysis", selezionare "A - AP standard Run" sotto "Script IBIS". Set 1.0 min per basale, 10.0 min per l'associazione, e 90 passaggi per dissociazione a velocità di 8 microlitri / s.
- All'interno delle "Cicli", iniettare PCSK9 umano uno concentrazione alla volta dopo cinque iniezioni tampone. Rigenerare le superfici mAb con glicina pH 2,0 tra ogni ciclo di rilegatura.
- Cinetica ciclo unico con array di anticorpi Fc-catturato
- Installare un nuovo chip sensore nel CFM. Ripetere i punti 5.2.3 a 5.2.5 sopra sostituendo i campioni mAb con proteina A / G.
- Rimuovi ilchip del sensore dalla MX96 e inserirla nella stampante CFM.
- Consegnare i mAbs nella metà superiore della piastra alla proteina A / G superficie del sensore usando 48 microcanali, e li ciclo sulla superficie usando un flusso bidirezionale per 10 min.
- Ripetere la stessa operazione per i restanti campioni mAb nella metà inferiore della piastra.
- Ancorare il chip sensore stampata nello strumento MX96. Nel software di acquisizione dati, fare clic su "File | Connetti | Aperto misura | Misura esistente | Avanti" e selezionare "proteina A + G kinetic7.30.14 vincolante".
- Clicca "camera" per visualizzare le matrici mAb e regolare il contrasto, se necessario. Posizionare le caselle quadrate rosse a circondare le macchie mAb, e spostare le caselle quadrate verdi ai interspots situati tra i punti mAb attivi per riferimento.
- In "Cycle Analysis", selezionare "A - AP standard Run" sotto "Script IBIS". Impostare "1.0 min" per linea di base, "10,0min" per l'associazione, e '' passi per dissociazione '10 / sec' velocità 8 microlitri.
- All'interno delle "Cicli", iniettare PCSK9 umano uno concentrazione alla volta dopo cinque iniezioni tampone. No rigenerazione viene effettuata tra ogni iniezione analita.
- Analisi dei dati utilizzando Sprint e Scrubber
- Nella sofware SprintX, fai clic su "File | Apri file Triangolo", selezionare tutte le iniezioni di esempio nella lista, e selezionare "Salva file SprintX", "Calibrazione", e "la determinazione RLL" prima di cliccare "Analizza".
NOTA: I valori RLL riferiscono alle immobilizzate catturate livelli / mAb sulla superficie del sensore. - Controllare "Riferimento" e selezionare "locale" per utilizzare i punti di riferimento adiacenti. Verificate anche "Allineare" sulla prima iniezione e quindi fare clic su "Start Automation."
- Nella scheda seriale, selezionare "Mostra righelli nella barra degli strumenti," regolarleper selezionare una piccola regione della linea di base immediatamente prima della prima iniezione del campione, e selezionare "zero".
- Generare un file .IBMX per l'analisi nel Scrubber selezionando "Esporta in IBMX file" e quindi fare clic su "Start Automation."
- Avviare Scrubber e caricare il file .IBMX. Inserire "100N" come "della conc" e "2", come il "fattore di diluizione".
- dati raccolti, rimuovere tutti basale prima dell'inizio dell'iniezione, e allineare le curve di legame all'inizio di associazione.
NOTA: Per l'esperimento di cinetica a ciclo unico, non si allineano le curve. - Vai alla scheda "Cinetica", regolare il righello per il tempo della fine dell'iniezione, selezionare "k d" e in forma, poi "fix k d". Selezionare "k a k d" e montare di nuovo.
- Float la colonna K D e montare di nuovo per perfezionare ulteriormente la vestibilità.
NOTA: Per il montaggio di ciclo unico curve di legame, Cliccare su "Opz" e deselezionare "Sottrarre", quindi selezionare "separato" e galleggiare la colonna "Begin Inj" per adattarsi ai profili di associazione di nuovo ad un origine teorica di base. - Rivedere i parametri cinetici montati nella scheda Risultati.
- Nella sofware SprintX, fai clic su "File | Apri file Triangolo", selezionare tutte le iniezioni di esempio nella lista, e selezionare "Salva file SprintX", "Calibrazione", e "la determinazione RLL" prima di cliccare "Analizza".
Risultati
La Figura 1 mostra l'immagine matrice anticorpo dal CFM ei livelli mAb macchiati con i due metodi di immobilizzazione, e la Figura 2 mostra i corrispondenti sensorgrams vincolanti generati nel MX96 dal multi-ciclo cinetico e il ciclo unico misurazioni cinetiche di queste matrici mAb . Il legame e le curve cineticamente muro di molteplici superfici rivestite di anticorpi generati nei quattro piattaforme biosensore tempo reale sono mostrati nelle figure 3 - 6. La figura 7 mostra il confronto dei tassi cinetici finali e vincolanti equilibrio costanti ricavate dall'analisi globale di queste curve di legame. L'individuo associazione (k a), dissociazione (k d), e l'equilibrio (K D) costanti di legame sono riportati in Tabella 1. Per dimostrare la variabilità dei set di dati generati all'interno dello stesso strumento, La Figura 8 mostra appezzamenti di k a, k d, e K D a diverse densità di superficie mAb, e la figura 9 confronta ulteriormente le attività di legame calcolati delle superfici mAb attraverso le piattaforme biosensore.
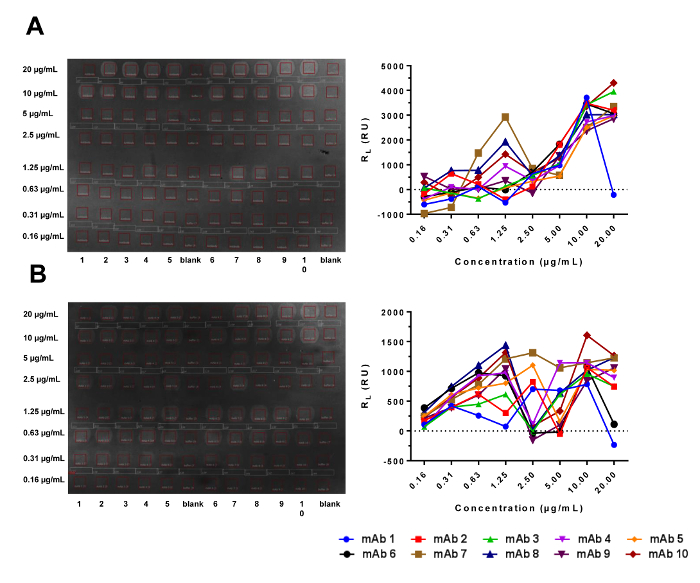
Figura 1. Multiplex Ligand Arrays di Amine-accoppiati (A) e Fc-catturato (B) Antibody superfici da CFM Stampa in IBIS MX96. Immagini delle matrici stampate sono mostrati nei pannelli di sinistra, dove le aree grigie racchiuse da quadrati rossi indicano la presenza di anticorpi. I interspots scure situate tra i punti di anticorpi attivi sono utilizzati per la sottrazione riferimento. Ogni colonna contiene un anticorpo stampato alle concentrazioni titolazione identificati sotto ea sinistra dell'immagine. Le quantità di anticorpi stampati quantificati calcolando la difference a turni di massa tra le posizioni attive e di riferimento sono presenti nei pannelli di destra. Si prega di cliccare qui per vedere una versione più grande di questa figura.
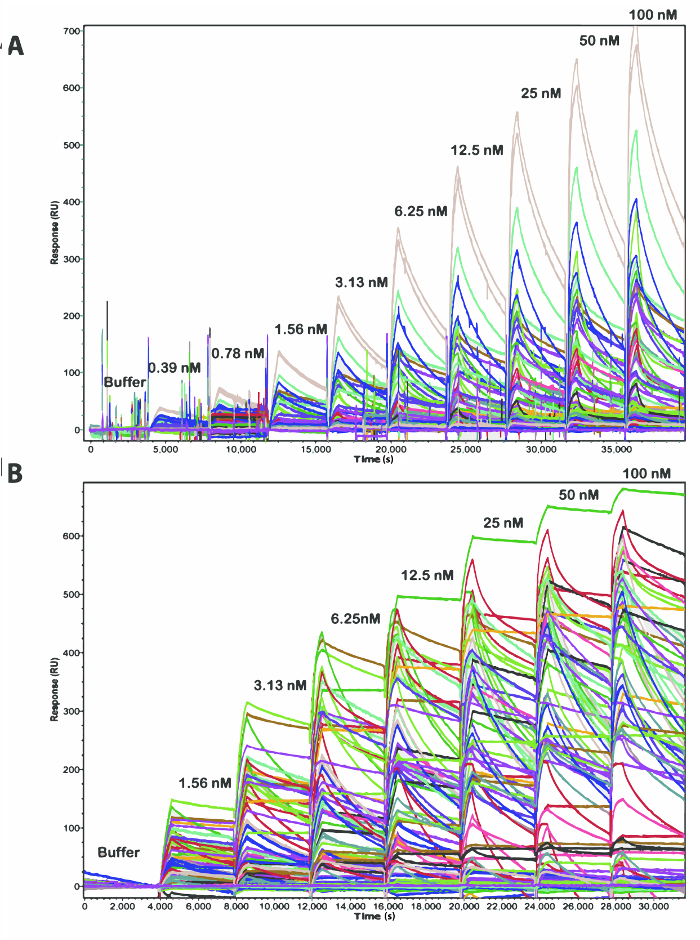
Figura 2. Confronto di tempo reale Binding Sensorgrams da Multi-ciclo (A) e ciclo singolo (B) Esperimenti cinetica nel IBIS MX96. Le iniezioni sequenziali di PCSK9 umana a concentrazioni crescenti attraverso le matrici 10 x 8 anticorpali sono indicati sopra ogni segmento sensorgram corrispondente. Si prega di cliccare qui per vedere una versione più grande di questa figura.
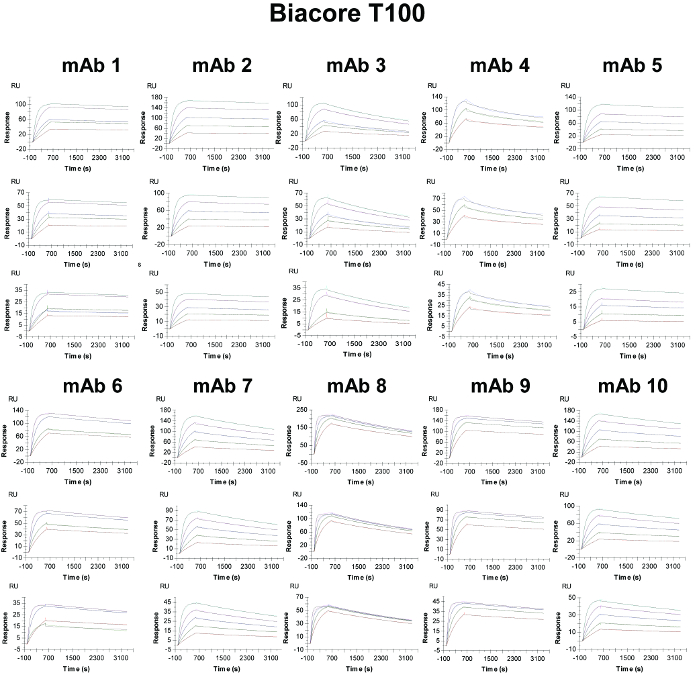
Figura 3. Binding Sensorgrams degli anticorpi catturati Interazione con PCSK9 umana e 1: 1 Kinetic Fit modello overlay nella Biacore T100. Le interazioni sono valutati su alta (pannelli superiori), medio (pannelli centrali) e bassa (pannelli inferiori) superfici densità. Le linee nere lisce sovrapposte rappresentano la misura cinetica dei segnali di risposta di legame a differenti concentrazioni PCSK9 umane (linee colorate) ad un 1: 1 modello di interazione. Si prega di cliccare qui per vedere una versione più grande di questa figura.
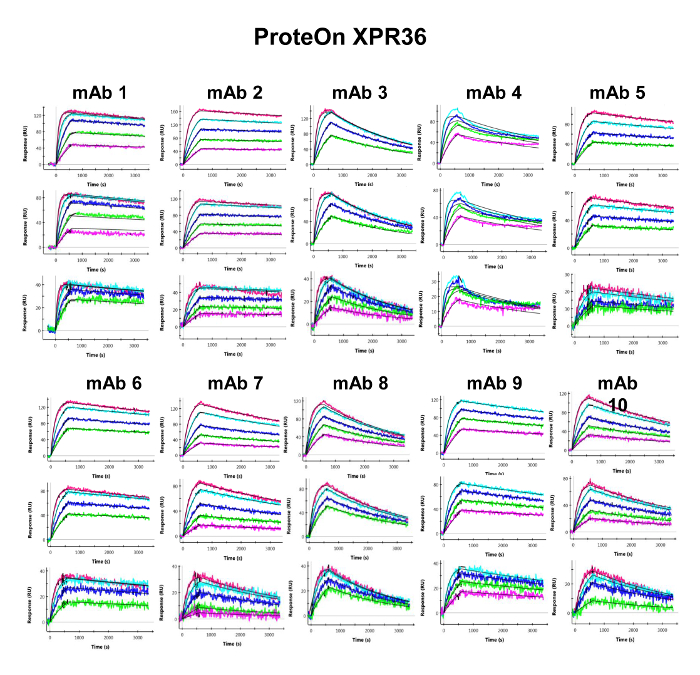
Figura 4. Binding Sensorgrams degli anticorpi catturati Interazione con PCSK9 umana e 1: 1 Kinetic Fit modello overlay nella ProteOn XPR36. Le interazioni sono valutate nel corso di alta (pannelli superiori), medium- (pannelli centrali) e bassa (pannelli inferiori) superfici densità. Le linee nere lisce sovrapposte rappresentano la misura cinetica dei segnali di risposta di legame a differenti concentrazioni PCSK9 umane (linee colorate) ad un 1: 1 modello di interazione. Si prega di cliccare qui per vedere una versione più grande di questa figura.

Figura 5. Binding Sensorgrams degli anticorpi catturati Interazione con PCSK9 umana e 1: 1 Kinetic Fit modello overlay nella Octet RED384. Le interazioni sono valutati su alta (pannelli superiori), medio (pannelli centrali) e bassa (pannelli inferiori) superfici densità. Le linee rosse sovrapposte rappresentano la misura cinetica dei segnali di risposta vincolanti a diverse concentrazioni PCSK9 umane (linee colorate) ad un 1: 1 modello di interazione. Si prega di cliccare qui per vedere una versione più grande di questa figura.
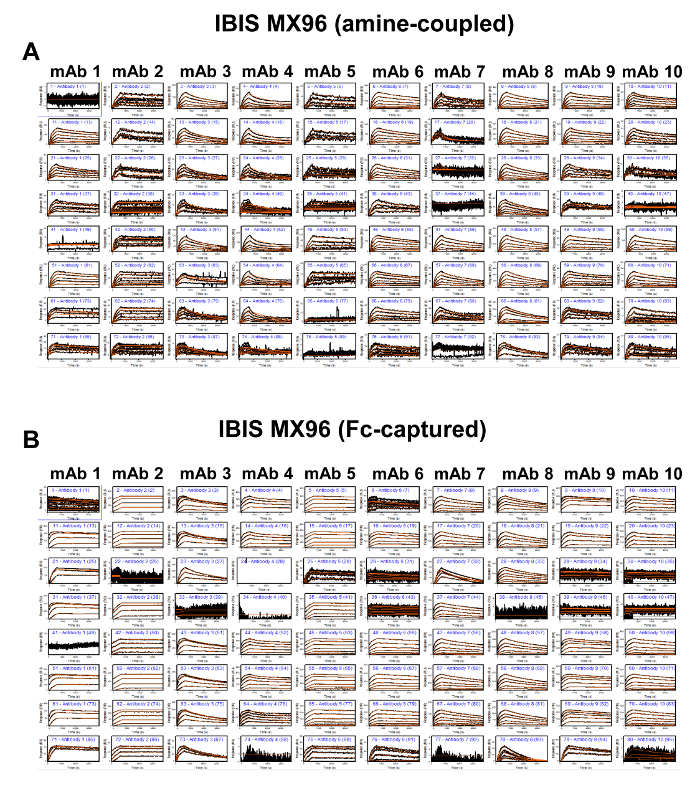
Figura 6. Binding Sensorgrams dell'ammina-accoppiati (A) e Fc-catturate (B) Anticorpi Interazione con PCSK9 umana e 1: 1 cinetiche modello Fit sovrapposizioni in IBIS MX96. I profili di legame sono organizzati come 10 x 8 pannelli che seguono la mappa piastra matrice di figura 1. Le linee nere rappresentano i segnali di risposta vincolanti registrati a diverse concentrazioni PCSK9 umani, e le linee rosse sovrapposte rappresentano le curve a muro. Si prega di cliccare qui per vedere una versione più grande di questa figura.
gura 7" src = "/ files / ftp_upload / 55659 / 55659fig7.jpg" />
Figura 7. Confronto dell'Associazione k a (A), dissociazione K D (B), ed Equilibrio K D (C) costanti di legame generati dalle piattaforme Four biosensore. I parametri cinetici derivano da analisi globale delle curve di legame delle Figure 3 - 6. Gli strumenti sono rappresentati: Biacore T100 (blu), ProteOn XPR36 (verde), Octet RED384 (rosso), IBIS MX96, ammina accoppiati (viola), e IBIS MX96, Fc-catturate (arancione). Si prega di cliccare qui per vedere una versione più grande di questa figura.
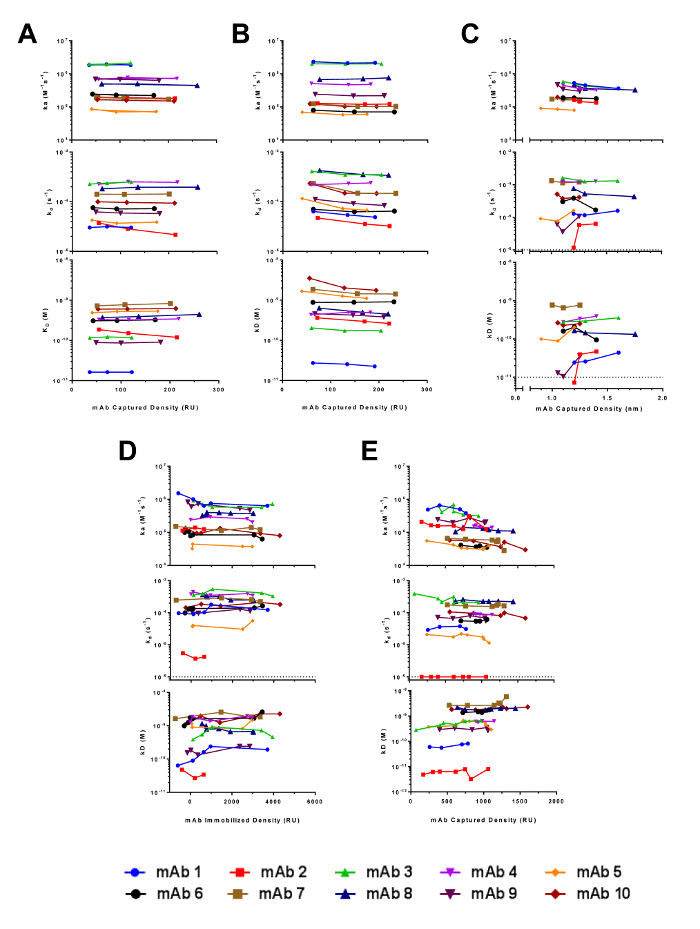
Figura 8. Confronto della Consistenza Kinetic Tasso costanti su più densità superficiale anticorpo nel Biac minerale T100 (A), ProteOn XPR36 (B), Octet RED384 (C), IBIS MX96, ammina-accoppiati (D), e IBIS MX96, Fc-catturato (E). I parametri cinetici k A (in alto sotto-pannelli), k d (middle sottoquadri), e K D (peggiori gruppi secondari) sono derivati da analisi di gruppo a singola densità superficiale anticorpo. Si prega di cliccare qui per vedere una versione più grande di questa figura.
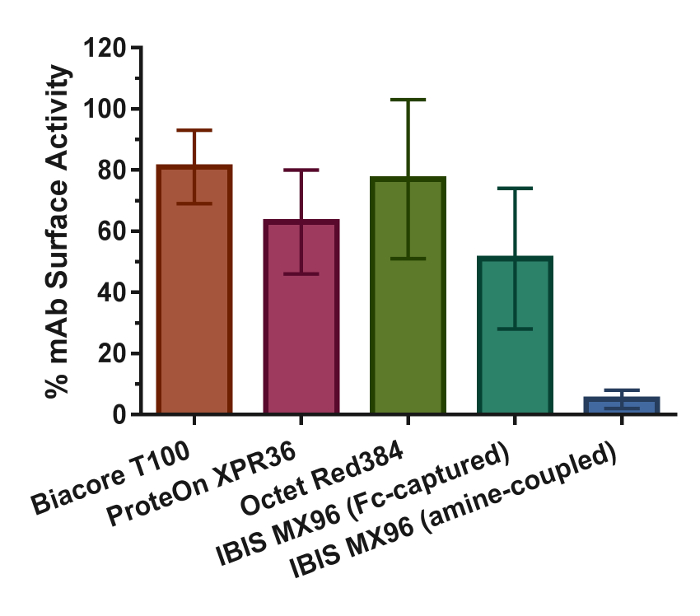
Figura 9. Attività legame dell'anticorpo superfici e loro deviazioni standard (barre di errore) nelle piattaforme Quattro biosensore. I valori sono calcolati usando le equazioni descritte negli articoli di ricerca 17.large.jpg" target = '_ blank'> Clicca qui per vedere una versione più grande di questa figura.
| k a | k d | K D | |
| (M -1 s -1) | (s -1) | (nm) | |
| mAb 1 | 11,7 (7,8) x 10 5 | 4,89 (4,18) x 10 -5 | 0.052 (0.05) |
| mAb 2 | 1,53 (0,28) x 10 5 | 1,30 (1,54) x 10 -5 | 0,092 (0,12) |
| mAb 3 | 11.4 (8.3) x 10 5 | 27.6 (11.0) x 10 -5 | 0.333 (0.20) |
| mAb 4 | 3.61 (1,6) x 10 5 | 20.1 (12.3) x 10 -5 | 0.659 (0.54) |
| mAb 5 | 0,59 (0,21) x 10 5 | 3,46 (2,50) x 10 -5 | 0,663 (0,46) |
| mAb 6 | 1,19 (0,78) x 10 5 | 7,21 (3,83) x 10 -5 | 0,894 (0,64) |
| mAb 7 | 1,29 (0,53) x 10 5 | 16,4 (5,63) x 10 -5 | 1.57 (1.02) |
| mAb 8 | 4.02 (2.23) x 10 5 | 22.8 (10.7) x 10 -5 | 0,768 (0.68) |
| mAb 9 | 4.20 (2.13) x 10 5 | 6,67 (4,3) x 10 -5 | 0,197 (0.16) |
| mAb 10 | 1,20 (0,49) x 10 5 | 12.5 (7,64) x 10 -5 | 1.27 (0.80) |
Tabella 1: Kinetic Prezzi ed equilibrio Binding costanti di 10 anticorpi monoclonali ottenuti inserendo globale delle curve vincolante da quattro biosensore Instruments al 1: 1 Interaction Model.
Discussione
Il nostro studio di confronto testa a testa mostra che ogni piattaforma biosensore ha i suoi punti di forza e di debolezza. Anche se i profili di legame degli anticorpi sono simili per confronto visivo (figure 3 - 6), e l'ordine di classificazione delle costanti di velocità cinetiche acquisiti sono altamente coerente tra gli strumenti (Figura 7), i nostri risultati dimostrano che gli strumenti SPR-based con continui fluidica flusso) sono meglio a risolvere le interazioni ad alta affinità con tassi di dissociazione lenti. Deriva verso l'alto durante la fase di dissociazione si osserva in set di dati (ad esempio, mAb 2, mAb 5, e mAb 9; figura 5) generato dal fluidica libere strumento BLI. Questo risultato può essere attribuito in gran parte per evaporazione campione nel tempo nella micropiastra, che è una limitazione principale del sistema. Con questa limitazione intrinseca, il tempo sperimentale è anche limitato a meno di 12 h; gli esperimenti sono stati quindi programmati con shvolte Orter (500 s associazione e 30 min dissociazione) rispetto a quelle delle altre piattaforme (10 min associazione e 45 min dissociazione). Tuttavia, accorciando il tempo sperimentale non sembra attenuare l'impatto di evaporazione sulla qualità dei dati / coerenza, come le costanti di velocità generate dallo strumento BLI basata show less linearità a causa delle fluttuazioni in alcune delle off-rate (Figura 8C ). Oltre a evaporazione del campione, le differenze di reagenti di cattura utilizzati possono hanno anche contribuito alle differenze nei risultati ottenuti. Mentre la proteina A / G è stato utilizzato in tutte e tre le piattaforme SPR fluidici basata, sensori AHC sono stati utilizzati nella piattaforma BLI non fluidica. Poiché proteina A / G può avere una debole affinità per i mAbs che fa l'AHC, una superficie biosensore a base di anticorpi, l'off-rate del complesso mAb-antigene da / superfici proteina A G può artificialmente apparire più veloci di quelli ottenuto dalla superficie AHC. Questa possibilità è supportata by i dati sperimentali che dimostrano che i valori off-rate generati dalla piattaforma BLI erano notevolmente inferiori a quelli ottenuti dagli altri strumenti (Figura 7, linea rossa). Tuttavia, la piattaforma di BLI ha diversi vantaggi rispetto alle altre piattaforme. Ad esempio, è altamente flessibile per quanto riguarda la scelta del sensore e configurazione test dovuti ai diversi sensori pre-rivestite per uso immediato. Nei nostri esperimenti, l'uso dei sensori AHC eliminato la necessità di passaggi ligando immobilizzazione, riducendo i tempi di preparazione. Inoltre, la piattaforma BLI richiede molto meno manutenzione rispetto alle altre piattaforme fluidica SPR, che dispongono di configurazioni di tubi e l'interruttore valore complicati. Questa caratteristica è un vantaggio per gli esperimenti che coinvolgono campioni grezzi che possono causare problemi di intasamento e di contaminazione.
Poiché la domanda per l'identificazione efficiente, rapida e accurata dei candidati aumenta terapeutici, la necessità di biil throughput osensor è anche in aumento. Tra i quattro piattaforme biosensore, il throughput dal biosensore capace di 96-ligando stampa matrice è la più alta, seguita dal biosensore accoppiato ad un crisscrossing formato 36-ligando e il biosensore BLI-based con lettura simultanea 16 canali, che alla fine aumenta il numero di interazioni misurati in un singolo ciclo di legatura a 96, 36 e 16, rispettivamente. Queste capacità di throughput sono significativamente superiori a quella della piattaforma tradizionale SPR, che è limitata da avere solo quattro celle di flusso collegati da un unico flusso seriale. Dal momento che i nostri esperimenti coinvolti un insieme relativamente piccolo campione di 10 anticorpi monoclonali valutati a più densità di superficie con tempi di dissociazione lunghi, i throughput strumentali hanno giocato un ruolo moderato nel determinare l'efficienza degli esperimenti. Non ci sono state differenze significative nei tempi sperimentali delle tre piattaforme high-throughput, e in tutti i casi gli esperimenti sono stati completati in un giorno. D'altra parte, i tradizionali esperimenti SPR flusso seriale necessari 3 giorni per completare, nonostante l'automazione walk-away di acquisizione dei dati dopo l'installazione. In altri studi che coinvolgono un gran numero di campioni (cioè nell'ordine delle centinaia o migliaia), per scopi di classifica off-rate / di screening cinetica o epitopo di categorizzazione, il throughput diventa un fattore critico.
Sebbene il throughput in IBIS MX96 è ordini di grandezza superiore a quella degli altri biosensori ed è quindi una scelta ottimale per questi scopi, ha alcuni inconvenienti. In particolare, la stampa di matrice dal CFM mostra grandi incongruenze superficiali (Figura 1) e ridotta riproducibilità dati (Figura 8D e 8E). Per misurazioni cinetiche accurate, la quantità di legante sulla superficie biosensore è un parametro critico che deve essere controllato per garantire che le risposte vincolanti non sono disturbati da fattori secondari cometrasferimento di massa o impedimento sterico. Per il Biacore T100 e ProteOn XPR36, i livelli ottimali L R sono stati determinati sulla base del calcolo standard dei valori massimi serie R come descritto nell'articolo di ricerca 17. D'altra parte, per le piattaforme Octet RED384 e IBIS MX96, i livelli di cattura mAb stati raggiunti empiricamente usando una serie di anticorpi 2 volte diluiti serialmente in tempi costanti. La mancanza di conoscenza o il controllo della fase di cattura comportato una superficie ad alta densità e segnali ad alta vincolanti di risposta (Figura 2) che possono aver compromesso l'accuratezza delle costanti cinetiche. Inoltre, la separazione della stampante dal rivelatore SPR presenta anche una sfida per lo svolgimento di misurazioni multiple ciclo di legame che coinvolgano rigenerazioni. L'unico modo per eseguire la configurazione cinetica cicli multipli era attraverso accoppiamento di ammina diretta dei mAbs, al contrario di catturare attraverso il mAb Fc dalla immobilizzato proteina A / Gsuperficie negli altri tre piattaforme biosensori. Come risultato, un esperimento di rigenerazione esplorazione è stato necessario per determinare le condizioni di rigenerazione ottimali. Il risultato di questa configurazione è stato collegato a un ~ 90% inferiore attività superficiale osservata rispetto al metodo di cattura Fc (Figura 9), in aggiunta a un tempo più lungo sperimentale. Per eseguire il metodo di cattura Fc, è stato adottato un approccio cinetico-singolo ciclo alternativo. Questo approccio comporta l'iniezione sequenziale di analiti a concentrazioni crescenti senza rigenerazione fra ogni iniezione (Figura 2). Questo approccio altamente conveniente, ma meno comunemente applicato non solo ridotto il tempo sperimentale e ridotto consumo di reagenti, ma anche prodotto costanti di velocità cinetiche molto simili a quelle degli altri biosensori (Figura 7). Pertanto, l'applicazione di questo approccio cinetica ciclo unico supera la limitazione intrinseca configurazione dello strumento e presenta unapossibilità di ottenere ad alta risoluzione costanti di velocità cinetiche in maniera high-throughput.
Nonostante il throughput essere un grosso limite nel Biacore T100, i nostri risultati dimostrano collettivamente che ha generato i dati più coerenti con la massima qualità. Questo è stato seguito dal ProteOn XPR36, che ha ~ 10 volte maggiore produttività. La loro capacità di generare dati di alta qualità diventa un vantaggio quando caratterizzazione alta affinità interazioni anticorpo-antigene che può essere tecnicamente difficile al raggiungimento dei limiti di rilevabilità degli strumenti. Mentre le limitazioni sistematiche nella strumentazione dell'ottetto RED384 ostacolano la misurazione accurata di costanti di velocità di dissociazione (cioè, diminuzione oltre la sensibilità per risolvere decadimento del segnale sufficiente per lenta off-tassi), sia il Biacore T100 e ProteOn XPR36 può fornire sensibile ed affidabile rilevamento per la differenziazione.
Divulgazioni
spese di pubblicazione di questo video-articolo sono stati pagati da Boehringer Ingelheim Pharmaceuticals.
Riconoscimenti
Gli autori ringraziano Noah Ditto e Adam Miles per l'assistenza tecnica sul IBIS MX96.
Materiali
| Name | Company | Catalog Number | Comments |
| Biacore T100 System | GE Healthcare | 28975001 | The T100 system has been upgraded to T200 |
| CM5 Sensor Chip | GE Healthcare | BR100012 | |
| BIAevaluation | GE Healthcare | Version 4.1 | |
| Biacore T100 Control Software | GE Healthcare | The T100 control software has been upgraded to T200 | |
| Amine Coupling Kit | GE Healthcare | BR100050 | It contains: 750 mg 1-ethyl-3-(3-dimethylaminopropyl)carbodiimide hydrochloride (EDC), 115 mg N-hydroxysuccinimide (NHS), 10.5 mL 1.0 M ethanolamine-HCl pH 8.5 |
| HBS-EP+ Buffer 10× | GE Healthcare | BR100669 | Concentrated stock solution |
| Plastic Vials, o.d. 7 mm | GE Healthcare | BR100212 | |
| Rubber Caps, type 3 | GE Healthcare | BR100502 | |
| Plastic Vials and Caps, o.d. 11 mm | GE Healthcare | BR100214 | |
| ProteOn XPR36 Protein Interaction Array System | Bio-Rad | 1760100 | |
| ProteOn Manager Software | Bio-Rad | 1760200 | Version 3.1.0.6 |
| GLM Sensor Chip | Bio-Rad | 1765012 | |
| Amine Coupling Kit | Bio-Rad | 1762410 | It includes EDAC (EDC), sulfo-NHS, ethanolamine HCl |
| Regeneration and Conditioning Kit and Buffers | Bio-Rad | 1762210 | It includes 1 each glycine buffer (pH 1.5, 2.0, 2.5, 3.0), and NaOH, SDS, HCl, phosphoric acid, NaCl; 50 mL each solution |
| 2 L PBS/Tween/EDTA buffer | Bio-Rad | 1762730 | It includes hosphate buffered saline (PBS), pH 7.4, 0.005% Tween 20, 3 mM EDTA |
| Octet RED384 System | FortéBio | ||
| Data Analysis Software | FortéBio | Version 9.0.0.4 | |
| Dip and Read Anti-hIgG Fc Capture (AHC) Biosensors | FortéBio | 18-5060 | One tray of 96 biosensors |
| 384-Well Tilted-Bottom Plate | FortéBio | 18-5080 | |
| Biosensor Dispenser | FortéBio | 18-5016 | |
| Kinetics Buffer 10x | FortéBio | 18-1092 | 10x concentration. Contains ProClin 300. |
| IBIS MX96 SPRi System | Wasatch Microfluidics | ||
| Microfluidics Continuous Flow Microspotter (CFM) Printer | Wasatch Microfluidics | Version 2.0 | |
| SensEye COOH-G chip | Wasatch Microfluidics | 1-09-04-004 | |
| Data Analysis Software | Wasatch Microfluidics | Version 6.19.3.17 | |
| SPRint Data Analysis Software | Wasatch Microfluidics | Version 6.15.2.1 | |
| Scrubber 2 | BioLogic Software | Version 2 | |
| Pierce Recombinant Protein A/G | ThermoFisher Scientific | 21186 |
Riferimenti
- Cooper, M. A. Optical biosensors in drug discovery. Nat Rev Drug Discov. 1 (7), 515-528 (2002).
- Van Regenmortel, M. H., et al. Measurement of antigen-antibody interactions with biosensors. J Mol Rec. 11 (1-6), 163-167 (1998).
- Canziani, G. A., Klakamp, S., Myszka, D. G. Kinetic screening of antibodies from crude hybridoma samples using Biacore. Anal Biochem. 325 (2), 301-307 (2004).
- Katsamba, P. S., et al. Kinetic analysis of a high-affinity antibody/antigen interaction performed by multiple Biacore users. Anal Biochem. 352 (2), 208-221 (2006).
- Abdiche, Y. N., Malashock, D. S., Pinkerton, A., Pons, J. Exploring blocking assays using Octet, ProteOn, and Biacore biosensors. Anal Biochem. 386 (2), 172-180 (2009).
- Abdiche, Y. N., et al. High-Throughput Epitope Binning Assays on Label-Free Array-Based Biosensors Can Yield Exquisite Epitope Discrimination That Facilitates the Selection of Monoclonal Antibodies with Functional Activity. PLoS ONE. 9 (3), e92451 (2014).
- Ecker, D. M., Jones, S. D., Levine, H. L. The therapeutic monoclonal antibody market. mAbs. 7 (1), 9-14 (2014).
- Rich, R. L., Myszka, D. G. Higher-throughput, label-free, real-time molecular interaction analysis. Anal Biochem. 361 (1), 1-6 (2007).
- Keighley, W. The need for high throughput kinetics early in the drug discovery process. Drug Disc World. 12 (3), 39-45 (2011).
- Lad, L., et al. High-throughput kinetic screening of hybridomas to identify high-affinity antibodies using bio-layer interferometry. J Biomol Screen. 20 (4), 498-507 (2015).
- Bravman, T., Bronner, V., Nahshol, O., Schreiber, G. The ProteOn XPR36TM Array System-High Throughput Kinetic Binding Analysis of Biomolecular Interactions. Cell Mol Bioeng. 1 (4), 216-228 (2008).
- Eddings, M. A., et al. Improved continuous-flow print head for micro-array deposition. Anal Biochem. 382 (1), 55-59 (2008).
- Abdiche, Y., Malashock, D., Pinkerton, A., Pons, J. Determining kinetics and affinities of protein interactions using a parallel real-time label-free biosensor the Octet. Anal Biochem. 377 (2), 209-217 (2008).
- Abdiche, Y. N., Lindquist, K. C., Pinkerton, A., Pons, J., Rajpal, A. Expanding the ProteOn XPR36 biosensor into a 36-ligand array expedites protein interaction analysis. Anal Biochem. 411 (1), 139-151 (2011).
- Rich, R. L., et al. A global benchmark study using affinity-based biosensors. Anal Biochem. 386 (2), 194-216 (2009).
- Yang, D., Singh, A., Wu, H., Kroe-Barrett, R. Comparison of biosensor platforms in the evaluation of high affinity antibody-antigen binding kinetics. Anal Biochem. 508, 78-96 (2016).
- Yang, D., Singh, A., Wu, H., Kroe-Barrett, R. Dataset of the binding kinetic rate constants of anti-PCSK9 antibodies obtained using the Biacore T100 ProteOn XPR36, Octet RED384, and IBIS MX96 biosensor platforms. Data in Brief. 8, 1173-1183 (2016).
- Bouvier, E. S. P., Koza, S. M. Advances in size-exclusion separations of proteins and polymers by UHPLC. Trends Anal Chem. 63, 85-94 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon