È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Modulazione reversibile mediata dalla luce del percorso proteico di kinasi attivato da Mitogen durante la differenziazione cellulare e
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Questo protocollo descrive una strategia optogenetica per modulare l'attivazione della proteina chinasi attivata dal mitogeno (MAPK) durante la differenziazione cellulare e lo sviluppo embrionale di Xenopus . Questo metodo consente l'attivazione reversibile del percorso di segnalazione MAPK nella coltura di cellule di mammiferi e negli organismi vivi multicellulari, come gli embrioni di Xenopus , con elevata risoluzione spaziale e temporale.
Abstract
L'attività cinasi è fondamentale per una pletora di funzioni cellulari, tra cui la proliferazione cellulare, la differenziazione, la migrazione e l'apoptosi. Durante il primo sviluppo embrionale, l'attività della chinasi è altamente dinamica e diffusa nell'embrione. Gli approcci farmacologici e genetici sono comunemente usati per sondare le attività di chinasi. Purtroppo, è difficile raggiungere una risoluzione spaziale e temporale superiore utilizzando queste strategie. Inoltre, non è possibile controllare l'attività cinasi in modo reversibile in cellule vive e organismi multicellulari. Tale limitazione rimane un collo di bottiglia per ottenere una comprensione quantitativa dell'attività cinasi durante lo sviluppo e la differenziazione. Questo lavoro presenta una strategia optogenetica che sfrutta un sistema bicistronic contenente proteine fotoattivatabili Arabidopsis thaliana cryptochrome 2 (CRY2) e il dominio N-terminale di citochrome-interazione di base elica-loop-elica (CIBN). ReversiL'attivazione del percorso di segnalazione della proteina chinasi attivata da mitogeni (MAPK) è ottenuta attraverso la traslocazione proteica mediata dalla luce in cellule vive. Questo approccio può essere applicato alle colture di cellule di mammiferi e agli embrioni di vertebrati vivi. Questo sistema bicistronic può essere generalizzato per controllare l'attività di altre chinasi con meccanismi di attivazione similari e può essere applicata ad altri sistemi di modello.
Introduzione
I fattori di crescita sono coinvolti in un ampio spettro di funzioni cellulari, tra cui la proliferazione, la differenziazione, la migrazione e l'apoptosi e svolgono ruoli chiave in molti eventi biologici, inclusi lo sviluppo embrionale, l'invecchiamento e la regolazione dello stato mentale 1 , 2 , 3 , 4 , 5 . Molti fattori di crescita segnano attraverso complesse cascate intracellulari di segnalazione. Questi eventi di segnalazione sono spesso gestiti da fosforilazione proteica reversibile in modo preciso regolato 6 , 7 . Pertanto, una comprensione dei risultati di segnalazione delle proteine chinasi, che sono responsabili della fosforilazione proteica, è fondamentalmente importante.
Diversi fattori di crescita agiscono attraverso una rete di segnalazione intracellulare piuttosto comune, anche se stimolano distRisposte cellulari inct 8 , 9 . I mediatori intracellulari comuni delle chinasi di tirosinici del recettore includono Ras, Raf, chinasi extracellulare-regolata (ERK), proteina chinasi attivata da mitogeni (MAPK) / ERK chinasi (MEK), fosfoinositide 3-chinasi (PI3K), Akt e gamma fosfolipasi C (PLCγ) 10 , 11 . L'accumulo di prove suggerisce che la diversità di segnalazione e la specificità dipendono dalla regolazione spaziale e temporale dell'attività di segnalazione 12 . Ad esempio, nelle cellule di phaocromocytoma di ratto (PC12), la stimolazione del fattore di crescita epidermico (EGF), che provoca la proliferazione cellulare, attiva temporaneamente il percorso ERK 9 . D'altra parte, la stimolazione con il fattore di crescita del nervo (NGF), che porta alla differenziazione cellulare, attiva il percorso ERK in modo sostenibile 9 , 13 . In colture rNei neuroni ippocampali, la segnalazione transitoria da un fattore neurotrofico derivato dal cervello (BDNF) favorisce la crescita primaria del neurite, mentre la segnalazione sostenuta porta ad una maggiore ramificazione del neurite 14 . Durante il primo sviluppo embrionale, l'attività ERK fosforilata è temporaneamente dinamica e diffusa nell'embrione 6 . Una recente schermata genetica durante l'embriogenesi iniziale di Xenopus ha mostrato che le cascate di segnalazione ERK e Akt, due percorsi di fattore di crescita primario a valle, mostrano i profili di attivazione specifici di fase 7 . Così, una comprensione dei risultati di segnalazione di chinasi richiede strumenti che possono sondare le caratteristiche spaziali e temporali dell'attività cinasi con una risoluzione sufficiente.
Approcci sperimentali convenzionali per sondare la natura dinamica della trasduzione del segnale durante lo sviluppo non hanno la desiderabile risoluzione spaziale e temporale. Ad esempio, gli approcci farmacologici utilizzano la piccola chemMolecole ical o biologiche per stimolare o sopprimere la trasduzione del segnale nelle cellule e nei tessuti. La natura diffusa di queste piccole molecole rende difficile limitare la loro azione ad una regione specifica di interesse. Gli approcci genetici ( ad esempio, transgenesi, il sistema Cre-Lox o mutagenesi) spesso portano all'attivazione o alla repressione irreversibili dell'espressione genica o dell'attività proteica 16 , 17 , 18 . Il sistema Tet-On / Tet-Off 19 offre un controllo temporale migliorato della trascrizione del gene, ma manca di un controllo spaziale rigoroso perché si basa sulla diffusione della tetraciclina. I recenti sviluppi nella dimerizzazione delle proteine chimicamente indotti dalla chimica 20 o nella foto-scoperta 21 , 22 , 23 , 24 hanno notevolmente miglioramentiEd il controllo temporale delle reti di segnalazione. Il controllo spaziale, tuttavia, rimane impegnativo a causa della diffusione dei prodotti chimici in gabbia.
Recenti approcci optogenetici emergenti, che sfruttano il potere di luce per controllare le interazioni proteine-proteine, consentono la modulazione di percorsi di segnalazione con elevata precisione spatiotemporale e reversibilità. Poco dopo il suo successo iniziale nel controllo del cervello neuronale 25 , 26 , 27 , l'ottogenetica è stata estesa per controllare altri processi cellulari, come la trascrizione di geni, la traduzione, la migrazione cellulare, la differenziazione e l'apoptosi 28 , 29 , 30 , 31 , 32 , 33 , 34 . Una strategia che utilizza il pProteina hotoattivatabile La proteina cripto- cromica 2 (CRY2) di Arabidopsis thaliana e il dominio N-terminale dell'elica a base di elicoidale (CIBN) che interagiscono con citochrome è stato recentemente sviluppato per controllare l'attività di Raf1-chinasi nelle cellule di mammiferi e negli embrioni Xenopus 35 . CRY2 si lega a CIBN su stimolazione azzurra-luce e il complesso proteico CRY2 / CIBN dissociata spontaneamente nel buio 34 . La luce blu eccita il cofattore CRY2, il dinucleotide di flavin adenina (FAD), che porta ad un cambiamento conformazionale in CRY2 e successivamente legato a CIBN. I mutanti costituenti attivamente attivi (W374A) e flavin-deficienti (D387A) di CRY2 possono essere prodotti attraverso mutazioni nella tasca di legame FAD: il mutante CRY2 W374A si lega a CIBN indipendente dalla luce, mentre il mutante CRY2 D387A non si lega a CIBN sotto l'azzurro Stimolazione luminosa 36 , 37 . Il sistema ottogenetico descritto iN questo protocollo utilizza il tipo di wild-type CRY2 e CIBN per indurre l'attivazione Raf1 mediata dalla traslocazione proteica in cellule vive. È noto che il reclutamento della membrana di Raf1 aumenta la sua attività 38 . In questo sistema, un modulo CIBN tandem è ancorato alla membrana plasmatica e CRY2-mCherry è fuso al terminale N di Raf1 35 . In assenza di luce blu, CRY2-mCherry-Raf1 rimane nel citoplasma, e Raf1 è inattivo. La stimolazione della luce blu induce il legame CRY2-CIBN e recluta Raf1 alla membrana plasmatica, in cui viene attivata Raf1. L'attivazione Raf stimola una cascata di segnalazione Raf / MEK / ERK. Entrambe le proteine di fusione CRY2 e CIBN sono codificate in un sistema genetico bicistronico. Questa strategia può essere generalizzata per controllare altre chinasi, come Akt, il cui stato di attivazione può essere attivato anche dalla traslocazione proteica nelle cellule 39 . Questo lavoro presenta protocolli dettagliati per l'implementazione di questa strategia optogenetica in cultu cellule di mammiferiRes e organismi multicellulari.
Access restricted. Please log in or start a trial to view this content.
Protocollo
La ricerca sugli animali è stata condotta in conformità con le linee guida stabilite dall'Istituto per l'Assicurazione e l'Utilizzazione degli Animali Istituzionali (IACUC) e dall'Università dell'Illinois Dipartimento di Risorse Animali (DAR).
1. Induzione ottogenetica della localizzazione della proteina nella cultura cellulare delle cellule BHK21
NOTA: i passaggi 1.1-1.3 forniscono un metodo per assemblare una camera di coltura cellulare per l'imaging con obiettivi di ingrandimento elevato ( ad es., 63X o 100X), che presentano tipicamente brevi distanze di lavoro. Questi obiettivi richiedono una copertura in vetro sottile ( ad esempio, spessore 1,5 mm, 170 μm) come substrato di imaging. In alternativa, può essere utilizzato un piatto / diapositiva di coltura a cellule in vetro. In questo caso, i passi 1.1-1.3 possono essere ignorati.
- Pulizia dei vetri in vetro
- Posizionare 30 coperchio di vetro in un supporto per copertina.
- In un bicchiere di plastica da 600 ml, aggiungere 20 g di detersivo e 400 ml di acqua calda del rubinetto. PlacIl bicchiere su un agitatore magnetico e mescolare fino a quando tutto il detergente è sciolto. Rimuovere la schiuma usando carta tissue.
- Utilizzare il fondo di un piatto di Petri di 100 mm x 15 mm come distanziatore dalla barra di stirata e come superficie per il supporto del coperchio. Per mantenere un adeguato flusso durante il processo di pulizia, fare 3-4 fori di drenaggio nel piatto Petri.
- Per fare i fori di drenaggio, scaldare un ferro saldato e bruciare la plastica in un cappuccio ventilato.
ATTENZIONE: non toccare il ferro saldato riscaldato con le mani nude. - Posizionare il portacoperta sul piatto Petri nel bicchiere. Mescolare per 2 ore fino a notte a temperatura ambiente.
- Lavare tre volte con 500 ml di acqua DI in un becher da 1.000 ml. La soluzione detergente può essere riutilizzata.
- Raccogli le copertine individuali usando le pinze. Immergere le copertine in etanolo a prova di 190% (95%) per 1 minuto.
- Asciugare le copertine all'interno di un cappuccio sterile. Conservare le copertine in un contenitore di plastica sterile fino all'uso.
- Realizzazione di coperchi rivestiti in poli-L-lisina (PLL)
- Creare un tampone di borato di 0,1 M sciogliendo 1,24 g di acido borico e 1,9 g di tetraborato di sodio in 400 ml di acqua. Regolare il pH a 8,5 con 10 M NaOH.
- Sciogliere PLL nel tampone di borato fino ad una concentrazione finale di 1 mg / mL. Aliquot la soluzione PLL in tubi sterilizzatori sterili da 50 ml.
- Aggiungere 50 ml di soluzione di rivestimento PLL a un piatto da 10 cm di coltura tissutale. Posizionare il piatto in un incubatore a 37 ° C per riscaldare la soluzione PLL.
- Aprire il contenitore del coperchio (passo 1.1.8) in un cappuccio sterile.
- Immersione dei coperchi puliti uno alla volta nella soluzione PLL pre-riscaldata nel piatto di coltura tissutale. Evitare la formazione di bolle per immergere tutte le superfici.
- Posizionare il piatto con la copertura in un incubatore di CO 2 a 37 ° C durante la notte. Togliere ogni coperchio e collocarlo in un portacandele sterile. Sciacquare tre volte con acqua ultrapure autoclavata (18,2 MΩ & #183; cm resistivity) in un becher da 1000 ml in un cappuccio sterile.
- Asciugare le copertine nel supporto del coperchio nel cappuccio sterile. Conservare le copertine in un contenitore di plastica sterile fino all'uso.
- Assemblaggio di camere di coltura cellulare polidimetilsilossano (PDMS)
- In un contenitore di plastica, pesare 40 g di base di PDMS e aggiungere 4 g di agente di tintura per ottenere un rapporto 10: 1 w / w di PDMS e agente di tintura. Mescolare l'agente PDMS / agente mescolando con una punta pipetta per 2 min. In alternativa, utilizzare un miscelatore centrifugo.
- Utilizzare un bicchiere con un diametro di 4 pollici come modello per realizzare un supporto con un foglio di alluminio. Posizionare il supporto di alluminio in un piatto di Petri da 15 cm. Posizionare un wafer di silicio da 4 pollici all'interno di questo portafoto in alluminio.
- Versare la soluzione mista PDMS nel supporto. Utilizzare una barra di vetro sottile per toccare delicatamente il bordo della wafer. Rimuovere tutte le bolle d'aria intrappolate sotto il wafer.
- Inserire il piatto di Petri da 15 cm in un camino a vuotoR per degas la soluzione PDMS. Continuare a degassare per circa 20 minuti, finché non si generano bolle. Nel frattempo, preriscaldare un forno a convezione a 65 ° C.
- Posizionare con cura il piatto di Petri da 15 cm nel forno. Incubare per 2 ore.
- Rimuovere la piastra dal forno. Utilizzare una barra di vetro per toccare delicatamente il bordo del PDMS e assicurare un collegamento reticolato completo. In caso contrario, incubare più a lungo, finché il PDMS non è solido e non appiccicoso.
- Rimuovere il foglio di alluminio dalla piastra e scollegarlo dal wafer di silicio. A partire dal bordo, scorri attentamente il PDMS curato dal wafer di silicio.
ATTENZIONE: Evitare di applicare troppa forza, in quanto può rompere il wafer. - Tagliare il PDMS in pezzi rettangolari da 20 mm x 30 mm per montare il coperchio da 24 mm x 40 mm con una lama di rasoio.
- Utilizzare una lama a scalpello per tagliare un'apertura rettangolare di 10 mm x 20 mm dal centro di ogni pezzo PDMS.
- Pulire le camere PDMS utilizzando il protocollo detergente descritto nel punto 1.1.
- Asciugare le camere PDMS in un cappuccio pulito. Posizionare le camere asciutte in un contenitore autoclavabile ricoperto di foglio di alluminio.
- Autoclave il contenitore con gravità (121 ° C, 15 psi) per 30 minuti e conservare il contenitore a temperatura ambiente.
- Coltura e transfezione di cellule BHK21
- Preparare 500 mL di terreno per la coltura di cellule BHK21 mescolando 445 mL di DMEM con 50 mL di FBS e 5 mL di 100x Penn-Strep-Glutamine (10.000 U / mL penicillina, 10 mg / mL streptomicina e 29.2 mg / mL L -Glutammina). Assicurarsi che la concentrazione finale di FBS sia del 10%.
- Aliquot 10 ml di mezzo indipendente di CO 2 non HEPES per l'imaging a cellule vive.
NOTA: il supporto indipendente di CO 2 supporta la crescita cellulare senza un incubatore di CO 2 ed è ideale per le cellule di imaging in condizioni atmosferiche. Per informazioni dettagliate, fare riferimento all'elenco Materiali. Oltre alla linea cellulare BHK21, anche altri tipi di cellule di mammiferi dovrebbero dareRisultati simili. - Assemblare un dispositivo di coltura di cellule posizionando una camera di PDMS sterilizzata autoclavata (punto 1.3) su una copertura di copertura sterile PLL (fase 1.2) in un cappuccio sterile.
- Posizionare ogni dispositivo in un piatto di Petri sterile da 60 mm.
- Utilizzare 0,5 mL di 0,25% di trypsina per staccare le cellule da un pozzetto di una piastra di coltura di tessuti a 12 pozzetti. Conta la densità cellulare con un emocitometro. Piastra 20.000 cellule BHK21 nella camera (circa 10.000 cellule / cm 2 ) con 200 μL di terreno di coltura cellulare.
- Preparare il plasmide DNA con un kit di preparazione del plasmide (vedere l'elenco dei materiali).
NOTA: Il disegno e la funzionalità del CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX sono mostrati nella Figura 1A - 1B . - Ventiquattro (24) h dopo la placcatura cellulare, trasfettano le cellule con 50-100 ng di plasmide CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX, secondo il protocollo del produttore.
- Tre (3) ore dopo la trasfezione, cambiare la mFino a 200 μl di mezzo di coltura fresco (punto 1.4.1) e consentire alle cellule di recuperare durante la notte incubando la coltura in un incubatore di CO 2 a 37 ° C.
- Imaging a cellule vive a fluorescenza
- Accendere un computer, una sorgente luminosa, un microscopio e una fotocamera. Utilizzare un microscopio confocale a fluorescenza (dotato di un obiettivo di immersione a olio 100X) per il resto del protocollo.
NOTA: può anche essere utilizzato un microscopio in epifluorescenza invertito. - Sostituire il mezzo di coltura cellulare con 200 μL di mezzo indipendente dal CO 2 .
- Impostare il protocollo di acquisizione dati prima di posizionare le celle sul microscopio. Usare un canale FITC di eccitazione 488 nm per la stimolazione ottogenetica. Utilizzare il canale TRITC di eccitazione di 561 nm per individuare le cellule trasfettate e monitorare la localizzazione cellulare della proteina con etichetta mCherry.
- Impostare il guadagno dei canali FITC e TRITC rispettivamente come 120 e 200. Utilizzare un tempo di dimensione pixel di 2,21 e la dimensione di 512 x 512 pixel.
- Misurare la potenza della luce di 488 nm mettendo un misuratore di potenza vicino alla finestra obiettivo. Un potere totale di 2 μW (circa 10.000 W / cm 2 al fuoco) è sufficiente per indurre l'associazione CIBN-CRY2PHR.
- Impostare un'acquisizione timestamp con un intervallo di 5 s e un tempo totale di acquisizione di 2 min.
- Applicare il materiale corrispondente dell'indice (olio di immersione) sulla finestra obiettiva. Utilizzare la modalità di contrasto di fase per concentrarsi sulle celle sulla superficie del coperchio.
- Individuare una cella trasfettata sotto la luce di 561 nm spostando lo stadio del microscopio.
ATTENZIONE: Evitare di utilizzare la luce blu durante questo passaggio poiché attiverà l'associazione tra le proteine fotoattivatabili. - Una volta localizzata una cella trasfettata, avviare l'acquisizione di dati.
NOTA: Le cellule trasfettate con successo devono mostrare fluorescenza in entrambi i canali FITC (da 2CIBN-GFP-CaaX) e TRITC (da CRY2-mCherry-Raf1) ( Figura 1C-D ). - Registrare una serie di immagini timbrate in entrambi i canali FITC e TRITC ( Figura 1D ).
- Per sondare la disintegrazione spontanea del complesso proteico CRY2-CIBN, registrare un altro timestamp con il canale Txred da solo per 20 minuti, con un intervallo di 30 s.
- Salvare l'immagine stampata in tempo per l'analisi dei dati.
- Accendere un computer, una sorgente luminosa, un microscopio e una fotocamera. Utilizzare un microscopio confocale a fluorescenza (dotato di un obiettivo di immersione a olio 100X) per il resto del protocollo.
- Analisi delle immagini
- Aprire le immagini con un software di analisi delle immagini che può estrarre l'intensità da un'immagine 40 .
- Selezionare due istantanee rappresentative: una prima dell'esposizione azzurra e l'altra esposizione post-blu-luce.
- Disegnare una linea attraverso la cella che attraversa lo sfondo, la membrana plasmatica e il citoplasma. Proietta l'intensità lungo questa linea. Salvare i valori e tracciarli per confrontare la differenza prima e dopo l'esposizione alla luce ( Figura 1D ).
- Per analizzare la cinetica dell'associazione proteica, selecUna regione rappresentativa di interesse (ROI) nella membrana plasmatica, un ROI nel citoplasma e un ROI in background.
- Acquisisci le intensità medie della membrana plasmatica (I PM ), il citoplasma (I CYT ) e lo sfondo (I BKD ) per lo stack di immagini.
- Calcolare il rapporto di intensità membrana / citosolica per ogni immagine utilizzando la seguente equazione:
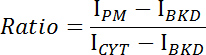
- Trama il rapporto rispetto al tempo e determina la cinetica di legame o dissociazione di CRY2-CIBN ( Figura 1E- F ).
2. Costruzione di un array LED per la stimolazione luminosa a lungo termine in un incubatore di CO 2
NOTA: Lo schema generale della configurazione sperimentale è mostrato in Figura 2A .
- Effettuare l'array LED inserendo 12 LED blu in due barriereDs e resistenze limitanti di corrente di collegamento.
NOTA: con un alimentatore a 30 V, quattro LED possono essere collegati in serie e la loro luminosità può essere controllata da una resistenza di limitazione di corrente. - Posizionare i pannelli in una scatola di alluminio.
NOTA: L'altezza della scatola di alluminio dovrebbe essere di 2 pollici. Questa altezza è ottimale per illuminare una piastra di coltura dei tessuti a 12 pozzetti, poiché la dimensione del punto di luce divergente è la stessa di quella di un singolo pozzetto. - Usare due fili metallici per collegarsi all'alimentatore. Assicurarsi che la lunghezza del filo sia sufficiente quando la scatola luminosa è posta all'interno di un incubatore di CO 2 .
- Utilizzare un diffusore di luce trasparente come coperchio della scatola luminosa ( figura 2C ).
- Calibrare l'uscita di potenza di ciascun LED ad una gamma di ingressi di tensione. Utilizzare un potere di 0,2 mW / cm 2 per il saggio di differenziazione delle cellule PC12 a 24 ore. Utilizzate un potere di 5 mW / cm 2 per embrioni o esplosioni Xenopus dal vivosaggi.
3. Induzione ottogenetica della differenziazione cellulare PC12
- Coltura e transfezione cellulare
- Preparare 500 ml di mezzo per una coltura di cellule di phaocromocytoma di ratto (PC12) mescolando 407,5 ml di F12K con 75 ml di siero di cavallo + 12,5 ml di FBS + 5 ml di 100 × Penn-Strep-Glutamine (concentrazioni finali del siero di cavallo e FBS : Rispettivamente del 15% e del 2,5%). Realizzare un substrato a basso tenore di sangue mescolando 1 volume di mezzo completo in 99 volumi di media F12K.
- Piastre PC12 in una piastra a 12 pozzetti ad una densità di 300.000 cellule / pozzetto o 75.000 cellule / cm 2 .
- Trasfettare le cellule con CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX 24 ore dopo la placcatura cellulare (simile al passaggio 1.4.7). Utilizzare 1,2 μg di DNA per ogni pozzetto. Lasciare le cellule recuperare durante la notte in terreno di coltura in un incubatore a 37 ° C completato con 5% di CO 2 . Controllare l'efficienza della trasfezione 16 h dopo la trasfezione.
NOTA: una trasfezione 30-50% effSi dovrebbe ottenere un'accurata necessità per garantire un numero sufficiente di cellule. - Cambiare il mezzo medio a basso contenuto di siero 24 h dopo la trasfezione. Utilizzare 1 ml di media per pozzetto di una piastra a 12 pozzetti.
- Differenziazione delle cellule PC12 indotta dalla luce
- Posizionare l'array LED in un incubatore CO 2 . Collegarlo all'alimentatore utilizzando un paio di fili. Impostare la potenza del LED a 0,2 mW / cm 2 . Posizionare la lastra a 12 pozzetti contenenti le cellule PC12 trasfettate (fase 3.1.3) sulla finestra dell'array LED. Applicare l'illuminazione continua della luce per 24 h in un incubatore a 37 ° C completato con 5% di CO 2 .
- Imaging di cellule vive in fluorescenza
- Seguire i punti 1.5.1-1.5.4 per il microscopio e la preparazione del campione.
- Impostare l'acquisizione di dati singola snapshot. Utilizza 200 ms per il canale GFP e Txred.
- Cattura immagini delle cellule trasfuse in entrambi i canali GFP e Txred. Registra circa 200 celLs per ogni condizione. Salvare i file per l'analisi dei dati.
- Analisi delle immagini
- Contare la percentuale di cellule differenziate sul numero totale di cellule trasfettate.
- Utilizzare qualsiasi modulo di conteggio delle celle nel software di analisi delle immagini per contare le celle.
- Contare manualmente le celle differenziate.
NOTA: le cellule differenziate sono definite come quelle in cui almeno un neurite è discernibilmente più lungo del corpo cellulare ( Figura 3A - 3B ). - Ripetere il passaggio 3.4.3 con le cellule indifferenziate.
- Calcola il rapporto di differenziazione utilizzando la seguente equazione:
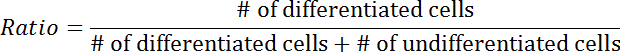
4. Controllo ottogenetico dell'attività kinasi negli embrioni Xenopus
- Preparazione dei tamponi
- Preparare 1 L di 1x Marc's Modified Ringer (MMR): 100 mMNaCl, 2 mM KCl, 1 mM MgCl2, 2 mM CaCl2 e 5 mM HEPES; PH 7,5.
- Preparazione di mRNA
- Digestare il DNA plasmidico CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX (2 μg) con 1 μl di ApaI (50 unità) a 37 ° C per 2 ore.
- Precipitare il DNA linearizzato in 60 μl di etanolo al 100%. Spinare a 12.000 × g per 5 minuti a temperatura ambiente per pelletare il DNA. Rimuovere con cautela il surnatante e lavare il pellet con 60 μl di etanolo al 70%. Spin a 12.000 × g per altri 5 minuti a temperatura ambiente. Rimuovere con cautela il surnatante e risospendere il pellet di DNA in 6 μl di H 2 O senza RNasi.
- Eseguire la trascrizione in vitro mediante incubazione di 1 μg di DNA linearizzato con 2 μl di polimerasi SP6 RNA in dotazione, una miscela ribonucleotide (10 mM ATP, CTP e UTP, 2 mM GTP e 8 mM cap analogo) e 20 μL di nucleasi nucleasi- Libera acqua a temperatura ambiente per 1 h.
- Rimuovere il DNA mediante digestione di DNasi I a 37 ° C per almeno 15 minuti e purificare l'RNA sintetizzato usando una colonna di spin di membrana di silice.
- Arrestare la reazione e precipitare l'RNA aggiungendo 30 μL di acqua priva di nucleasi e 30 μL di soluzione di precipitazione LiCl. Mescolare bene e raffreddare per 30 minuti a -20 ° C.
- Centrifugare a 4 ° C per 15 minuti a 12.000 × g per far pelliccare l'RNA.
- Rimuovere con cautela il surnatante. Lavare l'RNA una volta con 1 ml di etanolo al 70% e risospendere in 40 μL di acqua priva di nucleasi.
- Caricare 40 μL di RNA in una colonna di rotazione. Centrifugare a 4 ° C per 1 min a 12.000 × g per rimuovere i nucleotidi e le protezioni non incorporati.
- Harvest Xenopus embrioni
- Seguire il protocollo descritto da Sive et al. 41 per ottenere gli embrioni Xenopus .
- Microinjection di mRNA
- FabbricareGli aghi per la microinezione tirando i capillari di vetro con un tirante capillare. Impostare il programma di tiro su: calore = 355, forza di trazione = 80, velocità = 50 e tempo = 100.
- Rimuovere il cappotto di gelatina dagli embrioni trattandoli con 3% di cisteina (diluito in 0.2x MMR).
- Trasferire gli embrioni a 3% di polisolfuro e 0.5x di soluzione MMR per la microinezione. Iniettare 500 pg a 1 ng di CRY2-mCherry-Raf1-2A-2CIBN-GFP-CaaX RNA in ogni embrione.
NOTA: L'iniezione di una dose superiore a 1 ng produce un'attivazione indipendente dalla luce blu della segnalazione MAPK.
- Stimolazione ottogenetica dell'attività di chinasi
- Coltivare embrioni microinjected in 3% polysucrose, 0,5x soluzione MMR a temperatura ambiente fino a raggiungere la metà gastrula fase (fase 12). Quindi, coltivate gli embrioni in soluzione MMR 0,2 x.
- Trasferite gli embrioni o gli esplanti a una piastra a 12 pozzetti. Posizionare la piastra a 12 pozzetti sulla matrice LED a casa (passo 2.5) per il colore bluT (475 nm).
- Posizionare uno specchio sulla parte superiore della piastra a 12 pozzetti per assicurare un'illuminazione a pieno azzurro degli embrioni o delle esplosioni.
- Regolare la potenza della luce blu a 5 mW / cm 2 .
NOTA: Il trattamento con luce blu può essere eseguito in qualsiasi momento desiderato, sia in polisolfuro al 3%, in soluzione MMR da 0.5x, o in soluzione 0.2x MMR. - Raccogli gli embrioni in qualsiasi momento desiderato. Usalo per un'analisi istologica, Western blot o di espressione genica.
Access restricted. Please log in or start a trial to view this content.
Risultati
Espressione ratiometrica di coppie di proteine fotoattivatabili: la figura 1A mostra la progettazione di un costrutto ottogenetico bicistronico, CRY2-mCherry-Raf1-P2A-CIBN-CIBN-GFP-CaaX (denominato CRY2-2A-2CIBN), basato sulla teschovirum- 1 2A (P2A), che mostra la più alta efficacia di riduzione dei ribosomi tra le linee cellulari mammiferi 42 . Nel lavoro precedente, è stato stabilito che il rapporto ottimale per CIBN-GFP-Ca...
Access restricted. Please log in or start a trial to view this content.
Discussione
Quando si costruisce la scatola luminosa, è necessario misurare la potenza dei singoli LED. Sulla base dell'esperienza precedente, l'uscita di potenza può variare tra i singoli LED a causa della varianza di produzione. Selezionare un gruppo di LED che hanno una potenza entro il 10% dell'altro. Il numero di LED, la resistenza di limitazione corrente e l'ingresso di potenza possono essere modificati per diversi tipi di contenitori per la coltura cellulare ( ad esempio, una piastra a 6 oa un pozz...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari concorrenti.
Riconoscimenti
Questo lavoro è stato sostenuto dall'Università dell'Illinois a Urbana-Champaign (UIUC) e dagli Istituti Nazionali di Salute (NIGMS R01GM111816).
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Glass coverslip | VWR | 48393 230 | Substrate for live cell imaging |
| Coverslip holder | Newcomer Supply | 6817B | Holder for coverslips |
| Detergent | ThermoFisher | 16 000 104 | For cleaning coverslips |
| Boric acid | Sigma-Aldrich | B6768-500G | For making PLL buffer |
| Disodium tetraborate | Sigma-Aldrich | 71996-250G | For making PLL buffer |
| Plastic beaker | Nalgene | 1201-1000 | For cleaning coverslips |
| Sodium hydroxide | Sigma-Aldrich | 221465-2.5KG | For adjust pH |
| Poly-L-lysine hydrobromide | Sigma-Aldrich | P1274-500MG | For coating coverslip |
| Diethylpyrocarbonate (DEPC)-Treated Water | ThermoFisher Scientific | 750024 | For DNA preparation |
| Cover Glass Forceps | Ted Pella | 5645 | Cover glass handling |
| Tissue cutlure dish | Thermofisher | 12565321 | Cell culture dish |
| Sterile centrifuge tubes | ThermoFisher | 12-565-271 | Buffer storage |
| Transfection Reagent | ThermoFisher | R0534 | Transfection |
| CO2-independent medium | ThermoFisher | 18045088 | For live cell imaging |
| Polydimethylsiloxane (PDMS) | Ellsworth Adhesives | 184 SIL ELAST KIT 0.5KG | Form make cell chamber |
| Plasmid Maxiprep kit | Qiagen | 12965 | Plasmid preparation |
| DMEM medium | ThermoFisher | 11965-084 | Cell culturing medium component |
| F12K medium | ThermoFisher | 21127022 | Cell culturing medium component |
| Horse serum | ThermoFisher | 16050122 | Cell culturing medium component |
| Fetal Bovine Serum | Signa-Aldrich | 12303C-500 mL | Cell culturing medium component |
| Penicillin-Streptomycin-Glutamine | ThermoFisher | 10378016 | Cell culturing medium component |
| Trypsin (0.25%), phenol red | ThermoFisher Scientific | 15050065 | For mammalian cell dissociation |
| Agarose | Fisher Scientific | BP1356-100 | For DNA preparation |
| Ficoll PM400 | GE Heathcare Life Sciences | 17-5442-02 | For embryo buffer |
| L-Cysteine hydrochloride monohydrate | Sigma-Aldrich | 1.02839.0025 | Oocyte preparation |
| ApaI | ThermoFisher | FD1414 | For linearization of plasmids |
| Dnase I | ThermoFisher | AM2222 | For removing DNA template in the in vitro transcription assay |
| Index-match materials (immersion oil) | Thorlabs | MOIL-20LN | For matching the index between sample substrate and objective |
| Blue LED | Adafruit | 301 | Light source for optogenetic stimulation |
| Resistor kit | Amazon | EPC-103 | current-limiting resistor |
| Aluminum boxes | BUD Industries | AC-401 | light box |
| BreadBoard | Jekewin | 837654333686 | For making LED array |
| Hook up Wire | Electronix Express | 27WK22SLD25 | For making LED array |
| Relay Module | Jbtek | SRD-05VDC-SL-C | For intermittent light control |
| DC Power Supply | TMS | DCPowerSupply-LW-(PS-305D) | Power supply for LED |
| Silicon Power Head | Thorlabs | S121C | For light intensity measurement |
| Power meter | Thorlabs | PM100D | For light intensity measurement |
| Microscope | Leica Biosystems | DMI8 | For live cell imaging |
| BioSafety Cabinet | ThermoFisher | 1300 Series A2 | For mammalian cell handling |
| CO2 incubator | ThermoFisher | Isotemp | For mammalian cell culturing |
| Stereo microscope | Leica | M60 | For embryo micro-manipulation |
| Microinjector | Narishige | IM300 | For embryo microinjection |
| Micropipette puller | Sutter Instruments | P87 | Needle puller |
| in vitro transcription kit | ThermoFisher | AM1340 | For in vitro transcription. The kit includes nuclease-free water, SP6 RNA Polymerase, ribonucleotide mixture, cap analog, lithium choride precipitation solution, and spin column |
| RNA purfication kit | Qiagen | 74104 | Silica-membrane spin column for purification of synthesized RNA |
| Convection oven | MTI corporation | EQ-DHG-9015 | PDMS curing |
| Centrifugal mixer and teflon container | THINKY | AR310 | For mixing PDMS |
| Silicon wafer | UniversityWafer | 452 | Base for making PDMS devices |
| Blade | Techni Edge | 01-801 | For cutting PDMS |
| Capillary glass | Sutter Instruments | BF100-58-10 | For fabrication of injecting needles. |
Riferimenti
- Schlessinger, J., Ullrich, A. Growth factor signaling by receptor tyrosine kinases. Neuron. 9 (3), 383-391 (1992).
- Thisse, B., Thisse, C. Functions and regulations of fibroblast growth factor signaling during embryonic development. Dev Biol. 287 (2), 390-402 (2005).
- Perrimon, N., Pitsouli, C., Shilo, B. Z. Signaling mechanisms controlling cell fate and embryonic patterning. Cold Spring Harb Perspect Biol. 4 (8), a005975(2012).
- Salles, F. H., et al. Mental disorders, functional impairment, and nerve growth factor. Psychol Res Behav Manag. 10, 9-15 (2017).
- Basson, M. A. Signaling in cell differentiation and morphogenesis. Cold Spring Harb Perspect Biol. 4 (6), (2012).
- Schohl, A., Fagotto, F. beta-catenin, MAPK and Smad signaling during early Xenopus development. Development. 129 (1), 37-52 (2002).
- Zhang, S. W., Li, J. J., Lea, R., Amaya, E., Dorey, K. A Functional Genome-Wide In Vivo Screen Identifies New Regulators of Signalling Pathways during Early Xenopus Embryogenesis. PloS one. 8 (11), e79469(2013).
- Sweeney, C., et al. Growth factor-specific signaling pathway stimulation and gene expression mediated by ErbB receptors. J Biol Chem. 276 (25), 22685-22698 (2001).
- Marshall, C. J. Specificity of receptor tyrosine kinase signaling: transient versus sustained extracellular signal-regulated kinase activation. Cell. 80 (2), 179-185 (1995).
- Vandergeer, P., Hunter, T., Lindberg, R. A. Receptor Protein-Tyrosine Kinases and Their Signal-Transduction Pathways. Annu Rev Cell Biol. 10, 251-337 (1994).
- Lemmon, M. A., Schlessinger, J. Cell signaling by receptor tyrosine kinases. Cell. 141 (7), 1117-1134 (2010).
- Hunter, T. Signaling--2000 and beyond. Cell. 100 (1), 113-127 (2000).
- Qiu, M. S., Green, S. H. PC12 cell neuronal differentiation is associated with prolonged p21ras activity and consequent prolonged ERK activity. Neuron. 9 (4), 705-717 (1992).
- Ji, Y., et al. Acute and gradual increases in BDNF concentration elicit distinct signaling and functions in neurons. Nat Neurosci. 13 (3), 302-309 (2010).
- Luby-Phelps, K. Cytoarchitecture and physical properties of cytoplasm: volume, viscosity, diffusion, intracellular surface area. Int Rev Cytol. 192, 189-221 (2000).
- Sauer, B. Inducible gene targeting in mice using the Cre/lox system. Methods-a Companion to Methods in Enzymology. 14 (4), 381-392 (1998).
- Ling, M. M., Robinson, B. H. Approaches to DNA mutagenesis: an overview. Anal Biochem. 254 (2), 157-178 (1997).
- Gama Sosa, M. A., De Gasperi, R., Elder, G. A. Animal transgenesis: an overview. Brain Struct Funct. 214 (2-3), 91-109 (2010).
- Wanka, F., et al. Tet-on, or Tet-off, that is the question: Advanced conditional gene expression in Aspergillus. Fungal Genet Biol. 89, 72-83 (2016).
- Karginov, A. V., Ding, F., Kota, P., Dokholyan, N. V., Hahn, K. M. Engineered allosteric activation of kinases in living cells. Nat Biotechnol. 28 (7), 743-747 (2010).
- Liu, Q. Y., Deiters, A. Optochemical Control of Deoxyoligonucleotide Function via a Nucleobase-Caging Approach. Accounts of Chemical Research. 47 (1), 45-55 (2014).
- Arbely, E., Torres-Kolbus, J., Deiters, A., Chin, J. W. Photocontrol of tyrosine phosphorylation in mammalian cells via genetic encoding of photocaged tyrosine. J Am Chem Soc. 134 (29), 11912-11915 (2012).
- Gautier, A., Deiters, A., Chin, J. W. Light-activated kinases enable temporal dissection of signaling networks in living cells. J Am Chem Soc. 133 (7), 2124-2127 (2011).
- Nguyen, D. P., et al. Genetic Encoding of Photocaged Cysteine Allows Photoactivation of TEV Protease in Live Mammalian Cells. J Am Chem Soc. 136 (6), 2240-2243 (2014).
- Deisseroth, K. Optogenetics. Nat Methods. 8 (1), 26-29 (2011).
- Boyden, E. S., Zhang, F., Bamberg, E., Nagel, G., Deisseroth, K. Millisecond-timescale, genetically targeted optical control of neural activity. Nat Neurosci. 8 (9), 1263-1268 (2005).
- Banghart, M., Borges, K., Isacoff, E., Trauner, D., Kramer, R. H. Light-activated ion channels for remote control of neuronal firing. Nat Neurosci. 7 (12), 1381-1386 (2004).
- Zhang, K., Cui, B. Optogenetic control of intracellular signaling pathways. Trends in Biotechnology. 33 (2), 92-100 (2015).
- Tischer, D., Weiner, O. D. Illuminating cell signalling with optogenetic tools. Nat Rev Mol Cell Bio. 15 (8), 551-558 (2014).
- Kim, B., Lin, M. Z. Optobiology: optical control of biological processes via protein engineering. Biochemical Society Transactions. 41 (5), 1183-1188 (2013).
- Tucker, C. L. Manipulating cellular processes using optical control of protein-protein interactions. Prog Brain Res. 196, 95-117 (2012).
- Toettcher, J. E., Gong, D. Q., Lim, W. A., Weiner, O. D. Light Control of Plasma Membrane Recruitment Using the Phy-Pif System. Method Enzymol. 497, 409-423 (2011).
- Zoltowski, B. D., Gardner, K. H. Tripping the light fantastic: blue-light photoreceptors as examples of environmentally modulated protein-protein interactions. Biochemistry. 50 (1), 4-16 (2011).
- Kennedy, M. J., et al. Rapid blue-light-mediated induction of protein interactions in living cells. Nat Methods. 7 (12), 973-975 (2010).
- Krishnamurthy, V. V., et al. Reversible optogenetic control of kinase activity during differentiation and embryonic development. Development. 143 (21), 4085-4094 (2016).
- Liu, H., et al. Photoexcited CRY2 Interacts with CIB1 to Regulate Transcription and Floral Initiation in Arabidopsis. Science. 322 (5907), 1535-1539 (2008).
- Li, X., et al. Arabidopsis cryptochrome 2 (CRY2) functions by the photoactivation mechanism distinct from the tryptophan (trp) triad-dependent photoreduction. Proc Natl Acad Sci U S A. 108 (51), 20844-20849 (2011).
- Leevers, S. J., Paterson, H. F., Marshall, C. J. Requirement for Ras in Raf Activation Is Overcome by Targeting Raf to the Plasma-Membrane. Nature. 369 (6479), 411-414 (1994).
- Kohn, A. D., Takeuchi, F., Roth, R. A. Akt, a pleckstrin homology domain containing kinase, is activated primarily by phosphorylation. J Biol Chem. 271 (36), 21920-21926 (1996).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Sive, H. L., Grainger, R. M., Harland, R. M. Early Development of Xenopus laevis: A Laboratory Manual. , Cold Spring Harbor Laboratory Press. (2000).
- Kim, J. H., et al. High cleavage efficiency of a 2A peptide derived from porcine teschovirus-1 in human cell lines, zebrafish and mice. PloS one. 6 (4), e18556(2011).
- Ishimura, A., et al. Oncogenic Met receptor induces ectopic structures in Xenopus embryos. Oncogene. 25 (31), 4286-4299 (2006).
- Zhang, K., et al. Light-Mediated Kinetic Control Reveals the Temporal Effect of the Raf/MEK/ERK Pathway in PC12 Cell Neurite Outgrowth. PloS one. 9 (3), e92917(2014).
- Mohanty, S. K., Lakshminarayananan, V. Optical Techniques in Optogenetics. J Mod Opt. 62 (12), 949-970 (2015).
- Taslimi, A., et al. Optimized second-generation CRY2-CIB dimerizers and photoactivatable Cre recombinase. Nature chemical biology. 12 (6), 425-430 (2016).
- Kawano, F., Suzuki, H., Furuya, A., Sato, M. Engineered pairs of distinct photoswitches for optogenetic control of cellular proteins. Nat Commun. 6, 6256(2015).
- Wang, H., et al. LOVTRAP: an optogenetic system for photoinduced protein dissociation. Nat Methods. 13 (9), 755-758 (2016).
- Chang, K. Y., et al. Light-inducible receptor tyrosine kinases that regulate neurotrophin signalling. Nat Commun. 5, 4057(2014).
- Boulina, M., Samarajeewa, H., Baker, J. D., Kim, M. D., Chiba, A. Live imaging of multicolor-labeled cells in Drosophila. Development. 140 (7), 1605-1613 (2013).
- Liu, H., Gomez, G., Lin, S., Lin, C. Optogenetic control of transcription in zebrafish. PLoS One. 7 (11), e50738(2012).
- Buckley, C. E., et al. Reversible Optogenetic Control of Subcellular Protein Localization in a Live Vertebrate Embryo. Dev Cell. 36 (1), 117-126 (2016).
- Motta-Mena, L. B., et al. An optogenetic gene expression system with rapid activation and deactivation kinetics. Nature chemical biology. 10 (3), 196-202 (2014).
- Beyer, H. M., et al. Red Light-Regulated Reversible Nuclear Localization of Proteins in Mammalian Cells and Zebrafish. ACS Synth Biol. 4 (9), 951-958 (2015).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon