Method Article
Microscopia episcopica ad alta risoluzione (HREM) - protocolli semplici e robusti per la lavorazione e la visualizzazione di materiali organici
In questo articolo
Riepilogo
Forniamo protocolli semplici e robusti per la trasformazione di materiale bioptico di varie specie, embrioni di organismi del modello biomedico e campioni di altri tessuti organici per consentire la generazione di dati digitali di volume con il metodo di microscopia episcopica ad alta risoluzione.
Abstract
Forniamo semplici protocolli per la generazione di dati digitali di volume con il metodo di microscopia episcopica ad alta risoluzione (HREM). HREM è in grado di immaginare materiali organici con volumi fino a 5 x 5 x 7 mm 3 in risoluzioni numeriche tipiche tra 1 x 1 x 1 e 5 x 5 x 5 μm 3 . I campioni sono incorporati in resina di metacrilato e sezionati su un microtomo. Dopo ogni sezione un'immagine della superficie del blocco viene catturata con una videocamera digitale che siede sul phototube collegato alla testa del microscopio composto. L'asse ottico passa attraverso un cubo filtrante di proteine fluorescenti (GFP) verde ed è allineato con una posizione in cui il braccio del portabraccio si riposa dopo ogni sezione. In questo modo vengono prodotte una serie di immagini digitali allineate intrinsecamente, che mostrano le successive superfici di blocco. Caricare una simile serie di immagini nel software di visualizzazione tridimensionale (3D) facilita la conversione immediata in dati digitali di volume, che consentono diL'azione in diversi piani ortogonali e obliqui e la creazione di modelli di computer di volume e di superficie. Presentiamo tre semplici protocolli specifici per la lavorazione di diversi gruppi di esemplari organici, tra cui mouse, pulcino, quaglia, rana e zebra, materiale bioptico umano, materiale cartaceo e materiale per la sostituzione della pelle.
Introduzione
L'analisi strutturale dei materiali organici e anorganici è il primo passo nella comprensione delle proprietà fisiche e della loro funzione. La base di tale analisi è spesso informazioni bidimensionali (2D) ottenute con un'accurata osservazione delle sezioni istologiche con una varietà di metodi di imaging semplici e sofisticati che estraggono i dettagli dell'architettura dei tessuti, della morfologia cellulare e della topologia, della composizione molecolare e delle proprietà biomeccaniche 1 , 2 , 3 . Tuttavia, le informazioni 2D non sono adatte per la ricerca di accordi spazialmente complessi. Quindi un numero crescente di metodi in vivo e ex vivo che consentono la generazione di dati digitali di volume sono stati stabiliti negli ultimi decenni 4 e molti altri sono in fase di sviluppo.
Il principio metodico della maggior parte dei metodi di generazione dei dati di volume è la generazione di pile virtualiDi immagini digitali che visualizzano sezioni acquisite tramite la sezione virtuale o fisica di un oggetto. Se le immagini delle sezioni sono allineate correttamente, crea un volume che può essere ripartito in piani di sezione virtuale o utilizzato per creare modelli di superficie e volume resi 3D. Le tecniche popolari per la visualizzazione di esseri umani e di più grandi campioni biologici sono la tomografia a risonanza magnetica (MRT), la tomografia computerizzata (CT), la tomografia a positroni (PET) e la tomografia computerizzata per emissione a singolo fotone (SPECT). I piccoli esemplari sono di solito visualizzati utilizzando l'imaging micro-magnetico (μMRI), la tomografia di proiezione ottica (OPT), la tomografia di coerenza ottica (OCT), la tomografia fotoacoustica (PAT), i metodi basati sulla sezione istologica, la microscopia confocale e la tomografia elettronica 5 , 6 , 7 , 8 , 9 , 10 , 11 , 12 , 13 , 14 , 15 , 16 , 17 .
Una tecnica relativamente nuova di generazione dei dati di volume, che produce dati digitali di piccoli esemplari e campioni di tessuti istologici, è il metodo HREM, sviluppato in stretta collaborazione con Tim Mohun 18 , 19 . Si tratta di una semplice tecnica basata su microscopi, che genera dati digitali di volume da materiale incorporato in resina, sezionato su un microtomo. I dati facilitano l'analisi dettagliata dell'architettura dei tessuti e delle distribuzioni delle cellule, nonché l'analisi metrica delle piccole caratteristiche su un livello microscopico a luce intermedia.
HREM produce pile di immagini digitali intrinsecamente allineate che appaiono come se sono state catturate da eFrammenti istologici parzialmente colorati. Il contrasto del tessuto e la risoluzione dei dati rispetto al campo visivo superano quello dei dati prodotti con μCT, μMRI e OPT, ma sono inferiori a quelli raggiungibili con la microscopia confocale, di foglio leggero e di elettroni 20 . Tuttavia, contrariamente a quest'ultimo, HREM è in grado di visualizzare gli esemplari con volumi relativamente grandi fino a 5 x 5 x 7 mm 3 in qualità istologica. Alcuni recenti studi forniscono dettagliati caratterizzazioni e confronti di vantaggi e svantaggi delle singole tecniche di imaging e, per ragioni di obiettività, si riferiscono a quelle per ulteriori informazioni sulle loro limitazioni e potenziali campi di applicazione 4 , 21 , 22 , 23 , 24 .
Questo studio si concentra sul metodo di imaging HREM e mira a fornireProtocolli molto semplici per generare dati HREM di un ampio spettro di materiali organici, nonché esempi di applicazione. Il flusso di lavoro per la creazione di dati HREM è semplice e si applica a tutti i materiali che possono essere incorporati in resina di metacrilato ( Figura 1 ). Tuttavia ci sono differenze specifiche del tessuto nella preparazione del campione, che devono essere considerate. Di conseguenza, forniamo tre protocolli standard per la preparazione di vari campioni. I passaggi del protocollo di incorporamento e di generazione dati sono identici per tutti.
Protocollo
Tutte le procedure sono state eseguite secondo le linee guida etiche presso l'Università di Medicina di Vienna.
1. Preparazione del campione
- Preparazione di embrioni e tessuti embrionali (fino a 5 x 5 x 5 mm 3 )
- Fissare gli embrioni o parti degli embrioni in 4% di PFA / PBS o il fissante di Bouin a 4 ° C per almeno 16-24 ore sotto costante oscillazione.
NOTA: Possono essere utilizzati degli embrioni di diverse specie e di varie fasi di sviluppo, come il pesce zebra da 48 h, il pulcino e la quaglia da 19 h, e anche gli embrioni a base di prenatali, che sono stati utilizzati qui (vedere Risultati rappresentativi ). I campioni sono stati raccolti e trasferiti in PBS a temperatura ambiente per la preparazione, prima di essere messi in fissativo. Se necessario, tagliare gli esemplari con micro forbici o bisturi prima della fissazione per adattarsi agli stampi di incorporazione. - Rimuovere il fissativo e lavare il tessuto embrionale in PBS a 4 ° C sotto costante rockinG per 24 ore (2-3 cambi).
- Dehidrate i campioni in 70%, 80%, 90%, etanolo al 96% a 4 ° C per 2-3 ore ciascuno. Posizionare i tubi che contengono i campioni su un rotatore.
NOTA: per i piccoli campioni (<2 x 2 x 2 mm 3 ), l'intervallo di tempo di un campione in ogni etanolo può essere ridotto a 60 min. Per grandi campioni (> 5 x 5 x 5 mm 3 ), può essere esteso a 4 ore. - Eseguire le seguenti fasi di infiltrazione sotto una cappa di fumo che indossa guanti protettivi.
- Preparare la soluzione di infiltrazione aggiungendo 1,25 g di perossido di benzoile (catalizzatore plastificato) e 0,4 g di eosina a 100 ml di soluzione A (per maggiori dettagli, vedere tabella dei materiali ). Mescolare questo in un bicchiere su una piastra magnetica a 4 ° C finché l'eosina e il catalizzatore non sono completamente sciolti (2-3 ore).
- Posizionare i campioni per 12 - 24 h nella soluzione di infiltrazione. Tenere a 4 ° C in continua oscillazione o rotazione. Sostituire la soluzione di infiltrazione con soluzione fresca dopo 3 e 12h.
NOTA: estendere il tempo di infiltrazione a 48 h per gli embrioni di grandi dimensioni (> 1 cm di corona / lunghezza del fusto).
- Fissare gli embrioni o parti degli embrioni in 4% di PFA / PBS o il fissante di Bouin a 4 ° C per almeno 16-24 ore sotto costante oscillazione.
- Preparazione di campioni di tessuti adulti (fino a 5 x 5 x 7 mm 3 )
- Fissare i tessuti in 2% PFA / PBS contenente 4% di acido carbolico a 4 ° C per almeno 3 giorni.
NOTA: in questa soluzione possono essere fissati campioni di tessuto derivanti dalla pelle, dal fegato, dal pancreas, dal rene, dalla tiroide, dal cuore, dai muscoli striati, dal cervello, dai nervi e dai tumori di diversi umani neonatali e adulti, roditori, maiali, mosche di frutta e pesci di zebra E quindi elaborati per l'imaging HREM (dimostriamo la fissazione dei campioni; vedi i risultati rappresentativi ). Incise i campioni di tessuto con bisturi, forbici o pugni bioptici e li trasferisce direttamente in fissativo. Per la fissazione del tessuto nervoso utilizzare 4% PFA / PBS. - Rimuovere il fissativo e lavare il tessuto sotto acqua corrente per 3-6 h.
- Dehidrate i campioni successivamente placNel 70%, 80%, 90% e 96% di etanolo, mescolati con 0,4 g eosina per 100 ml (2-3 ore ciascuno). Conservare le soluzioni con campioni a 4 ° C sotto costante dondolo o su rotatore.
NOTA: per i piccoli campioni (<2 x 2 x 2 mm 3 ), l'intervallo di tempo di un campione in ogni etanolo può essere ridotto a 60 min. Per grandi campioni (> 5 x 5 x 5 mm 3 ), può essere esteso a 4-8 h. - Eseguire tutte le seguenti fasi di infiltrazione sotto una cappa aspirante che indossa guanti protettivi.
- Preparare la soluzione di infiltrazione aggiungendo 1,25 g di perossido di benzoile (catalizzatore plastificato) e 0,4 g di eosina a 100 ml di soluzione A (per maggiori dettagli, vedere tabella dei materiali ). Mescolare in un bicchiere su una piastra magnetica a 4 ° C finché l'eosina e il catalizzatore non sono completamente sciolti (2-3 ore).
- Posizionare i campioni in soluzione di infiltrazione per 24 - 36 h. Tenere a 4 ° C in continua oscillazione o rotazione. Sostituire la soluzione di infiltrazione con soluzione fresca dopo 3 e12 h.
NOTA: Per grandi campioni (> 5 x 5 x 5 mm 3 ) estendere il tempo di infiltrazione a 48 h.
- Fissare i tessuti in 2% PFA / PBS contenente 4% di acido carbolico a 4 ° C per almeno 3 giorni.
- Preparazione di altri materiali organici
NOTA: Il rivestimento della pelle sostituisce la carta e la carta alle dimensioni desiderate con le forbici ordinarie. Non è necessaria alcuna fissazione.- Salta la fase di fissaggio, lavaggio e disidratazione.
- Eseguire tutte le seguenti fasi di infiltrazione sotto una cappa aspirante che indossa guanti protettivi.
- Preparare la soluzione di infiltrazione aggiungendo 1,25 g di perossido di benzoile (catalizzatore plastificato) e 0,4 g di eosina a 100 ml di soluzione A (per maggiori dettagli, vedere tabella dei materiali ). Mescolare in un bicchiere a 4 ° C su una piastra di agitazione magnetica fino a quando l'eosina e il catalizzatore sono completamente sciolti (2-3 ore).
- Inserire i campioni in soluzione di infiltrazione per 3 - 12 h. Tenere a 4 ° C in continua oscillazione o rotazione. Sostituire la soluzione di infiltrazione con soluzione fresca dopo 1 e 2 - 4 h.
2. Embedding
NOTA: eseguire tutti i passaggi sotto una cappa aspirante che indossa guanti protettivi.
- Preparare la soluzione di infiltrazione fresca come sopra descritto (punto 1.1.4.1). Aggiungere 4 ml di soluzione B (per dettagli, vedere tabella dei materiali ) a 100 ml di soluzione di infiltrazione per preparare la soluzione di incorporazione.
NOTA: Se è necessario un orientamento preciso del campione negli stampi di incorporazione e per consentire l'inserimento di più campioni in parallelo, la polimerizzazione della soluzione di magazzino si rallenta mettendo il bicchiere in un bagno di ghiaccio. - Utilizzare i vassoi di copertura (per i dettagli, vedere tabella dei materiali ) con stampi aventi un volume di routine di 6 x 8 x 5 mm 3 o 13 x 19 x 5 mm 3 o stampi personalizzati con volumi fino a 8 x 10 x 15 mm 3 ( Figura 2 ).
- Riempire gli stampi con la soluzione di incisione e trasferire il campione nello stampo utilizzandoun cucchiaio. Durante il trasferimento assicurarsi che il campione sia completamente ricoperto da una soluzione di incasso per evitare la cattura d'aria.
NOTA: questo può essere fatto a temperatura ambiente se avviato immediatamente dopo la raccolta dei campioni e la soluzione di incasso dal frigorifero.- Orientare il campione all'interno dello stampo utilizzando aghi o pinze.
- Non appena la soluzione di incisione comincia a diventare viscosa, posizionare un elemento di bloccaggio (vedere tabella dei materiali ) sulla parte superiore dello stampo e aggiungere una soluzione di incasso attraverso il foro centrale del blocco supporto fino a quando la base del blocco di supporto è coperta da 1 a 2 Mm di soluzione di incasso.
- Sigillare il vassoio della coppa di copertura con la cera di paraffina liquida e aspettare fino ad essere indurito. In alternativa, posizionare un altro vassoio di coppa in scatola rivolto verso l'alto sul vassoio della coppa con i campioni e sigillarlo con nastro adesivo a prova d'aria.
- Consentire ai blocchi di terminare la polimerizzazione memorizzando i vassoi della tazza di stampaggio sigillati per 1 -2 giorni a temperatura ambiente.
- Per la lavorazione post-polimerizzazione, posizionare i vassoi con i blocchi polimerizzati in un forno di laboratorio standard e cuocere a 70-80 ° C per un minimo di 1-2 giorni fino a diventare rosso scuro. Raccogliere i vassoi dal forno e rimuovere i blocchi dallo stampo.
NOTA: La cottura può essere effettuata in normali condizioni ambientali. Il vassoio di copertura del rivestimento di copertura, che assicura la sigillatura dell'aria in prima fase dopo l'incasso, può essere rimosso per consentire il controllo del colore della resina dopo 10 h. Subito dopo la cottura, la resina è morbida ei blocchi possono essere facilmente rimossi dal vassoio della tazza. Entro due o tre ore sono duri e possono essere conservati a temperatura ambiente per diversi mesi.
3. Generazione di dati
NOTA: Il prototipo HREM 18 , 25 utilizzato qui comprende i seguenti elementi: (i) microtomo rotante con supporto bloccoChe si ferma dopo ogni taglio al suo punto di svolta superiore. (Ii) Coltello standard a microtomo non disponibile, metallo duro, profilo D (per dettagli, vedere tabella dei materiali ). (Iii) testa del microscopio composto da fluorescenza con rivoluzionario obiettivo e un cubo filtrante GFP (eccitazione 470/40; filtro emissione 525/50) nel suo asse ottico. L'asse ottico è disposto perpendicolarmente alla superficie appena tagliata di un blocco montato sul microtomo ed è tenuta da un dispositivo che consente il movimento verso l'alto e verso il basso. (Iv) Tabella incrociata motorizzata portante il microtomo. La tabella può essere spostata in direzione dell'asse ottico e lateralmente. V) Videocamera digitale collegata al microscopio fluorescente. (Vi) sorgente luminosa monocromatica (470 nm). (Vii) Computer collegato alla fotocamera, con software di generazione dati (per dettagli, vedere tabella dei materiali ).
- Procedura
- Indicare la regione di interesse.
- Usare il laboratorio standard li(Vedere tabella dei materiali ) per dirigere la luce bianca in modo obliquo alla superficie del blocco.
- Identificare le linee del materiale incorporato mentre si proietta alla superficie del blocco. Indicare la regione di interesse e il campo visivo successivo sulla superficie del blocco usando una penna o una lama di rasoio.
NOTA: Questo passaggio è obbligatorio per definire l'entità del campo visivo prima della sezione.
- Definisci il campo visivo.
- Montare il blocco della resina con il campione sul microtomo.
- Spostare il supporto bloccaggio nella sua posizione di arresto (punto di svolta superiore dell'escursione del blocco).
- Avviare il software della fotocamera digitale e della creazione di dati e acquisire un'immagine dal vivo.
- Scegliere una lente obiettivo, con un ingrandimento appropriato per coprire la regione di interesse indicata sulla superficie del blocco.
NOTA: vengono utilizzati obiettivi con ingrandimenti 2.5X, 5X, 10X, 20X e aperture numeriche tra 0.07 e 0.40. - Spostare l'ottico verso l'alto e verso il basso e il microtomo lateralmente utilizzando la tabella crociata motorizzata finché la regione di interesse non corrisponda al campo visivo visualizzato sullo schermo del computer.
- Messa a fuoco.
- Eseguire gradualmente la sequenza manuale fino a quando le prime strutture del campione diventano visibili.
- Disporre la superficie del blocco nel piano di messa a fuoco dell'ottica, spostando il microtomo in direzione dell'asse ottico.
- Scegli lo spessore della sezione; Sono raccomandati spessori di sezione da 0,5 a 5 μm.
- Indicare la regione di interesse.
- Generazione e elaborazione di dati
- Avviare la routine software che orchestra la sequenza e la cattura delle immagini. Seguire l'istruzione documentata del software di acquisizione delle immagini.
- Fermare la routine software e mettere una scaletta di scala davanti alla superficie del blocco non appena il campione è completamente tagliato.
- Cattura un'immagine in modo interattivo per la calibrazione successivazione.
NOTA: questo deve essere eseguito ogni volta che un obiettivo viene utilizzato in un nuovo insieme. - Convertire la serie di immagini in formato grigio a 8 bit in.jpg.
- Caricare la serie di immagini nel software di visualizzazione 3D e regolare i contrasti.
NOTA: la conversione delle immagini e la visualizzazione 3D possono essere eseguite con vari pacchetti software standard o liberamente disponibili, seguendo le istruzioni del rispettivo software.
Risultati
HREM genera una serie di immagini digitali intrinsecamente allineate con contrasti simili alle immagini delle sezioni istologiche macchiate di ematoxilina / eosina. Al contrario delle immagini a sezione 2D, le pile di immagini HREM consentono la visualizzazione e l'analisi dell'architettura dei tessuti, della morfologia e della topologia di una vasta gamma di materiali organici in 3D. L'elevato contrasto spesso consente una rapida e semplice visualizzazione 3D dei modelli di computer resi in volume e dei risultati del contorno semiautomatico per la generazione di modelli resi superficiali.
La dimensione dei dati HREM varia in base alla dimensione del target della fotocamera, alla modalità di acquisizione di immagini (8, 12, 16 bit, scala grigia, colore) e al numero di immagini a singolo blocco. I set di dati più piccoli di 1.000 immagini a scala di 8-bit.jpg di 2.048 pixel x 2.048 pixel hanno una dimensione di circa 900 MB. Grandi set di dati di 3.000 immagini a scala di 8-bit.jpg di 4.096 pIxel x 4.096 pixel hanno volumi di circa 20 GB.
I protocolli forniti sono semplici e robusti e durante l'ultimo decennio sono stati impiegati per la generazione di dati HREM di molti esemplari diversi. La sezione 1.1 del protocollo è stata ottimizzata per la trasformazione di embrioni interi di organismi modello biomedici con lunghezza fino a 1 cm e campioni di tessuti embrionali di dimensioni fino a 5 x 5 x 5 mm 3 ; Abbiamo usato questo metodo per la produzione di dati HREM di embrioni delle seguenti specie: muscoli ( Mus musculus) , pulcini ( Gallus gallus ), pesci zebra ( Danio rerio ), quaglia ( Coturnix coturnix) , rana afghana africana ( Xenopus laevis ) Equus ferus caballus ), lupo di latte ( Oncopeltus fasciatus ), coccodrillo ( Crocodylia ) e polpo ( Octopus vulgaris ). I dati di tutte le specie erano di qualità eccellente ( ad esempio , figura 3 )
3 e utilizzate per la visualizzazione di campioni di tessuti del tessuto Muscoli striati, cervello, nervi e modelli tumorali raccolti dall'uomo ( Homo sapiens ), topi ( Mus musculus ), ratti ( Rattus norvegicus ), suini ( Sus scrofa domestica ) furetti ( Mustela ), fegato, pancreas, rene, tiroide, Furo ), mosca di frutta ( Drosophila melanogaster ) e pesci zebra ( Danio rerio ). Mentre i risultati erano eccellenti con la maggior parte degli esemplari ( ad esempio , Figura 4 , Animazione 1 ), le parti molto centrali della pelle (con epidermide) e campioni cerebrali più grandi di 3 x 3 x 3 mm 3 spesso sono rimasti invariati a causa della insufficiente penetrazione di eosin attraverso Questi tessuti.
Protocollo sL'azione 1,3 è stata ottimizzata per l'elaborazione di materiale organico fibroso e impiegata per la visualizzazione dell'architettura in fibra di carta rivestita, carta non rivestita, materiale sostitutivo dermatologico nativo e materiale sostitutivo cutaneo a base di cellule staminali. Questi campioni sono stati semplici e veloci da elaborare. I dati della carta non rivestita e la maggior parte dei sostituti della pelle erano di buona qualità ( Figura 5 , Animazione 2 ). Si sono verificati problemi nell'elaborazione della carta rivestita, quando il materiale anorganico ostacolava la penetrazione dell'eosina. Un altro problema si è verificato durante l'elaborazione dei sostituti della pelle sulla base di agar perché sono diventati in parte digeriti dalla soluzione di infiltrazione.
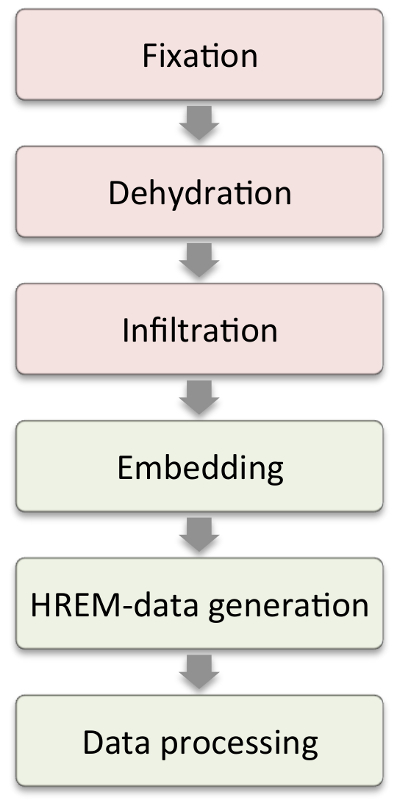
Figura 1: Flusso di lavoro. I passaggi visualizzati nelle caselle rossi richiedono modifiche secondo le caratteristiche del campione. I passaggi nelle caselle verdi sono quelli cheSono simili in tutti i campioni. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Stampi personalizzati. Le stampi possono essere adattate tagliando un foro nello stampo originale, inserendo la lampadina di una pipetta Pasteur e sigillandola con materiale di modellazione. Clicca qui per visualizzare una versione più grande di questa figura.
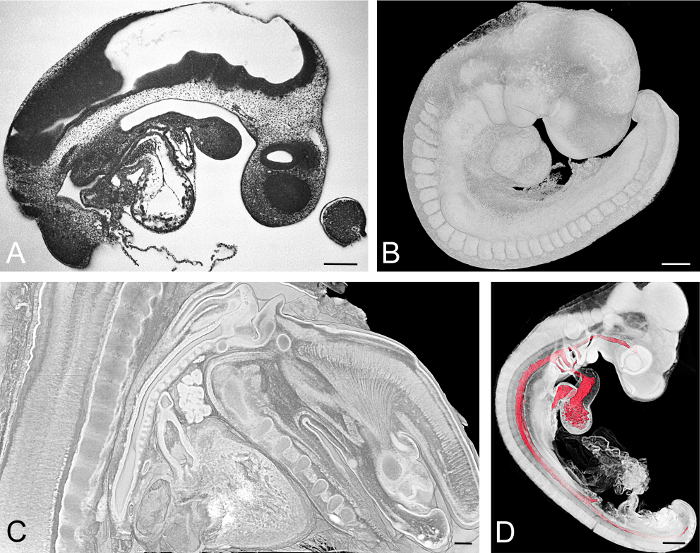
Figura 3: Embrioni esemplariamente visualizzati. (A, B) Embrione del mouse raccolto in giorno embrionale, E = 9,5. L'immagine della sezione HREM è mostrata in (A) . Il modello 3D di rendering di volume che mostra la superficie è mostrato in (B) . (C) Sezione virtuale sagittale attraverso un modello di volume reso del collo di un embrione di topo E15.5. (D) Embrione di pulcini nello sviluppo dello stadio Hamburger Hamilton (HH) 18. Modello di superficie della lumina dei componenti cardiovascolari combinato con il rendering di volume di tutti i tessuti embrionali. Barre di scala = 200 μm. Clicca qui per visualizzare una versione più grande di questa figura.
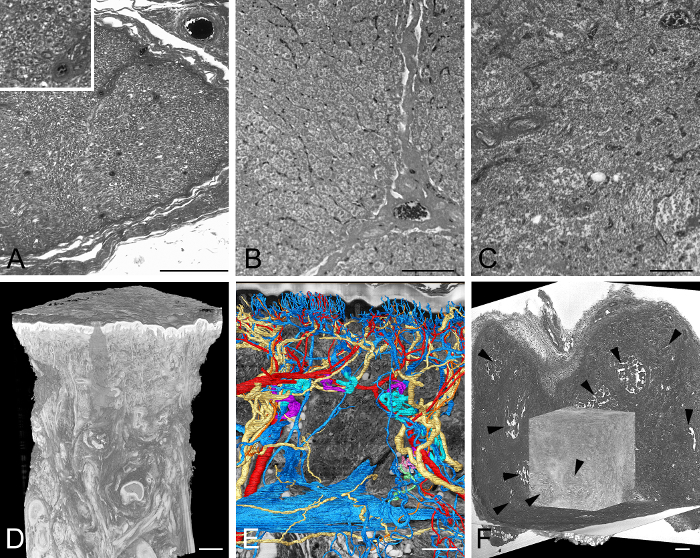
Figura 4: Esemplariamente visualizzati campioni di tessuti adulti. (A) Parte di un'immagine della sezione HREM attraverso un nervo umano. L'intarsio mostra più dettagliatamente una sezione dell'immagine. (B) Parte di un'immagine della sezione HREM attraverso il fegato di porcellini. Nota il nuclei. (C) Parte di un'immagine della sezione HREM del tessuto linfatico umano. (D, E) La pelle spessa di un pollice umano. Il volume ha reso il modello 3D dell'intera biopsia. (D) La superficie ha reso modelli di arterie, vene e nervi di fronte a una resezione virtuale attraverso i dati HREM (E) . (F) Tumore sperimentale nei tessuti adulti del topo. Il volume ha reso il modello 3D di fronte a tre sezioni virtuali attraverso i dati HREM. Notare le parti necrotiche (le frecce). Barre di scala = 200 μm. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 5: Materiali fibrosi esemplariamente visualizzati. (A, B) Immagine della sezione HREM di carta marrone non verniciata. (B) mostra una sezione di (A) . Si noti le fibre e il loro lume. (C) sezione HREM di carta rivestita. Si noti che le fibre rimangono non colorate. (D) Modello reso del volume di materiale nativo sostitutivo dermico. Si noti il diverso calibro e la forma delle fibre. Barre di scala = 100 μm. Clicca qui per visualizzare una versione più grande di questa figura.
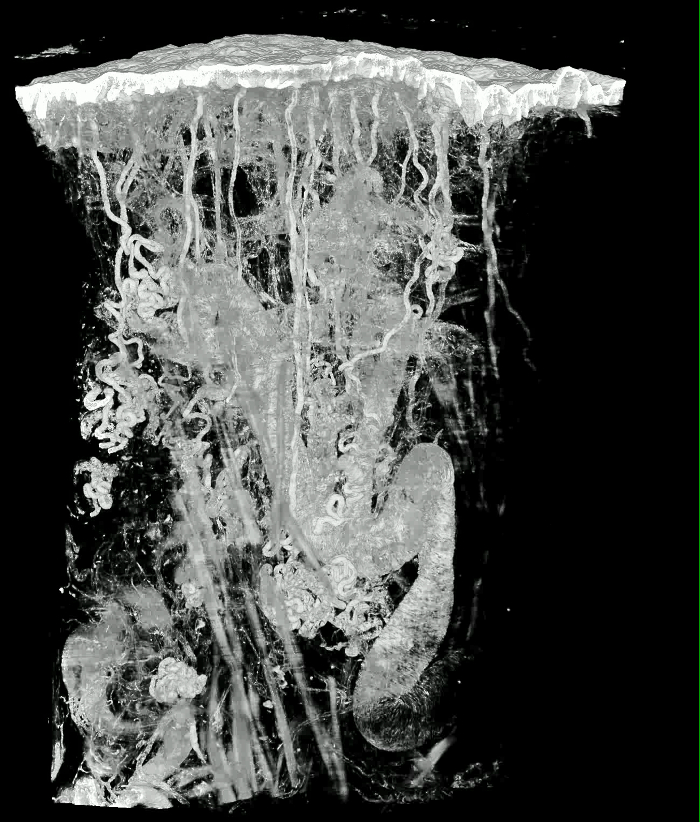
Animazione 1: Il volume ha reso il modello di pelle spessa di un pollice umano. La dimensione del blocco di tessuto è di circa 4,2 x 2,7 x 2,7 mm 3 . La dimensione del Voxel è 1,07 x 1,07 x 2 μm 3 . Clicca qui per visualizzare questo video. (Fare clic con il pulsante destro del mouse per scaricare.)
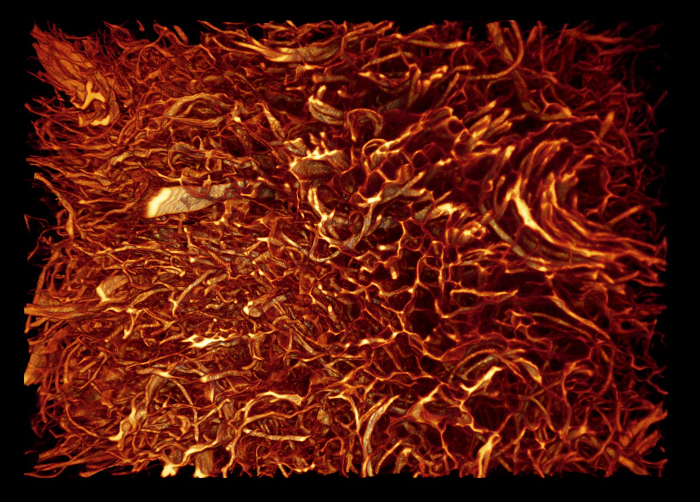
Animazione 2: Modello reso del volume del materiale nativo dermico nativo. La dimensione del campione è di circa 1,2 x 0,85 x 0,4 mm 3 . La dimensione del Voxel è di 0,54 x 0,54 x 2 μm 3 . Clicca qui per visualizzare questo video. (Fare clic con il pulsante destro del mouse per scaricare.)
Discussione
HREM è un metodo microscopico altamente robusto ideale per la visualizzazione di un ampio spettro di materiali organici impiegati in biomedicina e nell'industria 18 , 21 , 26 , 27 , 28 , 29 , 30 , 31 , 32 , 33 , 34 , 35 , 36 , 37 , 38 , 39 , 40 . Può essere impiegato come modalità di imaging esclusiva, attualmente utilizzata dal programma dei meccanismi di disturbi dello sviluppo (DMDD) 41 , 42 , 43 , 44 o come parte integrante delle tubazioni di imaging multimodale 45 .
Un apparecchio generatore di dati HREM completamente funzionante può essere assemblato da componenti convenzionali di laboratorio e comprende un microtomo motorizzato, un microscopio, una tavola incrociata motorizzata e un computer con un software appropriato 25 . È fondamentale l'uso di un microtomo dotato di un blocco di bloccaggio che riproducibilmente si ferma dopo ogni sezione in una posizione definita e cubi del filtro GFP all'interno del percorso ottico. Tuttavia, le soluzioni complete a pieno titolo possono essere acquistate da aziende come Indigo Scientific.
HREM affronta le stesse limitazioni di tutte le tecniche istologiche, tranne che non vengono introdotti manufatti durante il montaggio a sezione o sezione. Tuttavia, esistono limitazioni, che derivano dalla necessità di macchiare campioni prima della sezionamento eDalle caratteristiche del materiale incorporante. La penetrazione di eosina attraverso l'intero campione è necessaria per ottenere contatti tissutali sufficienti; Materiali molto denso, tessuti adiposi e sostanze anorganiche impediscono efficacemente la penetrazione di eosina e ciò provoca i tessuti non sottoposti al centro degli oggetti. Utilizzando speciali fissaggi aiuta a macchiare i campioni della pelle, ma non esiste ancora un metodo adeguato per superare completamente il problema. Un'altra limitazione è che le resine che bloccano più di 2 cm tendono a rompere durante la sezione. Ciò può essere parzialmente evitato tagliando separatamente i campioni e le parti di lavorazione.
Il posizionamento corretto di piccoli campioni o campioni con superfici irregolari negli stampi durante l'inserimento è spesso problematico. La copertura dei campioni con agarosio e la lavorazione dei blocchi di agarosio come descritto nel protocollo solitamente risolve questo problema 19 . Un approccio alternativo, che aiuta anche se i blocchi si rompono durante le sectiÈ quello di rimuovere il blocco già indurito dal suo supporto e riagganciare nuovamente, seguendo la procedura di incorporazione descritta.
Un set di dati HREM tipico comprende 500-3000 singole immagini. La sua risoluzione numerica è determinata dalla distanza tra le immagini successive (dallo spessore della sezione), dalla caratteristica del target della fotocamera e dalle proprietà delle ottiche utilizzate. Abbiamo utilizzato spessori di sezione compresi tra 1 μm e 5 μm e hanno ottenuto buoni risultati, anche se i protocolli presentati non eliminano completamente l'illuminazione dagli artefatti 20 , 46 . Questi artefatti sono causati da tessuti intensamente macchiati situati profondamente all'interno del blocco, con conseguente sfocatura delle informazioni sui tessuti sulle superfici del blocco.
Le telecamere hanno dimensioni target di 2,560 x 1,920 pixel 2 , 2,048 x 2,048 pixel 2 e 4,096 x 4,096 pixel 2 e sono stati combiCon obiettivi obiettivo 1.25X, 2.5X, 5X, 10X e 20X. Ciò ha portato a dimensione numerica di pixel tra 0,18 x 0,18 μm 2 e 5,92 x 5,92 μm 2 , che si è rivelato sufficiente per l'analisi 3D dell'architettura dei tessuti e delle forme cellulari e anche per la visualizzazione dei nuclei. Data l'elevata risoluzione numerica, altri organelli cellulari dovrebbero essere visibili pure. Contrasti insufficienti dovuti alla semplice colorazione di eosina e le proprietà ottiche degli obiettivi riducono drasticamente la possibilità di discriminare le strutture. La massima risoluzione spaziale massima dei dati HREM, che tiene conto dell'apertura numerica, è di circa 1 x 1 x 1 μm 3 e quindi consente solo una discriminazione effettiva di strutture superiori a circa 3 x 3 x 3 μm 3 .
Un problema comune a tutte le tecniche di digital imaging è il compromesso tra le dimensioni del campo di vista, che definisce la parte del campione che può essere visualizzataD sull'obiettivo della fotocamera e la risoluzione numerica dell'immagine. Maggiore è il campo visivo, minore è la risoluzione numerica massima possibile 46 . L'impostazione HREM qui utilizzata consente di generare dati HREM con un campo visivo compreso tra 0,74 x 0,74 mm 2 (obiettivo 20X) visualizzato in una risoluzione numerica di 0,18 x 0,18 μm 2 e 12,12 x 12,12 mm 2 (obiettivo di 1,25 X) visualizzata in Una risoluzione numerica di 2.96 x 2.96 μm 2 . I set-up alternativi e commercializzati possono fornire campi di vista più grandi, ma a costo della vera risoluzione. Tuttavia, essi forniscono risultati eccellenti, come evidente dai dati visualizzati sulla homepage del programma DMDD 47 .
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Gli autori ringraziano Tim Mohun per i suoi contributi invalute nello sviluppo di HREM e Petra Heffeter per la fornitura di campioni.
Materiali
| Name | Company | Catalog Number | Comments |
| JB-4 Plus Embedding Kit | Polysciences Europe GmbH | 18570-1 | includes Benzoyl Peroxide, Plasticized (Catalyst) and Solution A+B |
| Polyethylene Molding Cup Trays, 6 x 8 x 5 mm hexagon (9 cavities) | Polysciences Europe GmbH | 17177A-3 | |
| Polyethylene Molding Cup Trays, 13 x 19 x 5 mm (9 cavities) | Polysciences Europe GmbH | 17177C-3 | |
| JB-4 Plastic Block Holders | Polysciences Europe GmbH | 15899-50 | |
| Eosin | Waldeck GmbH & Co. KG, Division Chroma | 1A-196 | |
| Microtec CUT 4060E | rotary microtome | ||
| Leica DM LM, fluorescence compound microscope | Leica Mikrosysteme Handelges.m.b.H | ||
| GFP filter set | Leica Mikrosysteme Handelges.m.b.H | 11090937180000 | |
| Motorised cross table | Walter Uhl, technische Mikroskopie GmbH & CO. KG | KT5-LSMA | |
| Digital video camera SPOT-FLEX | Visitron Systems GmbH. | ||
| precisExcite High-Power LED | Visitron Systems GmbH. | light source | |
| VisiView 2.1.4 | Visitron Systems GmbH. | Image capturing software | |
| Hard metal knife (tungsten carbide), profile D | Leica Mikrosysteme Handelges.m.b.H | ||
| KL 2500 LCD | Schott AG | light source |
Riferimenti
- Murray, K. K., Seneviratne, C. A., Ghorai, S. High resolution laser mass spectrometry bioimaging. Methods. 104, 118-126 (2016).
- Holzlechner, M., et al. In Situ Characterization of Tissue-Resident Immune Cells by MALDI Mass Spectrometry Imaging. J Proteome Res. 16 (1), 65-76 (2017).
- Elsayad, K., et al. Spectrally coded optical nanosectioning (SpecON) with biocompatible metal-dielectric-coated substrates. Proc Natl Acad Sci U S A. 110 (50), 20069-20074 (2013).
- Norris, F. C., et al. A coming of age: advanced imaging technologies for characterising the developing mouse. Trends Genet. 29 (12), 700-711 (2013).
- Schneider, J. E., et al. High-resolution imaging of normal anatomy, and neural and adrenal malformations in mouse embryos using magnetic resonance microscopy. J Anat. 202 (2), 239-247 (2003).
- Sharpe, J. Optical projection tomography as a new tool for studying embryo anatomy. J Anat. 202 (2), 175-181 (2003).
- Metscher, B. D. MicroCT for developmental biology: A versatile tool for high-contrast 3D imaging at histological resolutions. Dev Dyn. 238 (3), 632-640 (2009).
- Syed, S. H., Larin, K. V., Dickinson, M. E., Larina, I. V. Optical coherence tomography for high-resolution imaging of mouse development in utero. J Biomed Opt. 16 (4), 046004 (2011).
- Zhang, E. Z., et al. Multimodal photoacoustic and optical coherence tomography scanner using an all optical detection scheme for 3D morphological skin imaging. Biomed Opt Express. 2 (8), 2202-2215 (2011).
- Singh, M., et al. Applicability, usability, and limitations of murine embryonic imaging with optical coherence tomography and optical projection tomography. Biomed Opt Express. 7 (6), 2295-2310 (2016).
- Weninger, W. J., Meng, S., Streicher, J., Müller, G. B. A new episcopic method for rapid 3-D reconstruction: applications in anatomy and embryology. Anat Embryol (Berl). 197 (5), 341-348 (1998).
- Weninger, W. J., Mohun, T. Phenotyping transgenic embryos: a rapid 3-D screening method based on episcopic fluorescence image capturing. Nat Genet. 30 (1), 59-65 (2002).
- Khairy, K., Keller, P. J. Reconstructing embryonic development. Genesis. 49 (7), 488-513 (2011).
- Medalia, O., et al. Macromolecular architecture in eukaryotic cells visualized by cryoelectron tomography. Science. 298 (5596), 1209-1213 (2002).
- Berrios-Otero, C. A., Wadghiri, Y. Z., Nieman, B. J., Joyner, A. L., Turnbull, D. H. Three-dimensional micro-MRI analysis of cerebral artery development in mouse embryos. Magn Reson Med. 62 (6), 1431-1439 (2009).
- Liu, M., et al. Dual modality optical coherence and whole-body photoacoustic tomography imaging of chick embryos in multiple development stages. Biomed Opt Express. 5 (9), 3150-3159 (2014).
- Mohun, T. J., Leong, L. M., Weninger, W. J., Sparrow, D. B. The morphology of heart development in Xenopus laevis. Dev Biol. 218 (1), 74-88 (2000).
- Weninger, W. J., et al. High-resolution episcopic microscopy: a rapid technique for high detailed 3D analysis of gene activity in the context of tissue architecture and morphology. Anat Embryol. 211 (3), 213-221 (2006).
- Mohun, T. J., Weninger, W. J. Embedding Embryos for High-Resolution Episcopic Microscopy (HREM). Cold Spring Harb Protoc. 2012 (6), 678-680 (2012).
- Geyer, S. H., Mohun, T. J., Weninger, W. J. Visualizing vertebrate embryos with episcopic 3D imaging techniques. ScientificWorldJournal. 9, 1423-1437 (2009).
- Bruneel, B., et al. Imaging the zebrafish dentition: from traditional approaches to emerging technologies. Zebrafish. 12 (1), 1-10 (2015).
- Adams, D., et al. Bloomsbury report on mouse embryo phenotyping: recommendations from the IMPC workshop on embryonic lethal screening. Dis Model Mech. 6 (3), 571-579 (2013).
- Heddleston, J. M., Chew, T. L. Light sheet microscopes: Novel imaging toolbox for visualizing life's processes. Int J Biochem Cell Biol. 80, 119-123 (2016).
- Powell, K. A., Wilson, D. 3-dimensional imaging modalities for phenotyping genetically engineered mice. Vet Pathol. 49 (1), 106-115 (2012).
- Geyer, S. H., Mohun, T. J., Kamolz, L. P., Weninger, W. J. High resolution episcopic microscopy - current applications. Current Biotechnology. 1 (4), 281-286 (2012).
- Weninger, W. J., Maurer, B., Zendron, B., Dorfmeister, K., Geyer, S. H. Measurements of the diameters of the great arteries and semi-lunar valves of chick and mouse embryos. J Microsc. 234 (2), 173-190 (2009).
- Geyer, S. H., Maurer, B., Dirnbacher, K., Weninger, W. J. Dimensions of the great intrathoracic arteries of early mouse fetuses of the C57BL/6J strain. The Open Anatomy Journal. 4 (1), 1-6 (2012).
- Geyer, S. H., Maurer, B., Pötz, L., Singh, J., Weninger, W. J. High-Resolution Episcopic Microscopy Data-Based Measurements of the Arteries of Mouse Embryos: Evaluation of Significance and Reproducibility under Routine Conditions. Cells Tissues Organs. 195 (6), 524-534 (2012).
- Geyer, S. H., Weninger, W. J. Some Mice Feature 5th Pharyngeal Arch Arteries and Double-Lumen Aortic Arch Malformations. Cells Tissues Organs. 196 (1), 90-98 (2012).
- Kee, H. J., et al. Ret finger protein inhibits muscle differentiation by modulating serum response factor and enhancer of polycomb1. Cell Death Differ. 19 (1), 121-131 (2012).
- Geyer, S. H., Nohammer, M. M., Tinhofer, I. E., Weninger, W. J. The dermal arteries of the human thumb pad. J Anat. 223 (6), 603-609 (2013).
- Geyer, S. H., Weninger, W. J. Metric characterization of the aortic arch of early mouse fetuses and of a fetus featuring a double lumen aortic arch malformation. Ann Anat. 195 (2), 175-182 (2013).
- Maurer, B., Geyer, S. H., Weninger, W. J. A Chick Embryo With a yet Unclassified Type of Cephalothoracopagus Malformation and a Hypothesis for Explaining its Genesis. Anat Histol Embryol. 42 (3), 191-200 (2013).
- Geyer, S. H., et al. High-Resolution Episcopic Microscopy (HREM): A Tool for Visualizing Skin Biopsies. Microsc Microanal. 20 (5), 1356-1364 (2014).
- Weninger, W. J., et al. Phenotyping structural abnormalities in mouse embryos using high-resolution episcopic microscopy. Dis Model Mech. 7 (10), 1143-1152 (2014).
- Geyer, S. H., et al. High-resolution episcopic microscopy (HREM): A useful technique for research in wound care. Ann Anat. 197, 3-10 (2015).
- Captur, G., et al. Morphogenesis of myocardial trabeculae in the mouse embryo. J Anat. 229 (2), 314-325 (2016).
- Wong, R., Geyer, S., Weninger, W., Guimberteau, J. C., Wong, J. K. The dynamic anatomy and patterning of skin. Exp Dermatol. 25 (2), 92-98 (2016).
- Jenner, F., et al. The embryogenesis of the equine femorotibial joint: The equine interzone. Equine Vet J. 47 (5), 620-622 (2015).
- Wiedner, M., et al. Simultaneous dermal matrix and autologous split-thickness skin graft transplantation in a porcine wound model: a three-dimensional histological analysis of revascularization. Wound Repair Regen. 22 (6), 749-754 (2014).
- Mohun, T., et al. Deciphering the Mechanisms of Developmental Disorders (DMDD): a new programme for phenotyping embryonic lethal mice. Dis Model Mech. 6 (3), 562-566 (2013).
- Wilson, R., et al. Highly variable penetrance of abnormal phenotypes in embryonic lethal knockout mice. Wellcome Open Res. 1, 1 (2016).
- Wilson, R., McGuire, C., Mohun, T. Deciphering the mechanisms of developmental disorders: phenotype analysis of embryos from mutant mouse lines. Nucleic Acids Res. 44 (D1), D855-D861 (2016).
- Dickinson, M. E., et al. High-throughput discovery of novel developmental phenotypes. Nature. 537 (7621), 508-514 (2016).
- Pieles, G., et al. microMRI-HREM pipeline for high-throughput, high-resolution phenotyping of murine embryos. J Anat. 211 (1), 132-137 (2007).
- Weninger, W. J., Geyer, S. H. Episcopic 3D Imaging Methods: Tools for Researching Gene Function. Curr Genomics. 9, 282-289 (2008).
- . Deciphering the Mechanisms of Developmental Disorders: DMDD Available from: https://dmdd.org.uk/ (2017)
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon