È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Analisi precisa, High-throughput di crescita batterica
In questo articolo
Riepilogo
Valutazione quantitativa della crescita batterica è essenziale per comprendere la fisiologia microbica come un fenomeno di livello dei sistemi. Un protocollo per la manipolazione sperimentale e un approccio analitico vengono introdotti, per consentire analisi precisa, ad alta velocità di crescita batterica, che è un tema chiave di interesse in biologia dei sistemi.
Abstract
Crescita batterica è un concetto centrale nello sviluppo della moderna fisiologia microbica, così come nelle indagini di dinamica cellulare a livello di sistemi. Studi recenti hanno riferito le correlazioni tra la crescita batterica e gli eventi di genoma, come riorganizzazione di riduzione e del trascrittoma di genoma. Analizzare correttamente la crescita batterica è cruciale per capire il coordinamento di crescita-dipendente delle funzioni geniche e componenti cellulari. Di conseguenza, la precisa valutazione quantitativa della crescita batterica in un modo ad alta velocità è richiesta. Gli sviluppi tecnologici emergenti offrono nuovi strumenti sperimentali che consentono aggiornamenti dei metodi utilizzati per studiare la crescita batterica. Il protocollo introdotto qui impiega un lettore di micropiastre con una procedura sperimentale altamente ottimizzata per la valutazione precisa e riproducibile di crescita batterica. Questo protocollo è stato usato per valutare la crescita di diversi ceppi di Escherichia coli in precedenza. Le fasi principali del protocollo sono come segue: la preparazione di un gran numero di cellule Stock in piccole fiale per ripetuti test con risultati riproducibili, l'uso di piastre a 96 pozzetti per valutazione di crescita ad alta velocità e il calcolo manuale delle due grandi parametri (cioè, crescita massima frequenza e densità di popolazione) che rappresenta il tasso di crescita. Rispetto ai tradizionali test di unità (CFU) formanti colonie, che conta le celle che sono coltivate in tubi di vetro nel tempo su piastre di agar, il presente metodo è più efficiente e fornisce documenti più dettagliati temporale dei cambiamenti di crescita, ma ha una più rigorosa limite di rilevabilità a bassa densità. In sintesi, il metodo descritto è vantaggioso per l'analisi di alto-rendimento preciso e riproducibile della crescita batterica, che può essere utilizzata per trarre conclusioni concettuale o effettuare osservazioni teoriche.
Introduzione
Studi microbiologici spesso iniziano con la cultura delle cellule batteriche e la valutazione delle curve di crescita batterica, che rappresentano un fenomeno fondamentale della fisiologia batterica1,2,3. Principi di cultura di base sono ampiamente disponibili nella letteratura di ricerca pubblicata e libri di testo perché la coltura batterica è una metodologia fondamentale. A livello di banco, notevole attenzione è stata concentrata tradizionalmente su ottimizzando la crescita media e condizioni di coltura, ma controllando il tasso di crescita, che sarebbe probabilmente fornire anche una maggiore comprensione della fisiologia microbica, non è stato estesamente studiato4. Per i batteri in crescita esponenziale, un parametro chiave dello stato cellulare è il tasso di crescita, che è stato segnalato per essere coordinato con il genoma, trascrittoma e proteoma5,6,7,8 . Pertanto, la valutazione quantitativa della crescita batterica è cruciale per capire la fisiologia microbica.
Per valutare la crescita batterica, i metodi sperimentali usati per stimare la biomassa sono affermati9,10 e si basano sulla rilevazione di parametri biochimici, fisici o biologici, come torbidità ottico. Inoltre, i metodi analitici utilizzati per catturare le proprietà dinamiche dei cambiamenti di crescita comunemente sono basati su modelli non lineari consolidati11,12,13, ad esempio, logistiche equazioni. Tasso di crescita è generalmente acquisite mediante campionamento temporizzato della crescita delle cellule in coltura da misura della torbidità ottico o eseguire dosaggi di formanti colonie (CFU) unità. La limitazione di questi metodi di rilevamento e di coltura è che i punti dati non sono un vero riflesso della dinamica di popolazione, poiché gli intervalli di misurazione sono spesso in ore e poiché la condizione di cultura (ad esempio, variazioni di temperatura e aerazione) è disturbato al momento del campionamento. Tecniche di analisi e cultura devono essere aggiornate utilizzando recenti sviluppi nella tecnologia e nella comprensione. Gli avanzamenti recenti in lettori di micropiastre consentono l'osservazione in tempo reale di crescita batterica e ridurre notevolmente i costi di manodopera. Utilizzando questi dispositivi avanzati, gli ultimi studi sulla crescita batterica sono riportati metodi analitici per misure ad alta velocità14,15.
Lo scopo del presente protocollo è quello di valutare la dinamica di crescita precise in un modo ad alta velocità, che si riveleranno utile per studi quantitativi che, infine, affrontare le questioni di come viene determinato il tasso di crescita e fattori che influenzano il tasso di crescita. Il protocollo risolve tutti i fattori che dovrebbero essere presi in considerazione per la quantificazione precisa e ripetibile di crescita batterica. Il metodo sperimentale e l'analisi sono descritti dettagliatamente nel testo principale. Questo metodo permette l'analisi precisa e riproducibile della crescita batterica in un modo ad alta velocità. Microbiologi possono utilizzare questo protocollo per derivare ulteriori risultati quantitativi da loro evidenza sperimentale. Questo protocollo è utilizzabile anche per gli studi in biologia dei sistemi che tentano di trarre conclusioni concettuale o per ottenere una panoramica teorica della crescita.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. preparare il mezzo di crescita
Nota: la composizione chimica di M63 medio minimo è il seguente: 62 mM K 2 HPO 4, 39mm KH 2 PO 4, 15mm (NH 4) 2 SO 4, 1,8 µM FeSO 4, 15 µM tiamina-HCl, 0,2 mM MgSO 4 e 22 millimetri di glucosio. M63 è fatto mescolando tre soluzioni di riserva: soluzione X cinque, 20% glucosio e soluzione di MgSO 4 della tiamina. Conservare tutte le soluzioni a 4 ° C.
- Preparare la soluzione di cinque X
- per preparare FeSO 4 soluzione, utilizzare una pipetta elettrica e una pipetta sierologica per aggiungere ddH 2 O a una provetta da centrifuga da 50 mL. Utilizzare un P-200 per aggiungere 0,06 mL HCl 36mg FeSO 4 M HCl. aggiungere 0.01-7h 2 O e mescolare bene.
- Misurare 160 mL ddH 2 O in un cilindro graduato e aggiungerlo a un becher da 500 mL. Aggiungere il codice seguente in questo ordine: 10,72 g K 2 HPO 4, 5,24 g KH 2 PO 4, 2,0 g (NH 4) 2 SO 4 e 0,5 mL di soluzione di 4 FeSO (preparata al punto 1.1.1).
- Utilizzando un misuratore di pH di precisione, regolare il pH a 7.0 con l'aggiunta di 2 M di KOH. Versare la soluzione in un cilindro graduato e aggiungere ddH 2 O a un volume totale di 200 mL. Sterilizzare la soluzione utilizzando un'unità di filtrazione da 250 mL (PVDF, da 0,22 µM).
- Preparazione della soluzione di glucosio 20%
- misurare 200 mL ddH 2 O in un cilindro graduato e versarlo in un recipiente di 500 mL. Continuando a mescolare con un agitatore magnetico, aggiungere 50 g di glucosio. Diluire la soluzione in un cilindro graduato per un volume totale di 250 mL. Sterilizzare la soluzione, come descritto al punto 1.1.3.
- Preparazione della soluzione di tiamina MgSO 4
- aggiungere 150 mL ddH 2 O in un becher da 500 mL. Continuando a mescolare con un agitatore magnetico, è necessario aggiungere 1,0 g di tiamina-HCl e 10 g di MgSO 4-7 H 2 O. Diluire la soluzione in un cilindro graduato per un volume totale di 200 mL. Sterilizzare la soluzione, come descritto al punto 1.1.3.
- Preparazione minima media M63
- posto la soluzione X cinque, la soluzione di glucosio 20%, la soluzione di MgSO 4 della tiamina, una pipetta elettronica, una pipetta P-200 e puntali per pipette 200-µ l su un banco pulito.
- Misurare 155,8 mL ddH 2 O in un cilindro graduato e versarlo in un becher da mL 500.
- Usando la pipetta elettronica e pipette sierologiche monouso, aggiungere 40 mL di soluzione X cinque e 4 mL di soluzione di 20% glucosio al ddH misurato 2 O. Quindi, aggiungere 0,2 mL di soluzione di MgSO 4 della tiamina con il P-200. Sterilizzare la soluzione, come descritto al punto 1.1.3.
2. Preparare il brodo di glicerolo
- cella cultura
Nota: I ceppi batterici (ad es., W3110 e suo genoma ridotto) sono disponibili da organizzazioni della banca di ceppo. I ceppi sono ottenuti solitamente sotto forma di colonie su piastre di agar.- Posto cinque tubi di vetro sterilizzati con tappi di gomma del silicone, una pipetta elettronica, P-1,000, puntali per pipette 1.000-µ l, P-200, puntali per pipette 200-µ l e i ceppi di destinazione (colonie su piastre) su un banco pulito.
- Esporre la bocca del tubo di vetro di un becco Bunsen prima di aprire il tappo di gomma del silicone. Esporre il tappo di gomma del silicone alla fiamma dopo il tubo è aperto e poi leggermente posizionare il tappo sul tubo di vetro.
- Utilizzare la pipetta elettronica e pipette sierologiche monouso per aggiungere uno dei tubi di vetro e 4,5 mL M63 nelle altre quattro provette 5 mL M63.
- Utilizzare la punta di P-200 per prendere una Colonia e inoculare e nel tubo di vetro contenente 5 mL M63.
- Vortex il tubo per fare una sospensione. Quindi, la soluzione diluita 10 volte da trasferire 0,5 mL di questa soluzione a uno dei quattro tubi contenente 4,5 mL M63.
- Ripetere il procedimento descritto al punto 2.1.5 per i rimanenti tubi. Una serie di diluizioni con cinque diverse concentrazioni (diluizioni di 1, 10, 100, 1000 e 10.000) è ora pronto.
- Sterilizzare le foci dei tubi di vetro e i tappi di gomma di silicone come descritto al punto 2.1.2. Le provette con i tappi di Cap. Non arricciando i tappi di gomma di silicone per evitare contaminazione.
- Posizionare i cinque tubi in un'incubatrice di agitazione pre-riscaldata a 37 ° C e agitare a 200 giri/min. Incubare la cultura durante la notte o per h. 10-30
- Selezione della cultura per lo stock di glicerolo
- inserire il mezzo di temperatura pre-riscaldato M63, P-1,000, puntali per pipette 1.000-µ l e una cuvetta monouso su un banco pulito.
- Aggiungere 1000 µ l M63 a una cuvetta monouso con un P-1, 000. Disponga la provetta monouso con uno spettrofotometro, avviare il programma a una lunghezza d'onda fissa di 600 nm e misura lo spazio in bianco.
- Spostare i tubi di cinque vetro dall'incubatrice che agita il banco pulito.
- Scartare M63 dalla cuvette monouso e aggiungere 1.000 µ l cultura per la stessa cuvetta monouso con un P-1, 000. Misurare la torbidità ottica della coltura delle cellule (OD 600) come descritto al punto 2.2.2.
Nota: Per evitare qualsiasi contaminazione e per ottenere una misura precisa, esporre i tubi di vetro e i tappi per la fiamma come descritto e vortice la cultura prima del campionamento. Per garantire risultati affidabili, misurazioni ripetute sono raccomandate, in particolare quando la densità cellulare è bassa. - Dei cinque colture cellulari, scegliere quello che è in fase di crescita esponenziale precoce (OD 600 = 0,01 - 0,05) per lo stock di glicerolo.
Nota: Se le colture multiple hanno densità all'interno della gamma ottima, quella più vicina a 0,05 è comunemente selezionata.
- Le scorte di glicerolo per ripetuti test.
Nota: Questo è descritto per la preparazione di dieci titoli. Quantità maggiori o minori possono essere fatti secondo i requisiti sperimentali.- Posto la soluzione di glicerolo 60% sterilizzata, dieci sterilizzato microprovette da 1,5 mL, P-1,000 e P-200, puntali per pipette 1.000 e 200 µ l e una microprovetta stare su un banco pulito.
- Aggiungere 250 µ l di soluzione di glicerolo di 60% e 750 µ l della cultura cella selezionata nella microprovetta 1,5 mL e mescolare sterilizzati pipettando.
Nota: Il volume di stock è variabile, ma sempre mantenere un rapporto di 1:3 di 60% glicerolo alla coltura delle cellule; Questo si traduce in una concentrazione finale di glicerolo al 15%. - Inserire i restanti nove microprovette nello stand conetti e pipettare 100 µ l della miscela preparata al punto 2.3.2 in ogni provetta. Ora ci sono dieci scorte di glicerolo identici per un utilizzo futuro.
- Conservare le scorte in un congelatore a -80 ° C.
3. Acquisendo le curve di crescita
- impostare il lettore di micropiastre.
Nota: I termini riportati in virgolette Visualizza il fraseggio specifico utilizzato il lettore della piastra utilizzato qui (Vedi la tabella materiali).- Apri il software. Aperto " protocolli " in " Task Manager " e scegliere " Crea nuovo ". Scegliere " protocollo Standard ".
- Open " Procedure " e regolare le impostazioni. Aperto " impostare la temperatura di " e selezionare " incubatore su ". Impostare " temperatura " a 37 ° C e " gradiente " a 0 ° C. Check " Preheat " prima di continuare con il passaggio successivo. Aperto " avviare Kinetic ", set " Runtime " per 24:00:00 o 48:00:00, e " intervallo " per 00:30:00 o 01:00.
Nota: Ci vuole circa 1 minuto per leggere un intero 96 piastra ben. - Open " Shake " e impostare " Shake modalità " come " lineare ". Verifica " Constitution Shake " e impostare " frequenza " al cpm 567. Aperto " lettura ", controllare " assorbanza ", " Endpoint/cinetica ", e " monocromatori. " Set " lunghezza d'onda " a 600.
- Clic " Validate " per confermare che la procedura è corretta. Fare clic su " salvare " per salvarlo come un nuovo programma per un uso futuro.
- Registrazione di crescita in tempo reale
- disegnare un pittogramma di piastra a 96 pozzetti (8 × 12 tavolo) per indicare le posizioni dei campioni inoculati cultura sulla piastra 96 pozzetti. Stampare la tabella e utilizzarlo come riferimento per l'esperimento.
Nota: I pozzetti situati sui bordi della micropiastra devono contenere solo mezzo vuoto a causa di evaporazione. - Posto un piatto sterilizzato di 96 pozzetti fondo micropiastra con coperchio, P-1000, puntali per pipette 1000-µ l, P-200, puntali per pipette 200-µ l, diversi microprovette da 1,5 mL, una pipetta multicanale 8, un serbatoio del reagente sterilizzato, temperatura ambiente M63 e glicerolo scorte (preparate al punto 2.3) su un banco pulito.
- Aggiungere circa 25 mL M63 per il serbatoio del reagente. Utilizzare questo stock di serbatoio per tutti i passaggi seguenti.
- µ L aggiungere 900 M63 per microprovette in preparazione per effettuare diluizioni seriali.
- Scongelare il brodo di glicerolo a temperatura ambiente. µ L 900 M63 alla Stock in glicerolo scongelati e vortice. Questo si traduce in una diluizione di 10 volte del titolo originale glicerolo.
- Trasferire 100 µ l della diluizione 10 volte ad un'altra microprovetta contenente 900 µ l di M63 e vortex. Questo si traduce in una diluizione di 100 volte.
- Ripetere il punto 3.2.6 fino al raggiungimento del numero desiderato di diluizioni.
- Riempire i pozzetti al bordo della micropiastra con 200 µ l M63 utilizzando una pipetta a 8 canali (P-200).
Nota: Questi pozzi possono essere utilizzati come il vuoto. - Carico 200 µ l di ogni campione preparato nei passaggi 3.2.4 - 3.2.7 dei pozzetti di micropiastra secondo la tabella di riferimento (punto 3.2.1) diluito. Vortice il priore di campioni diluiti a carico e carico lo stesso campione in pozzi multipli alle varie posizioni sulla piastra.
- Appoggiare la micropiastra a 96 pozzetti al lettore di piastra.
- Open " lettura ora " in " Task Manager " e scegliere il programma (sezione 3.1). Fare clic su " OK " per avviare la misurazione. Salvare la registrazione come un nuovo file sperimentale per l'analisi dei dati (sezione 4).
- disegnare un pittogramma di piastra a 96 pozzetti (8 × 12 tavolo) per indicare le posizioni dei campioni inoculati cultura sulla piastra 96 pozzetti. Stampare la tabella e utilizzarlo come riferimento per l'esperimento.
4. Analisi dei dati
- esportazione i risultati dei dati in tempo reale del tasso di crescita (sezione 3.2) da una memoria USB stick come un file di testo.
Nota: Un risultati formattati 96 pozzetti (curve) visualizzerà il lettore della piastra in tempo reale. I record orari (valori OD) possono essere esportati come un tavolo; le righe e le colonne rappresentano i numeri ben (ad esempio, A1, B1) e il tempo di misurazione (ad es., 00:00:59), rispettivamente. - Aprire il file di testo con un software di foglio di calcolo.
- Copiare le letture di 600 OD orarie della micropiastra 96 pozzetti in un nuovo foglio di lavoro per ulteriori analisi.
- Subtract background legge al tempo zero dalla legge orarie di ciascun campione ben.
Nota: Per comodità, il valore medio del bianco pozzetti contenenti M63 (punto 3.2.8) può essere utilizzato come il valore background. - Calcolare la media dei cinque consecutivi OD 600 letture per stimare la densità di popolazione massima.
Nota: Il valore medio più grande dei cinque consecutivi OD 600 legge è definito come il massimo OD 600 della curva corrispondente crescita. - Calcolare il tasso di crescita, µ (h -1), applicando la seguente equazione per tutte le coppie di valori consecutivi di OD 600:
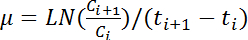
Nota: In questa equazione, C i e C i + 1 rappresentano i valori di 600 OD di due qualsiasi punti di volta consecutiva (t i e t i + 1, rispettivamente). LN indica il logaritmo naturale. - Calcola i cambiamenti nel tasso di crescita nel corso del tempo basati su orario OD 600 legge secondo punto 4.6. Calcolare la media e la deviazione standard dei cinque tassi di crescita consecutiva di stimare il tasso di crescita massima.
Nota: La media più grande con la più piccola deviazione standard è definita come il tasso di crescita massima della curva corrispondente crescita.
5. Confermando la tendenza globale del legge 96 pozzetti (opzionale)
Nota: entrambe la piastra reader e la piastra a 96 pozzetti consumabili possono causare misurazioni distorte. Per ottenere risultati quantitativi altamente precisi e riproducibili, confermando la tendenza globale della piastra 96 pozzetti è altamente raccomandato.
- Preparare la piastra a 96 pozzetti e il lettore di piastre per il bias test come descritto nella sezione 3.2.2.
- Aggiungere 20 mL M63 a una provetta da centrifuga da 50 mL sterilizzato. Aggiungere il brodo di glicerolo alla stessa provetta da centrifuga e vortice. Trasferire la soluzione sospesa ad un serbatoio di reagente sterilizzato.
- Utilizzare una pipetta a 8 canali per trasferire 200 µ l della soluzione sospesa la piastra a 96 pozzetti.
- Appoggiare la piastra a 96 pozzetti al lettore di piastra e avviare la misurazione.
- Record oraria OD 600 legge e analizzare come descritto nella sezione 4. Confronta la densità di tasso e popolazione di crescita massima calcolata di ciascun pozzetto per determinare la localizzazione bias del legge 96 pozzetti.
Access restricted. Please log in or start a trial to view this content.
Risultati
Il metodo descritto fornisce un mezzo per catturare la dinamica crescita batterica in modo continuo, ad alta velocità utilizzando un lettore di formato da 96 pozzetti che prende misurazioni multiple di densità ottica a vari intervalli di tempo (da minuti a ore o giorni). Le curve di crescita di un assortimento di ceppi di Escherichia coli che esprimono vari genomi possono essere acquisite proprio in un singolo esperimento (Figura 1A). In confronto ...
Access restricted. Please log in or start a trial to view this content.
Discussione
Fasi critiche nel protocollo prevedono la preparazione di un fondo comune delle cellule e la replica di stessi campioni in pozzi multipli in diverse posizioni sulla micropiastra in crescita esponenziale. Microbiologi avviato in precedenza, la cultura da una pre-cultura durante la notte. Mentre questo metodo può ridurre il tempo di ritardo di crescita batterica, è difficile da realizzare curve di crescita riproducibile. Come mostrato nella Figura 2, le misure indipendenti utilizzando le sco...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Ringraziamo Kohei Tsuchiya per fornire l'esempio di analisi CFU. Questo lavoro è stato parzialmente finanziariamente supportato da una sovvenzione per ricerca scientifica (C) n. 26506003 (con BWY) dal Ministero della pubblica istruzione, cultura, sport, scienza e tecnologia, Giappone.
Riconoscimenti
Gli autori non hanno nulla a rivelare.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| K2HPO4 | Wako | 164-04295 | |
| KH2PO4 | Wako | 166-04255 | |
| (NH4)2SO4 | Wako | 019-03435 | |
| MgSO4-7H2O | Wako | 138-00415 | |
| Thiamine-HCl | Wako | 201-00852 | |
| glucose | Wako | 049-31165 | |
| HCl | Wako | 080-01066 | |
| Iron (II) sulfate heptahydrate (FeSO4-7H2O) | Wako | 094-01082 | |
| KOH | Wako | 168-21815 | |
| Glycerol | Wako | 075-00611 | |
| Centrifuge tube (50 mL, sterilized) | WATSON | 1342-050S | |
| Pipette Tips, 200 µL | WATSON | 110-705Y | |
| Pipette Tips, 1,000 µL | WATSON | 110-8040 | |
| Microtube (1.5 mL) | WATSON | 131-715C | |
| 8 multichannel-pipette | WATSON | NT-8200 | |
| PASORINA STIRRER | AS ONE | 2-4990-02 | |
| Glass cylinder (200 mL) | AS ONE | 1-8562-07 | |
| Precision pH mater | AS ONE | AS800 / 1-054-01 | |
| Pipetman P-200 | GILSON | 1-6855-05 | |
| Pipetman P-1000 | GILSON | 1-6855-06 | |
| Disposable Serolocical Pipettes (10 mL) | SANPLATEC | SAN27014 | |
| Disposable Serolocical Pipettes (25 mL) | SANPLATEC | SAN27015 | |
| Microtube stand | BM Bio | 801-02Y | |
| Vortex | BM Bio | BM-V1 | |
| Corning Costar 96-well microplate with lid (Flat bottom, Clear) | Sigma-Aldrich | Corning, 3370 | |
| Corning Costar reagent reservoir (50 mL) | Sigma-Aldrich | Corning, 4870 | |
| Stericup GV PVDF (250 mL, 0.22 µM) | Merck Millipore | SCGVU02RE | |
| Pipet-Aid XP | DRUMMOND | 4-000-101 | |
| Bioshaker (BR-23UM MR) | TAITEC | 0053778-000 | |
| Disposal cell (1.5 mL) | Kartell | 1938 / 2-478-02 | |
| DU 730 Life Science UV/Vis Spectrophotometer | Beckman Coulter | A23616 | |
| EPOCH2 | BioTek | 2014-EP2-002 / EPOCH2T | |
| Beaker (500 mL) | IWAKI | 82-0008 | |
| BIO clean bench | Panasonic | MCV-B131F | |
| Glass tubes | NICHIDEN RIKA GLASS | P-10M~P-30 /101019 | |
| Silicone rubber stoppers | ShinEtsu Polymer | T-19 | |
| Bacterial strains | Strain bank organization; National Bio Resource Project (NBRP) in Japan |
Riferimenti
- Kovarova-Kovar, K., Egli, T. Growth kinetics of suspended microbial cells: from single-substrate-controlled growth to mixed-substrate kinetics. Microbiol Mol Biol Rev. 62 (3), 646-666 (1998).
- Soupene, E., et al. Physiological studies of Escherichia coli strain MG1655: growth defects and apparent cross-regulation of gene expression. J Bacteriol. 185 (18), 5611-5626 (2003).
- Sezonov, G., Joseleau-Petit, D., D'Ari, R. Escherichia coli physiology in Luria-Bertani broth. J Bacteriol. 189 (23), 8746-8749 (2007).
- Egli, T. Microbial growth and physiology: a call for better craftsmanship. Front Microbiol. 6, 287(2015).
- Kurokawa, M., Seno, S., Matsuda, H., Ying, B. W. Correlation between genome reduction and bacterial growth. DNA Res. 23 (6), 517-525 (2016).
- Matsumoto, Y., Murakami, Y., Tsuru, S., Ying, B. W., Yomo, T. Growth rate-coordinated transcriptome reorganization in bacteria. BMC Genomics. 14, 808(2013).
- Nahku, R., et al. Specific growth rate dependent transcriptome profiling of Escherichia coli K12 MG1655 in accelerostat cultures. J Biotechnol. 145 (1), 60-65 (2010).
- Dai, X., et al. Reduction of translating ribosomes enables Escherichia coli to maintain elongation rates during slow growth. Nat Microbiol. 2, 16231(2016).
- Madrid, R. E., Felice, C. J. Microbial biomass estimation. Crit Rev Biotechnol. 25 (3), 97-112 (2005).
- Harris, C. M., Kell, D. B. The estimation of microbial biomass. Biosensors. 1 (1), 17-84 (1985).
- Yates, G. T., Smotzer, T. On the lag phase and initial decline of microbial growth curves. J Theor Biol. 244 (3), 511-517 (2007).
- Kargi, F. Re-interpretation of the logistic equation for batch microbial growth in relation to Monod kinetics. Lett Appl Microbiol. 48 (4), 398-401 (2009).
- Peleg, M., Corradini, M. G. Microbial growth curves: what the models tell us and what they cannot. Crit Rev Food Sci Nutr. 51 (10), 917-945 (2011).
- Sprouffske, K., Wagner, A. Growthcurver: an R package for obtaining interpretable metrics from microbial growth curves. BMC Bioinformatics. 17, 172(2016).
- Hall, B. G., Acar, H., Nandipati, A., Barlow, M. Growth rates made easy. Mol Biol Evol. 31 (1), 232-238 (2014).
- Hermsen, R., Okano, H., You, C., Werner, N., Hwa, T. A growth-rate composition formula for the growth of E.coli on co-utilized carbon substrates. Mol Syst Biol. 11 (4), 801(2015).
- Engen, S., Saether, B. E. r- and K-selection in fluctuating populations is determined by the evolutionary trade-off between two fitness measures: Growth rate and lifetime reproductive success. Evolution. 71 (1), 167-173 (2017).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon