Method Article
Riconoscimento di sequenza-specifico e selettivo di Double-stranded RNA sopra Single-stranded RNA da modificati chimicamente acidi peptido nucleici
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Segnaliamo i protocolli per la sintesi e purificazione degli oligomeri di Peptide degli acidi nucleici (PNA) che incorporano per volta residui. Sono descritti i metodi biochimici e biofisici per la caratterizzazione del riconoscimento del RNA duplex dal PNAs modificate.
Abstract
RNAs stanno emergendo come biomarcatori importanti e bersagli terapeutici. Così, c'è grande potenziale nello sviluppo di sonde chimiche e terapeutiche ligandi per il riconoscimento della sequenza di RNA e struttura. Modificati chimicamente Peptide degli acidi nucleici (PNA) oligomeri sono state recentemente sviluppati in grado di riconoscere RNA duplex in modo sequenza-specifico. I PNA sono chimicamente stabili con una dorsale di peptide-come neutra. PNAs possono essere sintetizzati relativamente facilmente dal metodo di sintesi del peptide di fase solida Boc-chimica manuale. PNA è purificati mediante HPLC in fase inversa, seguita dalla caratterizzazione di peso molecolare in matrix-assisted laser desorption/ionizzazione-tempo di volo (MALDI-TOF). Non-denaturante del gel di poliacrilammide tecnica di elettroforesi (pagina) facilita l'imaging della formazione triplex, perché attentamente progettato gratis RNA duplex costruisce e PNA associato triplexes spesso tassi di migrazione diversi Visualizza. PAGINA non-denaturante con etidio bromuro post colorazione spesso è una tecnica facile ed informativa per caratterizzare l'affinità di legame e la specificità degli oligomeri PNA. In genere, più RNA forcine o duplex con singola coppia di basi mutazioni utilizzabile per caratterizzare le proprietà di associazione di PNA, come l'associazione affinità e specificità. 2-Aminopurine è un isomero di adenina (6-aminopurine); l'intensità di fluorescenza 2-aminopurine è sensibile ai cambiamenti di ambiente strutturale locale ed è adatto per il monitoraggio della formazione triplex con il residuo di 2-aminopurine incorporato nei pressi del sito di associazione di PNA. Titolazione fluorescenza 2-Aminopurine può essere utilizzato anche per confermare la selettività di associazione dei PNA modificate verso mirati RNA double-stranded (dsRNA) sopra RNA a singolo filamento (ssRNAs). Esperimenti di fusione termici UV-assorbanza-rilevato consentono la misura della stabilità termica di PNA-RNA duplex e PNA· Triplexes2 di RNA. Qui, descriviamo la sintesi e purificazione degli oligomeri PNA che incorporano modificato residui e descrivere i metodi biochimici e biofisici per la caratterizzazione del riconoscimento del RNA duplex dal PNAs modificate.
Introduzione
RNAs stanno emergendo come biomarcatori importanti e bersagli terapeutici, dovuto gli avanzamenti recenti nella catalisi di diversi processi biologici1,2,3e le scoperte dei ruoli degli RNA nel regolamento. Tradizionalmente, i fili antisenso sono stati utilizzati per associare a ssRNAs a Watson-Crick formazione duplex3,4,5,6,7,8, 9 , 10 , 11 , 12 , 13 , 14 , 15 , 16 , 17 , 18 , 19 , 20 , 21 , 22 , 23 , 24 , 25 , 26 , 27. recentemente, triplex-formando acidi peptido nucleici (TFPNAs) sono stati progettati per associare a dsRNA via (Figura 1)3,28,29di bonding dell'idrogeno di Hoogsteen. regioni di dsRNA sono presenti nella maggior parte dei tradizionali RNA antisenso-mirati, tra cui pre-mRNA e mRNA, pre- o pri-miRNA3e molti altri non-codificazione RNAs1,26,27. RNAds attraverso formazione di tripla elica usando TFPNAs di targeting può essere vantaggioso a causa della sua specificità di struttura ed è di grande potenziale per l'uso nel ripristinare le normali funzioni degli RNA, che sono dysregulated in malattie, per esempio.
La recente pubblicazione funziona da Rozners et al.e noi3,28,29,30,31,32,33, 34,35,36,37,38,39,40,41, segnalati gli sforzi sul miglioramento il legame selettivo di modificate TFPNAs verso dsRNA con maggiore affinità. Abbiamo sviluppato metodi di sintesi per razionalmente progettati monomeri PNA (Figura 2) tra cui thio-pseudoisocytosine (L) monomero30 e guanidina-modificato 5-metil citosina (Q) monomero31. Attraverso vari metodi di caratterizzazione biochimica e biofisica, abbiamo dimostrato che PNAs relativamente breve (6-10 residui) che incorpora L e Q residui mostrano migliore riconoscimento di coppie di basi di Watson-Crick G-C e C-G, rispettivamente, di dsRNA. Inoltre, rispetto ai non modificato PNAs, PNAs contenenti residui L e Q mostrano associazione più selettivi verso dsRNA sopra ssRNA e dsDNA. La funzionalità di guanidina42 nella base Q consente PNAs entrare di cellule HeLa31.
Nel nostro laboratorio, sintetizziamo PNAs dalla Boc-chimica manuale (Boc o t- Boc acronimo di tert-butyloxycarbony (Vedi Figura 2) fase solida del peptide sintesi metodo4. La sintesi del monomero PNA con Boc come l'ammina protezione gruppo è comoda come il gruppo di Boc è stericamente meno ingombrante rispetto al fluorenylmethyloxycarbonyl (Fmoc) ammina proteggere il gruppo, che può essere utile durante il monomero PNA accoppiamento supporto solido. Il gruppo di Boc è acido-labile e può essere facilmente rimosso il supporto solido di 20-50% di acido trifluoroacetico (TFA) in diclorometano (DCM) durante la sintesi PNA. Un sintetizzatore di peptidi automatizzato può essere impiegato per sintetizzare gli oligomeri PNA; Tuttavia, eccesso di 3-5 volte di monomero PNA è necessaria per un sintetizzatore di peptidi automatizzato. Sintesi manuale richiede significativamente meno monomero PNA (2-3 volte in eccesso), con ogni accoppiamento facilmente monitorato dal Kaiser prova43. Inoltre, molti sintetizzatori automatizzati non sono compatibili con la sintesi di strategia Boc dovute all'uso di corrosivo TFA durante la fase di rimozione di Boc.
Gli oligomeri PNA possono essere purificati mediante cromatografia liquida ad alte prestazioni a fase inversa (RP-HPLC) seguita dalla caratterizzazione di peso molecolare mediante MALDI-TOF (figure 3 e 4)30, 31. impieghiamo pagina non-denaturante per monitorare la formazione triplex, causa del fatto che libero duplex RNA costruisce e PNA associato triplexes spesso Mostra di migrazione diverse tariffe (Figura 5)30,31 . Nessuna etichettatura è necessaria se efficiente post-colorazione può essere raggiunto per entrambe le RNA duplex e PNA· Bande di triplex2 RNA. Una relativamente piccola quantità di campione è necessario per non-denaturante pagina esperimenti. Tuttavia, i buffer di caricamento (incubazione) e i buffer in esecuzione (pH 8.3) non possono essere lo stesso, con conseguente le misurazioni si limita a triplexes cineticamente stabile, perché un pH relativamente alto 8.3 significativamente potrebbe destabilizzare un triplex.
2-Aminopurine è un isomero di adenina (6-aminopurine); l'intensità di fluorescenza 2-aminopurine (con un picco di emissione a circa 370 nm) è sensibile ai cambiamenti di ambiente strutturale locale ed è adatto per il monitoraggio della formazione triplex con il residuo di 2-aminopurine incorporato vicino l'ANP associazione sito ( Figura 6) 31. a differenza di molti altri coloranti che mostrano l'emissione di fluorescenza nella gamma visibile, 2-aminopurine-labeled RNA possa essere esposti alla luce della stanza senza foto candeggio. A differenza di esperimento di pagina in cui è spesso necessario un buffer in esecuzione del pH 8.3, titolazione fluorescenza 2-aminopurine basata permette la misura di associazione in un'unica soluzione a pH specificato e così può consentire la misurazione e la rilevazione di relativamente debole e Associazione cineticamente instabile all'equilibrio.
Esperimenti di fusione termici UV-assorbanza-rilevato consentono la misura della stabilità termica del duplex (Figura 7)31 e triPlex30,32,44,45. A seconda della lunghezza e sequenza di composizione, lo scioglimento dei triplexes può o non può mostrare una chiara transizione. Parametri termodinamici possono essere ottenuti se le curve di riscaldamento e raffreddamento si sovrappongono. Parametri termodinamici accurati possono essere ottenuti isotermica di titolazione calorimetria (ITC)32; Tuttavia, relativamente grandi quantità di campioni sono generalmente richiesti per ITC.
Protocollo
1. manuale fase solida del Peptide sintesi di PNAs utilizzando Boc chimica
Nota: per il successo e la facilità della sintesi di oligomeri PNA desiderata, tutti i solventi e reagenti devono essere anidro. Aggiungere i setacci molecolari appropriati (4A, pellet del diametro di 1-2 mm) e occasionalmente spurgo gas azoto secco nelle bottiglie. Per la sintesi di monomeri PNA modificati, i protocolli riportati nei rispettivi riferimenti 30 , 31 possono essere utilizzati. Monomeri PNA non modificati possono essere acquistati da fonti commerciali. In tutti i passaggi di lavaggio, la giusta quantità di solvente è aggiunto alla resina, formando un impasto, prima è stata drenata off.
- Caricamento del primo monomero e tappatura le ammine primarie libera in eccesso sulla resina
- pesare 30 mg di cloridrato di 4-methylbenzhydrylamine (MBHA·HCl) polistirolo resina caricamento valore 0.7-1.4 (commercialmente disponibili; mmol/g; maglie di 100-200) e il trasferimento a un 5 mL vaso di reazione del peptide di fase solida dotato di un tappo di vetro e rubinetto.
- Ammollo la resina in una quantità appropriata di DCM per 1 h, permettendo la resina a gonfiare ed esporre le ammine (sale di HCl).
Nota: Perle di resina dovrebbero sempre essere completamente sommerso in solventi durante la sintesi. - Defluire il DCM applicando un delicato flusso di gas di azoto secco sopra la parte superiore del pallone. Aggiungere 1 mL di 50% (v/v) N, N-diisopropylethylamine (DIPEA) in DCM e lasciarlo per 15 min. Questo neutralizza il sale HCl attaccato alle ammine gratuite sulla resina.
- Ripetere passo 1.1.3. Nel frattempo, pesare 6 µmol di monomero e 6 µmol di esafluorofosfato (benzotriazol-1-yl-ossi) tripyrrolidinophosphonium (PyBOP) e trasferire in una provetta da 1,5 mL. Aggiungere 200 µ l di dimetilformammide (DMF) e 12 µmol di DIPEA. Vortex la soluzione di accoppiamento per un minimo di 3-5
Nota: Il valore di caricamento desiderato utilizzato è 0,2 mmol/g. Il primo monomero aggiunto è il monomero del C-terminale della sequenza desiderata di PNA. Il valore di caricamento del primo aminoacido può essere calcolato con il metodo acido picrico 46.
Soluzione - defluire il DIPEA da DCM. Lavare la resina con DCM (x 3), seguita da DMF (x 3) e chiudere il rubinetto. Aggiungere la soluzione di accoppiamento preparati alla resina e agitare delicatamente. Spingere la resina lungo le pareti interne del recipiente la soluzione di accoppiamento con l'uso di una spatola di acciaio inox pulito. Fissare il tappo di vetro e fissare la nave in un agitatore incubatore per 3 ore a 40 ° C.
Nota: In alternativa, il recipiente di reazione può essere mantenuto costante a temperatura ambiente per 6-8 h per consentire il completamento del peptide reazione di accoppiamento. - Preparare la soluzione di tappatura con 240 µmol di anidride acetica e 360 µmol di DIPEA in 200 µ l di DCM. Sgocciolare la soluzione di accoppiamento e lavare la resina con DMF (x 3) e DCM (x 3). Aggiungere nella soluzione di tappatura e lasciare la nave per 30 min, occasionalmente scuotendo delicatamente il vaso. Tappatura maschera i gruppi di ammina primaria libera in eccesso le resine di acetilazione.
- Ripetere passo 1.1.6. Asciugare la soluzione di tappatura e lavare la resina con DCM (x 3).
- Rimuovere una piccola aliquota di perle di resina, utilizzando un sottile tubo capillare e inserirli in un flaconcino di vetro piccolo 1,5 mL. Eseguire il test di Kaiser 43. Aggiungere 15 µ l di ciascuno del Kaiser soluzioni di test nel bicchiere fiala e utilizzando una pistola di calore di calore. Osservare il colore delle perline dopo il riscaldamento. Il colore delle perline dovrebbe rimanere invariato, che indica la mancanza di gruppi amminici liberi sulla resina.
Nota: Il kit di test Kaiser è commercialmente disponibile o può essere preparato secondo il protocollo segnalato. Soluzione a: ninidrina in etanolo; soluzione b: fenolo in etanolo; soluzione c: cianuro di potassio (KCN) in piridina.
Attenzione: KCN è altamente tossico; indossare abbigliamento protettivo adeguato e riscaldamento deve essere eseguita in una cappa ben ventilata, in assenza di solvente infiammabile o reagenti. - Ripetere il punto 1.1.6 se visualizzare le perle di resina colore blu blu o svenire.
- Rimozione del gruppo di protezione dell'ammina N-terminale
- scolare i solventi dal recipiente di reazione e aggiungere una soluzione di 50% (v/v) di acido trifluoroacetico (TFA) in DCM, assicurando che le resine sono completamente sommerse. Lasciare il vaso per 15 minuti, scuotendo ogni tanto per facilitare la deprotezione dei gruppi amminici. Ripetere 2 cicli più.
Attenzione: TFA è altamente corrosivo. Abbigliamento protettivo adeguato deve essere indossato quando si maneggia. - Lavare la resina con DCM (x 3), DMF (x 3) e DCM (x 3). Aggiungere una soluzione di 5% DIPEA in DCM. Lasciare il vaso per 15 min. Questo passo permette di attivare le ammine gratis neutralizzando gli anioni di contatore TFA. Ripetere un' volta.
- Scovare il DIPEA dalla soluzione DCM. Lavare la resina con DCM (x 3). Eseguire il test di Kaiser (punto 1.1.8).
Nota: Successo deprotezione dei gruppi amminici resa di sbiadimento blu di perline. Una volta eseguita deprotezione, accoppiamento successivo di monomeri di accoppiamento del peptide può essere effettuata.
- scolare i solventi dal recipiente di reazione e aggiungere una soluzione di 50% (v/v) di acido trifluoroacetico (TFA) in DCM, assicurando che le resine sono completamente sommerse. Lasciare il vaso per 15 minuti, scuotendo ogni tanto per facilitare la deprotezione dei gruppi amminici. Ripetere 2 cicli più.
- Di accoppiamento di monomeri successive
- pesare 18 µmol di voluto monomero (per il monomero di Boc-PNA-Q-OH, 13,2 mg di monomero viene pesato) e 18 µmol di PyBOP in una provetta da 1,5 mL. Aggiungere 200 µ l di DMF e 36 µmol di DIPEA nella provetta da 1,5 mL. Vortice fino a quando tutti i composti solidi disciolti.
- Lavare la resina con DMF (x 3). Aggiungere la soluzione di accoppiamento nel recipiente di reazione ed agitare delicatamente. Spingere la resina lungo le pareti interne del recipiente la soluzione di accoppiamento con l'uso di una spatola di acciaio inox pulito. Fissare il tappo di vetro e fissare la nave in un agitatore incubatore per 3 ore a 40 ° C.
- Scarico fuori la soluzione di accoppiamento e lavare la resina con DMF (x 3) e DCM (x 3). Eseguire il passaggio 1.1.8. Se il colore delle perline rimane invariato, ripetere i passaggi 1.2.1-1.3.3 finché non viene completata la sequenza desiderata di PNA. Se sbiadimento blu di perline è osservato dopo l'accoppiamento di un monomero, ripetere i passaggi 1.3.1-1.3.3. Se lo scolorimento persiste, eseguire nuovamente la tappatura (punto 1.1.6).
Nota: Estesa accoppiamento tempo (3-12 h) e/o uso di equivalente in eccesso del monomero e reagenti di accoppiamento sono consigliati se si verificano problemi con l'accoppiamento. - Una volta completata la sequenza desiderata di PNA, lavare la resina con DMF (x 3) e DCM (x 3). Asciugare completamente la resina applicando un flusso continuo di gas azoto secco per 15 min. Questa resina secca può essere diviso e utilizzata per collegare la lisina o un tag fluorescente ad esempio cianina 3 (Cy3) e carboxyfluorescein al N-terminale dell'ANP.
- Allegato del tag lisina o fluorescenti (Cy3, Cy5 o carboxyfluorescein) a N-terminale di PNA
- pesare 10 mg di resina, pre-caricata con la sequenza desiderata di PNA. Trasferire in un recipiente di reazione di 5 mL. Immergere la resina in DCM per 1 h.
- Per il fissaggio di lisina, eseguire passaggi 1.2.1-1.3.3, sostituendo il monomero con entrambi Boc-Lys (Z) - OH o Fmoc-Lys (Boc) - OH.
- Per il fissaggio di carboxyfluorescein, eseguire passaggi 1.2.1-1.3.3, con 10 volte l'eccesso di 5 6-carboxyfluorescein come il monomero, e N, N '-diisopropylcarbodiimide (DIPC) e parola (HOBt) come i reagenti di accoppiamento in DMF. Lasciare la reazione di accoppiamento durante la notte nello shaker incubatore a 40 ° C.
Nota: Come carboxyfluorescein è sensibile alla luce, il recipiente di reazione dovrebbe essere coperto con carta stagnola. - Per il fissaggio del colorante Cy3 o Cy5, etichetta dal metodo click chemistry, eseguire passaggi 1.2.1-1.3.3, sostituendo il monomero con N-Boc-2-propargyl-L-glycine. Questo functionalizes N-terminale dell'ANP con un gruppo di alchini. Eseguire la reazione di rame-catalizzata di clic per fissare il Cy3 contenenti azide o tintura fluorescente Cy5 12 , 47 , 48 , 49
- Fenditura PNA da supporto solido, purificazione e caratterizzazione
Nota: se qualsiasi gruppo amminico è protetto con la protezione gruppo Fmoc, prima deprotect il gruppo amminico trattando con 20% piperdine in DMF soluzione per 15 minuti (2 cicli). Lavare la resina accuratamente con DMF (x 3) seguita da DCM (x 3). Asciugare completamente la resina applicando un flusso continuo di gas azoto secco per 15 min.- Trasferire 5 mg di resina secca in una fialetta. Aggiungere 10 µ l di thioanisole e 4 µ l di 1,2-etanditiolo, assicurando che la resina è immersa nei reagenti. Lasciare il tubo a temperatura ambiente per 5 min.
Nota: Questi reagenti fungono da spazzini, che trappola specie cationiche reattive che si formano durante la rimozione della protezione dei gruppi in ANP. Spazzini appropriati possono essere scelto basato sulla catena laterale gruppi protettori. - Aggiungere 100 µ l del TFA nella provetta contenente la resina e spazzini. Delicatamente la miscela di vortice e soggetto a breve centrifugazione. Lasciare il tubo a temperatura ambiente per 10 min.
Attenzione: TFA è altamente corrosivo. Abbigliamento protettivo adeguato deve essere indossato quando si maneggia. - Attentamente aggiungere 20 µ l di Acido trifluorometansolfonico (TFMSA) al tubo. Prima di sottoporre a breve centrifugazione a temperatura ambiente, agitare delicatamente la miscela di reazione. Lasciare il tubo costante a temperatura ambiente per 2 h.
Attenzione: TFMSA è altamente corrosivo. Abbigliamento protettivo adeguato deve essere indossato quando si maneggia. - Filtro fuori il clivaggio cocktail in un pallone a sfondo sferico da 5 mL (RBF) con l'uso di un vetro Pasteur pipetta dotato di cotone. Utilizzare una piccola quantità di TFA per lavare la resina.
- Eliminare il gas azoto secco al filtrato raccolto fino a quando tutti i solventi volatili sono evaporati. Aggiungere 1 mL di etere etilico freddo in RBF.
Nota: L'etere etilico causerà l'ANP a precipitare.- Risciacquo RBF con il dietil etere più volte prima di trasferire la soluzione nuvolosa in una provetta da 1,5 mL. Sottoporre il tubo a centrifugazione per consentire il precipitato PNA a stabilirsi. Decantare i solventi e aggiungere 300-500 µ l di acqua in autoclave il precipitato. Vortice accuratamente per sciogliere il PNA.
- Purificare il campione PNA greggio via RP-HPLC usando water-acetonitrile-0.1% TFA come fase mobile. Raccogliere le frazioni corrispondenti, evaporare tutti i solventi utilizzando un concentratore a vuoto prima di ri-dissoluzione il PNA purificato in acqua autoclavato.
- Caratterizzano il PNA purificato tramite analisi MALDI-TOF con l'uso di acido α-ciano-4-idrossicinnamico (CHCA) come matrice di cristallizzazione del campione.
- Misurare l'assorbanza UV (260 nm) dell'ANP a 65 ° C. calcolare la concentrazione di PNA con l'equazione:

Nota: qui c è la concentrazione, A è la lettura di assorbanza ottenuta, ε è il coefficiente di estinzione della sequenza del RNA e l è la lunghezza del percorso ottico della provetta (1 cm). il coefficiente di estinzione della sequenza PNA è la somma del coefficiente di estinzione dei singoli monomeri 50. I coefficienti di estinzione di adenina, citosina, guanina e timina sono 15,4, 7.3, 11,7 e 8,8 mL/µmol·cm, rispettivamente. Il coefficiente di estinzione dei monomeri L e Q utilizzato è presupposto per essere la stessa di quella della citosina (C) base.
- Trasferire 5 mg di resina secca in una fialetta. Aggiungere 10 µ l di thioanisole e 4 µ l di 1,2-etanditiolo, assicurando che la resina è immersa nei reagenti. Lasciare il tubo a temperatura ambiente per 5 min.
2. PAGINA non-denaturante
- preparazione di soluzioni tampone richiesto
- preparare Incubation buffer (10 mL) utilizzando 116,88 mg NaCl (200 mM), 50 µ l di acido etilendiamminotetracetico 100mm (ed) (0,5 mM), 200 µ l di brodo di 1 M HEPES (20 mM), 9,75 m H 2 O; regolare di tampone a pH 7,5.
- Preparare 1 x buffer in esecuzione di Tris-Borato-EDTA (TBE) (1 L) utilizzando 100 mL 10 x Tris-Borato-EDTA a pH 8.3 e 900 mL H 2 O.
- Soluzione di preparare 10% ammonio persolfato (APS) (300 µ l) utilizzando 30mg persolfato dell'ammonio e 300 µ l H 2 O.
- Preparazione del gel di poliacrilammide del 12%
- pulire il pettine ben formatura lastre di colata di vetro e distanziali con etanolo; il pettine ben formatura e distanziali sono 1 mm di spessore. Impostare l'assembly di gel e sigillare con nastro di tenuta gel.
- Per un gel di poliacrilammide di 22 cm x 16,5 cm x 1 mm dimensione, 50 mL di soluzione di gel è adeguata. Pesare 5,7 g di acrilammide e 0,3 g di N, N '-methylenebisacrylamide (19:1) e trasferimento in una centrifuga di 50ml tube.
Attenzione: L'acrilammide è cancerogeno. Evitare di respirare polveri fumi da acrilammide e garantire che abbigliamento protettivo adeguato è indossato quando si maneggia. - Sciogliere composti solidi in tampone di corsa TBE 1x 50 mL 1 inserendo il tubo da centrifuga in un bagnomaria a 50 ° C per 15 minuti o fino a quando tutti i composti solidi si sono dissolti. Svolgere la centrifugazione (3.000 giri/min, 5 min, 25 ° C) per rimuovere tutte le bolle d'aria all'interno della soluzione. Lasciare agire la soluzione raffreddare a temperatura ambiente.
- Aggiungere 250 µ l di soluzione di 10% APS e 50 µ l di tetrametiletilendiammina (TEMED) nella soluzione di gel e mescolare delicatamente con una spatola. Versare immediatamente la soluzione tra le lastre di vetro, facendo attenzione a non introdurre eventuali bolle d'aria. Inserire il pettine ben formatura e lasciare il programma di installazione di gel a temperatura ambiente per almeno 60 minuti consentire la polimerizzazione si verifichi, prima di conservare a 4 ° C fino all'uso.
- Preparazione dei campioni
- rimuovere la quantità necessaria di hairpin RNA (1 µM) dal magazzino principale in una provetta pulita 1,5 mL. Asciugare la soluzione di RNA utilizzando un concentratore a vuoto.
Nota: Ogni campione contiene 1 µM di RNA in 20 µ l di tampone di incubazione. In genere, 13 campioni di RNA sono preparati per sperimentare una pagina. il RNA per tutti i campioni possa essere preparato insieme in un singolo tubo. - Rimuovere i volumi richiesti di PNA mirato (con la concentrazione finale di fino a 50 µM) dal magazzino principale e trasferimento in tubi separati 1.5 mL. Far evaporare l'acqua delle soluzioni PNA utilizzando un concentratore a vuoto.
- Aggiungere 260 µ l di tampone di incubazione in 1,5 mL provetta contenente secchi RNA e mescolare accuratamente per garantire che tutto il RNA è dissolute. Sottoporre il RNA per raffreddamento a scatto: posto il tubo in un blocco di calore (preriscaldato a 95 ° C) per 5 min, immediatamente trasferire in un bagno di ghiaccio e lasciare per 10 min.
- Aggiungere 20 µ l di RNA in ciascuno di 1,5 mL provette contenenti asciugato PNA e mescolare accuratamente. Eseguire ricottura: porre la provetta contenente RNA e PNA la miscela in un blocco di calore (preriscaldato a 65 ° C) per 10 min. Disabilita l'alimentazione del blocco di calore e lasciare raffreddare i campioni lentamente a temperatura ambiente. Incubare i campioni a 4 ° C durante la notte.
- rimuovere la quantità necessaria di hairpin RNA (1 µM) dal magazzino principale in una provetta pulita 1,5 mL. Asciugare la soluzione di RNA utilizzando un concentratore a vuoto.
- In esecuzione ed elaborazione di gel
- rimuovere il nastro di tenuta di gel sul lato inferiore della lastra di vetro. Montare e fissare la piastra di vetro sul supporto verticale gel utilizzando fascette in plastica.
Nota: Il gel in esecuzione viene effettuato in una cella frigorifera a 4 ° c circa. Tutte le attrezzature, i campioni e i buffer sono raffreddati a 4 ° C prima di eseguire il gel. - Riempire il serbatoio tampone inferiore con 1 x TBE buffer in esecuzione fino a quando la piastra è sommerso in circa 1-2cm di tampone di corsa. Riempire il serbatoio tampone superiore con tampone di corsa fino a quando il livello del buffer supera la parte superiore del gel di 1-2 cm. lentamente e rimuovere delicatamente il pettine ben formatura, permettendo il buffer in esecuzione riempire i pozzetti.
- Collegare il supporto di gel a un alimentatore. Pre-corsa il gel per almeno 30 min con una tensione costante di 250 V, che è ottimizzato per un 22 cm x 16,5 cm x 1 gel di mm.
- Nel frattempo, aggiungere 4 µ l (20% del volume del campione) di soluzione al 35% glicerolo a ciascuno dei campioni e mescolare delicatamente. Una volta completata pre-l'esecuzione, caricare 20 µ l di ciascun campione (tra cui un RNA da solo campione più campioni contenenti miscela di RNA e PNA) attentamente in fondo il bene usando una micropipetta e gel caricamento suggerimenti, facendo attenzione a non introdurre eventuali bolle d'aria. Esegua il gel a una tensione costante di 250 V, simile a pre-run, per 5 h.
- Interrompere l'alimentazione elettrica dopo 5 h e rimuovere la lastra di vetro dal supporto. Rimuovere il nastro di tenuta di gel rimanente e smontare la lastra di vetro. Rimuovere delicatamente e immergere il gel in un contenitore riempito con 350 mL di acqua deionizzata. Con attenzione aggiungere 35 µ l di bromuro di etidio (10 mg/mL) e posizionare il recipiente su un agitatore di piattaforma (bassa velocità) per 30 min.
Attenzione: Il bromuro di etidio è un agente mutageno. Abbigliamento protettivo adeguato deve essere indossato quando si maneggia. - Eliminare la soluzione di bromuro di etidio in un contenitore per rifiuti designato. Risciacquare il gel con 1,5-2 L di acqua distillata. Scansione il gel utilizzando un imager (Vedi Tabella materiali) con un laser verde di 532 nm e l'emissione del filtro insieme a 610 nm.
- rimuovere il nastro di tenuta di gel sul lato inferiore della lastra di vetro. Montare e fissare la piastra di vetro sul supporto verticale gel utilizzando fascette in plastica.
- Gel analisi
- quantificare l'intensità di banda gel utilizzando un software gratuito, GelQuant.NET (http://www.biochemlabsolutions.com/GelQuantNET.html). Normalizzare le intensità di banda secondo:

Nota: qui ho max duplex è l'intensità di banda dei tornanti RNA da solo senza l'aggiunta di PNA e Triplex max è l'intensità di banda triplex con la più alta concentrazione di PNA aggiunto.- Calcolare la frazione di formazione triplex secondo:

- Calcolare la frazione di formazione triplex secondo:
- trama la frazione della formazione triplex (Y) contro la concentrazione di PNA aggiunti ( ΜM). Adattare i dati all'equazione per ottenere la costante di dissociazione (K d):
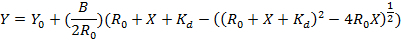
Nota: qui R 0 è la concentrazione di RNA hairpin (1 µM). Qui Y 0 e B sono l'iniziale e la massima variazione della frazione triplex, rispettivamente. Y è la frazione di triplex a varia concentrazione di PNA. X è la concentrazione totale di PNA e K d è la costante di dissociazione.
- quantificare l'intensità di banda gel utilizzando un software gratuito, GelQuant.NET (http://www.biochemlabsolutions.com/GelQuantNET.html). Normalizzare le intensità di banda secondo:
3. analisi di associazione di fluorescenza 2-Aminopurine
- preparazione dei campioni (contenente dsRNA)
- rimuovere la quantità necessaria di 2-aminopurine (2AP) etichettata dsRNA (1 µM per ciascuno filo) dal magazzino principale in una provetta pulita 1,5 mL. Evaporare l'acqua nelle soluzioni RNA utilizzando un concentratore a vuoto.
Nota: Ogni campione contiene 1 µM di RNA in 75 µ l di tampone di incubazione. In genere, sono preparati 13 campioni di RNA. RNA per tutti i campioni possa essere preparato insieme in un singolo tubo. - Rimuovere i volumi richiesti dell'ANP mirati per le varie concentrazioni dal magazzino principale e trasferimento in tubi separati 1.5 mL. Far evaporare l'acqua delle soluzioni PNA utilizzando un concentratore a vuoto.
- Aggiungere 975 µ l di tampone di incubazione in 1,5 mL provetta contenente secchi RNA e mescolare accuratamente per garantire che tutto il RNA è dissolto. Centrifugare la soluzione di RNA e sottoporre a ricottura: porre la provetta in un blocco di calore (preriscaldato a 95 ° C) per 10 min. Disabilita l'alimentazione del blocco di calore e lasciare raffreddare i campioni lentamente a temperatura ambiente.
- Aggiungere 75 µ l di RNA in ciascuno di 1,5 mL provette contenenti asciugato PNA e mescolare accuratamente. Lasciare i campioni a temperatura ambiente per almeno 1 h. Incubare i campioni a 4 ° C durante la notte.
- rimuovere la quantità necessaria di 2-aminopurine (2AP) etichettata dsRNA (1 µM per ciascuno filo) dal magazzino principale in una provetta pulita 1,5 mL. Evaporare l'acqua nelle soluzioni RNA utilizzando un concentratore a vuoto.
- Preparazione dei campioni (contenente ssRNA)
- rimuovere la quantità necessaria di ssRNA 2AP-etichettati (1 µM) dal magazzino principale nelle provette pulite 1,5 mL. Estrarre i volumi richiesti di PNA mirato per le varie concentrazioni dal magazzino principale e trasferimento nei tubi rispettivi 1,5 mL che contengono il ssRNA. Asciugare il composto di RNA e PNA utilizzando un concentratore a vuoto.
- Aggiungere 75 µ l di incubazione nel buffer in ciascuna delle provette da 1,5 mL e mescolare accuratamente. Sottoporre la miscela a ricottura: porre la provetta in un blocco di calore (preriscaldato a 95 ° C) per 10 min. Turn off il potere e lasciare che i campioni raffreddino lentamente a temperatura ambiente. Incubare i campioni a 4 ° C durante la notte.
- Misura e analisi
- utilizzare uno spettrofotometro di fluorescenza per misurare le emissioni sopra una gamma di lunghezza d'onda di 330-550 nm. Utilizzare una lunghezza d'onda di eccitazione di 303 nm.
Nota: I campioni sono misurati a temperatura ambiente e ogni campione viene misurata 3 volte, in cui viene presa la media.
Buffer di - trasferire 70 µ l di incubazione nella cuvetta 1 centimetro quadrato. Avviare la misurazione.
- Rimuovere il buffer dalla cuvette. Sciacquare la provetta con acqua distillata e spurgo gas di azoto ad asciugare. Ripetere per tutti i campioni. Sottrarre le misure tampone da tutti i campioni.
- Plot l'intensità di fluorescenza (a.u.) contro la lunghezza d'onda (nm). Registrare l'intensità della fluorescenza a 370 nm per tutti i campioni. Tracciare l'intensità della fluorescenza a 370 nm (a.u.) rispetto alla corrispondente concentrazione dell'ANP aggiunto (µM).
- Adattare i dati per l'equazione dal passaggio 2.5.2 per ottenere la costante di dissociazione (K d): Y = Y 0 + (B / (2R 0)) (R 0 + X + K d-((R 0 + X + K d) 2 -4R 0 X) 1/2), dove R 0 è la concentrazione di dsRNA 2AP-etichettati (1 µM).
Nota: Qui Y 0 e B sono il cambiamento iniziale e massimo della intensità di fluorescenza a 370 nm, rispettivamente. Y è l'intensità di fluorescenza a 370 nm a varia concentrazione di PNA. X è la concentrazione totale di PNA e K d è la costante di dissociazione.
- utilizzare uno spettrofotometro di fluorescenza per misurare le emissioni sopra una gamma di lunghezza d'onda di 330-550 nm. Utilizzare una lunghezza d'onda di eccitazione di 303 nm.
- preparazione dei campioni
Nota: misurare l'assorbanza UV (260 nm) del RNA a 95 ° C (per assicurarsi che le strutture secondarie del RNA sono interrotti). Calcolare la concentrazione del RNA con l'equazione:
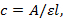
dove c è la concentrazione, A è la lettura di assorbanza ottenuta, ε è il coefficiente di estinzione della sequenza di RNA, e l è la lunghezza del percorso ottico della provetta (1 cm). Il coefficiente di estinzione del RNA viene calcolato in base ad un modello più simile utilizzando MeltWin 51 , 52. Il pacchetto del programma può essere fornito su richiesta. Tubi- Rimuovi la quantità necessaria di ssRNA (5 µM) dal magazzino principale in pulito 1,5 mL. Rimuovere i necessari volumi di PNA mirato (5 µM) dal magazzino principale e trasferimento nei tubi rispettivi 1,5 mL che contengono il ssRNA. Asciugare il composto di RNA e PNA utilizzando un concentratore a vuoto.
- Aggiungere 130 µ l di incubazione nel buffer in ciascuna delle provette da 1,5 mL e mescolare accuratamente. Sottoporre la miscela a ricottura: porre la provetta in un blocco di calore (preriscaldato a 95 ° C) per 10 min. Turn off il potere e lasciare che i campioni raffreddino lentamente a temperatura ambiente. Incubare i campioni a 4 ° C durante la notte.
- Misura e analisi
- utilizzare uno spettrofotometro UV-Vis per misurare l'assorbanza a 260 nm utilizzando una provetta 8-microcell con una lunghezza di percorso di 1 cm. misurare i campioni ' assorbanza all'aumentare della temperatura da 15 a 95 ° C, seguita da diminuzione della temperatura da 95 a 15 ° C a una velocità di rampa di 0,5 ° C/min
- Trasferire 130 µ l di campione in ciascuno del pozzo, facendo in modo che uno contiene ben il tampone di incubazione. Avviare la misurazione. Ripetere come richiesto.
- Normalizzare i valori di assorbanza con l'alta temperatura normalizzata all'unità di lettura e tracciare l'assorbanza normalizzata lettura contro la temperatura (° C). Tracciare la derivata prima delle curve. Ottenere le temperature di fusione inserendo le curve derivate prime a una funzione gaussiana.
4. UV-assorbanza-rilevato gli esperimenti di fusione termica
Risultati
HPLC di inverso-fase permette la purificazione degli oligomeri PNA. Possiamo ottenere puri oligomeri PNA con due turni di purificazione HPLC (Figura 3). L'identità dei PNA può essere confermata dalle analisi MALDI-TOF (Figura 4).
PAGINA non-denaturante è una tecnica facile ed informativa per caratterizzare l'affinità di legame e la specificità degli oligomeri PNA. In genere utilizziamo più RNA forcine o duplex con mutazioni di singola coppia di basi per caratterizzare le proprietà di associazione (Figura 5). I dati della pagina non-denaturante illustrati nella Figura 5 indicano chiaramente che il Q - e L-modificato PNA può riconoscere una regione di dsRNA con una C-G la coppia (Figura 5B, pannello inferiore) ma non l'uno senza un paio di C-G (Figura 5B, top pannello). Questo riconoscimento specifico e migliorato è attraverso il T· A-U, L · G-C ed è C-G PNA· Formazione di RNA2 triple base (Figura 1A, C, D). PNAs vari con le mutazioni di singole o multipli può essere utilizzato anche per dimostrare le proprietà di associazione migliorata di un PNA modificato. Abbiamo dimostrato che l'aggiunta di 2 mM Mg2 + in tampone di incubazione non pregiudica l'associazione significativamente31.
Abbiamo dimostrato di titolazione fluorescenza 2-aminopurine che un PNA Q - e L-modificato si lega a una regione di dsRNA mirati (Figura 6A, 6C, 6D) ma non ssRNA (Figura 6B, 6E, 6F). PNA P3 associa il dsRNA 2-aminopurine-etichettati con un valore did Kdi 0,8 ± 0,1 µM. L'intensità della fluorescenza a 370 nm per il 2-aminopurine-labeled ssRNA rimane relativamente costante con varia concentrazione P3, che indica la mancanza di associazione di PNA P3 per il ssRNA.
PNAs contenente Visualizza residui (P2 e P3) Q no thermal transizioni (Figura 7), non suggerendo vincolante per il ssRNA di fusione. Ciò è dovuto lo scontro sterico presente a Watson-Crick come Q-G la coppia. Rispetto al non modificato PNA P1, PNAs P4 e P5 contenente modificato L residui ma senza residui di Q, Visualizza diminuito temperature di fusione per i duplex RNA-PNA corrispondente a causa dello scontro sterico presente a Watson-Crick come L-G la coppia. I dati di fusione termici UV-assorbanza-rilevato sono coerenti con i dati di titolazione fluorescenza 2-aminopurine, che mostrano anche che un PNA contenenti residui di Q e L non associare a ssRNA sensibilmente (Figura 6B, 6E, 6F). Incorporando una base di Q è più destabilizzante di una base di L, una base di Q ha un scontro sterico più significativo nella formazione di una coppia di Watson-Crick-come Q-G (Figura 1F) rispetto ad una coppia di Watson-Crick-come L-G (Figura 1E ).
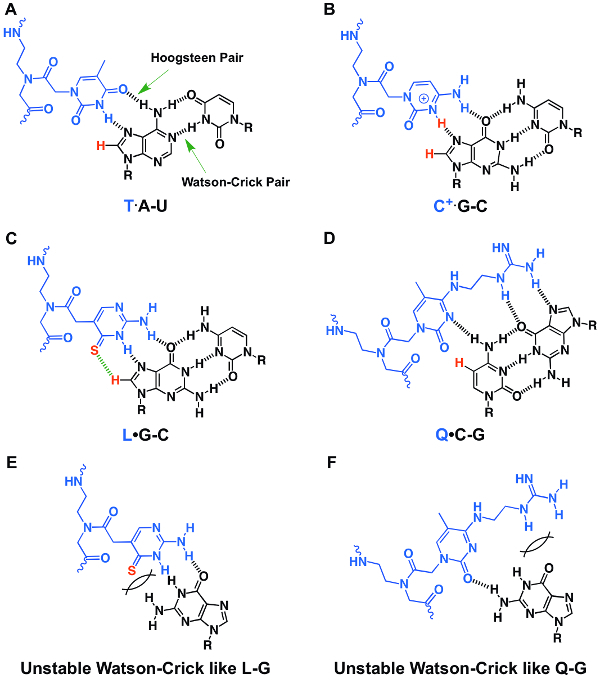
Figura 1 : Strutture chimiche delle strutture di base triple e instabile base stabile coppia. (A-D) Major-groove PNA· Triple base di RNA2 di T· A-U (A), C+· G-C (B), l · G-C (C) ed è C-G (D). (E, F) Watson-Crick instabile come coppie di basi di PNA-RNA di L-G (E) e Q-G (F). La lettera R rappresenta la spina dorsale di zucchero-fosfato del RNA. Legami dell'idrogeno sono indicate da linee nere tratteggiate. La figura è riprodotto dal riferimento31. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2 : Struttura chimica dei monomeri PNA. Vengono visualizzati quattro monomeri PNA (T, C, L e Q). Clicca qui per visualizzare una versione più grande di questa figura.
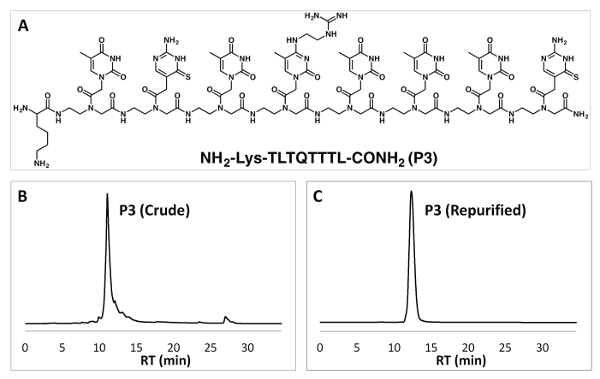
Figura 3 : Struttura chimica di un oligomero PNA e purificazione mediante RP-HPLC. (A) struttura chimica della sequenza PNA P3. (B, C) Dati di RP-HPLC di greggio PNA P3 (B) e ri-purificato PNA P3 (C). Clicca qui per visualizzare una versione più grande di questa figura.
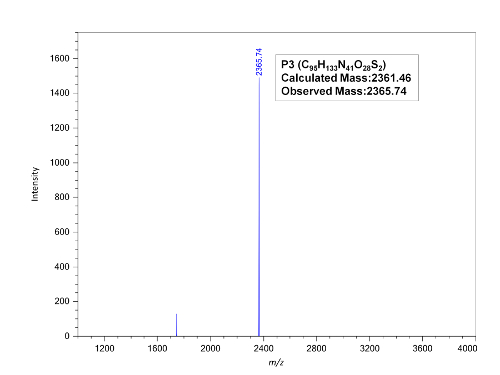
Figura 4 : Spettro MALDI-TOF di purificato PNA P3. Clicca qui per visualizzare una versione più grande di questa figura.
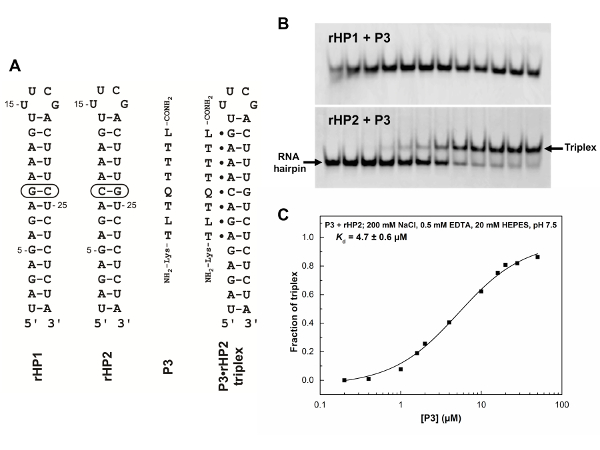
Figura 5 : Hairpin RNA e sequenze di PNA e caratterizzazione di associazione di non-denaturante pagina. Forcine per capelli (A), RNA (rHP1 e rHP2), PNA P3 e un PNA· RNA2 triplex formate tra PNA P3 e rHP2. (B) Non-denaturante pagina (12%) risultati per rHP1 e rHP2 associazione a PNA P3. Il tampone di incubazione è di 200 mM NaCl, 0,5 mM EDTA, 20 mM HEPES, pH 7.5. Le forcine di RNA caricate (rHP1 e rHP2) sono a 1 µM a 20 µ l. Le concentrazioni di PNA in corsie da sinistra a destra sono 0, 0,2, 0,4, 1, 1.6, 2, 4, 10, 16, 20, 28, e 50 µM. PNA P3 non associare a rHP1 (pannello superiore) ma si lega al rHP2 (pannello inferiore). (C) Kd determinazione per l'associazione di P3 a rHP2. La frazione di formazione triplex (Y) è stata tracciata contro la concentrazione di PNA. La figura è adattata da riferimento31. Clicca qui per visualizzare una versione più grande di questa figura.
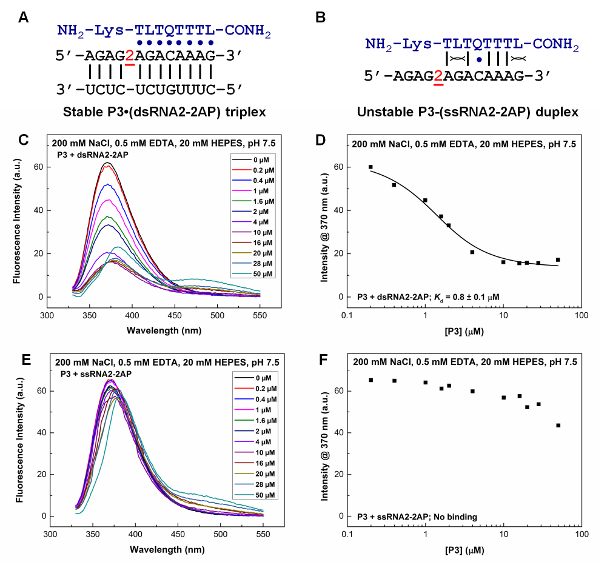
Figura 6 : Studio di titolazione fluorescenza di PNA P3 associazione2-aminopurine-labeled RNAs. Il residuo di 2-aminopurine è designato come '2' nella sequenza di RNA. Il tampone di incubazione è di 200 mM NaCl, 0,5 mM EDTA, 20 mM HEPES, pH 7.5. (A) un PNA· RNA2 triplex formate tra P3 e un 2-aminopurine-labeled dsRNA (dsRNA2-2AP). (B), un ipotetico duplex PNA-RNA formata tra P3 e un 2-aminopurine-labeled ssRNA (ssRNA2-2AP). (C, E) Spettri di emissione di fluorescenza per il 2-aminopurine-labeled RNA duplex (1 µM) e ssRNA (1 µM), rispettivamente, con varie P3 concentrazione a pH 7.5. Il picco a circa 475 nm è dovuto l'emissione di fluorescenza debole della base L in ANP. (D, F) K d determinazione basata su appezzamenti di intensità di fluorescenza 2-aminopurine (370 nm) di RNA duplex e ssRNA, rispettivamente, contro concentrazione di PNA P3. La figura è adattata da riferimento31. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 7 : Thermal fusione risultati per Duplex RNA-PNA. Il tampone di incubazione è di 200 mM NaCl, 0,5 mM EDTA, 20mm NaH2PO4, pH 7.5. Tutti i campioni contengono 5 µM di singoli filamenti di RNA (ssRNA1) e PNA in 130 µ l. (A) Single-stranded RNA (ssRNA1), PNAs (P1, P2, P3, P4 e P5) e un ipotetico duplex PNA-RNA formata tra PNA P3 e ssRNA1 in un orientamento parallelo. Lo scontro sterico è indicato per la Watson-Crick come Q-G e coppie di L-G. (B) fusione delle curve per diversi PNAs associazione a ssRNA1. Le temperature di fusione sono indicate per le curve con transizioni di fusione. La figura è adattata da riferimento31.
Discussione
Oligomeri PNA di RNA duplex-associazione (ad es., 10-mers) sono molecole di piccole e medie e così può mostrare spostamento di mobilità elettroforetica legandosi a RNA con una dimensione paragonabile o leggermente più grande (ad es., 50-mer o più piccoli). Se un RNA è significativamente maggiore di ANP, titolazione di PNA in RNA potrebbe non funzionare a causa di uno spostamento di mobilità limitata gel. Così, il grande RNA potrebbe essere troncato per le analisi di pagina non-denaturante. Titolazione di un grande RNA in un fluoroforo PNA consente il monitoraggio della formazione triplex da un non-denaturante del gel dell'agarosi con il campione caricato nel mezzo il gel40.
Per un esperimento di titolazione di non-denaturante pagina con una costante concentrazione totale di RNA, generalmente utilizziamo una concentrazione di RNA senza etichetta di 1 µM per efficiente post colorazione del RNA gratis e bande triplex di bromuro di etidio. Una concentrazione di RNA a partire da 0,2 µM può anche essere sufficiente a seconda del costrutto di RNA31. La concentrazione del RNA senza etichetta (0,2 µM) determina che i valori did Kpossono essere accuratamente misurati devono essere circa 0,2 µM o più grande. Altri coloranti colorazione possono essere utilizzati per migliorare l'efficienza di colorazione. In alternativa, i nostri dati non pubblicati suggeriscono che Cy3 tingere-contrassegnati RNAs utilizzabile in non-denaturante pagina esperimenti per misurare la stretta associazione di eventi.
Dovuto al fatto che è solo moderatamente fluorescente 2-aminopurine, titolazione fluorescenza 2-aminopurine è anche limitato alla misura dell'associazione con valori did Kvicino o sopra 0,2 µM31. il RNA o il PNA può essere marcato con un colorante relativamente luminoso per quantificare un'associazione relativamente stretto in soluzione mediante titolazione fluorescenza, se l'associazione si traduce in cambiamenti nella fluorescenza segnali53,54, 55.
La strategia di targeting strutture di RNA di dsRNA-associazione PNAs è stata testata per un numero limitato di RNA. È probabile che la proprietà binding può variare per dsRNA con diverse composizioni di sequenze e coppia di basi. Uno può scegliere sempre il filo di ricchi di purine di un duplex per la progettazione di TFPNAs. È fondamentale capire come consecutivi è C-G triple possono influenzare la stabilità di un triplex. Studi più approfonditi dipendente dalla sequenza chiaramente sono necessari per comprendere le proprietà di associazione dipendente dalla sequenza di TFPNAs.
L'affinità di legame di TFPNAs può essere ulteriormente migliorata aumentando la lunghezza e/o modificare ulteriormente le basi e dorsali56,57 di TFPNAs. Tuttavia, un'area duplex continua spesso non può consistere di più di 10 coppie consecutive di base senza la rottura di strutture non-Watson-Crick. Uno può coniugato TFPNAs con piccole molecole per il riconoscimento delle strutture non-Watson-Crick adiacenti alle regioni di dsRNA. In linea di principio, un coniugato di TFPNA-piccola molecola dovrebbe hanno maggiore affinità e specificità rispetto a un TFPNA o un piccola molecola da solo. Tuttavia, le proprietà chimiche e fisiche del linker per la coniugazione58,59,60,61,62,63,64 deve essere ottimizzato.
Il fatto che TFPNAs possono legarsi selettivamente a dsRNA sopra ssRNAs e dsDNAs suggerisce che è possibile sviluppare TFPNAs come sonde chimiche molto utili e terapeutici potenziali ligandi attraverso la regolazione della dinamica strutturale RNA ed interazioni con proteine e metaboliti. L'assorbimento cellulare di TFPNAs può essere facilitata attraverso la coniugazione con cellula-penetrante moiety come piccole molecole, peptidi e nanoparticelle o complessazione con strutture supramolecolari come liposomi5,6 ,12,17,25,31,41,65,66. Funzionalizzazione di TFPNAs con tag di Bioimmagini come fluorofori e radioisotopi può facilitare ulteriormente la rilevazione, di imaging e di targeting delle strutture funzionali di RNA negli organismi viventi.
Divulgazioni
È stata presentata una domanda di brevetto (PAT/179/14/15/PCT) sulla base dei lavori segnalati qui.
Riconoscimenti
Questo lavoro è stato supportato dal Ministero di Singapore di formazione (MOE) Tier 1 (RGT3/13 e 15/RG42 al G.C.) e MOE Tier 2 (MOE2013-T2-2-024 e MOE2015-T2-1-028 di G.C.).
Materiali
| Name | Company | Catalog Number | Comments |
| Molecular sieves, 4A, 1-2 mm diameter pellets | Alfa Aesar | 87956 | |
| 4-Methylbenzhydrylamine hydrochloride (MBHAŸHCl) | Sigma-Aldrich | 532444 | |
| N,N-diisopropylethylamine (DIPEA) | Alfa Aesar | A11801 | |
| (Benzotriazol-1-yl-oxy)tripyrrolidinophosphonium hexafluorophosphate (PyBOP) | Alfa Aesar | B25251 | |
| Acetic anhyride | Sigma-Aldrich | 320102 | |
| Kaiser Test Kit | Sigma-Aldrich | 60017 | |
| Trifluoroacetic acid (TFA) | Alfa Aesar | L06374 | |
| Unmodified PNA monomers | ASM Research Chemicals GmbH | 5004007, 5004008, 5004009, 5004010 | |
| Boc-Lys(Z)-OH / Fmoc-Lys(Boc)-OH | Sigma-Aldrich | B8389 / 47624 | |
| Thioanisole | Alfa Aesar | A14846 | |
| 1,2-Ethanedithiol | Alfa Aesar | L12865 | |
| Trifluoromethanesulfonic acid | Alfa Aesar | A10173 | |
| LiChrosper® 100 RP-18 endcapped (5 µm) LiChroCART® 250-4 | Merck Millipore | 150838 | |
| RNA Oligos | Sigma-Aldrich | Customized | |
| α-cyano-4-hydroxycinnamic acid (CHCA) | Sigma-Aldrich | 39468 | |
| Ethylenediaminetetraacetic acid (EDTA) | Alfa Aesar | J15694 | |
| HEPES | Lonza | 17-737E | |
| Acrylamide | Sigma-Aldrich | A8887 | |
| N.N'-methylenebisacrylamide | Sigma-Aldrich | 146072 | |
| Ammonium persulfate (APS) | Bio-rad | 161-0700 | |
| Tetramethylethylenediamine (TEMED) | Bio-rad | 161-0800 | |
| 10X Tris-Borate-EDTA (TBE) Buffer, pH 8.3 | 1st Base | BUF-3010-10X1L | |
| Glycerol | Promega | H5433 | |
| Ethidium bromide (10 mg/mL) | Bio-rad | 161-0433 | |
| High Precision Cell (Quartz Suprasil, 200-2500 nm) | Hellma Analytics | 105.250-QS |
Riferimenti
- Cech, T. R., Steitz, J. A. The noncoding RNA revolution-trashing old rules to forge new ones. Cell. 157 (1), 77-94 (2014).
- Velagapudi, S. P., Gallo, S. M., Disney, M. D. Sequence-based design of bioactive small molecules that target precursor microRNAs. Nat Chem Biol. 10, 291-297 (2014).
- Patil, K. M., Chen, G., Jurga, S., Erdmann, V. A., Barciszewski, J. . Modified Nucleic Acids in Biology and Medicine. , 299-317 (2016).
- Hyrup, B., Nielsen, P. E. Peptide nucleic acids (PNA): synthesis, properties and potential applications. Bioorg Med Chem. 4 (1), 5-23 (1996).
- Shiraishi, T., Nielsen, P. E. Improved cellular uptake of antisense peptide nucleic acids by conjugation to a cell-penetrating peptide and a lipid domain. Methods Mol Biol. 751, 209-221 (2011).
- Shiraishi, T., Nielsen, P. E. Nanomolar cellular antisense activity of peptide nucleic acid (PNA) cholic acid ("umbrella") and cholesterol conjugates delivered by cationic lipids. Bioconjugate Chem. 23 (2), 196-202 (2012).
- Khoo, B., Roca, X., Chew, S. L., Krainer, A. R. Antisense oligonucleotide-induced alternative splicing of the APOB mRNA generates a novel isoform of APOB. BMC Mol Biol. 8, 3 (2007).
- Stein, C. A., et al. Efficient gene silencing by delivery of locked nucleic acid antisense oligonucleotides, unassisted by transfection reagents. Nucleic Acids Res. 38 (1), 3 (2010).
- Kole, R., Krainer, A. R., Altman, S. RNA therapeutics: beyond RNA interference and antisense oligonucleotides. Nat Rev Drug Discov. 11 (2), 125-140 (2012).
- Peacey, E., Rodriguez, L., Liu, Y., Wolfe, M. S. Targeting a pre-mRNA structure with bipartite antisense molecules modulates tau alternative splicing. Nucleic Acids Res. 40 (19), 9836-9849 (2012).
- Stenvang, J., Petri, A., Lindow, M., Obad, S., Kauppinen, S. Inhibition of microRNA function by antimiR oligonucleotides. Silence. 3 (1), 1 (2012).
- Ma, X., et al. Intracellular delivery of antisense peptide nucleic acid by fluorescent mesoporous silica nanoparticles. Bioconjugate Chem. 25 (8), 1412-1420 (2014).
- Wojtkowiak-Szlachcic, A., et al. Short antisense-locked nucleic acids (all-LNAs) correct alternative splicing abnormalities in myotonic dystrophy. Nucleic Acids Res. 43 (6), 3318-3331 (2015).
- Lenartowicz, E., et al. Antisense Oligonucleotides Targeting Influenza A Segment 8 Genomic RNA Inhibit Viral Replication. Nucleic Acid Ther. 26 (5), 277-285 (2016).
- Avitabile, C., et al. Targeting pre-miRNA by peptide nucleic acids: a new strategy to interfere in the miRNA maturation. Artif DNA PNA XNA. 3 (2), 88-96 (2012).
- Barczak, A. K., et al. RNA signatures allow rapid identification of pathogens and antibiotic susceptibilities. Proc Natl Acad Sci USA. 109 (16), 6217-6222 (2012).
- Das, I., et al. A peptide nucleic acid-aminosugar conjugate targeting transactivation response element of HIV-1 RNA genome shows a high bioavailability in human cells and strongly inhibits tat-mediated transactivation of HIV-1 transcription. J Med Chem. 55 (13), 6021-6032 (2012).
- Fabani, M. M., et al. Efficient inhibition of miR-155 function in vivo by peptide nucleic acids. Nucleic Acids Res. 38 (13), 4466-4475 (2010).
- Riguet, E., et al. A peptide nucleic acid-neamine conjugate that targets and cleaves HIV-1 TAR RNA inhibits viral replication. J Med Chem. 47 (20), 4806-4809 (2004).
- Torres, A. G., et al. Chemical structure requirements and cellular targeting of microRNA-122 by peptide nucleic acids anti-miRs. Nucleic Acids Res. 40 (5), 2152-2167 (2012).
- Upadhyay, A., Dixit, U., Manvar, D., Chaturvedi, N., Pandey, V. N. Affinity capture and identification of host cell factors associated with hepatitis C virus (+) strand subgenomic RNA. Mol Cell Proteomics. 12 (6), 1539-1552 (2013).
- Wesolowski, D., et al. Basic peptide-morpholino oligomer conjugate that is very effective in killing bacteria by gene-specific and nonspecific modes. Proc Natl Acad Sci USA. 108 (40), 16582-16587 (2011).
- Armitage, B. A. The impact of nucleic acid secondary structure on PNA hybridization. Drug Discov Today. 8 (5), 222-228 (2003).
- Thomas, S. M., et al. Antitumor effects of EGFR antisense guanidine-based peptide nucleic acids in cancer models. ACS Chem Biol. 8 (2), 345-352 (2013).
- Bahal, R., McNeer, N. A., Ly, D. H., Saltzman, W. M., Glazer, P. M. Nanoparticle for delivery of antisense gammaPNA oligomers targeting CCR5. Artif DNA PNA XNA. 4 (2), 49-57 (2013).
- Adams, B. D., Parsons, C., Walker, L., Zhang, W. C., Slack, F. J. Targeting noncoding RNAs in disease. J. Clin. Invest. 127 (3), 761-771 (2017).
- Matsui, M., Corey, D. R. Non-coding RNAs as drug targets. Nat Rev Drug Discov. 16 (3), 167-179 (2017).
- Devi, G., Zhou, Y., Zhong, Z., Toh, D. -. F. K., Chen, G. RNA triplexes: from structural principles to biological and biotech applications. Wiley Interdiscip Rev RNA. 6 (1), 111-128 (2015).
- Rozners, E. Recent Advances in Chemical Modification of Peptide Nucleic Acids. J Nucleic Acids. 2012, 8 (2012).
- Devi, G., Yuan, Z., Lu, Y., Zhao, Y., Chen, G. Incorporation of thio-pseudoisocytosine into triplex-forming peptide nucleic acids for enhanced recognition of RNA duplexes. Nucleic Acids Res. 42 (6), 4008-4018 (2014).
- Toh, D. K., et al. Incorporating a guanidine-modified cytosine base into triplex-forming PNAs for the recognition of a C-G pyrimidine-purine inversion site of an RNA duplex. Nucleic Acids Res. 44 (19), 9071-9082 (2016).
- Li, M., Zengeya, T., Rozners, E. Short peptide nucleic acids bind strongly to homopurine tract of double helical RNA at pH 5.5. J Am Chem Soc. 132 (25), 8676-8681 (2010).
- Gupta, P., Zengeya, T., Rozners, E. Triple helical recognition of pyrimidine inversions in polypurine tracts of RNA by nucleobase-modified PNA. Chem Commun. 47 (39), 11125-11127 (2011).
- Zengeya, T., Li, M., Rozners, E. PNA containing isocytidine nucleobase: synthesis and recognition of double helical RNA. Bioorg Med Chem Lett. 21 (7), 2121-2124 (2011).
- Gupta, P., Muse, O., Rozners, E. Recognition of double-stranded RNA by guanidine-modified peptide nucleic acids. Biochemistry. 51 (1), 63-73 (2012).
- Zengeya, T., Gupta, P., Rozners, E. Triple-helical recognition of RNA using 2-aminopyridine-modified PNA at physiologically relevant conditions. Angew Chem Int Ed. 51 (50), 12593-12596 (2012).
- Muse, O., et al. Sequence selective recognition of double-stranded RNA at physiologically relevant conditions using PNA-peptide conjugates. ACS Chem Biol. 8 (8), 1683-1686 (2013).
- Hnedzko, D., Cheruiyot, S. K., Rozners, E. Using Triple Helix Forming Peptide Nucleic Acids for Sequence-selective Recognition of Double-stranded RNA. Curr Protoc Nucleic Acid Chem. 58, 61-64 (2014).
- Zengeya, T., Gupta, P., Rozners, E. Sequence selective recognition of double-stranded RNA using triple helix-forming peptide nucleic acids. Methods Mol Biol. 1050, 83-94 (2014).
- Endoh, T., Hnedzko, D., Rozners, E., Sugimoto, N. Nucleobase-Modified PNA Suppresses Translation by Forming a Triple Helix with a Hairpin Structure in mRNA In Vitro and in Cells. Angew Chem Int Ed. 55 (3), 899-903 (2016).
- Hnedzko, D., McGee, D. W., Karamitas, Y. A., Rozners, E. Sequence-selective recognition of double-stranded RNA and enhanced cellular uptake of cationic nucleobase and backbone-modified peptide nucleic acids. RNA. 23 (1), 58-69 (2017).
- Wexselblatt, E., Esko, J. D., Tor, Y. On guanidinium and cellular uptake. J Org Chem. 79 (15), 6766-6774 (2014).
- Kaiser, E., Colescott, R. L., Bossinger, C. D., Cook, P. I. Color test for detection of free terminal amino groups in the solid-phase synthesis of peptides. Anal. Biochem. 34 (2), 595-598 (1970).
- Roberts, R. W., Crothers, D. M. Stability and properties of double and triple helices: dramatic effects of RNA or DNA backbone composition. Science. 258 (5087), 1463-1466 (1992).
- Zhou, Y., et al. Recognition of RNA duplexes by chemically modified triplex-forming oligonucleotides. Nucleic Acids Res. 41 (13), 6664-6673 (2013).
- Gisin, B. F. The monitoring of reactions in solid-phase peptide synthesis with picric acid. Anal. Chim. Acta. 58 (1), 248-249 (1972).
- Gogoi, K., Mane, M. V., Kunte, S. S., Kumar, V. A. A versatile method for the preparation of conjugates of peptides with DNA/PNA/analog by employing chemo-selective click reaction in water. Nucleic Acids Res. 35 (21), 139 (2007).
- Shabanpoor, F., Gait, M. J. Development of a general methodology for labelling peptide-morpholino oligonucleotide conjugates using alkyne-azide click chemistry. Chem Commun. 49 (87), 10260-10262 (2013).
- Rostovtsev, V. V., Green, L. G., Fokin, V. V., Sharpless, K. B. A Stepwise Huisgen Cycloaddition Process: Copper(I)-Catalyzed Regioselective "Ligation" of Azides and Terminal Alkynes. Angew Chem Int Ed. 41 (14), 2596-2599 (2002).
- Haaima, G., Hansen, F. H., Christensen, L., Dahl, O., Nielsen, P. E. Increased DNA binding and sequence discrimination of PNA oligomers containing 2,6-diaminopurine. Nucleic Acids Res. 25 (22), 4639-4643 (1997).
- Schroeder, S. J., Turner, D. H. . Methods Enzymol. 468, 371-387 (2009).
- McDowell, J. A., Turner, D. H. Investigation of the Structural Basis for Thermodynamic Stabilities of Tandem GU Mismatches: Solution Structure of (rGAGGUCUC)2 by Two-Dimensional NMR and Simulated Annealing. Biochemistry. 35 (45), 14077-14089 (1996).
- Sato, T., Sato, Y., Nishizawa, S. Triplex-Forming Peptide Nucleic Acid Probe Having Thiazole Orange as a Base Surrogate for Fluorescence Sensing of Double-stranded RNA. J Am Chem Soc. 138 (30), 9397-9400 (2016).
- Cheruiyot, S. K., Rozners, E. Fluorescent 2-Aminopyridine Nucleobases for Triplex-Forming Peptide Nucleic Acids. ChemBioChem. 17 (16), 1558-1562 (2016).
- Sato, T., Sato, Y., Nishizawa, S. Optimization of the Alkyl Linker of TO Base Surrogate in Triplex-Forming PNA for Enhanced Binding to Double-Stranded RNA. Chem Eur J. 23 (17), 4079-4088 (2017).
- Virta, P. M., Tahtinen, V., Granqvist, L., Murtola, M., Stromberg, R. 19F NMR spectroscopic analysis of the binding modes in triple helical PNA/microRNA-complexes. Chem Eur J. , (2017).
- Zengeya, T., Gindin, A., Rozners, E. Improvement of sequence selectivity in triple helical recognition of RNA by phenylalanine-derived PNA. Artif DNA PNA XNA. 4 (3), 69-76 (2013).
- Moses, A. C., Huang, S. W., Schepartz, A. Inhibition of Rev.RRE complexation by triplex tethered oligonucleotide probes. Bioorg Med Chem. 5 (6), 1123-1129 (1997).
- Ben Gaied, N., Zhao, Z., Gerrard, S. R., Fox, K. R., Brown, T. Potent triple helix stabilization by 5',3'-modified triplex-forming oligonucleotides. ChemBioChem. 10 (11), 1839-1851 (2009).
- Grimm, G. N., Boutorine, A. S., Lincoln, P., Norden, B., Helene, C. Formation of DNA triple helices by an oligonucleotide conjugated to a fluorescent ruthenium complex. ChemBioChem. 3 (4), 324-331 (2002).
- Tran, T., et al. Targeting the r(CGG) Repeats That Cause FXTAS with Modularly Assembled Small Molecules and Oligonucleotides. ACS Chem Biol. , (2014).
- Gianolio, D. A., Segismundo, J. M., McLaughlin, L. W. Tethered naphthalene diimide-based intercalators for DNA triplex stabilization. Nucleic Acids Res. 28 (10), 2128-2134 (2000).
- Rumney, S., Kool, E. T. Structural Optimization of Non-Nucleotide Loop Replacements for Duplex and Triplex DNAs. J Am Chem Soc. 117, 5635-5646 (1995).
- Stafford, R. L., Dervan, P. B. The reach of linear protein-DNA dimerizers. J Am Chem Soc. 129 (45), 14026-14033 (2007).
- Gupta, A., Bahal, R., Gupta, M., Glazer, P. M., Saltzman, W. M. Nanotechnology for delivery of peptide nucleic acids (PNAs). J Control Release. 240, 302-311 (2016).
- Avitabile, C., et al. Incorporation of Naked Peptide Nucleic Acids into Liposomes Leads to Fast and Efficient Delivery. Bioconjugate Chem. 26 (8), 1533-1541 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon