Method Article
Seguita dalla singola cellula di rilevamento a Monitor biologia cellulare e la progressione di lignaggio di popolazioni neurali Multiple immagini dal vivo
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
È descritto un protocollo robusto per monitorare le popolazioni neurali di time-lapse video-microscopia seguita da basati su software di post-elaborazione. Questo metodo rappresenta un potente strumento per identificare gli eventi biologici in una popolazione selezionata durante esperimenti dal vivo di formazione immagine.
Abstract
La comprensione dei meccanismi che controllano gli eventi biologici critici delle popolazioni di cellule neurali, come proliferazione, differenziazione o le decisioni del destino cellulare, sarà cruciale per la progettazione di strategie terapeutiche per molte malattie che colpiscono il sistema nervoso. Attuali metodi per tenere traccia di popolazioni cellulari si basano sui loro risultati finali in immagini fisse e riescono in genere a fornire sufficiente risoluzione temporale per identificare caratteristiche comportamentali in singole cellule. Inoltre, variazioni nella morte delle cellule, eterogeneità del comportamento all'interno di una popolazione delle cellule, diluizione, diffusione o la scarsa efficienza degli indicatori utilizzati per analizzare le cellule sono tutti svantaggi importanti che porteranno a incomplete o errate letture dei risultati. Al contrario, eseguendo dal vivo imaging e singola cellula in condizioni appropriate di monitoraggio rappresenta un potente strumento per monitorare ciascuno di questi eventi. Qui, un protocollo di time-lapse video-microscopia, seguito da post-elaborazione, è descritto per tenere traccia di popolazioni neurali con risoluzione singola cella, utilizzando software specifici. I metodi descritti consentono ai ricercatori di indirizzo domande essenziali per quanto riguarda la progressione di biologia e il lignaggio di cella delle popolazioni neurali distinti.
Introduzione
Al fine di sviluppare nuove e più efficaci strategie terapeutiche per rigenerare popolazioni neurali, dobbiamo prima capire i meccanismi di base che mantengono le cellule con un potenziale di rigenerazione neurale. Perseguendo questo obiettivo richiede una conoscenza completa dei fattori che regolano gli equilibri tra quiescenza, proliferazione e differenziazione, la modalità e la tempistica della divisione, durata del ciclo cellulare, capacità migratoria, redditività, ecc. Anche se è un approccio tecnico che è stato impiegato per molti anni1, immagini dal vivo e osservazione diretta rimangono ancora l'opzione migliore per monitorare gli eventi sopra elencati. Al contrario di molti altri approcci centrati su letture di end-point, live imaging e singola cella rilevamento forniscono informazioni per tutta la lunghezza di un esperimento2,3,4,5, 6. quindi, l'aggiunta di risoluzione temporale permette di morte delle cellule, comportamento eterogenee delle cellule o le decisioni di destino delle cellule, così come molti altri eventi critici di identificare che altrimenti potrebbero passare inosservati. Idealmente, queste caratteristiche delle cellule dovrebbero essere controllate meglio presso la singola cella livello in vivo dove sia intrinseca (cell autonoma) ed estrinseche cues (nicchia delle cellule) sono presi in considerazione.
Tuttavia, anche se in vitro situazione eventi si verificano in un ambiente che non riproducono l'ambiente naturale, le condizioni della coltura a bassa densità in genere utilizzate in questi protocolli sono più adatte rivelare le caratteristiche intrinseche della cellule. Inoltre, un controllo più semplice dell'ambiente circostante, modificando semplicemente il mezzo di crescita, può costituire uno strumento prezioso per studiare il ruolo individuale di ogni fattore estrinseco che definisce la nicchia neurale, come pure i fattori ambientali che possono essere indotta in scenari patologici7,8,9,10,11,12,13. Pertanto, se configurato correttamente, come il protocollo proposto qui, live imaging fornisce una soluzione fattibile in vitro per affrontare la maggior parte delle domande precedentemente enumerate.
In breve, questo protocollo descrive l'hardware, software, condizioni di coltura e i passaggi principali necessari per eseguire con successo un esperimento di imaging dal vivo seguito dalla singola cellula di rilevamento. Questo approccio offre preziose informazioni che aiuta a svelare aspetti fondamentali della biologia e della progressione lignaggio, di popolazioni neurali multiple.
Protocollo
Le sezioni seguenti descrivono i passaggi necessari per eseguire dal vivo imaging seguita da rilevamento singola cella di popolazioni neurali multiple (Figura 1). Tutte le procedure che coinvolgono gli animali descritti nel presente protocollo devono essere effettuati conformemente agli orientamenti del Consiglio internazionale per la scienza degli animali di laboratorio (ICLAS).
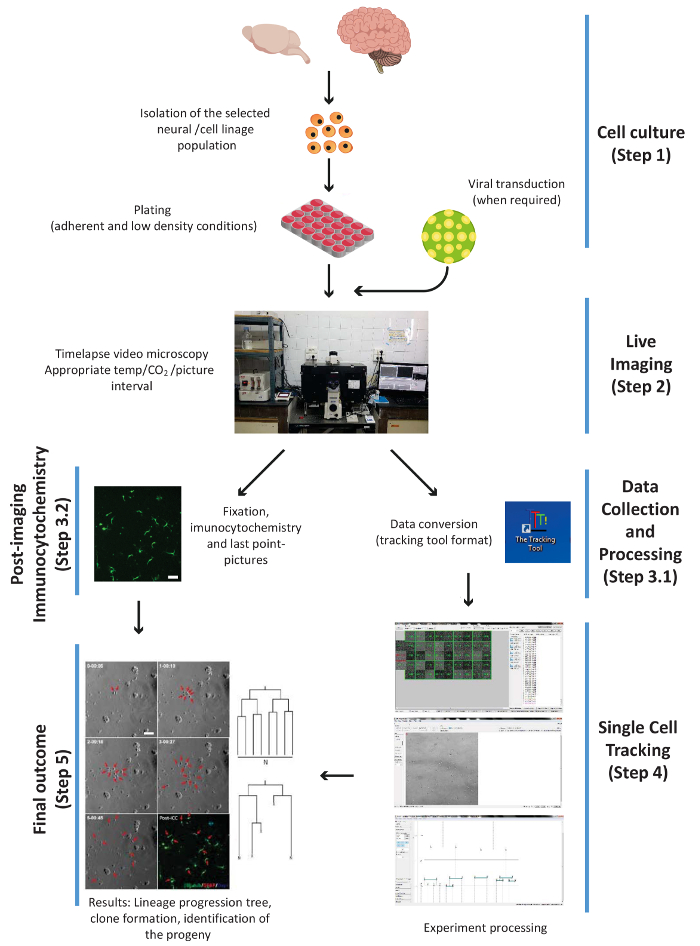
Figura 1. Schema che illustra i principali passaggi sperimentali della procedura, vale a dire: cella cultura, live imaging, PICC e raccolta dati, singola cella di rilevamento e il risultato finale. I passaggi sono numerati secondo il flusso di lavoro del protocollo. Clicca qui per visualizzare una versione più grande di questa figura.
1. cella cultura: Isolamento e placcatura dell'elemento selezionato in popolazione neurale o stirpe delle cellule
Nota: In combinazione con questo protocollo, sono riportati esempi della sua applicazione a popolazioni distinte delle cellule per convalidare la sua utilità per analizzare la biologia delle cellule neurali. Questi includono: le cellule staminali neurali adulte (aNSCs) derivato dal mouse SubEpendymal zona (SEZ) (per un isolamento dettagliato protocollo Vedi14); Astrociti corticali postnatali per studiare riprogrammazione neuronale (per un isolamento dettagliato protocollo Vedi15); Astrociti cerebellari postnatali (per una dettagliata isolamento metodo Vedi16); e la linea cellulare di Neuroblastoma di Mouse Neuro-2a (N2a).
- Seme le celle direttamente su poli-D-lisina 24 pozzetti piastre rivestite. Uso 1 mL di terreno di coltura per pozzetto. Incubare le piastre a 37 ° C e 8% CO2 per aNSCs, o a 37 ° C e 5% di CO2 per la linea di astrociti/cellulare per 2 h prima dell'imaging dal vivo. Evitare l'uso di vetrini coprioggetti per evitare movimenti indesiderati come il tavolino del microscopio motorizzato è spostato che rende impraticabile unicellulare di rilevamento.
Nota: Densità delle cellule e terreni di coltura utilizzati negli esperimenti sono: 30-40.000 cellule/pozzetto per aNSCs in Dulbecco modificato medio dell'Aquila (miscela nutriente DMEM:F12 medio); 20.000 cellule/pozzetto per cellule N2a in DMEM alto glucosio medium, 80.000 cellule/pozzetto per gli astrociti cerebellari in mezzo DMEM alto glucosio; e 55-65.000 cellule/pozzetto per gli astrociti postnatali in mezzo DMEM:F12 miscela di nutrienti. - Standardizzare il protocollo di cultura regolando la densità delle cellule della cultura per il minor numero di cellule fattibile. Ciò nonostante, la densità delle cellule deve essere sufficientemente alta per mantenere la vitalità della cultura.
Nota: Se la densità delle cellule è troppo alta, i detriti in eccesso o scarsa dissociazione (grumi) può ostacolare rilevamento delle singole cellule.
2. live Imaging di Time-Lapse Video-microscopia
- Accendere i sistemi microscopio, macchina fotografica, hardware e incubazione. Impostare la temperatura e pressione a 37 ° C e 8% CO2 per aNSCs, o a 37 ° C e 5% di CO2 per la linea di astrociti/cella d'aria. Consentire la temperatura e i livelli di CO2 per stabilizzare per 1-2 h.
Nota: Attrezzatura specifica è necessaria per eseguire l'analisi dei video time-lapse, tra cui: microscopi a contrasto/fluorescenza brillante campo/fase con componenti motorizzati; dispositivi di incubazione che controllano la temperatura, la CO2 e l'umidità; e infine, affidabile e sufficientemente potente hardware e software in grado di acquisire e gestire il volume di immagini ottenute durante esperimenti dal vivo di formazione immagini (si prega di controllare la Tabella materiali). - Una volta che le cellule sono saldamente fissate alla piastra (2 h dopo il placcaggio), è possibile utilizzare un pennarello permanente per fare un piccolo segno sul fondo di un pozzo che non verrà utilizzato per il rilevamento, cioè, un pozzo che non contengono celle.
Nota: Questo marchio verrà utilizzato come riferimento a zero le coordinate xyz, e può essere utilizzato in qualsiasi momento durante o dopo l'esperimento o tra le modifiche del mezzo, per tornare alla posizione zero. - Posizionare la piastra all'interno camera di incubazione del microscopio e fissare saldamente la piastra sul palcoscenico per evitare qualsiasi movimento indesiderato durante lo spostamento di fase motorizzata del microscopio.
- Lasciare che la temperatura del terreno di coltura cellulare per equilibrare nella camera per circa 20 min. Questo passaggio eviterà una perdita di messa a fuoco durante la registrazione a causa della dilatazione dei componenti.
- Avviare il software di live-formazione immagine e selezionare il modulo time-lapse per impostare l'esperimento.
- Impostare la durata totale dell'esperimento e i cicli di acquisizione immagine nel menu"scheda orario". A causa della fototossicità intrinseca dell'oggetto trasmesso o luce di fluorescenza utilizzato, è necessario definire un intervallo adeguato per l'equilibrio tra la risoluzione temporale dell'analisi e la morte delle cellule potenziali.
Nota: ad esempio, un totale di 120 ore è stato selezionato per aNSC culture, l'acquisizione di immagini di campo chiaro ogni 5 min. considera che l'acquisizione di 120 h di un singolo filmato in questa configurazione richiederà 120-150 gigabyte di spazio di archiviazione del dispositivo del computer. - Selezionare le posizioni di immagine definite da x e y coordinate e la distanza focale (coordinata z) nella "punti xyz tab menu". Includere il punto di riferimento (coordinate xyz zero) come la posizione iniziale al fine di recuperare le coordinate in qualsiasi momento.
- Solo o in combinazione con epifluorescenza eccitazione quando richiesto, selezionare il tipo di acquisizione in "lunghezza d'onda nella scheda menu di selezione", campo chiaro. Selezionare il tempo di esposizione. Tenete a mente che una eccessiva esposizione ai trasmessi, e soprattutto fluorescenti luce, può compromettere la vitalità cellulare (come indicato sopra).
- Per aNSCs, gli astrociti cerebellari e cellule N2a, selezionare campo chiaro (tempo di esposizione di 10-50 ms).
- Per gli astrociti corticali trasdotte selezionare campo chiaro (tempo di esposizione di 10-50 ms) in combinazione con fluorescenza rosso/verde, a seconda del reporter utilizzati per l'esperimento (lunghezza d'onda di eccitazione rosso: 550 nm e 400 ms tempo di esposizione; eccitazione verde lunghezza d'onda: 460-500 nm e tempo di esposizione di 100 ms).
- Definire il nome dell'esperimento e la cartella in cui verranno memorizzate le immagini. Salvare l'elenco delle posizioni per ricaricare l'esperimento in qualsiasi momento e dopo aver impostate tutte le condizioni, è possibile eseguire l'esperimento facendo clic sul pulsante "Esegui ora".
- Sospendere l'esperimento e ri-regolare le condizioni di messa a fuoco cliccando il pulsante z "sovrascrivere" una volta al giorno fino a quando l'esperimento è completato. Se durante l'imaging dal vivo sono necessarie modifiche nel mezzo, sospendere l'esperimento e recuperare la piastra dalla camera di lasso di tempo.
Nota: Le variazioni di pH del mezzo a causa di morte delle cellule o sovra-proliferazione, così come le variazioni di temperatura ambiente, possono influenzare la corretta messa a fuoco del microscopio sulle cellule. Per colture sensibili (ad esempio, aNSCs) si consiglia l'uso di supplementato con 4-(2-hydroxyethyl)-1-piperazineethanesulfonic acido (HEPES) (concentrazione finale: 1 mM).
3. post-imaging immunocitochimica (PICC), raccolta di dati e di elaborazione
- Una volta completato l'esperimento, mettere in pausa il software e recuperare la piastra di fissaggio e PICC, come descritto nei passaggi successivi.
- Eseguire la fissazione delle cellule: lavare le cellule una volta con salino di 1 mL tamponato fosfato (PBS) e aggiungere 500 µ l di paraformaldeide (PFA) (4% in PBS), incubare 10 min a temperatura ambiente (TA).
Attenzione: Paraformaldeide è un fissativo forte e dovrebbe essere maneggiato con cura per evitare il contatto con la pelle o gli occhi. Esso deve essere manipolato solo all'interno di una cappa aspirante. - Lavare le cellule tre volte con 1 mL di PBS e aggiungere 500 µ l di soluzione bloccante (PBS contenente il 2% (wt/vol) di albumina di siero bovino (BSA) e 0,2% (vol/vol) di un tensioattivo non ionico). Incubare 1h a RT.
- Rimuovere la soluzione bloccante e aggiungere 250-400 µ l della soluzione di anticorpi primari. Incubare 2 ore a RT. La soluzione di anticorpi primari contiene gli anticorpi primari diluiti in PBS contenente il 2% (wt/vol) di BSA e 0,2% (vol/vol) di un tensioattivo non ionico. Gli anticorpi utilizzati negli esperimenti descritti qui: GFAP (1: 500), βIII-tubulina (1:1, 000) e α-tubulina (1:1, 000). Poiché questo avviene direttamente nel pozzo, grandi volumi delle soluzioni sono necessarie (250-400 µ l) per coprire tutte le celle.
- Lavare tre volte con 1 mL di PBS e aggiungere 250-400 µ l della soluzione di anticorpi secondari (diluita come descritto al punto 3.4). Anticorpi secondari utilizzati negli esperimenti descritti qui: anti-Mouse della fluorescina (FITC) (1: 800), anti-coniglio Cy3 (1: 500). Incubare 1h a RT nel buio.
- Lavare tre volte in 1 mL di PBS. Mantenere le cellule in 1 mL di PBS per le fasi successive del protocollo.
- Posizionare la piastra torna sul palco microscopio e fissare saldamente allo stadio per evitare movimenti indesiderati durante lo spostamento di fase di microscopio motorizzato.
- Recuperare la xyz posizione zero utilizzando il marchio effettuato nel passaggio 2.2 e re-impostare le posizioni da questo punto di riferimento premendo il pulsante "Offset tutte le X, Y, Z". Re-impostare la distanza focale per ogni posizione.
- Acquisire un round finale di immagini, configurare le condizioni necessarie per l'emissione di fluorescenza nel "menu della scheda di selezione di lunghezza d'onda" al fine di rilevare gli antigeni precedentemente destinati alla PICC.
- In breve, oltre a campo chiaro, attivare FITC (eccitazione: 495 nm) e Cy3 (eccitazione: 550 nm) opzioni di acquisizione del software. Utilizzare 10-50 ms per l'esposizione di 400 ms campo chiaro per rilevare i fluorofori e premere il pulsante "1 ciclo di tempo", per acquisire un ultimo giro di immagini.
Nota: L'intensità della fluorescenza può differire a seconda dell'esito PICC. Regolare il tempo di esposizione per ottenere la qualità ottimale dell'immagine.
- In breve, oltre a campo chiaro, attivare FITC (eccitazione: 495 nm) e Cy3 (eccitazione: 550 nm) opzioni di acquisizione del software. Utilizzare 10-50 ms per l'esposizione di 400 ms campo chiaro per rilevare i fluorofori e premere il pulsante "1 ciclo di tempo", per acquisire un ultimo giro di immagini.
- Selezionare l'opzione di esportazione di file del software ed esportare le immagini in formato Joint Photographic Experts Group (Jpeg) o Tagged Image File Format (Tiff) in una cartella di destinazione pre-definiti.
- Convertire le immagini esportate nel formato richiesto dal software di rilevamento: strumento di Tracking The17 (tTt). Per raggiungere questo obiettivo, definire l'input e output cartella nello "strumento di conversione tTt" funzionamento finestra, come pure gli indicatori utilizzati per le posizioni (xy), canali (c) e intervalli di tempo (t) e premere il pulsante "Converti immagini".
Nota: Le immagini devono essere rinominate in conformità con le impostazioni specifiche e devono essere immagazzinati in singole cartelle per ogni posizione utilizzata nell'esperimento. Istruzioni per l'installazione, requisiti, ridenominazione di posizioni/immagini e l'uso dello strumento di rilevamento sono disponibili per il download: https://www.bsse.ethz.ch/csd/software/tTt-and-qtfy.html.
4. singola cella Tracking
- Dopo avere rinominato i dati, eseguire il software di tTt. Selezionare una cartella di lavoro utente nome e tTt.
Nota: Il rilevamento strumento lavoro cartella conterrà tutti i dati analizzati e i risultati esportati. La cartella di lavoro deve essere denominata tTtexport, contenente sottocartelle denominate "AVIexport", "Configurazioni", "TreeExport" e "tTtfiles". - L'esperimento da caricare nella "finestra cartella selezionare esperimento", che indica il percorso della cartella dove è memorizzato l'esperimento, di selezionare e fare clic sul pulsante "Load esperimento".
- Eseguire il convertitore di file di registro per trasformare le immagini caricate in un formato che può essere letto dal software di rilevamento (per esperimenti caricati per la prima volta, questo sarà richiesto automaticamente dal software).
- Selezionare una posizione per l'inseguimento cliccando sul relativo simbolo (dopo la conversione, una panoramica delle posizioni registrate durante gli esperimenti verrà visualizzato nella finestra"layout posizione"). Ogni posizione sarà rappresentata da un simbolo consistente in una foto della posizione e il numero corrispondente (Vedi Figura 1).
- Una volta che la posizione è stata selezionata e viene visualizzato un elenco delle immagini disponibili sulla destra della finestra"layout position", selezionarli e fare clic sul pulsante "Carica immagini".
- Una volta che il caricamento è completato e viene visualizzata la finestra"Editor delle celle", selezionare le lunghezze d'onda e intervallo di immagine deve essere rilevata nella finestra"Editor delle cellule". Lunghezza d'onda 0 corrisponde al campo chiaro, 1 corrisponde a FITC, 2 a Cy3 e 3 per DAPI. Negli esperimenti descritti qui, è stato utilizzato intervallo 1, vale a dire, tutte le immagini caricate. Per chiarire, intervallo 2 significa caricamento di ogni seconda immagine.
- Una volta che le immagini sono state caricate, tornare alla finestra"layout posizione" e fare doppio clic sull'icona che rappresenta la posizione precedentemente caricata. Finestra"film" apparirà che permette il tracciamento di singola cellula essere eseguita.
- Seguendo le istruzioni di strumento di verifica, procedere al rilevamento. Selezionare il canale 0 (corrispondente al campo chiaro) e regolare la luminosità e il contrasto ("regolare gamma pulsante"). Iniziare la registrazione premendo il tasto F2.
Nota: Durante il tracciamento, la cella cingolata seguiranno posizionando il puntatore del mouse su di esso e premendo il tasto "0". La divisione cellulare, l'apoptosi cellulare e cellulare perso pulsanti sono disponibili per monitorare questi eventi cellulari specifici.
5. esito
- Una volta che la singola cella di rilevamento è stata completata e la progenie identificata, salvare l'esperimento (scheda cella Editor finestra/File / Salva attuale albero come) e procedere per esportare i risultati.
- Esportare gli alberi di lignaggio e i dati di cella nel "menu Esporta" situato nella finestra"Editor delle celle". Allo stesso modo, esportare le immagini di cella e film tramite il menu"Esporta" accessibile attraverso la finestra"film". Le immagini, alberi di lignaggio, dati e filmati verranno esportati nella cartella di lavoro di tTt.
Risultati
Il metodo descritto consente domande critiche per quanto riguarda la biologia cellulare di popolazioni neurali multiple per essere risolto. Per esempio, è stato possibile monitorare la progressione del lignaggio neurogena e oligodendrogliogenic di aNSCs7,8,14,18. Tenendo traccia singola aNSCs e la loro progenie (Figura 2A, B), è stato possibile dimostrare che aNSCs isolato in vitro mantenere la loro natura neurogena, principalmente generando neuroblasti, e che seguono una sequenza proposta in vivo19 ma non precedentemente dimostrato a livello di singola cellula. Inoltre, questo sistema di coltura ammessi asimmetriche divisioni cellulari possano essere visualizzati per la prima volta nel lignaggio aNSC da SEZ (Figura 2B), fornendo un modello unico per studiare NSC autorinnovamento8,14. Allo stesso modo e indipendentemente dal lignaggio analizzati, è stato possibile ottenere dati importanti per quanto riguarda la crescita delle cellule, il giro di divisione, di attuabilità delle cellule o di durata del ciclo cellulare.
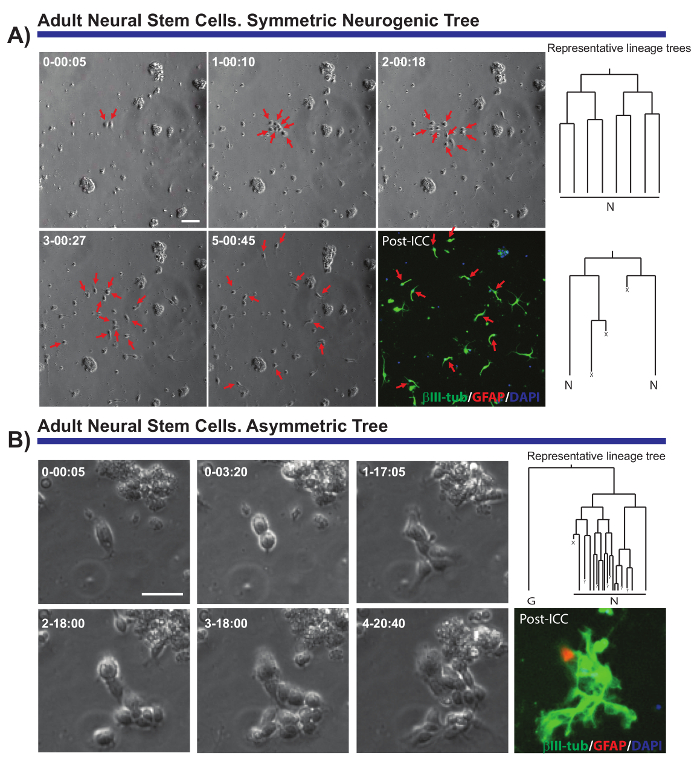
Figura 2. Esempio di aNSCs isolato dalla SEZ e analizzato dal vivo imaging e dalla singola cella di rilevamento. Immagini di contrasto di fase raffigurano la progressione del clone in diversi momenti (giorno-h: min). L'immagine finale corrisponde post-all'imaging immunocitochimica (PICC) per proteina silicea fibrillare Glial (GFAP, rosso), βIII-tubulina (verde) e 4', 6-diamidino-2-phenylindole (DAPI, blu). (A) analisi degli alberi simmetrici neurogeni attraverso diversi cicli di amplificazione divisioni per generare neuroblasti post-mitotici. Frecce rosse scegliere le celle incluse negli alberi simmetrici. Sulla destra, vengono visualizzati gli alberi di lignaggio corrispondente ai cloni e generati dal software di tTt. (B) esempio di un progenitore generando un albero neurogeno asimmetrico, con un ramo in fase di amplificazione divisioni per produrre neuroblasti mentre l'altra dà luogo a quiescente cellule positive di GFAP attraverso un evento potenziale di auto-rinnovamento. Sulla destra, l'albero di lignaggio generato dal software tTt è visualizzato. In tutti gli alberi del lignaggio: "N" raffigura post-mitotici neuroblasti; "G", cellule quiescenti di GFAP-positive; "X", la morte delle cellule; e "?" un cellulare perso. Barra della scala rappresenta 50 µm. Clicca qui per visualizzare una versione più grande di questa figura.
Immagini dal vivo e singola cella analisi di rilevamento inoltre fornisce una lettura accurata delle capacità migratorie di una popolazione neurale. Tali informazioni sono state ottenute dagli astrociti cerebellari postnatali presentati a una ferita scratch test20, generazione di informazioni per quanto riguarda la distanza media percorsa dai astrocytes quando chiusura della ferita (Figura 3). Inoltre, è stato possibile vedere che alcuni degli astrociti divisa durante il processo di guarigione, mentre gli altri rimangono inalterati in tutto l'esperimento. In maniera sconvolgente, quelli che dividono sembrano comportamenti migratori più prolifico rispetto ai loro omologhi di divisione (in viaggio due volte più lontano in media). Questo fenomeno suggerisce un'eterogeneità molto interessante della capacità di astrociti per formare una cicatrice dopo un danno, che sarebbe sono stato diluito nella lettura di un esperimento di analisi classica end-point.
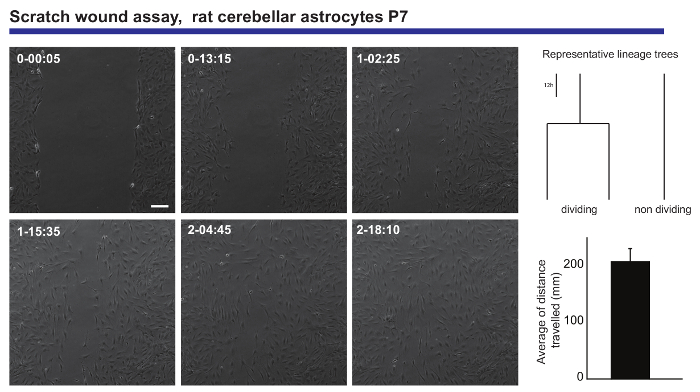
Figura 3. Analisi del comportamento migratorio dei astrocytes postnatale cerebellare in un graffio ferita dosaggio. Immagini di contrasto di fase raffigurano la ferita in diversi momenti (giorno-h: min). Alberi di lignaggio, generati dal software di tTt, illustrano il comportamento rappresentativo, in termini di divisione cellulare, dei astrocytes durante la chiusura della ferita. Istogramma mostra la distanza media percorsa dai astrocytes analizzati dalla singola cellula di rilevamento (media ± s.e.m.). Barra della scala rappresenta 50 µm. Clicca qui per visualizzare una versione più grande di questa figura.
Un'altra caratteristica interessante degli esperimenti di microscopia video time-lapse è la capacità di confrontare la proliferazione e la differenziazione in una popolazione delle cellule. Abbiamo testato le cellule N2a placcate in condizioni che promuovono la proliferazione (in presenza di 10% siero bovino fetale (FBS)) o differenziazione (in presenza di 0,5% FBS + acido arachidonico 10 µM). È stato possibile seguire la progressione di lignaggio di queste cellule in condizioni proliferative (Figura 4A), considerando che la differenziazione di cellule non proliferano e formano i neurites (Figura 4B). Notevolmente, singola cella rilevamento ammessi colonie con diversi proliferazione capacità di distinguere e allungamento dei neuriti (e retrazione) deve essere valutata, fornendo dati precisi e quantitativi che possono essere successivamente esportati.
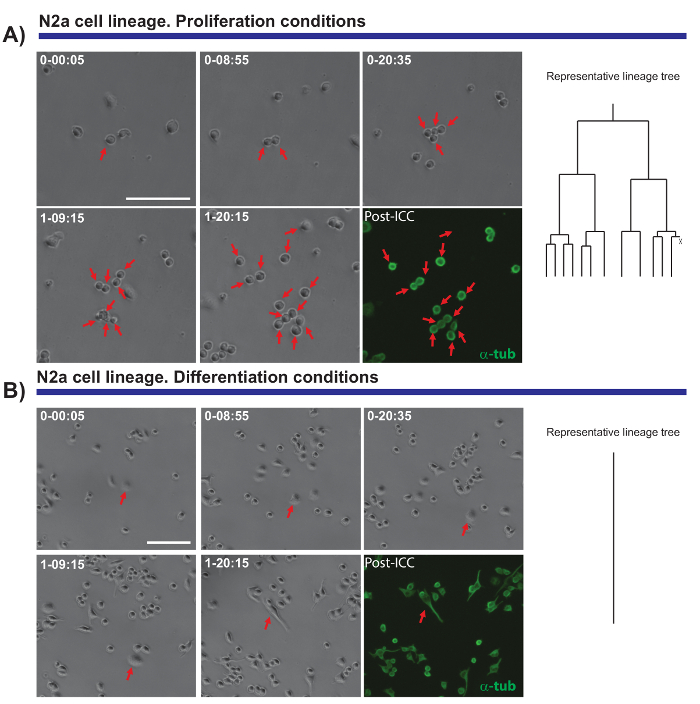
Figura 4. Il monitoraggio della biologia delle cellule N2a proliferazione (A) o condizioni di differenziazione (B). Immagini di contrasto di fase raffigurante la progressione del clone in diversi momenti (giorno-h: min). L'immagine finale corrisponde post-all'imaging immunocitochimica (PICC) per α-tubulina (verde). (A) singola cella di rilevamento permette il giro di divisione da monitorare, come pure l'eterogeneità nella risposta proliferativa delle cellule differenti. Sulla destra, l'albero di lignaggio generato dal software tTt illustra il comportamento proliferativo delle cellule N2a. (B) cellule in condizioni di differenziazione uscire il ciclo cellulare e generano neurites, un processo che può essere efficacemente misurato dall'analisi di post-formazione immagine. Singola tracciamento cellulare, rappresentato dall'albero della stirpe, sulla destra, illustra come le cellule N2a uscita ciclo cellulare e interrompere la divisione delle cellule in condizioni di differenziazione. Barra della scala rappresenta 50 µm.Clicca qui per visualizzare una versione più grande di questa figura.
Infine, immagini dal vivo e singola cella di rilevamento è estremamente utile per monitorare i cambiamenti morfologici e molecolari quando le cellule vengono sottoposti alla riprogrammazione. Formazione immagine diretta dei astrocytes postnatale trasformata con omologo Ascl4-scute 1 (Ascl1) fornisce dati importanti per quanto riguarda i cambiamenti morfologici che si verificano durante la riprogrammazione o il blocco della divisione cellulare quando gli astrociti vengono riprogrammati (Vedi Figura 5). Inoltre, quando Ascl1 trasduzione è combinato con la trasduzione di un costrutto che codifica per la proteina di fluorescenza verde (GFP) sotto il controllo del promotore doppie cortine (DCX), è possibile definire il punto di tempo preciso quando un neurone marcatori specifici cominciano a essere espressi nelle cellule riprogrammate (Figura 5). Time-lapse video-microscopia consente inoltre il numero di celle che completare con successo la riprogrammazione per essere quantificato e rispetto per le cellule che muoiono durante questo processo. Monitoraggio di tali eventi ha portato all'identificazione dei critici "checkpoint" nelle celle che sono stati riprogrammati con successo9.
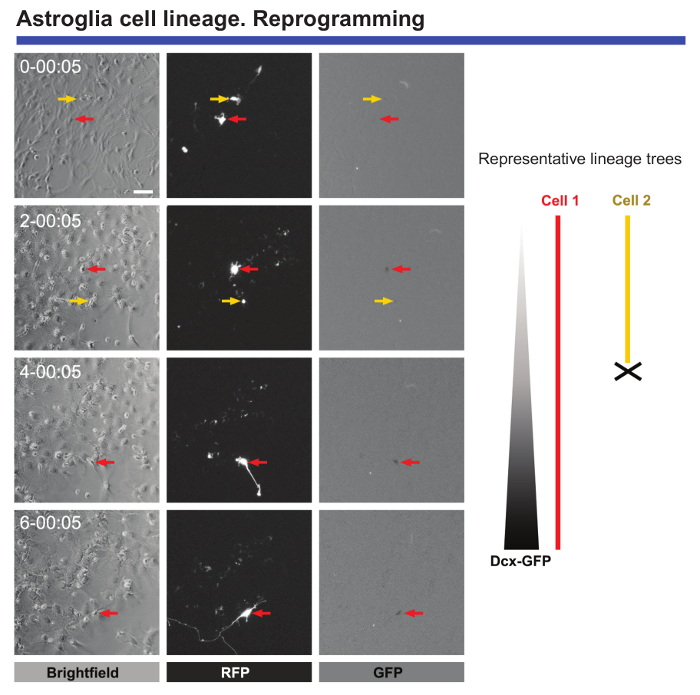
Figura 5. Analisi dei astrocytes corticali postnatale sottoposti a riprogrammazione neuronale. Riprogrammazione è stata indotta tramite trasduzione con vettori di proteina (RFP) di fluorescenza rosso-Ascl1 pro-neurogena. Conversione di un neurone è stata monitorata da co-trasduzione con un vettore codificante la GFP sotto il controllo di un promotore DCX. Immagini di contrasto di fase mostrano la progressione della riprogrammazione in diversi momenti (giorno-h: min). Immagini di fluorescenza di espressione RFP e GFP, rispettivamente. Immagini dal vivo e singola cella rilevamento ammessi eventi cruciali da seguire, ad esempio i cambiamenti morfologici, assenza di divisione delle cellule durante la riprogrammazione, morte cellulare, e può essere definito il tempo preciso quando cellule riprogrammate iniziano a esprimono marcatori neuronali . Barra della scala rappresenta 80 µm. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Uno dei valori più importanti di formazione immagine diretta è la possibilità di eseguire l'analisi accurata lignaggio, chiarire gli aspetti critici della progressione di lignaggio in una popolazione neurale. Lignaggio traccia è definito come l'identificazione e monitoraggio di tutti i discendenti di un singolo progenitore, dal fondatore del clone al clone successivo formata21. Notevolmente, metodi alternativi di lignaggio traccia (ad esempio, la trasduzione virale o reporter multicolor costrutti21) hanno uno svantaggio critico, per cui il risultato finale è basato su immagini fisse e non necessariamente costituiscono l'intera sequenza. Ciò significa che la morte delle cellule, eterogeneità nel comportamento della popolazione cellulare, diluizione, diffusione o scarsa efficienza dei marcatori, insieme con altri handicap importante, portare a incomplete o errate letture dei risultati2. Inoltre, live imaging consente al ricercatore di analizzare le caratteristiche importanti della biologia delle popolazioni neurali, come ad esempio la modalità e la tempistica di divisione cellulare, crescita cellulare, migrazione, proliferazione e differenziazione, durata del ciclo cellulare, neurite formazione, la complessità e la lunghezza, cella selezione destino (differenziazione), o conversione (riprogrammazione).
Inoltre, formazione immagine diretta può essere facilmente integrata con altre analisi destinato per ottenere dati da singole cellule come, ad esempio, sequenziamento di RNA. Tuttavia, per ottenere benefici combinati da formazione immagine dal vivo e di altre tecniche richiede che quelle cellule precedentemente monitorati nei film sono poi ri-identificati e raccolti individualmente per l'analisi secondaria. Ciò può essere ottenuto utilizzando microscopi che includono Coordinate posizionali, applicando reporter fluorescenti per celle specifiche o analizzando la distribuzione di gruppi di cellule come riferimenti. Infatti, la combinazione del profilo del trascrittoma e comportamento di singole celle possono rappresentare un potente percorso per delucidare nuovi spunti molecolari coinvolti nella biologia delle cellule.
Uno dei principali problemi che possono compromettere un esperimento di formazione immagine dal vivo è una densità di cultura cellulare inadeguato. Come indicato in precedenza, ad alta densità l'eccesso di detriti o scarsa dissociazione (formazione di ciuffo) può influenzare la qualità e la risoluzione spaziale delle immagini, rendendo impraticabile unicellulare di rilevamento. Quindi, le condizioni delle popolazioni distinte delle cellule in fase di studio dovrebbero essere regolate per il minor numero di celle possibile senza compromettere la redditività della coltura delle cellule.
La frequenza di acquisizione di immagini è inoltre cruciale e deve essere attentamente regolata, soprattutto quando viene utilizzata l'illuminazione a fluorescenza. Una eccessiva esposizione ai trasmessi e soprattutto luce di fluorescenza può compromettere la vitalità cellulare. In alternativa, un ritardo eccessivo tra l'acquisizione delle immagini può interferire con la risoluzione temporale dell'analisi.
Un altro passaggio fondamentale durante l'esperimento di imaging dal vivo è l'adeguamento periodico di messa a fuoco. Fallimento nella corretta impostazione/re-setting della distanza focale può ostacolare rilevamento singola cella. Inoltre, è necessario controllare attentamente che la camera di incubazione consente di mantenere la temperatura adeguata, l'umidità e livelli di CO2 , che modifica variazioni indesiderate che possono indurre la morte delle cellule.
Infine, una volta che è stato eseguito il PICC, è importante recuperare correttamente la posizione xyz zero prima dell'ultimo turno di acquisizione immagine. Non corretto re-impostazione degli xyz posizione zero renderà difficile da eguagliare le immagini di contrasto di fase e di immunofluorescenza, impedendo l'identificazione della progenie delle cellule.
Anche se questo approccio ha molti aspetti positivi, persistono ancora alcune limitazioni per l'imaging in vivo delle popolazioni neurali. Per esempio, la densità delle cellule basso richiesta per eseguire il rilevamento di successo singola cella di aNSCs rende impossibile impiegare saggi biochimici, come ad esempio Western blotting14. Inoltre, monitoraggio veloce dividendo popolazioni come astrociti cerebellari o cellule N2a è temporalmente limitata come è spesso troppo difficile tenere traccia di cellule come le culture nei pressi della confluenza. Inoltre, molti metodi di coltura, come pure le limitazioni biologiche inerente connesse con l'isolamento di cellule, spesso compromettono la vitalità cellulare per lunghi periodi, limitando la durata degli esperimenti dal vivo imaging. Infine, isolando le cellule dal loro ambiente naturale ha effetti sia positivi che negativi. Cellule isolate da loro nicchia fisiologica potrebbero non riuscire a ricevere segnali importanti che modulano il loro comportamento, mentre allo stesso tempo, rappresenta un potente mezzo per testare l'effetto di quei segnali individualmente nella progressione lignaggio di specifici neurale popolazioni.
Date le limitazioni sopra descritte, è chiaro che lo scenario metodologico perfetto sarebbe quella di eseguire esperimenti di tracciamento cellulari live imaging e singolo sotto condizioni fisiologiche normali in vivo. Tuttavia, le attuali tecniche sono in grado di seguire le singole cellule per lunghi periodi di tempo in regioni profonde del cervello2. Pertanto, il futuro dell'imaging dal vivo dovrebbe concentrarsi sul superamento di questa limitazione, con l'obiettivo di analizzare completamente la biologia cellulare di singole cellule in vivo con le più piccole possibili interferenze dell' ambiente fisiologico3.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Ringraziamo Beatriz Gascon per il suo lavoro di assistenza e arte nella Figura 1. Ringraziamo anche Dr. C. Norris per la sua assistenza. Il lavoro qui presentato è stato sostenuto da borse di ricerca, "Red de excelencia Consolider Ingenio spagnolo canale ionico iniziativa" (BFU2015-70067REDC), Fundación Ramon, BRADE-CM (S2013/ICE-2958), UCM-Santander (PR26/16-18B-3) e MEC (BFU2014-53654-P) Programma Areces Grant (PR2018/16-02). Felipe Ortega riconosce il Ramon y Cajal programma del Ministero spagnolo dell'economia e competitività (MEC: RYC-2013-13290).
Materiali
| Name | Company | Catalog Number | Comments |
| Poly-D-lysine | Sigma | P0899 | Working solution 0.02 mg mL-1 |
| 24 wells plate | Falcon | 352047 | |
| Dulbecco’s modified Eagle’s medium (DMEM): F12 Nutrient Mixture medium (-L Glutamine) | Invitrogen | 21331-020 | |
| DMEM High Glucose medium | Sigma | D6546 | |
| Bovine Serum Albumin | Sigma | A6003 | |
| Triton X-100 | Merck | 11869 | non-ionic surfactant |
| Mouse anti-β III Tubulin | Sigma | T8660 | |
| Rabbit anti-GFAP | DakoCytomation | Z0334 | |
| Mouse anti-α Tubulin | Sigma | T5168 | |
| Anti-Mouse FITC | Jackson Laboratories | 715-095-150 | |
| Anti-Rabbit Cy3 | Jackson Laboratories | 711-165-152 | |
| Brightfield/Phase contrast/fluoresence microscope | Nikon | TE-2000-E | |
| CFI PLAN FLUOR DLLL 10X objetives | Nikon | Ref 280MRH10101 | |
| CFI SUPER PLAN FLUOR ELWD AMD 20X objetives | Nikon | Ref 280MRH48230 | |
| pE-300 LED fluorescence | Cool LED | Ref Number 1981 | |
| 310M-201 Incubation system (temperature) | OKO-Lab | Serial Nº VOF007307 | |
| Pro-ScanII Motorized stage system | Prior | Serial Nº 60018 | |
| High precision microscope camera version 4.2 | ANDOR Zyla | VSC-03650 | |
| Specifc software for live imaging with timelapse module: NIS-Elements AR4.5 | Nikon | NIS-Elements AR4.5 -Hasp ID: 13CE819E | |
| OKO touch Incubation system (CO2) | OKO-lab | Serial number 1716 | |
| Murine Neuro-2a Neuroblastoma Cell line | ATCC | ATCCCCL131 | |
| HEPES buffer solution 1 M | Invitrogen | 15630-056 |
Riferimenti
- Conklin, E. G. The Mutation Theory From the Standpoint of Cytology. Science. 21 (536), 525-529 (1905).
- Ortega, F., Costa, M. R. Live Imaging of Adult Neural Stem Cells in Rodents. Front Neurosci. 10, 78 (2016).
- Skylaki, S., Hilsenbeck, O., Schroeder, T. Challenges in long-term imaging and quantification of single-cell dynamics. Nat Biotechnol. 34 (11), 1137-1144 (2016).
- Schroeder, T. Tracking hematopoiesis at the single cell level. Ann N Y Acad Sci. 1044, 201-209 (2005).
- Schroeder, T. Imaging stem-cell-driven regeneration in mammals. Nature. 453 (7193), 345-351 (2008).
- Etzrodt, M., Endele, M., Schroeder, T. Quantitative single-cell approaches to stem cell research. Cell Stem Cell. 15 (5), 546-558 (2014).
- Ortega, F., et al. Oligodendrogliogenic and neurogenic adult subependymal zone neural stem cells constitute distinct lineages and exhibit differential responsiveness to Wnt signalling. Nat Cell Biol. 15 (6), 602-613 (2013).
- Costa, M. R., et al. Continuous live imaging of adult neural stem cell division and lineage progression in vitro. Development. 138 (6), 1057-1068 (2011).
- Gascon, S., et al. Identification and Successful Negotiation of a Metabolic Checkpoint in Direct Neuronal Reprogramming. Cell Stem Cell. 18 (3), 396-409 (2016).
- Karow, M., et al. Reprogramming of pericyte-derived cells of the adult human brain into induced neuronal cells. Cell Stem Cell. 11 (4), 471-476 (2012).
- Kleiderman, S., et al. Conversion of Nonproliferating Astrocytes into Neurogenic Neural Stem Cells: Control by FGF2 and Interferon-gamma. Stem Cells. 34 (12), 2861-2874 (2016).
- Bunk, E. C., et al. Prox1 Is Required for Oligodendrocyte Cell Identity in Adult Neural Stem Cells of the Subventricular Zone. Stem Cells. 34 (8), 2115-2129 (2016).
- Aravantinou-Fatorou, K., et al. CEND1 and NEUROGENIN2 Reprogram Mouse Astrocytes and Embryonic Fibroblasts to Induced Neural Precursors and Differentiated Neurons. Stem Cell Reports. 5 (3), 405-418 (2015).
- Ortega, F., et al. Using an adherent cell culture of the mouse subependymal zone to study the behavior of adult neural stem cells on a single-cell level. Nat Protoc. 6 (12), 1847-1859 (2011).
- Heinrich, C., et al. Generation of subtype-specific neurons from postnatal astroglia of the mouse cerebral cortex. Nat Protoc. 6 (2), 214-228 (2011).
- Jimenez, A. I., et al. Potentiation of ATP calcium responses by A2B receptor stimulation and other signals coupled to Gs proteins in type-1 cerebellar astrocytes. Glia. 26 (2), 119-128 (1999).
- Hilsenbeck, O., et al. Software tools for single-cell tracking and quantification of cellular and molecular properties. Nat Biotechnol. 34 (7), 703-706 (2016).
- Ortega, F., Berninger, B., Costa, M. R. Primary culture and live imaging of adult neural stem cells and their progeny. Methods Mol Biol. 1052, 1-11 (2013).
- Kriegstein, A., Alvarez-Buylla, A. The glial nature of embryonic and adult neural stem cells. Annu Rev Neurosci. 32, 149-184 (2009).
- Yu, A. C., Lee, Y. L., Eng, L. F. Astrogliosis in culture: I. The model and the effect of antisense oligonucleotides on glial fibrillary acidic protein synthesis. J Neurosci Res. 34 (3), 295-303 (1993).
- Kretzschmar, K., Watt, F. M. Lineage tracing. Cell. 148 (1-2), 33-45 (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon