Method Article
Caratterizzazione biochimica e strutturale del carboidrato trasporto substrato-proteina-SP0092
In questo articolo
Riepilogo
È presentato un protocollo ottimizzato per l'esecuzione di un'ampia caratterizzazione biochimica e strutturale di una proteina carboidrato substrato da Streptococcus pneumoniae .
Abstract
Sviluppo di nuovi agenti antimicrobici e vaccini per Streptococcus pneumoniae (pneumococco) sono necessarie per fermare la rapida ascesa in ceppi più resistenti. Proteine che legano il substrato del carboidrato (SBPs) rappresentano obiettivi vitali per lo sviluppo di nuovi agenti antimicrobici e vaccini basati su proteine a causa della loro localizzazione extracellulare e la centralità dell'importazione del carboidrato per metabolismo pneumococco, rispettivamente. È descritto qui un razionalizzato protocollo integrato per svolgere una caratterizzazione completa di SP0092, che può essere estesa a altri carboidrati SBPs da pneumococco e altri batteri. Questa procedura può aiutare la progettazione basata su struttura di inibitori per questa classe di proteine. Presentati nella prima parte di questo manoscritto sono protocolli per l'analisi biochimica di analisi termica dello spostamento, multi angolo di dispersione della luce (MALS) e le dimensioni di cromatografia di esclusione (SEC), che ottimizzano la stabilità e omogeneità del campione diretto a prove di cristallizzazione e quindi migliorare la probabilità di successo. La seconda parte di questa procedura descrive la caratterizzazione dei cristalli SBP mediante una beamline di sincrotrone sintonizzabile lunghezza d'onda anomala diffrazione e protocolli di raccolta di dati per dati di misurazione che possono essere utilizzati per risolvere la proteina cristallizzata struttura.
Introduzione
S. pneumoniae (pneumococco) è un batterio gram-positivo asintomatico che risiedono nelle vie aeree superiori dell'apparato respiratorio umano con la capacità di migrare a nicchie normalmente sterile causando otite, polmonite, sepsi, setticemia, e la meningite1,2. Inoltre, l'infezione da pneumococco è la principale causa di polmonite comunità-acquistata, che sta contribuendo a un onere clinici ed economici in tutto il mondo3,4. Ceppi antibiotico-resistenti di S. pneumoniae sono diffuse in tutto il mondo e anche se un sette-Valente e un vaccino coniugato pneumococcico proteina di tredici-Valente hanno contribuito a riducono il tasso di resistenza agli antimicrobici, ceppi di ricambio da uso del vaccino sono emersi e hanno portato a crescenti richieste per la ricerca nello sviluppo di nuovi trattamenti per le malattie da pneumococco5,6,7,8.
Lo pneumococco è dipende da zuccheri importati dall'host come un carbonio fonte9,10; infatti dedica il 30% dei suoi macchinari di importazione per il trasporto di 32 diversi carboidrati11,12,13. Questi importatori includono almeno otto trasportatori di ABC13. In trasportatori di ABC, la SBPs extracellulare gioca un ruolo fondamentale nel determinare la specificità per il ligando e presentarla al trasportatore di membrana integrale per l'assorbimento nella cella. SBPs rappresentano destinazioni valide per la progettazione di nuovi vaccini e antimicrobici perché sono proteine di superficie e il loro ruolo vitale nei processi cellulari.
Caratterizzazione di proteine bersaglio e descrizione dettagliata delle caratteristiche strutturali, come ligando tasche e flessibilità tra domini, è possibile fornire uno strumento utile per droga basata su struttura design14,15. Cristallografia a raggi x è il metodo di scelta per la caratterizzazione strutturale di proteine al vicino a risoluzione atomica, ma il processo di cristallizzazione è imprevedibile, che richiede tempo e non sempre un successo. Metodi sistematici hanno migliorato il tasso di successo e fattori importanti sono la qualità del campione e la stabilità. Il tasso di successo di cristallizzazione è influenzato dalle proprietà chimiche della proteina e metodologia di preparazione del campione. L'effetto di questi possa essere valutato e informato dalla caratterizzazione biochimica16,17.
Un'ulteriore complicazione per progettazione basata sulla struttura è il problema di fase cristallografica, che dovrà essere affrontato. Come altre strutture proteiche sono diventati disponibili, molte strutture possono essere risolto con il metodo di sostituzione molecolare, che richiede una struttura omologa18. Come SBPs presentano una struttura di dominio flessibile, sostituzione molecolare potrebbe rivelarsi anche impegnativo19. Se un modello strutturale che è sufficientemente simile alla proteina bersaglio non è disponibile, una serie di tecniche consente di ottenere la fase sperimentale20. Tra questi, il metodo di singolo-lunghezza d'onda anomala dispersione (SAD) è emerso come la tecnica principale è stato ampiamente utilizzato per risolvere il problema di fase21. L'utilizzo del metodo triste è stato ulteriormente avanzato con miglioramenti in hardware e software, nonché strategie di raccolta dei dati per consentire la rilevazione e l'uso di deboli segnali anomali phasing-22,23, 24. Inoltre, avanza in metodi diretti per risolvere strutture delle macromolecole, che in passato ha richiesto dati di diffrazione a risoluzione atomica, possono ora essere utilizzati combinando ad esempio stereochimica conoscenza per come è implementato nel programma ARCIMOLDO25. Una recensione utile di metodi per risolvere il problema di fase in cristallografia è dato da Taylor26.
Qui presentiamo un protocollo razionalizzato per la caratterizzazione del trasporto dei carboidrati SBP, SP0092 di S. pneumoniae, integrando tecniche biochimiche e strutturali (Figura 1). Questo protocollo dettagliato fornisce un utile esempio test case delle strategie per migliorare il tasso di successo di studi strutturali su SBPs in generale, che si trovano in tutti i regni della vita. In particolare, il protocollo sottolinea l'importanza di caratterizzare lo stato oligomerico più stabile della proteina in soluzione in un metodo veloce ed efficace e permette l'identificazione delle specie migliori al follow-up per esperimenti di cristallizzazione. Anche se ci sono oltre 500 strutture SBP segnalati nell' Protein Data Bank27, sostituzione molecolare può essere difficile a causa della intrinseca natura flessibile dei due domini α/β, che sono collegati da una cerniera regione19. Così, la seconda parte del protocollo viene descritto utilizzando il metodo triste per l'eliminazione da ioni metallici associati, che è comune a SBPs, nonché l'incorporazione di selenometionina e uso di selenio (Se) in phasing triste.
Protocollo
Nota: la sequenza codificante per il quale viene eliminato il peptide segnale viene clonata nel vettore pOPINF seguendo un protocollo standard in fusione; la proteina nativa è espressa come una fusione di His-tag in cellule di Escherichia coli BL21 Rosetta 28 , 29. la selenometionina etichettata variante è espresso seguendo metodi standard secondo il produttore 30. Il ricombinante SBP è purificata come precedentemente descritto 31 , 32.
1. caratterizzazione biochimica
- Thermal shift assay
- 48 preparare soluzioni tampone con un pH che vanno da 4.0 a 9.5 e concentrazione di NaCl da 0 a 0,5 M e dispensare 40 µ l in una piastra a 96 pozzetti, come descritto Tabella 1 33.
- aggiungere a ogni ben 5 µ l della soluzione alla concentrazione di 1-2 mg/mL SBP.
- Aggiungere a ogni ben 5 µ l di colorante fluorescente per le proteine (Vedi Tabella materiali): 20x.
- Mescolare le soluzioni di pipettaggio, sigillare la piastra con una pellicola adesiva trasparente e spin per 2 min a 112 x g a temperatura ambiente.
- Eseguire l'esperimento su una macchina in tempo reale: impostare una rampa di temperatura di 3 ° C/min da 4 a 99 ° C con un tempo di attesa 10 s e leggere la fluorescenza ogni 0,5 ° C con eccitazione a 483 nm ed emissione a 568 nm.
- Analizzare le curve di emissione di fluorescenza per ogni condizione di buffer, che identifica la temperatura di fusione (T m) come il minimo del derivato.
Nota: Questa procedura dà una buona indicazione della migliore soluzione tampone per aiutare nel miglioramento della stabilità proteica. Scegliere l'intervallo di pH e concentrazione salina sulla base della condizione di buffer con il più alto T m e aggiungere 2,5% (v/v) di glicerolo e 0,5 mM di tris(2-carboxyethyl) fosfina (TCEP) per definire la soluzione tampone per i seguenti passaggi (SEC-buffer).
- Malles Venosta e analitici SEC
- eseguire un lavaggio due colonne volume della colonna di filtrazione gel pre-confezionati con acqua filtrata degassato (utilizzare una colonna di 24 mL di vasta gamma peso molecolare).
- Collegare la colonna per il rilevatore di dispersione della luce ed equilibrare la colonna con il SEC-buffer determinato del dosaggio di spostamento termico.
- Iniettare 100 µ l di SBP purificata a 5 mg/mL nel volume SEC equilibrato colonna e flusso una colonna del SEC-buffer.
- Controllare il cromatogramma di eluizione per singolo o picchi multipli ed esaminare i dati di scattering utilizzando il software di analisi, ottenendo la massa molare e l'indice di polidispersione per tutte le specie.
Nota: Per ogni picco corrispondente ad una specie oligomerica, è utile controllare la polidispersità indice calcolato come massa molare ponderata sulla massa di massa/molare ponderata sul numero di molecole (Mw/Mn). Un valore di polidispersione vicino a 1 indica un picco di monodispersed.
Nota: Una volta definite tutte le specie di oligomerizzazione, è utile studiare la loro dipendenza dalla concentrazione di proteine, come le più alte concentrazioni potrebbero favorire la formazione più grande oligomero. - Eseguire più analitica SEC esecuzioni; iniettare 100 µ l di SBP purificato all'aumento delle concentrazioni (0,1 - 10 mg/mL) nella colonna di filtrazione equilibrato gel (uso una colonna di 5 mL di vasta gamma peso molecolare) e un volume di colonna del SEC-buffer di flusso ogni volta.
- Controllare il cromatogramma di eluizione per singolo o picchi multipli e analizzare le intensità di assorbanza a 280 nm delle curve corrispondenti a differenti concentrazioni di proteina di partenza. Se le intensità relative delle diverse cime rimangono costanti, quindi nessun interconversione tra le specie oligomeriche è presente. Le intensità relative a differenti concentrazioni di variazione è un'indicazione dell'interconversione tra le diverse specie oligomeriche che dipende la concentrazione nella proteina.
Nota: Questo passaggio di caratterizzazione è fondamentale per definire quali specie sono più adatte per cristallizzazione. Infatti, sono più inclini a cristallizzare se sono in un costante stato di oligomerico a una concentrazione definita specie distinte monodispersed.
2. Preparazione della proteina e cristallizzazione
- preparativa SEC
- dopo un volume di una colonna di lavaggio con acqua filtrata degassato, equilibrare con SEC-buffer la colonna di filtrazione preparativa gel pre-confezionati (una vasta gamma molecolare peso 120 mL colonna).
- Iniettare 1-5 mL di SBP purificata a 5-50 mg/mL nel volume di una colonna equilibrato gel filtrazione colonna e flusso di buffer di SEC.
- Raccogliere il flusso-attraverso la colonna in frazioni di 1 mL.
- Piscina le frazioni corrispondenti ad un picco singolo monodispersi; spin il campione nei filtri di centrifugazione di 15 mL a 1.500 g e quantificare la concentrazione fino a raggiungere la concentrazione desiderata (in genere 50-100 mg/mL per SP0092).
- Cristallizzazione
- determinare la gamma di concentrazione ottimale per l'esperimento di cristallizzazione con il Test di pre-cristallizzazione come descritto dal costruttore:
- Dispensare 0,5 - 1,0 mL di ciascuno del test quattro cristallizzazione soluzioni in un serbatoio diverso di una seduta di 24 pozzetti goccia piatto cristallizzazione.
- Preparare le gocce di cristallizzazione mescolando 1 µ l di soluzione proteica con 1 µ l di soluzione di serbatoio, sigillare la piastra e incubare a temperatura ambiente per non meno di 1 h (risultati più affidabili sono ottenuti dopo incubazione overnight).
- Verifica la qualità di goccia per ogni concentrazione utilizzando un microscopio di ingrandimento di 10x.
Nota: Test concentrazione almeno tre diverse proteine: (I) la maggior parte delle gocce restanti chiaro indicano che la proteina è troppo diluita per cristallizzazione; (II) la presenza di precipitato pesante nella maggior parte delle gocce implica una concentrazione troppo elevata; e (III) un'equilibrata presenza di gocce chiare sia precipitato (meglio se leggero) è una buona indicazione che la concentrazione sperimentata è favorevole per la cristallizzazione.
- Preparare la goccia di seduta di 96 pozzetti piastre cristallizzazione; Pipettare 100 µ l di soluzione di cristallizzazione commerciali differenti in ciascun serbatoio ben 34 , 35 , 36 , 37.
- erogare 100 nL del campione della proteina alla concentrazione definita in precedenza (punto 1.1.6) in tutti i pozzetti di goccia di cristallizzazione utilizzando un sistema robotico cristallizzazione.
- Erogare 100 nL del serbatoio diverso soluzioni per la cristallizzazione corrispondente goccia pozzi per mescolare con proteina erogata al punto 2.2.3. Sigillare la piastra di cristallizzazione per evitare l'evaporazione e abilitare l'equilibramento della cristallizzazione goccia con il serbatoio.
- Verifica periodicamente le gocce (inizialmente ogni 1-2 giorni, poi ogni settimana) utilizzando un 10x (almeno) microscopio per valutare la crescita e la formazione di cristalli.
Nota: Un sistema di imaging automatizzato per cristallizzazione della proteina può essere utilizzato per gocce di visualizzazione e catturando.
- determinare la gamma di concentrazione ottimale per l'esperimento di cristallizzazione con il Test di pre-cristallizzazione come descritto dal costruttore:
- cristallo montaggio
- preparare la soluzione di crioprotettore aggiungendo 25% (v/v) di glicerolo (concentrazione finale) per la condizione di cristallizzazione (sostituendo così il 25% dell'acqua nella fase iniziale miscela) 38.
- Riempire una schiuma dewar con azoto liquido e posto la parte di recinzione del campione di un uni-puck nel dewar. Attendere che si raffreddi alla temperatura dell'azoto liquido.
- Tagliare e rimuovere la sigillatura con nastro adesivo la piastra di cristallizzazione sopra la goccia dove si formano i cristalli.
- Mettere una goccia di 1 µ l di soluzione crioprotettore su un coverslide posizionato in prossimità della goccia destinazione.
- Trasferire il cristallo selezionato dal menu originale per il calo di crioprotettore soluzione utilizzando un nylon cryo-loop su una base standard della colonna vertebrale, montata su una bacchetta magnetica 39 , 40.
- Trasferire velocemente il cristallo dal menu crioprotettore all'azoto liquido, ponendo il ciclo nella prima posizione vuota del supporto del campione di uni-puck.
- Ripetere (dal passo taglio nastro sigillante) fino a quando tutti i cristalli desiderati vengono raccolte e memorizzati nel supporto del campione ed uni-puck.
- Posto il uni-puck basare il uni-puck utilizzando la bacchetta di puck e prendere il disco per la beamline (in condizioni di azoto liquido).
Attenzione: A bassissima temperatura! - Utilizzare il puck e il cryo-pinze strumento di caricamento di dewar per caricare il uni-puck(s) nel robot beamline campione changer dewar. Il portacampioni uni-puck si staccherà dal coperchio base lasciando i portacampioni ciclo verticale nel robot esempio dewar e così esposta all'azoto liquido e accessibile al robot changer campione.
- Caratterizzazione di cristallo
Nota: I cristalli raccolti allora possono essere selezionati per qualità di diffrazione usando una sorgente di raggi x di laboratorio o presso una sorgente di raggi x di sincrotrone (SR). Beamlines di cristallografia macromolecolare (MX) forniscono una fonte di energia sintonizzabile per sfruttare la diffrazione anomala per la soluzione della struttura. Nella sezione seguente, una serie di istruzioni di lavoro in linea con le capacità sperimentale di Diamond Light Source MX beamlines è proposto, ma queste linee guida possono anche essere adattate per MX beamlines presso altri sincrotroni in tutto il mondo.- Come primo passo, è utile confermare o identificare scatterers anomala nel cristallo. Ciò può essere convenientemente ottenuto misurando un spettro di fluorescenza di raggi x dal cristallo. In primo luogo garantire l'energia dei raggi x della beamline è impostato abbastanza alto per eccitare tutti gli elementi in genere previsti per cristalli macromolecolari (14 keV o superiore).
- Utilizzare il software di controllo beamline per selezionare il campione per essere montato dal robot changer campione; il ciclo verrà automaticamente centrato nel fascio di raggi x e la centratura di cristallo può essere confermata manualmente utilizzando il software di controllo beamline.
- Registrare uno spettro di fluorescenza di raggi x utilizzando il software di controllo beamline: l'utente seleziona un tempo di esposizione e avvia la misurazione (impostazione di spettro di fluorescenza/fluorescenza, → esegue, Figura 2A). Il software di controllo beamline posiziona automaticamente i raggi x, rivelatore a fluorescenza nel posto e determina l'incidente minimo flusso di raggi x per ottenere un segnale leggibile sul rivelatore di fluorescenza. Lo spettro acquisito viene poi analizzato in modo automatico con picchi di emissione montati naturali elementi biologici. Queste routine sono disponibili all'interno della beamline controllo software interfaccia utente grafica (GUI) - GDA (www.opengda.org) presso la nostra azienda e sincrotroni in tutta Europa 41 , 42.
- Se gli elementi adatti sono identificati che può essere sfruttata per un esperimento di diffrazione anomala, eseguire una scansione di energia bordo assorbimento dei raggi x per determinare la lunghezza d'onda ottimale per la raccolta di dati di diffrazione anomala dal cristallo: sulla beamline software di controllo, selezionare l'elemento e iniziare la scansione (impostazione di analisi di fluorescenza/fluorescenza, → nome di Atom, → esegue, Figura 2B).
Nota: Per un esperimento di diffrazione anomala singolo, il picco del bordo di assorbimento viene utilizzato per ottimizzare la dispersione anomala. Considerazioni sperimentali per l'esecuzione di un esperimento di diffrazione anomala di multi-lunghezza d'onda sono state precedentemente dettagliate 43. - Per determinare i parametri di cella di cristallo centrale, simmetria e limite di diffrazione, misurare tre modelli di diffrazione di raggi x ad intervalli di 45° con il metodo di oscillazione (raccolta dati/Screening → eseguire attuale, Figura 2). Il software di controllo beamline fornisce all'utente una scelta dell'angolo di oscillazione e tempo di esposizione e la possibilità di impostare la percentuale di trasmissione del fascio di raggi x. Per uno screening di cristallo standard, si consiglia un angolo di oscillazione di 0,5 °, un tempo di esposizione di 0,5 s e la trasmissione di fascio di raggi x 5%. Le immagini di diffrazione misurato automaticamente vengono analizzate tramite la pipeline di EDNA e restituire un set di strategie per la raccolta di un set di dati completo 22.
- Dei raggi x raccolta dati
Nota: l'utente può selezionare per raccogliere dati in modalità oscillazione standard o fascio inversa, che permette di Friedel compagni essere registrato nelle vicinanze nel tempo e dose di raggi x e con circa lo stesso comportamento di assorbimento consentendo una più accurata misurazione delle differenze anomale. Quest'ultimo è utile soprattutto se piccole differenze anomale sono attesi e/o i campioni sono radiazione sensibili nello svolgimento di un esperimento di diffrazione anomala. Considerazioni sulle migliori strategie di raccolta di dati da utilizzare sono state ampiamente Recensito il 22 , 23 , 44 , 45 , 46 , 47.- utilizzando il software di controllo beamline, importare i parametri della raccolta dati come suggerito dal programma di strategia che fornirà:
i. inizio posizione dell'asse di rotazione ω
ii. Larghezza di oscillazione della rotazione ω-asse angolo per ogni immagine di diffrazione
iii. Tempo di esposizione per ogni immagine di diffrazione
iv. Numero di immagini per un set di dati completo (indirettamente definendo l'angolo di rotazione totale ω-asse per la raccolta di dati interi)
v. percentuale di attenuazione del fascio di raggi x per evitare danni di sovraesposizione e radiazione
Nota: alla nostra istituzione, per esegue la raccolta dei dati, le pipeline software destinati all'elaborazione automatizzata dei dati raccolti sono ben stabilite: (i) la diffrazione di dati sono ridotti (indicizzata, integrato e in scala) tramite la pipeline di xia2, che genera i file mtz di riflessione per l'utente come input per soluzione e affinamento graduale e struttura 48. (ii) quando viene rilevato un segnale anomalo significativo durante l'analisi dei dati, una prima struttura automatizzata veloce soluzione pipeline (fast_ep) utilizzando IGNAZIOXAXA tenta di risolvere la sottostruttura di atomi pesanti di sperimentale phasing, fornendo una mappa di densità dell'elettrone graduale ove possibile. Una seconda pipeline di soluzione più completa struttura si impegna automaticamente tentativi di risolvere e costruire la llare utilizzando software indipendenti suites 49 , 50 , 51 , 52; nei casi in cui questo è successo, l'utente verrà fornito con una mappa di densità di elettrone e modello iniziale. Questo fornisce la base per l'utente completare la raffinatezza e convalidare il modello con la suite di software cristallografici di scelta.
- utilizzando il software di controllo beamline, importare i parametri della raccolta dati come suggerito dal programma di strategia che fornirà:
Risultati
Questo protocollo integrato ha dimostrato di avere successo con quattro (due pubblicati e due inediti strutture) di sei carboidrati proteine obiettivi vincolanti da pneumococco analizzati finora32,53. In questa sezione presentiamo la caratterizzazione biochimica e strutturale di SP0092 come un risultato rappresentativo per guidare gli studi strutturali di SBPs in generale.
Dopo il SP0092 di SBP è stato espresso e purificato come definito in precedenza32, la proteina purificata è stata analizzata per la stabilità di buffer usando un'analisi di spostamento termico: SP0092 esibisce un aumento Tm a pH 6,5 e in presenza di NaCl in 0 - 0.2 M intervallo di concentrazione (Figura 3A). Alla luce di ciò, la soluzione tampone per la procedura seguente è stata definita come: 0,02 M MES pH 6.5, 0,2 M NaCl, 2.5% (v/v) glicerolo, 0,5 mM TCEP. La massa molare assoluta degli Stati diversi oligomerizzazione di SP0092 è stata misurata da MALS accoppiato al SEC misurazione un peso molecolare di 187,2, 140,8, 97,0 e 49,4 kDa, corrispondente alla specie tetramerica, trimerici, dimeriche e monomeriche, rispettivamente ( Figura 3B). L'analisi del profilo SEC a diluizioni differenti della proteina ha rivelato che l'oligomerizzazione è innescato da concentrazione aumentata nella proteina, suggerendo che gli oligomeri più grandi sono più stabili alle più alte concentrazioni di tipicamente utilizzati in cristallizzazione. Infatti, più grande specie oligomeriche purificata diretto ai processi di cristallizzazione, cristalli di proteina prodotta con successo mentre il monomero specie non ha fatto.
L'ottimizzata cristalli ottenuti dal nativo e Se-metionina con etichettata forme di SP0092 furono caratterizzati dalla diffrazione dei raggi x. Misura della fluorescenza a raggi x da questi cristalli ha rivelato in entrambi i casi picchi di emissione per Zn essendo legato alle proteine, mentre Se è stata rilevata solo per i cristalli di Se-metionina come previsto. Più tardi, sono state eseguite scansioni di assorbimento di raggi x ai bordi Se e Zn, che ha fornito i dati sperimentali diretti per sintonizzare la lunghezza d'onda dei raggi x incidente per i bordi di assorbimento di raggi x di Zn o Se presentano nei cristalli per massimizzare il segnale anomalo ottenibili dai dati risultanti (Figura 4A–B).
Dopo tre modelli di diffrazione a bassa trasmissione di misura, un insieme completo di dati anomalo è stato ottenuto utilizzando la strategia di raccolta dati suggerita da EDNA (Figura 4). Il segnale anomalo presente innesca la pipeline di fasatura automatica presso la beamline per determinare la sub- struttura e basato sulle iniziali fasi sperimentali derivate, produce iniziale mappe e modelli, che possono poi essere raffinati e convalidati (Figura 4 ).
In sintesi, potenziali insidie della tecnica sono incentrate principalmente sulla qualità e disponibilità di cristallo. Ottimizzazione delle condizioni di buffer per migliorare la stabilità proteica, nonché individuazione dello stato oligomerico più adatto della proteina da utilizzare, quando più di un oligomero è identificato in soluzione, può ridurre il rischio di fallimento presso la cristallizzazione fase. Sfruttando l'uso di ioni metallici associati identificato in una fase precoce può accelerare la soluzione della struttura ed evitare inutili produzione di selenometionina etichettata proteine quando sostituzione molecolare metodi falliscono.
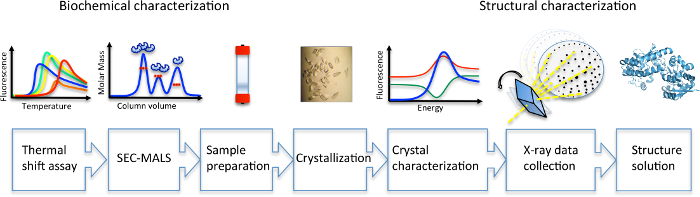
Figura 1. Diagramma di flusso di lavoro per la caratterizzazione biochimica e strutturale del carboidrato SBPs. Clicca qui per visualizzare una versione più grande di questa figura.
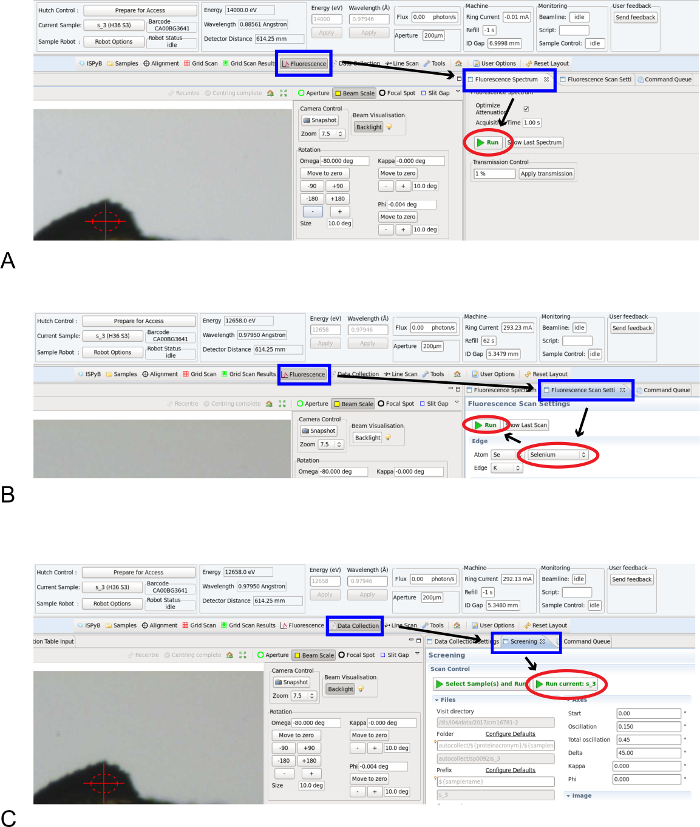
Figura 2. Caratterizzazione di cristallo in GDA. (A) Screenshot della scheda per il controllo di fluorescenza a raggi x del software di controllo beamline GDA. (B) Screenshot della scheda di controllo a raggi x bordo-scansione dei energia GDA. (C) schermata della scheda dati della collezione crystal screening GDA. Clicca qui per visualizzare una versione più grande di questa figura.

Nella figura 3. Caratterizzazione biochimica di SP009239-491. (A) grafico 3D-superficie tramando la temperatura di fusione di SP009239-491 in funzione del pH e della concentrazione di NaCl della soluzione tampone. (B) SEC e Malles Venosta risultati per SP009239-491. Assorbimento a 280 nm è indicata in blu. Le masse molari degli Stati diversi oligomerizzazione sono mostrate in rosso. Pannello (B) è stato modificato da32. Clicca qui per visualizzare una versione più grande di questa figura.
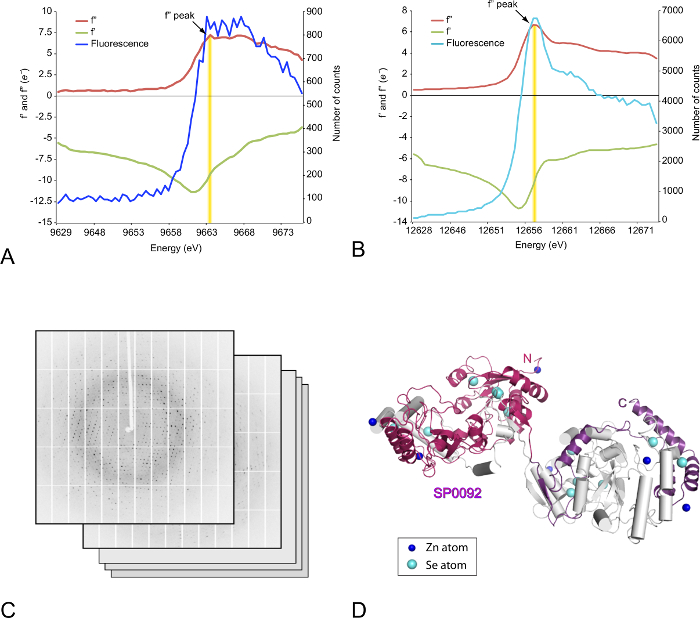
Nella figura 4. Caratterizzazione strutturale di SP009239-491. (A) e l'energia di assorbimento di raggi x (B) scansione al bordo dello Zn e del Se per SP009239-491 cristalli. La fluorescenza misurata dal Zn e Se contenenti cristalli è mostrata in blu e ciano, il calcolato f' e f "dispersione anomala frazioni sono in verde e rosso, rispettivamente. (C) esempi di diffrazione a raggi x raccolti da SP009239-491 cristalli. (D) del fumetto di rappresentazione della struttura del dimero SP009239-491 . Uno protomer è colorato di bianco e l'altro magenta (residui 39-366) e viola (residui 367-491), e gli atomi di dispersione anomala vengono mostrati come sfere blu e ciano per Zn e Se, rispettivamente. Clicca qui per visualizzare una versione più grande di questa figura.
| 0.1 acido citrico M a pH 4.0 | 0.1 acido citrico M a pH 4,5 | 0,1 fosfato M a pH 5.0 | 0.1 M citrato pH 5.5 | 0,1 Bis-tris-M a pH 6 | 0.1 M a pH ADA 6.5 | 0,1 MOPS M a pH 7 | 0.1 M HEPES, pH 7.5 | 0.1 M a pH imidazolo 8.0 | 0.1 M Tris-HCl pH 8,5 | 0.1 M a pH CHES 9 | 0.1 M a pH CHES 9.5 | |
| NaCl 0 M | 40 ΜL | 40 ΜL | 40 ΜL | 40 ΜL | 40 ΜL | 40 ΜL | 40 ΜL | 40 ΜL | 40 ΜL |
Tabella 1. Composizione di buffer per analisi termica dello spostamento.
Discussione
In questo articolo, descriviamo e convalidare un protocollo integrato per la caratterizzazione biochimica e strutturale del carboidrato SBPs con una particolare enfasi sulle proteine da S. pneumoniae. Tuttavia, questo utilizzabile come una procedura standard per l'analisi di altri SBPs da organismi differenti e anche altre proteine solubili non correlati.
La prima parte del protocollo è focalizzata sulla fornitura di informazioni biochimiche sulla stabilità proteica e struttura quaternaria, che può essere sfruttata nella preparazione dei campioni della proteina per la cristallizzazione. Nella sezione analisi di spostamento termico, descriviamo solo il pH e le variazioni delle concentrazioni di NaCl per mantenere la natura generale di questa procedura. Nonostante questo, molte altre condizioni di buffer possono essere testati in un modo simile, ad esempio, compreso qualsiasi composto chimico utilizzato come additivo stabilizzante: in particolare l'effettivi ligandi che si legano a un specifico SBP sono notevolmente efficaci nell'aumentare la Tm di pochi gradi Celsius31. In alcuni casi, le curve di denaturazione possono essere mal definite a causa del segnale basso o uno sfondo alta fluorescenza, che è causato da aggregazione proteica o parziale svolgimento. Per evitare questo, una titolazione di proteina: tintura può essere eseguita per ottimizzare le curve di denaturazione poco chiare. Se non si ottengono miglioramenti, vari additivi che possono migliorare la stabilità della proteina di screening è consigliato e schermi adatti sono stati descritti in precedenza54.
In genere, la maggior parte SBPs sono monomerico nel loro ambiente naturale, ma come illustrato di seguito può verificarsi multimerization alle più alte concentrazioni utilizzate in esperimenti di cristallizzazione, quindi la caratterizzazione del comportamento di oligomerizzazione fornita da Malles Venosta e SEC è essenziale per valutare lo stato di oligomerizzazione monodispersi stabile più favorevole per la cristallizzazione. Tuttavia, è difficile prevedere l'effetto di diverse sostanze chimiche incluse nella condizione cristallizzazione sul comportamento di oligomerizzazione delle proteine. Se l'esame SEC e Malles Mostra vasta aggregazione del campione della proteina, vi consigliamo i seguenti per ridurre la probabilità che si verifichi questo: utilizzare esempio di proteina fresco (non congelare-sciolto) ed espandere l'analisi di stabilizzazione eseguita con termica analisi di spostamento, test di eventuali additivi e detergenti delicati come ultima risorsa, per ridurre al minimo l'aggregazione. In questa carta, presentiamo le linee guida di base per la cristallizzazione mediante lo screening di matrice sparsa di alto-rendimento commerciale cristallizzazione per mantenere la natura generale del presente protocollo. Tuttavia, ottenere cristalli della proteina di diffrazione di raggi x ad alta risoluzione potrebbe essere necessario iterativa messa a punto per ottimizzare le condizioni di cristallizzazione rispetto alla concentrazione precipitante, pH, aggiunta di additivi chimici, temperature diverse, e altri fattori mutevoli dinamiche di equilibrio tra il cristallo goccia e serbatoio16,17.
La seconda parte del protocollo descrive la caratterizzazione dei cristalli della proteina al fine di definire la strategia ottimale per la raccolta di dati di diffrazione di raggi x con un focus specifico sull'acquisizione di dati anomali phasing-triste. Anche se SBPs mantenere una simile architettura generale (e ci sono molte strutture 3D depositati potenzialmente utilizzabile come modelli di partenza), graduale di queste proteine con il metodo di sostituzione molecolare non è sempre semplice a causa della variabilità della elementi di struttura secondaria e la flessibilità intrinseca di queste proteine. Quindi, proponiamo il metodo triste ed evidenziare che queste proteine possono già avere intrinsecamente legata metalli o legame infatti non specifico dei metalli dalle condizioni di buffer di cristallizzazione, che possono fornire una gamma di elementi anomali diffrangano come un passo standard nel nostro protocollo generale.
In conclusione, il presente protocollo definisce un flusso di lavoro guidata standard di procedure che consentano la descrizione dettagliata delle caratteristiche biochimiche e strutturali di SBPs che può essere sfruttata per aumentare il tasso di successo di determinazione di struttura, nonché di accelerare la caratterizzazione strutturale di SBPs in generale.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Riconosciamo OPPF-UK per assistenza nella clonazione, Gemma Harris per SEC-centri commerciali e gli scienziati di beamlines I03 e I04 presso Diamond Light Source.
Materiali
| Name | Company | Catalog Number | Comments |
| SelenoMethionine Medium Complete | Molecular Dimensions | MD12-500 | Based on a synthetic M9 minimal media supplemented with glucose, vitamins and amino acids with the exception of L-methionine. Other equivalent products are commercially available by other companies. |
| MicroAmp Optical 96-Well plate | Applied Biosystems | 4306737 | The Applied Biosystems MicroAmp Optical 96-Well Reaction Plate with Barcode is optimized to provide unmatched temperature accuracy and uniformity for fast, efficient PCR amplification. This plate, constructed from a single rigid piece of polypropylene in a 96-well format, is compatible with Applied Biosystems® 96-Well Real-Time PCR systems and thermal cyclers. |
| SYPRO Orange | Molecular Probes | S6651 | SYPRO Orange Protein Gel Stain is a sensitive, ready-to-use fluorescent stain for proteins in 1D gels. Quite universal and well-established protein dye for hydrophobic regions. |
| MicroAmp Optical Adhesive film | Applied Biosystems | 4311971 | The Applied Biosystems MicroAmp Optical Adhesive Film reduces the chance of well-to-well contamination and sample evaporation when applied to a microplate. It is ideal for optical measurement, because it gives low background. |
| 7500 Fast Real-time PCR System | Applied Biosystems | 4362143 | The Applied Biosystems 7500 Fast Real-Time PCR System enables standard 96-well format high speed thermal cycling, significantly reducing your run time for quantitative real-time PCR applications, delivering results in about 30 minutes. The Upgrade Kit is available to upgrade a standard 7500 Real-Time PCR System to the Fast Configuration via a field service installation. Other RT-PCR machine can be used. Data export not easy for the old data analysis software. |
| Superdex 200 increase 10/300 GL column | GE Healthcare | 28990944 | Superdex 200 Increase 10/300 GL is a versatile, prepacked column for size exclusion chromatography in small-scale (mg) preparative purification as well as for characterization and analysis of proteins with molecular weights between 10 000 and 600 000, such as antibodies. Optimal separation ideal for high resolution biophysical techniques. |
| DAWN HELEOS II | Wyatt | DAWN HELEOS II is the premier Multi-Angle static Light Scattering (MALS) detector for absolute characterization of the molar mass and size of macromolecules and nanoparticles in solution. The DAWN offers the highest sensitivity, the widest range of molecular weight, size and concentration, and the largest selection of configurations and optional modules for enhanced capabiliites. Other MALS detecting systems from other companies apart from Wyatt have not been tested, so no additional feedback can be provided. | |
| Superdex 200 5/150 GL | GE Healthcare | 28906561 | Superdex 200 are prepacked size exclusion chromatography columns for high-resolution small-scale preparative and analytical separations of biomolecules. Superdex 200 has a separation range for molecules with molecular weights between 10 000 and 600 000. The peak separation is not as optimal as for the "increase" version but this model is ideal for the standard day by day use. |
| HiLoad 16/600 Superdex 200 pg | GE Healthcare | 28989335 | HiLoad 16/600 Superdex 200 prep grade are prepacked XK columns designed for high-resolution preparative gel filtration chromatography. |
| 24 Well "Big" Sitting Drop Crystallization Plate | MiTeGen | XQ-P-24S-A | The 24 well "big" crystallization plate is used mainly for protein crystal screening by sitting drop vapor diffusion techniques, and for crystallization condition optimization. It has quite large reservoir well and sample container, which favor manual handling and big sized protein crystal growth. In addition, its flat surfaces are easily to be sealed by transparent tape or cover slips. |
| PCT Pre-Crystallization Test | Hampton | HR2-140 | The PCT Pre-Crystallization Test is used to determine the appropriate protein concentration for crystallization screening. |
| 96 Well CrystalQuick | Greiner bio-one | 6098xx | Square-well plates have three crystallization wells per reservoir, making it possible to test 288 samples per plate. Generally used only for the inital screening because their squared edge make crystals fishing difficult. |
| Uni-Puck | Molecular Dimensions | MD7-601 | The Universal V1-Puck (Uni-puck) is a sample pin storage and shipping container for use with the majority of automated sample mounting systems worldwide - includes the ACTOR, SAM and CATS systems amongst others (Diamond, Soleil, SPring-8, Photon Factory, CLSI and across the USA) |
| Standard Foam Dewar | Molecular Dimensions | MD7-35 | 5.7" diameter by 2.8" deep. 800mL capacity. |
| Mounted CryoLoop - 20 micron | Hampton | HR4-955 | Mounted CryoLoops with 20 micron diameter nylon. These nylon loops are bonded to hollow, stainless steel MicroTubes™ that are used to mount, freeze, and secure the crystal during cryocrystallographic procedures and X-ray data collection. Different sizes exist and they can be adapted in lenght. They are quite versatile tools. |
| CryoWand | Molecular Dimensions | MD7-411 | |
| Puck dewar loading tool | Molecular Dimensions | MD7-607 | This tool is used to separate uni-pucks to load them into the robot dewar. It consists of two pieces: a Teflon tube part and a metal rod part. |
Riferimenti
- Austrian, R. Prevention of pneumococcal infection by immunization with capsular polysaccharides of Streptococcus pneumoniae: current status of polyvalent vaccines. J Infect Dis. 136, S38-S42 (1977).
- Bogaert, D., De Groot, R., Hermans, P. W. Streptococcus pneumoniae colonisation: the key to pneumococcal disease. Lancet Infect Dis. 4 (3), 144-154 (2004).
- van Mens, S. P., et al. Longitudinal analysis of pneumococcal antibodies during community-acquired pneumonia reveals a much higher involvement of Streptococcus pneumoniae than estimated by conventional methods alone. Clin Vaccine Immunol. 18 (5), 796-801 (2011).
- Weycker, D., Strutton, D., Edelsberg, J., Sato, R., Jackson, L. A. Clinical and economic burden of pneumococcal disease in older US adults. Vaccine. 28 (31), 4955-4960 (2010).
- Doern, G. V. Antimicrobial use and the emergence of antimicrobial resistance with Streptococcus pneumoniae in the United States. Clin Infect Dis. 33, S187-S192 (2001).
- Vasoo, S., et al. Increasing antibiotic resistance in Streptococcus pneumoniae colonizing children attending day-care centres in Singapore. Respirology. 16 (8), 1241-1248 (2011).
- Black, S., et al. Efficacy, safety and immunogenicity of heptavalent pneumococcal conjugate vaccine in children. Northern California Kaiser Permanente Vaccine Study Center Group. Pediatr Infect Dis J. 19 (3), 187-195 (2000).
- Hanage, W. P., et al. Diversity and antibiotic resistance among nonvaccine serotypes of Streptococcus pneumoniae carriage isolates in the post-heptavalent conjugate vaccine era. J Infect Dis. 195 (3), 347-352 (2007).
- Burnaugh, A. M., Frantz, L. J., King, S. J. Growth of Streptococcus pneumoniae on human glycoconjugates is dependent upon the sequential activity of bacterial exoglycosidases. J Bacteriol. 190 (1), 221-230 (2008).
- King, S. J. Pneumococcal modification of host sugars: a major contributor to colonization of the human airway?. Mol Oral Microbiol. 25 (1), 15-24 (2010).
- Tettelin, H., et al. Complete genome sequence of a virulent isolate of Streptococcus pneumoniae. Science. 293 (5529), 498-506 (2001).
- Buckwalter, C. M. Pneumococcal carbohydrate transport: food for thought. Trends Microbiol. 20 (11), 517-522 (2012).
- Bidossi, A., et al. A functional genomics approach to establish the complement of carbohydrate transporters in Streptococcus pneumoniae. PLoS One. 7 (3), e33320 (2012).
- Zheng, X., Gan, L., Wang, E., Wang, J. Pocket-based drug design: exploring pocket space. AAPS J. 15 (1), 228-241 (2013).
- Singh, S., Malik, B. K., Sharma, D. K. Molecular drug targets and structure based drug design: A holistic approach. Bioinformation. 1 (8), 314-320 (2006).
- Dessau, M. A., Modis, Y. Protein crystallization for X-ray crystallography. J Vis Exp. (47), (2011).
- Bergfors, T. . Protein crystallization: techniques, strategies, and tips. A laboratory manual. , (1999).
- Scapin, G. Molecular replacement then and now. Acta Crystallogr D Biol Crystallogr. 69 (Pt 11), 2266-2275 (2013).
- Mao, B., Pear, M. R., McCammon, J. A., Quiocho, F. A. Hinge-bending in L-arabinose-binding protein. The "Venus's-flytrap" model. J Biol Chem. 257 (3), 1131-1133 (1982).
- McCoy, A. J., Read, R. J. Experimental phasing: best practice and pitfalls. Acta Crystallogr D Biol Crystallogr. 66 (Pt 4), 458-469 (2010).
- Dodson, E. Is it jolly SAD?. Acta Crystallogr D Biol Crystallogr. 59 (Pt 11), 1958-1965 (2003).
- Incardona, M. F., et al. EDNA: a framework for plugin-based applications applied to X-ray experiment online data analysis. J Synchrotron Radiat. 16 (Pt 6), 872-879 (2009).
- Popov, A. N., Bourenkov, G. P. Choice of data-collection parameters based on statistic modelling. Acta Crystallogr D Biol Crystallogr. 59 (Pt 7), 1145-1153 (2003).
- Terwilliger, T. C., et al. Can I solve my structure by SAD phasing? Planning an experiment, scaling data and evaluating the useful anomalous correlation and anomalous signal. Acta Crystallogr D Struct Biol. 72 (Pt 3), 359-374 (2016).
- Millan, C., Sammito, M., Uson, I. Macromolecular ab initio phasing enforcing secondary and tertiary structure. IUCrJ. 2 (Pt 1), 95-105 (2015).
- Taylor, G. L. Introduction to phasing. Acta Crystallogr D Biol Crystallogr. 66 (Pt 4), 325-338 (2010).
- Berman, H. M., et al. The Protein Data Bank. Nucleic Acids Res. 28 (1), 235-242 (2000).
- Bird, L. E., et al. Green fluorescent protein-based expression screening of membrane proteins in Escherichia coli. J Vis Exp. (95), e52357 (2015).
- Berrow, N. S., et al. A versatile ligation-independent cloning method suitable for high-throughput expression screening applications. Nucleic Acids Res. 35 (6), e45 (2007).
- Hendrickson, W. A., Horton, J. R., LeMaster, D. M. Selenomethionyl proteins produced for analysis by multiwavelength anomalous diffraction (MAD): a vehicle for direct determination of three-dimensional structure. EMBO J. 9 (5), 1665-1672 (1990).
- Culurgioni, S., Harris, G., Singh, A. K., King, S. J., Walsh, M. A. Structural Basis for Regulation and Specificity of Fructooligosaccharide Import in Streptococcus pneumoniae. Structure. , (2016).
- Culurgioni, S., Tang, M., Walsh, M. A. Structural characterization of the Streptococcus pneumoniae carbohydrate substrate-binding protein SP0092. Acta Crystallogr F Struct Biol Commun. 73 (Pt 1), 54-61 (2017).
- Ericsson, U. B., Hallberg, B. M., Detitta, G. T., Dekker, N., Nordlund, P. Thermofluor-based high-throughput stability optimization of proteins for structural studies. Anal Biochem. 357 (2), 289-298 (2006).
- Wooh, J. W., Kidd, R. D., Martin, J. L., Kobe, B. Comparison of three commercial sparse-matrix crystallization screens. Acta Crystallogr D Biol Crystallogr. 59 (Pt 4), 769-772 (2003).
- Gorrec, F. Protein crystallization screens developed at the MRC Laboratory of Molecular Biology. Drug Discov Today. 21 (5), 819-825 (2016).
- Jancarik, J., Scott, W. G., Milligan, D. L., Koshland, D. E., Kim, S. H. Crystallization and preliminary X-ray diffraction study of the ligand-binding domain of the bacterial chemotaxis-mediating aspartate receptor of Salmonella typhimurium. J Mol Biol. 221 (1), 31-34 (1991).
- Newman, J., et al. Towards rationalization of crystallization screening for small- to medium-sized academic laboratories: the PACT/JCSG+ strategy. Acta Crystallogr D Biol Crystallogr. 61 (Pt 10), 1426-1431 (2005).
- Garman, E. F., Mitchell, E. P. Glycerol concentrations required for cryoprotection of 50 typical protein crystallization solutions. Journal of Applied Crystallography. 29 (5), 584-587 (1996).
- Teng, T. -. Y. Mounting of crystals for macromolecular crystallography in a free-standing thin film. Journal of Applied Crystallography. 23 (5), 387-391 (1990).
- Cipriani, F., et al. Automation of sample mounting for macromolecular crystallography. Acta Crystallogr D Biol Crystallogr. 62 (Pt 10), 1251-1259 (2006).
- Gabadinho, J., et al. MxCuBE: a synchrotron beamline control environment customized for macromolecular crystallography experiments. J Synchrotron Radiat. 17 (5), 700-707 (2010).
- Leonard, G. A., et al. Online collection and analysis of X-ray fluorescence spectra on the macromolecular crystallography beamlines of the ESRF. Journal of Applied Crystallography. 42 (2), 333-335 (2009).
- Walsh, M. A., Evans, G., Sanishvili, R., Dementieva, I., Joachimiak, A. MAD data collection - current trends. Acta Crystallogr D Biol Crystallogr. 55 (Pt 10), 1726-1732 (1999).
- Dauter, Z. Data-collection strategies. Acta Crystallogr D Biol Crystallogr. 55 (Pt 10), 1703-1717 (1999).
- Finke, A. D., et al. Advanced Crystallographic Data Collection Protocols for Experimental Phasing. Methods Mol Biol. 1320, 175-191 (2016).
- Gonzalez, A. Optimizing data collection for structure determination. Acta Crystallogr D Biol Crystallogr. 59 (Pt 11), 1935-1942 (2003).
- Leslie, A. G. The integration of macromolecular diffraction data. Acta Crystallogr D Biol Crystallogr. 62 (Pt 1), 48-57 (2006).
- Winter, G., Lobley, C. M., Prince, S. M. Decision making in xia2. Acta Crystallogr D Biol Crystallogr. 69 (Pt 7), 1260-1273 (2013).
- Sheldrick, G. M. Experimental phasing with SHELXC/D/E: combining chain tracing with density modification. Acta Crystallogr D Biol Crystallogr. 66 (Pt 4), 479-485 (2010).
- Skubak, P., Pannu, N. S. Automatic protein structure solution from weak X-ray data. Nat Commun. 4, 2777 (2013).
- Terwilliger, T. C., et al. Decision-making in structure solution using Bayesian estimates of map quality: the PHENIX AutoSol wizard. Acta Crystallogr D Biol Crystallogr. 65 (Pt 6), 582-601 (2009).
- Vonrhein, C., Blanc, E., Roversi, P., Bricogne, G. Automated structure solution with autoSHARP. Methods Mol Biol. 364, 215-230 (2007).
- Culurgioni, S., Harris, G., Singh, A. K., King, S. J., Walsh, M. A. Structural Basis for Regulation and Specificity of Fructooligosaccharide Import in Streptococcus pneumoniae. Structure. 25 (1), 79-93 (2017).
- Boivin, S., Kozak, S., Meijers, R. Optimization of protein purification and characterization using Thermofluor screens. Protein Expression and Purification. 91 (2), 192-206 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon