È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Applicazione di microscopia di velocità ad alta velocità super-risoluzione in Live cilio primario
In questo articolo
Riepilogo
Recentemente abbiamo mappato le posizioni spaziali (3D) tridimensionale delle vie di trasporto per varie proteine dispostamento all'interno ciliari in cellule vive. Qui questo dettagli di carta la messa a punto sperimentale, il processo di campioni biologici e l'analisi dei dati per la fluorescenza 3D Super-resolution imaging approccio appena applicati in live ciliari.
Abstract
Il cilio primario è una protrusione microtubule-basato sulla superficie di molte cellule eucariotiche e contiene un unico complemento delle proteine tale funzione criticamente nella motilità cellulare e di segnalazione. Poiché le ciglia sono incapaci di sintetizzare le proprie proteine, quasi 200 unici ciliare proteine devono essere oggetto di traffico tra il citosol e ciglia primarie. Tuttavia, è ancora una sfida tecnica per mappare le tridimensionale (3D) posizioni delle vie di trasporto per queste proteine in vivo ciliari a causa delle limitazioni attualmente esistenti tecniche. Per vincere la sfida, recentemente abbiamo sviluppato e impiegato una microscopia ad alta super-risoluzione 3D virtuale ad alta velocità, definita microscopia Sub-diffrazione (velocità) singolo punto bordo-eccitazione, per determinare la posizione spaziale 3D delle vie di trasporto per entrambi citosolici e proteine di membrana in ciglia primarie di cellule vive. In questo articolo, ci dimostrerà la configurazione dettagliata di microscopia di velocità, la preparazione delle cellule che esprimono proteine ciliari fluorescenza-proteina-identificate, l'inseguimento in tempo reale di singola molecola di singole proteine in vivo Cilio e il raggiungimento di densità di probabilità spaziale 3D mappe delle vie di trasporto per le proteine ciliari.
Introduzione
Quanto affermato da Ernst Abbe nel 1873, la risoluzione di microscopia chiara convenzionale è stata creduta di essere limitato a circa 200 nm a causa della diffrazione della luce dall' obiettivo1,2. Attualmente, le tecniche di microscopia di Super-risoluzione rompono questa limitazione e permettono di catturare immagini dinamiche con risoluzione sub-diffrazione (< 200 nm). Le tecniche rientrano generalmente in due grandi categorie: stimolato gli approcci di microscopia basata di svuotamento (STED) emissione, che generano volume di illuminazione di Sub-diffrazione dovuto la risposta ottica non lineare di fluorofori in campioni3; e fotoattivazione microscopia (PALM) e microscopia di ricostruzione ottica stocastica (STORM)-basato su tecniche di Super-risoluzione, che utilizzano funzioni matematiche per localizzare i centroidi di fluorofori e quindi ricostituire questi centroidi per formare super-risoluzione immagini4,5. Attualmente, a causa del relativamente semplice setup ottico, PALM e tempesta sono ampiamente impiegati attivando solo un piccolo sottoinsieme di fluorofori in ogni fotogramma di un video lungo di una preparazione biologica. Questo consente per la localizzazione più precisa di raccordo 2D gaussiana dello spot fluorescente, definito la funzione di diffusione del punto (PSF), di proteine fluorescente etichettati in ogni fotogramma del video. La posizione 2D di ogni molecola fluorescente etichettati quindi possa essere sovrapposti su un unico piano di formazione immagine per produrre un'immagine di Super-risoluzione del biologico preparazione1,2. Mentre questi localizzazione singola molecola, approcci di Super-risoluzione per la microscopia certamente rivoluzionato come imaging di campioni biologici è stata eseguita, ci sono ancora sfide da superare. Ad esempio, tempesta e PALM può raggiungere i loro migliori risoluzioni spaziali dopo la fissazione dei campioni biologici e presentano quindi una rappresentazione statica delle proteine con etichetta fluorescente, che è una limitazione simile di microscopia elettronica. Inoltre, per ottenere un'alta risoluzione spaziale per ogni proteina fluorescente-labeled in cellule vive, i campioni devono essere imaged al framerate molto lungo che non sono in grado di catturare proteina dinamiche. Pertanto, è necessario per superare questi ostacoli tecnici principali.
Per ottenere un'alta risoluzione spazio-temporale che è particolarmente adatta per la rilevazione di proteine veloci o RNA in cellule vive, abbiamo sviluppato la microscopia di Super-risoluzione velocità nel nostro laboratorio (Figura 1)6,7, 8. diverse importanti progressi tecnici nella microscopia di velocità precedentemente ci hanno permesso di monitorare con successo trasporto nucleocytoplasmic di piccole molecole, proteine, mRNA e virus tramite nativo nucleare poro complessi (NPC)6, 7 , 8. brevemente, le seguenti funzionalità di microscopia di velocità verrà utilizzata per tenere traccia di macromolecole veloci attraverso le strutture simmetria rotazionale sub-micrometrica in cellule vive, come NPC e ciglia primarie: (1) un inclinato o un illuminazione verticale PSF consente l'eccitazione di singole molecole all'interno di un volume piccolo limite di diffrazione nel piano focale (Figura 1); (2), il PSF inclinato può evitare notevolmente out-of-focus fluorescenza e quindi migliorare il rapporto segnale-rumore. (3), la densità ottica di 100-500 kW / cm2 nell'illuminazione PSF permette a migliaia di fotoni per essere raccolti dal singolo fluorofori con velocità veloce rilevamento (> 500 Hz). Velocità di rilevazione (4), il digiuno inoltre notevolmente riduce l'errore di localizzazione spaziale di singola molecola (< 10 nm) nel determinare le traiettorie spaziali di molecole fluorescenti in movimento in cellule vive, perché la diffusione molecolare è uno dei principali fattori causando le imperfezioni della singola molecola localizzazione per lo spostamento di molecole. Algoritmi di trasformazione (5) ben consolidate 2D a 3D ci permettono di fornire mappe di densità di probabilità spaziale 3D delle vie di trasporto per le molecole in NPC o il cilio primario. È interessante nota che il nostro processo di conversione tra il cartesiano e il sistema di coordinamento cilindrico viene utilizzato per generare una densità di probabilità spaziale 3D mappa anziché 3D tracking della singola molecola (Figura 2). In precedenza, dati di microscopia elettronica hanno rivelato che la NPC9,10 e il cilio primario11 entrambi hanno una struttura simmetria rotazionale. In linea di principio, in modo casuale diffondere molecole si muove attraverso il NPC o il cilio primario dovrebbe anche avere distribuzioni simmetriche rotazionale. Come illustrato nella Figura 2, un elevato numero di molecole all'interno del cilindro di diffusione in modo casuale genererebbe simmetriche rotazionale distribuzioni la vista di sezione trasversale come quello nel NPC, ulteriore conseguente un circa uniforme spaziale distribuzione all'interno di ogni sub-regione molto piccola tra due anelli adiacenti (Figura 2E). Questa distribuzione uniforme porta che la distribuzione spaziale lungo dimensione θ nel sistema cilindrico è costante. Quindi le coordinate 3D (X, R, θ) possono essere semplificate per essere le coordinate 2D (R, X, costante). In realtà, il nostro processo di conversione tra il cartesiano e i sistemi cilindrici è da 2D (X, Y) a 2D (R, X, costante). Il θ costante, la densità spaziale p in Figura 2,Esi riferisce, viene calcolata utilizzando l'equazione A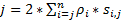 .
.
In ultima analisi, rilevamento di singola molecola ha un'ampia applicazione nella ricerca biologica, così, è naturale che una pletora di tecniche sarà sviluppata per riempire nicchie biologiche specifiche12,13,14. Tale è il caso con microscopia di velocità. In precedenza, quando accoppiato con un algoritmo di trasformazione 3D, questa tecnica è stata sviluppata per risolvere i percorsi di trasporto 3D di transito attraverso gli NPC, una struttura biologica rotationally simmetrici e sub diffraction-dimensioni6molecole. In questa carta, ciglia primarie è dimostrato di essere organelli eccellente modello pure. Ciliari sono cilindrici, antenna-come organelli (raggio di ~ 125 nm) che proiettano dalla superficie delle cellule più mammiferi15,16,17. Sono responsabili per la ricezione di segnali esterni e trasmettere una risposta intracellulare in genere associata a crescita e metabolismo15,16. Di conseguenza, flusso di proteine strutturali, riciclo di recettori transmembrana e trasmissione di messaggeri intracellulari è responsabilità vitale delle ciglia primarie. Nel punto di giunzione tra le ciglia primarie e il corpo cellulare è una barriera di selettività critico, chiamata la zona di transizione o TZ, attraverso il quale tutto questo trasporto di proteine deve verificarsi11,18,19, 20. Oltre alla funzione di gating del TZ, almeno due processi di trasporto, trasporto intraflagellare e diffusione passiva, si pensiero siano responsabili del movimento della proteina attraverso questa regione16,21, 22. dal punto di vista della salute umana, la perdita delle ciglia primarie e conseguente deregolamentazione di segnalazione a valle è caratteristica di molti cancri. Inoltre, molte malattie genetiche, come la sindrome di Bardet-Biedl e malattia del rene policistico, sono associati con proteina difettosa trasporto23. Sia la dimensione del limite di Sub- diffrazione e il complesso processo di trasporto selettivo di proteine attraverso la TZ rendono le ciglia primarie un obiettivo primario per questa tecnica. In questa carta di metodi, dimostreremo il tracciamento di una proteina transmembrana ciliare, recettore della somatostatina 3 (SSTR3)24, etichettati esternamente con Alexa Fluor 647 e un componente dell'IFT, IFT2025, etichettati con una molecola GFP fusa.
Protocollo
1. NIH 3T3-preparazione delle cellule per microscopia di velocità da stock
- 1,5 settimane in anticipo l'esperimento, recuperare una coltura fresca delle cellule NIH 3T3 da uno stock congelato scongelamento a 37 ° C e trasferendo le cellule di un matraccio di cultura cellulare2 25 cm con 3 mL dell'Aquila per volta di Dulbecco Medium (DMEM) addizionata di 110 mg/mL piruvato di sodio, la glutamina 2mm, 10% siero bovino fetale e 1% di penicillina/streptomicina.
- Incubare le cellule a 37 ° C in un incubatore di 5% CO2 .
- Dividere le celle al confluency di 80%, circa ogni due giorni, almeno tre volte prima di giorno sperimentale per garantire omogeneità del ciclo cellulare. Tripsinizzano cellule con tripsina 0,25% per 2 min a 37 ° C, aspirare tripsina e sostituirlo con 2 mL di terreno. Pipetta il medium più volte per spezzare mazzi delle cellule, rimuovere il numero di celle desiderato e portare il volume totale dei media indietro fino a 3 mL.
Nota: NIH-3T3 precedentemente erano geneticamente per esprimere NPHP-4, una proteina che si localizza alla TZ26, fusa al capolinea C a mCherry. mCherry è un fluoroforo che possa essere eccitato con illuminazione a 561 nm per localizzare quantitativamente la barriera di selettività TZ e orientare le ciglia primarie. - Due giorni prima dell'esperimento, piastra le cellule in un fondo di vetro 35mm piatto al confluency di 60-70% con 1,5 mL di terreno stesso come passo 1.1 e restituiscono le cellule nell'incubatore.
- Un giorno prima dell'esperimento, transfect chimicamente le cellule con il plasmide desiderata. Mescolare 500-1000 ng del plasmide desiderato (vedere la Nota qui sotto) in un rapporto di 1: 2,5 con reagente di transfezione in 0,25 mL di media riduttori del siero senza antibiotici per 30 min media aspirato da 35 mm vetro piatto fondo e sostituirlo con il plasmide di 0,25 mL / mix di reagente di transfezione più un extra 1,25 mL di media riduttori del siero senza antibiotici. Riduttore del siero media serve allo scopo facilitare una successo transfezione, che inducono la crescita di ciglia primarie, come pure mantenere le cellule viva abbastanza a lungo per eseguire l'esperimento. Riporre le cellule nell'incubatore per l'esperimento il giorno seguente.
Nota: Quando si esegue il rilevamento di singola molecola di IFT20, un plasmide contenente che un IFT20 geneticamente fuso al suo capolinea C GFP è usato25. Durante l'esecuzione di singola molecola rilevamento di SSTR3, un plasmide contenente un SSTR3 geneticamente fuso al suo capolinea N a un dominio di peptide (AP) acceptor e C terminale GFP è usato22. Oltre il costrutto SSTR3, un plasmide contenente la ligasi di biotina BirA deve essere co-espressi e i media di transfezione deve essere integrati con la biotina 10 µM. BirA attribuisce quindi biotina al dominio AP di recentemente sintetizzati molecole AP-SSTR3-GFP a livello dell'ER. Alexa647 coniugato a tre dei quattro siti di biotina-legante su streptavidina, in media, quindi può essere completata ai media prima di imaging a fluorescente etichetta le molecole di AP-SSTR3-GFP sulla superficie esterna della cella22,27 . GFP e AlexaFluor647 vengono utilizzati in questo metodo; Tuttavia, altre sonde fluorescenti possono essere utilizzate se hanno similmente alta fotostabilità e resa quantistica. - Se utilizzando il costrutto SSTR3 etichettati esternamente, rimuovere il supporto dal fondo di vetro piatto 1 h prima di esperimento, lavare la cella 5 volte con 1 mL di tampone fosfato salino (PBS) e aggiunta 1 mL di siero riduttore terreno completati con 1 µM Alexa647 coniugato streptavidina.
- Non più di 15 min prima dell'esperimento, rimuovere il supporto dal vetro piatto fondo e lavare le cellule trasfettate ed etichettate 5 volte con 1 mL di PBS.
- Posizionare 1 mL di tampone di imaging (20 mM HEPES, 110 mM KOAc, 5 mM NaOAc, 2 mM MgOAc, 1 mM EGTA, pH 7,3) nel piatto fondo di vetro.
Nota: Nel buffer di imaging, le cellule sono vitali per non più di 3 h. Di conseguenza, solo 2 h di esperimenti vengono eseguite su ogni piatto.
2. microscopia di velocità
Nota: L'impostazione della velocità della microscopia include un microscopio di fluorescenza invertito dotato di un obiettivo apocromatico di 1,4-NA 100 × immersione in olio, un 35 mW 633 nm laser a He-Ne, 50 mW 488 nm e 561 nm laser a stato solido, un guadagno di moltiplicazione su chip charge-coupled device fotocamera e un pacchetto di software di microscopio per acquisizione dati ed elaborazione (Figura 1). Per l'imaging di singolo canale, GFP, mCherry e Alexa647 sono eccitati da 488 nm, 561 nm, o 633 nm laser, rispettivamente. Per il rilevamento di singola molecola, unico punto illuminazione viene utilizzato per tenere traccia di singole molecole fluorescente etichettati. Per l'imaging di epifluorescenza, una lente concava viene inserita nel percorso di illuminazione laser per espandere il raggio in un campo uniforme di illuminazione. Le emissioni di fluorescenza sono raccolti dall'obiettivo stesso, filtrate da un filtro dicroico (405/488/561/635) e un filtro di emissione (405/488/561/635) e ripreso con la telecamera sopra a 500 Hz per l'inseguimento di singola molecola o 2 Hz per formazione immagine di epifluorescenza.
- Appone la piastra inferiore di vetro sul palco del microscopio e individuare una cellula che esprime correttamente i costrutti desiderati. Una volta che è stato trovato un cellulare adeguata, allineare la macchia NPHP4-mCherry alla base delle ciglia primarie con la posizione sul piano formazione immagine che corrisponde all'illuminazione di singolo punto del laser.
- Acquisire un'immagine di epifluorescenza del NPHP4-mCherry e IFT20-GFP o AP-SSTR3-GFP utilizzando la funzione "Snap" nella scheda "Fotocamera" della finestra "Focus controlli" se utilizza il pacchetto software di microscopia digitale (Vedi Tabella materiali).
Nota: Queste immagini agirà come un riferimento per le posizioni successive singola molecola. - Una volta ottenute le immagini di riferimento, localmente di ridurre la concentrazione di molecole singole con etichettate. Foto-candeggina la TZ con illuminazione di laser 1 mW per 20 s o fino a quando l'intensità di fluorescenza è vicina a quella di fluorescenza di fondo.
Nota: Quando la concentrazione precisa può essere controllata, 0.1-1 nM etichettato singole molecole sono utilizzati. - Per preparare per il rilevamento di singola molecola, ridurre la potenza di illuminazione del laser a ~0.15 mW per singole molecole etichettati con GFP o ~0.5 mW per molecole etichettata con Alexa647.
- Non appena la potenza del laser e parametri di imaging sono insieme, guadagno massimo e intensificazione e 2 ms frame rate, per l'imaging di singola molecola, coinvolgere il laser illuminazione appropriata e registrare non-photobleached, etichettato singole molecole che vengono trasportati attraverso la regione di photobleached del TZ facendo clic sul pulsante "Flusso" nella scheda "Fotocamera" della finestra "Controlli di messa a fuoco".
Nota: Non più di 2 min di video devono essere acquisite per ridurre al minimo gli effetti di deriva ciliare ad un livello trascurabile. - Dopo la cattura del video di singola molecola, elaborare i video utilizzando un algoritmo di raccordo gaussiana 2D, come scorcio dal laboratorio Gelles, che localizza con precisione il centroide di eccitazione di ogni singola molecola PSF in un'area che comprenda di interesse (AOI).
- Selezionare tutte le posizioni di singola molecola con precisione < 10 nm e correggere il centro delle ciglia basato sulla distribuzione delle posizioni di singola molecola dotata di una funzione gaussiana 2D.
Nota: Utilizzando 2D all'algoritmo di trasformazione 3D, 3D trasporto visualizzati i percorsi delle vie IFT20-GFP e AP-SSTR3 sono chiaramente sulla membrana ciliare axonemal o ciliare, rispettivamente.
3. 2D a 3D trasformazione
- Una volta mille diverse localizzazioni per molecole (segnale-rumore rapporto > 11) in transito nel Cilio sono raccolti, è possibile selezionare l'asse lungo del cilio come la dimensione X. Fare un istogramma di dimensione Y delle posizioni e procurarsi le somme bin in incrementi di 10 nm.
Nota: Da 2D a 3D trasformazione possono essere valutati da mano o qualsiasi software o linguaggio di programmazione. Gli autori hanno implementato con successo la trasformazione in Matlab e Python 2.7.
Risultati
Questa sezione illustra i dati ottenuti dall'esecuzione di microscopia di velocità presso la TZ delle ciglia primarie per studiare il percorso di trasporto di SSTR3 collegato da un linker esterno di nm ~ 15 a Alexa647 (Figura 3A). Esso ha il duplice scopo di verificare l'algoritmo di trasformazione 3D. Alexa647 dovrebbe solo etichetta la superficie esterna del cilio primario e di conseguenza, la via di trasporto 3D dovrebbe rivelare una via ...
Discussione
Questo protocollo descrive l'applicazione di microscopia di velocità per il cilio primario, un organello di segnalazione cellulare che è altamente affidamento sul trasporto efficiente della proteina. Microscopia di velocità in grado di fornire alta risoluzione posizioni (< 10 nm) per molecole fluorescente etichettati come passano attraverso l'illuminazione di singolo punto centrato su TZ. In precedenza è stato applicato per studiare la proteina traffico attraverso il NPC6,
Divulgazioni
Gli autori non dichiarano conflitti di interesse.
Riconoscimenti
Ringraziamo il dottor Kristen Verhey (University of Michigan, Ann Arbor) e Dr. Gregory Pazour (Università del Massachusetts Medical School) per la fornitura di alcuni plasmidi. Il progetto è stato sostenuto da sovvenzioni dal National Institutes of Health (NIH GM097037, GM116204 e GM122552 a WY).
Materiali
| Name | Company | Catalog Number | Comments |
| 25 cm2 tissue culture dish | Corning | VV-01936-00 | |
| Penicillin/streptomycin | ThermoFisher | 15140122 | |
| Fetal bovine serum | ThermoFisher | 10438018 | |
| DMEM | ThermoFisher | 10566-016 | |
| OPTIMEM | ThermoFisher | 31985062 | |
| Trypsin | ThermoFisher | 25300054 | |
| Phosphate buffered saline | Sigma-Aldrich | P3813-1PAK | |
| Transit LT1 | Mirus | MIR 2300 | |
| 35 mm glass bottom dish | MatTek | P35GCOL-0-14-C | |
| AlexaFluor 647-conjugated streptavidin | ThermoFisher | S21374 | |
| Biotin | Sigma-Aldrich | B4501-100MG | |
| 633 nm He-Ne laser | Melles Griot | 25-LHP-928-249 | |
| 561 nm solid state laser | Coherent | OBIS 561-50 LS | |
| 488 nm solid state laser | Coherent | 1185053 | |
| Inverted fluorescence microscope | Olympus | IX81 | |
| 1.4-NA 100× oil-immersion apochromatic objective | Olympus | UPLSAPO 100× | |
| On-chip multiplication gain charge-coupled-device camera | Roper Scientific | Cascade 128+ | |
| Dichroic filter | Semrock | Di01- R405/488/561/635-25x36 | |
| Emission filter | Semrock | NF01-405/488/561/635-25X5.0 | |
| Slidebook 6.0 | Intelligent Imaging Innovations | digital microscopy software |
Riferimenti
- Huang, B., Bates, M., Zhuang, X. Super-resolution fluorescence microscopy. Annu Rev Biochem. 78, 993-1016 (2009).
- Leung, B. O., Chou, K. C. Review of super-resolution fluorescence microscopy for biology. Appl Spectrosc. 65, 967-980 (2011).
- Willig, K. I., Rizzoli, S. O., Westphal, V., Jahn, R., Hell, S. W. STED microscopy reveals that synaptotagmin remains clustered after synaptic vesicle exocytosis. Nature. 440, 935-939 (2006).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313, 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nat Meth. 3, 793-796 (2006).
- Ma, J., Yang, W. Three-dimensional distribution of transient interactions in the nuclear pore complex obtained from single-molecule snapshots. Proc Natl Acad Sci USA. 107, 7305-7310 (2010).
- Ma, J., Goryaynov, A., Sarma, A., Yang, W. Self-regulated viscous channel in the nuclear pore complex. Proc Natl Acad Sci USA. 109, 7326-7331 (2012).
- Ma, J., et al. High-resolution three-dimensional mapping of mRNA export through the nuclear pore. Nat Comm. 4, (2013).
- Akey, C. W., Radermacher, M. Architecture of the Xenopus nuclear pore complex revealed by three-dimensional cryo-electron microscopy. J Cell Biol. 122, 1-19 (1993).
- Akey, C. W. Interactions and structure of the nuclear pore complex revealed by cryo-electron microscopy. J Cell Biol. 109, 955-970 (1989).
- Czarnecki, P. G., Shah, J. V. The ciliary transition zone: from morphology and molecules to medicine. Trends Cell Biol. 22, 201-210 (2012).
- Elf, J., Li, G. -. W., Xie, X. S. Probing transcription factor dynamics at the single-molecule level in a living cell. Science. 316, 1191-1194 (2007).
- Anzalone, A., Annibale, P., Gratton, E. 3D orbital tracking in a modified two-photon microscope: an application to the tracking of intracellular vesicles. J Vis Exp. , (2014).
- Ritter, J. G., Veith, R., Veenendaal, A., Siebrasse, J. P., Kubitscheck, U. Light sheet microscopy for single molecule tracking in living tissue. PloS one. 5, 11639 (2010).
- Marshall, W. F., Nonaka, S. Cilia: tuning in to the cell's antenna. Curr Biol. 16, 604-614 (2006).
- Scholey, J. M., Anderson, K. V. Intraflagellar transport and cilium-based signaling. Cell. 125, 439-442 (2006).
- Yang, T. T., et al. Superresolution pattern recognition reveals the architectural map of the ciliary transition zone. Sci Rep. 5, 14096 (2015).
- Craige, B., et al. CEP290 tethers flagellar transition zone microtubules to the membrane and regulates flagellar protein content. J Cell Biol. 190, 927-940 (2010).
- Kee, H. L., et al. A size-exclusion permeability barrier and nucleoporins characterize a ciliary pore complex that regulates transport into cilia. Nat Cell Biol. 14, 431-437 (2012).
- Najafi, M., Maza, N. A., Calvert, P. D. Steric volume exclusion sets soluble protein concentrations in photoreceptor sensory cilia. Proc Natl Acad Sci USA. 109, 203-208 (2012).
- Nachury, M. V., Seeley, E. S., Jin, H. Trafficking to the ciliary membrane: how to get across the periciliary diffusion barrier. Annu Rev Cell Dev Biol. 26, 59-87 (2010).
- Ye, F., et al. Single molecule imaging reveals a major role for diffusion in the exploration of ciliary space by signaling receptors. Elife. 2, 00654 (2013).
- Ross, A. J., et al. Disruption of Bardet-Biedl syndrome ciliary proteins perturbs planar cell polarity in vertebrates. Nat Genetics. 37, 1135-1140 (2005).
- Handel, M., et al. Selective targeting of somatostatin receptor 3 to neuronal cilia. Neuroscience. 89, 909-926 (1999).
- Follit, J. A., Tuft, R. A., Fogarty, K. E., Pazour, G. J. The intraflagellar transport protein IFT20 is associated with the Golgi complex and is required for cilia assembly. Mol Biol Cell. 17, 3781-3792 (2006).
- Awata, J., et al. NPHP4 controls ciliary trafficking of membrane proteins and large soluble proteins at the transition zone. J Cell Sci. 127, 4714-4727 (2014).
- Howarth, M., Ting, A. Y. Imaging proteins in live mammalian cells with biotin ligase and monovalent streptavidin. Nat Protoc. 3, 534-545 (2008).
- Huang, B., Wang, W., Bates, M., Zhuang, X. Three-dimensional super-resolution imaging by stochastic optical reconstruction microscopy. Science. 319, 810-813 (2008).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon