Method Article
Aderenza dei batteri per piantare superfici misurate in laboratorio
In questo articolo
Riepilogo
Un metodo semplice per la misurazione e la caratterizzazione di adesione batterica alle piante, in particolare radici e germogli, è descritto in questo articolo.
Abstract
Questo manoscritto descrive un metodo per misurare l'associazione batterica alle superfici axeniche pianta al microscopio ottico e attraverso l'utilizzo di conteggi delle cellule vitali. Materiali vegetali utilizzati includono radici, germogli, foglie e tagliare la frutta. I metodi descritti sono poco costoso, facile e adatto per campioni di piccole dimensioni. Il legame è misurato in laboratorio e una varietà di media di incubazione e condizioni può essere utilizzata. L'effetto degli inibitori può essere determinato. Possono essere valutati anche situazioni che promuovono e inibiscono il grippaggio. In alcuni casi è possibile distinguere se varie condizioni alterano associazione principalmente dovuto i loro effetti sulla pianta o sui batteri.
Introduzione
La misura di associazione batterica per piantare le superfici è diventato importante in tre situazioni disparate. La prima situazione è l'esame della trasmissione di agenti patogeni umani su pianta superfici1,2,3. L'obiettivo è di impedire l'associazione batterica o di rimuovere o uccidere i batteri associati e quindi a ridurre la trasmissione della malattia da materiale vegetale. La seconda situazione è l'esame dell'associazione degli agenti patogeni della pianta per pianta superfici4. Ancora una volta l'obiettivo è di impedire l'associazione o di rimuovere o uccidere i batteri associati e quindi per ridurre la malattia. La terza situazione è l'esame dell'associazione di batteri simbiotici o pianta-growth-promoting5,6. L'obiettivo è quello di promuovere batterica vincolante e quindi di aumento di salute delle piante e delle colture produce.
Le tecniche per la misurazione associazione batterica per piantare le superfici descritte in questo articolo sono poco costoso e relativamente facile da realizzare. Gli unici requisiti sono un microscopio e materiali trovati generalmente in un laboratorio di batteriologia. Per alcune tecniche un sonicatore vasca è utile. Le tecniche descritte sono progettate per l'associazione di esperimenti condotti utilizzando campioni di relativamente piccole dimensioni. Misure di associazione sono effettuate in laboratorio, anche se potrebbe essere possibile modificare alcune di queste tecniche per l'utilizzo in serra o in campo.
Queste tecniche sono state usate per misurare l'associazione batterica a radici, germogli, foglie di taglio, taglio frutta e pomodorini intatti nel laboratorio7,8,9,10,11, 12,13,14,15. Essi sono stati utilizzati anche per misurare la colonizzazione della radice delle piante che crescono nel terreno o sabbia nel laboratorio16. Le tecniche sono state utilizzate con molte specie batteriche come Agrobacterium tumefaciens, Sinorhizobium meliloti, Escherichia coli, Salmonella enterica, Pseudomonas fluorescens. Un'utile descrizione dei metodi per valutare l'interazione di a. tumefaciens con superfici può essere trovata in Morton e Fuqua (2012)17. In tutti i casi le dimensioni del campione coinvolte erano piccole, generalmente meno di 25-50 piante. Le tecniche descritte sono adatte per l'utilizzo con agenti patogeni umani che hanno bisogno di essere tenuto confinato durante gli esperimenti.
Protocollo
1. preparazione del materiale vegetale axenica
- Preparare piantine coltivate in acqua.
- Posizionare un piccolo numero di semi (meno di 30) in una 30, 50, 100 o Becher vetro da 150 mL. Abbiamo usato pomodoro, erba medica, Arabidopsis thaliana, pisello, fagiolo, tabacco, lattuga e semi di carota con questo protocollo.
Nota: Per determinare quanti semi possono essere sterilizzati insieme senza contaminazione sta diffondendo da un seme in un altro, vedere il punto 1.1.2. - Coprire i semi con 80% di etanolo e agitare brevemente. Lasciare i semi in ammollo per 1 min.
- Versare fuori l'etanolo e coprire i semi con una soluzione di 50% di candeggina commerciale volume (NaClO) e 0,1% Triton X-100 nell'acqua del rubinetto. Lasciare i semi in ammollo per 20 min.
Nota: Se i semi sono grandi, come i semi di fagiolo, può essere necessario allungare il tempo di macerazione completamente uccidere i funghi sulla superficie del seme. - Versare la miscela di candeggina e lavare i semi 3 volte con acqua sterile. Lasciare i semi in ammollo in acqua per 1 minuto per ogni lavaggio.
- Per ottenere piantine axeniche aggiungono una piccola quantità di acqua sterile tra 5 e 25 mL a seconda delle dimensioni dei semi. Versare i semi e l'acqua in una piastra Petri sterile per la germinazione dei semi.
Nota: L'acqua deve coprire il fondo del piatto ma non coprire i semi per favorire la formazione di peli radicali. Axeniche descrive una cultura in cui soltanto una singola specie di organismo è presente. - Incubare al buio fino a quando le piantine raggiungono la taglia desiderata tra 1 e 10 cm (circa 5 giorni per pomodoro e a. thaliana e 1 a 3 giorni per erba medica). Per i semi più grandi è necessario utilizzare un contenitore sterile coperto con una capacità di più di 100 mL ad esempio di un piatto di vetro coperto con un foglio.
- Posizionare un piccolo numero di semi (meno di 30) in una 30, 50, 100 o Becher vetro da 150 mL. Abbiamo usato pomodoro, erba medica, Arabidopsis thaliana, pisello, fagiolo, tabacco, lattuga e semi di carota con questo protocollo.
- Determinare la frequenza di contaminazione di un particolare lotto con microrganismi che rimangono vitali su o all'interno i semi dopo la superficie-sterilizzazione.
Nota: Ciò è necessario perché occasionale semi veicolare microrganismi sotto il cappotto di seme che non può essere ucciso da superficie-sterilizzazione. Utilizzare la frequenza di semi contaminati in un sacco di seme per determinare quanti semi per sterilizzare in una sola volta senza un elevato rischio del gruppo di semi diventando contaminato a causa di un seme con batteri o funghi che sopravvivono al trattamento.- Eseguire i punti da 1.1.1 attraverso 1.1.3.
- Mettere i semi su un agar nutriente di Petri. Mettere circa 10-30 semi in ogni piatto distanziando così che essi possono essere segnati singolarmente. Sigillare i piatti con nastro o sigillatura film.
- Incubare per 3-5 giorni a 25 ° C, segnando ogni giorno per la conseguenza visibile di microrganismi dai semi.
- Preparare piantine coltivate in sabbia. I semenzali possono essere coltivati in sabbia per uso negli esperimenti di adesione batterica.
- Eseguire i punti da 1.1.1 attraverso 1.1.3.
- Sterilizzare la sabbia di quarzo o mare in autoclave. Entrambi questi contengono alcune sostanze organiche. Se questo influenzerà l'esperimento, lavare la sabbia coprendolo con due volte il suo volume di 0.1 M HCl. Mix per 10 min. Consenti la sabbia per stabilirsi e versare il liquido. Sciacquare la sabbia 3 volte con acqua, e una volta con 80% etanolo seguita da due risciacqui di acqua supplementare utilizzando lo stesso protocollo per quanto riguarda la 0.1 M HCl. sterilizzare la sabbia lavata in autoclave.
- Sterilizzare i contenitori immergendo loro in candeggina al 50% per 5 min e risciacquarli 5 volte con acqua sterile da immersione. Lasciarli asciugare e la parte inferiore della guarnizione con pellicola sigillante. Il film come da parte del produttore è generalmente gratuito di microrganismi all'interno lato che deve essere posizionato rivolto verso l'interno del contenitore.
- Mescolare la sabbia sterile con acqua abbastanza sterile bagnarlo abbastanza che la sabbia bastoni insieme. La quantità dipenderà la sabbia è come asciutto. 10-35% del volume della sabbia è solitamente sufficiente. Disporre la sabbia bagnata nel contenitore permettendo profondità sufficiente per la lunghezza delle radici necessario e sufficiente spazio sopra la sabbia per la crescita delle riprese. Le distanze esatte dipendono dalla specie e della varietà della pianta viene utilizzata.
- Semi di piante.
- Fare un buco poco profondo nella sabbia con una bacchetta di vetro sterile solo abbastanza profonda per posizionare il seme sotto la superficie della sabbia (circa 1-5 mm). Posizionare il seme nel foro. Coprire con un sottile strato di sabbia. Sigillare la parte superiore del contenitore con la pellicola per evitare la perdita di acqua e ingresso di microbi ulteriori sigillante.
- Crescere le piante in laboratorio sotto una luce o in serra a una temperatura adeguata e la lunghezza del giorno per le specie e varietà di pianta. Per il pomodoro, erba medica, o Arabidopsis thaliana utilizzare temperatura e cicli di luce/buio di 12 h.
- Piantare piantine in un buco nella sabbia (1-2 mm di diametro e profondità sufficiente che la radice sarà coperto con la sabbia). Fare il buco con una bacchetta di vetro sterile. Guida con attenzione la radice nel foro utilizzando un sterili in acciaio inox uncinetto se necessario e riempire i lati del foro con sabbia.
- In alternativa, di coltivare piante axenically o in agar contenente una miscela di sali come sali di MS. Utilizzare i germogli di piante axeniche in agar direttamente. Evitare le radici coltivate con agar come agar si attacca alla superficie della pianta sia i batteri. Ciò potrebbe comportare una falsa impressione di adesione batterica alla superficie della pianta.
2. preparazione di altro materiale vegetale
- Lavare il materiale vegetale coltivato in serra con acqua prima dell'uso per ridurre il numero di microbi indigeni. Ci saranno i batteri e funghi sulle piante. Protozoi sarà presenti nel suolo, sulle radici ed eventualmente sulle foglie. Lavaggio con candeggina o etanolo altererà la superficie della pianta e non è raccomandato.
- Se dopo il lavaggio in acqua un numero significativo di microbi rimanga, trattare le piante con sapone liquido diluito o diluire perossido di idrogeno (0,01%) di rimuovere o uccidere i microbi. Questo è generalmente meno dannoso per la pianta di candeggina o etanolo.
- Come pianta materiale acquistato presso un mercato locale è difficile da decontaminare, scegliere materiale che sembra non sono state oggetto di immagazzinaggio di lunga durata. Evitare le parti del materiale che appaiono danneggiati o marrone.
- Lavarlo con acqua e fare tagli freschi sulle estremità del materiale che sono state tagliate in precedenza prima di utilizzarlo, a meno che le interazioni dei batteri prova con i batteri (ed eucarioti) che avrà accumulato presso siti di taglio sono di interesse. Non lavare con candeggina o etanolo come venga alterato la superficie della pianta.
- Trattare siti ferita. Spesso i siti ferita forniscono batteri accesso all'interno della pianta materiale18.
- Bloccare o ridurre batterica allegato a e movimento attraverso, ferita o tagliati siti creati nel preparare il materiale immergendo il bordo tagliato in paraffina fusa o dipingendo il sito con paraffina fusa, usando una piccola spazzola. Non utilizzare una spatola o un qualsiasi attrezzo affilato come questo può danneggiare il tessuto.
- Guarnizione stelo cicatrici sulla frutta come i pomodori con paraffina. Alcuni batteri nuoterà nella zona sotto la paraffina, ma i loro numeri sono generalmente di piccole dimensioni.
- Se batterica entrata in siti ferita è di interesse, stima la distanza che i batteri possono muoversi portata da corsi d'acqua o diffusione osservando il movimento di un colorante aggiunto alla soluzione. Contrassegnare i batteri mobili con l'introduzione di un gene che codifica per una proteina fluorescente come proteina fluorescente verde (GFP) in loro e li traccia utilizzando la microscopia a fluorescenza descritta in passaggio 6.1.219.
3. preparazione dei batteri
- I batteri di crescere. Uso di un mezzo che più si avvicina le condizioni i batteri sono suscettibili di essere stati esposti a immediatamente prima che si verifichi la pianta nel mondo reale di fuori del laboratorio. Fonti di carbonio e azoto così come la presenza di ioni, specialmente bivalenti cationi (Ca, Mg, Fe, Mn e Zn) e fosfato e il pH del terreno sono importanti.
- Utilizzare Media AB minimo o brodo di Luria per a. tumefaciens e altri di batteri del suolo20. Per e. coli, che possono avere origine nell'intestino, utilizzare brodo di Luria.
- Aggiungere induttori come essudati o estratti di piante commerciali come soytone o zuccheri come il saccarosio o xilosio al media21. Se queste sostanze sono usate come induttori, aggiungere una concentrazione bassa, ad esempio 0,01%. Se essi sono fonti di carbonio usato, aggiungere una maggiore concentrazione, ad esempio 0,1%.
- Preparare l'inoculo batterico. Diluire i batteri in acqua sterile o nel mezzo in cui l'incubazione avviene e aggiungerli al materiale vegetale. La diluizione appropriata è discusso ai punti 4.2 e 4.3.
- Per rimuovere il supporto di crescita prima di utilizzare i batteri, centrifugare la sospensione batterica a 10.000 x g per 2 minuti, versare il sovranatante e risospendere i batteri nel Vortex li nello stesso mezzo che verrà utilizzato per l'incubazione con il materiale vegetale. Questo metodo può rimuovere o diminuire il numero o la quantità di materiale extracellulare e appendici come esopolisaccaridi, capsule, flagellae e pili. Se è importante che queste strutture superficiali rimangono intatte, quindi semplicemente diluire i batteri prima di inoculare loro o utilizzare il metodo alternativo descritto al punto 3.2.2.
- Come metodo alternativo per rimuovere il mezzo di crescita, raccogliere i batteri su un filtro di nitrocellulosa o policarbonato con un diametro dei pori di 0,2 micron o meno. Lavare i batteri con il medium di incubazione e risospendere loro agitazione delicata o tramite vortex del filtro in un recipiente sterile del mezzo.
4. inoculazione dei batteri
- Determinare il numero di batteri per essere inoculati con riferimento la misura da utilizzare per determinare l'adesione batterica e la lunghezza del tempo di incubazione.
- Per gli studi microscopici che coinvolgono incubazione volte meno di 1 giorno inoculare un numero relativamente elevato di batteri. Aggiungere una quantità di cultura per raggiungere una concentrazione batterica finale di più di 106 batteri / mL. Tempi di incubazione più lunghi è necessario ridurre la dimensione di inoculo batterico.
- Per gli studi in cui adesione batterica sarà misurato mediante conta di cellule vitali, aggiungere una quantità di cultura per raggiungere una concentrazione batterica finale di 103 106 batteri / mL.
- Evitare di aggiungere così tanti batteri che loro metabolismo cambia la concentrazione di pH o ossigeno nel medium di incubazione. Misura del pH utilizzando carta pH o un elettrodo. Misurare la concentrazione di ossigeno utilizzando un elettrodo di ossigeno.
- Per piante coltivate in sabbia, inoculare in tre modi possibili.
- Inoculare il seme con i batteri prima di piantare il seme per 1 min con ammollo in una sospensione in acqua di circa 106 batteri / mL.
- Inoculare la radice di semenzali axeniche di germinazione, come descritto nella sezione 1 e quando la radice del semenzale è lunga circa 1 cm immergendolo o immissione il semenzale tutto in una sospensione di 106 batteri / mL in acqua per 1 min.
- Inoculare sabbia mescolando i batteri con la sabbia prima di piantare per ottenere una concentrazione finale di circa 103 batteri per mL o da annaffiare la piantina con una sospensione di 106 batteri / mL dopo la piantatura.
5. incubazione dei batteri con il materiale vegetale
- Per incubazione in mezzi liquidi, incubare i batteri con la materia vegetale in acqua sterile o saccarosio e sali minerali o vegetali terreno di coltura del tessuto (ad esempio un 01:10 diluizione dei sali MS)22,23.
- Utilizzare un contenitore a cui i batteri non aderiscono. Cercare di mantenere la superficie della pianta continuamente coperto da immersione o da agitazione delicata. Agitazione vigorosa può impedire l'adesione o anche rimuovere i batteri dalla superficie della pianta.
- Osservare il materiale vegetale al microscopio ottico dopo diversi intervalli di tempo per determinare quando interrompere l'incubazione e fare misurazioni. Un corso di tempo dell'adesione è spesso prezioso con campioni prelevati ogni 1 a 4 ore o ogni giorno a seconda della velocità dell'interazione.
- Per incubare in sabbia, si applicano le stesse considerazioni descritte per l'incubazione in medium liquido al punto 5.1.
6. misurazione dell'aderenza usando la microscopia
- Effettuare le misure al microscopio con ottica Nomarski o contrasto di fase per una facile visualizzazione dei batteri sulle superfici. Tuttavia, può essere utilizzato qualsiasi microscopio luminoso-campo con ingrandimento di 20 X o superiore.
- Utilizzare osservazione al microscopio per determinare se i batteri sono casualmente distribuiti o situati in siti specifici. Anche di verificare se essi sono vincolati singolarmente o in grappoli. Cercare la presenza di microcolonies suggerendo crescita batterica o allettamento dopo l'adesione. Verifica se i batteri sembrano formare un biofilm sulla superficie.
Nota: Un biofilm è un gran numero di batteri associati alla superficie e circondato da una matrice extracellulare23,24,25. La struttura può essere liscia e uniforme o hanno un'architettura più complessa. Metodi per lo studio di biofilm associati con superfici delle piante sono state descritte17. - Usare i batteri contrassegnati con un marcatore fluorescente. Se sono presenti altri batteri e i batteri di interesse sono identificati da un tag fluorescente come proteina fluorescente verde, è possibile utilizzare microscopia di fluorescenza per determinare la presenza dei batteri taggati in grappoli di altri batteri26. Per GFP, utilizzare un filtro con 490 nm eccitazione e 520 nm emissione.
- Controllare che l'etichetta fluorescente non incidono negativamente i batteri osservando l'aderenza al materiale axenica di una miscela uguale di batteri di tipo selvaggio e taggati batteri dello stesso ceppo utilizzando ottiche di fluorescenza e di Nomarski. Se i batteri fluorescenti e scuri sono casualmente mista e presenti nei numeri uguali il tag non ha interferito con il test.
- Utilizzare osservazione al microscopio per determinare se i batteri sono casualmente distribuiti o situati in siti specifici. Anche di verificare se essi sono vincolati singolarmente o in grappoli. Cercare la presenza di microcolonies suggerendo crescita batterica o allettamento dopo l'adesione. Verifica se i batteri sembrano formare un biofilm sulla superficie.
- Determinare il numero di batteri associati.
Nota: È molto difficile determinare il numero di batteri associati al microscopio. Quando l'associazione viene a superfici di pianta irregolare, generalmente non è possibile ottenere una misura quantitativa. La microscopia (non descritto in questo articolo) può essere utilizzata per effettuare tali misurazioni.- Quando i batteri sono associati a una superficie liscia come una radice dei capelli, contare il numero di batteri associati al bordo dei capelli radice per radice dei capelli di lunghezza di mm. Fate attenzione ad per usare peli radicali di all'incirca le stesse dimensioni e forma nel confronto tra misure.
- Per determinare le dimensioni degli oggetti nel microscopio, è possibile utilizzare una diapositiva commerciale con misurate marcature su di esso. Osservare e fotografare questa diapositiva con le stesse impostazioni come quello usato per il materiale sperimentale e utilizzare le immagini risultanti per determinare la dimensione degli oggetti nelle microfotografie.
- Preparare il campione per microscopia.
- Lavare il campione. Spostare il campione in una goccia d'acqua o incubazione medio su un vetrino da microscopio e osservarlo direttamente.
Nota: Questo ha il vantaggio che se c'era alcuna crescita batterica o batteriche morte reale c' non è improbabile che siano molti batteri liberi. Prendere l'assenza di batteri liberi come un segnale di avvertimento che ci può essere stato morte batterica o associazione batterica al contenitore in cui è stata effettuata l'incubazione. L'effetto di lavaggio nell'esempio è illustrato nella Figura 1. - Lavare delicatamente il campione in mezzo acqua o incubazione inserendola in un flacone di liquido e capovolgendo delicatamente il flaconcino. Quindi mettere il campione sul vetrino da microscopio nel liquido fresco per l'osservazione.
- Inserire il campione nel liquido utilizzando un normale vetrino coprioggetto e un vetrino da microscopio.
- Se il campione è spesso e quindi avrebbe un rigonfiamento sotto il vetrino coprioggetto, utilizzare un vetrino coprioggetto press-apply. Questi vetrini coprioggetto hanno un anello di gomma o di plastica intorno al bordo del vetrino coprioggetti. Inserire il liquido e il campione in bene dentro il vetrino coprioggetto e quindi porre il vetrino in cima e premete dolcemente per sigillare il vetrino coprioggetto alla diapositiva. Invertire ed esaminare.
- In alternativa utilizzare un alghe conteggio diapositiva e coprioggetto in modo simile. Si noti che le diapositive con questa profondità generalmente non possono essere esaminato con un obiettivo di maggiore di 20 ingrandimenti.
- Lavare il campione. Spostare il campione in una goccia d'acqua o incubazione medio su un vetrino da microscopio e osservarlo direttamente.

Figura 1 : Fa un passo nella preparazione di un campione per la determinazione del numero di batteri associati. A. tumefaciens associazione di peli radicali di pomodoro (A, B e C) e di fili di nylon (D, E e F). Nei campioni montati in acqua senza lavare entrambi associati batteri (frecce nere) e batteri liberi (frecce bianche) possono essere visto (A e D). Dopo lavaggio che i batteri associati rimangono, ma i batteri liberi non sono più presenti (B ed E). Dopo sonicazione i batteri associati sono stati rimossi dalla superficie del campione (C e F). Clicca qui per visualizzare una versione più grande di questa figura.
- Se sono stati utilizzati batteri fluorescenti, esaminare il campione al microscopio dopo la sabbia è stata rimossa. Le radici delle piante coltivate in sabbia, generalmente, non sono adatte per microscopia come le particelle di sabbia interferiscano con l'osservazione dei batteri.
- Rimuovere l'impianto da un contenitore di crescita secondo il passaggio 7. Mettere le radici in un contenitore di acqua e mescolare delicatamente per rimuovere la sabbia che si depositerà sul fondo del contenitore.
- Togliere la pianta dall'acqua di lavaggio e montare il campione come descritto al punto 6.3.3.
7. misurazione dell'aderenza utilizzando cellule vitali conta
- Determinare il numero di batteri vitali di allegati utilizzando un sonicatore.
- Rimuovere i batteri non associati. Il campione viene posto in un flaconcino con acqua sufficiente, il buffer o incubazione media per coprire il materiale vegetale e capovolgendo il flacone di lavaggio.
- Se c'erano più di 103 gratis batteri / mL presenti in incubazione iniziale, effettuare lavaggi sequenziali per rimuovere tutti loro. Check-in un microscopio ottico (Vedi punto 6.3) per determinare la presenza di batteri liberi.
- Lavare fino a quando c'è una sostanziale riduzione del numero di batteri liberi, ma ricordate che c'è un equilibrio tra batteri associati e quindi il numero di batteri liberi non può mai ridursi a zero.
- Rimuovere l'esempio dal flacone di detersivo con un forcipe o una spatola.
- Determinare il numero di batteri associati nelle incubazioni di piante in un liquido.
- Sospendere il campione lavato in un flaconcino e coprirlo con una determinata quantità di acqua, medium di incubazione o tampone di lavaggio. Usare liquido a sufficienza per coprire il campione. Collocare il flaconcino in un sonicatore vasca e Sonicare esso per un min.
- Rimuovere l'esempio ed esaminarlo al microscopio luce per determinare se i batteri associati sono stati rimossi dalla pianta. La rimozione dei batteri da un campione di sonicazione è illustrata nella Figura 1. Se ci sono ancora vincolati batteri presenti continuare sonicating il campione fino ad un momento che nessun batterio associato rimanga sulla superficie del campione. Se i batteri associati non possono essere rimosso tramite sonicazione, aggiungere sabbia di quarzo sterile 1-10 mg/mL e ripetere l'esame al microscopio e sonicazione.
- Determinare che la procedura di sonicazione che appare più efficace nel passaggio 7.1.3.2 non riduce la vitalità dei batteri sospendendo i batteri da una cultura liquida nella soluzione da utilizzare per sonicazione (aggiungere sabbia di quarzo, se fosse necessario). Determinare il numero di cellule vitali. Sottoporre ad ultrasuoni i batteri e determinare il numero di cellule vitali nuovamente.
Nota: Se c'è una riduzione del conteggio di cellule vitali, modificare la procedura modificando la composizione del liquido e/o sonicazione tempo fino a quando il trattamento non ha alcun effetto sul conteggio di cellule vitali. - Determinare che il tampone di diluizione utilizzato per la conta di cellule vitali non pregiudica la vitalità dei batteri che sono state incubate nelle circostanze usate confrontando i conteggi delle cellule vitali realizzati con buffer diversa diluizione e/o acqua.
- Rimuovere i batteri non associati. Il campione viene posto in un flaconcino con acqua sufficiente, il buffer o incubazione media per coprire il materiale vegetale e capovolgendo il flacone di lavaggio.
- Determinare il numero di batteri vitali allegati, mediante omogeneizzazione.
- Il campione viene posto in un mortaio sterile o frullatore o altro dispositivo di omogeneizzazione. Coprire con un volume misurato di acqua sterile, medium di incubazione o27di tampone di lavaggio. Utilizzare un volume sufficiente a coprire il campione. Per un frullatore utilizzare 100 mL.
- Macinare il campione fino a quando esso è ben omogeneizzato. Controllare il pH dopo omogeneizzazione per essere sicuri che acido rilasciato dal tessuto della pianta non ha causato una forte diminuzione nel pH inferiore a 7. Se il pH è sceso è possibile utilizzare un buffer come tampone fosfato nel liquido omogeneizzazione per mantenere il pH.
- Determinare il numero di cellule vitali.
- Usare i batteri contrassegnati con resistenza agli antibiotico.
- In situazioni in cui è presente più di un tipo di batterio, contrassegnare ceppi batterici utilizzando spontanea resistenza agli antibiotici (in genere rifampicina e acido nalidixico).
- Determinare il livello di antibiotico da utilizzare. Se culture degli altri organismi dovuti essere presenti in incubazione sono disponibili, piastra di queste colture cresciute sotto le condizioni dell'incubazione previsto con piante su piastre contenenti un intervallo di concentrazioni del scelto antibiotiche. Determinare la concentrazione più bassa che non permette la crescita di uno qualsiasi di questi organismi. Questa è la più bassa concentrazione di antibiotico a cui i batteri di prova dovrà essere resistente.
- Ottenere spontanee mutanti resistenti agli antibiotici. Crescere una cultura dei batteri nel terreno ricco registro tardo o fase stazionaria. Piastra di 0,1 mL non diluito su un piatto che contiene l'antibiotico desiderato a una concentrazione appropriata. Determinare le concentrazioni da utilizzare come descritto nel passaggio 7.3.1.1. Mantenere e purificare i batteri che crescono e sono così resistenti agli antibiotici.
- Determinare che la resistenza agli antibiotico non riduce la crescita dei batteri facendo una curva di crescita in terreno liquido della controllante e ceppi antibiotico-resistenti. Determinare che i batteri resistenti agli antibiotici mostrano lo stesso livello di colonizzazione del materiale vegetale axeniche come ceppo, se questo è possibile.
- In situazioni in cui è presente più di un tipo di batterio, contrassegnare ceppi batterici utilizzando spontanea resistenza agli antibiotici (in genere rifampicina e acido nalidixico).
- Determinare il numero di batteri vitali associato a piante coltivate in sabbia.
- Rimuovere la pianta dal contenitore e dalla sabbia. Per rimuovere piante dai contenitori, prima di rimuovere il materiale sigillante sulla parte superiore e inferiore del contenitore. Posizionare il contenitore su un pezzo di carta sterile e rimuovere delicatamente la sabbia intero o suolo e pianta come un grande cilindro conico battendo delicatamente il contenitore contro una superficie per allentare il materiale.
- Se necessario, utilizzare una spatola o una barra per allentare il materiale intorno ai bordi andando in attraverso il fondo del contenitore.
- Quando il cilindro di sabbia o terreno contenente l'impianto è libero sulla carta, è possibile dividerlo giù al centro per rivelare la radice della pianta. Se lo si desidera, è possibile prelevare campioni di sabbia da vicino al bordo del contenitore, così come vicino alla radice. Questo può essere utile per determinare la diffusione (e accumulazione e crescita) dei batteri.
- Misurare la lunghezza della radice. Prendere la radice e rimuovere la sabbia e batteri di aderire liberamente alla radice (il materiale di rizosfera) immergendo la radice in un volume misurato di acqua o di buffer e scuotendolo delicatamente. Determinare il numero di cellule vitali dei batteri la sospensione risultante dalla placcatura su un supporto adatto come agar Luria. Questo rappresenta il numero di batteri strettamente associata con la radice.
- Rimuovere la pianta dal contenitore e dalla sabbia. Per rimuovere piante dai contenitori, prima di rimuovere il materiale sigillante sulla parte superiore e inferiore del contenitore. Posizionare il contenitore su un pezzo di carta sterile e rimuovere delicatamente la sabbia intero o suolo e pianta come un grande cilindro conico battendo delicatamente il contenitore contro una superficie per allentare il materiale.
- Rimuovere i batteri strettamente associati di sonicazione e determinare i loro numeri come descritto al punto 7.1.
- In alternativa, per determinare la posizione sulla radice dei batteri strettamente associati posto la radice lavata sulla superficie di una piastra Petri contenente agar nutriente o altro mezzo idoneo. Osservare la posizione delle colonie batteriche sulla radice i prossimi 3 giorni usando un microscopio da dissezione o la lente di ingrandimento.
- Esprimere i risultati come numero di batteri per pianta, per cm2 di superficie, per lunghezza della radice cm o per grammo di peso fresco del tessuto. Fare più replicati il giorno stesso e anche fare repliche in giorni diversi, utilizzando differenti colture batteriche e diversi lotti di materiale vegetale.
8. determinare se un effetto di condizioni di incubazione su adesione è dovuto una risposta di batteri o la pianta
- Utilizzare materiale vegetale morto o ucciso.
- Sottoporre l'impianto di materiale ad una varietà di prodotti chimici, fissativi o altri trattamenti come il calore per ucciderlo. Lavare accuratamente il materiale vegetale in acqua e incubazione medio dopo l'uso di uno qualsiasi di questi trattamenti. Quindi inoculare i batteri. Questo non distruggerà la superficie della pianta ma lo renderà metabolicamente inattivo affinché non può rispondere ai batteri.
- Misurare, adesione batterica come descritto nei passaggi 6 e/o 7. Inoltre determinare il numero di cellule vitali aggiunto per l'incubazione con il materiale vegetale all'inizio dell'incubazione e il numero di cellule vitali presenti alla fine dell'incubazione per garantire che nessun prodotti chimici tossici erano presente durante l'incubazione.
- Utilizzare materiale inanimato.
- L'adesività batterica uso di materiale inanimato, per determinare se un effetto visto su aderenza batterica di materiale vegetale è a causa di un effetto sulla pianta o i batteri. Scegliere un materiale inanimato a cui associare i batteri e che è simile nella forma e dimensioni al materiale vegetale studiato. Le possibilità includono filtri di carta di tutti i tipi (cellulosa, nitrocellulosa, fibra di vetro, policarbonato, ecc.), filati (nylon, cotone, poliestere, lana di vetro, ecc.), vetro o lamelle di plastica, in acciaio inox coupon e dialisi membrane.
- Lavare il materiale inanimato accuratamente in acqua ed il mezzo in cui l'incubazione avviene e sterilizzare prima dell'uso. La maggior parte dei materiali elencati al punto 8.2.1 sono stabile alla sterilizzazione in autoclave.
- Incubare il materiale inanimato sotto le condizioni desiderate e punteggio come descritto nei passaggi 6 e 7. Un esempio dell'uso di fili di nylon per determinare che il grippaggio ridotto di a. tumefaciens ai peli radicali alle concentrazioni di calcio alto è dovuto (almeno in parte) per un effetto di calcio sui batteri è mostrato nella Figura 428.
Risultati
A. tumefaciens colonizza superfici radicolari. Al fine di determinare se la produzione batterica di cellulosa svolge un ruolo nella colonizzazione della radice, gli effetti delle mutazioni batteriche che impediscono la sintesi di cellulosa sono stati esaminati16. Le tecniche descritte nei passaggi 1.3 e 7.1 sono state usate. Semi di pomodoro erano superficie sterilizzato e germinati in acqua sterile. Quando le radici erano lunghi circa 2 cm erano immersi in una sospensione di 105 batteri / mL e piantati nel terreno pastorizzato in contenitori. Le piante sono state coltivate per 14 giorni a 25 ° C su un ciclo di 12 h luce/12 h scuro. Dopo l'indicato volte le piante sono stati rimossi dai contenitori. Le radici sono state lavate e sonicate in un sonicatore vasca per rimuovere i batteri associati. Numeri batterici sono stati determinati utilizzando i conteggi delle cellule vitali. La figura 2 Mostra l'effetto di due mutazioni differenti di cellulosa-meno la capacità dei batteri di colonizzare le radici di pomodoro. Anche se le deviazioni standard di alcune misurazioni erano alti come 0,9 log10 (un problema comune con questo tipo di misura) la riduzione nell'associazione dei mutanti cellulosa-meno è chiaramente evidente e possiamo concludere che la produzione batterica di cellulosa aiuta i batteri nella colonizzazione delle radici di pomodoro.
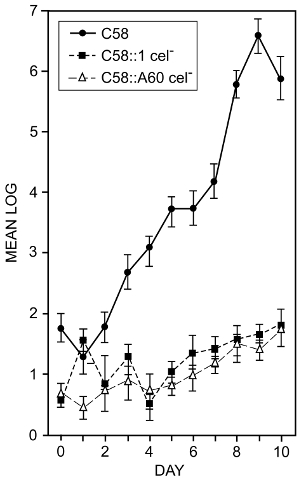
Figura 2 : Root colonizzazione da wild-type e mutanti di cellulosa-meno di a. tumefaciens. Numero totale di log10 dei batteri per cm lunghezza della radice ha recuperato dalle radici di pomodoro inoculati con wild-type a. tumefaciens strain C58 e mutanti di cellulosa-meno C58:1 e C58:A60. I numeri indicati sono il mezzo da un minimo di quattro esperimenti separati. Le barre indicano le deviazioni standard dei mezzi. Le radici sono state inoculate immergendoli in una sospensione di 105 batteri / mL per un minuto. Le piante sono state coltivate in contenitori e i batteri senza bloccare aderenti sono stati rimossi lavando le radici in buffer in un flaconcino di vetro di lavaggio. Strettamente aderenti batteri sono stati rimossi usando un sonicatore vasca e la sospensione risultante placcato per determinare cellule vitali conta16. Questa figura è stata modificata da Matthysse e McMahan. Clicca qui per visualizzare una versione più grande di questa figura.
Il ruolo di esopolisaccaridi nell'associazione di e. coli e altri batteri di germogli di erba medica è stato esaminato. Alcuni focolai di malattia diarroica dovuto Escherichia coli O157: H7 sono stati rintracciati a germogli contaminati. Associazione dei batteri wild type e mutanti in grado di fare vari esopolisaccaridi è stata misurata utilizzando i metodi descritti ai punti 1.1, 5.1 e 7.2. Germogli di erba medica sono stati superficie sterilizzato e germinato per un giorno in acqua sterile a 25 ° C al buio. Quattro germogli con annesso tegumenti sono stati collocati in piatti di plastica sterili contenente 5 mL di acqua. I batteri cresciuti in brodo di Luria sono stati aggiunti a una concentrazione finale di circa 5 x 103 millilitri. I germogli inoculati sono stati incubati a 25 ° C al buio per 3 giorni. I germogli sono stati lavati due volte in 5 mL di acqua sterile in un flaconcino di vigorosa inversione e omogeneizzati in buffer utilizzando un omogeneizzatore di vetro Teflon a motore di lavaggio. Precedenti esperimenti utilizzando batteri contrassegnato con un plasmide che trasporta il gene GFP ha mostrato nessun batteri interiorizzato anche batteri dalla superficie facilmente sono stati osservati. I risultati sono mostrati nella tabella 1. Sono stati esaminati due ceppi di e. coli O157: H7. In entrambe le razze la produzione di acid(PGA) Poli-β-1,6-glucuronico è apparso per rendere il più grande contributo all'associazione di patogeni di Escherichia coli per piantare le superfici. Acido colico ha anche svolto un ruolo significativo nell'associazione. Mentre la riduzione nell'associazione in cellulosa-minus mutanti era significativa non era grande come quella per gli altri due polisaccaridi.
| Effetti delle mutazioni nei geni di produzione di esopolisaccaridi sull'associazione di E. coli O157: H7 per germogli e aperti tegumenti | |||
| Ceppo batterico | Mutazione o genotipo (fenotipo pertinente) | 10 numero di batteri associati al germoglio o tegumento log | |
| Germogli di erba medicab | Aperti tegumenti | ||
| 86-24 | Nessuno (wild type) | 4,7 ± 0.6 | 5,6 ± 0,2 |
| 8624N | yhjN (cellulosa-minus) | 2,9 ± 0,7c | 3,5 ± 0.6c |
| 8624C | wcaD (meno acido colico) | 1,8 ± 0,7c | 2.4 ± 0.5c |
| 8624P | pgaC (PGA-minus) | < 1.0c | 1,0 ± 1.0c |
| DEC4A | Nessuno (wild type) | 5,6 ± 0,2 | 6,1 ± 0,3 |
| DEC4AN | yhjN (cellulosa-minus) | 4,8 ± 0,8d | 4,1 ± 0,8d |
| DEC4AC | wcaD (meno acido colico) | 3,9 ± 0.5c | 4,8 ± 0,8d |
| DEC4AP | pgaC (PGA-minus) | < 1.0c | 1,2 ± 0,7c |
| media ± deviazione standard di un minimo di tre misure del numero (registro10) di batteri associati dopo 3 giorni. | |||
| Germogli di b sono stati lavati prima misurazione. | |||
| c significativamente differenti dal wild type: P < 0.01. | |||
| d significativamente differenti dal wild type: P < 0.05. | |||
| Questa tabella è stata modificata da Matthysse, Deora, Mishra e Torres10. | |||
Tabella 1: effetti delle mutazioni nei geni di produzione di esopolisaccaridi sul legame di Escherichia coli O157: H7 per germogli. Al fine di determinare il ruolo delle varie esopolisaccaridi e lipopolisaccaride nell'associazione di patogeni Escherichia coli O157: H7 ceppi di germogli di erba medica, l'associazione di un insieme di mutanti per i germogli e aperti tegumenti è stato esaminato usando i metodi descritto nel passaggio 6. I risultati hanno mostrato che poli-ß-1, 6 -N-acetil-D-glucosamina (PGA) sembra essere essenziale per l'associazione di germogli e che l'acido sia cellulosa e colanic sono necessari per legame massimo di e. coli O157. Questa tabella è stata modificata da Matthysse, Deora, Mishra e Torres10.
Al fine di determinare se la produzione di PGA è sufficiente per causare associazione batterica piantare le superfici, un plasmide (pMM11) che trasportano l'operone clonato codifica i geni richiesti per PGA produzione è stato introdotto in due diversi tipi di batteri che non normalmente essere in grado di legarsi al pomodoro radici10. A. tumefaciens A1045 è un mutante del ceppo selvaggio tipo C58 che non riesce a rendere ciclico-β-1,2 glucano e inoltre non riesce ad associare a piantare superfici29. Sinorhizobium meliloti 1021 che forma noduli radice su erba medica non riesce ad associare ai non-legumi tra cui pomodoro12. Le tecniche descritte nei passaggi 1.1, 5.1, 6.3 e 7.1 sono state utilizzate per determinare se la capacità di fare PGA generalmente aumentato associazione batterica a superfici radicolari. Semi di pomodoro erano superficie sterilizzato e germinati in acqua sterile. Le radici sono state tagliate in segmenti di 1 cm di lunghezza e disposto in acqua sterile e i batteri sono stati inoculati. Come queste due specie di batteri crescono a ritmi diversi, associazione è stata misurata in momenti diversi per consentire all'incirca uguali quantità di crescita batterica. La presenza del plasmide pMM11 causato un simile aumento significativo del numero di batteri associati di entrambi di specie (tabella 2)10. Un aumento significativo nell'associazione è stato anche visto al microscopio ottico, ma l'associazione è stata molto diversa per le due specie (Figura 3). A. tumefaciens A1045 associato come singoli batteri per la superficie della radice. S. meliloti rilegato in cluster di grandi dimensioni in cui solo alcuni dei batteri fossero collegati direttamente alla radice e la maggior parte dei batteri sono stata fissata ad altri batteri. Questo esempio mostra che semplicemente analizzando il numero di batteri associati senza includere le osservazioni al microscopio può dare un'impressione ingannevole dei risultati di un esperimento. Sebbene entrambi i metodi (conta delle cellule vitali e osservazioni al microscopio) mostrano che pMM11 aumentato associazione batterica alle radici di pomodoro, il tipo di associazione causato dalla produzione di PGA era differente per le due specie batteriche10.
| L'effetto del plasmide pMM11 sui Binding dei batteri alle radici di pomodoro | ||
| Ceppo batterico | Plasmide | Numero di batteri associata a lunghezza della radice mm |
| A. tumefaciens A1045un | nessuno | 0,25 x 103 ± 0.25 x 103 |
| pBBR1mcs (vettore) | 0,25 x 103 ± 0.25 x 103 | |
| pMM11 (sintesi di PGA) | 10 x 103 ± 0.25 x 103 | |
| S. meliloti 1021b | nessuno | nessuno rilevato |
| pBBR1mcs (vettore) | nessuno rilevato | |
| pMM11 (sintesi di PGA) | 50 x 103 ± 5 x 103 | |
| un'associazione batterica è stata misurata dopo 2 ore | ||
| b Associazione batterica era misura dopo 18 ore | ||
| Questa tabella è stata modificata da Matthysse, Deora, Mishra e Torres10. | ||
Tabella 2: l'effetto di un plasmide portatori di geni per la sintesi di PGA sul legame di A. tumefaciens A1045 e S. meliloti 1021 a segmenti di radice di pomodoro. Al fine di esaminare la possibilità di poli-ß-1, 6 -N-acetil-D-glucosamina (PGA) per promuovere l'associazione di batteri per piantare radici, l'effetto di un plasmide che attribuisce la capacità di fare PGA (pMM11) l'associazione di due ceppi di batteri pianta-collegato a radici di pomodoro è stato esaminato. Nessun ceppo di batteri ha mostrato significativa associazione alle radici di pomodoro in assenza del plasmide o in presenza del plasmide senza i geni che codificano la sintesi di PGA (pBBR1mcs). L'aggiunta del plasmide portatori di geni di sintesi PGA aumentato associazione di entrambi i tipi di batteri. Perché a. tumefaciens cresce più velocemente di quanto S. meliloti associazione è stata misurata dopo 2 h di incubazione per a. tumefaciens e dopo 18 h per S. meliloti. Le tecniche utilizzate sono quelle descritte nel passaggio 7. Questa tabella è stata modificata da Matthysse, Deora, Mishra e Torres10.
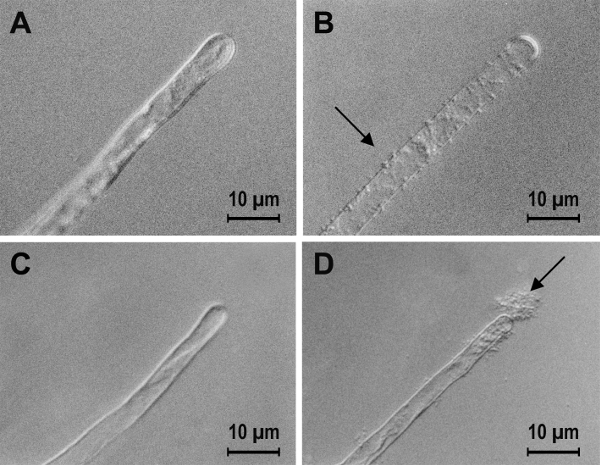
Figura 3 : L'effetto del pMM11 di plasmidi che trasportano i geni di biosintesi di PGA sul legame di a. tumefaciens A1045 e S. meliloti 1021 di peli radicali pomodoro. Associazione di pomodoro di peli radicali della A) a. tumefaciens A1045, B) a. tumefaciens A1045 pMM11, C) S. meliloti 1021 e D) S. meliloti 1021 pMM11. Anche se l'aumento nell'associazione delle due specie batteriche è approssimativamente simile i dettagli dell'associazione come visto al microscopio ottico sono molto diversi. Le tecniche utilizzate sono quelle descritte nel passaggio 6. Questa figura è stata modificata da Matthysse, Deora, Mishra e Torres10. Clicca qui per visualizzare una versione più grande di questa figura.
A volte è possibile utilizzare l'associazione per le superfici non-biologici per aiutare a distinguere il contributo dei batteri e della pianta in un'interazione specifica. Il polysaccharide(UPP) unipolare di a. tumefaciens ha dimostrato di essere in grado di mediare associazione batterica ad una varietà di entrambe le superfici biologiche e non biologiche30. Calcio è stato osservato per inibire il legame di a. tumefaciens piantare superfici mediate dal UPP28. Al fine di determinare se l'inibizione di ioni del calcio di associazione batterica per piantare le superfici è dovuta ad un effetto sui batteri o sulla superficie della pianta, l'associazione dei batteri a fili di nylon è stata esaminata. Le tecniche vengono descritti nel passaggio 8.2 sono stati usati. Semi di pomodoro erano superficie sterilizzato e germinati in acqua come descritto nel passaggio 1. I batteri sono stati coltivati in terreno minimo con saccarosio e aggiunto al pomodoro radici o fili di nylon ad una concentrazione finale di circa 105/ml come descritto al punto 5.1. L'effetto di aggiunta CaCl2 sull'associazione batterica alle radici di pomodoro e fili di nylon è stato esaminato al microscopio. La figura 4 Mostra una simile inibizione di associazione di calcio utilizzando sia superficiale, suggerendo che l'effetto di calcio è principalmente sui batteri10.

Figura 4 : L'effetto di calcio sul legame di a. tumefaciens al pomodoro di peli radicali e fili di nylon. A. tumefaciens è stato incubato con radici di pomodoro (A e B) o fili di nylon (C e D) per 24 h in un 01:10 diluizione di MS sali e un 01:20 diluizione di terreno minimo di AB, 0,4% di saccarosio (A e C) o in un 01:10 diluizione di MS sali e 01:20 diluizione del terreno minimo AB , 0,4% saccarosio contenente 60 mM CaCl2 (B e D)31. L'aggiunta di CaCl2 ha inibito il grippaggio batterica sia radici e fili di nylon, suggerendo che l'inibizione era principalmente a causa di un effetto sui batteri piuttosto che sulla superficie della pianta. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
È importante essere consapevoli di tutte le superfici a cui i batteri possono aderire durante l'esperimento. Così i batteri che sono in grado di legarsi al vetro possono essere sottovalutati se i conteggi delle cellule vitali sono fatte utilizzando provette e pipette. Se le piante sono cresciute in agar o terreno e alcuni dell'agar o terreno rimane sulle piante i batteri possono associare il materiale aderente anziché alle piante. D'altra parte, lavare la superficie della pianta, specialmente nel caso di radici, può rimuovere rivestimenti di superficie naturali come mucose e quindi alterare i risultati delle prove di aderenza.
È importante essere certi che i batteri aggiunti alla miscela di incubazione rimangono vivi durante l'esperimento. Così i conteggi delle cellule vitali dei batteri liberi così come allegati ordinariamente dovrebbero essere fatta. Alcuni trattamenti o mutazioni batteriche possono ridurre il tasso di crescita batterica o effettivamente causare la morte di una frazione della popolazione batterica. Batteri vivi e morti non siano distinguibili nel microscopio a meno che non vengono utilizzati coloranti speciali. C'è un kit di macchia utile per live/dead batteri che dipende l'esclusione di coloranti dai batteri viventi. Tuttavia, se una popolazione mista di specie batteriche è presente quindi i conteggi delle cellule vitali delle specie di interesse rischia di essere il metodo più semplice per determinare se l'incubazione ha provocato la morte batterica.
Composizione media influenzerà la crescita e la sopravvivenza batterica. Radice dell'essudato e materiali rilasciato dalla ferita e tagliare siti fornirà il substrato per supportare la modesta crescita batterica. Fosfato, azoto e ferro tendono ad essere limitante in queste condizioni. Cationi bivalenti come calcio e magnesio possono influenzare l'adesione. In alcuni casi fonte di carbonio possa influenzare adesione. pH è anche importante. In generale il pH della rizosfera è tra 5.5 e 6.5.
È necessario prestare attenzione nell'utilizzo di batteri contrassegnati con resistenza agli antibiotico. Gli antibiotici più spesso utilizzati sono la rifampicina e acido nalidixico. Resistenza a questi antibiotici è in genere dovuta a mutazioni in geni cromosomici (RNA polimerasi e girasi, rispettivamente) e pertanto non possono essere facilmente trasferiti ad un altro ceppo durante l'incubazione. Questo tipo di resistenza anche non provocare la degradazione o la modifica dell'antibiotico. Marcatura di batteri con un plasmide-sopportate gene marcatore non è raccomandato a meno che il plasmide non può essere trasferito a qualsiasi altri batteri. La resistenza agli antibiotici non deve essere dovuto modifica degradazione o chimica dell'antibiotico come antibiotici batteri sensibili sarà quindi in grado di crescere su piastre antibiotici se essi si trovano vicino i batteri resistenti.
I metodi descritti in questa carta sono utili per campioni di piccole dimensioni e/o esperimenti dove i campioni devono essere contenuti (per esempio, esperimenti che coinvolgono agenti patogeni umani). Per campioni di grandi dimensioni (superiori a 100 g di materiale o di più di 50 piante) altri metodi o drastica modificazione di questi metodi sarebbero necessari10,19,32,19,33, 34 , 35 , 36. la presenza di un gran numero di microrganismi diversi le specie oggetto di studio può anche porre problemi significativi. Possibili soluzioni includono l'uso di batteri contrassegnato con una proteina fluorescente o la resistenza agli Antibiotico, come descritto nei passaggi 6.1.2 e 7.3. Tuttavia, quando i batteri di interesse sono rari individui in una grande popolazione di altri microrganismi questi indicatori non siano sufficienti per consentire una valutazione univoca dei numeri dei batteri in fase di studio.
Tutti i metodi descritti qui sono metodi di laboratorio di base. Modifiche minori sarebbero necessari per gli studi di serra. Altre importanti modifiche sono suscettibili di essere necessario per gli studi sul campo dove protozoi, insetti e altri animali predazione e clima variazione complicare la fornitura di condizioni definite per gli esperimenti. In futuro questi metodi possono essere estesa per includere le interazioni di due o più microrganismi sulla superficie della pianta.
Divulgazioni
L'autore dichiara che non ha nessun concorrenti interessi finanziari.
Riconoscimenti
L'autore ringrazia Susan Whitfield per la preparazione delle figure e Camille Martin e Hillary Samagaio per assistenza con alcuni esperimenti.
Materiali
| Name | Company | Catalog Number | Comments |
| light microscope | any | N/A | phase contrast or Nomarski optics are helpful, for studies using fluorescent markers a fluorescence microscope is required. |
| seeds | any | N/A | make a note of the seed lot number and the cultivar |
| bleach | any | N/A | |
| bath sonicator | any scientific supply company | N/A | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| nutrient agar | Difco | 001-01-8 | |
| soytone | Difco | 24360 | |
| Sand | sea sand Fisher Scientific | S25 | |
| sand | Sigma-Aldrich | S9887 | |
| conetainers | Stuewe & Sons, Inc. | Ray-Leach cone-tainers | many different sizes are available to suit the type of plant you wish to grow |
| parafilm | any scientific supply company | N/A | |
| MS salts | Sigma-Aldrich | M5524 | |
| parrafin | any | N/A | |
| centrigfuge | any scientific company | N/A | to pellet most bacteri only a small centrifuge with a max force of 10,000 XG is needed |
| vortex mixer | any scientific company | ||
| micrometer | ACCU-SCOPE Accessories | A3145 | |
| Sedgwick-rafter counting cell | Hauser Scientific | HS3800 | |
| probe-clip press-seal incubatin chamber | Sigma-Aldrich | Z359483 | |
| rifampicin | Sigma-Aldrich | R3501 | |
| naladixic acid | Sigma-Aldrich | n8878 | |
| Live/dead stain | In Vitrogen Molecular Bioprobes | L7007 |
Riferimenti
- Heaton, J. C., Jones, K. Microbial contamination of fruit and vegetables and the behaviour of enteropathogens in the phyllosphere: a review. J. Appl. Microbiol. 104, 613-626 (2008).
- Beuchat, L. R. Ecological factors influencing survival and growth of human pathogens on raw fruit and vegetables. Microbes Infect. 4, 413-423 (2002).
- Berger, C. N., et al. Fresh fruit and vegetables as vehicles for the transmission of human pathogens. Environ. Microbiol. 12, 2385-2397 (2010).
- Lugtenberg, B. J., Chin, A. W. T., Bloemberg, G. V. Microbe-plant interactions: principles and mechanisms. Antonie Van Leeuwenhoek. 81, 373-383 (2002).
- Preston, G. M., Haubold, B., Rainey, P. B. Bacterial genomics and adaptation to life on plants: implications for the evolution of pathogenicity and symbiosis. Curr. Opin. Microbiol. 1, 589-597 (1998).
- Compant, S., Duffy, B., Nowak, J., Clement, C., Barka, E. A. Use of Plant Growth-Promoting Bacteria for Biocontrol of Plant Diseases: Principles, Mechanisms of Action, and Future Prospects. Appl. Environ. Microbiol. 71, 4951-4959 (2005).
- Mathews, S. L., Smith, R. B., Matthysse, A. G. A comparison of the retention of pathogenic Escherichia coli. O157 by sprouts, leaves and fruits. Microb. Biotechnol. 7, 570-579 (2014).
- Fuqua, C., Matthysse, A. G. Methods for studying bacterial biofilms associated with plants. Methods Enzymol. 337, 3-18 (2001).
- Torres, A. G., Jeter, C., Langley, W., Matthysse, A. G. Differential binding of Escherchia coli.O157:H7 to alfalfa, human epithelial cells, and plastic is mediated by a variety of surface structures. Appl. Environ. Microbiol. 71, 8008-8015 (2005).
- Matthysse, A. G., Deora, R., Mishra, M., Torres, A. G. Polysaccharides cellulose, poly-beta-1,6-n-acetyl-D-glucosamine, and colanic acid are required for optimal binding of Escherichia coli O157:H7 strains to alfalfa sprouts and K-12 strains to plastic but not for binding to epithelial cells. Appl. Environ. Microbiol. 74, 2384-2390 (2008).
- Matthysse, A. G., et al. The effect of cellulose overproduction on binding and biofilm formation on roots by Agrobacterium tumefaciens. Mol. Plant Microbe Interact. 18, 1002-1010 (2005).
- Matthysse, A. G., Kijne, J. W., Spaink, H. P., Kondorosi, A., Hooykaas, P. J. J. Attachment of Rhizobiaceae to plant cells. The Rhizobiaceae. , 235-249 (1998).
- Matthysse, A. G. Conditioned medium promotes the attachment of Agrobacterium tumefaciens.strain NT1 to carrot cells. Protoplasma. 183, 131-136 (1994).
- Matthysse, A. G., McMahan, S. The effect of the Agrobacterium tumefaciens. attR mutation on attachment and root colonization differs between legumes and other dicots. Appl. Environ. Microbiol. 67, 1070-1075 (2001).
- Jeter, C., Matthysse, A. G. Characterization of the binding of diarrheagenic strains of E. coli.to plant surfaces and the role of curli in the interaction of the bacteria with alfalfa sprouts. Mol. Plant Microbe Interact. 18, 1235-1242 (2005).
- Matthysse, A. G., McMahan, S. Root colonization by Agrobacterium tumefaciens is reduced in cel, attB, attD, and attR mutants. Appl. Environ. Microbiol. 64, 2341-2345 (1998).
- Morton, E. R., Fuqua, C. Phenotypic analyses of Agrobacterium. Curr. Protoc. Microbiol. , (2012).
- Brandl, M. T. Plant lesions promote the rapid multiplication of Escherichia coli O157:H7 on postharvest lettuce. Appl. Environ. Microbiol. 74, 5285-5289 (2008).
- Franz, E., et al. Quantification of contamination of lettuce by GFP-expressing Escherichia coli O157:H7 and Salmonella enterica serovar Typhimurium. Food Microbiol. 24, 106-112 (2007).
- Morton, E. R., Fuqua, C. Laboratory maintenance of Agrobacterium. Curr. Protoc. Microbiol. , (2012).
- Lugtenberg, B. J., Kravchenko, L. V., Simons, M. Tomato seed and root exudate sugars: composition, utilization by Pseudomonas biocontrol strains and role in rhizosphere colonization. Environ. Microbiol. 1, 439-446 (1999).
- Murashige, T., Skoog, F. A Revised Medium for Rapid Growth and Bio Assays with Tobacco Tissue Cultures. Physiologia Plantarum. 15, 473-497 (1962).
- Danhorn, T., Fuqua, C. Biofilm formation by plant-associated bacteria. Annu. Rev. Microbiol. 61, 401-422 (2007).
- Branda, S. S., Vik, S., Friedman, L., Kolter, R. Biofilms: the matrix revisited. Trends Microbiol. 13, 20-26 (2005).
- O'Toole, G., Kaplan, H. B., Kolter, R. Biofilm formation as microbial development. Annu. Rev. Microbiol. 54, 49-79 (2000).
- Charkowski, A. O., Barak, J. D., Sarreal, C. Z., Mandrell, R. E. Differences in growth of Salmonella enterica and Escherichia coli O157:H7 on alfalfa sprouts. Appl. Environ. Microbiol. 68, 3114-3120 (2002).
- Loper, J., Suslow, T., Schroth, M. Lognormal distribution of bacterial populations in the rhizosphere. Phytopathology. 74, 1454-1460 (1984).
- Matthysse, A. G. Attachment of Agrobacterium to plant surfaces. Front Plant Sci. 5, 252 (2014).
- Douglas, C. J., Halperin, W., Nester, E. W. Agrobacterium tumefaciens mutants affected in attachment to plant cells. J. Bacteriol. 152, 1265-1275 (1982).
- Tomlinson, A. D., Fuqua, C. Mechanisms and regulation of polar surface attachment in Agrobacterium tumefaciens. Curr. Opin. Microbiol. 12, 708-714 (2009).
- Matthysse, A. G. Attachment of Agrobacterium to plant surfaces. Front Plant Sci. 5, (2014).
- Wright, K. M., et al. The endophytic lifestyle of Escherichia coli O157:H7: quantification and internal localization in roots. Phytopathology. 103, 333-340 (2013).
- Berger, C. N., et al. Fresh fruit and vegetables as vehicles for the transmission of human pathogens. Environ. Microbiol. 12, 2385-2397 (2010).
- Erickson, M. C., et al. Internalization and Fate of Escherichia coli O157:H7 in Leafy Green Phyllosphere Tissue Using Various Spray Conditions. J. Food Prot. 77, 713-721 (2014).
- McKellar, R. C., et al. Evaluation of different approaches for modeling Escherichia coli O157:H7 survival on field lettuce. Int. J. Food Microbiol. 184, 75-85 (2014).
- Wu, F. M., et al. Factors influencing the detection and enumeration of Escherichia coli O157:H7 on alfalfa seeds. Int. J. Food Microbiol. 71, 93-99 (2001).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon