È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Replica di ordinato, Nonredundant biblioteca del ceppo di Pseudomonas aeruginosa PA14 trasposone inserimento mutanti
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
L'infezione di Pseudomonas aeruginosa causa la morbosità significativa in host vulnerabili. La biblioteca di mutante di inserimento trasposone nonredundant di p. aeruginosa ceppo PA14, designato come PA14NR insieme, facilita l'analisi della funzionalità del gene in numerosi processi. Presentato qui è un protocollo per generare copie di alta qualità della libreria mutante PA14NR impostare.
Abstract
Pseudomonas aeruginosa è un batterio gram-negativo fenotipicamente e geneticamente vario e adattabile onnipresente in ambienti umani. P. aeruginosa è in grado di formare biofilm, sviluppano resistenza agli Antibiotico, produrre fattori di virulenza ed evolversi rapidamente nel corso di un'infezione cronica. Così p. aeruginosa può causare sia acuta e cronica, difficile da trattare infezioni, con conseguente morbosità significativa in alcune popolazioni di pazienti. Ceppo di p. aeruginosa PA14 è un isolato clinico umano con una struttura di genoma conservato che infetta una varietà di mammiferi e nonvertebrate padroni di casa rendendo PA14 un ceppo attraente per lo studio di questo agente patogeno. Nel 2006, è stata generata una libreria di mutante inserimento nonredundant trasposone contenente 5.459 mutanti corrispondente a 4.596 geni PA14 predetti. Da allora, la distribuzione della libreria PA14 ha permesso alla comunità di ricerca comprendere meglio la funzione dei singoli geni e vie complesse di p. aeruginosa. Mantenimento dell'integrità della biblioteca attraverso il processo di replica richiede tecniche di corretta gestione e precise. A tal fine, questo manoscritto presenta protocolli che descrivono in dettaglio i passaggi coinvolti nella replica di biblioteca, libreria di controllo di qualità e una corretta conservazione dei singoli mutanti.
Introduzione
Pseudomonas aeruginosa è un batterio gram-negativo fenotipicamente e geneticamente vario e adattabile presente nel suolo, acqua e ambienti più umani, come pure della pelle microflora. Rispetto a molte specie batteriche, p. aeruginosa ha un genoma relativamente grande di 5.5-7 Mbp con alto contenuto G + C (65-67%). Inoltre, una percentuale significativa dei suoi geni sono coinvolta nella capacità di adattamento metabolico e fanno parte di reti di regolazione, consentendo una grande flessibilità in risposta a stress ambientali1. P. aeruginosa esprime una miriade di fattori di virulenza, esibisce la propensione a forma di biofilm, possiede la capacità di coordinare le risposte attraverso più ' quorum sensing vie e visualizza una notevole capacità di sviluppare resistenza agli Antibiotico e tolleranza2,3,4,5,6,7,8. Questi attributi presenti sfide significative per il trattamento di infezioni causate da p. aeruginosa.
Croniche infezioni da p. aeruginosa può verificarsi in numerosi Stati di malattia. Fibrosi cistica (CF), una malattia genetica causata dalla mutazione del gene Regolatore di conduttanza del Transmembrane fibrosi cistica (CFTR) , provoca secrezioni inspissated, infetti all'interno delle vie respiratorie, bronchiettasia progressiva e, in definitiva, morte da guasto respiratorio9. Nell'età adulta, la maggior parte dei pazienti con i CF è cronicamente infettata con p. aeruginosa, che svolge un ruolo chiave nella morbosità e mortalità connesse con questa malattia10. Inoltre, i pazienti con ustioni gravi lesioni11, i tracheostomies12, protesi articolari13o di cateteri indwelling14 sono a rischio per l'infezione di p. aeruginosa legata alla capacità dei batteri di forma biofilm e fuga host le risposte infiammatorie15. Ulteriormente, la colonizzazione si verifica senza concorrenza dopo aver selezionata una popolazione multi-antibiotico-resistente o tollerante a trattamento antimicrobico ad ampio spettro, sequenziale12,16,17 , 18. migliore comprensione della patogenesi di p. aeruginosa avrà implicazioni significative per numerosi Stati di malattia.
Diversi isolati clinici di p. aeruginosa , compresi i ceppi PAO1, PA103, PA14 e PAK, sono stati studiati estesamente per studiare le diverse caratteristiche della patogenesi di p. aeruginosa . PA14 ceppo è un isolato clinico che appartiene a uno dei più comuni gruppi clonale in tutto il mondo19,20 e non è stato ampiamente attraversato in laboratorio. PA14is altamente virulenti nei vertebrati dell'infezione, con un notevole endotossina profile21, pili struttura22,23, delle isole di patogenicità tipo sistema di secrezione di III (SS3), citotossicità verso mammalian cells24 e profili di antibiotico resistenza e persistenza25. Inoltre, PA14 è anche altamente virulento in numerosi sistemi di modello ospite-patogeno, compresi foglia pianta infiltrazione modelli26,27,elegans di Caenorhabditis infezione modelli28, 29, insetto modelli30,31, così come polmonite del mouse modelli32,33 , e ustioni cutanee modelli34.
Genoma mutante raccolte sono insiemi di isogeniche mutanti nei geni non essenziali che costituiscono strumenti molto potenti per capire la biologia di un organismo consentendo analisi della funzione genica su scala genomica. Due vicino a saturazione trasposone inserimento mutante librerie costruite in p. aeruginosa sono attualmente disponibili per la distribuzione. I siti di inserimento dei trasposoni sono stati determinati per entrambe le librerie. Queste librerie nonredundant cosiddette facilitano studi di genoma di ceppi batterici diminuendo notevolmente il tempo e costo coinvolti screening includevano mutanti trasposone casuale. La libreria mutante p. aeruginosa PAO1 trasposone, costruita nel MPAO1 isolare di ceppo PAO1 utilizzando trasposoni ISphoA/ Ah ed èlacZ/ hah35, è curata dal laboratorio Manoil, Università di Washington. La libreria è costituita da una collezione di sequenza-verificato di 9.437 trasposone mutanti che fornisce una copertura ampia genoma e include due mutanti per la maggior parte dei geni36. Informazioni sulla libreria mutante di p. aeruginosa PAO1 trasposone sono disponibile sul sito pubblico, accessibile da internet Manoil laboratorio presso http://www.gs.washington.edu/labs/manoil/libraryindex.htm. P. aeruginosa ceppo PA14 trasposone nonredundant inserimento mutante Set di libreria (PA14NR) costruito nel ceppo PA14 utilizzando trasposoni MAR2xT7 e TnphoA37 è attualmente distribuito dal dipartimento di pediatria presso il Massachusetts General Hospital. Il PA14NR Set comprende una collezione di più di 5.800 mutanti con inserimenti di singolo trasposone in geni non essenziali37. Dettagli sulla costruzione del Set PA14NR sono descritti in http://pa14.mgh.harvard.edu/cgi-bin/pa14/home.cgi?section=NR_LIB il sito pubblico, accessibile da internet, che contiene anche una varietà di strumenti di ricerca online per facilitare l'uso della PA14NR Impostare.
Il Set di PA14NR originale comprendeva 5.459 mutanti, selezionati da una libreria completa di circa 34.000 mutanti inserimento del trasposone casuale, che corrispondono a geni PA14 4.596 predetti che rappresentano il 77% di tutti predetto PA14 geni37. Dopo la costruzione della biblioteca nel 2006 sono stati aggiunti nuovi mutanti, e attualmente il PA14NR Set include più di 5.800 mutanti38 che rappresentano circa 4.600 PA14 geni. La maggior parte dei mutanti PA14 trasposoni sono stata generata nel tipo selvaggio sfondo37. Dettagli riguardanti ogni membro della libreria mutante, tra cui background genetico, sono disponibili attraverso la ricerca del database online o scaricando il foglio di calcolo di Nonredundant biblioteca, entrambe le funzionalità disponibili nel sito Web PA14 (http:// PA14.MGH.Harvard.edu/cgi-bin/PA14/Home.cgi). La maggior parte dei mutanti sono stata creata utilizzando il MAR2xT7 trasposoni (MrT7), con un piccolo set creato utilizzando il trasposone TnPhoA (phoA)37. Ogni trasposone ha una cassetta di resistenza agli antibiotici, che consente la selezione di mutanti utilizzando gentamicina (MrT7) o kanamicina (phoA). Il set di PA14NR di mutanti viene memorizzato in piastre da 96 pozzetti sessantatre e comprende due ulteriore controllo 96 pozzetti piastre, che consistono di tipo selvaggio PA14 inoculati e pozzi non inoculati intercalato in un pattern predefinito. Il formato di piastra a 96 pozzetti accoppiato con gli strumenti di ricerca online notevolmente facilita lo sviluppo personalizzato di analisi che consentono agli utenti di identificare facilmente i geni connessi con i fenotipi mutanti di screening. Gli strumenti di ricerca online facilitano anche la ricerca e selezione di mutanti pertinenti aggiuntive necessarie per ulteriori studi.
Il PA14 PAO1 trasposone mutante sono le librerie e le risorse globali molto importante per la comunità scientifica, e si completano a vicenda nella convalida la funzione dei geni sconosciuto e le vie di questo agente patogeno batterico. Per coincidenza, dopo la costruzione delle librerie di mutazione trasposone PAO1 e PA14, analisi di sequenziamento del DNA di pieno-genoma di molti isolati di p. aeruginosa ha dimostrato che PAO1 e PA14 appartengono a diversi subclades principali del P. aeruginosa phylogeny7,39,40,41. Perché gli isolati clinici di p. aeruginosa sono trovati distribuiti in tutta la filogenesi, il fatto che PAO1 e PA14 appartengono a diversi p. aeruginosa sottogruppi aumenta il valore delle due librerie di mutazione trasposone per comparativa studi.
Pubblicazioni che descrivono la costruzione e screening di librerie mutante batteriche, tra cui p. aeruginosa librerie35,37,42, sono facilmente disponibili in letteratura. Tuttavia, al meglio della nostra conoscenza, nessun protocolli pubblicati che descrivono le procedure dettagliate e tecniche utilizzate per la replica, con manutenzione e convalida delle librerie mutante batteriche sono disponibili.
La metodologia descritta nella presente pubblicazione descrive un insieme di tre protocolli che semplificano l'utilizzo e la manutenzione del Set PA14NR. Il primo protocollo descrive la replica della libreria come raccomandato ai destinatari del Set PA14NR. Il secondo protocollo include linee guida per striature, la crescita e la memorizzazione di singoli mutanti identificati utilizzando il Set di PA14NR. Il terzo protocollo descrive le tecniche di controllo di qualità, tra cui l'amplificazione di PCR dei frammenti da mutanti trasposoni e successivo sequenziamento per confermare identità mutanti. Questo insieme di protocolli può anche essere adattato per la replica e la manutenzione di altre librerie mutante batteriche o collezioni. La replica delle librerie mutante batteriche o collezioni è altamente consigliata per preservare l'integrità della copia del"Maestro" (copia originale ricevuto). Replica di diverse copie del Set PA14NR per uso di laboratorio di routine minimizza la probabilità di contaminazione interwell la copia master.
Protocollo
Attenzione: Utilizzare misure di sicurezza standard BSL-2 durante la manipolazione di p. aeruginosa, un agente patogeno umano. Se siete un individuo immunocompromised o avere qualsiasi condizione medica che aumenta la suscettibilità alle infezioni batteriche, prendere speciali precauzioni quando si lavora con P. aeruginosa. Consultare l'ufficio di biosicurezza nel tuo istituto e ottenere l'approvazione del vostro medico prima di lavorare con il PA14 NR impostato o librerie mutante di batteri patogeni.

Figura 1: Panoramica del protocollo i: replica della PA14NR impostata. Giorno 1: Replicare colture congelate mutante da "copia master" di PA14NR Set in LB agarizzati e crescere mutanti durante la notte a 37 ° C. 2 ° giorno: Trasferimento crescita mutante da LB agarizzati a profonda ben blocchi contenenti brodo liquido LB, crescere durante la notte a 37 ° C con agitazione a 950 giri/min. Giorno 3: Mescolare pernottamento LB culture con glicerolo, poi trasferimento a piastre a 96 pozzetti destinazione per l'archiviazione a lungo termine. Posto 96 piatti piatto in congelatore a-80 ° C. Clicca qui per visualizzare una versione più grande di questa figura.
Figura 2: configurazione consigliata. Sterilità e flusso di lavoro deve essere mantenute tramite l'utilizzo di opportune precauzioni. Clicca qui per visualizzare una versione più grande di questa figura.
1. protocollo i: replica del PA14NR impostato
Nota: La replica della libreria può essere ottenuta dividendo il PA14NR Set in quattro sottogruppi di sedici piastre ciascuno che possono essere elaborati in quattro settimane consecutive. Generazione di 1 a 6 copie aderisce al flusso di lavoro settimanale indicato nella tabella 1, mentre la generazione di più di 6 copie segue il flusso di lavoro settimanale indicato nella tabella 2. Per generare 12 copie del Set PA14NR, inoculare lo stesso sottoinsieme di PA14NR in liquido LB media il giorno 2 e nuovamente il giorno 3 (dallo stesso set di piastre di agar replicato il giorno 1) e trasferimento pernottamento mutante culture in lastre di copia il giorno 3 e 4 giorno rispettivamente.
| Giorno 0 | 1 ° giorno | 2 ° giorno | 3 ° giorno |
| Giorno della preparazione della | Crescita dei mutanti PA14NR impostato su LB agarizzati | Crescita di PA14NR impostare mutanti nei mezzi liquidi LB | Trasferimento di culture mutante PA14NR Set di piastre di destinazione |
Tabella 1: pianificazione di replica di 1-6 copie del Set PA14NR. Replica di un piccolo numero di copie può aderire a un flusso di lavoro settimanale.
| Giorno 0 | 1 ° giorno | 2 ° giorno | 3 ° giorno | 4 ° giorno |
| Giorno della preparazione della | Crescita dei mutanti PA14NR impostato su LB agar | Crescere di mutanti PA14NR impostato in terreno liquido LB | Trasferimento di giorno 3 culture mutante per generare 1 ° set di 6 copie di PA14NR Set | Trasferimento di giorno 4 culture mutante per generare 2 ° set di 6 copie di PA14NR Set |
| Crescita di PA14NR impostare mutanti nei mezzi liquidi LB |
Tabella 2: pianificazione di replica di fino a 12 copie del Set PA14NR. Replica di un numero maggiore di copie richiederà la stratificazione all'interno di un flusso di lavoro settimanale.
- Giorno 1: Crescita di PA14NR Set Master copia su piastre di agar LB
- Indossare guanti, camice e mascherina per gestire il Set di PA14NR.
- Chiara panca e pulire la superficie con etanolo al 70%.
- Preparare LB agar media43 e sterilizzare in autoclave per 25 min. Cool media a 55 ° C in acqua del bagno e aggiunta o gentamicina 15 µ g/mL, per mutanti contenenti MAR2xT7 trasposone inserzioni, o 200 kanamicina µ g/mL, per mutanti contenenti TnphoA inserimenti.
- Versare fuso LB agar in piastre rettangolari con circa 60 mL di supporti per piastra. Secco piastre in una cappa sterile per circa 1 h prima di utilizzano. Assicurarsi che non c'è nessuna condensazione di acqua sulla superficie dell'agar. Conservare le piastre a 4 ° C, se necessario.
- Creare un campo sterile con un becco Bunsen su etanolo-spazzato da banco e impostare la pagina di contenitori con soluzioni appropriate per la sterilizzazione di pin replicator (Vedi punto 1.1.9). Uso aprire contenitori di plastica (5,25" L x 4,25" W x 1.75 "H dimensioni) per la sterilizzazione di pin replicator. Contenitori di plastica di autoclave prima dell'uso.
Nota: Il calore della fiamma bruciatore di Bunsen crea una corrente di convezione che riscalda lo spazio sopra la fiamma e solleva le particelle nell'aria alto e lontano l'aria più fresca sotto, mantenendo la zona di lavoro sterile. - Rimuovere un massimo di quattro piastre master PA14NR impostato dal congelatore-80 ° C (per evitare inutili scongelamento) e metterli su ghiaccio secco in vaschetta ghiaccio 4 L.
- Prendere la piastra 96 pozzetti master per essere replicati da ghiaccio secco e posizionarlo sulla panchina per consentire lo scongelamento breve, che avrà solo il tempo necessario per la sterilizzazione dei perni replicator (circa 3-4 min) (vedere il punto 1.1.8). Segnare la posizione della coordinata A1 sulla piastra di agar prima timbratura. Allineare i master piatto e piastra di agar con la coordinata di A1 di entrambe le piastre in alto a sinistra.
- Sterilizzare il replicatore "perni" attenendosi alla procedura descritta di seguito. Toccare leggermente replicator dopo ogni passaggio per rimuovere il liquido in eccesso.
- Immergere i perni in 250 mL di 0.3-0.5% ipoclorito di sodio (candeggina 10%) per 30 secondi. Minimizzare il contatto con soluzione di ipoclorito di sodio, come si può portare a danni di pin. Immergere i perni in 250 mL ddH sterile2O o acqua ultrapura sterile per 10 secondi, quindi in 250 mL di etanolo al 70% per 30 secondi, quindi in 250 mL di etanolo al 95% per 2 min.
- Fiamma sterilizzare perni replicator holding replicator perpendicolarmente alla fiamma bruciatore di Bunsen, lentamente si avvicina la fiamma fino a produzione di etanolo, quindi ritirarla immediatamente dalla fiamma. Fiamma si spegne una volta tutte le ustioni di etanolo fuori. Tenere un coperchio o un contenitore simile vicino a soffocare la combustione dell'etanolo, se necessario.
Nota: Usare estrema cautela quando si lavora con etanolo vicino a una fiamma. NON tenere replicator direttamente sopra la fiamma. - Perni di cool da appuntare su un piatto rettangolare sterile inutilizzato che contiene LB agarizzati per 30 secondi.
- Brevemente in alluminio caldo seal da PA14NR Set master piatto con la mano prima si staccava. Fare questo durante il raffreddamento perni replicator. Rimuovere con attenzione per impedire che il sigillo ritocco la piastra di alluminio di tenuta. Utilizzare una pinzetta per togliere eventuali residui della guarnizione in alluminio.
- Inserire gli spinotti replicator nei master piatto, spingendo delicatamente e replicator oscillante nelle quattro direzioni per assicurarsi che i perni faccia contatto con colture batteriche congelate in ciascuno dei 96-pozzi. Prestare particolare attenzione ai pozzi situato sul bordo esterno della piastra premendo perni replicator contro colture congelate nei pozzetti situati al bordo della piastra.
- Posizionare delicatamente perni replicator sulla superficie della piastra di agar. Replicator si muovono in un leggero movimento circolare a forma di mini-prati di circa 4-5 mm per ogni ceppo mutante. Evitare la possibilità che i mini-prati possono sovrapporsi, per evitare la contaminazione incrociata.
- Guarnizione PA14NR Set master piatto con una nuova guarnizione di alluminio sterile. Non toccare il lato adesivo della guarnizione di alluminio in qualsiasi punto per evitare la contaminazione. Assicurarsi che ogni pozzetto e i bordi della piastra sono completamente sigillati utilizzando un rullo di piastra. Piastra 96 ritorno a ghiaccio secco.
- Ripetere la procedura per ogni lastra.
- Pulire tutte le superfici di lavoro con etanolo al 70% dopo aver maneggiato il Set di PA14NR.
- Trasferimento replicati piastre di agar in incubatore a 37 ° C ed incubare per una notte.
- Giorno 2: Crescita della copia PA14NR Set Master in brodo liquido LB
- Preparare LB brodo liquido43 contenenti gentamicina o 15 µ g/mL, per mutanti contenenti MAR2xT7 trasposone inserzioni, o 200 kanamicina µ g/mL, per mutanti contenenti inserimentiphoA Tn.
- Deselezionare la cappa a flusso laminare di attrezzature inutili e accendere ventilatore cappa per un minimo di 10 min prima di iniziare il lavoro. Pulire le superfici di cappuccio ed eventuali oggetti posizionati in cappa utilizzando etanolo al 70%.
- Riempire 2 mL profondo pozzo blocchi con 525 µ l di brodo liquido LB contenente antibiotici appropriati in cappa a flusso laminare usando una pipetta elettronica 50-1200 µ l 12 canali. Quindi, trasferire media-riempito pozzo profondo blocchi per etanolo-spazzato da banco. Riutilizzare, suggerimenti, purché condizioni sterili sono mantenute.
- Indossare guanti, camice e mascherina per gestire PA14NR impostare.
- Chiara panca e pulire la superficie con etanolo al 70%.
- Portare le piastre di agar con ceppi mutanti coltivati durante la notte alla parte superiore del banco.
- Creare un campo sterile con un becco Bunsen su etanolo-spazzato da banco e impostare la pagina di contenitori con soluzioni appropriate per la sterilizzazione di pin replicator (Vedi punto successivo).
- Sterilizzare i perni"replicatore" seguendo la procedura descritta di seguito. Toccare leggermente replicator dopo ogni immersione per eliminare il liquido in eccesso.
- Immergere i perni in 250 mL di 0.3-0.5% ipoclorito di sodio per 30 secondi. Minimizzare il contatto di pin di replicator con soluzione di ipoclorito di sodio, come può portare a danni di pin. Immergere quindi, perni in 250 mL ddH sterile2O o acqua ultrapura per 10 secondi, poi in 250 mL di etanolo al 70% per 30 secondi, quindi in 250 mL di etanolo al 95% per 2 min.
- Fiamma sterilizzare perni replicator, poi raffreddare perni da appuntare inutilizzato piastra rettangolare contenente LB agarizzati per 30 secondi.
Nota: Usare estrema cautela quando si lavora con etanolo vicino a una fiamma (Vedi 1.1.8).
- Delicatamente posto perni replicator sulla piastra di agar contenente crescita mutante e controllare che piedini siano in contatto con tutti i 96 mutanti sulla piastra di agar, quindi immergere perni in pozzi profondi contenenti brodo liquido LB. Evitare di toccare i lati dei pozzetti con i perni.
- Guarnizione del pozzo profondo blocco con una membrana sigillante traspirante sterile. Utilizzare un rullo piastra per garantire che ogni individuo ben correttamente è sigillato.
- Ripetere la procedura per ogni piastra di agar.
- Pulire tutte le superfici di lavoro con etanolo al 70% dopo aver maneggiato il Set di PA14NR.
- Coltivare colture inoculate liquide per 15-16 ore a 37° C a 950 giri/min, utilizzando un agitatore ad alta velocità, se disponibile.
Nota: Se un agitatore ad alta velocità non è disponibile, l'incubazione di pozzi profondo blocchi in agitatore a 250-300 giri/min è fattibile. Tuttavia, c'è una maggiore possibilità di piccola colonia varianti (SCV)25 emergenti in condizioni di bassa ossigenazione. Di conseguenza, è altamente consigliabile mantenere incubazione volte sotto i 15-16 h quando culture in pozzi profondo blocchi a velocità inferiori di shaker in crescita. Indesiderate proliferazione di SCV a wells mutante può alterare fenotipi mutanti quando si utilizza la libreria per eseguire schermi genetici.
Alcuni pozzi nel PA14NR Set contengono crescendo/non-crescita lenta mutanti, mancanti di cloni, o contengono contenuti multimediali non inoculato. La posizione di questi pozzi è stata registrata e può essere trovata nel file supplementari PA14NR impostare Wells informazioni incluso con questa pubblicazione.
- Giorno 3: Trasferimento di PA14NR Set notte culture in lastre di destinazione
- Deselezionare la cappa a flusso laminare e accendere ventilatore cappa per un minimo di 10 min prima di iniziare il lavoro. Pulire le superfici di cappuccio ed eventuali oggetti posizionati in cappa utilizzando etanolo al 70%.
- Stampare etichette adesive impermeabile (Vedi file supplementari PA14NR impostare etichette per modello). Rimuovere 96 pozzetti in plastica avvolgono all'interno di una cappa a flusso laminare. Piastra di Peel-off label e posizionarlo lungo il bordo della piastra più vicina alla A1 a H1 pozzetti della piastra, destinazione. Sollevare il coperchio della piastra destinazione leggermente e posizionare l'etichetta sul bordo inferiore per visualizzare l'etichetta quando la piastra è coperto con il coperchio.
- 3,5 L di glicerolo al 60% (v/v) di preparare e sterilizzare 20 min in autoclave.
- Indossare guanti, camice e mascherina per gestire il Set di PA14NR. Rimuovere i blocchi pozzi profondo da agitatore ad alta velocità o in uno shaker regolare.
- Trasferire blocchi di profondo-bene di cappa a flusso laminare sterile e rimuovere con cura la membrana sigillante traspirante. Sostituire con sigillo in alluminio. Selezione verso il basso i blocchi di profondo-bene a velocità molto bassa per raccogliere la condensa (30 secondi a 50-150 x g, poi decelerare quicky avendo centrifuga freno su).
- Trasferire blocchi di profondo-bene torna a cappuccio sterile e utilizzando una pipetta elettronica 50-1200 µ l 12 canali e suggerimenti filtrati sterile aggiungere 525 µ l di miscela di glicerolo/LB brodo liquido (parti uguali LB brodo e 60% glicerolo soluzione liquida) in ciascun pozzetto. Mescolare pipettando delicatamente 300 µ l su e giù 3 volte con pipetta elettronica. Toccare punte a lato del pozzo prima di espellere suggerimenti per evitare gocciolamenti. Espellere i puntali e continuare con la riga successiva fino a quando fatto con pozzo profondo tutto il blocco.
- Utilizzando la pipetta ripetitivo elettronica 12 canali e suggerimenti filtrate, per prevenire la contaminazione interwell durante aliquotare, tirare su 900 µ l di cultura mutante e dispensare 150 µ l in ogni piastre 96 pozzetti destinazione per generare 6 copie della piastra biblioteca. Evitare fuoriuscite toccando la parete dei pozzetti con suggerimenti prima di iniziare il trasferimento della cultura nelle piastre di destinazione. Se non ripetere pipetta è disponibile, è possibile utilizzare una pipetta multicanale per dispensare 150 µ l in ciascuna delle sei piastre 96 pozzetti, utilizzando la tecnica descritta sopra per evitare gocciolamenti.
- Utilizzare guarnizioni in alluminio sterile per coprire le piastre e utilizzare un rullo piastra completamente i bordi della piastra di tenuta e tutti i pozzetti. Assicurarsi di non coprire l'etichetta con il sigillo di alluminio. Non scuotere le piastre, cultura può spruzzare sui lati dei pozzetti o il sigillo in alluminio.
- Rimuovere piastre sigillati da piastre cappuccio e posto su un piatto, superficie uniforme nel congelatore-80 ° C.
- Pulire tutte le superfici di lavoro con etanolo al 70% dopo la gestione della libreria.
- Eseguire controlli di qualità dopo la replica di biblioteca e dopo usando la libreria per eseguire schermi genetici (Vedi protocollo III).
2. protocollo II: Movimentazione e stoccaggio di singoli mutanti dal Set PA14NR
-
Giorno 1: Streak mutante di interesse
- Identificare la posizione del mutante di interesse attraverso la PA14NR impostare link http://pa14.mgh.harvard.edu/cgi-bin/pa14/search.cgi?searchType=SEARCH_PLATE_POSITIONS o utilizzando il file Nonredundant Library.xls scaricato da link http:// PA14.MGH.Harvard.edu/cgi-bin/PA14/downloads.cgi). Prendere nota dell'antibiotico necessario per selezionare ogni mutante particolare (gentamicina o kanamicina).
- Preparare LB agarizzati, sterilizzare in autoclave per 20-25 minuti e raffreddare a 55 ° C in bagnomaria. Aggiungere 15 gentamicina µ g/mL per mutanti contenenti MAR2xT7 trasposone inserimenti e kanamicina di 200 µ g/mL per mutanti contenenti gli inserimenti TnphoA trasposoni. Versare LB agarizzati sulle piastre (piastre rotonde o rettangolari sono sufficienti). Secco piastre in cappa sterile per circa 30 min a 1 h prima di utilizzano.
- Autoclave tutti plasticware e forniture non sterili prima dell'uso.
- Indossare guanti, camice e mascherina per gestire il Set di PA14NR. Deselezionare la panchina e pulire la superficie con etanolo al 70% prima di lavorare con il Set di PA14NR.
- Creare un campo sterile con un becco Bunsen.
- Rimuovere PA14NR Set piastra a 96 pozzetti con mutante di interesse dal congelatore a-80 ° C e posizionarlo sul ghiaccio secco, prendere il contenitore di ghiaccio secco in panchina e brevemente posto piastra a 96 pozzetti in cima panca per consentire lievi scongelamento (circa 1-2 min).
Nota: Tenere un registro di tutte le piastre da 96 pozzetti PA14NR impostare letta mutanti singoli striscia, come più accesso alle piastre di biblioteca è correlata con un maggior rischio per contaminazione interwell. - Sigillo in alluminio caldo con mano prima staccava, facendo attenzione a impedire il sigillo di ritocco la piastra. Utilizzare una pinzetta per togliere eventuali residui della guarnizione in alluminio.
- Individuare il mutante di interesse sulla piastra 96 pozzetti. Utilizzare il bastone di legno sterile o punta sterile per prendere una piccola quantità di cultura congelato dall'individuo ben contenente il mutante di interesse.
- Striatura congelato cultura sulla piastra di agar per singoli mutante colonie come segue: delicatamente diffondere i batteri sopra una sezione della piastra per creare striscia 1, utilizzando una fresca, sterile in legno stick o pipetta punta, trascinare attraverso striscia 1 e sviluppa i batteri su una seconda sezione del la piastra, per creare striscia 2. Utilizzando un terzo legno bastone o pipetta punta sterile, trascinare attraverso vena 2 e diffondere i batteri sopra l'ultima sezione della piastra, per creare striscia 3.
- Sigillare la piastra di origine con una nuova guarnizione di alluminio sterile. Non toccare il lato adesivo della guarnizione di alluminio in qualsiasi punto per evitare la contaminazione. Assicurarsi che ciascun pozzetto e i bordi della piastra sono completamente sigillati utilizzando un rullo di piastra. Piastra 96 ritorno a ghiaccio secco e poi il congelatore a-80 ° C.
- Incubare la piastra di agar in incubatore a 37 ° C durante la notte.
- Pulire tutte le superfici di lavoro con etanolo al 70% dopo la gestione della libreria.
-
Giorno 2: Crescita del mutante di interesse in brodo liquido LB
- Preparare il brodo LB liquido contenente 15 µ g/mL Gentamicina o 200 kanamicina µ g/mL, secondo inserimento trasposoni.
- Indossare guanti, camice e mascherina per gestire p. aeruginosa.
- Chiara panca e pulire la superficie con etanolo al 70%. Creare un campo sterile con un becco Bunsen.
- Trasferire 3-5 mL di brodo LB con antibiotico adatto in coltura sterile provetta con tappo.
- Utilizzando un applicatore sterile o una pipetta sterile, prendere una singola colonia di ceppo mutante e inoculare in media LB.
- Incubare colture liquide LB a 37 ° C in un agitatore a 225-250 giri/min per una notte.
- Pulire tutte le superfici di lavoro con etanolo al 70% dopo la gestione di p. aeruginosa.
-
Giorno 3: Memorizzare mutante di interesse in congelatore a-80 ° C
- Cryovial etichetta con nome mutante, antibiotico aggiunto al brodo LB e la data di deposito.
- Preparare 500 mL di 50% (v/v) glicerolo e sterilizzare in autoclave.
- Indossare guanti, camice e mascherina per gestire p. aeruginosa.
- Deselezionare la panchina e/o cappuccio di flusso laminare e pulire la superficie con etanolo al 70%.
- Rimuovere la provetta contenente il mutante cultura da shaker.
- Preparare un piccolo contenitore con ghiaccio secco.
- Utilizzare una cappa a flusso laminare o creare un campo sterile su etanolo-spazzato da banco con bruciatore di Bunsen.
- Aggiunge la stessa quantità di coltura batterica e glicerolo al 50% per il cryovial con etichetta utilizzando condizioni di sterilità e mescolare delicatamente con la pipetta (volume finale 1-2 mL/fiala a seconda delle dimensioni del cryovials utilizzato). Cryovial posto su ghiaccio secco per Quick-Freeze.
- Cryovial posto nella casella etichettata in congelatore a-80 ° C.
- Pulire tutte le superfici di lavoro con etanolo al 70% dopo la manipolazione p. aeruginosa.
3. Protocollo III: controllo di qualità di PA14NR Set
- Selezionare un insieme casuale di mutanti da piastre appena replicati per rilevare eventuale contaminazione interwell (prova di 30-40 mutanti è consigliato).
Nota: In casi dove è necessario confermare l'identità di un mutante viene utilizzato per la caratterizzazione di un gene specifico, si consiglia di eseguire le amplificazioni di PCR utilizzando primers gene-specifico progettato per la nota sequenza del gene che contiene il inserimento di trasposoni. Anche se più impegnativo, i vantaggi dell'utilizzo arbitrario gli iniettori di PCR, piuttosto che gli iniettori di PCR del gene-specifico, quando amplyfing DNA frammenti dai mutanti trasposoni sono la facilità di conferma mutante su larga scala e la capacità di rilevare la presenza di potenziale contaminanti. Ai fini del controllo di qualità, non è necessario ottenere dati di sequenziamento PCR di alta qualità per tutti in modo casuale slected mutanti, fino a quando un numero sufficiente di mutanti vengono analizzato per valutare il tasso di errore. - Seguire "Protocollo II" per striscia e coltivare ceppi mutanti.
- Autoclave tutti plasticware e forniture non sterili prima dell'uso.
- Isolare il DNA genomico mediante metodo preferito. Per l'analisi descritta in questo lavoro, un kit di isolamento del DNA genomico è stato utilizzato seguendo protocolli del produttore. Ceppi mutanti multipli possono essere analizzati simultaneamente.
- Misurare la concentrazione di DNA genomica utilizzando uno spettrofotometro di microvolume. Regolare la concentrazione del DNA genomica a circa 100 ng / µ l.
- Utilizzare il DNA di genomic come modello per eseguire "PCR1 reazione" come descritto nel passo 1 della tabella 3, generando frammenti di PCR Arb1 (Figura 3). Gli iniettori per questo passaggio sono elencati nella tabella 4.
- Aggiungere 0,5 µ l di 10 x caricamento buffer da 5 µ l di reazione PCR1, caricare in un gel di agarosio 1.5-2% ed eseguire il gel alla 80-150 V.
Nota: Una lunghezza di frammento specifico o un intervallo particolare frammento non è previsto, e più di una banda può essere presente in cui il primer arbitrario può associare a più posizioni. - Utilizzare il DNA da reazione di PCR1 come modello per eseguire "PCR2 reazione" come descritto nel passaggio 2 della tabella 3, generando frammenti di PCR Arb2 (Figura 3). Gli iniettori per questo passaggio sono elencati nella tabella 4.
- Aggiungere 0,5 µ l di 10x buffer da 5 µ l di reazione di PCR2 di carico, caricare in un gel di agarosio 1.5-2% ed eseguire il gel alla 80-150 V.
Nota: Una lunghezza di frammento specifico o un intervallo particolare frammento non è previsto, e più di una banda può essere presente in cui il primer arbitrario può associare a più posizioni. - Invia PCR2 reazione con apposito trasposone specifici primer per il sequenziamento.
- Analize sequenziamento risultati tipo loro contro il genoma completo PA14 utilizzando il link BLAST forniti sul sito Web di PA14NR Library (http://pa14.mgh.harvard.edu/cgi-bin/pa14/blast.cgi) o da spedendoli direttamente contro la sequenza del gene specifico di il mutante di interesse.
Nota: Informazioni su mutanti selezionati è reperibile consultando il sito Web PA14NR impostato (ricerca http://pa14.mgh.harvard.edu/cgi-bin/pa14/search.cgi?searchType=SEARCH_PLATE_POSITIONS o Scarica il Library.xls Nonredundant il file da http:/ il link / pa14.mgh.harvard.edu/cgi-bin/pa14/downloads.cgi).
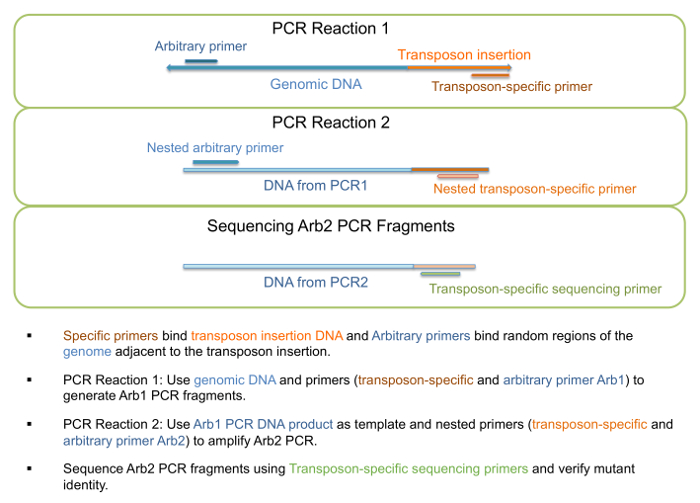
Figura 3: PCR amplificazione e sequenziamento di mutanti di inserzione del trasposone. Rappresentazione schematica dei passaggi coinvolti nella amplificazione di PCR e sequenziamento per la verifica dell'identità mutanti. Clicca qui per visualizzare una versione più grande di questa figura.
| Set-up reazione di PCR | |
| Passo 1 | Passo 2 |
| Reazione di PCR1 : | Reazione di PCR2 : |
| Acqua 23,25 µ l (grado di biologia molecolare) | 19.15 µ l acqua (grado di biologia molecolare) |
| Della polimerasi di Taq 3µL 10x Buffer | 5 µ l 10 x Buffer Taq polimerasi |
| 0.5µL Taq polimerasi | 0,6 µ l Taq polimerasi |
| 0,625 µ l 20 µM primer Arb1D (tabella 4) | 0,625 µ l 20 µM primer Arb2A (tabella 4) |
| Iniettore di 0,625 µ l 20 µM transposon-specific (PMFLGM. GB 3a o Tn5Ext) (tabella 4) | Iniettore di 0,625 mL 20 µM Transposon-specific (PMFLGM. GB 2a o Tn5Int2) (tabella 4) |
| 1 µ l 10 mM dNTPs | 1 µ l 10 mM dNTPs |
| 1 µ l DNA genomico, 100 ng | Reazione di PCR1 5 µ l |
| Volume di reazione finale di 30 µ l | Volume di reazione finale di 30 µ l |
| Impostazioni della reazione di PCR | |
| PCR1 Thermocycler condizioni: | PCR2 Thermocycler condizioni: |
| 95 ° C – 2 min | 95 ° C – 2 min |
| Ripetere 5 cicli: | Ripetere 30 cicli: |
| 95 ° C – 30 s | 95 ° C – 30 s |
| 30 ° C – 1 min | 54 ° C – 30 s |
| 72 ° C – 1 min | 72 ° C – 1,5 min |
| Ripetere 30 cicli: | 72 ° C – 10 min |
| 95 ° C – 30 s | 4 ° C – Hold |
| 45 ° C – 30 s | |
| 72 ° C – 1 min | |
| 72 ° C – 10 min | |
| 4° C – Hold |
Tabella 3: Condizioni set-up e thermocycler reazione di PCR usate per la PCR arbitrario. Reazioni di PCR arbitrarie vengono eseguite in sequenza, e frammenti generati durante la reazione di PCR1 sono utilizzati come modello nella reazione PCR2. Thermocycler specifiche impostazioni vengono utilizzate per ogni set di reazioni.
| Nome di primer | Sequenza più superelegante |
| MAR2xT7 trasposone specifici Primers | |
| PMFLGM. GB-3a | TACAGTTTACGAACCGAACAGGC |
| PMFLGM. GB-2a | TGTCAACTGGGTTCGTGCCTTCATCCG |
| Primer di sequenziamento Transposon MAR2xT7 | |
| PMFLGM. GB-4a | GACCGAGATAGGGTTGAGTG |
| TNphoA trasposone specifici Primers | |
| Tn5Ext | GAACGTTACCATGTTAGGAGGTC |
| Tn5Int2 | GGAGGTCACATGGAAGTCAGATCCTGG |
| TNphoA trasposone Primer di sequenziamento | |
| Tn5Int | CGGGAAAGGTTCCGTTCAGGACGC |
| Primer arbitrario | |
| ARB1D | GGCCAGGCCTGCAGATGATGNNNNNNNNNNGTAT |
| ARB2A | GGCCAGGCCTGCAGATGATG |
Tabella 4: elenco dei Primers utilizzati nel controllo di qualità Esperimenti. Primer usati per l'amplificazione di PCR e sequenziamento del trasposone inserimento mutanti per confermare identità mutanti.
Risultati
Dodici nuove copie del PA14NR Set sono state replicate mediante protocollo ho e una valutazione di controllo della qualità delle copie del nuove generato è stato condotto utilizzando il protocollo III.
PA14NR Set piastre mutante con placche di comando, che consistono di tipo selvaggio PA14 inoculato e non inoculato pozzi intercalati in un pattern predefinito (Figura 4A), sono stati replicati in se...
Discussione
Il P. aeruginosa PA14NR è una risorsa preziosa per la comunità scientifica. Secondo marzo 2017 dataset dal database degli Clarivate Analitica indicatori principali di scienza, Liberati et al. (2006) 37, che descrive la costruzione del Set PA14NR, è classificato nella top 1% delle pubblicazioni di microbiologia. Google Scholar segnala oltre 600 citazioni del Liberati et al. manoscritto originale (2006) a partire dal agosto 2017. La biblioteca ha svolto un ruol...
Divulgazioni
Gli autori non segnalano conflitti di interessi finanziari. Eliana Drenkard e Frederick Ausubel partecipato alla creazione della libreria di mutante nonredundant trasposone PA14. Bryan Hurley e Lael Yonker attualmente casa e distribuire la libreria mutante come parte del dipartimento di pediatria presso il Massachusetts General Hospital.
Riconoscimenti
Vorremmo ringraziare Lisa Philpotts della MGH Treadwell Virtual Library per la sua guida la ricerca nel database. Questo lavoro è stato supportato dalla Fondazione fibrosi cistica (YONKER16G0 e HURLEY16G0) e NIH NIAID (BPH e ADE: R01 A1095338).
Materiali
| Name | Company | Catalog Number | Comments |
| Materials for Library Replication | |||
| Sterile 96-well Tissue-culture treated, case of 50 | Corning Life Sciences | 353072 | via Fisher Scientific |
| Sterile 96 Well Clear V-Bottom 2000μL Deep Well Plates, case of 25 | Corning Life Sciences | 3960 | via Fisher Scientific |
| Nunc OmniTray (rectangular plates), case of 60 | Thermo Scientific Rochester | 242811 | via Fisher Scientific |
| Rectangular Ice Pan, Midi (4L) | Corning Life Sciences | 432104 | via Fisher Scientific |
| Secure-Gard Cone Mask, case of 300 | Cardinal Health | AT7509 | via Fisher Scientific |
| AluminaSeal, pack of 100 | Diversified Biotech | ALUM-100 | via Fisher Scientific |
| Breathe-Easy membrane, pack of 100 | Diversified Biotech | BEM-1 | via Sigma-Aldrich |
| Sterile, individually wrapped, 50mL Solution Trough/Reagent Reservoir, case of 100 | Sorenson | S50100 | via Westnet Incorporated |
| Plate roller | VWR | 60941-118 | via VWR |
| Cryo Laser Labels - CRYOLAZRTAG 2.64" x 0.277", pack of 16 sheets | GA International | RCL-11T1-WH | via Labtag.com (template for printing also available from Labtag.com) |
| 96-well replicator | V & P Scientific, Inc. | Custom 407C, 3.18mm pin diameter, 57mm long | via V & P Scientific, Inc. |
| Multitron Pro, 3mm Shaking incubator | Infors HT | l10003P | via Infors HT |
| Picus 12 Channel 50-1200μL Electronic Pipette | Sartorius | 735491PR | via Sartorius |
| Filter Tips 50-1200μL, pack of 960 | Biohit | 14-559-512 | via Fisher Scientific; use electronic multichannel-compatible tips |
| Dry Ice | User-specific vendor | ||
| Materials for Individual Mutant Storage | |||
| Fisherbrand Premium Microcentrifuge Tubes: 1.5mL | Fisher Scientific | 05-408-130 | via Fisher Scientific |
| Pipettes (P1000, P200, P20, P2) | Gilson | F167370 | via Gilson |
| Materials for Quality Control PCR | |||
| Fisherbrand Premium Microcentrifuge Tubes: 1.5mL | Fisher Scientific | 05-408-130 | via Fisher Scientific |
| NanoDrop | Thermo Scientific | ND-2000 | via ThermoFisher |
| PCR Thermocycler | |||
| Omnistrips PCR Tubes with domed lids | Thermo Scientific | AB0404 | via Fisher Scientific |
| ART Barrier low-retention pipette tips (10 uL, 100 uL, 1000 uL) | Molecular BioProducts, Inc. | Z676543 (10 uL), Z676713 (100 uL), Z676802 (1000 uL) | via Sigma-Aldrich |
| Pipettes (P1000, P200, P20, P2) | Gilson | F167370 | via Gilson |
| Fisherbrand Premium Microcentrifuge Tubes: 1.5mL | Fisher Scientific | 05-408-130 | via Fisher Scientific |
| MasterPure DNA Purification Kit | Epicentre | MCD85201 | via Epicentre Technologies Corp |
| GeneRuler 1 kb Plus DNA Ladder, ready-to-use | Thermo Scientific | SM1333 | via ThermoFisher |
| RediLoad Loading Buffer | Invitrogen | 750026 | via ThermoFisher |
| Chemicals | |||
| Chemicals for Library and Individual Mutant Storage | |||
| Glycerol MB Grade, 1L | Sigma Aldrich | G5516 | via Sigma-Aldrich |
| LB Broth | Per 1L dH2O: 10g tryptone, 5g yeast extract, 5g NaCl, 1ml 1N NaOH (Current Protocols in Molecular Biology. Wiley, 1994.) | ||
| Tryptone | Sigma Aldrich | T7293 | via Sigma-Aldrich |
| Yeast Extract | Sigma Aldrich | Y1625 | via Sigma-Aldrich |
| Sodium Chloride | Sigma Aldrich | S7653 | via Sigma-Aldrich |
| Sodium Hydroxide | Sigma Aldrich | S8045 | via Sigma-Aldrich |
| LB agar | See preparation above, add 15g Bacto Agar | ||
| Bacto Agar | Sigma Aldrich | A5306 | via Sigma-Aldrich |
| Gentamicin sulfate, 10g | BioReagent | 1405-41-0 | via Sigma-Aldrich |
| Kanamycin sulfate | Gibco | 11815024 | via ThermoFisher |
| Ethanol, 190 proof | Decon | 04-355-221 | via Fisher Scientific |
| Chemicals for Quality Control PCR | |||
| Primers | User-preferred vendor | See primers listed in Table 3 | |
| Corning cellgro Molecular Biology Grade Water | Corning | 46000CV | via Fisher Scientific |
| Taq Polymerase Buffer | Invitrogen | 10342020 | via ThermoFisher |
| Taq DNA Polymerase, recombinant | Invitrogen | 10342020 | via ThermoFisher |
| dNTPs | Invitrogen | 10297018 | via ThermoFisher |
| Agarose | Sigma | A9539 | via Sigma-Aldrich |
Riferimenti
- Moradali, M. F., Ghods, S., Rehm, B. H. Pseudomonas aeruginosa lifestyle: A paradigm for adaptation, survival, and persistence. Front Cell Infect Microbiol. 7, 39 (2017).
- Bleves, S., et al. Protein secretion systems in Pseudomonas aeruginosa: A wealth of pathogenic weapons. Int J Med Microbiol. 300 (8), 534-543 (2010).
- Breidenstein, E. B., de la Fuente-Nunez, C., Hancock, R. E. Pseudomonas aeruginosa: all roads lead to resistance. Trends Microbiol. 19 (8), 419-426 (2011).
- Flynn, K. M., et al. Evolution of ecological diversity in biofilms of Pseudomonas aeruginosa by altered cyclic diguanylate signaling. J Bacteriol. 198 (19), 2608-2618 (2016).
- Hazan, R., Maura, D., Que, Y. A., Rahme, L. G. Assessing Pseudomonas aeruginosa persister/antibiotic tolerant cells. Methods Mol Biol. 1149, 699-707 (2014).
- Klockgether, J., et al. Genome diversity of Pseudomonas aeruginosa PAO1 laboratory strains. J Bacteriol. 192 (4), 1113-1121 (2010).
- Mathee, K., et al. Dynamics of Pseudomonas aeruginosa genome evolution. Proc Natl Acad Sci U S A. 105 (8), 3100-3105 (2008).
- Taylor, P. K., Yeung, A. T., Hancock, R. E. Antibiotic resistance in Pseudomonas aeruginosa biofilms: towards the development of novel anti-biofilm therapies. J Biotechnol. 191, 121-130 (2014).
- Flume, P. A., Van Devanter, D. R. State of progress in treating cystic fibrosis respiratory disease. BMC Med. 10, 88 (2012).
- . Cystic Fibrosis Foundation Patient Registry 2015 Annual Data Report Available from: https://www.cff.org/Our-Research/CF-Patient-Registry/2015-Patient-Registry-Annual-Data-Report.pdf (2016)
- Church, D., Elsayed, S., Reid, O., Winston, B., Lindsay, R. Burn wound infections. Clin Microbiol Rev. 19 (2), 403-434 (2006).
- Sadikot, R. T., Blackwell, T. S., Christman, J., Prince, A. Pathogen-host interactions in Pseudomonas aeruginosa pneumonia. Am J Respir Crit Care Med. 171 (11), 1209-1223 (2005).
- Song, Z., et al. Prosthesis infections after orthopedic joint replacement: the possible role of bacterial biofilms. Orthop Rev (Pavia). 5 (2), 65-71 (2013).
- National Nosocomial Infections Surveillance, S. National Nosocomial Infections Surveillance (NNIS) System Report, data summary from January 1992 through June 2004, issued October 2004. Am J Infect Control. 32 (8), 470-485 (2004).
- Cohen, T. S., Parker, D., Prince, A. . Pseudomonas aeruginosa Host Immune Evasion. 7, 3-23 (2014).
- Fernandes, A., Dias, M. The microbiological profiles of infected prosthetic implants with an emphasis on the organisms which form biofilms. J Clin Diagn Res. 7 (2), 219-223 (2013).
- Khosravi, A. D., Ahmadi, F., Salmanzadeh, S., Dashtbozorg, A., Montazeri, E. A. Study of Bacteria Isolated from Orthopedic Implant Infections and their Antimicrobial Susceptibility Pattern. Res J of Microbiol. 4 (4), 6 (2009).
- Roemhild, R., Barbosa, C., Beardmore, R. E., Jansen, G., Schulenburg, H. Temporal variation in antibiotic environments slows down resistance evolution in pathogenic Pseudomonas aeruginosa. Evol Appl. 8 (10), 945-955 (2015).
- Fischer, S., et al. Intraclonal genome diversity of the major Pseudomonas aeruginosa clones C and PA14. Environ Microbiol Rep. 8 (2), 227-234 (2016).
- Wiehlmann, L., et al. Population structure of Pseudomonas aeruginosa. Proc Natl Acad Sci U S A. 104 (19), 8101-8106 (2007).
- Lam, J. S., Taylor, V. L., Islam, S. T., Hao, Y., Kocincova, D. Genetic and functional diversity of Pseudomonas aeruginosa lipopolysaccharide. Front Microbiol. 2, 118 (2011).
- Choi, J. Y., et al. Identification of virulence genes in a pathogenic strain of Pseudomonas aeruginosa by representational difference analysis. J Bacteriol. 184 (4), 952-961 (2002).
- He, J., et al. The broad host range pathogen Pseudomonas aeruginosa strain PA14 carries two pathogenicity islands harboring plant and animal virulence genes. Proc Natl Acad Sci U S A. 101 (8), 2530-2535 (2004).
- Mikkelsen, H., McMullan, R., Filloux, A. The Pseudomonas aeruginosa reference strain PA14 displays increased virulence due to a mutation in ladS. PLoS One. 6 (12), e29113 (2011).
- Drenkard, E., Ausubel, F. M. Pseudomonas biofilm formation and antibiotic resistance are linked to phenotypic variation. Nature. 416 (6882), 740-743 (2002).
- Rahme, L. G., et al. Common virulence factors for bacterial pathogenicity in plants and animals. Science. 268 (5219), 1899-1902 (1995).
- Rahme, L. G., et al. Use of model plant hosts to identify Pseudomonas aeruginosa virulence factors. Proc Natl Acad Sci U S A. 94 (24), 13245-13250 (1997).
- Kirienko, N. V., Cezairliyan, B. O., Ausubel, F. M., Powell, J. R. Pseudomonas aeruginosa PA14 pathogenesis in Caenorhabditis elegans. Methods Mol Biol. 1149, 653-669 (2014).
- Mahajan-Miklos, S., Tan, M. W., Rahme, L. G., Ausubel, F. M. Molecular mechanisms of bacterial virulence elucidated using a Pseudomonas aeruginosa-Caenorhabditis elegans pathogenesis model. Cell. 96 (1), 47-56 (1999).
- Limmer, S., et al. Pseudomonas aeruginosa RhlR is required to neutralize the cellular immune response in a Drosophila melanogaster oral infection model. Proc Natl Acad Sci U S A. 108 (42), 17378-17383 (2011).
- Miyata, S., Casey, M., Frank, D. W., Ausubel, F. M., Drenkard, E. Use of the Galleria mellonella caterpillar as a model host to study the role of the type III secretion system in Pseudomonas aeruginosa pathogenesis. Infect Immun. 71 (5), 2404-2413 (2003).
- Coleman, F. T., et al. Hypersusceptibility of cystic fibrosis mice to chronic Pseudomonas aeruginosa oropharyngeal colonization and lung infection. Proc Natl Acad Sci U S A. 100 (4), 1949-1954 (2003).
- Pazos, M. A., et al. Pseudomonas aeruginosa ExoU augments neutrophil transepithelial migration. PLoS Pathog. 13 (8), e1006548 (2017).
- Maura, D., Hazan, R., Kitao, T., Ballok, A. E., Rahme, L. G. Evidence for direct control of virulence and defense gene circuits by the Pseudomonas aeruginosa quorum sensing regulator, MvfR. Sci Rep. 6, 34083 (2016).
- Jacobs, M. A., et al. Comprehensive transposon mutant library of Pseudomonas aeruginosa. Proc Natl Acad Sci U S A. 100 (24), 14339-14344 (2003).
- Held, K., Ramage, E., Jacobs, M., Gallagher, L., Manoil, C. Sequence-verified two-allele transposon mutant library for Pseudomonas aeruginosa PAO1. J Bacteriol. 194 (23), 6387-6389 (2012).
- Liberati, N. T., et al. An ordered, nonredundant library of Pseudomonas aeruginosa strain PA14 transposon insertion mutants. Proc Natl Acad Sci U S A. 103 (8), 2833-2838 (2006).
- Feinbaum, R. L., et al. Genome-wide identification of Pseudomonas aeruginosa virulence-related genes using a Caenorhabditis elegans infection model. PLoS Pathog. 8 (7), e1002813 (2012).
- Stewart, L., et al. Draft genomes of 12 host-adapted and environmental isolates of Pseudomonas aeruginosa and their positions in the core genome phylogeny. Pathog Dis. 71 (1), 20-25 (2014).
- Thrane, S. W., et al. The widespread multidrug-resistant serotype O12 Pseudomonas aeruginosa clone emerged through concomitant horizontal transfer of serotype antigen and antibiotic resistance gene clusters. MBio. 6 (5), e01396-e01315 (2015).
- van Belkum, A., et al. Phylogenetic Distribution of CRISPR-Cas Systems in Antibiotic-Resistant Pseudomonas aeruginosa. MBio. 6 (6), e01796-e01715 (2015).
- Lewenza, S., et al. Construction of a mini-Tn5-luxCDABE mutant library in Pseudomonas aeruginosa PAO1: a tool for identifying differentially regulated genes. Genome Res. 15 (4), 583-589 (2005).
- . . Current Protocols in Molecular Biology. , (1994).
- Breidenstein, E. B., Khaira, B. K., Wiegand, I., Overhage, J., Hancock, R. E. Complex ciprofloxacin resistome revealed by screening a Pseudomonas aeruginosa mutant library for altered susceptibility. Antimicrob Agents Chemother. 52 (12), 4486-4491 (2008).
- Musken, M., Di Fiore, S., Dotsch, A., Fischer, R., Haussler, S. Genetic determinants of Pseudomonas aeruginosa biofilm establishment. Microbiology. 156 (Pt 2), 431-441 (2010).
- Schurek, K. N., et al. Novel genetic determinants of low-level aminoglycoside resistance in Pseudomonas aeruginosa. Antimicrob Agents Chemother. 52 (12), 4213-4219 (2008).
- Oumeraci, T., et al. Comprehensive MALDI-TOF biotyping of the non-redundant Harvard Pseudomonas aeruginosa PA14 transposon insertion mutant library. PLoS One. 10 (2), e0117144 (2015).
- Yeung, A. T., et al. Swarming of Pseudomonas aeruginosa is controlled by a broad spectrum of transcriptional regulators, including MetR. J Bacteriol. 191 (18), 5592-5602 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon