È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Titolazione ELISA come un metodo per determinare la costante di dissociazione dell'interazione ligando del recettore
In questo articolo
Riepilogo
Un protocollo dettagliato per eseguire una titolazione ELISA è descritto. Inoltre, un algoritmo di romanzo è presentato per valutare titolazione ELISAs e ottenere una costante di dissociazione di legame di un ligando solubile ad un recettore di immobilizzato piastra di microtitolo.
Abstract
La costante di dissociazione viene descritta l'interazione tra due partner nell'equilibrio associazione ed è una misura della loro affinità. È un parametro fondamentale per confrontare diversi ligandi, per esempio, inibitori competitivi, isoforme della proteina e mutanti, per la loro forza vincolante ad un partner di associazione. Costanti di dissociazione sono determinate tracciando le concentrazioni di associato contro gratis ligando come curve di associazione. Al contrario, le curve di titolazione, in cui un segnale che è proporzionale alla concentrazione del ligando associato viene tracciato contro la concentrazione totale di ligando aggiunto, sono molto più facili da registrare. Il segnale può essere rilevato spettroscopicamente e dall'analisi enzima-collegata dell'immunosorbente (ELISA). Questo è esemplificato in un protocollo per una titolazione test ELISA che misura l'associazione del rhodocetin di derivata da veleno di serpente al proprio dominio di destinazione immobilizzato dell'integrina α2β1. Titolazione ELISAs sono versatile e ampiamente utilizzati. Qualsiasi coppia di proteine interagenti utilizzabile come immobilizzato recettore e ligando solubile, purché entrambe le proteine sono pure, e le loro concentrazioni sono noti. La difficoltà è stata finora per determinare la costante di dissociazione da una curva di titolazione. In questo studio, è stato introdotto una funzione matematica sottostante curve di titolazione. Senza qualsiasi stima errori grafico di una resa di saturazione, questo algoritmo consente l'elaborazione dei dati grezzi (intensità del segnale a differenti concentrazioni di ligando aggiunto) direttamente dalla valutazione matematica tramite regressione non lineare. Così, diverse curve di titolazione possono essere registrate simultaneamente e trasformate in un set di parametri caratteristici, tra cui la costante di dissociazione e la concentrazione del recettore vincolante-attivo, e possono essere valutati statisticamente. Quando combinato con questo algoritmo, titolazione ELISAs guadagnare il vantaggio di presentare direttamente la costante di dissociazione. Pertanto, essi possono essere utilizzati in modo più efficiente in futuro.
Introduzione
La costante di dissociazione K è un parametro fondamentale per descrivere l'affinità di un recettore (R) per il suo ligando (L). Basato sulla legge di azione di massa, K è definito per l'equilibrio, in cui il RL complesso recettore-ligando si dissocia nel recettore R e il ligando l:
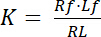 Equazione 1
Equazione 1
con gli indici f che indica lo stato di libero/non legata del recettore e ligando. La concentrazione del complesso recettore-ligando, RL, è identica alla concentrazione del ligando del recettore associato Lb. Come la concentrazione totale dei recettori Rt è la somma del recettore libero Rf e recettore-ligando Rb = Lb, la costante di dissociazione può essere scritta anche come:
 Equazione 2
Equazione 2
Quindi, la saturazione rendimento Y, definita come la frazione di ligando associato Lb in relazione alla concentrazione totale di recettore Rt,
 Equazione 3
Equazione 3
dipende dalla concentrazione del ligando libero Lf:
 Equazione 4
Equazione 4
Questa relazione iperbolica descrive la curva di associazione di un'interazione recettore-ligando e sua trama indica la concentrazione del ligando associato Lb in funzione della concentrazione del ligando libero Lf. Dalla curva di associazione, la costante di dissociazione K può essere derivata come la concentrazione del ligando libero allo snervamento mezzo massima saturazione. Inoltre, sono stati istituiti diversi algoritmi per linearizzare le curve di associazione, come la trama di doppio-reciproco di Klotz1,2, o trasformazioni secondo Scatchard o Hanes (Recensito da Bisswanger3). Tuttavia, tutti gli algoritmi soffrono il problema che il valore massimo del rendimento saturazione, che si avvicina asintoticamente ad alte concentrazioni di ligando libero della curva di associazione, deve essere stimato in una pre-valutazione grafica e pertanto è soggetta a errori.
Inoltre, la determinazione di una curva di associazione richiede la quantificazione del ligando libero e rilegato durante l'equilibrio di associazione. A tal fine, il ligando libero deve essere separato dal ligando-recettore associato e quantificato. Di conseguenza, il ligando e recettore necessario differiscono nelle loro proprietà, ad esempio un ligando non proteici in contrasto con un ricevitore della proteina. Se entrambi i partner di associazione sono proteine, devono essere distinguibili nelle loro dimensioni, spese o altre caratteristiche molecolari. Tuttavia, la quantificazione delle concentrazioni di ligando in approcci di associazione su piccola scala è un compito difficile. Etichettatura radioattivo del ligando spesso è stato necessario per rilevare la bassa concentrazione del ligando associato, soprattutto se notevoli quantità di recettori non erano disponibili o accessibili. Inoltre, il ligando del recettore associato può dissociare durante e dopo l'isolamento in maniera non trascurabile. Quindi, metodi complessi, come equilibrio gel filtrazione4, elettroforesi capillare5e impulso proteolisi6, sono tenuti a quantificare il ligando del recettore associato e separarlo dal ligando libero.
In contrasto con queste analisi obbligatorie, esperimenti di titolazione non richiedono la separazione quantitativa del ligando legato e libero. A tal fine, un recettore ad una concentrazione costante viene titolato con differenti concentrazioni di ligando aggiunto. Legandosi al recettore, il ligando associato dispone di una proprietà biofisica che lo distingue dal ligando libero ed è misurabile con, per esempio, fotometria, fluorimetria o rilevazione dell'anticorpo. Così, un segnale S, che è proporzionale alla saturazione resa Y e di conseguenza anche alla concentrazione del ligando del recettore associato (Lb), è rilevato come una funzione della concentrazione totale di aggiunto ligand (Lt). Entrambi i parametri, il segnale S e la concentrazione totale di ligando aggiunto sono quantificati in modo diretto e agevole rispetto le concentrazioni di ligando legato e libero. In particolare, la rilevazione del ligando del recettore associato di analisi enzima-collegata dell'immunosorbente (ELISA) ha permesso la riduzione dei volumi di campione di sotto 100 µ l e misurazioni parallele di diverse concentrazioni di ligando anche in multi-pozzetto di microtitolo. In una titolazione ELISA, un recettore è fisicamente adsorbito alla micropiastra alla stessa concentrazione e titolato con legante solubile. Il recettore è immobilizzato per la superficie di plastica essenzialmente mediante adsorbimento idrofobo. La concentrazione della superficie di recettore immobilizzato correla con la concentrazione di rivestimento del recettore in una relazione non lineare, probabile secondo Langmuir´s adsorbimento isoterma7. Oltre al numero totale di molecole adsorbite recettore, il loro stato di attività è un altro parametro importante per le analisi di titolazione. Solo immobilizzato i ricevitori che hanno mantenuto l'attività di legame del ligando, sono rilevanti per il dosaggio di titolazione e infine contribuiscono alla concentrazione totale di recettori attivi Rt del test di titolazione, che non può essere determinato direttamente.
Siti sulla superficie di plastica, che non sono coperti dal recettore immobilizzato sono inclini ad assorbire altre proteine, come il ligando. Adsorbimento fisico del ligando a tali siti di superficie in plastica si tradurrebbe in un segnale simile come il ligando del recettore associato, ma in un modo non specifico. Per ridurre questo segnale aspecifico, i siti di superficie plastica delle piastre micrometodi che non sono state rivestite con proteina ancora verranno bloccate con albumina di siero bovino (BSA). Tuttavia, per alcuni test di titolazione del recettore-ligando, segnali aspecifici sfondo possono essere osservati. Poi, altri agenti di blocco, ad esempio una soluzione di 0,2% di gelatina o di 0,04% Tween 20, sono raccomandati.
Dopo il legame al recettore, il ligando libero viene rimosso mediante due passaggi del lavaggio. Ligando associato rimane con il recettore, che è immobilizzato per la superficie di plastica del pozzo microtitolo e facoltativamente rinforzato tramite la fissazione chimica. Per i successivi covalenti legame incrociato di associato ligando e recettore immobilizzato con glutaraldeide, la sostanza tampone TRIS è sostituita per HEPES, senza alcun cambiamento nel grippaggio del ligand. HEPES, in contrasto con TRIS, non inattivare glutaraldeide. Il legame covalente incrociato con glutaraldeide corregge il ligando associato con il suo recettore e impedisce la sua dissociazione durante i passaggi successivi di lavaggio e di incubazione. Così, l'interazione recettore-ligando chimicamente è congelato e garantisce una curva di titolazione che risente delle successive fasi di lavaggio e di incubazione. Tuttavia, la fissazione di glutaraldeide può modificare chimicamente il ligando e recettore in modo tale che la loro interazione è ridotto o abolito. Inoltre, modifica degli epitopi entro il ligando può modificare l'affinità di legame dell'anticorpo di rilevazione, soprattutto se un anticorpo monoclonale è utilizzato per quantificare il ligando associato. Anche se nessuno di questi effetti avversi della fissazione in glutaraldeide si verifica in questa titolazione ELISA, la sensibilità del test verso glutaraldeide dovrà essere testata per ogni interazione recettore-ligando prima dell'esperimento di titolazione. Dopo la fissazione, glutaraldeide in eccesso viene rimosso in tre lavaggi con tampone TRIS-contenente. TRIS inattiva restanti gruppi di aldeide, che non specifico potrebbero reagire con il rilevamento di anticorpi nel passaggio successivo.
La quantità di legante associato viene quantificata con gli anticorpi enzima-collegata, che forniscono un segnale fotometrico di ELISA S. Ciò è tracciata contro il ligando totale concentrazione Lt aggiunti a ciascun pozzetto. Nonostante la sua più facile acquisizione, la curva di titolazione non è una funzione iperbolica in contrasto con la curva di associazione. Inoltre, esso è stato poco chiaro come calcolare la costante di dissociazione K da una curva di titolazione. Anche se gli algoritmi per linearizzare le curve di titolazione spettroscopicamente acquisite sono stati riportati indipendentemente da Stockell8 e Heyn e Weischet9, sono caduto breve a causa della loro incertezza di stimare il segnale massimo valore che il resa di saturazione si avvicina alle alte concentrazioni di ligando aggiunto.
Qui, una titolazione ELISA e un algoritmo di regressione non lineare sono descritti per ricavare la costante di dissociazione K per un'interazione ligando del recettore da una curva di titolazione. Questo protocollo è esemplificato per l'interazione tra il dominio di A collagene-associazione dell'integrina α2β1 con un inibitore di derivati da veleno di serpente. Le integrine sono molecole di adesione cellulare, che mediano l'ancoraggio delle cellule alla matrice extracellulare circostante o il sottostante membrana basale10,11. Inoltre, le integrine trasmettono segnali importanti tra cellule e matrice extracellulare con l'assunzione di ulteriori molecole di segnalazione e formando nuovi organelli delle cellule, adhesomes, su cellula-matrice interazione12,13, 14. il collagene, il ligando di integrina α2β1, è la proteina più abbondante del corpo umano ed è un componente fondamentale dell'armatura del tessuto connettivo15. L'interazione tra integrina α2β1 e collagene è mediata dal dominio della subunità α2 integrine. L'integrina α2A-dominio contiene un catione bivalente, che è necessaria per l'associazione di collagene e stabilizza la sua struttura. Il selvaggio-tipo modulo nonché mutanti del dominio α2A, ad esempio quella in cui il residuo di superficie-esposta Y216 era stato sostituito per una glicina, possono essere facilmente prodotta recombinantly in un sistema di espressione batterica e isolati tramite loro oligo-His-tag con un NiNTA colonna di SuperFlow con una successiva dialisi contro di 2 mM MgCl2contenente soluzione fisiologica tamponata (TBS; 50 mM TRIS/HCl, pH 7.4, 150 mM NaCl)16. Le loro concentrazioni sono state determinate con l'analisi di acido bicinconinico (BCA) e loro purezze sono testate da convenzionale SDS-PAGE e macchiate con Coomassie Brilliant Blue R250.
L'interazione tra integrina α2β1 e collagene è bloccato dall'associazione del componente di veleno di serpente, rhodocetin, dalla vipera di pozzo malese (Calloselasma rhodostoma)16,17. Usato come un legante solubile in questa titolazione ELISA, rhodocetin è stato purificato dal greggio veleno come descritto in precedenza16. Esso è disciolto in soluzione salina tamponata HEPES (HBS; 10 mM HEPES/NaOH, pH 7.4, 150 mM NaCl) ed è conservato congelati a-20 ° C. La sua concentrazione è stata determinata da BCA e la sua purezza è stata dimostrata da SDS-PAGE. Come antagonista, rhodocetin non solo blocca collagene vincolanti per l'integrina α2β1 dominio, ma inoltre stabilizza la conformazione inattiva dell'integrina impedendo qualsiasi segnalazione da collagene nelle cellule o piastrine18. È di grande importanza biomedica per determinare la costante di dissociazione di rhodocetin con il suo obiettivo di recettore e svelare così il meccanismo molecolare e farmaceutici potenziali ad es., come agente antitrombotico. A tal fine, una titolazione ELISA è descritto compreso la sua valutazione, che è applicabile a quasi qualsiasi interazione recettore-ligando con una stechiometria 1:1 interazione.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. magazzino soluzioni
- Per preparare 100 mL di 10 x TBS soluzione a pH 7,4, sciogliere 6,06 g di TRIS e 8,77 g di NaCl in 90 mL di acqua deionizzata, regolare il pH a 7.4 con soluzione di HCl 37%, riempire il volume a 100 mL con acqua deionizzata e filtrare la soluzione.
- Per preparare 100 mL di 1 M HEPES/NaOH, soluzione a pH 7,4, sciogliere g 23,83 di HEPES in 90 mL di acqua deionizzata, regolare il pH a 7.4 con 1,5 M NaOH, riempire il volume a 100 mL con acqua deionizzata e filtrare la soluzione.
- Per preparare 100 mL di soluzione NaCl 5m, sciogliere 29,2 g di NaCl in 90 mL di acqua deionizzata. Riempire il volume a 100 mL con acqua deionizzata e filtrare la soluzione.
- Per preparare 100 mL di soluzione 1 M MgCl2 , sciogliere 20,33 g di MgCl2 · 6H2O in 90 mL di acqua deionizzata. Riempire il volume a 100 mL con acqua deionizzata e filtrare la soluzione.
- Per preparare 5% BSA inattivati in acqua, pesano 2,5 g di BSA (frazione V, pH 7.0) in una provetta da 50 mL e dissolverlo in 45 mL di acqua deionizzata. Riempire la soluzione a 50 mL con acqua deionizzata e riscaldare la soluzione in un bagno di acqua a 68 ° C per 45 min. raffreddare in un bagno di ghiaccio e conservare a-20 ° C.
- Preparare la soluzione acquosa di glutaraldeide al 25%.
Attenzione: La glutaraldeide è nocivo se ingerito, tossico per inalazione e provoca ustioni. Indossare indumenti protettivi, guanti e protezione per occhi/volto. - Sollevare l'antisiero di coniglio come descritto in precedenza19. Il titolo dell'antisiero è stato determinato secondo protocolli standard20.
- Preparare gli anticorpi dell'immunoglobulina di anti-coniglio da capra coniugato con fosfatasi alcalina.
- Per preparare 100 mL di soluzione 0,1 M di glicina, sciogliere 0,75 g di glicina in 90 mL di acqua deionizzata. Regolare il pH a 10.4 con 1,5 M NaOH soluzione, riempire il volume a 100 mL con acqua deionizzata e filtrare la soluzione.
- Per preparare 100 mL di 0.5 M Zn (II)-soluzione di acetato, sciogliere 10,98 g di Zn (II)-acetato · 2H2O in 90 mL di acqua deionizzata. Riempire il volume a 100 mL con acqua deionizzata e filtrare la soluzione.
- Per preparare 100 mL di soluzione di NaOH di 1,5 M, sciogliere 6,0 g di NaOH in 90 mL di acqua deionizzata. Riempire il volume a 100 mL con acqua deionizzata e filtrare la soluzione.
2. preparare il buffer e soluzioni di lavoro
- Diluire 5 mL di 10 x TBS pH 7.4, con 45 mL di acqua deionizzata e aggiungere 100 µ l di una soluzione 1 M MgCl2 . Tenerlo a temperatura ambiente. La TBS, pH 7.4, 2 mM MgCl2 soluzione è per l'immobilizzazione di recettore e lavaggio micropiastre.
- Diluire 10 mL di 5% di BSA inattivati in acqua e 5 mL di 10 x TBS, pH 7.4 con acqua deionizzata a 50 mL. Aggiungere 100 µ l di soluzione stock di 1 M MgCl2 e mescolare bene la soluzione. Tenere sul ghiaccio e memorizzarlo per ulteriori esperimenti a-20 ° C. Nota che 2,5 mL di 1% BSA in TBS, pH 7.4, 2 mM MgCl2 sono necessari per ogni curva di titolazione.
- Diluire 2,5 mL di 1 M HEPES/NaOH, pH 7,4 e 1.5 mL 5m NaCl soluzione a 50 mL con acqua deionizzata, aggiungere 100 µ l 1 M MgCl2 soluzione e mescolare accuratamente per preparare 50 mL di HBS, pH 7.4: 50 mM HEPES/NaOH , 150 mM NaCl.
- Aggiungere 5 µ l di soluzione madre di 1M MgCl2 e 2 µ l 0,5 M Zn (II)-soluzione di riserva di acetato di 5 mL soluzione di glicina 0,1 M, pH 10.4 per preparare il tampone di fosfatasi alcalina (AP) (soluzione di glicina 0,1 M, pH 10.4, 1 mM MgCl2, 0,2 mM Zn(II)-acetate).
3. immobilizzazione del recettore (integrina α2A-dominio) per micropiastra
- Diluire la soluzione madre di integrina α2A-dominio in TBS, pH 7.4, 2 mM MgCl2 per una concentrazione finale di 5 µ g/mL.
Nota: Il volume della soluzione di rivestimento è 650 µ l per una curva di titolazione, composto da ben 12-fila di mezza zona-micropiastra. - Riempire ogni pozzetto di una riga su una mezza zona-micropiastra con 50 µ l/pozzetto della soluzione di rivestimento 5 µ g/mL di integrina α2A-dominio (wild-type o mutanti). Eseguire almeno ogni riga di titolazione in duplicati (in questo esempio come quartine; Vedi layout della piastra per microtitolazione in Figura 1).
- Sigillare la piastra con un foglio o chiudere con un coperchio. Lasciare la piastra a 4 ° C durante la notte.
4. lavaggio pozzetti rivestiti della piastra due volte con TBS, pH 7.4, 2 mM MgCl2
- Per rimuovere il recettore solubile molecole, che non sono state immobilizzate per la superficie di plastica di adsorbimento fisico, rimuovere la soluzione di rivestimento e riempiono ogni bene con 50 µ l di TBS, pH 7.4, 2 mM MgCl2.
- Rimuovere la soluzione di lavaggio. Assicurarsi che i pozzi non diventi secco. Di conseguenza, non toccare la piastra per microtitolazione su un panno di tessuto per rimuovere il liquido residuo. Utilizzare una pipetta multistep o una pipetta multicanale per riempire i pozzetti rapidamente.
- Ripetere questo passaggio di lavaggio una volta.
5. bloccare i siti di legame non specifico
- Aggiungere 50 µ l di soluzione di BSA 1% in TBS, pH 7.4, 2 mM MgCl2 in ciascun pozzetto.
- Sigillare i pozzetti con stagnola o chiudere con un coperchio.
- Incubare per 1 h a temperatura ambiente.
Nota: I passaggi di incubazione del presente protocollo può essere eseguiti su una piattaforma a dondolo o agitazione. Tuttavia, questo non è necessario e non altera l'esito dell'esperimento.
6. preparazione di una riga di diluizione seriale del ligando, Rhodocetin
- Variare la concentrazione iniziale di rhodocetin e il fattore di diluizione della diluizione seriale, per ottenere un'appropriata gamma di concentrazioni di ligando e registrare una curva di titolazione completa con un segnale minimo e massimo. In questo esperimento, impiegano una concentrazione iniziale di 243 nM rhodocetin e un fattore di diluizione di 2.3. Diluire la soluzione di riserva di rhodocetin per la più alta concentrazione di ligando della riga diluizione seriale. Per ogni curva di titolazione con un fattore di diluizione di 2.3, preparare 115 µ l di soluzione di rhodocetin 243 nM (cioè, 15,2 µ g/mL) in 1% BSA/TBS, pH 7.4, 2 mM MgCl2 in provetta #1.
- Riempire 65 µ l di soluzione di BSA 1% in TBS, pH 7.4, 2 mM MgCl2 in 10 provette, etichettato #2 e #11.
- Trasferire 50 µ l della diluizione rhodocetin da provetta #1 alla provetta 2 #, mescolare entrambe le soluzioni (volume totale: 115 µ l; fattore di diluizione: 1:2. 3) di triturazione e quindi trasferire 50 µ l da questa miscela alla provetta #3, ecc.
- Continuare questa diluizione seriale fino a provetta #11.
Nota: I volumi dati nei passaggi 6.1-6.4 sono sufficienti per una curva di titolazione. Moltiplicare il numero delle ripetizioni di questi volumi. In questo caso, eseguire otto curve di titolazione (quartine di due forme di α2A-dominio) e preparare i seguenti volumi: 920 µ l di soluzione di rhodocetin al più alta concentrazione in provetta #1; 520 µ l di BSA 1% in TBS, pH 7.4, 2 mM MgCl2 per riempire in ciascuna delle provette #2 e #11; e 400 µ l di volume di trasferimento da un tubo di quello successivo.
7. associazione del ligando (Rhodocetin) a differenti concentrazioni di immobilizzato recettore (integrina α2A-dominio)
- Rimuovere la soluzione bloccante da pozzetti della piastra da una linea di vuoto.
- Immediatamente aggiungere 50 µ l della soluzione rhodocetin in 1% BSA/TBS, pH 7,4/MgCl2 soluzione di provetta #1 nei pozzetti della colonna 1, soluzione della provetta #2 pozzetti di colonna 2,ecc. aggiungere 50 µ l di BSA 1% in TBS, pH 7.4 , 2 mM MgCl2 (tampone di diluizione e di blocco) come un controllo privo di ligando pozzetti di colonna 12 (Vedi layout della piastra per microtitolazione, Figura 1).
- Sigillare i pozzetti con stagnola o chiudere con un coperchio.
- Incubare i pozzetti per 1,5 h a temperatura ambiente (circa 20-22 ° C).
8. lavaggio pozzetti della piastra due volte con HBS, pH 7.4, 2 mM MgCl2
- Per rimuovere molecole leganti non associati, rimuovere la soluzione di rilegatura e riempire ogni bene con 50 µ l di HBS, pH 7.4, 2 mM MgCl2. Quindi, rimuovere la soluzione di lavaggio.
- Fare attenzione che i pozzi non diventino asciutti. Di conseguenza, non toccare la piastra per microtitolazione su un panno di tessuto per rimuovere il liquido residuo. Utilizzare una pipetta multistep o una pipetta multicanale per riempire i pozzetti rapidamente.
- Ripetere questo passaggio di lavaggio una volta.
9. Fissare il ligando del recettore associato con 2,5% glutaraldeide in HBS, pH 7.4, 2 mM MgCl2
- Preparare una soluzione di glutaraldeide 2,5% fresca con 1 parte di soluzione di glutaraldeide al 25% e 9 parti di HBS, pH 7.4, 2 mM MgCl2.
- Riempire ogni pozzetto della piastra microtiter con 50 µ l della soluzione di glutaraldeide al 2,5% in HBS, pH 7.4, 2 mM MgCl2. Incubare la piastra per microtitolazione per 10 min a temperatura ambiente.
10. lavare pozzetti della piastra tre volte con 50 µ l/pozzetto di TBS, pH 7.4, 2 mM MgCl2
- Per rimuovere e inattivare glutaraldeide in eccesso, rimuovere la soluzione di fissazione e riempire ogni bene con 50 µ l di TBS, pH 7.4, 2 mM MgCl2. Quindi rimuovere la soluzione di lavaggio.
Nota: Versare la soluzione di fissazione contenenti glutaraldeide dalla micropiastra in un piatto e scartare la soluzione di fissazione dopo che è stato inattivato da un uguale volume di TBS, pH 7,4. - Fare attenzione che i pozzi non diventino asciutti. Di conseguenza, non toccare la piastra per microtitolazione su un panno di tessuto per rimuovere il liquido residuo. Utilizzare una pipetta multistep o una pipetta multicanale per riempire i pozzetti rapidamente.
- Ripetere questo passaggio di lavaggio due volte.
11. quantificazione del ligando del recettore associato da ELISA
- Aggiungere 50 µ l/pozzetto di soluzione di anticorpo primario in 1% BSA in TBS, pH 7.4, 2 mM MgCl2. La soluzione di anticorpo primario è un antisiero di coniglio contro rhodocetin19, diluito 1:2,000 in 1% BSA in TBS, pH 7.4, 2 mM MgCl2.
- Incubare la piastra per 75-90 min a temperatura ambiente. Lavare tutti i pozzetti della piastra tre volte con 50 µ l/pozzetto di TBS, pH 7.4, 2 mM MgCl2. Durante le tre fasi di lavaggio, maschiatura della micropiastra su un tessuto panno non è necessaria.
- Aggiungere 50 µ l/pozzetto di soluzione di anticorpo secondario in 1% BSA in TBS, pH 7.4, 2 mM MgCl2. A tal fine, diluire l'anticorpo secondario, anticorpi di capra dell'immunoglobulina-targeting di coniglio coniugati con fosfatasi alcalina, per 1:2,000 in 1% BSA in TBS, pH 7.4, 2 mM MgCl2. Incubare la piastra per 75-90 min a temperatura ambiente.
- Preparare la soluzione di rilevamento AP sciogliendo una compressa da 5 mg contenenti 4-nitrofenil fosfato disodico sale esaidrato (substrato fosfatasi) in 5 mL di tampone di AP (soluzione di glicina 0,1 M, pH 10.4, contenente 1 MgCl2 e 0,2 mM Zn(II)-acetate).
- Lavare tutti i pozzetti della piastra tre volte con 50 µ l/pozzetto di TBS, pH 7.4, 2 mM MgCl2, immediatamente prima di eseguire il passaggio successivo. Toccare la piastra per microtitolazione su un panno di tessuto dopo l'ultimo passaggio di lavaggio per rimuovere tutte le tracce di liquido.
- Aggiungere 50 µ l/pozzetto di soluzione AP-rilevazione dei pozzetti di micropiastre. Aggiungere soluzione AP-rilevazione tempestivamente a tutti i pozzetti per avviare la conversione enzimatica simultaneamente come possibile. Pertanto, utilizzare una pipetta multicanale.
- Incubare la piastra a temperatura ambiente fino a quando la soluzione nei pozzetti con la più alta concentrazione di ligando ingialliscono.
Nota: Il tempo di incubazione può variare tra 5 min e 1 h a seconda dell'intensità di segnale. - Interruzione della conversione del substrato fosfatasi aggiungendo 50 µ l/pozzetto di soluzione di NaOH di 1,5 M. Lasciare la piastra per alcuni minuti affinché le striature di miscelazione di entrambe le soluzioni. Nell'ottica di garantire lo stesso periodo di incubazione in tutti i pozzetti, utilizzare una pipetta multicanale e aggiungere il 1,5 M NaOH nello stesso ordine in aggiunta ai pozzetti come quello del substrato AP-rilevazione nel passaggio 11,8.
- Misurare la densità ottica (OD) a 405 nm di ciascun pozzetto da un lettore ELISA.
12. valutazione dei segnali titolazione
- Aprire la tabella di dati grezzi, i valori di OD405nm , con Excel. Come questi valori di segnale delle curve di titolazione sono leggere nelle righe, recepire i valori di una colonna e l'etichetta con le concentrazioni di ligando aggiunto in un'altra colonna.
- Aprire il Graphpad Prism 5 (versione 5.0). Aprire un nuovo file di progetto nel menu principale. Scegliere il formato XY sotto il grafico & nuovi dati. Scegliere l'opzione invio e trama di un punto unico per ogni valore dell'asse y.
- Copiare le due colonne, concentrazione del ligando aggiunto e segnalare valori (OD405nm ) da excel il file e incollarli nella scheda di dati di GraphPad Prism come valori X e Y, rispettivamente.
- Aprire il sub-programma di analisi di GraphPad Prism 5 e scegliere l'opzione di regressione Non lineare sotto XY-analisi. Scegli equazione definiti dall'utente e premere il pulsante nuovo per creare una nuova equazione.
- Digitare l'equazione curva di titolazione in forma: Y =(Smax-Smin)*((X+R+K)-sqrt((X+R+K)^2-4*R*X)) /(2*R) + Smin + B * X nel foglio del modello aperto di recente, con Y essendo il valore del segnale S, X è la concentrazione del ligando aggiunto L , R è la concentrazione di immobilizzato recettore, K è la costante di dissociazione, e B è lo sfondo del pendio. Definire i vincoli appropriati, ad esempio K > 0 e R > 0.
Nota: Questa equazione è la stessa equazione della equazione 9 in una forma diversa. - Analizzare i valori del foglio dati scegliendo l'equazione definito dall'utente, che è stata appena creata. Aprire la tabella con i valori di approssimazione calcolata (K, R,t, Smax, Smine B) che sono mostrati sotto la sezione dei risultati sul lato sinistro della schermata del software.
Nota: Il software determina i 5 parametri di montaggio iterativo della regressione non lineare solo se la curva di titolazione è costituito da dati di almeno 5 punti. - Valutare i parametri K, R,t, Smax, Smine B per ogni gruppo di curve di titolazione statisticamente e correlare i parametri con la caratteristica specifica del gruppo (ligando mutato o modificati chimicamente o del ricevitore).
Access restricted. Please log in or start a trial to view this content.
Risultati
Dopo il test ELISA è stato sviluppato, il colore giallo del substrato fosfatasi alcalina convertito, para- nitrofenolato di sodio, indica che la quantità di legante associato rhodocetin diminuisce con la diminuzione delle concentrazioni di rhodocetin aggiunto da colonne da 1 a 11 (Figura 1). L'incolore wells nei pozzetti privo di rhodocetin nella colonna 12 Visualizza un segnale di basso fondo.
Access restricted. Please log in or start a trial to view this content.
Discussione
La titolazione ELISA è un sistema versatile per determinare la dissociazione di un'interazione recettore-ligando. Come la titolazione ELISA elude la necessità di separare i leganti liberi e rilegati in modo efficace e per analizzare quantitativamente, le loro concentrazioni sostanzialmente più studi e pubblicazioni hanno impiegato titolazione ELISAs invece di registrare curve di associazione . Inoltre, titolazione ELISAs sono facili da eseguire e richiedono ragionevolmente basse quantità di recettore e ligando. Per u...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
L'autore non ha nulla di divulgare.
Riconoscimenti
Il protocollo e l'algoritmo sono stati sviluppati all'interno di un progetto finanziato dalla Deutsche Forschungsgemeinschaft (DFG grant SFB1009 A09 ed EB177/13-1). L'autore ringrazia Barbara Schedding e Felix Schmalbein per il supporto tecnico e Dr. Niland per leggere criticamente il manoscritto.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| TRIS | neoFrox | 1125KG001 | |
| HEPES | Sigma-Aldrich | H4034 | |
| NaCl | Applichem | 1,316,591,214 | |
| MgCl2 | Merck | 172571 | |
| integrin a2A, wild-type and mutant, recombinant | isolated in author's lab | ||
| NiNTA superflow column | Qiagen, Germany | 30821 | |
| Coomassie-Brilliant Blue R250 | Serva | 35050 | |
| bicinchoninic acid assay (BCA), protein concentration determination kit | Fisher Scientific | 23225 | |
| bovine serum albumine (BSA), fraction V | Applichem | A1391 | |
| 25 % solution of glutaraldehyde | Merck | 354400 | |
| anti-rabbit immunglobulin-antibodies from goat, conjugated with alkaline phosphatase | Sigma-Aldrich | A9919 | |
| Glycine | Applichem | A1377 | |
| Zn(II)-acetate | Applichem | A4324 | |
| NaOH | Applichem | A1551 | |
| Alkaline phosphatase substrate tablet (5 mg) | Sigma-Aldrich | S0942 | |
| Costar half-area microtiter plate | Thermo Scientific | Corning 3690 | |
| micro reaction tubes | Eppendorf | 30120086 | |
| Microplate ELISA reader | BioTek | Synergy HT |
Riferimenti
- Klotz, I. M. The application of the law of mass action to binding by proteins; interactions with calcium. Arch Biochem. 9, 109-117 (1946).
- Klotz, I. M. Ligand-receptor complexes: origin and development of the concept. J Biol Chem. 279 (1), 1-12 (2004).
- Bisswanger, H. Ch. 1: Multiple Equilibria, Principles and Derivations. Enzyme Kinetics: Principles and Methods. , Wiley VCH Verlag GmbH. 1-26 (2017).
- Shimura, K., Kasai, K. Affinity gel titration: quantitative analysis of the binding equilibrium between immobilized protein and free ligand by a continuous titration procedure. Anal Biochem. 149 (2), 369-378 (1985).
- Gong, M., Nikcevic, I., Wehmeyer, K. R., Limbach, P. A., Heineman, W. R. Protein-aptamer binding studies using microchip affinity capillary electrophoresis. Electrophoresis. 29 (7), 1415-1422 (2008).
- Hanes, M. S., Ratcliff, K., Marqusee, S., Handel, T. M. Protein-protein binding affinities by pulse proteolysis: application to TEM-1/BLIP protein complexes. Protein Sci. 19 (10), 1996-2000 (2010).
- Latour, R. A. The Langmuir isotherm: a commonly applied but misleading approach for the analysis of protein adsorption behavior. J Biomed Mater Res A. 103 (3), 949-958 (2015).
- Stockell, A. The binding of diphosphopyridine nucleotide by yeast glyceraldehyde-3-phosphate dehydrogenase. J Biol Chem. 234 (5), 1286-1292 (1959).
- Heyn, M. P., Weischet, W. O. Circular dichroism and fluorescence studies on the binding of ligands to the α subunit of tryptophan synthase. Biochem. 14 (13), 2962-2968 (1975).
- Campbell, I. D., Humphries, M. J. Integrin structure, activation, and interactions. Cold Spring Harb Perspect Biol. 3 (3), (2011).
- Zeltz, C., Gullberg, D. The integrin-collagen connection--a glue for tissue repair? J Cell Sci. 129 (4), 653-664 (2016).
- Kanchanawong, P., et al. Nanoscale architecture of integrin-based cell adhesions. Nature. 468 (7323), 580-584 (2010).
- Luo, B. H., Carman, C. V., Springer, T. A. Structural basis of integrin regulation and signaling. Annu Rev Immunol. 25, 619-647 (2007).
- Luo, B. H., Springer, T. A. Integrin structures and conformational signaling. Curr Opin Cell Biol. 18 (5), 579-586 (2006).
- Ricard-Blum, S. The collagen family. Cold Spring Harb Perspect Biol. 3 (1), a004978(2011).
- Eble, J. A., Tuckwell, D. S. The α2β1 integrin inhibitor rhodocetin binds to the A-domain of the integrin α2 subunit proximal to the collagen-binding site. Biochem J. 376 (Pt 1), 77-85 (2003).
- Eble, J. A., et al. The α2β1 integrin-specific antagonist rhodocetin is a cruciform, heterotetrameric molecule. FASEB J. 23 (9), 2917-2927 (2009).
- Eble, J. A., et al. Dramatic and concerted conformational changes enable rhodocetin to block α2β1 integrin selectively. PLoS Biol. 15 (7), e2001492(2017).
- Bracht, T., Figueiredo de Rezende, F., Stetefeld, J., Sorokin, L. M., Eble, J. A. Monoclonal antibodies reveal the alteration of the rhodocetin structure upon α2β1 integrin binding. Biochem J. 440 (1), 1-11 (2011).
- Harlow, E., Lane, D. Chapter 5: Immunizations. Antibodies, a laboratory manual. 5, Cold Spring Harbor Laboratory. 53-138 (1988).
- Lu, D. Analyzing interactions between SSB and proteins by the use of fluorescence anisotropy. Methods Mol Biol. 922, 155-159 (2012).
- Fielding, L. NMR methods for the determination of protein-ligand dissociation constants. Curr Top Med Chem. 3 (1), 39-53 (2003).
- Leavitt, S., Freire, E. Direct measurement of protein binding energetics by isothermal titration calorimetry. Curr Opin Struct Biol. 11 (5), 560-566 (2001).
- McDonnell, J. M. Surface plasmon resonance: towards an understanding of the mechanisms of biological molecular recognition. Curr Opin Chem Biol. 5 (5), 572-577 (2001).
- Rich, R. L., Myszka, D. G. Advances in surface plasmon resonance biosensor analysis. Curr Opin Biotechnol. 11 (1), 54-61 (2000).
- Pesquero, N. C., et al. Real-time monitoring and kinetic parameter estimation of the affinity interaction of jArtinM and rArtinM with peroxidase glycoprotein by the electrogravimetric technique. Biosens Bioelectron. 26 (1), 36-42 (2010).
- Scheuermann, T. H., Padrick, S. B., Gardner, K. H., Brautigam, C. A. On the acquisition and analysis of microscale thermophoresis data. Anal Biochem. 496, 79-93 (2016).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon