Method Article
Scansione automatizzata Slide e segmentazione nei tessuti fluorescente-identificati utilizzando un sistema di analisi di Widefield ad alto contenuto
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Qui descriviamo un protocollo per la segmentazione automatica dei tessuti fluorescente contrassegnati su diapositive utilizzando un sistema di analisi ad alto contenuto di widefield (WHCAS). Questo protocollo ha vaste applicazioni in qualsiasi campo che coinvolge la quantificazione di marcatori fluorescenti in tessuti biologici, tra cui le scienze biologiche, ingegneria medica e scienze della salute.
Abstract
Automatizzato di scansione di diapositive e segmentazione dei tessuti con etichetta fluorescente è il modo più efficace per analizzare tutta diapositive o sezioni di tessuto grande. Purtroppo, molti ricercatori spendono grandi quantità di tempo e risorse per sviluppare e ottimizzare i flussi di lavoro che sono rilevanti solo per i loro esperimenti. In questo articolo, descriviamo un protocollo che può essere utilizzato da coloro che hanno accesso ad un sistema di analisi ad alto contenuto di widefield (WHCAS) per qualsiasi tessuto montata, con opzioni di personalizzazione all'interno dei moduli pre-costruiti, trovate il software associato di immagine. Non originariamente inteso per la scansione di diapositive, le operazioni descritte in questo articolo rendono possibile acquisire diapositive scansione delle immagini in WHCAS che può essere importato nel software associato. In questo esempio, la segmentazione automatizzata di diapositive del tumore di cervello è dimostrata, ma la segmentazione automatizzata di qualsiasi marcatore fluorescente etichettati nucleare o citoplasmatica è possibile. Inoltre, ci sono una varietà di altri moduli di software quantitativi compresi saggi per la localizzazione della proteina/traslocazione, proliferazione/sopravvivenza/apoptosi cellulare e l'angiogenesi che può essere eseguito. Questa tecnica sarà risparmiare fatica e tempo di ricercatori e creare un protocollo automatizzato per l'analisi di diapositiva.
Introduzione
La quantificazione accurata e precisa dei tessuti con etichetta fluorescente su diapositive è una tecnica molto ricercata in molti campi scientifici. Tuttavia, i ricercatori spesso manualmente contano esemplari o spendono una considerevole quantità di tempo a sviluppare tecniche esoteriche automatizzate per raggiungere questo obiettivo. Qui, forniamo un protocollo per la scansione automatizzata di diapositiva e quantificazione delle cellule usando un WHCAS e il suo software associato, con cellule dell'immunità innata nelle sezioni del tumore di cervello umano congelato come un esempio. Il software associato offre una vasta gamma di built-in moduli personalizzabili da neuriti contando alla differenziazione della cellula tipi1,2,3,4,5, 6. l'obiettivo di questo metodo è quello di fornire ai ricercatori con un protocollo di inizio-fine, facilmente riproducibile per acquisire immagini di e quantificare con etichetta fluorescente entità in qualsiasi tessuti montata.
In questo protocollo, il WHCAS pricipalmente è usato per piastre per l'analisi successiva del software associato di imaging, anche se una scheda di diapositiva e le nozioni di base per far scorrere la scansione7 erano disponibili. Era proibitivo per diapositive di immagini perché l'attenta calibratura spaziale dell'area di acquisizione, la selezione di riviste appropriati, la creazione di armamenti su misura e un collegamento con i rappresentanti di prodotto sono stati richiesti. Nel più ampio corpo di letteratura, invece l'acquisto di un imaging diapositiva dedicata e analisi apparato8, un precedente rapporto tecnologico con accesso a questo software eluse l'acquisizione di immagini di diapositive sul WHCAS complessivamente9. Esecuzione di analisi di immagine o acquisizione immagine su diverse piattaforme richiede un lavoro aggiuntivo per garantire che ognuno è compatibile con l'altro.
La possibilità di utilizzare il WHCAS e il relativo software per l'acquisizione immagine vorrei evitare le complicazioni inutili di ricerca o lo sviluppo di un alieno di flusso di lavoro per questi strumenti. In questo articolo, i passaggi necessari per creare una scansione Panoramica di basso ingrandimento e le corrispondenti immagini di alto ingrandimento trattando la diapositiva come un piatto, e le successive analisi utilizzando il modulo di segmentazione di multi-lunghezza d'onda delle cellule segnando permettono per la riuso della WHCAS. Questo protocollo prontamente utilizzabile fornisce un vantaggio rispetto a tecniche alternative perché non esiste alcuna necessità di sviluppare algoritmi o multi-step conteggio protocolli10,11 , una volta che le immagini sono acquisite sulla WHCAS. Questo protocollo consente di ridurre il tempo necessario per ottimizzare una tecnica di quantificazione, è più preciso12 ed efficiente rispetto al conteggio manuale e massimizza l'uso della WHCAS. Questo protocollo può essere ampiamente e facilmente utilizzato poiché essa consente l'imaging e l'analisi di eventuali tessuti fluorescente etichettati sulle diapositive.
Protocollo
Gli esemplari del tumore sono stati ottenuti secondo il protocollo approvato dal comitato di revisione istituzionale locale amministrazione e di etica e condotta secondo le normative nazionali. Il WHCAS e il suo software associato utilizzato in questo articolo sono elencati nella Tabella materiali.
1. l'importazione delle riviste
- Aprire il software associato.
- Scarica la suite di journal indicata nella Tabella materiali.
- Importare la suite di giornale di registrazione in una directory adatta cliccando sul menu principale voce Journal, selezionando Importazione Gazzetta Suite...e facendo clic su Importa.
2. creazione impostazioni anteprima scansione acquisizione
- Nella finestra di dialogo Setup di acquisizione di piastra , passare la scheda obiettivo e fotocamera Selezionare 4 X come l'ingrandimento, 1 come la fotocamera binning e 2 come il guadagno.
- Ingresso le impostazioni in Figura 1A sotto la scheda piastra – Slidescanning.
Nota: Queste impostazioni sono state create per consentire all'utente di visualizzare e navigare fino a 3 scivoli. Ogni diapositiva è stata suddivisa in 3 pozzetti adiacenti per la navigazione facile e 'live' visualizzazione del fettine di tessuto o cellule. - Nella scheda siti da visitare , riempire i pozzetti uniformemente con i siti.
- Fare clic su Modifica piastra inferiore impostazioni... e immettere i valori visualizzati in Figura 1B.
- Sotto la scheda di Acquisizione Loop , immettere la lunghezza d'onda desiderata (qualsiasi macchia nucleare in questo esempio) per la scansione di anteprima. Per il miglior contrasto e basate su immagine messa a fuoco, utilizzare il segnale fluorescente più brillante (ad es., spesso una macchia nucleare).
Nota: Colorazione di tessuti con una macchia luminosa come una macchia nucleare è consigliato poiché questo fornisce un elevato contrasto rispetto allo sfondo. Non solo sarà questo aiuto nella posizione del campione, ma quando più di un fluoroforo nella successiva acquisizione di alto ingrandimento di imaging, utilizzerà il colore più brillante su cui basare l'imaging offset degli altri colori. - Attivare La correzione di Shading in modalità di acquisizione di piastra per cucire insieme le immagini.
- Salvare queste impostazioni come impostazione A.
3. creazione di impostazioni per l'acquisizione di diapositiva ad alto ingrandimento
- Nella finestra di dialogo Setup di acquisizione di piastra , passare la scheda obiettivo e la fotocamera seleziona 40 X come l'ingrandimento, 1 come la fotocamera binning (per la massima risoluzione digitale) e 2 come il guadagno (per aumentare il segnale e la luminosità delle immagini).
Nota: Un'impostazione di ingrandimento obiettivo inferiore può essere utilizzata, ma gli autori hanno trovato 40 X essere l'impostazione ottimale per l'analisi di umana microglia e macrofagi nel tessuto del tumore di cervello utilizzando il modulo Multi-lunghezza d'onda delle cellule segnando . - Garantire tutte le impostazioni per le schede piastra – Slidescanning e Modifica piastra inferiore impostazioni... sono la stessa di quella per impostazione A (vedi passo 2).
- Se le immagini ottenute non sono nitide, cambiare la profondità, altezza piastrae Piastra inferiore impostazioni per regolare la messa a fuoco. Se c'è ancora un problema con lo stato attivo, scegliere Laser con recupero di immagine nella sezione Opzioni di messa a fuoco automatica .
- Sotto la scheda di Acquisizione Loop , immettere il numero di lunghezze d'onda presenti nel campione. Controllare le opzioni attiva basate su laser messa a fuoco e attiva basata su immagine di messa a fuoco (per il recupero di acquisizione o laser) .
- Sotto la scheda di messa a fuoco automatica , selezionare Focus sulla piastra e ben inferiore.
- Nella scheda di riviste , selezionare le opzioni come indicato in Figura 2, assicurando che Impedisci hardware asincrono si muove anche è selezionata.
- Salvare queste impostazioni come impostazione B.
4. Posizionare la diapositiva nel sistema di analisi Widefield ad alto contenuto
- Premere F4 per arrivare al Menu principale. Selezionare scansioni vetrini. Fare clic su porta aperta – espellere Slide e inserire nelle diapositive e la scheda di presentazione (che può contenere fino a tre diapositive) in WHCAS (Figura 3A e 3B). Orientare la diapositiva con il coprioggetto rivolto verso il basso e l'etichetta sul lato la tacca nella scheda diapositiva (Figura 3A).
Nota: Le slides utilizzate in questo esperimento sono stati standard 25 x 75 x diapositive di 1 mm. - Fare clic su Chiudi la porta – scivolo di carico.
5. l'acquisizione di una scansione di anteprima
- Sotto il titolo di Screening , selezionare piastra di acquisizione e controllo... e Piastra acquisizione Setup... e caricare impostazione A.
- Sotto la scheda di pozzetti da visitare , spostare il bene contenente il campione, con il pulsante destro del mouse. Sotto la scheda di siti da visitare , spostare diversi siti con la cattura dell'immagine Live fino a quando le cellule si trovano. Concentrarsi sul campione o utilizzare l' Autofocusmanualmente. Utilizzare Snap per catturare l'immagine.
Nota: Il campione è più facile da individuare con più siti e montando i siti per il pozzo (Vedi punto 2.3). - Nel menu principale, selezionare Anteprima diapositive.
- Nella finestra di dialogo che si apre automaticamente, selezionare l'opzione che riflette quante diapositive stanno per essere analizzati. Scansione di una diapositiva alla volta. Premere OK. Selezionare 4 X come l'ingrandimento. Premere OK. Selezionare coprioggetto giù (0,17 mm) come l'orientamento della diapositiva (utilizzare una correzione del collare se viene utilizzato uno spessore diverso vetrino coprioggetti). Premere OK. Fare clic su continuare.
Nota: Se l'utente desidera acquisire scansioni di anteprima per 2 o più diapositive, ogni scansione di anteprima deve essere aperti individualmente prima dell'acquisizione dell'immagine corrispondente ad alto ingrandimento. - Sotto Carico impostazione di Slide di analisi, garantire che le impostazioni di acquisizione e calibrazioni caselle siano selezionate. Premere Annulla. Quando la casella di Impostazioni di scansione Slide riapparirà, selezionare continua.
- Quando viene visualizzata la finestra di dialogo Scansione diapositiva , regolare le impostazioni come suggerito nella Figura 4A-4 C. Selezionare Chiudi al termine.
Nota: Aumentando la fotocamera binning e diminuendo il tempo di esposizione della fotocamera si tradurrà in una scansione più rapida, anche se a un inferiore digitale ad alta risoluzione e gamma dinamica, rispettivamente, quindi diminuendo la qualità dell'immagine. Correzione dell'ombreggiatura garantirà un'intensità uniforme attraverso un unico campo di vista. Girando su Hardware e immagine messa a fuoco automatica assicura la scansione è a fuoco ma rallenterà la scansione. Nell'interesse di risparmio di tempo, è consigliabile avere ombreggiatura correzione Hardware e immagine messa a fuoco automatica fuori mentre stava acquistando la scansione a basso ingrandimento, ma di avere queste opzioni acceso per passaggio 6 durante l'imaging ad alto ingrandimento. La risoluzione della scansione basso ingrandimento non influenzerà la risoluzione della scansione ad alto ingrandimento. - Selezionare una directory per la diapositiva dati di scansione. Fare clic su OK. Immettere un nome per il file. Fare clic su OK.
- Automaticamente verrà visualizzata la finestra di dialogo Disegna regione . Utilizzare lo strumento di area rettangolare (Figura 5A) per selezionare l'area di scansione intero anteprima come mostrato in figura 5B. Fare clic su continuare.
- Verrà visualizzata la finestra di dialogo Installazione completata . Selezionare continuare.
Nota: La scansione di diapositive anteprima inizierà ora. - Quando l'anteprima è completata, apparirà la finestra di dialogo Scansione completata . Selezionare continuare. Non chiudere la finestra di scansione di anteprima (in questo esempio la finestra di Scan DAPI ; Vedi Figura 6).
Nota: L'installazione e il processo di acquisizione di ingrandimento basso scansione richiederà circa 20 min. Il tempo di acquisizione immagine successiva ad alto ingrandimento dipenderà il numero di siti scelti così come i tempi di esposizione per ogni canale.
6. alto ingrandimento scansione
- Caricare l'impostazione di B.
- Determinare quali pozzi sono essere imaged. Sotto la scheda di pozzetti da visitare , spostare il bene contenente il campione, con il pulsante destro del mouse.
Nota: La tecnica è illustrata qui di ben 1 di imaging. - Sotto la scheda di siti da visitare , spostare diversi siti con la funzionalità Live fino a quando le cellule o i tessuti si trova. Concentrarsi sul campione o utilizzare l' Autofocusmanualmente. Utilizzare Snap per catturare l'immagine.
Nota: Per aiutare a localizzare il campione ad un alto ingrandimento, utilizzare la finestra di dialogo piastra di acquisizione e controllo per regolare le dimensioni di passaggio ovunque da 5 a 100 µm per trovare l'esempio se messa a fuoco automatica non può. - Sotto la scheda W1 DAPI , fare clic su Auto esporre. Garantire che l'immagine Piastra acquisizione Snap - DAPI è croccante.
- Il menu principale, fare clic su Giornale di rivelare il pull-down. Selezionare Esegui Journal....
- Fare doppio clic sulla rivista Installazione di acquisizione regione diapositiva per lanciarlo. Quando viene visualizzata la finestra di dialogo Installazione Slide regione acquisizione , selezionare continua.
- Quando richiesto, selezionare correttamente il DAPI Scan per selezionare l'immagine della diapositiva a basso ingrandimento. Premere OK. Allo stesso modo, evidenziare la piastra acquisizione Snap - DAPI quando ha chiesto per lo snap di acquisizione di piastra. Fare clic su OK.
- Quando viene visualizzata la casella Crea o regioni di carico , è possibile utilizzare lo strumento di area rettangolare per selezionare (a) della regione di interesse nella DAPI Scan (Figura 6). Premere continua.
Nota: La superficie massima che può essere selezionata a 40x è 45 colonne di 35 righe. Se sono selezionate più aree di interesse, le regioni verranno ridimensionate alla casella con l'area più grande. Le caselle possono essere riposizionate dopo il ridimensionamento. - Selezionare No sotto la finestra di dialogo Aree di carico .
Nota: Selezionando Sì carica precedentemente salvato regioni per lotti di diapositive con la regione stessa dei luoghi di interesse. - Utilizzando lo strumento Locator , evidenziare la più grande regione di interesse e premere continua sotto la finestra di dialogo Selezionare l'area . Quando appare la finestra di dialogo Confirm regioni , selezionare continua. Decidere se la regione deve essere salvato quando viene visualizzata la finestra di dialogo Salva regioni .
- Nella finestra successiva, Sito spaziatura, permette all'utente di scegliere o in piastrelle, che si traduce in un'immagine senza sovrapposizione, o 10% si sovrappongono. Scegliere un'opzione e fare clic su OK. Gli autori hanno scelto maiolica.
- Tre finestre di dialogo verranno visualizzati in questo momento. Premendo continuapuò essere chiusa la finestra di Installazione completata . Mantenere la casella di Configurazione del sito di acquisizione per 40 X (figura 7A) aperto per ora poiché rende chiaro il numero di colonne e righe devono essere immessi nella finestra di Setup di acquisizione di piastra (figura 7B). Tenere la scatola di WellPositions (Figura 7) che appare nell'angolo inferiore sinistro aperto per tutta la durata dell'imaging.
- Nella finestra Acquisizione piastra di supporto di quest'ultima, sotto Wells da visitare, lo stesso numero di pozzi come regioni di interesse selezionati in precedenza, deve essere evidenziata (Figura 7).
Nota: Gli autori hanno sviluppato questo protocollo per un massimo di nove regioni di interesse per ogni diapositiva. Se ci sono più, semplicemente aumentare il numero di righe o colonne nella scheda di piatto - SlideScanning per riflettere il numero delle regioni di interesse. Questi pozzi spazialmente non rappresentano la posizione delle regioni di interesse in qualsiasi modo, ma il numero appropriato deve essere evidenziata (tasto sinistro del mouse). - Nella finestra di Setup di acquisizione di piastra , vai a della scheda di siti da visitare immettere le colonne suggerite e le righe (figura 7B). Ricordatevi di cliccare su siti di piastrelle.
- Per eliminare le congetture di trovare i siti contenente il campione, è necessario premere La piastra di acquisire sotto la scheda di Riepilogo per posizionare il tavolino presso l'area pre-selezionata di interesse. Una volta che la telecamera su un sito che contiene le celle, premere Annulla l'angolo inferiore destro per interrompere l'acquisizione dell'immagine. Garantire la fase è ora situato sopra il campione.
- Ottimizzare le impostazioni della lunghezza d'onda per garantire che si ottiene un concentrato e un'immagine di alta gamma dinamica. Quando siete soddisfatti, fare clic sulla scheda Riepilogo e Piastra di acquisire.
7. image Analysis
- Selezionare il modulo personalizzato desiderato.
Nota: Gli autori hanno scelto il modulo Multi-lunghezza d'onda delle cellule segnando per dimostrare una segmentazione automatizzata le diapositive del tumore di cervello. - Eseguire l'analisi su tutti i siti. Se i parametri di analisi sono gli stessi per molte diapositive, l'analisi può essere incluso nella finestra di Setup di acquisizione piastra sotto la scheda di Acquisizione Post .
- Una volta terminata l'analisi, è necessario escludere tutti i siti di immagini di scarsa qualità per non falsare i risultati. Poiché l'obiettivo di questa analisi è quello di quantificare la microglia e macrofagi nel parenchima del tumore, seleziona la regione di interesse entro i bordi del campione del tumore. Inoltre, escludere i siti che sono fuori fuoco e/o contengono tessuto pieghe o bolle e lacrime nel tessuto. Per una dimostrazione, vedere i Risultati di rappresentante. In alternativa, utilizzare il Punteggio di messa a fuoco del Laser generato per ogni sito in base all'ampiezza della riflessione laser come una soglia di sotto quali sono i siti da escludere (la soglia è personalizzabile e dipende da parametri quali il tempo di esposizione e la tipo di supporto utilizzato).
Nota: Le immagini di alto ingrandimento rendono possibile escludere specificamente strutture, quali i grandi vasi sanguigni e aree di necrosi, se necessario. È anche possibile includere solo le aree di interesse, come quelle che sono hypercellular. In questo modo, la specificità e l'accuratezza delle analisi può essere aumentate.
Risultati
Le immagini possono essere visualizzate all'interno del software WHCAS. Figura 8 Mostra le miniature di alto ingrandimento di tutti i siti all'interno dell'area definita di interesse. Scrivi una recensione su ogni sito per identificare quelli che dovrebbero essere esclusi dall'analisi (esempi sono mostrati a basso e alto ingrandimento in Figura 8 e Figura 9, rispettivamente). Ad esempio, sito 149 è fuori fuoco (Figura 9A), sito 219 ha bolle (figura 9B) e 54 sito contiene una piega (Figura 9) e dovrebbero essere esclusi. È tipico di escludere il 10-15% di tutti i siti imaged. Nell'esempio fornito, il 15,3% dei siti sono stati esclusi (25/300 aveva bolle, 17/300 erano fuori fuoco, 3/300 sono stati piegati e 1/300 era sul bordo). Figura 10A è un'immagine rappresentativa scelta per l'analisi (sua miniatura corrispondente basso ingrandimento è illustrato nella Figura 8). Qui, le sovrapposizioni corrispondente generate dal modulo multi-lunghezza d'onda delle cellule segnando dimostrano i risultati della segmentazione automatica eseguita sul sito 59, su misura per le specifiche degli autori (larghezza minima nuclei = 2,5 µm, larghezza massima = 7,5 µm, e intensità sopra sfondo locale = 35 graylevels; Larghezza minima le cellule CD11b-positive = 4 µm, larghezza massima = 18 µm, minima zona macchiata = 15 µm2e l'intensità sopra sfondo locale = 310 graylevels; e larghezza minima le cellule CD45-positive = 4 µm, larghezza massima = 18 µm, minima zona macchiata = 15 µm2e l'intensità sopra sfondo locale = 50 graylevels). Dopo la segmentazione, i dati quantitativi della proporzione di cellule macchiatura positiva per ogni indicatore (6,2 ± 5,1% e 3,8 ± 2,1% di CD11b+ e CD45+ le cellule, rispettivamente), entrambi gli indicatori (3,5 ± 2,1% CD11b+CD45+ cellule), nessun marcatori (86,6 ± 9,0% CD11b–CD45 cellule– ; Figura 10B), significa zona macchiata (40,0 µm ± 9.2 e 36,7 ± 7,6 µm di CD11b+ e CD45+ le cellule, rispettivamente; Figura 10) e significa che l'intensità di fluorescenza (408,9 ± 40,3 fluorescenza relativa unità e 373,9 ± 38,1 relativa fluorescenza di CD11b+ e CD45+ le cellule, rispettivamente; Figura 10) possono essere ottenuti.
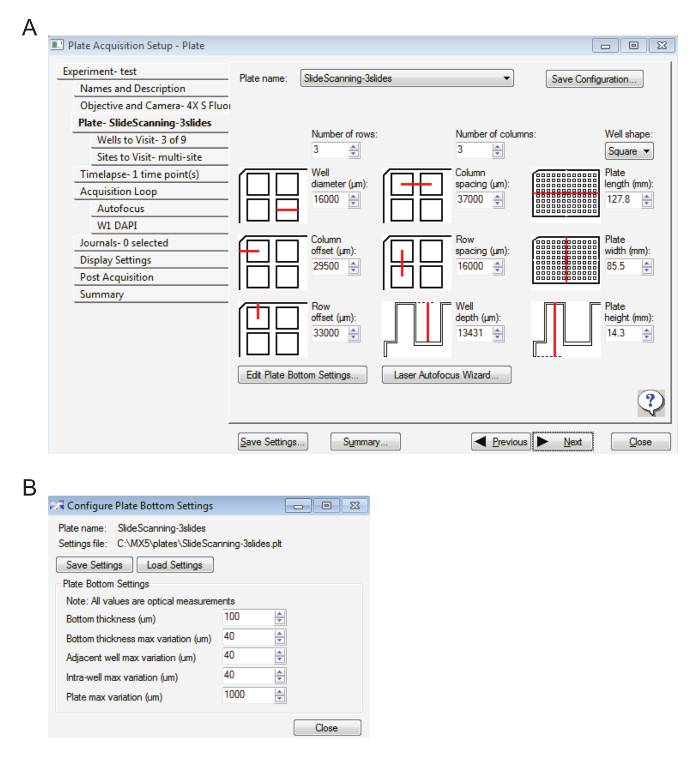
Figura 1: impostazioni per l'acquisizione di diapositiva a basso ingrandimento. A. questa immagine vengono visualizzate le impostazioni di piastra. B. qui, in cui vengono visualizzate le impostazioni di fondo piatto. Clicca qui per visualizzare una versione più grande di questa figura.
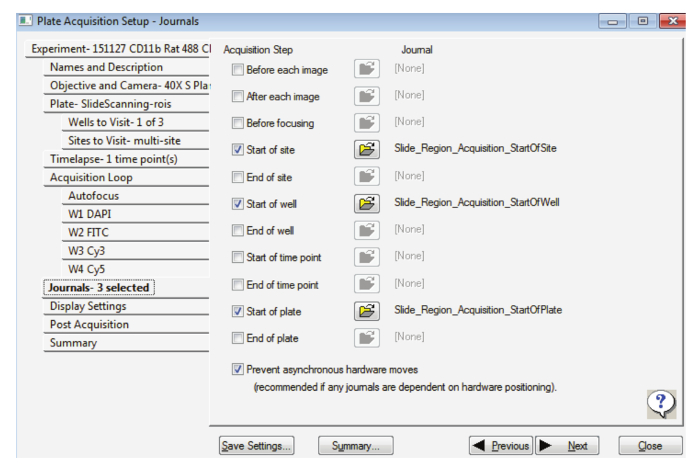
Figura 2: impostazioni per l'acquisizione di diapositiva a forte ingrandimento. Questa figura mostra la selezione delle riviste appropriate. Clicca qui per visualizzare una versione più grande di questa figura.
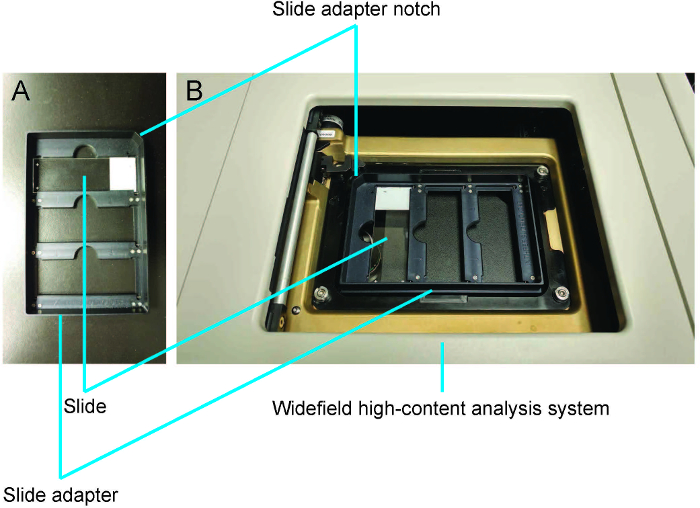
Figura 3: dimostrazione di come caricare diapositive utilizzando la scheda diapositiva. A. la diapositiva viene inserita coprioggetto giù con l'etichetta sul lato la tacca di adattatore di diapositiva. B. la scheda diapositiva viene caricata il sistema di analisi ad alto contenuto di widefield. Clicca qui per visualizzare una versione più grande di questa figura.
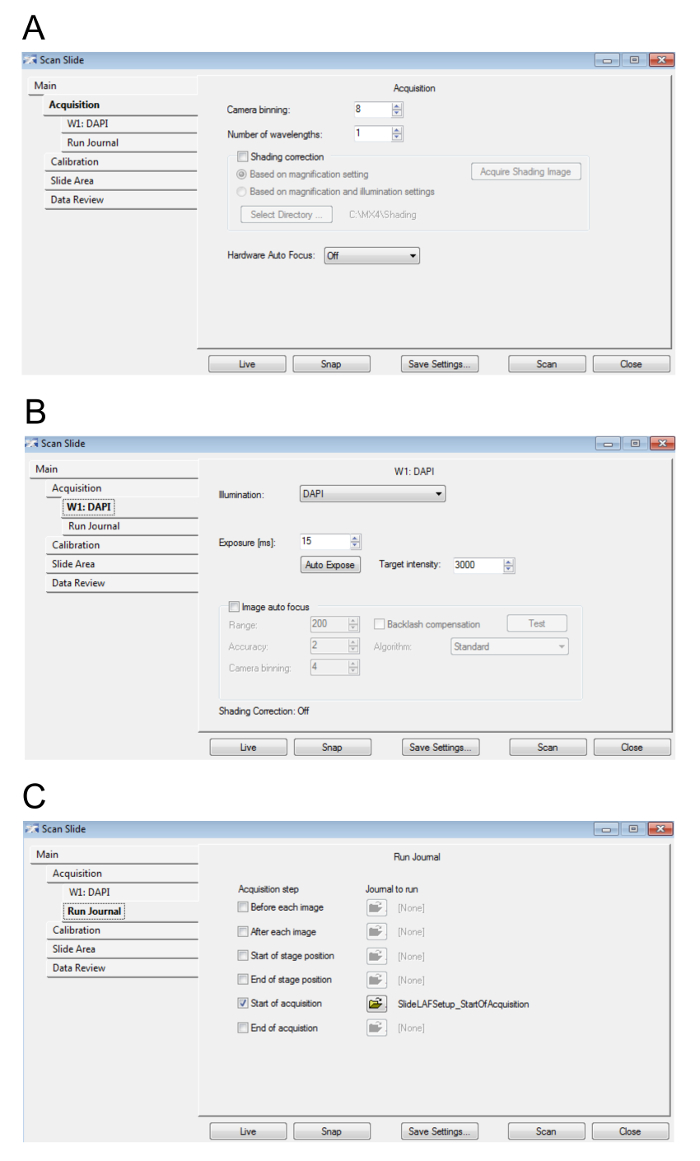
Figura 4: impostazioni di anteprima diapositiva. A. questa cifra consente di visualizzare i parametri per l'acquisizione. B. qui, vengono visualizzati i valori per le impostazioni di lunghezza d'onda di Hoescht/DAPI. C. questa immagine mostra le impostazioni del journal. Clicca qui per visualizzare una versione più grande di questa figura.
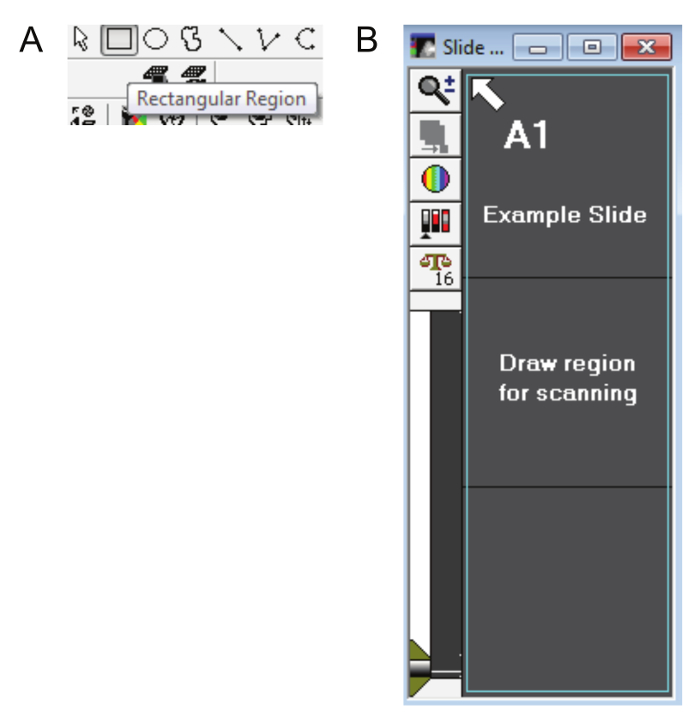
Figura 5: disegno di una regione di diapositiva. A. lo strumento area rettangolare (non possono essere utilizzati altri strumenti di disegno) viene utilizzato per selezionare l'area di scansione di anteprima e creare aree di interesse sull'esplorazione di DAPI. B. il contorno di teal in questa figura viene illustrato come deve essere selezionata l'intera area della diapositiva (compresa l'etichetta). Clicca qui per visualizzare una versione più grande di questa figura.

Figura 6: creazione di aree di interesse sulla scansione anteprima. L'anteprima o scansione DAPI rappresenta l'area intera diapositiva. In questo esempio, ci sono tre sezioni di tessuto di serie del cervello (i contorni rettangolare bianchi solidi). L'area sopra queste sezioni rappresenta le etichette circolari sulla diapositiva. Accordi di sezione diversa non limiterà la successiva selezione delle regioni di interesse. La regione di interesse in questo particolare esempio è mostrata con un contorno rettangolare bianco tratteggiato. La barra della scala = 1 mm. per favore clicca qui per visualizzare una versione più grande di questa figura.
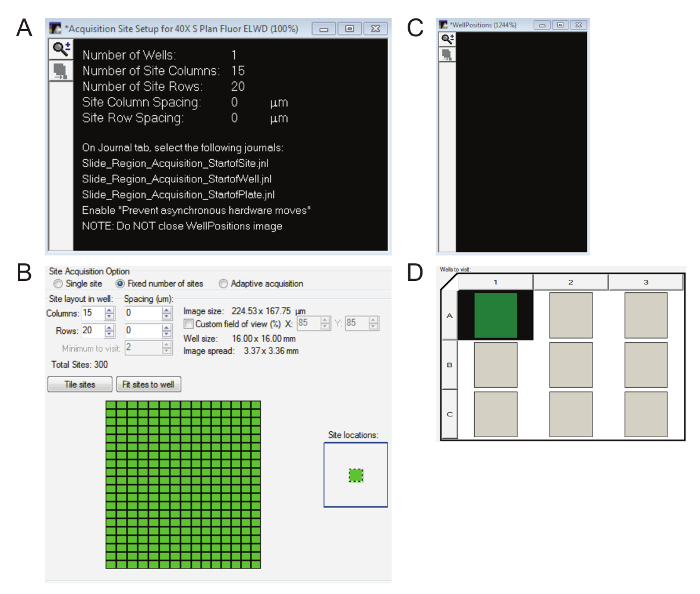
Figura 7: acquisizione di immagini di windows essenziali per elevati ingrandimenti. A. acquisizione sito Setup finestra mostrerà quante righe e colonne sono all'interno di regioni di interesse. B. assicurare il numero corretto di colonne e righe indicate in figura 7A sono immessi nella casella di acquisizione piastra di supporto e i siti sono piastrellati. C. è necessario tenere la finestra di WellPositions aperta per tutta la durata dell'acquisizione immagine, anche se è vuota. D. il numero appropriato di regioni di interesse deve essere evidenziato. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 8: immagine rappresentativa della regione di interesse. Ogni sito possa essere visualizzato in una composito miniatura della regione di interesse. I siti rappresentativi che sono state escluse (contrassegnati con caselle rosse) e un esempio di un sito incluso (contrassegnato con una casella verde) è mostrato ad un più alto ingrandimento. Si raccomanda di esaminare ogni sito per verificare che è di qualità sufficiente per l'analisi. La barra della scala = 1 mm. per favore clicca qui per visualizzare una versione più grande di questa figura.
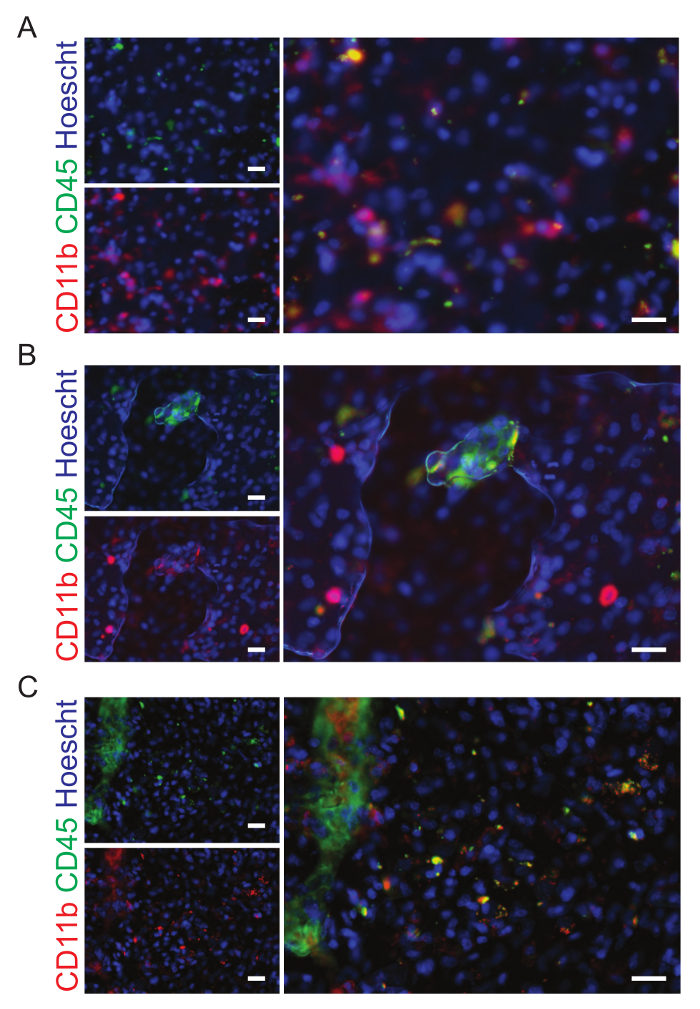
Figura 9: esempi di siti che dovrebbero essere esclusi dall'analisi. A. le sezioni del tumore di cervello umano sono state colorate con CD11b (verde) e CD45 (rosso), gli indicatori di microglia e macrofagi. Questa immagine è fuori fuoco ed è stata esclusa dall'analisi. B. le bolle sono presenti in questa immagine che è stata esclusa dall'analisi finale. C. Questo sito è stato escluso dall'analisi a causa della piega nel tessuto. Le barre di scala sono 20 µm. Clicca qui per visualizzare una versione più grande di questa figura.
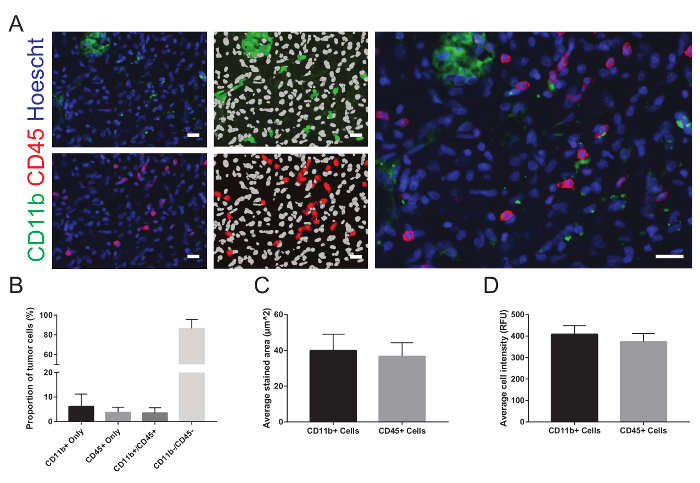
Figura 10: analisi e immagini rappresentative. A. ogni immagine viene visualizzata accanto alle sovrapposizioni che rappresenta i risultati della segmentazione automatizzata. Nella sovrapposizione, i nuclei sono visualizzati in bianco e cellule marcate positivamente sono mostrate in verde per CD11b e rosso per CD45. Dopo i siti di qualità inadeguata l'esclusione dall'analisi e combinando i risultati di tutti i siti rimanenti, B. le proporzioni medie del numero totale delle cellule in ogni sito che hanno macchiato il positivo per ogni indicatore e co-identificato per entrambi gli indicatori, C. la media macchiato zona e D. l'intensità media delle celle sono rappresentate graficamente. RFU = fluorescenza relativa unità. I dati sono espressi come media ± deviazione standard. Le barre di scala sono 20 µm. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Un comune ancora problema che ostacolano l'efficienza della ricerca scienza biologica è lo sviluppo di protocolli per la quantificazione precisa, accurata e imparziale dei tessuti fluorescente-identificati e le loro strutture all'interno. Notevoli quantità di tempo e fatica sono orientate a trovare modi per analizzare i vetrini di tessuto una volta che sono stato ripreso. Molti metodi esistenti forniscono algoritmi agli utenti di ricreare all'interno di programmi12,13,14. Questi metodi sono accettabili, ma il significato di questo rapporto è che esso consente all'utente di facilmente e rapidamente stabilire un protocollo di analisi e acquisizione di immagini olistico se l'utente ha accesso a un WHCAS. Saggi che differenziano i tipi di cellule e quantificare molteplici strutture e processi, analisi del ciclo cellulare e traslocazione nucleare, ad esempio, sono già disponibili nel software associato.
L'installazione richiede pochi passaggi critici. In primo luogo, i parametri spaziali della diapositiva sono definiti come se si trattasse di un piatto. In secondo luogo, una scansione di Panoramica di basso ingrandimento viene creata da cui regioni di interesse sono selezionate per l'imaging ad alto ingrandimento. Infine, sono esclusi i siti che riguardano l'accuratezza e la precisione delle analisi successive. Il più grande limite di questa tecnica è che la sua applicabilità dipende se l'utente ha accesso a un WHCAS. Tuttavia, con la crescente necessità di sistemi di analisi ad alto contenuto, molte istituzioni stanno fornendo questi ai loro ricercatori a rimanere competitivo15. Risoluzione dei problemi è necessario, più comunemente, quando le sezioni di tessuto non hanno lo stesso spessore. Se sono selezionate più aree di interesse, alcuni saranno a fuoco mentre altri non lo faranno. Idealmente, durante il sezionamento, l'utente sarebbe preso cura di creare campioni omogenei. Tuttavia, se i campioni sono incoerenti, l'attenzione per la lunghezza d'onda di macchia nucleare (o fluorophore più brillanti dell'utente), che viene utilizzato per mettere a fuoco, può essere regolata per ogni regione di out-of-focus di interesse e anche per ogni campo di vista. Come le altre lunghezze d'onda sono semplicemente offset dalla lunghezza d'onda di macchia nucleare, solo questa lunghezza d'onda ha bisogno di riaggiustamento.
In questo rapporto, abbiamo dettaglio come eseguire la scansione e analizzare diapositive utilizzando un WHCAS e il software associato. Il modulo multi-lunghezza d'onda delle cellule segnando consente all'utente di contare automaticamente eventuali marcatori nucleari o citoplasmatici fluorescente etichettati. Dopo la regolazione delle impostazioni di messa a fuoco e definizione delle caratteristiche cellulari come larghezza e area per personalizzare il modulo al tessuto imaged, non vi è più necessità di intervento dell'utente ottenere le diapositive imaged e dati quantitativi. Fino a tre diapositive possa essere imaged in un momento e possono essere definite più aree di interesse. Questo consente di protocollo WHCAS gli utenti che hanno bisogno di analizzare diapositive sfruttare personalizzabile, multiuso, automatizzato dei flussi di lavoro che non richiedono poca o nessuna ottimizzazione e può essere applicati in tutti i progetti in futuro che coinvolgono l'analisi istologica del tessuto.
Divulgazioni
Gli autori non hanno nessun interessi finanziari prodotti descritti in questo manoscritto e nient'altro da divulgare.
Riconoscimenti
Questo progetto è stato finanziato da una sovvenzione dal istituti canadesi di ricerca sanitaria e Alberta Innova - Health Solutions/Alberta Cancer Foundation. Gli autori desiderano riconoscere l'unità di rigenerazione in funzione di memoria per l'utilizzo della propria attrezzatura, il lavoro di Paula Gedraitis nella costruzione della Fondazione su cui scorrono scansione sul WHCAS è stato reso possibile e il creatore dei prodotti neurobiologia citati in questo articolo, Molecular Devices.
Materiali
| Name | Company | Catalog Number | Comments |
| ImageXpress MicroXLS | Molecular Devices | NA | Apparatus for image acquisition |
| MetaXpress 5.1 | Molecular Devices | NA | Associated software for ImageXpress MicroXL (runs on a PC with the Windows operating system). |
| Slide adapter | Molecular Devices | NA | Metal slide holder that fits into ImageXpress MicroXL |
| Slide_Region_Acquisition_revA.jzp | Molecular Devices | NA | The journal can be obtained from metamorph.moleculardevices.com/forum/showthread.php?tid=218&highlight=slide or from contacting a Molecular Devices representative |
| Slide_Region_Acquisition _Setup.JNL | Molecular Devices | NA | Select this journal in Step 6.6. |
Riferimenti
- Hua, Y., Shun, T. Y., Strock, C. J., Johnston, P. A. High-content positional biosensor screening assay for compounds to prevent or disrupt androgen receptor and transcriptional intermediary factor 2 protein-protein interactions. Assay and Drug Development Technologies. 12 (7), 395-418 (2014).
- Rishal, I., et al. WIS-NeuroMath enables versatile high throughput analyses of neuronal processes. Developmental Neurobiology. 73 (3), 247-256 (2013).
- Kanungo, J., Lantz, S., Paule, M. G. In vivo imaging and quantitative analysis of changes in axon length using transgenic zebrafish embryos. Neurotoxicology and Teratology. 33 (6), 618-623 (2011).
- Schurmann, C., et al. Analyzing illumina gene expression microarray data from different tissues: methodological aspects of data analysis in the MetaXpress Consortium. PLoS ONE. 7 (12), e50938 (2012).
- Vogt, A., Codore, H., Day, B. W., Hukriede, N. A., Tsang, M. Development of automated imaging and analysis for zebrafish chemical screens. Journal of Visualized Experiments. (40), e1900 (2010).
- Bravo-San Pedro, J. M., et al. High-throughput quantification of GFP-LC3+ dots by automated fluorescence microscopy. Methods in Enzymology. , 71-86 (2017).
- Varga, V. S., et al. Automated multichannel fluorescent whole slide imaging and its application for cytometry. Cytometry Part A. 75 (12), 1020-1030 (2009).
- Narayan, P. J., et al. Assessing fibrinogen extravasation into Alzheimer's disease brain using high-content screening of brain tissue microarrays. Journal of Neuroscience Methods. 247, 41-49 (2015).
- Du, Y., Budman, H. M., Duever, T. A. Segmentation and quantitative analysis of apoptosis of Chinese hamster ovary cells from fluorescence microscopy images. Microscopy and Microanalysis. 23 (3), 569-583 (2017).
- Mueller, J. L., et al. Quantitative segmentation of fluorescence microscopy images of heterogeneous tissue: application to the detection of residual disease in tumor margins. PLoS One. 8 (6), e66198 (2013).
- Donnelly, D. J., Gensel, J. C., Ankeny, D. P., van Rooijen, N., Popovich, P. G. An efficient and reproducible method for quantifying macrophages in different experimental models of central nervous system pathology. Journal of Neuroscience Methods. 181 (1), 36-44 (2009).
- Kozlowski, C., Weimer, R. M. An automated method to quantify microglia morphology and application to monitor activation state longitudinally in vivo. PLoS One. 7 (2), e31814 (2012).
- Fish, K. N., Sweet, R. A., Deo, A. J., Lewis, D. A. An automated segmentation methodology for quantifying immunoreactive puncta number and fluorescence intensity in tissue sections. Brain Research. 1240, 62-72 (2008).
- Zock, J. M. Applications of high content screening in life science research. Combinatorial Chemistry & High Throughput Screening. 12 (9), 870-876 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon