È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Selezionando più biomarcatore sottoinsiemi con similmente efficace classificazione binaria spettacoli
In questo articolo
Riepilogo
Algoritmi esistenti generano una soluzione per un set di dati di rilevamento biomarcatore. Questo protocollo dimostra l'esistenza di molteplici soluzioni similmente efficaci e presenta un software user-friendly per aiutare i ricercatori biomedici indagare il loro set di dati per la sfida proposta. Gli informatici possono anche fornire questa funzionalità nel loro biomarcatore algoritmi di rilevamento.
Abstract
Rilevamento di biomarcatore è una delle più importanti domande biomedicale per i ricercatori di high-throughput "omiche", e quasi tutti gli algoritmi di rilevamento biomarcatore esistenti generano un sottoinsieme di biomarcatore con la misura di prestazioni ottimizzate per un determinato set di dati . Tuttavia, un recente studio ha dimostrato l'esistenza di più sottoinsiemi di biomarcatore con esibizioni di classificazione similmente efficaci o addirittura identici. Questo protocollo presenta una metodologia semplice e diretta per la rilevazione di sottoinsiemi di biomarcatore con esibizioni di classificazione binaria, meglio di un cut-off definito dall'utente. Il protocollo consiste di preparazione dei dati e caricamento, Riepilogo informazioni di base, parametro tuning, lo screening biomarcatore, visualizzazione dei risultati e interpretazione, biomarcatore gene annotazioni ed esportazione di risultato e la visualizzazione a qualità di pubblicazione. Il biomarcatore proposto strategia di screening è intuitivo e dimostra una regola generale per lo sviluppo di algoritmi di rilevamento del biomarcatore. Un'interfaccia grafica utente (GUI) è stata sviluppata utilizzando il linguaggio di programmazione Python, permettendo i ricercatori biomedici di avere accesso diretto ai loro risultati. Il manuale di kSolutionVis e il codice sorgente può essere scaricati da http://www.healthinformaticslab.org/supp/resources.php.
Introduzione
Classificazione binaria, uno dei più comunemente studiato e dati impegnativi problemi in ambito biomedico, di data mining viene utilizzato per costruire un modello di classificazione addestrato su due gruppi di campioni con la più accurata discriminazione potenza1, 2 , 3 , 4 , 5 , 6 , 7. Tuttavia, le grandi quantità di dati generati in campo biomedico ha l'intrinseca "grande p piccolo n" paradigma, con il numero di caratteristiche solitamente molto più grande rispetto al numero di campioni6,8,9. Pertanto, i ricercatori biomedici necessario ridurre la dimensione caratteristica prima che utilizza gli algoritmi di classificazione per evitare l'overfitting problema8,9. Biomarcatori di diagnosi sono definiti come un sottoinsieme delle caratteristiche rilevate che separa i pazienti di una determinata malattia da sani di controllo campioni10,11. I pazienti sono solitamente definiti come i campioni positivi, e i controlli sani sono definiti come i campioni negativi12.
Studi recenti hanno suggerito che esiste più di una soluzione con prestazioni identiche o similmente efficace classificazione per un set di dati biomedici5. Quasi tutti gli algoritmi di selezione funzionalità sono algoritmi deterministici, producendo solo una soluzione per lo stesso dataset. Gli algoritmi genetici possono generare simultaneamente molteplici soluzioni con prestazioni simili, ma cercano ancora di selezionare un'unica soluzione con la migliore funzione di fitness come output per un dato set di dati13,14.
Algoritmi di selezione funzionalità possono essere approssimativamente raggruppati come filtri o wrapper12. Un algoritmo di filtro sceglie le caratteristichek top - classificate dalla loro associazione significativa individuali con le etichette di classe binario basato sul presupposto che dispone sono indipendente da ogni altro15,16,17 . Anche se questo presupposto non vale per quasi tutti i dataset reali, la regola di filtro euristico esegue bene in molti casi, per esempio, l'algoritmo di mRMR (ridondanza minima e massima rilevanza), Wilcoxon test basato funzionalità filtro (WRank) algoritmo e la trama ROC (caratteristica di funzionamento ricevitore) basato su algoritmo di filtraggio (ROCRank). mRMR, è un algoritmo efficiente filtro perché si approssima il problema combinatorio stima con una serie di problemi molto più piccoli, confronto con l'algoritmo di selezione funzione massimo-dipendenza, ognuna delle quali prevede solo due variabili, e utilizza pertanto le probabilità di joint pairwise che sono più robusti18,19. Tuttavia, mRMR può sottovalutare l'utilità di alcune caratteristiche non misura le interazioni tra caratteristiche che possono aumentare la pertinenza e così non trova alcune combinazioni di funzionalità che sono individualmente inutile ma sono utili solo quando combinato. L'algoritmo di WRank calcola un punteggio non parametrica di come discriminante una caratteristica è tra due classi di campioni ed è noto per la sua robustezza per outlier20,21. Inoltre, l'algoritmo di ROCRank valuta come significativo l'Area Under the ROC curva (AUC) di una particolare caratteristica è per la classificazione binaria indagate prestazioni22,23.
D'altra parte, un wrapper valuta le prestazioni del classificatore pre-definiti di un sottoinsieme di funzionalità specificato, in modo iterativo generato da una regola euristica e crea il sottoinsieme di funzionalità con le migliori prestazioni misura24. Un wrapper generalmente supera un filtro delle prestazioni di classificazione ma corre più lento25. Ad esempio, l'algoritmo di27 26,foresta casuale regolarizzata (RRF) utilizza una regola di avida, valutando le caratteristiche su un sottoinsieme dei dati di training a ogni nodo casuale foresta, cui punteggi di caratteristica importanza vengono valutate tramite l'indice di Gini . La scelta di una nuova funzionalità sarà penalizzata se suo guadagno informazioni non migliora che delle caratteristiche selezionate. Inoltre, l'analisi di previsione per i Microarrays (PAM)28,29 algoritmo, anche un algoritmo di wrapper, calcola un centroide per tutte le etichette di classe e quindi seleziona funzionalità per compattare i centroidi gene verso la totale centroide di classe. PAM è robusta per caratteristiche periferiche.
Molteplici soluzioni con le prestazioni di classificazione superiore possono essere necessarie per qualsiasi set di dati specificato. In primo luogo, l'obiettivo di ottimizzazione di un algoritmo deterministico è definito da una formula matematica, ad esempio, tasso di errore minimo30, che non è necessariamente ideale per campioni biologici. In secondo luogo, un set di dati possono avere soluzioni significativamente differenti, multiplo, con simili prestazioni efficaci o addirittura identici. Quasi tutti gli algoritmi di selezione funzionalità esistenti selezionerà casualmente una di queste soluzioni come l' uscita31.
Questo studio introdurrà un protocollo analitico di informatica per la generazione di soluzioni di selezione funzionalità multiple con prestazioni simili per qualsiasi set di dati di classificazione binaria dato. Considerando che i ricercatori biomedici più non familiarità con tecniche informatiche o computer di codifica, un'interfaccia grafica utente (GUI) è stata sviluppata per facilitare l'analisi rapida dei set di dati biomedici classificazione binaria. Il protocollo analitico consiste di caricamento dei dati e che riassume, parametro tuning, l'esecuzione della pipeline e interpretazioni di risultato. Con un semplice click, il ricercatore è in grado di generare il biomarcatore sottoinsiemi e pubblicazione-qualità visualizzazione grafici. Il protocollo è stato testato utilizzando i trascrittomi di classificazione binaria base dati di leucemia linfoblastica acuta (LLA), vale a dire, ALL1 e ALL212. I set di dati di ALL1 e ALL2 sono stati scaricati dal Broad Institute genoma Data Analysis Center, disponibile presso http://www.broadinstitute.org/cgi-bin/cancer/datasets.cgi. ALL1 contiene 128 campioni con 12.625 caratteristiche. Di questi esempi, 95 sono B-cellula tutti e 33 sono cellule T tutti. ALL2 include 100 campioni con 12.625 caratteristiche pure. Di questi esempi, ci sono 65 pazienti che hanno sofferto di ricaduta e 35 pazienti che non hanno fatto. ALL1 era un set di dati di facile classificazione binaria, con una precisione minima di quattro filtri e quattro wrapper essendo 96.7% e 6 degli algoritmi di selezione 8 funzionalità raggiungimento 100%12. Mentre ALL2 era un dataset più difficile, con gli algoritmi di selezione 8 caratteristica sopra raggiungere nessuno meglio di 83,7% precisione12. Questa precisione migliore è stata realizzata con 56 caratteristiche rilevate dall'algoritmo wrapper, funzionalità di selezione basata sulla correlazione (CFS).
Protocollo
Nota: Il seguente protocollo descrive i dettagli del procedimento analitico informatica e pseudo-codici dei moduli principali. Il sistema di analisi automatica è stato sviluppato utilizzando Python versione 3.6.0 e i Panda di moduli Python, abc, numpy, scipy, sklearn, sys, PyQt5, sys, Jamy, matematica e matplotlib. I materiali utilizzati in questo studio sono elencati nella Tabella materiali.
1. preparare la matrice di dati e le etichette di classe
- Preparare il file di matrice di dati come un file delimitato da tabulazioni o da virgole di matrice, come illustrato in Figura 1A.
Nota: Ogni riga contiene tutti i valori di una funzione, e il primo elemento è il nome della funzionalità. Una caratteristica è un ID di probeset per il set di dati di microarray-basata del trascrittoma o può essere un altro valore ID come un residuo di cisteina con il relativo valore di metilazione in un dataset di methylomic. Ogni colonna dà i valori della funzione di un dato campione, con il primo elemento è il nome del campione. Una riga è suddiviso in colonne da una scheda (Figura 1B) o una virgola (Figura 1). Un file delimitato da tabulazioni matrice viene riconosciuto dal TSV di estensione di file, e un file delimitato da virgole di matrice ha l'estensione. csv. Questo file può essere generato da una matrice di risparmio sia come il formato TSV o CSV da software come Microsoft Excel. La matrice di dati può essere generata anche da computer di codifica. - Preparare il file di etichette di classe come un delimitato da tabulazioni o da virgole file matrice (Figura 1), simile al file di matrice di dati.
Nota: La prima colonna indica i nomi di esempio, e l'etichetta di classe di ogni campione è dato nella colonna intitolata classe. Massima compatibilità è considerato nel processo di codifica, in modo che possono essere aggiunti ulteriori colonne. Il file di etichette di classe potrebbe essere formattato come un file TSV o CSV. I nomi nella colonna classe possono essere tutti i termini, e ci possono essere più di due classi di campioni. L'utente può scegliere due delle classi per la seguente analisi.
2. caricare la matrice di dati e le etichette di classe
- Caricare le etichette dati di matrice e classe nel software. Fare clic sul pulsante Datamatrix di carico per scegliere il file di matrice di dati specificati dall'utente. Fare clic sul pulsante etichette di classe di carico di scegliere il corrispondente file di etichette di classe.
Nota: Dopo che entrambi i file vengono caricati, kSolutionVis condurrà un routine schermo della compatibilità tra i due file. - Riassumere le caratteristiche e i campioni dal file di matrice di dati. Stimare le dimensioni del file di matrice dati.
- Riepilogare i campioni e le classi da file di etichetta della classe. Stimare le dimensioni del file di etichetta della classe.
- Verificare se ogni campione dalla matrice dei dati ha un'etichetta di classe. Riepilogare i numeri dei campioni con le etichette di classe.
3. riepilogare e visualizzare le statistiche di base del Dataset
- Fare clic sul pulsante riepiloga, senza alcun input di parola chiave specificata, e il software visualizzerà 20 caratteristiche indicizzate e i corrispondenti nomi di funzioni.
Nota: Gli utenti devono specificare il nome della funzionalità che desiderano trovare per vedere le sue statistiche base e distribuzione del valore corrispondente tra tutti i campioni di input. - Fornire una parola chiave, ad esempio "1000_at", nella casella di testo funzionalità per trovare una funzionalità specifica che si desidera riepilogare. Fare clic sul pulsante riepiloga per ottenere le statistiche di base per questa determinata funzionalità.
Nota: La parola chiave può apparire ovunque nei nomi di funzione obiettivo, facilitando il processo di ricerca per gli utenti. - Fare clic sul pulsante riepiloga per trovare più di una funzione con la parola chiave specificata e quindi specificare l'ID di caratteristica unica per procedere con il passo precedente di riassumere una caratteristica particolare.
4. determinare le etichette di classe e il numero di caratteristiche Top-ranked
- Scegliere i nomi delle classi di negativo ("N (95)") e positivi ("P (33)") nelle caselle a discesa Classe positivo e Negativo di classe, come mostrato nella Figura 2 (medio).
Nota: Si consiglia di scegliere che un dataset di classificazione binaria bilanciata, cioè, la differenza tra i numeri dei campioni positivi e negativi è minimo. Il numero di campioni è anche dato tra parentesi dopo il nome di ogni etichetta di classe nelle due caselle a discesa. - Scegliere 10 come il numero di funzioni di posizionamento (parametro pTopX) nella casella a discesa Top_X (?) per una schermata completa del sottoinsieme di funzionalità.
Nota: Il software si colloca automaticamente tutte le funzionalità del P-valore calcolato da un t-test di ogni caratteristica di confronto tra le classi di positive e negative. Una caratteristica con un più piccolo P-valore ha un potere meglio discriminante tra le due classi di campioni. Il modulo completo screening è computazionalmente intensivo. Il parametro pTopX è 10 per impostazione predefinita. Gli utenti possono modificare questo parametro nell'intervallo da 10 a 50, fino a trovare soddisfacente dispongono di sottoinsiemi con spettacoli di buona classificazione.
5. ottimizzare i parametri di sistema per prestazioni differenti
- Scegliere la misura delle prestazioni (pMeasurement), precisione (Acc) nella casella a discesa Acc/bAcc (?) per il classificatore selezionato macchina di apprendimento Extreme (ELM). Un'altra opzione di questo parametro è la misurazione precisione equilibrato (bAcc).
Nota: Lasciare che il TP, FN, TN, e FP il numero di veri positivi, falsi negativi, veri negativi e falsi positivi, rispettivamente. La misurazione Acc è definita come (TP+TN)/(TP+FN+TN+FP), che funziona meglio su un dataset equilibrato6. Ma un classificatore ottimizzato per Acc tende ad assegnare tutti i campioni alla classe negativa se il numero di campioni negativi è più grande di quello di quelli positivi. Il bAcc è definito come (Sn + Sp) / 2, dove Sn = TP/(TP+FN) e Sp = TN/(TN+FP) sono le tariffe correttamente previste per il positivo e negativo campioni, rispettivamente. Pertanto, bAcc normalizza le prestazioni di stima sopra le due classi e può portare a una performance equilibrata pronostico su due classi sbilanciate. ACC è la scelta predefinita di pMeasurement. Il software utilizza il classificatore ELM per impostazione predefinita per calcolare le prestazioni di classificazione. L'utente può anche scegliere un classificatore SVM (Support Vector Machine), KNN (k Nearest Neighbor), Decision Tree o Naïve Bayes. - Scegliere il valore di cutoff 0.70 (parametro pCutoff) per la misurazione delle prestazioni specificato nella casella di input pCutoff:.
Nota: Sia Acc e bAcc variano fra 0 e 1, e l'utente può specificare un valore pCutoff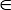 [0, 1] come il cut-off per visualizzare le soluzioni abbinate. Il software effettua un completo funzionalità-sottoinsieme di screening, e una scelta appropriata di pCutoff farà la visualizzazione 3D più intuitivo ed esplicite. Il valore predefinito per pCutoff è 0,70.
[0, 1] come il cut-off per visualizzare le soluzioni abbinate. Il software effettua un completo funzionalità-sottoinsieme di screening, e una scelta appropriata di pCutoff farà la visualizzazione 3D più intuitivo ed esplicite. Il valore predefinito per pCutoff è 0,70.
6. eseguire la Pipeline e produrre i risultati visualizzati interattivi
- Fare clic sul pulsante Analyze per eseguire la pipeline e generare i grafici di visualizzazione, come illustrato nella Figura 2 (in basso).
Nota: La tabella a sinistra dà tutti i sottoinsiemi di funzionalità e loro pMeasurement calcolato dalla strategia di validazione incrociata 10 volte del classificatore ELM, come descritto in precedenza5. Due grafici a dispersione 3D e due-linea trame vengono generati per la procedura di screening del sottoinsieme di funzionalità con le impostazioni correnti. - Scegliere 0,70 come il valore predefinito di pMeasurement taglio (parametro piCutoff, casella di input valore) e 10 come il valore predefinito del numero di sottoinsiemi di migliore funzionalità (parametro piFSNum).
Nota: La pipeline viene eseguita utilizzando i parametri pTopX, pMeasurement e pCutoff. La funzionalità rilevata sottoinsiemi possono essere ulteriormente proiettato utilizzando il taglio piCutoff, tuttavia piCutoff non può essere minore di pCutoff. Pertanto, piCutoff viene inizializzato come pCutoff e saranno visualizzati solo i sottoinsiemi di funzione con le prestazioni misura ≥ piCutoff . Il valore predefinito di piCutoff è pCutoff. A volte kSolutionVis rileva molte soluzioni e solo i migliori piFSNum (impostazione predefinita: 10) sottoinsiemi di funzionalità verranno visualizzate. Se il numero dei sottoinsiemi di funzionalità rilevato dal software è minore di piFSNum, saranno visualizzati tutti i sottoinsiemi di funzionalità. - Raccogliere e interpretare le caratteristiche rilevate dal software, come mostrato nella Figura 3.
Nota: La tabella nella finestra di sinistra mostra i sottoinsiemi di funzionalità rilevate e le loro misurazioni delle prestazioni. I nomi delle prime tre colonne sono "F1", "F2" e "F3". Le tre caratteristiche in ogni sottoinsieme di funzionalità sono date nel loro ordine di classifica in una riga (F1 < F2 < F3). L'ultima colonna dà la misura di prestazioni (Acc o bAcc) di ogni sottoinsieme di funzionalità, e il nome della colonna (Acc o bAcc) è il valore di pMeasurement.
7. interpretare Scatter 3D trame-visualizzare e interpretare i sottoinsiemi di funzionalità con performance di classificazione binaria similmente efficaci utilizzando grafici a dispersione 3D
- Fare clic sul pulsante Analyze per generare il grafico a dispersione 3D dei sottoinsiemi 10 ampiamenti con le migliori performance di classificazione (Acc o bAcc) rilevato dal software, come mostrato in Figura 3 (casella centrale). Ordinare le tre funzioni in un sottoinsieme di funzionalità in ordine crescente di loro ranghi e utilizzare i ranghi delle tre caratteristiche come gli assi di F1/F2/F3, vale a dire, F1 < F2 < F3.
Nota: Il colore di un puntino rappresenta le prestazioni di classificazione binaria del sottoinsieme di funzionalità corrispondente. Un set di dati può avere più sottoinsiemi di funzionalità con allo stesso modo le misurazioni delle prestazioni efficaci. Pertanto, occorre un grafico a dispersione interattiva e semplificata. - Modificare il valore di 0,70 nella casella di input pCutoff: e fare clic sul pulsante Analyze per generare il grafico a dispersione 3D dei sottoinsiemi caratteristica con prestazioni misura ≥ piCutoff, come si vede in Figura 3 (casella a destra). Fare clic sul pulsante 3D tuning per aprire una nuova finestra per sintonizzare manualmente gli angoli di visualizzazione del grafico a dispersione 3D.
Nota: Ogni sottoinsieme di funzionalità è rappresentata da un punto nello stesso modo come sopra. Il grafico a dispersione 3D è stata generata nell'angolo di default. Per facilitare la visualizzazione 3D e tuning, una finestra separata si aprirà facendo clic sul pulsante 3D tuning. - Fare clic sul pulsante Riduci per ridurre la ridondanza dei sottoinsiemi caratteristica rilevata.
Nota: Se gli utenti desiderano ulteriormente selezionare le triplette di funzionalità e ridurre al minimo la ridondanza dei sottoinsiemi caratteristica, il software fornisce anche questa funzione utilizzando l'algoritmo di selezione funzionalità mRMR. Dopo aver cliccato il pulsante Riduci , kSolutionVis rimuoverà quelle caratteristiche ridondanti in terzine la caratteristica e rigenerare la tabella e le due vincenti piazzole di cui sopra. La funzionalità rimosse di terzine caratteristica verrà sostituita dalla parola chiave nella tabella. I valori None nell'asse F1/F2/F3 verranno indicati come valore della piFSNum (l'intervallo del valore normale di F1/F2/F3 è [1, top_x]). Di conseguenza, i puntini che includono un valore None sembra essere terreni puntini "outlier" in 3D. Le trame 3D manualmente sintonizzabile potrebbero trovarsi in "Sintonizzazione manuale delle trame 3D dot" nel materiale supplementare.
8. trovare Gene le annotazioni e le loro associazioni con malattie umane
Nota: I passaggi da 8 a 10 illustrerà come annotare un gene dal livello di sequenza di DNA e di proteine. In primo luogo, la sigla del gene di ogni ID di biomarcatore dei passaggi precedenti verrà recuperato dal database DAVID32, e poi due rappresentative web server saranno usati per analizzare questa sigla del gene dai livelli di DNA e proteine, rispettivamente. Il server GeneCard fornisce un'annotazione funzionale completa di un simbolo dato gene e l'eredità mendeliana in linea nel database di uomo (OMIM) fornisce il curation più completo delle associazioni del gene-malattia. Il server UniProtKB è uno del più completo database di proteine, e il server basato su gruppo Prediction System (GPS) predice la segnalazione fosforilazione per una lista molto ampia di chinasi.
- Copiare e incollare il collegamento web del database DAVID in un browser web e aprire la pagina web di questo database. Clicca sul link Gene ID conversione visto in Figura 4A e ingresso la funzionalità IDs 38319_at/38147_at/33238_at del primo sottoinsieme biomarcatore del dataset ALL1 (Figura 4B). Clicca sul link Gene elenco e fare clic su Elenco di presentare come mostrato in Figura 4B. Recuperare le annotazioni di interesse e fare clic su Visualizza elenco di Gene (Figura 4). Ottenere l'elenco dei simboli di gene (Figura 4).
Nota: I simboli di gene estratto qui verranno essere utilizzati per ulteriori annotazioni funzionali nei passaggi successivi. - Copiare e incollare il collegamento web del database Gene carte in un browser web e aprire la pagina web di questo database. Nome di un gene CD3D di ricerca nella casella di input di query di database e trovare le annotazioni di questo gene da Gene carte33,34, come mostrato in tabella 1 e Figura 5A.
Nota: Carte di Gene è un gene completa knowledgebase, fornendo nomenclatura, genomica, proteomica, localizzazione subcellulare e vie coinvolte e altri moduli funzionali. Fornisce inoltre collegamenti esterni a varie altre banche dati biomediche come PDB/PDB_REDO35, Entrez Gene36, OMIM37e UniProtKB38. Se il nome della caratteristica non è un simbolo standard del gene, è possibile utilizzare il database di ENSEMBL per convertirlo39. CD3D è il nome del gene della catena Delta T-Cell Receptor T3. - Copiare e incollare il collegamento web del database OMIM in un browser web e aprire la pagina web di questo database. Cerca il nome di un gene CD3D e trovare le annotazioni di questo gene dal database OMIM37, come mostrato in tabella 1 e figura 5B.
Nota: OMIM ora serve come una delle fonti più autorevole ed esaustivo delle connessioni del gene umano con malattie ereditarie. OMIM è stata iniziata da Dr. Victor McKusick A. per catalogare le mutazioni genetiche associate a malattia40. OMIM ora copre oltre 15.000 geni umani e oltre 8.500 fenotipi, a partire da dicembre 1st 2017.
9. annotare le proteine codificate e le modifiche post-traduzionali
- Copiare e incollare il collegamento web del database UniProtKB in un browser web e aprire la pagina web di questo database. Nome di un gene CD3D di ricerca nella casella di input di query di UniProtKB e trovare le annotazioni di questo gene dal database38, come mostrato in tabella 1 e nella figura 5.
Nota: UniProtKB raccoglie una ricca fonte di annotazioni per le proteine, tra cui nomenclatura sia informazioni funzionali. Questo database fornisce anche link esterni ad altri database utilizzati, inclusi PDB/PDB_REDO35, OMIM37e Pfam41. - Copiare e incollare il collegamento web del server web GPS in un browser web e aprire la pagina web di questo server web. Recuperare la sequenza della proteina codificata dal gene biomarcatore CD3D dal database UniProtKB38 e prevedere residui di modificazione post-traduzionale (PTM) della proteina utilizzando il tool online GPS, come mostrato in tabella 1 e nella figura 5.
Nota: Un sistema biologico è dinamico e complicati, e i database esistenti raccolgono solo le informazioni conosciute. Pertanto, strumenti online Pronostico biomedica, nonché programmi offline possono fornire elementi utili per integrare un meccanismo supposto. GPS è stato sviluppato e migliorato per oltre 12 anni7,42 e può essere usato per predire residui PTM di una proteina in un determinato peptide sequenza43,44. A disposizione anche strumenti per vari temi di ricerca, tra cui la previsione di una proteina subcellulare posizione45 e trascrizione fattore vincolante motivi 46 tra gli altri.
10. annotazione di interazioni proteina-proteina e loro moduli funzionali arricchiti
- Copiare e incollare il collegamento web del server web stringa in un browser web e aprire la pagina web di questo server web. Ricerca nell'elenco per i geni CD3D e P53 e trovare loro immobili orchestrate utilizzando il database String47. La stessa procedura può essere effettuata utilizzando un altro server web, DAVID32.
Nota: Oltre le suddette annotazioni per singoli geni, ci sono molti strumenti informatici su larga scala disponibili per studiare le proprietà di un gruppo di geni. Un recente studio ha dimostrato che i geni marcatori individualmente male potrebbero costituire un gene molto migliorato set5. Pertanto, vale la pena il costo computazionale per schermo per biomarcatori più complicati. Il database String può visualizzare le connessioni di interazione noto o previsto, e il server di David potrebbe rilevare i moduli funzionali con fenotipo-associazioni significative nei geni interrogato47,32. A disposizione anche vari altri strumenti di analisi informatica su larga scala.
11. esportare i sottoinsiemi di biomarcatore generato e le trame di visualizzazione
- Esportare i sottoinsiemi di biomarcatore rilevato come un file di testo di TSV o CSV per ulteriori analisi. Fare clic sul pulsante Esporta la tabella sotto il tavolo di tutti i sottoinsiemi di biomarcatore rilevati e scegliere il formato di testo per salvare come.
- Esportare i grafici di visualizzazione come file di immagine. Fare clic sul pulsante Salva sotto ogni trama e scegliere il formato di immagine per salvare come.
Nota: Il software supporta il pixel formato. png e il vettore formato. svg. Le immagini di pixel sono buone per la visualizzazione sullo schermo del computer, mentre le immagini vettoriali possono essere convertite in qualsiasi risoluzione richiesta a fini di pubblicazione journal.
Risultati
L'obiettivo di questo flusso di lavoro (Figura 6) è quello di rilevare più sottoinsiemi di biomarcatore con efficienze simili per un set di dati di classificazione binaria. L'intero processo è illustrato da due set di dati esempio ALL1 e ALL2 estratte da un rilevamento di biomarcatore recentemente pubblicato lo Studio12,48. Un utente può installare kSolutionVis seguendo le istruzioni riportate nei...
Discussione
Questo studio presenta un protocollo di individuazione e caratterizzazione di biomarcatore multi-soluzione facile da seguire per un set di dati di classificazione binaria specificata dall'utente. Il software mette l'accento sulla facilità d'uso e interfacce flessibili di importazione/esportazione per vari formati di file, permettendo un ricercatore biomedico indagare il loro set di dati facilmente utilizzando la GUI del software. Questo studio evidenzia anche la necessità di generare più di una soluzione con prestazio...
Divulgazioni
Non abbiamo conflitti di interesse relativi alla presente relazione.
Riconoscimenti
Questo lavoro è stato supportato dal programma di ricerca priorità strategica dell'Accademia cinese delle scienze (XDB13040400) e la concessione di avvio dalla Università di Jilin. Utenti anonimi e utenti test biomedici sono stati apprezzati per i loro commenti costruttivi per migliorare l'usabilità e la funzionalità di kSolutionVis.
Materiali
| Name | Company | Catalog Number | Comments |
| Hardware | |||
| laptop | Lenovo | X1 carbon | Any computer works. Recommended minimum configuration: 1GB extra hard disk space, 1 GB memory, 2.0MHz CPU |
| Name | Company | Catalog Number | Comments |
| Software | |||
| Python 3.0 | WingWare | Wing Personal | Any python programming and running environments support Python version 3.0 or above |
Riferimenti
- Heckerman, D., et al. Genetic variants associated with physical performance and anthropometry in old age: a genome-wide association study in the ilSIRENTE cohort. Scientific Reports. 7, 15879 (2017).
- Li, Z., et al. Genome-wide association analysis identifies 30 new susceptibility loci for schizophrenia. Nature Genetics. 49, 1576-1583 (2017).
- Winkler, T. W., et al. Quality control and conduct of genome-wide association meta-analyses. Nature Protocols. 9, 1192-1212 (2014).
- Harrison, R. N. S., et al. Development of multivariable models to predict change in Body Mass Index within a clinical trial population of psychotic individuals. Scientific Reports. 7, 14738 (2017).
- Liu, J., et al. Multiple similarly-well solutions exist for biomedical feature selection and classification problems. Scientific Reports. 7, 12830 (2017).
- Ye, Y., Zhang, R., Zheng, W., Liu, S., Zhou, F. RIFS: a randomly restarted incremental feature selection algorithm. Scientific Reports. 7, 13013 (2017).
- Zhou, F. F., Xue, Y., Chen, G. L., Yao, X. GPS: a novel group-based phosphorylation predicting and scoring method. Biochemical and Biophysical Research Communications. 325, 1443-1448 (2004).
- Sanchez, B. N., Wu, M., Song, P. X., Wang, W. Study design in high-dimensional classification analysis. Biostatistics. 17, 722-736 (2016).
- Shujie, M. A., Carroll, R. J., Liang, H., Xu, S. Estimation and Inference in Generalized Additive Coefficient Models for Nonlinear Interactions with High-Dimensional Covariates. Annals of Statistics. 43, 2102-2131 (2015).
- Li, J. H., et al. MiR-205 as a promising biomarker in the diagnosis and prognosis of lung cancer. Oncotarget. 8, 91938-91949 (2017).
- Lyskjaer, I., Rasmussen, M. H., Andersen, C. L. Putting a brake on stress signaling: miR-625-3p as a biomarker for choice of therapy in colorectal cancer. Epigenomics. 8, 1449-1452 (2016).
- Ge, R., et al. McTwo: a two-step feature selection algorithm based on maximal information coefficient. BMC Bioinformatics. 17, 142 (2016).
- Tumuluru, J. S., McCulloch, R. Application of Hybrid Genetic Algorithm Routine in Optimizing Food and Bioengineering Processes. Foods. 5, (2016).
- Gen, M., Cheng, R., Lin, L. . Network models and optimization: Multiobjective genetic algorithm approach. , (2008).
- Radovic, M., Ghalwash, M., Filipovic, N., Obradovic, Z. Minimum redundancy maximum relevance feature selection approach for temporal gene expression data. BMC Bioinformatics. 18, 9 (2017).
- Ciuculete, D. M., et al. A methylome-wide mQTL analysis reveals associations of methylation sites with GAD1 and HDAC3 SNPs and a general psychiatric risk score. Translational Psychiatry. 7, e1002 (2017).
- Lin, H., et al. Methylome-wide Association Study of Atrial Fibrillation in Framingham Heart Study. Scientific Reports. 7, 40377 (2017).
- Wang, S., Li, J., Yuan, F., Huang, T., Cai, Y. D. Computational method for distinguishing lysine acetylation, sumoylation, and ubiquitination using the random forest algorithm with a feature selection procedure. combinatorial chemistry & high throughput screening. , (2017).
- Zhang, Q., et al. Predicting Citrullination Sites in Protein Sequences Using mRMR Method and Random Forest Algorithm. combinatorial chemistry & high throughput screening. 20, 164-173 (2017).
- Cuena-Lombrana, A., Fois, M., Fenu, G., Cogoni, D., Bacchetta, G. The impact of climatic variations on the reproductive success of Gentiana lutea L. in a Mediterranean mountain area. International journal of biometeorology. , (2018).
- Coghe, G., et al. Fatigue, as measured using the Modified Fatigue Impact Scale, is a predictor of processing speed improvement induced by exercise in patients with multiple sclerosis: data from a randomized controlled trial. Journal of Neurology. , (2018).
- Hong, H., et al. Applying genetic algorithms to set the optimal combination of forest fire related variables and model forest fire susceptibility based on data mining models. The case of Dayu County, China. Science of the Total Environment. 630, 1044-1056 (2018).
- Borges, D. L., et al. Photoanthropometric face iridial proportions for age estimation: An investigation using features selected via a joint mutual information criterion. Forensic Science International. 284, 9-14 (2018).
- Kohavi, R., John, G. H. Wrappers for feature subset selection. Artificial intelligence. 97, 273-324 (1997).
- Yu, L., Liu, H. Efficient feature selection via analysis of relevance and redundancy. Journal of machine learning research. 5, 1205-1224 (2004).
- Wexler, R. B., Martirez, J. M. P., Rappe, A. M. Chemical Pressure-Driven Enhancement of the Hydrogen Evolving Activity of Ni2P from Nonmetal Surface Doping Interpreted via Machine Learning. Journal of American Chemical Society. , (2018).
- Wijaya, S. H., Batubara, I., Nishioka, T., Altaf-Ul-Amin, M., Kanaya, S. Metabolomic Studies of Indonesian Jamu Medicines: Prediction of Jamu Efficacy and Identification of Important Metabolites. Molecular Informatics. 36, (2017).
- Shangkuan, W. C., et al. Risk analysis of colorectal cancer incidence by gene expression analysis. PeerJ. 5, e3003 (2017).
- Chu, C. M., et al. Gene expression profiling of colorectal tumors and normal mucosa by microarrays meta-analysis using prediction analysis of microarray, artificial neural network, classification, and regression trees. Disease Markers. , 634123 (2014).
- Fleuret, F. Fast binary feature selection with conditional mutual information. Journal of Machine Learning Research. 5, 1531-1555 (2004).
- Pacheco, J., Alfaro, E., Casado, S., Gámez, M., García, N. A GRASP method for building classification trees. Expert Systems with Applications. 39, 3241-3248 (2012).
- Jiao, X., et al. DAVID-WS: a stateful web service to facilitate gene/protein list analysis. Bioinformatics. 28, 1805-1806 (2012).
- Rappaport, N., et al. Rational confederation of genes and diseases: NGS interpretation via GeneCards, MalaCards and VarElect. Biomedical Engineering OnLine. 16, 72 (2017).
- Rebhan, M., Chalifa-Caspi, V., Prilusky, J., Lancet, D. GeneCards: integrating information about genes, proteins and diseases. Trends in Genet. 13, 163 (1997).
- Joosten, R. P., Long, F., Murshudov, G. N., Perrakis, A. The PDB_REDO server for macromolecular structure model optimization. IUCrJ. 1, 213-220 (2014).
- Maglott, D., Ostell, J., Pruitt, K. D., Tatusova, T. Entrez Gene: gene-centered information at NCBI. Nucleic Acids Research. 39, D52-D57 (2011).
- Amberger, J. S., Bocchini, C. A., Schiettecatte, F., Scott, A. F., Hamosh, A. OMIM.org: Online Mendelian Inheritance in Man (OMIM(R)), an online catalog of human genes and genetic disorders. Nucleic Acids Research. 43, D789-D798 (2015).
- Boutet, E., et al. the Manually Annotated Section of the UniProt KnowledgeBase: How to Use the Entry View. Methods in Molecular Biology. 1374, 23-54 (2016).
- Zerbino, D. R., et al. Ensembl 2018. Nucleic Acids Res. , (2017).
- McKusick, V. A., Amberger, J. S. The morbid anatomy of the human genome: chromosomal location of mutations causing disease. Journal of Medical Genetics. 30, 1-26 (1993).
- Finn, R. D., et al. The Pfam protein families database: towards a more sustainable future. Nucleic Acids Research. 44, D279-D285 (2016).
- Xue, Y., et al. GPS: a comprehensive www server for phosphorylation sites prediction. Nucleic Acids Research. 33, W184-W187 (2005).
- Deng, W., et al. GPS-PAIL: prediction of lysine acetyltransferase-specific modification sites from protein sequences. Scientific Reports. 6, 39787 (2016).
- Zhao, Q., et al. GPS-SUMO: a tool for the prediction of sumoylation sites and SUMO-interaction motifs. Nucleic Acids Research. 42, W325-W330 (2014).
- Wan, S., Duan, Y., Zou, Q. HPSLPred: An Ensemble Multi-Label Classifier for Human Protein Subcellular Location Prediction with Imbalanced Source. Proteomics. 17, (2017).
- Zhang, H., Zhu, L., Huang, D. S. WSMD: weakly-supervised motif discovery in transcription factor ChIP-seq data. Scientific Reports. 7, 3217 (2017).
- Szklarczyk, D., et al. STRING v10: protein-protein interaction networks, integrated over the tree of life. Nucleic Acids Research. 43, D447-D452 (2015).
- Chiaretti, S., et al. Gene expression profile of adult T-cell acute lymphocytic leukemia identifies distinct subsets of patients with different response to therapy and survival. Blood. 103, 2771-2778 (2004).
- Rowley, J. D., et al. Mapping chromosome band 11q23 in human acute leukemia with biotinylated probes: identification of 11q23 translocation breakpoints with a yeast artificial chromosome. Proceedings of the National Academy of Sciences of the United States of America. 87, 9358-9362 (1990).
- Rabbitts, T. H., et al. The chromosomal location of T-cell receptor genes and a T cell rearranging gene: possible correlation with specific translocations in human T cell leukaemia. Embo Journal. 4, 1461-1465 (1985).
- Yin, L., et al. SH2D1A mutation analysis for diagnosis of XLP in typical and atypical patients. Human Genetics. 105, 501-505 (1999).
- Brandau, O., et al. Epstein-Barr virus-negative boys with non-Hodgkin lymphoma are mutated in the SH2D1A gene, as are patients with X-linked lymphoproliferative disease (XLP). Human Molecular Genetics. 8, 2407-2413 (1999).
- Burnett, R. C., Thirman, M. J., Rowley, J. D., Diaz, M. O. Molecular analysis of the T-cell acute lymphoblastic leukemia-associated t(1;7)(p34;q34) that fuses LCK and TCRB. Blood. 84, 1232-1236 (1994).
- Taylor, G. M., et al. Genetic susceptibility to childhood common acute lymphoblastic leukaemia is associated with polymorphic peptide-binding pocket profiles in HLA-DPB1*0201. Human Molecular Genetics. 11, 1585-1597 (2002).
- Wadia, P. P., et al. Antibodies specifically target AML antigen NuSAP1 after allogeneic bone marrow transplantation. Blood. 115, 2077-2087 (2010).
- Wilson, D. M., et al. 3rd et al. Hex1: a new human Rad2 nuclease family member with homology to yeast exonuclease 1. Nucleic Acids Research. 26, 3762-3768 (1998).
- O'Sullivan, R. J., et al. Rapid induction of alternative lengthening of telomeres by depletion of the histone chaperone ASF1. Nature Structural & Molecular Biology. 21, 167-174 (2014).
- Lee-Sherick, A. B., et al. Aberrant Mer receptor tyrosine kinase expression contributes to leukemogenesis in acute myeloid leukemia. Oncogene. 32, 5359-5368 (2013).
- Guyon, I., Elisseeff, A. An introduction to variable and feature selection. Journal of machine learning research. 3, 1157-1182 (2003).
- John, G. H., Kohavi, R., Pfleger, K. . Machine learning: proceedings of the eleventh international conference. , 121-129 (1994).
- Jain, A., Zongker, D. Feature selection: Evaluation, application, and small sample performance. IEEE transactions on pattern analysis and machine intelligence. 19, 153-158 (1997).
- Taylor, S. L., Kim, K. A jackknife and voting classifier approach to feature selection and classification. Cancer Informatics. 10, 133-147 (2011).
- Andresen, K., et al. Novel target genes and a valid biomarker panel identified for cholangiocarcinoma. Epigenetics. 7, 1249-1257 (2012).
- Guo, P., et al. Gene expression profile based classification models of psoriasis. Genomics. 103, 48-55 (2014).
- Xie, J., Wang, C. Using support vector machines with a novel hybrid feature selection method for diagnosis of erythemato-squamous diseases. Expert Systems with Applications. 38, 5809-5815 (2011).
- Zou, Q., Zeng, J., Cao, L., Ji, R. A novel features ranking metric with application to scalable visual and bioinformatics data classification. Neurocomputing. 173, 346-354 (2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon