È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
In Situ Microscopia per la determinazione in tempo reale della morfologia a singola cellula nei bioprocessi
In questo articolo
Riepilogo
È stato sviluppato un dispositivo di microscopia foto-ottica in situ per monitorare le dimensioni delle singole cellule direttamente nella sospensione cellulare. La misurazione in tempo reale viene effettuata accolando la sonda sterilizzabile fotoottica a un'analisi automatizzata delle immagini. I cambiamenti morfologici compaiono con dipendenza dallo stato di crescita e dalle condizioni di coltivazione.
Abstract
Il monitoraggio in situ nei bioprocessi microbici è per lo più limitato alle proprietà chimiche e fisiche del mezzo(ad esempio,il valore di pH e la concentrazione di ossigeno disciolto). Tuttavia, la morfologia delle cellule può essere un indicatore adatto per condizioni ottimali, poiché cambia con la dipendenza dallo stato di crescita, l'accumulo di prodotto e lo stress cellulare. Inoltre, la distribuzione delle dimensioni delle singole cellule fornisce non solo informazioni sulle condizioni di coltivazione, ma anche sull'eterogeneità della popolazione. Per ottenere tali informazioni, è stato sviluppato un dispositivo di microscopia in situ foto-ottico1 per consentire il monitoraggio della distribuzione delle dimensioni delle singole cellule direttamente nelle sospensioni cellulari nei bioreattori. Un'analisi automatizzata delle immagini viene accoppiata alla microscopia basata su un modello di rete neurale, sottoposto a training con immagini con annotato dall'utente. Diversi parametri, che si ottengono dalle catture del microscopio, sono correlati per elaborare le caratteristiche rilevanti delle cellule, come la loro attività metabolica. Fino ad ora, la serie di sonde di microscopia in situ è stata applicata per misurare la dimensione del pellet nelle sospensioni filamentose dei funghi. È stato utilizzato per distinguere le dimensioni delle singole cellule nella coltivazione delle microalghe e metterle in relazione con l'accumulo di lipidi. La forma delle particelle cellulari era correlata al germogliamento nelle colture di lievito. L'analisi della microscopia può essere generalmente suddivisa in tre fasi: (i) acquisizione di immagini, (ii) identificazione delle particelle e (iii) analisi dei dati, rispettivamente. Tutte le misure devono essere adattate all'organismo, e quindi sono necessarie informazioni specifiche con annotato per ottenere risultati affidabili. La capacità di monitorare i cambiamenti nella morfologia cellulare direttamente in linea o on line (in un by-pass) consente valori in tempo reale per il monitoraggio e il controllo, nello sviluppo dei processi e nella scala di produzione. Se i dati off line sono correlati ai dati in tempo reale, le misurazioni off line correnti con influenze sconosciute sulla dimensione della cellula diventano inutili.
Introduzione
Le caratteristiche morfologiche delle cellule sono spesso legate allo stato fisiologico, esiste una connessione tra forma e funzione per molte applicazioni. La morfologia di una singola cellula è influenzata dallo stato di crescita, dall'età della cellula, dalle sollecitazioni osmotiche e da altre potenziali sollecitazioni cellulari o dall'accumulo di prodotti. I cambiamenti morfologici delle cellule sono spesso una misura della vitalità di crescita di una cultura. Sintesi del prodotto intracellulare, accumulo di lipidi nelle alghe e formazione del corpo di inclusione nei batteri, tra gli altri, sono correlati con la dimensione della cellula pure. L'agglomerazione cellulare può essere un altro fattore che vale la pena indagare come riassunto di recente2.
Le eterogeneità della popolazione possono essere quantificate sulla base delle caratteristiche morfologiche delle singole cellule. Gli studi hanno dimostrato che l'eterogeneità all'interno di una cultura potrebbe essere significativa, ad esempio, in condizioni di produzione su larga scala3 la resa complessiva potrebbe essere influenzata da una bassa performance delle sottopopolazioni4.
Di solito, la valutazione delle caratteristiche morfologiche delle cellule viene eseguita mediante campionamento manuale o con una camera di flusso by-pass accoppiata a un dispositivo foto-ottico. Questo porta a diverse restrizioni: la quantità limitata di dati acquisiti difficilmente può fornire misurazioni statisticamente affidabili; il ritardo tra il campionamento e l'accessibilità dei risultati può essere troppo lungo rispetto alle dinamiche del processo; e, cosa più importante, la procedura di campionamento (posizione della porta di campionamento, pretrattamento del campione prima della misurazione, condizioni sfavorevoli nel tubo di campionamento o bypass) può innescare un errore di parte in quanto la procedura di campionamento stessa può già influenzare la cellula Morfologia. Infine, esiste sempre un alto rischio di contaminazione durante il campionamento o nelle soluzioni by-pass, se non sono sterilizzabili in posizione.
L'applicazione della microscopia in situ (ISM) può aggirare molti di questi problemi. Se le cellule vengono rilevate automaticamente, è possibile esaminare una corretta identificazione delle loro caratteristiche morfologiche5. Fino ad ora, i principali limiti di questo metodo erano (i) il tempo di valutazione delle immagini, che era troppo lungo per le applicazioni in situ, e (ii) la scarsa risoluzione delle immagini, soprattutto ad alta densità di cellule. Sebbene le prime soluzioni di ISM includessero il campionamento meccanico, la diluizione della sonda o fossero limitate a un sistema by-pass6,7, ulteriori approcci consentono l'acquisizione direttamente della sospensione cellulare8.
I recenti progressi nell'ISM consentono il monitoraggio in linea o on line delle cellule su base a singola cellula, che fornisce la distribuzione dei parametri morfologici in tempo reale direttamente nelle sospensioni cellulari a concentrazioni cellulari notevolmente elevate. Attraverso analisi off-line dei parametri chiave delle cellule, è possibile identificare le correlazioni con le informazioni fornite dal rilevamento automatico delle cellule accoppiato e dall'ISM. Quindi, vengono ottenuti nuovi progetti di sensori morbidi, in cui un parametro non misurabile viene stimato con la morfologia a cella singola.
In questo rapporto, l'ISM è condotto acunmando una sonda foto-ottica a un'analisi automatizzata delle immagini. L'ISM è costituito da una sonda sensore a singola asta che consente l'acquisizione di immagini all'interno di un intervallo di messa a fuoco noto in un gap di misura regolabile con una telecamera CCD ad alta risoluzione [MM-Ho - CCD GT2750 (2750x2200) e MM 2.1 - CMOS G507c (2464x2056)]. L'illuminazione della luce flash avviene tramite trasmissione. Pertanto, la luce proviene dal lato opposto della fotocamera9 e la sua intensità può essere regolata. Le cellule passano continuamente attraverso questo divario con il flusso di liquido. Di conseguenza, si ottiene una popolazione campione rappresentativa. La sonda può essere montata direttamente al bioreattore in modo che raggiunga la sospensione cellulare, oppure può essere utilizzata in un by-pass sterilizzabile. Il guscio del sensore è collegato al sistema prima della sterilizzazione, le parti ottiche vengono successivamente montate nel guscio.
Fino ad ora, i microrganismi industriali rilevanti, ad esempioi funghi filamentosi (diametro fino a oltre 200 m), le microalghe eterotrofiche Criptodinium cohnii (diametro medio delle cellule di 20 m), e il lievito Saccharomyces cerevisiae (diametro medio delle cellule di 5 m), sono stati studiati con questo o dispositivi simili, che è brevemente descritto.
I funghi filamentosi tendono a formare pellet in determinate condizioni di coltivazione. Questi sono di una dimensione di diverse centinaia di m. Le ife delle cellule fungine sviluppano diverse lunghezze di dipendenza dallo stress idrodinamico nella fase fluida. Questo ha un'influenza sull'attività metabolica e di crescita, l'assorbimento del substrato e il rilascio del prodotto. ISM è stato applicato per identificare la distribuzione delle dimensioni del pellet e la larghezza delle zone di minore densità di biomassa ai bordi dei pellet (dati inediti).
La dimensione di C. cohnii cambia tra 15 e 26 m quando le cellule accumulano l'acido grasso polinsaturico docosaexaenoico acido (DHA) sotto limiti di azoto. Questo processo di produzione di DHA biotecnologico è costituito da due parti, la fase di crescita, in cui le cellule si dividono e diventano più piccole, e la fase di produzione, in cui le cellule accumulano il prodotto e quindi diventano più grandi. Pertanto, la dimensione della cella è stata utilizzata per determinare lo stato del processo, in cui la crescita o la produzione di DHA era favorevole. Infine, è stata trovata una correlazione tra la dimensione della cella e il contenuto DHA. In questo caso, ISM permette di monitorare l'accumulo di DHA intracellulare in tempo reale senza il requisito di campionamento, interruzione delle cellule, e l'analisi cromatografia gas comune10.
Il lievito di busparazione è di solito di dimensioni comprese tra 3 e 8 m. La percentuale di cellule che sono nello stato di maturazione alla volta, come descritto con l'indice in erba (BI), fornisce informazioni sulla vitalità della crescita11,12, e anche una relazione con la secrezione proteica ricombinante è stata dimostrata13. Con l'aiuto di ISM, le cellule di lievito in erba e non in erba (cellule con e senza gemme) si sono distinte14. Le condizioni di stress possono anche portare a una variazione più ampia delle dimensioni delle cellule all'interno di una popolazione di lieviti, come recentemente mostrato nelle coltivazioni scale-down, in cui le condizioni di coltivazioni alimentate con limitato nutrimento su larga scala sono state imitate3.
Pertanto, ISM ha il potenziale per monitorare la vitalità della crescita e la formazione del prodotto a livello di singola cellula durante tutte le fasi di un bioprocesso per l'identificazione di condizioni di coltivazione ottimali o ai fini del controllo dei processi. I metodi qui descritti si concentrano sulle applicazioni microbiche con singole cellule, ma sono applicabili anche a particelle più grandi come cellule umane e animali, agglomerati cellulari e pellet di organismi mentosi.
Protocollo
NOTA: I seguenti passaggi sono necessari per adattare i parametri alle rispettive condizioni di microrganismo e coltura. La regolazione delle impostazioni della sonda dura circa 20 min per un utente esperto. Una descrizione dettagliata degli strumenti e dei passaggi è fornita nel manuale della sonda corrispondente di SOPAT GmbH. In generale, gli strumenti che sono presentati nel seguente protocollo sono necessari: (i) Probe Controller per le regolazioni probe e l'acquisizione dell'immagine; (ii) Figi (ImageJ) per le annotazioni sulle immagini acquisite; (iii) Supporto SOPAT per la formazione della rete neurale artificiale (ANN) e la creazione di flussi di lavoro; (iv) Batcher per l'elaborazione batch di dati utilizzando immagini già acquisite con un flusso di lavoro; (v) Analizzatore dei risultati per la visualizzazione dei risultati e la valutazione delle immagini elaborate in batch; e (vi) Monitorare la misurazione automatizzata in tempo reale e la visualizzazione dei risultati.
1. Impostazione dei parametri hardware
- Preparare una coltura con la più alta concentrazione cellulare che potrebbe essere raggiunta durante l'esperimento o centrifugare e rispendere il pellet al fine di raggiungere questa concentrazione. In questo caso, sono stati scelti 65 g di L-1 di concentrazione di biomassa secca per le coltivazioni S. cerevisiae.
- Preparare diverse diluizioni, che vanno dalla più alta alla più bassa concentrazione in modo che l'intervallo previsto sia completamente coperto. Si raccomandano un minimo di 4 diverse concentrazioni.
- Identificare l'intervallo di dimensioni delle cellule del microrganismo con la microscopia convenzionale. Definire il diametro massimo previsto (dmax) delle rispettive celle. Questo valore è impostato su 8 m in caso di S. cerevisiae.
- Scegliere due spazi di misura di 5x e 10x del previsto dmax delle celle.
- Scegliere la massima intensità stroboscopico. Scegliere le intensità dello stroboscopio per entrambe le lacune con la più alta concentrazione di cellule in modo che le celle siano ancora visibili sulle immagini con l'intensità di luce più bassa (immagini più scure).
- Scegliere l'intensità minima stroboscopico, quindi scegliere le intensità stroboscopico per entrambi gli spazi in modo che le celle siano ancora visibili sulle immagini con la massima intensità di luce (immagini più luminose). Utilizzare la concentrazione cellulare più bassa, che probabilmente appare durante il periodo di misurazione.
- Scegliere una posizione di messa a fuoco, che restituisce le immagini più nitide per ogni spazio di misurazione, sia per le intensità stroboscopico che per l'intervallo di concentrazione che devono essere testati (vedere il passaggio 2 per i dettagli sulla messa a fuoco). Mettere a fuoco le celle in modo appropriato in modo che i dati dell'immagine possano essere annotati in un secondo momento (vedere il passaggio 4).
- Misurare la serie di diluizione precedentemente preparata della concentrazione cellulare (vedi passo 2) con le larghezze del divario e le intensità dello stroboscopio.
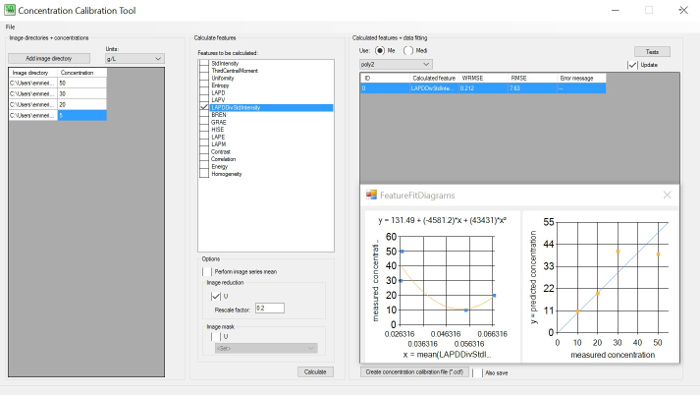
Figura 1: Strumento di calibrazione della concentrazione. GUI sinistra: impostare le directory delle immagini (minimo 3) con concentrazioni note; INTERFACCIA grafica centrale: scegliere le funzioni da calcolare sulla directory delle immagini; GUID a destra: scegliere l'errore quadrato medio radice ponderato (WRMSE) per identificare il minimo. WRMSE e la migliore correlazione tra qualsiasi caratteristica dell'immagine e la concentrazione cellulare. Fare clic qui per visualizzare una versione più grande di questa figura.
- Valutare l'esperimento della serie di diluizione.
- Utilizzare lo strumento Concentration Calibration (CoCa) (vedere la figura 1) per identificare la correlazione ottimale tra le caratteristiche dell'immagine estratta (luminosità o nitidezza) e le concentrazioni precedentemente misurate fornite dall'utente, ad esempiola biomassa secca o il numero di cellule. Seguire le istruzioni nel manuale del software per ulteriori dettagli.
- Identificare la correlazione ottimale tra le informazioni estratte dalle caratteristiche dell'immagine a varie concentrazioni rispetto a qualsiasi misurazione off line. Vedere la legenda della figura 1.
- Scegliere il divario di misurazione più ragionevole e le intensità dello stroboscopio in considerazione della curva di correlazione della concentrazione con le caratteristiche che si traducono nel più piccolo errore quadrato media radice ponderata (WRMSE).
NOTA: Il divario di misurazione viene fissato durante l'esperimento, mentre l'intensità dello stroboscopio può essere adattata in base alla concentrazione cellulare.
2. Misurazione fuori linea
- Regolare lo spazio di misurazione desiderato in base al punto 1 con l'aiuto di un misuratore di spessore.
- Aprire l'interfaccia utente grafica Probe Control nel dashboard SOPAT.
- Collegare la sonda desiderata all'amplificatore nella sottosezione software Azioni e premere Connetti.
- Premere il pulsante Riproduci per avviare lo streaming (Live View).
- Pulire il divario di misura spruzzando etanolo nello spazio e pulire con attenzione qualsiasi polvere o sporco con una carta ottica. Verificare che il vetro del sensore sia privo di particelle con la vista Dal vivo nel CamControl.
NOTA: Le particelle e la polvere disturbano le misurazioni e l'identificazione automatica delle celle. - Posizionare una carta ottica asciutta nello spazio di misurazione. Aprire il controllo Sonda scheda e regolare l'intensità stroboscopio per visualizzare la carta. Girare la vite di rilegatura fino a vedere chiaramente le singole fibre della carta.
- Riempire un tubo con brodo di coltura. Immergere il microscopio nel brodo di coltura in modo che il divario sia completamente coperto con sospensione cellulare. Aprire il controllo Sonda scheda e regolare l'intensità stroboscopio desiderata in base alla sezione 1. Concentrarsi sulle celle mettendo a punto la vite di legame della messa a fuoco. La messa a fuoco non deve più essere cambiata durante l'esperimento
NOTA: 5-6 mL di brodo di coltura vengono aggiunti a un tubo centririco conico conico da 50 mL per far galleggiare sufficientemente il divario di misura. - Definire il numero di fotogrammi per punto temporale [-] nell'interfaccia utente nel menu Attivazione nei frame GUI per trigger. Impostate il numero di fotogrammi su 200 fotogrammi per trigger.
NOTA: il numero di fotogrammi può essere ridotto al valore più basso, necessario per un risultato statistico affidabile. Ciò dipende dalla dimensione del campione necessaria per ottenere una distribuzione rappresentativa delle dimensioni morfologiche delle cellule (vedere anche il passaggio 5). - Definire la frequenza fotogrammi [Hz] nel menu Attivazione nella frequenza fotogrammiGUI . Scegliete una frequenza fotogrammi che garantisca che le particelle in movimento da un fotogramma precedente non verranno visualizzate nel fotogramma seguente.
NOTA: Questo può essere dimostrato con un trigger di prova con 200 fotogrammi. Ispezionare le immagini per le particelle, che vengono catturate ripetutamente. In questo caso, diminuire la frequenza fotogrammi. Per le misurazioni off line, si consiglia 1 Hz. - Impostare la directory in cui verranno salvate le immagini acquisite, nel menu Generale.
- Eseguire un'acquisizione dell'immagine attivando il pulsante Avvia acquisizione trigger immagine. Spostare il tubo con sospensione della coltura delicatamente su e giù per indurre un flusso attraverso il divario di misurazione.
- Ripetere il passaggio 2.5 dopo ogni misurazione.
- Controllare le immagini acquisite. Le celle devono essere abbastanza nitide per l'annotazione. Ispezionare le immagini per le particelle, che vengono catturate ripetutamente. In questo caso, diminuire la frequenza fotogrammi.
- Salvare le impostazioni selezionando il percorso seguente: C:
3. Identificazione delle particelle
- Annotare le particelle per la formazione della rete neurale artificiale (ANN) (set di formazione).
- Caricare le immagini acquisite nello strumento di annotazione "Fiji, ImageJ" trascinando e rilasciando il file nella finestra principale "ImageJ" (vedere GUI "Fiji" strumento figura 2)
- Aprire Gestione ROI selezionando: Analizza Proprietà Tools (Strumenti) Gestione ROI.
- Scegliere uno strumento di selezione. Sono consigliate selezioni di bacchetta (tracciamento), ovali o ellittiche.
- Disegnare un cerchio intorno alla particella che deve essere annotato con gli strumenti di selezione menzionati in precedenza e quindi rifinirlo con lo strumento pennello.
- Aggiungere l'annotazione a Gestione ROI premendo Aggiungi [t].
- Contrassegnare tutti gli oggetti di interesse (celle da identificare) su circa 15 immagini.
NOTA: Per coprire tutte le informazioni necessarie, ovvero forme, dimensioni diverse, concentrazione delle celle, luminosità, ecc.,utilizzare cinque immagini dall'inizio, cinque immagini dall'inizio e cinque immagini dalla fine dell'esperimento. - Decidere, se le cellule devono essere classificate in sottoclassi diverse a causa della loro forma(ad esempio,fasi diverse di un ciclo cellulare), o se tutte le celle sono della stessa classe.
- Modificare di conseguenza il nome di ogni particella selezionata. Impostare un nome o un'abbreviazione per ogni classe e un contatore per ogni particella della classe(ad esempio,cell_1, cell_2 e così via). Annotare almeno 50 particelle per classe.
- Non annotare gli oggetti, che non devono essere rilevati, perché non sono rilevanti per il processo, come bolle di gas o altre particelle come componenti multimediali non disciolti.
NOTA: tali eventi non saranno inclusi nella procedura di formazione per l'ANN e saranno considerati come sfondo. - Non annotare le celle sfocate.
- Annotare le immagini nel modo più coerente possibile. In caso di dubbi, è possibile applicare l'etichetta Ignora. Si raccomanda vivamente di non abusare del suo utilizzo, dal momento che l'ANN riconoscerà solo le strutture che sono etichettate.

Figura 2: Interfaccia utente dello strumento Fiji. Viene creato un set di formazione con le immagini con annotato. Viene rappresentata un'annotazione manuale composta da due classi, l'elenco delle particelle con annotazioni viene visualizzato in Gestione ROI. È possibile impostare nomi e colori diversi per classi diverse. Fare clic qui per visualizzare una versione più grande di questa figura.
- Salvare gli oggetti annotati e l'immagine in un formato zip e inviare il file alla rete di formazione, sia tramite caricamento sulla piattaforma o inviando il file zip via e-mail.
NOTA: Di solito, ci vogliono un certo numero di turni di formazione iterativo per identificare le previsioni adeguate degli oggetti classificati sulle immagini. Ogni ciclo di formazione porta a un flusso di lavoro restituito dal programma. - Utilizzare il flusso di lavoro (con estensione wf) con l'algoritmo di riconoscimento degli oggetti addestrati per analizzare le immagini di test con il programma Batch batch batch di batch di dati che può essere avviato nel dashboard.
- Controllare il rilevamento degli oggetti sulle immagini di prova attraverso la quantificazione di eventi falsi positivi e negativi.
- Quantificare la rilevazione di eventi falsi positivi: particelle erroneamente rilevate come cellule, cellule non classificate correttamente e cellule di cui il contorno non è stato ben identificato.
- Quantificare gli eventi falsi negativi (celle non riconosciute come tali).
- Visualizzare i risultati nell'analizzatore dei risultati dello strumento avviando il programma nel dashboard.
- Importare i file dei risultati desiderati con File Importazione di un file o di un file Importa cartelle.
- Visualizzare i risultati per Grafico Creare un grafico nella GUI di Charts.
- Selezionare una delle seguenti opzioni: Grafico di distribuzione, Grafico sensibilità, Caratteristica nel tempo, Caratteristica sui punti di rilievo e Feature contro feature.
NOTA: un manuale relativo all'utilizzo del Analizzatore di risultati viene fornito con il sistema ed è disponibile anche dal supporto. - Se i risultati sono accettabili, eseguire il flusso di lavoro in Batcher su tutte le immagini acquisite dell'esperimento. Allo stesso tempo, il programma di monitoraggio può essere creato combinando le impostazioni salvate dal Controller probe (.pcfg) con il flusso di lavoro (.wf), vedere anche il manuale.
NOTA: il flusso di lavoro può essere utilizzato anche per monitorare esperimenti futuri per questo supporto di coltura. - Se i risultati non sono accettabili, controllare l'annotazione sul set di training e/o continuare con un altro ciclo di formazione iterativo (vedere il passaggio 4.2).
4. Quantificazione delle dimensioni del campione
- Impostare la deviazione standard (), che è accettabile tra le particelle rilevate.
NOTA: la deviazione standard cambia in parallelo all'omogeneità delle dimensioni delle celle. La deviazione standard massima indica il campione con il più alto grado di eterogeneità di dimensioni. - Impostare l'ampiezza dell'intervallo di confidenza o la precisione desiderata in relazione alla varianza prevista delle misurazioni (e).
- Impostare l'errore ammesso tra il 5% (z1-z/2 - 1,96) e il 10% (z1-z/2 - 1,64).
- Calcolare il numero di celle da identificare da ogni classe dall'equazione 1.
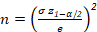 [Equazione 1]
[Equazione 1]
NOTA: In base al numero di celle, il numero di immagini che devono essere acquisite può essere definito per ogni punto dati. - Eseguire un'analisi di sensibilità sui punti temporali casuali dell'esperimento per verificare che l'analisi di n particelle porti ad una variabilità del diametro medio di Feret e del Dv90 inferiore al 5%. Può essere calcolato automaticamente nell'Analizzatore deirisultati.
5. Misurazione in linea (by-pass) o in linea
- Eseguire prima la procedura di misurazione off line (vedere il passaggio 2) per impostare le impostazioni hardware e software in funzione dell'organismo e del processo (concentrazione o supporto).
- Caricare le impostazioni salvate dalla sezione precedente selezionando il pulsante Carica e selezionare il percorso seguente: C:
- Collegare la sonda alla cella di flusso o al bioreattore.
NOTA: Le misurazioni in situ possono essere eseguite con una flangia di pizzicamento. - Eseguire la sterilizzazione.
NOTA: Solo il materiale bagnato dello strumento è sterilizzabile attraverso la sterilizzazione del vapore. La lunghezza bagnata della sonda può andare da 6 a 222 mm (Figura 3).
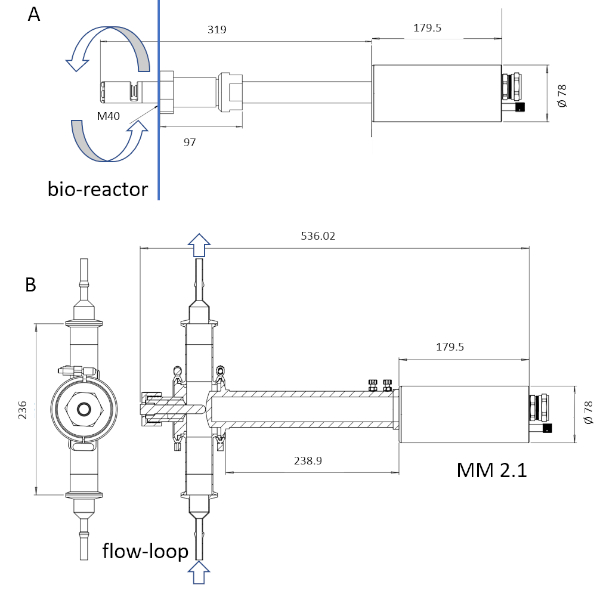
Figura 3: Schizzo dei dispositivi ISM. La sonda MM-Ho (A) è installabile direttamente nel bioreattore, mentre la sonda MM 2.1 (B) può essere utilizzata come by-pass. La circolazione del brodo culturale è contrassegnata da frecce in ogni immagine. I fattori di conversione sono 0,166 - m pix-1 per MM-Ho e 0,087 -m pix-1 per MM 2.1. Fare clic qui per visualizzare una versione più grande di questa figura.
- Definire la velocità di acquisizione dell'immagine nella GUI Triggering nel campo Trigger interval [s].
NOTA: a seconda delle dinamiche di processo, il tasso di acquisizione dell'immagine può essere adattato. Ad esempio, se si prevede una fase di ritardo di 3 ore, il tasso di acquisizione può essere inferiore rispetto a quando si deve monitorare un cambiamento del metabolismo o l'accumulo di un prodotto. Di solito, questo richiede un tempo di acquisizione molto più breve nell'intervallo di minuti. Ad esempio, una sequenza di acquisizione di immagini compresa tra 5 e 10 min durante una coltivazione di lievito in batch fornisce informazioni sufficienti per acquisire le dinamiche di processo. - Definire la frequenza fotogrammi come spiegato nel passaggio 2.10.
NOTA: nelle misurazioni on line e in linea, la frequenza fotogrammi può essere aumentata, poiché l'agitazione meccanica può aumentare la portata attraverso il divario. - Avviare l'acquisizione dell'immagine attivando il pulsante Avvia streaming della sonda selezionata.
- Interrompere l'acquisizione al termine dell'esperimento con il pulsante Interrompi streaming del probe selezionato.
NOTA: Per la corsa 1st, avviare l'acquisizione poco prima dell'inoculazione della cultura e procedere al passaggio 4. Per le esecuzioni seguenti, aprire il programma Monitoraggio nel dashboard e selezionare il flusso di lavoro creato (vedere il passaggio 4). Avviare il monitoraggio appena prima dell'inoculazione della cultura premendo il pulsante Riproduci.
Risultati
Il rilevamento delle dimensioni delle cellule nelle colture di lievito con l'ISM e il rilevamento automatico delle immagini per distinguere tra cellule in erba e non in erba è stato condotto con successo. Sia l'intensità dello stroboscopio che la scelta del divario di misurazione hanno una gamma di tolleranza, in cui l'identificazione delle particelle non è influenzata. Ad esempio, le cellule di S. cerevisiae sono state misurate con varie intensità dello stroboscopio all'inte...
Discussione
ISM come presentato qui con lo stesso o molto simili dispositivi è stato utilizzato per misurare la dinamica morfologica di funghi, microalghe e lieviti, che ha permesso la determinazione dell'attività di crescita, e in caso di alghe, l'accumulo di prodotti intracellulari. Il sensore non ha parti mobili ed è direttamente applicabile in qualsiasi bioreattore di serbatoio agitato standard, sia attraverso una porta standard o in un by-pass sterilizzabile. Poiché il lievito è molto più piccolo delle alghe, la riduzione...
Divulgazioni
Gli autori non hanno nulla da dichiarare.
Riconoscimenti
Gli autori sono grati per il sostegno del Ministero federale tedesco dell'economia e dell'energia nell'ambito del progetto "Smart Process Inspection", grant no. 4184201CR5.
Materiali
| Name | Company | Catalog Number | Comments |
| Sensor MM 2.1 - MFC | SOPAT GmbH, Germany | n.a. | Inline Monocular Microscopic probe Version 2.1 with a Mirco Flow Cell |
| Sofware version v1R.003.0092 | SOPAT GmbH, Germany | n.a. | |
| Thickness gauge | n.n. | It can be any supplier, DIN 2275:2014-03 | |
| Ethanol 70% | n.n. | It can be any supplier | |
| SOPAT manual Version 2.0.5 | SOPAT GmbH, Germany | ||
| Optical lense paper | VWR | 470150-460 | |
| Fiji, ImageJ | open source | ||
| 50 mL conical centrifuge tubes | It can be any supplier |
Riferimenti
- Maaß, S., Rojahn, J., Hänsch, R., Kraume, M. Automated drop detection using image analysis for online particle size monitoring in multiphase systems. Computers & Chemical Engineering. 45, 27-37 (2012).
- Lemoine, A., Delvigne, F., Bockisch, A., Neubauer, P., Junne, S. Tools for the determination of population heterogeneity caused by inhomogeneous cultivation conditions. Journal of biotechnology. 251, 84-93 (2017).
- Marbà-Ardébol, A. M., Bockisch, A., Neubauer, P., Junne, S. Sterol synthesis and cell size distribution under oscillatory growth conditions in Saccharomyces cerevisiae scale-down cultivations. Yeast. 35 (2), 213-223 (2017).
- Xiao, Y., Bowen, C. H., Liu, D., Zhang, F. Exploiting nongenetic cell-to-cell variation for enhanced biosynthesis. Nature chemical biology. 12 (5), 339-344 (2016).
- Beutel, S., Henkel, S. In situ sensor techniques in modern bioprocess monitoring. Applied microbiology and biotechnology. 91 (6), 1493 (2011).
- Belini, V. L., Wiedemann, P., Suhr, H. In situ microscopy: A perspective for industrial bioethanol production monitoring. Journal of microbiological methods. 93 (3), 224-232 (2013).
- Havlik, I., et al. Monitoring of microalgal cultivations with on-line, flow-through microscopy. Algal Research. 2 (3), 253-257 (2013).
- Suhr, H., Herkommer, A. M. In situ microscopy using adjustment-free optics. Journal of biomedical optics. 20 (11), 116007 (2015).
- Panckow, R. P., Reinecke, L., Cuellar, M. C., Maaß, S. Photo-Optical In-Situ Measurement of Drop Size Distributions: Applications in Research and Industry. Oil Gas Sci. Technol. - Rev. IFP Energies. 72 (3), 14 (2017).
- Marbà-Ardébol, A. -. M., Emmerich, J., Neubauer, P., Junne, S. Single-cell-based monitoring of fatty acid accumulation in Crypthecodinium cohnii with three-dimensional holographic and in situ microscopy. Process Biochemistry. 52, 223-232 (2017).
- Porro, D., Vai, M., Vanoni, M., Alberghina, L., Hatzis, C. Analysis and modeling of growing budding yeast populations at the single cell level. Cytometry. Part A : the journal of the International Society for Analytical Cytology. 75 (2), 114-120 (2009).
- Brauer, M. J., et al. Coordination of growth rate, cell cycle, stress response, and metabolic activity in yeast. Molecular biology of the cell. 19 (1), 352-367 (2008).
- Puxbaum, V., Gasser, B., Mattanovich, D. The bud tip is the cellular hot spot of protein secretion in yeasts. Applied microbiology and biotechnology. 100 (18), 8159-8168 (2016).
- Marbà-Ardébol, A. M., Emmerich, J., Neubauer, P., Junne, S. Vol. P2. Prozessmesstechnik. , 222-225 (2017).
- Marbà-Ardébol, A. -. M., Emmerich, J., Muthig, M., Neubauer, P., Junne, S. Real-time monitoring of the budding index in Saccharomyces cerevisiae batch cultivations with in situ microscopy. Microbial cell factories. 17 (1), 73 (2018).
- Marquard, D., Schneider-Barthold, C., Düsterloh, S., Scheper, T., Lindner, P. Online monitoring of cell concentration in high cell density Escherichia coli cultivations using in situ Microscopy. Journal of biotechnology. 259, 83-85 (2017).
- Marquard, D., et al. In situ microscopy for online monitoring of cell concentration in Pichia pastoris cultivations. Journal of biotechnology. 234, 90-98 (2016).
- Camisard, V., Brienne, J., Baussart, H., Hammann, J., Suhr, H. Inline characterization of cell concentration and cell volume in agitated bioreactors using in situ microscopy: application to volume variation induced by osmotic stress. Biotechnology and bioengineering. 78 (1), 73-80 (2002).
- Böhm, A., Ücker, A., Jäger, T., Ronneberger, O., Falk, T. ISOODL: Instance segmentation of overlapping biological objects using deep learning. , 1225-1229 (2018).
- Davey, H. M. Life, Death, and In-Between: Meanings and Methods in Microbiology. Applied and environmental microbiology. 77 (16), 5571-5576 (2011).
- Lodolo, E. J., Kock, J. L., Axcell, B. C., Brooks, M. The yeast Saccharomyces cerevisiae-the main character in beer brewing. FEMS yeast research. 8 (7), 1018-1036 (2008).
- Albertin, W., et al. Population size drives industrial Saccharomyces cerevisiae. alcoholic fermentation and is under genetic control. Applied and environmental microbiology. 77 (8), 2772-2784 (2011).
- Gomes, J., Chopda, V. R., Rathore, A. S. Integrating systems analysis and control for implementing process analytical technology in bioprocess development. Journal of Chemical Technology and Biotechnology. 90 (4), 583-589 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon