Method Article
Dissezione della funzione Enhancer usando Multiplex basati su CRISPR Enhancer interferenze nelle linee cellulari
In questo articolo
Riepilogo
Questo protocollo descrive i passaggi necessari per progettare e realizzare targeting multiplex di esaltatori con la proteina di fusione disattivazione SID4X-dCas9-KRAB, noto anche come interferenza di enhancer (Enhancer-i). Questo protocollo consente l'identificazione dei rinforzatori che regolano l'espressione genica e facilita la dissezione delle relazioni tra esaltatori di un gene bersaglio comune di regolazione.
Abstract
Rinforzatori più spesso regolano un dato gene, ma per la maggior parte dei geni, rimane poco chiaro quali stimolatori sono necessari per l'espressione genica, e come questi rinforzatori si combinano per produrre una risposta trascrizionale. Come milioni di rinforzatori sono stati identificati, ad alta produttività strumenti sono necessari per determinare la funzione enhancer su scala genomica. Attuali metodi per lo studio della funzione enhancer includono fare genetiche eliminazioni utilizzando Cas9 nucleasi-competente, ma è difficile studiare gli effetti combinatoriali di esaltatori di più utilizzando questa tecnica, come più righe successive clonale delle cellule devono essere generati. Qui, presentiamo Enhancer-i, un metodo basato su interferenza di CRISPR che permette per l'interrogatorio funzionale di esaltatori di multipli contemporaneamente ai loro loci endogeni. Enhancer-i fa uso di due domini repressivi fuse a nucleasi-carenti Cas9, SID e KRAB, raggiungere disattivazione enhancer tramite deacetilazione ai loci mirati. Questo protocollo utilizza trasfezione transiente di guida RNAs per abilitare inattivazione transitoria delle regioni mirate ed è particolarmente efficace nel bloccare trascrizionali inducibili risposte agli stimoli nelle impostazioni di coltura del tessuto. Enhancer-i è altamente specifico sia nella sua targeting genomica e suoi effetti sull'espressione genica globale. Risultati ottenuti da questo protocollo aiutano a capire se un rinforzatore contribuisca all'espressione genica, l'entità del contributo, e come il contributo è influenzato da altri esaltatori di nelle vicinanze.
Introduzione
Su larga scala di sequenziamento progetti come codifica1e tabella di marcia epigenomica2FANTOM3 sono identificati milioni di esaltatori di presunti all'interno del genoma umano attraverso centinaia di tipi di cellule. Si stima che ogni promotore si associa con una media di 4,9 esaltatori e ogni enhancer contatti una media di 2,4 geni3, suggerendo che l'espressione genica è spesso il risultato dell'integrazione di molteplici interazioni di regolamentazione distribuite. Una sfida significativa restante è quello di definire non solo come singoli rinforzatori contribuiscono all'espressione genica, ma come essi si combinano per colpire espressione. Approcci genetici sono comunemente usati per identificare le relazioni tra esaltatori in organismi modello da drosofila4 a topi5. Tuttavia, questi esperimenti sono basso-rendimento per lo studio di esaltatori di più geni multipli e che richiede tempo.
Un approccio per lo studio della funzione enhancer su larga scala prevede saggi reporter parallelismo massivo. Queste analisi consentono la selezione simultanea di migliaia di sequenze di DNA per la loro capacità di guidare l'espressione di un reporter gene6. Mentre queste analisi hanno indicato che sequenza di DNA da solo può essere sufficiente per trasmettere il gene regolamento informazioni7, tornano con i caveat di viene eseguita all'esterno del contesto di cromatina nativo e con un promotore eterologo. Inoltre, la dimensione della sequenza di DNA analizzato nelle analisi di parallelismo massivo reporter è di solito meno di 200 basi, che possono escludere la sequenza rilevante circostante. Cosa importante, come reporter saggi solo misurano l'attività di una sequenza in un momento, non tengono conto le relazioni complesse che possono esistere tra rinforzatori. Così, mentre saggi reporter massicciamente parallela possono essere informativi circa l'attività intrinseca di una sequenza di DNA, essi non necessariamente ci informa della funzione di quella sequenza di DNA nel contesto del genoma.
Recentemente sviluppati strumenti di CRISPR/Cas98 hanno facilitato lo studio della regolazione genica poiché consentono l'eliminazione di esaltatori del locus endogeno. Tuttavia, l'eliminazione di esaltatori di multipli simultaneamente può portare a instabilità genomica ed è molto tempo per generare le eliminazioni successive del rinforzatore in una linea singola cella. Inoltre, nuova sequenza genomica è creato presso il sito dell'eliminazione dopo la riparazione, e questa sequenza può avere funzione regolatrice. Una versione alternativa di Cas9 è stata sviluppata specificamente per la modulazione dell'espressione genica, basandosi su fusioni di attivazione9,10 o reprimere11,12 domini alla forma di nucleasi-carenti di Cas9 (dCas9). Queste proteine di fusione sono ideali per studiare i luoghi multipli simultaneamente come fisicamente non alterano la sequenza del DNA e invece modulare epigenetica per interrogare una regione regolatrice. La fusione repressiva più ampiamente usata è KRAB, che recluta il complesso co-repressore KAP1, promuovendo la deposizione di repressione-collegata istone H3 lisina 9 trimethylation (H3K9me3)13. dCas9-KRAB, noto anche come CRISPR interferenza14, è stato usato per destinazione e schermo rinforzatori individuali per i loro contributi a gene espressione15,16; Tuttavia, esso non è stato ottimizzato per il targeting più aree contemporaneamente. Una versione di interferenza di CRISPR multiplex per rinforzatori, mosaico-seq17, utilizza singola cella RNA-seq come una lettura, ma questa tecnologia è costoso e adatto solo per lo studio dei geni altamente espressi a causa della bassa sensibilità della singola cella RNA-seq.
Abbiamo cercato di sviluppare un metodo di base di interferenza di CRISPR per la dissezione funzione combinatoria enhancer nel contesto di una risposta trascrizionale ad estrogeno. Circa la metà dei geni estrogeno-rispondente contengono esaltatori di 2 o più vincolati dal recettore degli estrogeni (ER) è vicino a18, suggerendo che esaltatori di più possono essere partecipanti nella risposta dell'estrogeno e comprendere che la logica di regolamentazione richiederebbe targeting rinforzatori multipli contemporaneamente. Come gli studi di iniziale con interferenza CRISPR a promotori ha suggerito che non tutti i promotori sono ugualmente reattivi KRAB-mediata repressione19, abbiamo ragionato che l'aggiunta di un dominio repressivo distinto per dCas9 può facilitare la disattivazione della esaltatori di diversi. Abbiamo scelto il Sin3a interagendo dominio di Mad1 (in SID)20 come conduce al reclutamento di istone deacetilasi21, che rimuovono i gruppi acetile sugli istoni che sono associati con l'attività trascrizionale. D'importanza, il dominio SID è stato efficace nel ridurre l'espressione genica quando fusa a dCas922 e TALEs23e Sin3a ha dimostrato di essere un co-fattore potente di repressivo in una varietà di contesti di enhancer sequenza24. Abbiamo usato SID4x-dCas9-KRAB (Enhancer-i) a 10 diversi rinforzatori vincolati al pronto soccorso di destinazione e identificare siti di legame di ER (erbe) che sono necessari per la risposta trascrizionale dell'estrogeno alle 4 geni18. Siamo presi di mira anche le combinazioni di rinforzatori per identificare i siti che collaborano nella produzione della risposta trascrizionale dell'estrogeno. Abbiamo trovato che fino a 50 siti possono potenzialmente essere mirate simultaneamente con cambiamenti di espressione di gene rilevabile. Utilizzando ChIP-seq e RNA-seq, abbiamo dimostrato che Enhancer-i è una tecnica altamente specifica per studiare rinforzatori multipli contemporaneamente.
In questo protocollo, descriviamo i passaggi coinvolti nell'esecuzione di Enhancer-i, una tecnica flessibile che consente lo studio funzionale di esaltatori di multipli simultaneamente in un ambiente di coltura del tessuto. Enhancer-i è altamente correlato con delezione genetica ma fornisce disattivazione transitoria che dipende dell'istone deacetilasi (HDAC). Fornendo guida RNAs tramite trasfezione transiente al contrario stabile integrazione tramite vettori virali, questo protocollo evita la deposizione e la potenziale diffusione del H3K9me3. Questo protocollo Dettagli Guida RNA design e clonazione tramite Assemblea di Gibson, la transfezione di guida RNAs utilizzando lipofezione, e l'analisi dell'espressione genica risultante cambia da qPCR. Includiamo anche i metodi per valutare la specificità di Enhancer-i targeting a livello del genoma e del trascrittoma. Mentre questa tecnica è stata sviluppata per studiare regolamento del gene di ER associato rinforzatori in linee cellulari tumorali umane, è applicabile per la dissezione di qualsiasi mammifero enhancer.
Protocollo
1. generazione di Cell Lines che esprimono stabilmente SID4X-dCas9-KRAB
Nota: Le condizioni di transfezione e le concentrazioni di farmaco qui presentate sono state ottimizzate per le cellule di Ishikawa, una linea cellulare di cancro dell'endometrio, coltivate in RPMI 1640 media completati con 10% FBS e 1% penicillina/streptomicina (RPMI completo). Altre linee cellulari possono richiedere condizioni diverse di transfezione e concentrazioni nella droga. Gli utenti possono anche eseguire esperimenti di trasfezione transiente in cellule wild-type, invece di generare una linea cellulare stabile, con un plasmide esprimente SID4X-dCas9-KRAB insieme a RNA guida esprimendo plasmidi; Tuttavia, i risultati da transfezioni transienti potrebbero essere difficili da riprodurre come livelli di SID4X-dCas9-KRAB possono variare dalla transfezione.
- Piastra di cellule di Ishikawa in almeno 2 pozzetti di una piastra di 6 pozzetti di confluenza 30-50% (circa 300.000 cellule di Ishikawa) in 3 mL di RPMI completo.
- Aspirare i media dalle cellule. Lavare le cellule una volta con PBS 1X (pH 7.4). Aspirare il PBS e aggiungere tripsina (4 mL per un piatto di 10 cm o 5 mL per un pallone da T-75).
- Incubare le cellule per ~ 5 min a 37 ° C, controllando ogni 2 min per cellule indipendente e delicatamente scuotendo il vaso.
- Una volta che le cellule hanno staccato, pipetta tripsinizzate cellule su e giù per un paio di volte e pipetta inclinate leggermente il bordo del recipiente per rilasciare eventuali cellule allegate.
- Trasferire le cellule in una provetta conica da 15 mL e rallentare le cellule per 5 min a 250 x g.
- Aspirare la tripsina e risospendere le cellule in 5-10 mL di media. Utilizzare una pipetta P1000 per dissociare ciuffi di cella, se necessario.
- Contare le celle e determinare il volume necessario per piastra ~ 300.000 cellule per pozzetto in un volume totale di 3 mL. Aggiungere le celle 2 pozzetti separati di una piastra a 6 pozzetti. Riempire ogni bene a 3 mL con RPMI completo.
- Scuotere delicatamente la piastra ogni 5 min nel primo 15 min dopo placcatura per garantire che le cellule siano distribuite uniformemente sulla piastra. Utilizzare un microscopio per garantire che le cellule sono disperse dalla metà del pozzo.
- Entro 24 h dalla placcatura, eseguire i seguente transfezioni utilizzando un reagente di transfezione appropriato per la linea cellulare di interesse. Per cellule di Ishikawa, è necessario utilizzare la procedura come descritto di seguito.
Nota: Il presente protocollo si presuppone l'utilizzo di reagenti di transfezione di base di liposomi cationici. Elettroporazione fornisce un metodo alternativo per tipi di cellule che sono molto sensibili a questi reagenti o che esibiscono l'efficienza di trasfezione basso con lipofezione. Condizioni di transfezione devono essere ottimizzate per la linea cellulare di interesse prima di tentare di esperimenti Enhancer-i.- In un 1,7 mL provetta Eppendorf, diluire 2,5 μg di plasmide SID4X-dCas9-KRAB e 800 ng di plasmide esprimente una proteina fluorescente in terreni privi di siero in modo tale che il volume finale del tubo è µ l 155 e la concentrazione finale del plasmide è 0.020 µ g / µ l.
- In un altro tubo, diluire 3,3 µ g di un plasmide che non contengono una cassetta di resistenza di neomicina, quali pCMV-GFP, in media del siero senza tale che il volume finale del tubo è µ l 155 e la concentrazione finale del plasmide è 0.020 µ g / µ l.
- Vortice ogni tubo brevemente e rotazione verso il basso utilizzando un microfuge.
- Aggiungere 9,9 µ l di reagente di transfezione (Tabella materiali) in ogni provetta. Mescolare nel Vortex brevemente a bassa velocità. Rallentare i tubi con un microfuge.
- Incubare le provette a temperatura ambiente per almeno 5 minuti, ma non più di 20 min.
- Nella cappa di biosicurezza, aggiungere 150 µ l della miscela del DNA: reagente preparato goccia a goccia in un pozzetto sulla piastra 6 pozzetti. Ripetere per l'altro tubo di mix di DNA: reagente preparato. Miscelare le piastre agitando delicatamente e riporre la piastra nell'incubatore.
- Transfezione di post del giorno 2, cambiare il supporto e completare con G418 a una concentrazione finale di 600 ng/μL. Questa concentrazione potrebbe essere necessario essere ottimizzato per il tipo di cella.
- Modificare multimediale completo RPMI e completare con G418 ogni altro giorno per 2-4 settimane fino a quando il controllo transfettata le cellule sono morti e i pozzetti contenenti SID4X-dCas9-KRAB diventano confluenti. L'esatta quantità di tempo necessario per recuperare le cellule dipenderà il tempo di raddoppiamento delle cellule.
- Quando le cellule diventare confluenti, passaggio a una nave T-25 o T-75 in completano RPMI con una dose inferiore di G418 (300 ng/μL per cellule di Ishikawa). Durante questo passaggio, fare 2 aliquote di ~ 100.000 cellule ogni (circa 1/10th di una piastra a 6 pozzetti) in 2 provette Eppendorf di separata 1,7 mL per l'isolamento di RNA e DNA, rispettivamente. Girare questi tubi verso il basso (5 min, 250 x g), rimuovere tripsina pipettando e congelare le provette a-20 ° C per un uso futuro.
- Isolare il DNA di genomic usando i kit disponibili in commercio ed eseguire PCR utilizzando il "pAC95_PCR" o "SID4X_PCR" primer (tabella 1) per verificare la presenza della proteina di fusione all'interno nella linea cellulare. Utilizzare il DNA genomic estratto della linea parentale come controllo negativo e il DNA del plasmide SID4x-dCas9-KRAB come controllo positivo. Utilizzare un mix master della polimerasi ad alta fedeltà con 50-100 ng di DNA genomico e le seguenti condizioni in bicicletta: 98 ° C per 30 s, 25 cicli di (98 ° C per 10 s, 58 ° C per 30 s, 72 ° C per 2 min), 72 ° C per 5 min , tenere a 4 ° C.
- Per verificare l'espressione della proteina di fusione a livello di RNA, eseguire qPCR con RNA estratto dalla linea cellulare usando i kit disponibili in commercio. Utilizzare il primer "dCas9_qPCR" (tabella 1) e il protocollo di qPCR One-Step fornito nel passaggio 6.3 del presente protocollo.
- Per verificare l'espressione del livello della proteina di fusione, eseguire un Western blot su lisati dalla linea cellulare. Utilizzare anti-FLAG o anti-HA gli anticorpi per rilevare la proteina di fusione.
2. Guida RNA Design
Nota: Questo protocollo è progettato per uso con il RNA di guida U6 clonazione vettoriale creata dal laboratorio Chiesa e disponibile su Addgene (Addgene 41824). Per creare una versione di questo vettore contenente resistenza con puromicina che ha permesso per la stessa strategia clonazione 41824, ci siamo spostati i polylinker da questo vettore nel vettore pGL3-U6-sgRNA-PGK-con puromicina (Addgene 51133). Addgene 41824 o la nostra versione con con puromicina (Addgene 106404) sono compatibili con la clonazione strategia descritta di seguito.
- Ottenere 600-900 basi di sequenza di DNA di ogni regione regolatrice di interesse. Utilizzare siti di legame del fattore di trascrizione e/o l'accessibilità della cromatina per indicazioni su dove andare a definire l'area di interesse (Figura 2A).
Nota: Mentre nell'esempio in Figura 2A caratteristiche esaltatori di upstream e downstream, è anche possibile di destinazione elementi regolatori che si trova all'interno di introni. - Posizionare tutte le sequenze ottenute in un unico file di testo utilizzando il formato FASTA.
- Identificare almeno una regione di controllo negativo che non si prevede di passare a condizioni sperimentali, come promotore di un gene che non è espresso nella linea cellulare di interesse. Ottenere la sequenza del DNA per questa regione e aggiungerlo al file di testo in formato FASTA.
Nota: Abbiamo usato la guida RNAs targeting il promotore IL1RN 25 come controllo negativo per tutte le regioni che ci rivolgiamo. Gli utenti possono anche selezionare sequenza intergenica vicino alla regione di interesse che non contiene siti di legame del fattore di trascrizione come controllo negativo. Tuttavia, se più loci sono presi di mira contemporaneamente, una regione di singolo controllo negativo semplifica disegno sperimentale e interpretazione dei risultati. Se il rinforzatore mirato è intronic, può essere utile a destinazione una regione intronic nello stesso locus, che non contiene un elemento regolatore putativo come un ulteriore controllo negativo, come la fusione di dCas9 può interferire con la trascrizione. - Identificare le aree di controllo positivo, quali promotori che sono il bersaglio putativo di nelle regioni regolatrici di interesse o promotori di geni che sono altamente trascritti nella linea cellulare di interesse. Ottenere la sequenza del DNA per queste regioni e aggiungerlo al file di testo in formato FASTA.
- Utilizzare un programma come e-croccante26 (http://www.e-crisp.org/E-CRISP/) sulle sequenze del DNA generate per cercare Guida RNAs con basso off-target (idealmente 0-3). Guida RNAs consistono di 20 nucleotidi a Monte di un motivo adiacente protospacer (PAM), che assume la forma "NGG" per il dCas9 da S. pyogenes.
- Sul sito Web e-croccante, selezionare l'organismo di interesse utilizzando il menu a discesa. L'Assemblea di genoma appare a destra del nome della specie.
- Selezionare il pulsante di opzione Input è FASTA sequenza . Copiare le sequenze FASTA dall'alto e incollarli nella finestra di dialogo. Verificare che un'intestazione FASTA è inclusa per ogni sequenza.
Nota: Fino a 50 sequenze possono essere interrogati simultaneamente. - Nel menu a discesa, selezionare il pulsante di opzione Medium e unico disegno .
- Fare clic sul pulsante Avvia ricerca sgRNA. Si aprirà una nuova scheda del browser, e i risultati verranno visualizzati. Scarica le sequenze di candidati facendo clic sul pulsante Scarica un report tabella Excel formattato per tutte le sequenze di query insieme.
- Aprire il report tabulare utilizzando Excel o un editor di testo.
- Utilizzare il browser genoma UCSC BLAT sequenze di Full-Length gRNA candidato (23 basi) al genoma.
- In un browser, passare al sito del browser UCSC genoma (http://genome.ucsc.edu). Sotto la sezione i nostri strumenti, individuare la parola BLAT e fare clic su di esso. Verrà aperto lo strumento di ricerca BLAT.
- Utilizzare i menu a discesa situati sotto il testo BLAT ricerca genoma per selezionare l'assembly organismo e genoma di interesse.
- Copiare le sequenze di RNA guida dal report tabulare generato dal e-crisp e incollarli nella finestra di dialogo. Garantire che ogni sequenza ha un'unica intestazione FASTA, quindi fare clic su Invia nella parte inferiore della finestra di dialogo.
Nota: Fino a 25 sequenze possono essere esaminati in una sola volta. Nella pagina dei Risultati di ricerca BLAT , allineamenti di ogni sequenza di RNA guida apparirà, con ogni riga che rappresenta un allineamento. Idealmente, dovrebbe esserci un allineamento per ogni guida di RNA, che indica l'unicità di tale guida RNA. - Evitare le guide che si allineano a più posizioni nel genoma, se possibile.
- Per esaminare la guida RNA localizzazione e distribuzione all'interno della regione di interesse, clicca sul link Browser nella sezione azioni per uno degli RNA guida richiesto. Il Genome Browser verrà visualizzato e sarà centrato sulla guida selezionata RNA. Utilizzare i pulsanti Zoom indietro nella parte superiore della pagina per visualizzare la distribuzione di altre guida RNAs identificato da e-croccante all'interno della regione di interesse.
- Selezionare 4 preferibilmente non sovrapposte guida sequenze di RNA che vengono distribuiti in tutta la regione di interesse (Figura 2B). Se la regione di interesse è superiore a 600 bp, considerare l'aggiunta di 1-2 guide aggiuntive. Evitare di guida RNAs con tratti di omopolimero e contenuto di GC estrema, come queste caratteristiche possono ostacolare il RNA guida processo di duplicazione e ridurre guida RNA efficienza di targeting.
- Una volta che le guide sono state selezionate, creare un file contenente la guida completa sequenza di RNA (23 nucleotidi) per ogni guida desiderata e quindi rimuovere il nucleotide 5' così come il PAM (NGG) dall'estremità 3'. Questo passaggio facilita l'ordinazione oligo.
- Aggiungere la seguente sequenza al 5' conclusione della sequenza del oligonucleotide: GTGGAAAGGACGAAACACCG.
- Aggiungere la seguente sequenza all'estremità 3' della sequenza del oligonucleotide: GTTTTAGAGCTAGAAATAGC.
Nota: La sequenza finale dovrebbe essere 59 nucleotidi lungo e senti come questo: GTGGAAAGGACGAAACACCG-destinazione (19 nt)-GTTTTAGAGCTAGAAATAGC. - Assicurarsi che ogni elemento normativo per essere mirate con Enhancer-i con almeno 4 oligonucleotidi unici progettati per esso. Ordinare queste sequenze insieme con gli iniettori di "U6_internal" elencati nella tabella 1.
3. Guida RNA clonazione
Nota: Guida RNA clonazione tramite Assemblea Gibson ha dimostrato di essere altamente efficiente nelle nostre mani, producendo centinaia di colonie per piastra, con pochi eventuali colonie presenti nel controllo unico vettore. Tale efficienza è fondamentale per mantenere la complessità durante la clonazione in pool. Un altro vantaggio dell'Assemblea di Gibson clonazione è che gli utenti non devono preoccuparsi circa la presenza di un enzima di restrizione tagliata sito nella Guida RNA stanno cercando di inserire il vettore di clonazione U6. Tuttavia, questo protocollo può essere adattato per tradizionale degli enzimi di limitazione basato clonazione se lo si desidera.
- Ricostituire i oligos guida RNA ad una concentrazione finale di 100 μM in acqua ultrapura (privo di RNasi, DNasi-free). Ci dovrebbe essere almeno 4 Guida separata RNA oligos per ogni area di interesse.
- Per ogni regione regolatrice di interesse, creare un pool di tutti i oligos corrispondente alla regione di interesse. In una provetta Eppendorf, unire 5 μL di ogni oligo RNA guida ricostituito individuale per ogni regione. Mescolare bene la piscina Vortex, poi rimuovere 1 μL e diluire questa aliquota 1: 200 in acqua ultrapura.
Nota: Se lo si desidera, queste piscine targeting singole regioni regolatorie possono essere ulteriormente combinate per generare una complessa piscina targeting più regioni. Fino a 50 regioni regolarici possono essere mirate simultaneamente in un singolo pool (Figura 3). - Eseguire una breve PCR con primers U6 per allegare regioni di omologia oligos prima del montaggio di Gibson. Circa 40 basi verranno aggiunto a ogni oligo, ottenendo un prodotto di ~ 100 bp che contiene sufficiente omologia al vettore U6 su entrambe le estremità.
- Per ogni guida di pool di RNA, impostare un 20 μL PCR con un mix master della polimerasi ad alta fedeltà e i seguenti componenti: 1 μL di oligo diluito piscina dal punto 3.2, 1 μL di U6 forward primer (10 μM), 1 μL di U6 reverse primer (10 μM) e fino a 20 μL di acqua.
- Incubare in un termociclatore con le seguenti condizioni: 98 ° C per 30 s, 10 cicli di (98 ° C per 10 s, 55 ° C per 30 s, 72 ° C per 2 min), 72 ° C per 5 min e tenere a 4 ° C.
- Eseguire 5 μL della reazione su gel di agarosio 1-2% con una scala di basso peso molecolare. Il prodotto finale dovrebbe essere ~ 100 basi (Figura 2).
- Ripulire la reazione di estensione con un kit di purificazione di DNA basata su colonne ed eluire in 20 μL di tampone di eluizione fornito nel kit.
Nota: Poiché il prodotto è breve, evitare di utilizzare basati su tallone clean-up, che sono progettati per escludere piccoli frammenti meno di 100 bp. - Quantificare il DNA purificato utilizzando un fluorimetro o spettrofotometro (previsto resa è 10-20 ng/μL). Guida RNA inserti possono essere conservati a-20 ° C, o possono essere utilizzati immediatamente in Assemblea di Gibson con un vettore linearizzato U6.
- Per preparare il destinatario U6 vettore di clonazione per assemblaggio di Gibson, impostare un digest di enzima di restrizione. Se molte reazioni di Gibson assembly devono essere eseguiti, impostare più digest per garantire sufficiente rendimento di taglio vettoriale.
- Utilizzare 20 unità di enzima AflII e 1 μg di plasmide in una reazione di 20 µ l con il buffer di enzima di restrizione appropriato. Incubare a 37 ° C per 1-2 ore.
- Ripulire il digest con perline o un kit basato su colonna per purificazione del DNA ed eluire in 20 μL di tampone di eluizione. Quantificare il DNA purificato utilizzando un fluorimetro o spettrofotometro. I campioni possono essere congelati a-20 ° C per un uso successivo.
- Eseguire il montaggio di Gibson sul vettore del preparato e inserire.
- Impostare Gibson Assemblea reazioni su ghiaccio. Uso 50 ng del vettore e 7 ng dell'inserto in una reazione di 20 μL. Diluire l'inserti 01:10 in acqua ultrapura per agevolare il pipettaggio. Impostare una vettore solo Gibson Assemblea reazione, utilizzando 50 ng del vettore e sostituire l'inserto con l'acqua.
- Incubare le reazioni di assemblaggio di Gibson per 15 min a 50 ° C, seguita da una sospensione a 4 ° C.
- Trasferire i prodotti assemblati in ghiaccio. Diluire i prodotti assemblati 1:4 in acqua ultrapura sul ghiaccio. Ad esempio, aggiungere 5 µ l di prodotto di assemblaggio di Gibson a 15 μL di acqua ultrapura.
- Trasformare i prodotti di assemblaggio Gibson diluiti.
- Scongelare le cellule competenti di alta efficienza sul ghiaccio e fare 25 aliquote del μL per ogni trasformazione. Se si desidera una complessa piscina più siti di targeting, scongelare abbastanza cellule in diverse provette per eseguire trasformazioni più indipendente della stessa piscina complesso gRNA.
- Per ogni prodotto diluito, aggiungere 1 μL di questa diluizione a un 1,7 mL provetta Eppendorf contenente 25 μL di cellule competenti. Mescolare brevemente sfogliando il tubo. Incubare le provette in ghiaccio per 30 min.
- Scossa di calore le cellule per 30 s a 42 ° C, quindi trasferire immediatamente in ghiaccio per 2 min.
- Aggiungere 300 μL SOC media (tryptone di 2%, 0,5% di Estratto di lievito, 10 millimetri di NaCl, 2.5 mM KCl, 10mm MgCl2, 10mm MgSO4e 20 mM glucosio) e lasciare che le celle recuperare per 1h a 37 ° C con agitazione (300 giri/min). Durante questo tempo, riscaldare le piastre di agar con ampicillina/carbenicillina a 37 ° C in un incubatore. Utilizzare una piastra per ogni trasformazione.
- Piastra 50 μL di cellule e posizionare le piastre in un incubatore a 37 ° C durante la notte. Per i pool di targeting per singoli siti, posto direttamente in 3-5 mL di brodo LB (Tabella materiali) contenente ampicillina/carbenicillina (1 mg/mL) e incubare per una notte con agitazione a 250 giri/min a 37 ° C per minipreps.
- Raccogliere le cellule e isolare il DNA.
- Per le librerie di RNA di grande guida targeting più siti, è possibile utilizzare un raschietto piatto per raccogliere tutte le colonie da ogni singola piastra in una maxiprep (coltura liquida 150 mL). Questo può essere facilitato da versando ~ 5 mL di LB con l'antibiotico adatto in un tubo falcon 50 mL e raschiare le colonie nel tubo. Per le raccolte siti individuali di targeting, raschiare le piastre in un miniprep (coltura liquida 3-5 mL).
- Incubare queste culture con l'antibiotico adatto per 3-5 h a 37 ° C con agitazione a 250 giri/min.
- Eseguire l'estrazione di DNA utilizzando un kit che si traduce in preps privo di endotossina.
- Quantificazione di DNA usando un fluorimetro o spettrofotometro. Plasmidi possono essere utilizzati immediatamente nella transfezione o conservati a-20 ° C per un uso futuro.
- Per confermare la presenza della sequenza di RNA di guida all'interno del vettore di U6 per piccole piscine targeting per singoli siti, utilizzare Sanger sequenziamento sul miniprep preparato con il primer "U6_PCR_R" elencati nella tabella 1. A causa di messa in comune di guida RNAs, alla sequenza di destinazione del 19 basepair gRNA produrrà basi miste, ma il promotore U6 e impalcatura di RNA guida che circondano questa sequenza dovrebbe essere intatti.
4. la transfezione di Enhancer-i
Nota: Per il successo del blocco di un estrogeno risposta utilizzando Enhancer-i in cellule di Ishikawa, è necessario privare le cellule di estrogeno per 5-7 giorni prima della trasfezione di mantenerle in rosso fenolo libero RPMI con 10% carbone-spogliato FBS e 1% penicillina/streptomicina. Le cellule dovrebbero essere coltivate in questa media durante e dopo la trasfezione se cercando di bloccare una risposta di estrogeno. Si consiglia l'uso di rosso fenolo libero tripsina per passaggio di cellule in rosso fenolo completa gratuita RPMI.
- Il giorno prima di transfezione, piastra le cellule (wild-type o che esprimono stabilmente SID4X-dCas9-KRAB) in una piastra a 24 pozzetti al confluency di 30-50% (~ 60.000 cellule per pozzetto per cellule di Ishikawa). Piastra abbastanza cellule tale che transfezioni possono essere eseguite in duplicato e includono pozzi per trasfettate con guida controllo RNAs. Assicurarsi che le celle sono distribuite uniformemente tra il pozzo agitando delicatamente la piastra dopo il placcaggio di cella come descritto al punto 1.1.7.
Nota: Il presente protocollo si presuppone l'utilizzo di reagenti di transfezione di base di liposomi cationici. Elettroporazione fornisce un metodo alternativo per i tipi di cella che sono molto sensibili a questi reagenti. Condizioni di transfezione devono essere ottimizzate per la linea cellulare di interesse prima di tentare di esperimenti Enhancer-i. - Il giorno seguente, preparare transfezioni seguendo le istruzioni del reagente di transfezione di scelta. Per cellule di Ishikawa, uso 550 ng di plasmide totale per ciascun pozzetto di una piastra a 24 pozzetti. Diluire i plasmidi a una concentrazione finale di 0.020 μg/μL in media privo di siero (1,1 μg di DNA in 52 μL di volume totale per trasfezione 2 pozzi). Utilizzare 3 μL di reagente di transfezione per ogni 1 μg di DNA, vortex e incubare come descritto al punto 1.2. Aggiungere 25 µ l della miscela finale ad ogni pozzetto.
Nota: Per combinazioni di destinazione dei siti, utilizzare lo stesso peso del plasmide per ogni singolo sito e quindi riempire il peso rimanente con un plasmide di controllo (RNA guida vuoto clonazione vettoriale o guida RNAs destinazione è un'area di controllo negativo come IL1RN promotore). Per transfezioni transienti, utilizzare un rapporto di plasmide di RNA di 3:2 Cas9 fusione: Guida. Plasmidi contenenti reporter fluorescenti possono essere aggiunti per monitorare l'efficienza di trasfezione. - A transfezione di post 36H, cambiare il supporto utilizzando rosso fenolo RPMI con 10% carbone-spogliato FBS e 1% penicillina/streptomicina (per cellule di Ishikawa) e la fornitura gratuita con puromicina (concentrazione finale: 1 μg/mL) e neomicina (concentrazione finale: 300 ng/mL). Se le cellule sono sensibili al reagente di transfezione, il supporto può essere modificato in precedenza, ma antibiotici devono essere aggiunto non prima di transfezione post 24 h.
Nota: Attendere almeno 24 ore dopo l'aggiunta di antibiotico prima della raccolta di cellule. Cambiamenti di espressione a causa di Enhancer-i possono essere rilevati come presto come 48h post transfezione e fino a 5 giorni dopo trasfezione. Se lavora con cellule di Ishikawa che sono state private di estrogeno, eseguire un'induzione 8-h 10 nM 17 β-estradiolo (E2) il giorno dopo il trattamento antibiotico e quindi raccogliere le cellule immediatamente.
5. cellula di raccolta ed estrazione del RNA
- Preparare il tampone di lisi con 1% β-mercaptoetanolo (BME). Assicurarsi che ci sia abbastanza miscela di lisi-BME (300 μL per ogni bene che la raccolta).
- Aspirare il supporto utilizzando un aspiratore.
- Lavare le cellule una volta con un volume uguale di PBS 1X (500 μL) e aspirare a rimuovere quanto più PBS come possibile.
- Aggiungere 300 μL di soluzione di lisi-BME ad ogni pozzetto usando una pipetta multicanale. Dispensare la soluzione di lisi su e giù 8 - 10 volte e il trasferimento a un profondo-piastra o 1,7 mL Eppendorf tubi sul ghiaccio. RNA possa essere estratti immediatamente, o lisati possono essere congelati a-80 ° C per elaborazioni future.
- Per estrarre RNA da lisati, utilizzare un kit disponibile in commercio che include un trattamento della dnasi. Eluire nel più piccolo volume consigliato di acqua ultrapura (privo di RNasi, DNasi-free) o eluizione buffer e quantificare il RNA. Per un piccolo numero di campioni, utilizzare un fluorimetro o spettrofotometro. Per un gran numero di campioni, utilizzare una sonda fluorescente che rileva RNA e misura su un lettore di piastra. I campioni possono essere congelati a-80 ° C prima o dopo la quantificazione.
6. quantificare i cambiamenti di espressione genica utilizzando uno stadio qPCR e RNA-seq
- Ottenere qPCR primer per i geni di interesse e per le pulizie almeno un gene che è espresso vicino al livello del gene targeting e non cambia in condizioni sperimentali. Idealmente, questi iniettori si estendono in una giunzione esone-esone per evitare l'amplificazione di DNA genomic.
- Prova questi iniettori su RNA ottenuto dalla linea cellulare di interesse. Uso sciogliere l'analisi delle curve per verificare la produzione di un singolo prodotto. Se non viene prodotto un unico prodotto, prova coppie di primer aggiuntive.
- Per ogni campione di RNA trattato Enhancer-i e controllo di guida, identificare quanti geni devono essere dosati in quel campione. Questo insieme di geni dovrebbe includere geni housekeeping, quali CTCF o GAPDH.
- Impostare qPCR reazioni.
- Diluire i campioni tutti alla stessa concentrazione in acqua, tale che 50 ng di RNA totale è facilmente dispensato e c'è abbastanza diluito RNA per ogni reazione. Ad esempio, diluire RNA a ~16.6 ng/μL e utilizzare 3 μL di RNA in ogni reazione. Tenere RNA su ghiaccio durante l'impostazione master mix.
- Preparare il mix master separati per ogni gene deve essere misurata usando i corredi qPCR commercialmente disponibile One-Step. Utilizzare un volume di reazione di 20 μL con 1 μL di ogni primer (soluzione madre di 10 μM). Impostare queste reazioni su ghiaccio.
- In una piastra di reazione che è appropriata per il termociclatore, aggiungere campioni di RNA seguiti da master mix. Sigillare con un foglio sigillante e mescolare delicatamente nel Vortex o pipettaggio. Centrifugare brevemente la piastra (140 x g per 60 s) affinché quel liquido è presso il fondo dei pozzetti.
- Incubare la piastra in un termociclatore come segue (o come kit incarica): 48 ° C per 30 min, 95 ° C per 10 min, 40 cicli di (95 ° C per 15 s, 60 ° C per 1 min).
- Ottenere i valori di Ct per ogni gene misurata all'interno di ogni campione. Utilizzare il metodo comparativo di Ct per identificare i cambiamenti nell'espressione genica.
- Sottrarre il Ct del gene delle pulizie da Ct di ogni gene di interesse per ogni campione per generare valori normalizzati di Ct.
- Per i campioni di controllo trattato, prendere una media dei valori di Ct normalizzati per ciascun gene. Registro base 2-scala pieghevole repressione quindi può essere calcolato sottraendo l'esempio Enhancer-i trattati normalizzata Ct per ogni gene dallo stesso valore per il campione di controllo trattato per il gene corrispondente.
- Per determinare cambiamenti nell'espressione genica Enhancer-i trattamento seguente, è necessario preparare i campioni per la sequenza di RNA utilizzando un kit disponibile in commercio compatibile con tecnologia di sequenziamento dell'utente. Uso ~ 500 ng di RNA per il materiale di partenza e preparare le librerie per almeno 2 repliche biologiche.
7. Verifica di Targeting genomici specifici di SID4X-dCas9-KRAB utilizzando ChIP-seq
Nota: La proteina di fusione SID4X-dCas9-KRAB contiene sia un tag di epitopo FLAG e un tag di epitopo HA, ma migliori risultati per ChIP-seq sono stati ottenuti con gli anticorpi anti-FLAG. Se lo si desidera, l'utente può eseguire ulteriori esperimenti di ChIP-seq per fattori di trascrizione potenzialmente interessati da Enhancer-i, o per H3K27ac, un segno di attività enhancer che è diminuito da Enhancer-io. Tuttavia, ogni esperimento ChIP-seq richiede 10 x 106 celle, quindi pianificare di conseguenza.
- Transfect cellule con piscine Enhancer-i.
- Piastra 10 x 106 cellule in un piatto di coltura del tessuto di 15 cm. Ogni piatto rappresenta 1 ChIP-seq esperimento per 1 fattore di interesse.
- Il giorno seguente, transfect le celle utilizzando 20 μg di DNA totale per ogni piatto. Per transfezioni nelle linee cellulari che esprimono stabilmente SID4X-dCas9-KRAB, il DNA deve essere un pool di plasmide di guida RNAs targeting per tutti i siti di interesse e, facoltativamente, un plasmide di espressione della proteina fluorescente. Per transfezioni transienti, utilizzare un rapporto di pool di RNA proteina: Guida di 3:2 dCas9 fusione. Per ChIP-seq di altri TFs o modificazioni istoniche, eseguire almeno una trasfezione di guida RNA di controllo aggiuntivo su un altro piatto.
- Trattare i piatti con con puromicina (1 μg/mL) e neomicina (300 ng/mL) a transfezione post 24-48 h. Attendere almeno 24 ore prima della raccolta della cromatina.
- Cromatina raccolto dai piatti.
Nota: Per studiare gli effetti di Enhancer-i su associazione genomiche di ER in cellule di Ishikawa, eseguire un trattamento di 1 ora e 10 nM E2 su piatti transfected con guida controllo RNAs ed Enhancer-i prima della vendemmia. Per studiare gli effetti di Enhancer-i su H3K27ac, eseguire un 8 h 10 nM E2 induzione su piatti transfected con guida controllo RNAs ed Enhancer-i prima della vendemmia.- Applicare 500 μL di formaldeide al 37% per ogni piatto (concentrazione finale dell'1%). Agitare brevemente le piastre. Lasciate che le piastre riposare a temperatura ambiente per 10 min.
- Aggiungere 1 mL di 2,5 M glicina (concentrazione finale di 125 mM). Agitare brevemente le piastre.
- Versare i media con formaldeide e glicina. Aggiungere un volume equivalente (~ 20 mL) di PBS 1X freddo.
- Versare il PBS. Aspirare con un aspiratore per rimuovere quanto più PBS come possibile. Mettere i piatti sul ghiaccio.
- Aggiungere 3-5 mL di freddo 1X PBS o Farnham buffer di lisi (5 millimetri tubi pH 8.0, 85 mM KCl, 0,5% NP-40) con 1 x inibitore della proteasi (aggiunta appena prima dell'uso) per ogni piatto. Raschiare il piatto con un raschietto piatto e trasferire la soluzione in una provetta conica da 15 mL sul ghiaccio.
- A pellet della cromatina di filatura giù tubi in una centrifuga per 5 min a 4 ° C a 1000 x g. gettare il surnatante e memorizzare i pellet a-80 ° C per un uso futuro o procedere con il protocollo del ChIP-seq di scelta utilizzando un anticorpo anti-FLAG o anticorpi targeting altro fattori di trascrizione o le modifiche dell'istone di interesse (H3K27ac, H3K9me3).
Risultati
La figura 1 Mostra uno schema del flusso di lavoro descritto nel protocollo. Per determinare i contributi di esaltatori di ER associato vicino al gene estrogeno-regolate MMP17, che ha 3 siti di legame nelle vicinanze come definito dal ChIP-seq (Figura 2A), guidano di RNA sono stati progettati per ogni regione. Per progettare guida RNAs, una finestra di bp di 600-900 di sequenza che circonda ogni ER sito di legame di interesse è stato selezionato e messo in un programma di progettazione del RNA di guida. RNA sequenze risultanti guida con 0-2 preveduto fuori target siti sono stati allineati al genoma umano utilizzando BLAT. Quattro non sovrapposte guida RNAs che attraversava l'area definita da ChIP-seq e ipersensibilità di DNaseI sono stati scelti per il targeting (Figura 2B). Ulteriore sequenza (tabella 1) è stato aggiunto a ciascuna estremità per facilitare la clonazione a valle e la conseguente 59 frammenti del nucleotide sono state ordinate. All'arrivo, guida RNAs erano diluiti e riuniti dal sito e un breve PCR è stata effettuata per aggiungere aree di omologia prima del montaggio di Gibson. Figura 2 Mostra il prodotto di RNA Guida previsto dopo un breve PCR utilizzando i primer di "U6_internal" (tabella 1), che aggiungeranno 20 basi di sequenza a ogni estremità della del basepair 59 guida frammento di RNA, risultanti in una sequenza del basepair ~ 100. A seguito di Assemblea Gibson, queste piscine guida RNA sono state trasformate in batteri e plasmide minipreps sono stati preparati il giorno seguente. Figura 2D Mostra i risultati da un esperimento di dissezione di enhancer, dove più rinforzatori è vicino a MMP17 sono mirati da solo e in combinazione con Enhancer-i. Siti mirati da Enhancer-io sono indicati con un esagono nero. Guida RNA plasmidi targeting i siti indicati sono stati trasfettati in una linea cellulare Ishikawa estrogeno-sfavoriti che esprimono stabilmente SID4X-dCas9-KRAB. Due giorni dopo, i media è stato cambiato e con puromicina è stato aggiunto per arricchire per cellule trasfettate. Il giorno seguente, le cellule sono state raccolte dopo un trattamento di estradiolo di 8 h 10 nM. RNA è stato isolato, e una qPCR One-Step è stato effettuato. In questo esempio, siti 1 e 2 sono necessari per una risposta estrogenica completa di MMP17, mentre sito 3 non contribuisce in queste condizioni (Figura 2D, corsie ii-iv). Quando sono attivi solo i siti 2 o 3 (vi e vii), la risposta di estrogeno è simile a quando nessun sito è attivo (viii), suggerendo che questi siti non possono contribuire in modo indipendente. Sito 1 può contribuire qualche espressione di sé (v), ma la più grande attività è visto quando siti 1 e 2 sono attivi (iv).
Per manipolare simultaneamente 10 rinforzatori vicino a 4 diversi geni (Figura 3A), complesse piscine di guidano RNAs è stato generato contenente 42 enhancer guide e 16 promotore. Guida RNA oligos sono stati riuniti prima della Guida iniziale estensione RNA PCR (punto 3.3), e i prodotti PCR ottenuti sono stati purificati e combinati con il vettore di clonazione U6 con puromicina vuoto utilizzando assembly di Gibson. A seguito dell'Assemblea di Gibson, più trasformazioni indipendenti erano eseguite e placcate. Le piastre sono state raschiate in LB e ha permesso di crescere fuori per 2-4 h prima del maxiprep. Figura 3B Mostra rappresentative riduzioni nell'espressione del gene di qPCR quando questi pool di RNA di guida sono stati trasfettati in una linea cellulare Ishikawa estrogeno-sfavoriti che esprimono stabilmente SID4X-dCas9-KRAB e trattati come descritto in precedenza (Figura 2D). Riduzioni da Enhancer-i sono simili a quelli ottenuti prendendo di mira il promotore del gene bersaglio putativo. Figura 3 illustra gli effetti di diluizione della Guida RNAs su riduzione della risposta dell'estrogeno utilizzando Enhancer-i. Un 01:50 diluizione di un pool di guida RNA targeting il rinforzatore vicino G0S2 ancora produce una riduzione significativa nell'espressione genica, suggerendo che Enhancer-i può essere utilizzato per indirizzare fino a 50 siti in una volta. Tuttavia, la disattivazione può essere diluita, che indica che centinaia di siti non può essere destinati allo stesso tempo a meno che non sono impiegati metodi di rilevazione più sensibili.

Figura 1. Protocollo schematico per dissezione enhancer multiplex utilizzando Enhancer-i. Guida RNAs (rosso e blu) sono progettati utilizzando e-croccante e selezionato utilizzando il browser del genoma UCSC. Guida quattro RNAs sono scelti che coprono le regioni di interesse (trascrizione fattore associazione siti come definito da ChIP-seq). Oligonucleotidi da RNA guida che hanno messo in comune dalla regione di interesse (rosso e blu) sono sottoposti a una PCR per aggiungere regioni di omologia (arancione) prima della Assemblea di Gibson e trasformazione. Risultante piscine plasmide transfected via lipofezione in linee cellulari che esprimono stabilmente SID4X-dCas9-KRAB o in cellule wild-type in combinazione con il plasmide SID4X-dCas9-KRAB. Guida RNA plasmide piscine possono essere transfected individualmente per un sito di destinazione in un momento, o in combinazione per indirizzare più siti contemporaneamente. Cellule transfettate sono trattate con antibiotici per arricchire per celle contenenti guida RNAs. A transfezione di post ~ 72 h, le cellule vengono raccolte. Acidi nucleici può essere estratta per qPCR, RNA-seq o ChIP-seq Clicca qui per visualizzare una versione più grande di questa figura.
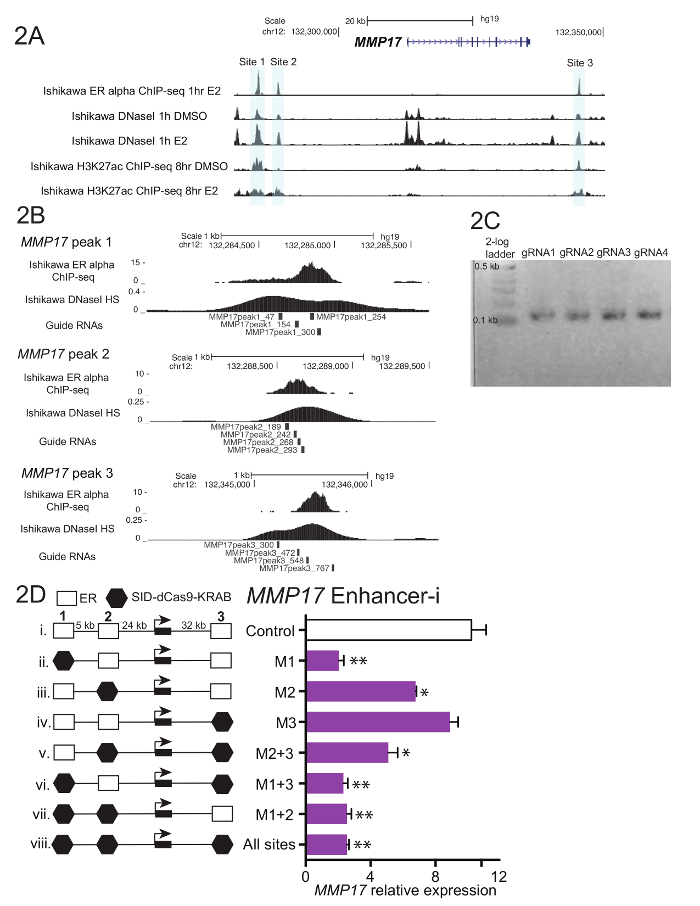
Nella figura 2. Guida di dissezione di design e rinforzatore di RNA per MMP17. (A) Genome browser screenshot di esaltatori di alfa-associazione di ER (grigio) ad essere bersaglio vicino MMP17. Questa figura è stata modificata da Carleton, et al. 18. (B) guida RNA progetta per i 3 siti18di associazione. Il sito di legame per ER come definito da ChIP-seq è il target e la piastrella di RNAs 4 Guida in tutta questa regione. Il segnale di sensibilità di DNaseI, che si estende per il sito di legame, è utilizzabile anche per definire la sequenza di destinazione per guida design di RNA. ChIP-seq sia dati di DNaseI HS sono stati ottenuti da cellule di Ishikawa trattate con 10 estradiolo nM per 1 h. (C) rappresentante guida sequenze di RNA sono pronti per il montaggio di Gibson, avendo subito un breve PCR per aggiungere regioni di omologia. (D) l'espressione relativa di MMP17 misurato via qPCR seguendo il targeting di specifiche regioni con Enhancer-i e un trattamento di estradiolo nM 8-h10. Espressione è relativo livello CTCF ed espressione di MMP17 in cellule non trattate con estradiolo. Guida di controllo RNA bersaglio il promotore di IL1RN. Tutte le barre di errore rappresentano SEM, doppi asterischi indicano p < 0.01 e singoli asterischi indicano p < 0.05 in un t-test accoppiato. Questa figura è stata modificata da Carleton, et al. 18. Clicca qui per visualizzare una versione più grande di questa figura.
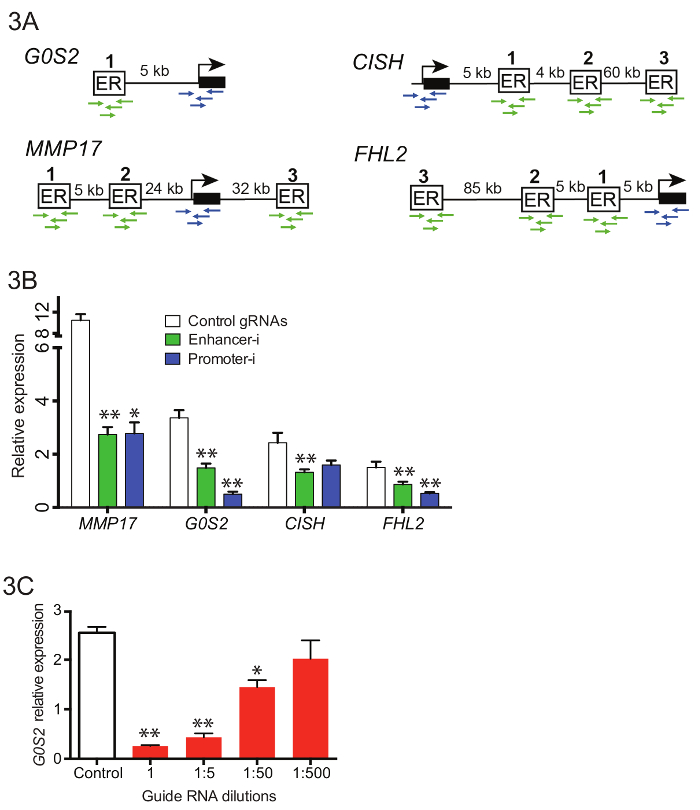
Figura 3. Esaltatori di più vicino a geni differenti simultaneamente con il targeting in pool Enhancer-i. (A) schema dei siti di legame e promotori di essere mirati in pool Enhancer-i. (B) gli effetti sull'espressione come misurato da qPCR dopo trattamento E2 su Ishikawa cellule transfected con piscina di plasmide Enhancer-i (verde), promotore-i plasmidi piscina (blu) o controllo gRNAs (bianco)18. Una riduzione significativa a tutti i geni è osservata con Enhancer-i. Questa figura è stata modificata da Carleton, et al. 18. (C) gli effetti sui livelli di espressione di G0S2 dopo il trattamento E2 in Ishikawa cellule trasfettati con diverse quantità di guida RNAs G0S2di targeting. Una riduzione significativa può essere visto anche con piccole quantità di guida RNA (01:50 diluizione), suggerendo che fino a 50 siti possono mirati contemporaneamente. Tutte le barre di errore rappresentano SEM, doppi asterischi indicano p < 0.01 e singoli asterischi indicano p < 0.05 in un t-test accoppiato. Clicca qui per visualizzare una versione più grande di questa figura.
| Nome | Sequenza |
| U6_internal_F | TTTCTTGGCTTTATATATCTTGTGGAAAGGACGAAACACCG |
| U6_internal_R | GACTAGCCTTATTTTAACTTGCTATTTCTAGCTCTAAAAC |
| U6_PCR_F | CCAATTCAGTCGACTGGATCCGGTA |
| U6_PCR_R | AAAAAAAGCACCGACTCGGTGCCA |
| gRNA_qPCR_F | GCTAGAAATAGCAAGTTAAAATAAGGCTAGTCCG |
| gRNA_qPCR_R | AAAAAGCACCGACTCGGTGCC |
| dCas9_qPCR_F | GTGACCGAGGGAATGAGAAA |
| dCas9_qPCR_R | AGCTGCTTCACGGTCACTTT |
| pAC95_PCR_F | AGAAGAGAAAGGTGGAGGCC |
| pAC95_PCR_R | CGTCACCGCATGTTAGAAGG |
| SID4X_PCR_F | CAATAGAAACTGGGCTTGTCG |
| SID4X_PCR_R | TCGTGCTTCTTATCCTCTTCC |
Tabella 1. Primer usati per estensione della guida della RNA e sequenziamento, qPCR e rilevazione della proteina di fusione.
Discussione
Questo protocollo descrive un metodo semplice e flessibile per la dissezione funzione enhancer del locus genomico endogeno senza alterare fisicamente la sequenza del DNA. Mentre simile nel concetto a protocolli di interferenza CRISPR precedentemente pubblicati utilizzando dCas9-KRAB27, Enhancer-i differisce da questi protocolli in 3 modi principali. In primo luogo, Enhancer-i utilizza il dominio interagente SIN3A di MAD120 per raggiungere disattivazione enhancer. Disattivazione di Enhancer possa essere recuperati utilizzando inibitori HDAC, suggerendo che il meccanismo primario di disattivazione è dipendente di HDAC. A differenza di interferenza CRISPR con dCas9-KRAB, Enhancer-i non porta alla deposizione di H3K9me3. Questo è probabilmente dovuto al fatto che Enhancer-i si basa sull'introduzione transitoria della Guida RNAs, con le cellule essendo raccolte alle 3 giorni post transfezione. In interferenza CRISPR, è osservato un aumento H3K9me3 a 7 giorni post trasduzione12. Infine, il protocollo di Enhancer-i fornisce una strategia per indirizzare più siti contemporaneamente e monitorare l'efficienza di targeting. Mosaico-seq17, dCas9-KRAB è utilizzato per indirizzare rinforzatori multipli simultaneamente, ma questa tecnica si basa sul sequenziamento di RNA di singola cellula per identificare i cambiamenti di espressione, e molti geni (quali geni estrogeno-rispondente) passano inosservati a causa della bassa sensibilità di singole cellule RNA-seq. Enhancer-i fornisce un metodo affidabile per studiare rinforzatori individualmente e in associazione per qualsiasi gene.
La fase più critica di Enhancer-i è transfezione, che dovrebbe essere ottimizzata per la linea cellulare di interesse. Questo protocollo si basa su un trattamento con puromicina arricchire per cellule trasfettate, ma è possibile che guida co-trasfettando RNAs con una proteina fluorescente e l'ordinamento per cellule fluorescenti mediante citometria a flusso può rivelarsi un metodo migliore di arricchimento per alcuni cellulari tipi. Si consiglia di monitorare il livello di espressione della Guida RNAs e SID4x-dCas9-KRAB di qPCR per risolvere i problemi e confermare la transfezione. Se i livelli guida RNA sono bassi (ciclo soglia > 30), gli utenti possono anche considerare alternative guida strategie di produzione di RNA come in vitro trascrizione28. È anche possibile che, malgrado i livelli elevati gRNA, guida targeting RNA della proteina SID4x-dCas9-KRAB è inefficiente, nel qual caso selezionando diversi guida sequenze di RNA può essere necessario. Eseguendo ChIP-seq la proteina di fusione con cromatina dalle cellule Enhancer-i trattati, l'efficienza di targeting può essere monitorato. Se c'è segnale alto di SID4x-dCas9-KRAB presso la regione di interesse e viene rilevata alcuna modifica di espressione nel suo gene bersaglio putativo, quindi la regione probabile non contribuisce all'espressione di tale gene sotto le condizioni studiato.
Una potenziale limitazione di Enhancer-i è che gli effetti fuori bersaglio possono accumularsi se troppi siti sono destinate allo stesso tempo. Tuttavia, CRISPR interferenza strategie per atterramento hanno meno effetti di destinazione oltre RNAi29, particolarmente quando viene utilizzata una linea di policlonale delle cellule che esprimono dCas9-KRAB. Mentre abbiamo visto fuori bersaglio genomic associazione di SID4X-dCas9-KRAB quando la destinazione 10 siti contemporaneamente, non abbiamo individuato i cambiamenti di espressione genica a seguito di quegli eventi di associazione. Come alcuni rinforzatori possono contattare promotori multipli e/o altri esaltatori, è possibile che molti geni possono cambiare espressione al momento di targeting di un enhancer singolo, anche se non è chiaro se questa forma di regolazione genica è comune. Per confermare che i cambiamenti di espressione osservati sono dovuti a un rinforzatore specifico di targeting e non gli effetti fuori bersaglio, gli utenti possono eseguire Enhancer-i con due insiemi distinti di guida non sovrapposte RNAs targeting della stessa regione. Inoltre, la delezione genetica della regione utilizzando Cas9 nucleasi-competente può confermare ulteriormente suoi effetti sull'espressione genica.
Come funzioni Enhancer-i attraverso deacetilazione, è possibile che le sue abilità di disattivazione sono limitate ai rinforzatori che hanno livelli apprezzabili di acetilazione dell'istone. Ci sono una varietà di fusioni repressive alternative che può essere più efficace alle specifiche esaltatori di targeting. Fusioni di methyltransferase del DNA di dCas9 possono essere utilizzate per ridurre l'espressione genica quando mirati a rinforzatori distale30, ma questa repressione spesso non è temporanea. Un'altra fusione repressivo utilizza il dominio di amico di GATA1 (FOG1), che porta a istone H3 lisina 27 trimethylation e reprime l'espressione genica a livelli simili a dCas9-KRAB attraverso una varietà di cellula linee e promotori31. È interessante notare che, aggiunta di ulteriori copie di FOG1 a dCas9 ridotto il potenziale repressivo a promotori, suggerendo che una singola copia del dominio SID può fornire ulteriori disattivazione potenziatore delle 4 copie attualmente utilizzato in Enhancer-i. È possibile che alcuni loci possono trarre vantaggio dal targeting dual da diverse combinazioni delle fusioni dCas9 sopra. Ad esempio, stabile a lungo termine repressione può avvenire attraverso la trasduzione simultanea di dCas9-DNMT3a e dCas9-KRAB32. La maggior parte di queste fusioni repressive sono stata indirizzata solo a un singolo locus in un momento, e rimane poco chiaro che è più efficace nel manipolare rinforzatori multipli contemporaneamente.
Enhancer-i, mentre un metodo adatto per lo studio di combinazioni di rinforzatori per una manciata di geni, è ancora un po ' limitato nella velocità effettiva se l'utente desidera studiare putativi rinforzatori per centinaia di geni. Future applicazioni di questa tecnica di integrare tecnologie basate su formazione immagine per quantificare i geni multipli in campioni multipli contemporaneamente. D'importanza, queste tecnologie sono compatibili con rilevamento diretto di molecole di RNA da lisato, eliminando la necessità di isolamento del RNA che richiede tempo. Questi adattamenti faciliterà l'interrogatorio del set più grandi dei rinforzatori.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Questo lavoro è stato supportato da NIH/NHGRI R00 HG006922 e NIH/NHGRI R01 HG008974 a J.G. e il Huntsman Cancer Institute. J.B.C. è stato sostenuto dal programma di formazione di NIH in genetica T32GM007464.
Materiali
| Name | Company | Catalog Number | Comments |
| ZR 96-well Quick-RNA Kit | Zymo Research | R1053 | |
| Power SYBR Green RNA-to-CT 1-Step | Applied Biosystems | 4389986 | |
| AflII restriction enzyme | NEB | R0520S | |
| Phusion High-Fidelity PCR Master Mix with HF Buffer | NEB | M0531L | |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | |
| FuGENE HD | Promega | E2312 | |
| DNA Clean & Concentrator Kit | Zymo Research | D4013 | |
| Buffer RLT Plus | Qiagen | 1053393 | |
| b-estradiol | Sigma-Aldrich | E2758 | |
| Human: Ishikawa cells | ECACC | 99040201 | |
| H3K27ac rabbit polyclonal | Active Motif | 39133 | |
| H3K9me3 rabbit polyclonal | Abcam | ab8898 | |
| FLAG mouse monoclonal | Sigma-Aldrich | F1804 | |
| ER alpha rabbit polyclonal | Santa Cruz | sc-544 | |
| pGL3-U6-PGK-Puro plasmid | Addgene | 51133 | Shen et al., 2014 |
| gRNA_cloningVector plasmid | Addgene | 41824 | Mali et al., 2013 |
| AflII U6 puromycin plasmid | Addgene | 106404 | Carleton et al., 2017 |
| SID4X-dCas9-KRAB plasmid | Addgene | 106399 | Carleton et al., 2017 |
| 2-Mercaptoethanol | Sigma-Aldrich | M6250-10ML | |
| UltraPure DNase/RNase-Free Distilled Water | ThermoFisher Scientific | 10977-023 | |
| Opti-MEM I Reduced Serum Medium | Gibco | 31985070 | |
| KAPA Stranded mRNA-Seq Kit, with KAPA mRNA Capture Beads | Kapa Biosytems | KK8420 | |
| Pierce Protease and Phosphatase Inhibitor Mini Tablets | ThermoFisher Scientific | A32959 | |
| Formaldehyde solution | Sigma-Aldrich | 252549-25ML | |
| Geneticin Selective Antibiotic (G418 Sulfate) (50 mg/mL) | ThermoFisher Scientific | 10131035 | |
| LB Broth | ThermoFisher Scientific | 10855001 | |
| Quick-DNA Miniprep Kit | Zymo Research | D3020 | |
| Quick-Load Purple 2-Log DNA Ladder | NEB | N0050S |
Riferimenti
- Consortium, E. P. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Roadmap Epigenomics, C., et al. Integrative analysis of 111 reference human epigenomes. Nature. 518 (7539), 317-330 (2015).
- Andersson, R., et al. An atlas of active enhancers across human cell types and tissues. Nature. 507 (7493), 455-461 (2014).
- Perry, M. W., Boettiger, A. N., Bothma, J. P., Levine, M. Shadow enhancers foster robustness of Drosophila gastrulation. Curr Biol. 20 (17), 1562-1567 (2010).
- Lam, D. D., et al. Partially redundant enhancers cooperatively maintain Mammalian pomc expression above a critical functional threshold. PLoS Genet. 11 (2), e1004935 (2015).
- Patwardhan, R. P., et al. Massively parallel functional dissection of mammalian enhancers in vivo. Nat Biotechnol. 30 (3), 265-270 (2012).
- Savic, D., et al. Promoter-distal RNA polymerase II binding discriminates active from inactive CCAAT/ enhancer-binding protein beta binding sites. Genome Res. 25 (12), 1791-1800 (2015).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Cheng, A. W., et al. Multiplexed activation of endogenous genes by CRISPR-on, an RNA-guided transcriptional activator system. Cell Res. 23 (10), 1163-1171 (2013).
- Hilton, I. B., et al. Epigenome editing by a CRISPR-Cas9-based acetyltransferase activates genes from promoters and enhancers. Nat Biotechnol. , (2015).
- Kearns, N. A., et al. Functional annotation of native enhancers with a Cas9-histone demethylase fusion. Nat Methods. 12 (5), 401-403 (2015).
- Thakore, P. I., et al. Highly specific epigenome editing by CRISPR-Cas9 repressors for silencing of distal regulatory elements. Nat Methods. 12 (12), 1143-1149 (2015).
- Groner, A. C., et al. KRAB-zinc finger proteins and KAP1 can mediate long-range transcriptional repression through heterochromatin spreading. PLoS Genet. 6 (3), e1000869 (2010).
- Qi, L. S., et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell. 152 (5), 1173-1183 (2013).
- Fulco, C. P., et al. Systematic mapping of functional enhancer-promoter connections with CRISPR interference. Science. 354 (6313), 769-773 (2016).
- Joo, J. Y., Schaukowitch, K., Farbiak, L., Kilaru, G., Kim, T. K. Stimulus-specific combinatorial functionality of neuronal c-fos enhancers. Nat Neurosci. 19 (1), 75-83 (2016).
- Xie, S., Duan, J., Li, B., Zhou, P., Hon, G. C. Multiplexed Engineering and Analysis of Combinatorial Enhancer Activity in Single Cells. Mol Cell. 66 (2), 285-299 (2017).
- Carleton, J. B., Berrett, K. C., Gertz, J. Multiplex Enhancer Interference Reveals Collaborative Control of Gene Regulation by Estrogen Receptor alpha-Bound Enhancers. Cell Syst. 5 (4), 333-344 (2017).
- Gilbert, L. A., et al. CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell. 154 (2), 442-451 (2013).
- Ayer, D. E., Laherty, C. D., Lawrence, Q. A., Armstrong, A. P., Eisenman, R. N. Mad proteins contain a dominant transcription repression domain. Mol Cell Biol. 16 (10), 5772-5781 (1996).
- Alland, L., et al. Role for N-CoR and histone deacetylase in Sin3-mediated transcriptional repression. Nature. 387 (6628), 49-55 (1997).
- Konermann, S., et al. Optical control of mammalian endogenous transcription and epigenetic states. Nature. 500 (7463), 472-476 (2013).
- Rennoll, S. A., Scott, S. A., Yochum, G. S. Targeted repression of AXIN2 and MYC gene expression using designer TALEs. Biochem Biophys Res Commun. 446 (4), 1120-1125 (2014).
- Stampfel, G., et al. Transcriptional regulators form diverse groups with context-dependent regulatory functions. Nature. 528 (7580), 147-151 (2015).
- Perez-Pinera, P., et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors. Nat Methods. 10 (10), 973-976 (2013).
- Heigwer, F., Kerr, G., Boutros, M. E-CRISP: fast CRISPR target site identification. Nat Methods. 11 (2), 122-123 (2014).
- Parsi, K. M., Hennessy, E., Kearns, N., Maehr, R. Using an Inducible CRISPR-dCas9-KRAB Effector System to Dissect Transcriptional Regulation in Human Embryonic Stem Cells. Methods Mol Biol. , 221-233 (2017).
- Romanienko, P. J., et al. A Vector with a Single Promoter for In Vitro Transcription and Mammalian Cell Expression of CRISPR gRNAs. PLoS One. 11 (2), e0148362 (2016).
- Smith, I., et al. Evaluation of RNAi and CRISPR technologies by large-scale gene expression profiling in the Connectivity Map. PLoS Biol. 15 (11), e2003213 (2017).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247 (2016).
- O'Geen, H., et al. dCas9-based epigenome editing suggests acquisition of histone methylation is not sufficient for target gene repression. Nucleic Acids Res. 45 (17), 9901-9916 (2017).
- Amabile, A., et al. Inheritable Silencing of Endogenous Genes by Hit-and-Run Targeted Epigenetic Editing. Cell. 167 (1), 219-232 (2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon