Method Article
Un sistema di coltura timica tridimensionale per generare emigrati timici derivati da murine
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Questo articolo descrive un nuovo metodo per generare emigranti timici (iTE) derivati da cellule staminali specifiche dell'antigene tumorale del tumore da un sistema di coltura timica tridimensionale (3D). ITE sono un sottoinsieme omogeneo di cellule T strettamente correlate alle cellule T ingenue con la capacità di proliferazione, formazione della memoria, e la soppressione del tumore.
Abstract
L'eredità dei recettori delle cellule T pre-riorganizzati (TCR) e il loro ringiovanimento epigenetico rendono le cellule T derivate da cellule staminali pluripotenti indotte (iPSC) una fonte promettente per la terapia adottiva delle cellule T (ACT). Tuttavia, i metodi classici in vitro per la produzione di cellule T rigenerate da iPSC generano cellule T innate o differenziate terminalmente, che sono fenotipicamente e funzionalmente distinte dalle cellule T ingenue. Recentemente, è stato sviluppato un nuovo sistema di coltura timica tridimensionale (3D), che include la capacità di proliferazione, la formazione della memoria, le cellule T specifiche dell'antigene, con un fenotipo funzionale ingenuo simile a cellule T, compresa la capacità di proliferazione, la formazione della memoria , e la soppressione del tumore in vivo. Questo protocollo evita i destini di sviluppo aberranti, consentendo la generazione di cellule T di identità clinicamente rilevanti, designate come emigranti timici derivati da iPSC (iTE), fornendo anche un potente strumento per chiarire le successive funzioni necessarie per la maturazione delle cellule T dopo la selezione timica.
Introduzione
La terapia con cellule T adottive (ACT) può essere un trattamento efficace per alcuni pazienti con cancro avanzato. Sfortunatamente, molti pazienti non sperimentano la regressione tumorale e le cellule trasferite non riescono a persistere dopo l'infusione. Ciò può essere dovuto alla qualità delle cellule T infuse. Un modello murino ACT ha mostrato che rispetto alle cellule T della memoria centrale ingenue o meno differenziate, le cellule efumani differenziate terminalmente sono meno potenti a causa della scarsa persistenza in vivo1, un'osservazione supportata anche dai dati clinici2, 3.
Nel tentativo di migliorare l'efficacia dell'attuale ACT, le cellule staminali pluripotenti indotte derivate dalle cellule T (T-iPSC) sono state studiate ampiamente4,5. Quando le cellule T vengono riprogrammate in T-iPSC e ridifferenziate in cellule T, la configurazione riorganizzata dei geni TCR viene ereditata da T-iPSC e successivamente dalle cellule T ridifferenziate. Pertanto, la capacità di T-iPSC di sottoporsi a un'espansione illimitata in vitro consente la riproduzione efficiente di cellule T immature che trasportano i recettori delle cellule T specifiche del neoantigeno (TCR) quando tali cellule sono progettate da cellule T specifiche dell'antigene tumorale6 ,7. Tuttavia, il metodo preciso per la differenziazione di T-iPSC in cellule T mature, che consentirebbe la produzione di cellule T specifiche dell'antigene del cancro con un fenotipo meno differenziato e una migliore potenza anti-tumorale, rimane da chiarire.
La differenziazione T-iPSC che impiega la co-coltura delle cellule stromali murine OP9 che esprimono sovraesprimenti le DLL del ligando umano NotchDLL1 è un metodo consolidato per produrre cellule T in vitro6,7. Nei topi e negli esseri umani, questo sistema di co-cultura può differenziare costantemente iPSC, ricapitolando così gli eventi di sviluppo dallo stadio di blastocisti fino all'immaturo lignaggio delle cellule Tfase 6,7. Nonostante questi progressi biotecnologici, la differenziazione fisiologica dopo lo stadio CD4-CD8- doppio positivo (DP) è ancora difficile da raggiungere. Uno dei motivi è che nel timo vengono generate cellule T in vivo CD4-CD8- cd8- singolo positivo (SP), un organo responsabile della maturazione e della selezione delle cellule T che hanno specificità antigene estranea ma non auto-reattività8. Questi processi selettivi sono definiti rispettivamente come selezione positiva e negativa. Tuttavia, la maggior parte dei meccanismi molecolari necessari per far maturare le cellule T nel timo non sono ancora pienamente compresi, rendendo difficile ricostruire questo processo in vitro. Nel tentativo di superare questo ostacolo fisiologico, diversi gruppi hanno stimolato il complesso TCR usando anticorpi anti-CD3 o peptidi agonisti. Queste tecniche in vitro generano prodotti cellulari che esprimono i marcatori cellulari chiave t, come CD3, CD8, TCR e CD62L, pur mantenendo la specificità dell'antigene tumorale. Sfortunatamente, le cellule T generate da questi metodi extratimici costituiscono un'ampia popolazione eterogina di cellule caratterizzate da una selezione positiva incompleta, caratteristiche innate, uccisione non specifica del TCR, incapacità per la formazione della memoria e effetti antitumorali non persistenti in vivo8,9,10,11. Queste anomalie hanno sollevato preoccupazioni sul fatto che tali cellule potrebbero innescare una varietà di effetti collaterali, tra cui linfoma e anomalie cutanee e ossee, se utilizzato per applicazioni terapeutiche12,13,14 .
Per ricreare i segnali fisiologici mancanti negli attuali sistemi di differenziazione in vitro, l'iPSC specifico dell'antigene tumorale è stato differenziato utilizzando un timo raccolto. Il sistema classico di coltura dell'organo fetale (FTOC), progettato per studiare lo sviluppo intra-timico delle cellule T, è stato migliorato utilizzando un sistema di coltura 3D che ha prodotto con successo cellule T che hanno completato l'educazione timica. Queste cellule T post-timiche, designate come emigranti timici derivati da iPSC (iTE), mostravano proprietà ingenue15. iTE ha mostrato la proliferazione, formazione della memoria, e adeguati effetti anti-tumore in un modello murino contro stabilito B16 tumori del melanoma. In questo articolo viene descritto in dettaglio il protocollo di questo nuovo sistema FTOC utilizzando un sistema di colture 3D (Figura 1).
Protocollo
Tutti gli esperimenti sugli animali sono stati approvati dai Comitati istituzionali per la cura e l'uso degli animali del National Cancer Institute (NCI) ed eseguiti in conformità con le linee guida NIH.
1. Preparazione delle celle OP9/DLL1 per la co-cultura con iPSC
- Cellule OP9/DLL1 di coltura nei supporti OP9 (mezzo essenziale minimo di z [z-MEM] - 20% di siero bovino fetale non caldo inattivato [FBS] - 1x penicillina-streptomycin - acido ascorbico [50 ng/mL] e mono-thioglycerol [100 nM]) a 37 . Quando le cellule OP9/DLL1 raggiungono l'80-95% di confluenza, lavate una volta con 1x magnesio, calcio e fenolo rosso fosfato libero buffered salina (in seguito indicato come PBS).
- Aggiungere 4 mL di 0,05% di prova e incubare per 5 min a 37 gradi centigradi. Quindi aggiungere 4 mL di supporto OP9, dissociare lo strato di cella pipettando per fare una sospensione a cella singola.
- Trasferire la sospensione cellulare in un tubo conico da 50 mL attraverso un colino cellulare da 100 m. Centrifuga a 300 x g per 5 min a 4 gradi centigradi, aspira il super-natante e risolvono di 12 mL di supporti OP9.
- Piastra 2 mL di sospensione delle celle OP9/DLL1 su una nuova parabola Petri a coltura cellulare di 10 cm e aggiungere ulteriori 8 mL di supporti OP9. Ripetere il passaggio ogni 2-3 giorni.
NOT: La qualità delle condizioni di applicazione e di impostazioni cultura di FBS è fondamentale per mantenere l'espansione delle celle OP9/DLL1 senza perdere la capacità di supportare la differenziazione iPSC. Pertanto, si raccomanda di prevalutare il lotto di FBS e il passaggio in modo coerente all'80% di confluenza per prevenire la differenziazione cellulare e la senescenza. È anche importante fare abbastanza scorte congelate di cellule OP9/DLL1 e scongelare un nuovo stock ogni 4-6 settimane.
2. Differenziazione in vitro di iPSC in cellule T immature
-
Il giorno 0, inizia la co-cultura iPSC su piatti confluenti OP9/DLL1.
- Raccogliere iPSC come una sospensione a singola cellula per tripsinizzazione (5 min in 0.05% trypsin a 37 gradi C), raccogliere le cellule, e centrifugare a 300 x g per 5 min a 4 gradi centigradi.
- Aspirati il supernatante e risospendono nuovamente le cellule a 1,0 x 105 iPSC per 10 mL di supporti OP9. Piastra 1,0 x 105 iPSC su un piatto confluente OP9/DLL1 10 cm.
NOT: I piatti OP9/DLL1 10 cm vengono utilizzati per la differenziazione iPSC quando raggiungono la confluenza del 90-100%. Le differenze di confluenza possono influire sull'efficienza della differenziazione iPSC.
- Il giorno 3, aspirare vecchi supporti e sostituire con 10 mL di nuovi supporti OP9.
-
Il giorno 6, passare le cellule.
- Lavare ogni piatto OP9 confluente di 10 cm con 10 mL di PBS. Aggiungere 3 mL di 0,05% di cipina per piatto e incubare per 3-5 min a temperatura ambiente (RT).
- Aggiungere 4 mL di supporti OP9 e raccogliere le cellule con pipettaggio delicato. Passare le cellule attraverso un colino a 100 m e centrifugare a 300 x g per 5 min a 4 gradi centigradi. Scartare supernatante.
- Resospendi le celle in 10 mL di supporti di differenziazione (supporti OP9 con 5 ng/mL mouse Flt3 ligando [FLT3L] e 5 mouse ng/mL IL-7) e piastre di sospensione delle celle su una nuova tela confluente OP9/DLL1 da 10 cm.
- Il giorno 9, aspirare vecchi supporti e sostituire con 10 mL di supporti di differenziazione freschi.
-
Il giorno 11, quando i cardiomiociti sono osservati nelle colonie iPSC, staccarle meccanicamente le cellule non aderenti pipettando e filtrare attraverso un colino cellulare di 100 m. Girare a 300 x g per 5 min a 4 gradi centigradi.
- Aspirare il supernatante e resuspend in 24 mL di supporti di differenziazione. Piastra iPSC in una piastra confluente OP9/DLL1 6 pozzetti (4 mL/pozzetto).
-
Il giorno 15, raccogliere tutte le cellule non aderenti e filtrare attraverso un colino di 40 m.
- Girare a 300 x g per 5 min a 4 gradi centigradi.
- Continuare a passare passando le celle non aderenti ogni 3-4 giorni ripetendo il passaggio 2.5.1.
3. 3D cultura dell'organo timica per generare iTE
- Raccogliere lobi timici fetali toro e distribuire linfociti endogeni con trattamento deoxyguanosine (dGUO) come descritto in precedenza16.
- Il giorno 7 del trattamento dGUO, prendere quattro nuovi piatti da 10 cm e riempirli ciascuno con 20 mL di supporti completi (Rosswell Park Memorial Institute Media 1640 [RPMI 1640] - 10% FBS - 1x L-alanyl-L-glutamine , 1x piastre di sodio , mezzo minimo essenziale con amminoacidi non essenziali (MEM-NEAA) - 1x penicillina-streptomicina - [1:1000] 2-mercapto etanolo).
- Trasferire tutte le membrane di nitrocellulosa con lobi timici in un piatto di 10 cm. Staccare i singoli lobi dalla membrana con pinze, permettendo loro di essere sommersi dai media. Scartare le membrane. Incubare per 1 h a RT.
- Trasferire i lobi timici in un nuovo piatto di 10 cm con supporti completi e incubare per 1 h a RT. Ripetere questo passaggio altre 2 volte.
- Usando le pinze, fissate i lobi timici al piatto (uno alla volta), e con l'altra mano fate un'incisione profonda 100-200 m al centro e estendendo la metà del diametro del lobo per facilitare la migrazione dei progenitori delle cellule T nel lobo.
- Trasferire i lobi timici in un nuovo piatto di 10 cm riempito con un supporto di differenziazione completo (media completi : 5 mouse ng/mL IL-7 - 5 mouse ng/mL FLT3L - 5 ng/mL SCF).
- Facoltativamente, se si utilizzano piastre di coltura 3D con griglie di livello inferiore e superiore, riempire entrambe le griglie con PBS sterile per evitare l'evaporazione e l'essiccazione delle gocce appese.
- Trasferire 30 - L di supporto completo contenente un lobo timico trattato con dGuo dal passo 3.6 in ogni pozzetto della piastra di coltura 3D.
- Raccogliere celle di lignaggio T non aderenti (cellule T immature derivate da iPSC) dalla cocoltura OP9/DLL1 (giorni 16-21) (passaggio 2.6.2) e risospendere a 2–5 x 103 celle di lignaggio T per mezzo supporto.
- Aggiungere 20 litri di t leccatura a ogni lobo timico nella piastra di coltura 3D. Incubare pernottamento a 37 gradi centigradi con il 5% di CO2.
- Impostare il pisconto P200 a 30 gradi e aspirare il supporto dopo aver derubato più volte da ogni pozzo per rimuovere tutte le cellule che circondano i lobi timici. Eliminare i supporti e aggiungere 30 - L di supporto completo. Ripetere questa procedura 5-7 volte per rimuovere eventuali cellule T extra immature che non migrano nei lobi. Modificare 25-30 - L di media ogni giorno in seguito.
- Confermare la formazione di un alone di emigranti timici derivati da iPSC (iTE) intorno ai lobi a partire dal giorno 4-5 mediante microscopia leggera.
- Raccogli iTE ogni giorno pipetting media senza interruzioni del lobo. Cambia i media ogni giorno e continua la raccolta fino a circa 12 giorni.
- Gli iTE raccolti sono pronti per l'uso per le analisi molecolari (Figura 2, Figura 3, Figura 4e Figura 5) o per esperimenti di trapianto in vivo.
4. Preparazione delle celle di presentazione dell'antigene (APC)
- Sacrificare un topo C57BL/6 per dislocazione cervicale e posizionarlo su un tappetino per l'ammollo di laboratorio come descritto sopra.
- Rimuovere la milza e posizionarla su un colino da 100 m. Comprimere la milza sul colino utilizzando uno stantuffo di siringa da 12 mL per effettuare una sospensione a cella singola.
- Trasferire la sospensione cellulare attraverso un colino sterile da 40 m. Centrifugare la sospensione a 300 x g per 5 min a 4 gradi centigradi per pelletare le cellule.
- Aspirare il supernatante e risospendere il pellet cellulare in 2 mL di ammonio-cloride-potassio (ACK) lysis buffer per escludere i globuli rossi (RBC). Incubare per 5 min a RT.
- Sottocedi il buffer di lisi ACK aggiungendo 10 mL di PBS. Pellet le cellule per centrifugazione a 300 x g per 5 min a 4 gradi centigradi.
- Aspirare il supernatante e risospendere il pellet cellulare in 10 mL di supporti completi e trasferire in un piatto Petri sterile 10 cm.
- Gli splenociti irradiati con 3500 rad utilizzando un dispositivo di irradiazione (radiazione z) per prevenire la proliferazione cellulare.
- Riportare immediatamente le cellule irradiate a un incubatore e a una coltura di 37 gradi centigradi durante la notte.
- Utilizzare celle irradiate come APC o congelare in banchiere cellulare.
5. APC pulsa con antigene
- Contare APC irradiato dal vivo utilizzando un emocitometro Neubauer e trypan tintura blu. Incubare APC con peptidi (hgp100) o nucleoproteine per 30 min a 37 gradi centigradi.
- Lavare aPC due volte con 10 mL di PBS per rimuovere qualsiasi peptide in più.
- Contare iTE e mescolare con APC in un rapporto 1:1 in supporto completo con 100 IU IL-2 e 5 ng/mL IL-7. Aliquota 100 L della miscela di cellule (concentrazione totale: 1 x 106 cellule / mL) in ogni pozzo di un attacco ultra-basso U inferiore 96 bene piastra e coltura per 48 h a 37 .
- Dopo 48 h, trasferire le celle a una nuova piastra utilizzando una pipetta multicanale e passare ogni 2-3 giorni successivi.
-
Il giorno 3, analizzare il profilo di secrezione di citochine macchiando le cellule con anticorpo intracellulare e analizzare per citometria di flusso (Figura 3).
- Aggiungete 0,67 l/mL di inibitore del trasporto proteico (ad esempio, GolgiStop) e incubate a 37 gradi centigradi per 6 h per migliorare l'accumulo intracellulare di citochine. Lavare con 10 mL di PBS.
- Risospendere le cellule in 3 mL di PBS freddo (4 gradi centigradi) e aggiungere lentamente 1 mL di soluzione fredda 4% paraformaldeide (PFA).
- Dopo 10 min, ruotare le cellule a 300 x g per 5 min a 4 gradi centigradi, eliminare supernatali e lavare con 10 mL di PBS.
- Risospendere le celle in PBS da 1 mL : 1% FBS - 0,1% di surfactant nonionico, e mettere in 4 gradi centigradi per 10-15 min.
- Aggiungere anticorpi, proteggere i campioni dalla luce e mettere in 4 gradi centigradi per 30 min.
- Abbassare le cellule a 300 x g per 5 min a 4 gradi centigradi, eliminare supernatali e lavare con 10 mL di PBS.
- Abbassare le celle a 300 x g per 5 min a 4 gradi centigradi e risospendere le cellule in 1 mL di PBS. Le cellule sono pronte per essere analizzate in un citometro di flusso.
Risultati
I timoli fetali co-coltivati sono stati sezionati per analizzare se le cellule di lignaggio T derivate da iPSC possono migrare nei lobi timici. I lobi di controllo non semiati avevano un'architettura tissutale caratterizzata da un'astrocitica web timica17, schierata dacellule endogene CD3. D'altra parte, i lobi timici seminati con cellule T immature derivate da iPSC sono stati ripopolati con cellule mononucleari CD3e che indicavano la migrazione di cellule T immature derivate da iPSC nei lobi (Figura 2A).
Le cellule T che sono migrate e maturate all'interno del microambiente timico sono poi emigrate come iTE. Per testare la loro caratterizzazione fenotipica, è stata eseguita l'analisi citometrica del flusso dei timociti C57BL6, delle cellule T immature derivate da Pmel iPSC (extratimiche) e delle cellule che sono emerse dai lobi timici (iTE). Le cellule T extratimetiche su OP9/DLL1 mostravano le cellule T CD4,CD8, e le cellule T CD8-SP senza espressione del marcatore di selezione positiva MHC-I, mentre iTE aveva una popolazione chiara di fenotipo della cella CD8-SP MHC-I passaggio riuscito attraverso la selezione positiva prima di uscire dai lobi timici. iTE esprimono costantemente MHC-I e CD62L, che sono marcatori associati ad alta competenza proliferale, produzione di citochine, sopravvivenza periferica, e linfoide homing18,19,20. Questo fenotipo è coerente con i timociti M2 SP che sono la popolazione più matura di singole cellule T positive nel timo20, il che suggerisce che iTE sono passati attraverso un normale programma di sviluppo timico (Figura 3). Per monitorare l'efficienza della generazione di iTE, le cellule che erano evolate dai singoli lobi timici sono state isolate. Il giorno 7, i lobi timici generavano in media 1 x 103 CD8SP LIVE CD45.1, CD3, iTE al giorno (Figura 3B). Un tasso simile di produzione iTE si osserva dal giorno 6 al giorno 12 della co-cultura timica 3D.
L'attivazione dipendente dall'antigene e la secrezione delle citochine sono state analizzate per osservare le proprietà funzionali delle cellule T iPSC timiceri. In presenza di un peptide irrilevante (nucleoproteina), Pmel-iTE non ha rilasciato quantità significative di TNF-z, IL-2, o IFN-z. Quando viene stimolato con il peptide cognato per le cellule Pmel T (hgp100), Pmel-iTE ha rilasciato robuste quantità diTNF-z e IL-2, producendo anche basse quantità di IFN-z (Figura 4), indicando che iTE timicicamente istruiti possono riconoscere il loro pepgnato cognato emare citochine eseftuali con un profilo simile a quello degli emigranti timici recenti naturali (RTE).
Per esaminare le differenze trascrizionali tra cellule di lignaggio T derivate da iPSC differenziate su OP9/DLL1 con o senza educazione timica (cioè cellule T extratimiche), l'analisi RNA-seq è stata eseguita su queste due popolazioni e confrontata a quella delle cellule di lignaggio DP T differenziate utilizzando le cellule OP9/DLL1 (DP) e le cellule CD8- Pmel T ingenui primarie. L'espressione di 102 geni che svolgono ruoli cruciali nell'ontogenesi delle cellule T, nell'attivazione dei timociti e nella formazione della memoria sono stati analizzati15,20,21,22. Un'analisi componente principale di queste quattro popolazioni studiate ha dimostrato che le cellule DP e T generate extratinicamente si sono raggruppate, mentre iTE si sono raggruppate più vicino alle cellule T ingenue (Figura 5). Collettivamente, questi dati dimostrano che gli iTE hanno un fenotipo più vicino alle cellule T ingenue rispetto alle cellule di lignaggio T generate da metodi extratimici.
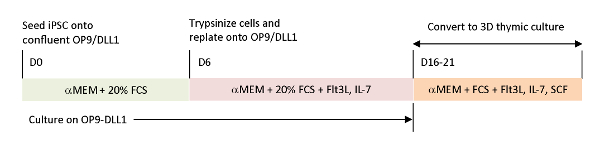
Figura 1 : Panoramica schematica della differenziazione di iPSC a iTE utilizzando OP9/DLL1 e la cultura timica 3D. Il protocollo prevede tre fasi di differenziazione distinte; (Sinistra) dalle cellule iPSC alle cellule di lignaggio ematopoietico su OP9/DLL1 (giorno 0 a 6), (medio) dalle cellule di lignaggio ematopoietico alle cellule T immature su OP9/DLL1 con citochine (giorno 6 a 16–21) e (a destra) dalle cellule T immature (giorno 16–21) alle iTE utilizzando un sistema di coltura timica 3D. Fare clic qui per visualizzare una versione più grande di questa figura.
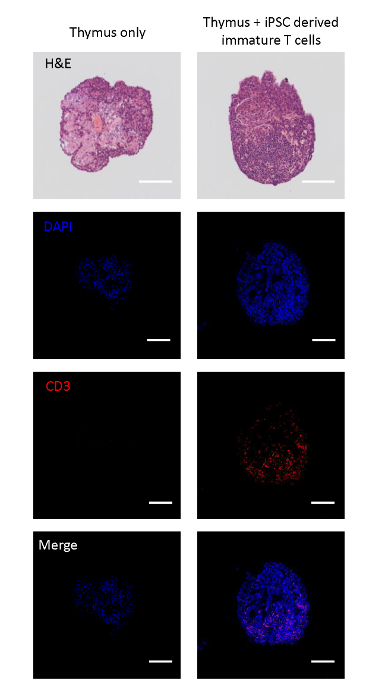
Figura 2 : Immuno-istochimica dei lobi timici seminati con cellule T immature derivate da iPSC. In alto: colorazione H&E di un lobo timico con e senza semina di cellule T immature derivate da iPSC. Dal secondo verso l'alto verso il basso: immagini confocali dei lobi sezionati macchiati con DAPI (nucleo), CD3 (cellula T) e merge. Barre di scala : 100 m. Fare clic qui per visualizzare una versione più grande di questa figura.
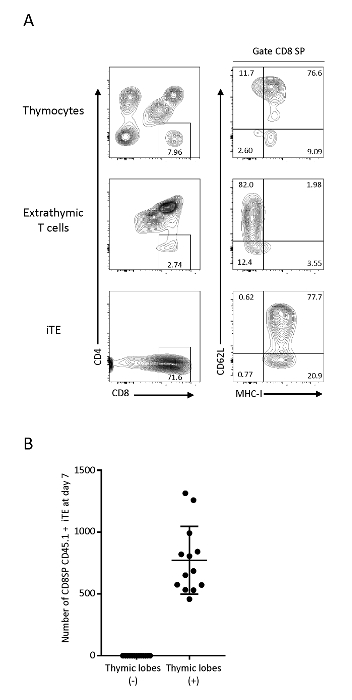
Figura 3 : iTE mostrano un fenotipo delle cellule T post-timiche. (A) Analisi FACS di timociti, cellule T extratimiche (sistema di cocoltura OP9/DLL1) e Pmel-iTE. Le cellule vive sono state gated su CD45 congenico. Le popolazioni CD8 SP sono state ulteriormente analizzate per l'espressione CD62L e MHC-I. (B) Numero medio di CD8SP CD45.1 iTE prodotti durante la notte per lobo 7 giorni dopo la pre-seeding. Sono stati raccolti dati da 12 esperimenti indipendenti. Fare clic qui per visualizzare una versione più grande di questa figura.
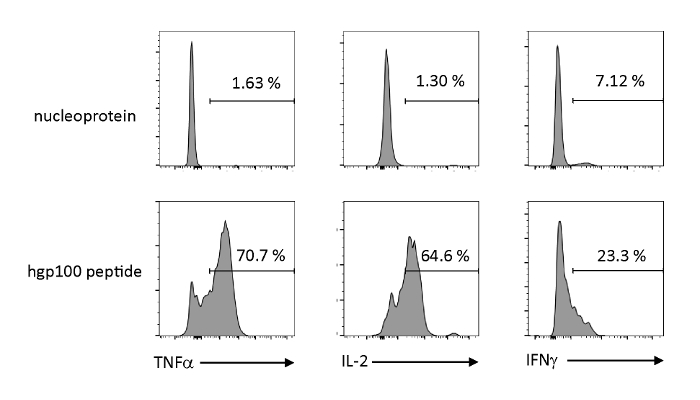
Figura 4 : iTE producono varie citochine mediante stimolazione specifica dell'antigene. Analisi FACS della produzione intracellulare di citochine da parte di iTE. Gli iTE sono stati co-coltivati con APC precaricati con peptide irrilevante (nucleoproteina) o cognato (hgp100) per tre giorni. I numeri visualizzati nei quadranti in alto a destra indicano le percentuali di citochina che produce iTE. Fare clic qui per visualizzare una versione più grande di questa figura.
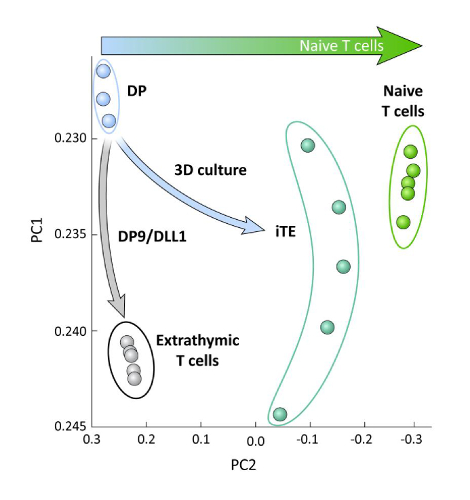
Figura 5 : L'analisi dell'intero trascrittoma rivela uno spostamento nell'espressione genica iTE verso un CD8 ingenuo + Programma a cellule T. Analisi dei componenti principali (PCA) dei dati RNA-seq provenienti da cellule T DP, extratimiche CD8 SP, iTE e ingenue. (Analisi di 102 geni correlati alla differenziazione timica utilizzando il database pubblico GSE105110) 15. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
L'utilizzo di T-iPSC per rigenerare le cellule T specifiche dell'antigene tumorale può superare molti degli attuali ostacoli di ACT generando cellule giovani con una maggiore persistenza. Sebbene siano stati segnalati diversi metodi che utilizzano il sistema di cocoltura OP9/DLL1 per generare cellule CD8 SP6,7,10,13 che esprimono molecole CD8 e TCR specifici dell'antigene tumorale, gene globale i modelli di espressione e l'analisi funzionale mostrano che queste cellule CD8 SP rigenerati in modo extratimicamente sono diverse dalle cellule T ingenue (Figura 4). Qui, descriviamo un sistema di coltura timica 3D in grado di generare emigranti timici derivati da iPSC (iTE) con alta fedeltà e omogeneità da murini T-iPSC. iTE assomigliano cellule T ingenue nel modello di espressione genica globale e nella funzionalità, come la formazione della memoria e l'effetto anti-tumorale in vivo contro il tumore stabilito15.
Il sistema FTOC classico è un modo per ricapitolare la selezione timica in vitro. È stato utilizzato per lo studio dello sviluppo intra-timico di timociti23, e ci sono alcuni rapporti di FTOC utilizzati per generare RTE24. Tuttavia, il sistema FTOC ha diverse limitazioni. Per affrontare la mancanza di ossigeno in una coltura di organi artificiali, diversi gruppi hanno utilizzato una coltura semi-secca basata sulla membrana23,o sistemi di coltura ad alta immormerazione dell'ossigeno25. Tuttavia, nessun metodo attuale può generare costantemente una popolazione omogenea di cellule T post-timiche. Per superare i limiti del sistema FTOC classico, abbiamo progettato un sistema di coltura timica 3D che fornisce miglioramenti tecnici rispetto ai metodi convenzionali15. Ad esempio, utilizzando il nostro metodo di coltura timica 3D, lo scambio massimo di ossigeno e l'assenza di stress meccanico superficie-lobo mantengono i lobi timici in un ambiente più fisiologico. Inoltre, la coltura a lungo termine permette alle cellule T mature di uscire naturalmente dai lobi timici. Infine, l'osservazione in tempo reale e la micro-manipolazione consentono lo scambio di media e una costante raccolta di iTE senza disturbare fisicamente i lobi timici. Così, il metodo di coltura timica 3D fornisce miglioramenti tecnici significativi, nonché un modo per studiare timicamente selezionati cellule T ingenue che non era precedentemente disponibile.
Ci sono diversi punti chiave per la generazione di successo di iTE utilizzando questo sistema di coltura timica 3D. La qualità delle condizioni di applicazione e di impostazioni cultura di FBS è fondamentale per mantenere l'espansione delle celle OP9/DLL1 senza perdere la capacità di supportare la differenziazione iPSC. Pertanto, si consiglia di pre-valutare il lotto FBS e di passare costantemente all'80% di confluenza per prevenire la differenziazione cellulare e la senescenza. Inoltre, è necessaria una coltura CONfluente OP9/DLL1 per la differenziazione in vitro di iPSC in cellule T immature, poiché le differenze di confluenza possono influire sulla loro efficienza. Infine, l'età embrionale dei lobi timici è cruciale per la generazione di iTE. Si consiglia di utilizzare Lobi timiti E14.5 - 15.5.
Come con qualsiasi nuovo protocollo, questo metodo ha limitazioni ed è soggetto a miglioramenti. La tecnica di coltura qui presentata genera circa 1000 iTE per lobo timico al giorno per un periodo di due settimane. L'aumento della generazione di iTE può essere possibile con ulteriori modifiche, tra cui l'ottimizzazione della concentrazione di ossigeno, il volume dei supporti e il tipo di piastra di coltura 3D. L'aggiunta o la rimozione di citochine, così come i cambiamenti nella concentrazione di citochine, possono anche contribuire a una migliore resa iTE.
Dato che il sistema di coltura timica 3D qui presentato può generare emigranti timici in un sistema completamente ex vivo, questa tecnica può essere applicata a una varietà di progetti di ricerca sul trasferimento di cellule immunologiche e adottive, tra cui, ma non limitato a T differenziazione cellulare, maturazione delle cellule T post-timiche e generazione di cellule T specifiche dell'antigene da progenitore ematopoietico o cellule staminali. Sebbene questo metodo non sia direttamente applicabile ai campioni umani, l'iTE e il sistema di coltura timica 3D hanno un grande potenziale per chiarire i meccanismi molecolari di selezione positiva e negativa e possono facilitare la creazione di un sistema di coltura che consenta la generazione di cellule T ingenue specifiche dell'antigene tumorale clinicamente rilevanti per ACT.
Divulgazioni
Gli autori Raul Vizcardo, Nicholas D. Klemen e Nicholas P. Restifo sono inventori della domanda di brevetti internazionale PCT/US2017/65986, depositata il 13 dicembre 2017, dal titolo "Metodi di preparazione di una popolazione isolata o purificata di cellule emigranti timiche e Metodi di trattamento con lo stesso."
Riconoscimenti
Ringraziamo Hiroshi Kawamoto e Kyoko Masuda per aver gentilmente fornito la linea di celle OP9/DLL1. Ringraziamo Alan B. Hoofring ed Erina. Lui per l'assistenza grafica. Questa ricerca è stata sostenuta dal Programma di Ricerca Intramurale del National Cancer Institute degli Stati Uniti e dal programma Cancer Moonshot per il Center for Cell-based Therapy presso il NCI, NIH. Il lavoro è stato sostenuto anche dalla Milstein Family Foundation.
Materiali
| Name | Company | Catalog Number | Comments |
| Chemicals, Peptides and Recombinant Proteins | |||
| 2-deoxyguanosine | Sigma-Aldrich | 312693-72-4 | |
| 2-Mercaptoethanol (1,000x) | Thermo Fisher Scientific | 21985-023 | |
| ACK Lysing Buffer | Gibco | A1049201 | |
| Ascorbic acid | Sigma-Aldrich | A8960 | |
| Blasticidin | Thermo Fisher Scientific | R21001 | |
| FBS | Gemini | 100-500 | |
| Flt-3 ligand | R&D Systems | 427-FL | |
| GlutaMAX (100x) | Thermo Fisher Scientific | 35050-061 | |
| hgp100 | Genscript | 282077-1, KVPRNQDWL | |
| Interleukin-2 | R&D Systems | 402-ML | |
| Interleukin-7 | R&D Systems | 407-ML | |
| MEM Non-Essential Amino Acids Solution | Gibco | 11140050 | |
| MEM powder | Gibco | 61100061 | |
| Monothioglycerol | Sigma-Aldrich | M-6145 | |
| Nucleoprotein | Global Peptides | ASNENMETM | |
| Penicillin/streptomycin | Thermo Fisher Scientific | 15140-122 | |
| Phosphate buffered saline pH 7.4 (1x) | Thermo Fisher Scientific | 10010-023 | |
| Puromycin | Thermo Fisher Scientific | A1113803 | |
| RPMI 1640 | Gibco | 11875093 | |
| Sodium Pyruvate | Thermo Fisher Scientific | 11360-070 | |
| Stem Cell Factor (SCF) | R&D Systems | 455-MC | |
| Stemfactor LIF, Mouse Recombinant | STEMGENT | 03-0011-100 | |
| Trypsin-EDTA (0.05%), phenol red | Thermo Fisher Scientific | 25300-062 | |
| Cell Culture Vessels and others | |||
| 10 cm dish | Corning, Inc. | 353003 | |
| 12 mL Syringe | Covidien Monoject | 22-652-090 | |
| 6 well plate | Corning/Coster | 3516 | |
| Cell strainer 100 μm | Fisher Scientific | 22-363-549 | |
| Cell strainer 40 μm | Fisher Scientific | 22-363-547 | |
| Forceps | DUMONT | 0108-5PO | |
| Lab soaker mat | Versi-Dry | Cat. EF2175CX 74018-00 | |
| Membrane filters ( 0.8 μm, 47diam) | Whatman | WHA7408004 ALDRICH | |
| Perfecta3D Hanging Drop Plate | Sigma-Aldrich | HDP1096 | |
| U Bottom 96 well plate | Corning/Coster | 3799 | |
| Experimental Cell lines | |||
| CD3-iPSC | Vizcardo et al., Cell Report 2018 | N/A | |
| MEF-iPSC | Vizcardo et al., Cell Report 2018 | N/A | |
| Mouse Embryonic Fibroblasts (MEF) | ATCC | SCRC-1040; RRID:MGI:5007926 | |
| OP9/N-DLL1 | Riken Bioresource center | Cat# RCB2927; RRID:CVCL_B220 | |
| Pmel-iPSC | Vizcardo et al., Cell Report 2018 | N/A | |
| Experimental mouse models | |||
| B6.SJL-PtprcaPepcb/BoyCrCrl | Charles River | Strain Code 564; RRID:IMSR_CRL:564 | |
| C57BL/6N | NCI/Charles River | N/A | |
| Pmel-1 mice | Overwijk et al. | J Exp Med 198(4):569-80 | |
| Antibodies | |||
| Anti-aTCR | Biolegend | 109202; RRID:AB_313425 | |
| Anti-CD3 | abcam | ab11089; RRID:AB_369097 | |
| Anti-CD4 | BD Biosciences | 553730; RRID:AB_395014 | |
| Anti-CD44 | BD Biosciences | 559250; RRID:AB_398661 | |
| Anti-CD45.1 | BD Biosciences | 553775; RRID:AB_395043 | |
| Anti-CD45.2 | BD Biosciences | 553772; RRID:AB_395041 | |
| Anti-CD62L | BD Biosciences | 560516; RRID:AB_1645257 | |
| Anti-CD69 | BD Biosciences | 552879; RRID:AB_394508 | |
| Anti-CD8a | BD Biosciences | 557959; RRID:AB_396959 | |
| Anti-CD8b | BD Biosciences | 550798; RRID:AB_393887 | |
| Anti-H-2Kb | BD Biosciences | 553570; RRID:AB_394928 | |
| Anti-IFN-g | BD Biosciences | 557998; RRID:AB_396979 | |
| Anti-IL-2 | BD Biosciences | 554428; RRID:AB_395386 | |
| Anti-TCRb | Thermo Fisher Scientific | 35-5961-81; RRID:AB_469741 | |
| Anti-TCRVb13 | BD Biosciences | 553204; RRID:AB_394706 | |
| Anti-TNFa | BD Biosciences | 557644; RRID:AB_396761 |
Riferimenti
- Gattinoni, L., et al. Wnt signaling arrests effector T cell differentiation and generates CD8+ memory stem cells. Nature Medicine. 15 (7), 808-813 (2009).
- Rosenberg, S. A., et al. Durable complete responses in heavily pretreated patients with metastatic melanoma using T-cell transfer immunotherapy. Clinical Cancer Research. 17 (13), 4550-4557 (2011).
- Singh, N., Perazzelli, J., Grupp, S. A., Barrett, D. M. Early memory phenotypes drive T cell proliferation in patients with pediatric malignancies. Science Translational Medicine. 8 (320), 320-323 (2016).
- Crompton, J. G., Clever, D., Vizcardo, R., Rao, M., Restifo, N. P. Reprogramming antitumor immunity. Trends of Immunology. 35 (4), 178-185 (2014).
- Takahashi, K., Yamanaka, S. Induction of pluripotent stem cells from mouse embryonic and adult fibroblast cultures by defined factors. Cell. 126 (4), 663-676 (2006).
- Vizcardo, R., et al. Regeneration of human tumor antigen-specific T cells from iPSCs derived from mature CD8(+) T cells. Cell Stem Cell. 12 (1), 31-36 (2013).
- Nishimura, T., et al. Generation of rejuvenated antigen-specific T cells by reprogramming to pluripotency and redifferentiation. Cell Stem Cell. 12 (1), 114-126 (2013).
- Takada, K., Kondo, K., Takahama, Y. Generation of Peptides That Promote Positive Selection in the Thymus. Journal of Immunology. 198 (6), 2215-2222 (2017).
- Yamagata, T., Mathis, D., Benoist, C. Self-reactivity in thymic double-positive cells commits cells to a CD8 alpha alpha lineage with characteristics of innate immune cells. Nature Immunology. 5 (6), 597-605 (2004).
- Themeli, M., et al. Generation of tumor-targeted human T lymphocytes from induced pluripotent stem cells for cancer therapy. Nature Biotechnology. 31 (10), 928-933 (2013).
- Serwold, T., Hochedlinger, K., Inlay, M. A., Jaenisch, R., Weissman, I. L. Early TCR expression and aberrant T cell development in mice with endogenous prerearranged T cell receptor genes. Journal of Immunology. 179 (2), 928-938 (2007).
- Lei, F., et al. In vivo programming of tumor antigen-specific T lymphocytes from pluripotent stem cells to promote cancer immunosurveillance. Cancer Research. 71 (14), 4742-4747 (2011).
- Maeda, T., et al. Regeneration of CD8alphabeta T Cells from T-cell-Derived iPSC Imparts Potent Tumor Antigen-Specific Cytotoxicity. Cancer Research. 76 (23), 6839-6850 (2016).
- Serwold, T., et al. T-cell receptor-driven lymphomagenesis in mice derived from a reprogrammed T cell. Proceedings of the National Academy of Sciences of the United States of America. 107 (44), 18939-18943 (2010).
- Vizcardo, R., et al. Generation of Tumor Antigen-Specific iPSC-Derived Thymic Emigrants Using a 3D Thymic Culture System. Cell Reports. 22 (12), 3175-3190 (2018).
- Jenkinson, W., Jenkinson, E., Anderson, G. Preparation of 2-dGuo-treated thymus organ cultures. Journal of Visualized Experiments. (18), (2008).
- Hamazaki, Y., Sekai, M., Minato, N. Medullary thymic epithelial stem cells: role in thymic epithelial cell maintenance and thymic involution. Immunological Reviews. 271 (1), 38-55 (2016).
- Vivier, E., et al. Innate or adaptive immunity? The example of natural killer cells. Science. 331 (6013), 44-49 (2011).
- Rosen, S. D. Ligands for L-selectin: homing, inflammation, and beyond. Annual Review of Immunology. 22, 129-156 (2004).
- Hogquist, K. A., Xing, Y., Hsu, F. C., Shapiro, V. S. T Cell Adolescence: Maturation Events Beyond Positive Selection. Journal of Immunology. 195 (4), 1351-1357 (2015).
- Best, J. A., et al. Transcriptional insights into the CD8(+) T cell response to infection and memory T cell formation. Nature Immunology. 14 (4), 404-412 (2013).
- Schmitz, I., Clayton, L. K., Reinherz, E. L. Gene expression analysis of thymocyte selection in vivo. International Immunology. 15 (10), 1237-1248 (2003).
- Nitta, T., Ohigashi, I., Takahama, Y. The development of T lymphocytes in fetal thymus organ culture. Methods in Molecular Biology. 946, 85-102 (2013).
- Ueno, T., et al. Role for CCR7 ligands in the emigration of newly generated T lymphocytes from the neonatal thymus. Immunity. 16 (2), 205-218 (2002).
- Watanabe, Y., Katsura, Y. Development of T cell receptor alpha beta-bearing T cells in the submersion organ culture of murine fetal thymus at high oxygen concentration. European Journal of Immunology. 23 (1), 200-205 (1993).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon